Method Article
Rápido Neuronal diferenciação de células estaminais pluripotentes induzidas para medir a atividade de rede na Arrays Micro-eletrodo
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Nós modificar e implementar um protocolo publicado anteriormente descrevendo a diferenciação rápida, reprodutível e eficiente dos humanos pluripotentes induzidas (células-tronco hiPSCs) em neurônios corticais excitatórios 12. Especificamente, nossa modificação permite o controle da densidade de células neuronais e uso em matrizes de micro-eléctrodos para medir as propriedades eletrofisiológicas no nível da rede.
Resumo
Neurônios derivados de células-tronco pluripotentes induzidas pelo homem (hiPSCs) fornecer uma nova ferramenta promissora para o estudo de distúrbios neurológicos. Na última década, muitos protocolos de diferenciação em neurónios hiPSCs têm sido desenvolvidos. No entanto, estes protocolos são muitas vezes lento, com alta variabilidade, baixa reprodutibilidade e baixa eficiência. Além disso, os neurônios obtidos com estes protocolos são muitas vezes imaturo e falta de atividade funcional adequada, tanto a nível de uma única célula e de rede, a menos que os neurônios são cultivadas por vários meses. Parcialmente devido a estas limitações, as propriedades funcionais das redes neuronais hiPSC derivados não são ainda bem caracterizada. Aqui, nós adaptar um protocolo recentemente publicado que descreve a produção de neurónios humanos de hiPSCs por expressão forçada do factor de transcrição neurogenin-2 12. Este protocolo é rápida (produzindo neurônios maduros dentro de 3 semanas) e eficiente, com quase 100% de eficiência de conversão de transduced células (> 95% de células são positivas DAPI-MAP2 positivo). Além disso, o protocolo produz uma população homogénea de neurónios excitatórios que permitiriam que a investigação das contribuições específicas do tipo de células para doenças neurológicas. Nós modificamos o protocolo original, gerando células hiPSC estavelmente transduzidas, dando-nos o controle explícito sobre o número total de neurônios. Estas células são então utilizados para gerar redes neuronais hiPSC-derivados de matrizes micro-eléctrodos. Desta forma, a actividade electrofisiológica espontânea de redes neuronais hiPSC-derivados pode ser medido e caracterizado, mantendo consistência interexperimental em termos de densidade celular. O protocolo apresentado é amplamente aplicável, especialmente para estudos sobre os mecanismos e farmacológicos em redes neuronais humanas.
Introdução
O desenvolvimento de humanos induzida pluripotentes células estaminais (hiPSCs) protocolos de diferenciação para gerar neurônios humanos in vitro tem proporcionado uma nova e poderosa ferramenta para o estudo de distúrbios neurológicos. Até recentemente, o estudo desses distúrbios foi gravemente prejudicado pela falta de sistemas modelo usando neurônios humanos. Apesar de roedores podem ser usadas para estudar doenças neurológicas, os resultados de tais estudos não pode ser facilmente traduzidos para os seres humanos 1. Dadas estas limitações, os neurónios hiPSC-derivados são uma alternativa promissora modelo que pode ser utilizado para elucidar os mecanismos moleculares subjacentes e distúrbios neurológicos in vitro para rastreio de fármacos.
Na última década, vários protocolos para converter hiPSCs em neurónios foram desenvolvidos 2-8. No entanto, estes protocolos são ainda limitados de muitas maneiras. Em primeiro lugar, os protocolos são muitas vezes demorado: geração de neurônios com a maturação adequada (ou seja synapformação se) e da atividade funcional requer meses de procedimentos de cultivo, o que torna os estudos de grande escala difíceis 9. Além disso, a eficiência de conversão hiPSC-a-neurónio é baixo. Protocolos frequentemente produzir uma população heterogénea de neurónios, e, portanto, não permitem estudos de subconjuntos específicos de células neuronais. Além disso, os protocolos não são reproduzíveis, produzindo resultados diferentes para diferentes linhas de IPSC 10,11. Por fim, a fase de maturação e propriedades funcionais dos neurónios resultantes são também variáveis 10.
Para resolver esses problemas, Zhang et al. (2013) 12 desenvolvido um protocolo que rapidamente e reprodutivelmente gera neurónios humanos de hiPSCs por sobre-expressam o factor de transcrição neurogenin-2. Como reportado pelos autores, diferenciação ocorre de forma relativamente rápida (apenas 2-3 semanas após a indução da expressão de neurogenin-2), o protocolo é reprodutível (propriedades neuronais são independentes dos starting linha hiPSC), e a conversão hiPSC-a-neurónio é altamente eficiente (cerca de 100%). A população de neurónios gerados com o seu protocolo é homogénea (que se assemelha-camada superior neurónios corticais), que permite a investigação de contribuições específicas do tipo de célula para distúrbios neuronais. Além disso, seus neurônios hiPSC derivados exibiram propriedades maduros (por exemplo, a capacidade de formar sinapses e atividade funcional robusta) depois de apenas 20 d.
Caracterização das propriedades eletrofisiológicas de neurônios hiPSC derivados no nível de rede é um requisito importante antes que a tecnologia hiPSC pode ser explorada para o estudo de doenças humanas. Por esta razão, muitos grupos de pesquisa começaram recentemente a investigar neurônios derivados de células-tronco em nível de rede utilizando rede de micro-eletrodo (MEA) dispositivos (sistemas multicanal, Reutlingen, Alemanha) 13-16. Os eléctrodos de um MEA são embutidos em um substrato sobre o qual as células neuronais podem ser cultivadas.MEAs podem ser utilizados para explorar as propriedades electrofisiológicas de redes neuronais e o desenvolvimento in vitro da sua actividade. Actualmente, os AMA são utilizados apenas em combinação com os protocolos de diferenciação que levar vários meses a produzir redes maduros. Assim, combinando AAM com um protocolo de diferenciação rápida deve facilitar o uso desta tecnologia em larga escala estudos de distúrbios neurológicos.
Aqui, nós apresentamos uma modificação do Zhang et al. (2013) 12 de protocolo e adaptá-lo para uso em AAM. Em particular, em vez de confiar em um transdução lentiviral aguda, nós em vez disso criou linhas hiPSC expressam de forma estável rtTA / NGN2 antes de induzir a diferenciação. Fizemos isso principalmente para ter o controle reprodutível sobre a densidade celular neuronal, uma vez que a densidade celular neuronal é fundamental para a formação da rede neuronal, e por um bom contato entre os neurônios e os eléctrodos do MEA 17,18. although o Zhang et al. protocolo é muito eficiente no que diz respeito à conversão de hiPSCs transduzidas, é inerentemente variável no que diz respeito ao rendimento final de neurónios do número de hiPSCs chapeado inicialmente (ver Figura 2E em Zhang et ai.) 12. Com uma linha estável, eliminamos muitos problemas que causam variabilidade, tais como toxicidade lentiviral e eficiência de infecção. Em seguida, os parâmetros optimizados que produzem de forma fiável redes neuronais hiPSC-derivados de AAM, obtendo-se a maturação da rede (por exemplo, eventos síncronos que envolvem a maioria dos canais), por a terceira semana. Este protocolo rápido e de confiança deve permitir comparações directas entre os neurónios derivados de diferentes linhas hiPSC (isto é, específicos do paciente), bem como fornecer a consistência robusta para estudos farmacológicos.
Protocolo
Todos os experimentos em animais foram realizados de acordo com os cuidados com os animais e utilizar as orientações aprovadas da Comissão de Cuidados com Animais, Radboud University Medical Centre, na Holanda, (RU-DEC-2011-021, protocolo número: 77073).
1. Glia isolamento de células e Cultura
NOTA: O protocolo aqui apresentado é baseado na obra de McCarthy e de Vellis 19 anos, e um protocolo detalhado muito semelhante para os astrócitos do mouse está disponível 20. Para gerar culturas primárias de astrócitos corticais embrionárias a partir de cérebros de rato (E18), uma ratazana grávida tem de ser sacrificados, os embriões devem ser colhidas a partir do útero, e os cérebros precisa de ser isolado a partir dos embriões. Para encher um frasco T75, os córtices de 2 cérebros embrionários precisam ser combinados. Como uma alternativa, os astrócitos e purificados congelados disponíveis comercialmente podem ser comprados.
- Prepara-se o balão de cultura T75
- Dilui-se com poli-D-lisina (PDL) emestéril, água ultrapura até uma concentração final de 10 ug / mL. Adicionar 5 ml da PDL diluída para o frasco de cultura T75. Swish ao redor delicadamente para molhar a superfície de crescimento inteira. Colocar o balão numa atmosfera humidificada a 37 ° C incubadora durante 3 h.
- Aspirar o PDL do frasco. Lavar as 3x frasco com 5 mL de água estéril para remover PDL não ligado. Aspirar a água completamente. Deixar o frasco a secar numa câmara de fluxo laminar ou usadas imediatamente.
- A dissecação dos córtices
- Preparar 50 ml de meio de dissecção: meio de Lebovitz G-15 com 2% (v / v) de B-27 suplemento. Manter em gelo.
- Anestesiar o rato profundamente com isoflurano em uma câmara de indução (caixa de pequenas Plexiglas) até que a respiração cessa (~ 5-8 min). Remover de rato a partir da câmara de indução e imediatamente sacrificados por deslocamento cervical.
- Pulverizar o abdómen do rato com etanol 70% e limpe o excesso. Expor e remover o útero da barragem via seç cesarianasobre o uso de um par de tesouras 21.
- Corte embriões individuais de suas bolsas amnióticas com uma tesoura, a transferência para uma placa de Petri estéril preenchido com meio de dissecção fria e manter em gelo.
- Transferir embriões novamente para uma nova e estéril seis centímetros de Petri com meio cheio dissecção fria. Extrair cérebros de embriões sob um microscópio estéreo. Para expor o cérebro, delicadamente descascar a pele e crânio usando uma pinça. Suavemente retire todo o cérebro e transferir para um 35 milímetros placa de Petri com meio de dissecação limpa e fria.
NOTA: cérebros inteiros foram dissecados a partir de embriões podem ser armazenadas em meio de dissecção em gelo durante muitas horas sem perder viabilidade celular. - Separar os dois hemisférios do cérebro cada cortando a linha média com uma tesoura de mola de ponta fina ou um bisturi. Cuidadosamente retirar as meninges com uma pinça de ponta fina retas.
NOTA: É muito importante para remover as meninges completamente. ºé contaminação por fibroblastos previne a cultura de astrócitos. Os fibroblastos são células que se dividem rapidamente e, eventualmente, irá deslocar as outras células. - Remova o mesencéfalo / estriado e bulbo olfativo com uma tesoura de mola e um bisturi. Também certifique-se de remover o hipocampo (estrutura em forma de C que é perimedian e caudal em relação ao córtex) com uma tesoura de mola e um bisturi. Recolher os hemisférios corticais em um tubo de 15 mL de centrífuga preenchido com 5 meio de dissecção mL. Manter em gelo.
- A dissociação dos córtices
- Preparar 2 mL de Ca 2+ / Mg 2+ livre de Solução Salina Equilibrada de Hank (HBSS) com tripsina a 0,25% (meio de dissociação). Preparar 50 ml de elevada glicose de Eagle Modificado Meio de Dulbecco (DMEM) com 15% (v / v) de Soro Fetal Bovino (FBS) e 1% (v / v) de penicilina / estreptomicina (meio de cultura) e esterilizar filtro.
- Deixe o tecido depositar-se no fundo do tubo de centrifugação. cuidado,pirata tanto do meio de dissecção quanto possível a partir de cima do tecido. Lavar o tecido com 5 mL de Ca 2+ / Mg 2+ livre de HBSS (sem tripsina) e permitir que o tecido para assentar no fundo do tubo.
- Aspirar cuidadosamente a HBSS. Adicionar 2 mL de meio de dissociação e apertar o tubo suavemente para misturar a enzima em torno do tecido. Incubar em um banho de água a 37 ° C durante 5-10 min. Flick o tubo algumas vezes durante a incubação para agitar o tecido.
- Imediatamente triturar o tecido usando uma ponteira de 1.000 mL. Definir o volume de pipeta para cerca de 800 mL. Aspirar as peças e ejectar força para o lado do tubo, imediatamente acima da linha de fluido. No entanto, tentar minimizar bolhas ou espuma. Repetir até que o tecido é suficientemente dissociadas, cerca de 15 - 20x. Adiciona-se 8 mL de meio de cultura para inactivar a tripsina. Misture suavemente invertendo o tubo várias vezes.
- Passar a suspensão de células através de um coador de células de 70 uM colocado no topo de uma 50 mG tubo de centrífuga. Lavar o tubo de 15 mL com meio de cultura e o meio filtrar através do filtro de células para recolher o meio no tubo de 50 mL com a suspensão de células. Lavar o filtro de células algumas vezes com meio de cultura. Após lavagem, o volume final deve ser de cerca de 20 - 25 ml.
- Agregar as células a 200 xg durante 10 min. Aspirar cuidadosamente tanto forma possível, sem tocar no sedimento celular. Ressuspender as células em 1 mL de meio de cultura usando uma pipeta de 1000 mL. Adicionar 11 mL de meio de cultura previamente aquecido e misturar suavemente (para evitar bolhas) utilizando uma pipeta de 10 mL.
- Lavar o balão T75 PDL revestido de uma vez com 5 mL de meio de cultura. Aspirar o meio e transferir a suspensão de célula para o balão. Todas as células estão em suspensão são plaqueadas, e considera-se geralmente desnecessário de contá-las, uma vez que os astrócitos não podem ser diferenciados a partir de outras células na suspensão. Colocar o balão em atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2 Fou dois dias.
- Expansão e manutenção dos astrócitos
- Substituir a forma inteira pela primeira vez 2 d após o plaqueamento inicial. Substituição de todo o meio depois a cada 3 d. Sempre Pré-aquecer o meio fresco a 37 ° C antes de adicionar às células.
NOTA: Os astrócitos exigem 7-10 d para atingir aproximadamente 90% de confluência (astrócitos aparecer como uma monocamada tessellated densamente, com microglia e oligodendrócitos deitado em cima e misturados). - Quando os astrócitos atingir cerca de 90% de confluência, agite o frasco para remover as células gliais contaminantes:
- Retirar o balão da incubadora e aperte a tampa (fenólica) ou cubra a porta (filtrado). Para remover a microglia, Agitar o frasco sobre uma plataforma orbital a 180 rpm durante 1 h. Aspirar o meio. Lavar uma vez com 5 mL de pré-aquecido meio de cultura, aspirado e substituir com 12 mL de meio de cultura.
- Removeros oligodendrócitos, devolver o balão para a plataforma orbital e agitar a 250 rpm, 37 ° C durante um mínimo de 7 horas, mas de preferência O / N.
- Aspirar o meio. Lavar uma vez com 5 mL de pré-aquecido meio de cultura, aspirado e substituir com 12 mL de meio de cultura. Devolver o frasco para a incubadora.
- Quando 100% confluentes, dividir os astrócitos, utilizando procedimentos padrão, na proporção de 1: 3 a 1: 2 com ácido etilenodiaminotetra-tripsina a 0,05% (EDTA). Um frasco T75 em 100% de confluência, normalmente rendimento de cerca de 4,0 X 10 6 células no total. Sob este esquema, as culturas geralmente pode ser dividido uma vez por semana.
Nota: Quando o astrócitos atingem a confluência, podem ser recolhidos e utilizados para a diferenciação hiPSC como descrito abaixo no passo protocolo 3.4. Os astrócitos podem ser divididos pelo menos uma vez sem uma perda notável de viabilidade. Eles podem ser mantidos durante até 2 meses em cultura. A partir da experiência, principal embrionária dia 18 de astrócitos de rato progressively se tornar terminalmente diferenciadas e / ou perder a viabilidade após a divisão repetida. Embora seja possível congelar os astrócitos para uso futuro, prefere-se isolar os astrócitos do cérebro embrionárias fresco quando necessário.
- Substituir a forma inteira pela primeira vez 2 d após o plaqueamento inicial. Substituição de todo o meio depois a cada 3 d. Sempre Pré-aquecer o meio fresco a 37 ° C antes de adicionar às células.
2. Geração de rtTA / NGN2 -positivo hiPSCs
NOTA: Os hiPSCs utilizados para nossos experimentos foram gerados in-house por transdução lentiviral de fibroblastos humanos com a reprogramação fatores cMyc, SOX2, OCT4 e KLF4.
NOTA: Para a geração de rtTA / NGN2 hiPSCs -positivas, vectores lentivirais são usados para integrar estavelmente os transgenes no genoma das hiPSCs. O protocolo para a produção do lentivírus foi publicada previamente 22. Os detalhes dos vectores lentivirais de embalagem que são utilizados para produzir as partículas de lentivírus rtTA NGN2 e são fornecidos na Tabela de materiais / equipamentos. O vector de transferência usado para o lentivírus rtTA é pLVX-EF1α- (Tet-On-Avançada) -IRES-G418 (R); ou seja, este vector codifica um transactivador Tet-On avançada sob controlo de um promotor constitutivo EF1α e confere resistência ao antibiótico G418. O vector de transferência usado para o lentivírus NGN2 é pLVX- (TRE-thight) - (RATO) NGN2-PGK-puromicina (R); ou seja, este vector codifica o gene para neurogenin-2 murino sob o controlo de um promotor controlado por Tet e o gene de resistência à puromicina sob o controlo de um promotor constitutivo de PGK. Assim, utilizando estes dois vectores de transferência, uma linha hiPSC pode ser criado para o qual a expressão de neurogenin-2 de murino pode ser induzida por suplementação do meio com doxiciclina. Para a transdução dos hiPSCs, o sobrenadante com as partículas de lentivírus é usado (referido como "suspensão lentivírus 'no restante do texto), isto é, saberHout concentrar as partículas utilizando ultracentrifugação.
- Placa os hiPSCs (dia 1)
NOTA: Os volumes que são mencionados neste protocolo assumem que as hiPSCs são cultivadas numa placa de 6 poços e que as células de um poço são colhidas. Além disso, presume-se que as células são plaqueadas subsequentemente em 12 cavidades de uma placa de 12 poços.- Preparar 10 ml de DMEM frio / F12 com 1% (v / v) de membrana basal da matriz (BMM) para se obter BMM diluído. Adicionar 800 mL de BMM diluída por poço de uma placa de 12 poços. Incuba-se durante pelo menos 1 h em atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2. Antes da utilização, incubar a placa durante 1 h à TA.
- Meio Essencial quente 15 ml 8 (E8) com 1% (v / v) de penicilina / estreptomicina, 9 ml de DMEM / F12 e uma solução de separação celular mL (CDS) para a temperatura ambiente. Suplementar o meio E8 com a proteína quinase Rho-associado inibidor (rock).
- Aspirar o meio gasto das hiPSCs umd adicionar 1 mL de CDS para os hiPSCs. Incubar 3-5 min numa atmosfera humidificada de 37 ° C incubadora com uma atmosfera de 5% de CO 2. Verificar se ao microscópio as células são separar um do outro.
- Adicionar 2 ml de DMEM / F12 no poço, suavemente suspender as células com uma pipeta de 1000 mL e transferência das células para um tubo de 15 mL. Adicionar 7 ml de DMEM / F12 para a suspensão de células. Rodar as células a 200 xg durante 5 min.
- Aspirar o sobrenadante e adicionar 2 mL do meio E8 preparado. Se obter uma suspensão de células em que os hiPSCs são dissociados (não formam aglomerados de células), colocando a ponta de uma pipeta de 1000 mL de encontro ao lado do tubo de 15 mL e ressuspensão das células suavemente. Verificar sob o microscópio se as células são dissociadas.
- Determinar o número de células (células / mL) utilizando uma câmara de hemocitómetro.
NOTA: Uma placa de 6 poços bem a 80 - 90% de confluência, normalmente obtendo-se 3,0 - 4,0 x 10 6 células no total. - Aspiraro BMM diluído a partir das cavidades da placa de 12 poços. Dilui-se as células para se obter uma suspensão de células de 3,0 x 10 4 células / ml. Placa 1 mL da suspensão de células por poço da placa de 12 poços. Colocar a placa de 12 poços O / N numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2.
- Transdução as células iPS com rtTA e NGN2 lentivírus (dia 2)
- 12 mL de meio E8 morna com 1% (v / v), penicilina / estreptomicina até à temperatura ambiente. Suplementar o meio com E8 inibidor de ROCK e polibreno a uma concentração final de 8 ug / mL para a forma E8.
- Descongelar as alíquotas com suspensão lentivírus. Adicionar polibreno a uma concentração final de 8 ug / mL para a suspensão lentivírus. Aspirar o meio gasto e adicionar 1 mL do meio E8 preparado a cada poço.
- Realizar a transdução com diferentes quantidades do rtTA - e suspensões NGN2 -lentivirus. Por exemplo, transduCE O hiPSCs pela adição de 100 ul de ambas as -lentivirus rtTA e suspensão -lentivirus NGN2 a um poço da placa de 12 poços. Para os outros poços, utilizar 200 ul, 300 ul, 400 ul e 500 ul de suspensão de lentivírus, em vez de 100 uL. Os hiPSCs de dois dos poços da placa de 12 poços não deve ser transduzidas; eles vão servir como controles durante a seleção.
NOTA: As transduções são de preferência realizadas em duplicado, de modo que a eficiência de transdução pode ser estimado com maior precisão depois de o início da selecção (ver passo protocolo 2.2.4). A quantidade de suspensão de lentivírus que é necessário para transduzir eficientemente a maior parte dos hiPSCs depende do título da suspensão lentivírus e a linha hiPSC que é usado. Neste estudo, nós usamos geralmente 100 - 500 mL de suspensão de lentivírus para transduzir as hiPSCs. - Colocar a placa de 12 poços numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO2 durante 6 h. Antes do fim do período de incubação de 6 horas, o meio quente 12 mL E8 com 1% (v / v) de penicilina / estreptomicina e 12 ml de Dulbecco fosfato-salino tamponado (DPBS) à TA. Suplementar o meio E8 com inibidor de ROCK.
- Aspirar o meio E8 gasto. Lavar cada poço com 1 mL de DPBS. Adicionar 1 mL do meio E8 preparado a cada poço. Colocar a placa de 12 poços O / N numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2.
- Refrescar o meio E8 (dia 3)
- 12 mL de meio E8 morna com 1% (v / v), penicilina / estreptomicina até à TA. Aspirar o meio consumido a partir dos poços da placa de 12 poços e adicionar 1 mL do meio E8 preparado a cada poço. Colocar a placa de 12 poços O / N numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2.
- Realizar a seleção com puromicina e G418 (dia 4-8)
NOTA: Dependendo da taxa de divisão celular do quadrilA linha SC e a eficiência da transdução de lentivírus, as células podem chegar a 70-80% de confluência, durante o período de selecção, no ponto em que as culturas precisam ser divididas. Uma vez que o momento do desdobramento não pode ser prevista em avanço, não será mencionado no protocolo. No entanto, em vez de refrescar o meio suplementado com E8 as concentrações mencionadas de puromicina e G418, pode-se dividir a cultura hiPSC como uma cultura normais hiPSC (incluindo plaqueamento das células em placas revestidas com vitronectina). A única exceção é que o meio E8 deve ser complementada com as concentrações mencionadas dos antibióticos para continuar a seleção.- 12 mL de meio E8 morna com 1% (v / v), penicilina / estreptomicina até à temperatura ambiente. Adicionar puromicina e G418 para selecção; quantidades diferentes dos antibióticos são adicionados durante o período de selecção (Tabela 1).
- Estimar a eficiência da transdução calculando a percentagem de G418- e puromicinacélulas resistentes ao. Para estimar a percentagem de células resistentes, uma estimativa da percentagem de células mortas (células não resistentes) para as diferentes condições (as culturas transduzidas com as diferentes quantidades de suspensão lentivírus) e para as células nontransduced (as células que servem como um controlo de selecção). Calcula-se a percentagem de células resistentes quanto [100% - (percentagem de células mortas)].
NOTA: Se as transduções com as diferentes quantidades de suspensão lentivírus foram realizadas em duplicado, a eficácia de transdução pode ser estimado com maior precisão. A condição com as células não transduzidas serve como um controlo de selecção; as percentagens de células mortas para as culturas transduzidas com as diferentes quantidades de suspensão deve ser menor do lentivírus. A percentagem estimada de células resistentes é utilizado para escolher as que são susceptíveis hiPSCs positivo para ambos os transgenes. Em geral, nós escolhemos as hiPSCs a partir da condição de transdução foram> 90% da células sobreviver ao período de seleção 5 d. - Aspirar o meio gasto das hiPSCs e adicionar 1 mL do meio E8 preparado para os poços. Colocar a placa de 12 poços O / N numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2.
| A concentração final de G418 | A concentração final de puromicina | |
| dia 4 | 250 ug / mL | 2 ug / mL |
| dia 5 | 250 ug / mL | 2 ug / mL |
| dia 6 | 250 ug / mL | 1 ug / mL |
| dia 7 | 250 ug / mL | 1 ug / mL |
| dia 8 | 250 ug / mL | 1 ug / mL |
Tabela 1: concentrações de antibióticos durante o período de seleção. As concentrações da puromicina e G418 durante o 5 d o período de selecção.
- Pare a seleção e iniciar a cultura regulares (dia 9 e posterior)
- Após o período de selecção 5 d, a cultura das hiPSCs -positivas rtTA / NGN2 como hiPSCs normais, com a excepção de que a forma E8 das células é suplementado com G418 para uma concentração final de 50 ug / mL e com puromicina a uma concentração final de 0,5? g / mL.
NOTA: As células podem agora ser congelado (de acordo com protocolos convencionais para a criopreservação de células) para servir como uma reserva. Este é um passo importante para a reprodutibilidade do protocolo de diferenciação, uma vez que permite a utilização do mesmo lote de rtTA / NGN2 hiPSCs -positivas para muitas experiências futuras de diferenciação.
- Após o período de selecção 5 d, a cultura das hiPSCs -positivas rtTA / NGN2 como hiPSCs normais, com a excepção de que a forma E8 das células é suplementado com G418 para uma concentração final de 50 ug / mL e com puromicina a uma concentração final de 0,5? g / mL.
3. Diferenciação de rtTA / NGN2 hiPSCs -positivas aos neurônios em 6 poçosAAM e lamelas de vidro
NOTA: Neste protocolo, os detalhes são fornecidos para diferenciar rtTA / NGN2 hiPSCs -positivas em dois diferentes substratos, ou seja AMA 6 poços (dispositivos compostos por 6 poços independentes com 9 gravação e 1 de referência microeletrodos incorporados por poço) e lamelas de vidro em os poços de uma placa de 24 poços. Os protocolos, contudo, podem ser facilmente adaptados para substratos maiores (por exemplo, para os poços de placas de 12 ou 6 cavidades), dimensionando-se os valores mencionados de acordo com a área da superfície.
- Prepare os AAM ou lamelas de vidro (dia 0 e no dia 1)
- O dia antes do início da diferenciação, esterilizar os MEAs de acordo com a recomendação do fabricante.
- Dilui-se a proteína de adesão de poli-L-ornitina (PLO) em água ultrapura esterilizada até uma concentração final de 50 ug / mL. Cubra a área de eletrodo ativo da AAM de 6 poços, colocando um 100 Lqueda da OLP diluído em cada poço. Coloque as lamelas nos poços da placa de 24 poços usando pinças estéreis. Adicionar 800 ul da PLO diluído em cada poço. Impedir que as lamelas de flutuar, empurrando-os para baixo com a ponta da pipeta 1000 mL.
- Incubar as MEAs de 6 poços e 24 bem placa S / N numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2. No dia seguinte, aspirar o PLO diluída. Lave as superfícies de vidro dos MEAs de 6 poços e as lamelas duas vezes com água ultrapura estéril.
- Diluir laminina em frio DMEM / F12 a uma concentração final de 20 ug / mL (para as MEAs de 6 poços) e 10 ug / mL (para as lamelas de vidro). Imediatamente revestir a área do eléctrodo activo de MEAs de 6 poços, colocando uma gota 100 uL em cada poço. Da mesma forma, adicionar 400 uL da laminina diluído em cada poço da placa de 24 poços para revestir as lamelas. Impedir que as lamelas de flutuar, empurrando-os para baixo com a ponta da pipeta de 1000 mL.
- Incubar as MEAs de 6 poços e placa de 24 poços numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2 durante pelo menos 2 h.
- Placa os hiPSCs (dia 1)
NOTA: Os volumes que são mencionadas nos passos 3.2.1 - 3.2.4 assumir que os hiPSCs -positivas rtTA / NGN2 são cultivadas numa placa de 6 poços e que as células de um poço são colhidas. Os volumes que são necessários para o plaqueamento das células sobre as MEAs de 6 poços e / ou as lamelas depende do número de AAM de 6 poços e / ou o número de lamelas que são utilizadas na experiência; os números especificados nas etapas 3.2.6 - 3.2.8 permitem dimensionar a diferentes tamanhos de experiência.- Aqueça DMEM / F12, CDS e forma E8 com 1% (v / v) de penicilina / estreptomicina a R / T. Adicionar doxiciclina para uma concentração final de 4 ug / mL e inibidor ROCHA para a forma E8.
- Aspirar o meio gasto dos hiPSCs -positivas rtTA / NGN2 e adicionar 1 mL CDS aos hiPSCs. Incubar 3-5 min numa atmosfera humidificada de 37 ° C incubadora com uma atmosfera de 5% de CO 2. Verificar se ao microscópio as células são separar um do outro.
- Adicionar 2 ml de DMEM / F12 no poço, suavemente suspender as células com uma pipeta de 1000 mL e transferência das células para um tubo de 15 mL. Adicionar 7 ml de DMEM / F12 para a suspensão de células. Rodar as células a 200 xg durante 5 min.
- Aspirar o sobrenadante e adicionar 2 mL do meio E8 preparado. Dissociar a hiPSCs, colocando a ponta de uma pipeta de 1000 mL de encontro ao lado do tubo de 15 mL e ressuspensão das células suavemente. Verificar sob o microscópio se as células são dissociadas.
- Determinar o número de células (células / mL) utilizando uma câmara de hemocitómetro.
NOTA: Uma placa de 6 poços bem a 80 - 90% de confluência irá tipicamente produzir 3,0 - 4,0 x 10 6 células no total. - Aspirar a laminina diluída. Para as MEAs de 6 poços, diluir as células para se obter um cesuspensão ll de 7,5 x 10 5 células / mL. Placa as células por adição de uma gota de 100 mL da suspensão de células sobre a área do eléctrodo activo, em cada poço de MEAs de 6 poços. Para as lamelas, diluir as células para se obter uma suspensão de células de 4,0 x 10 4 células / ml. Placa as células por adição de 500 uL da suspensão de células para os poços da placa de 24 poços.
NOTA: A densidade celular final sobre as MEAs é mais elevada do que nas lamelas (Figura 1A e B). Descobrimos que esta alta densidade celular foi necessário para a gravação adequada da atividade da rede. No protocolo, os números são, desde que acabou por ser ideal para os ensaios. - Coloque as MEAs de 6 poços e placa de 24 poços numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2 durante 2 h (MEA) ou O / N (placa de 24 poços).
- Após 2 h, adicionar cuidadosamente 500 ul do meio E8 preparado a cada poço das AAM de 6 poços. Coloque a 6 nósLL AAM O / N numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2.
- Alterar o meio (dia 2)
- No dia seguinte, preparar DMEM / F12 com 1% (v / v) suplemento N-2, 1% (v / v) de aminoácidos não essenciais e 1% (v / v) de penicilina / estreptomicina. Adicionar humana recombinante neurotrofina-3 (NT-3) a uma concentração final de 10 ng / mL, o factor derivado do cérebro humano recombinante neurotrófico (BDNF) a uma concentração final de 10 ng / mL, e doxiciclina para uma concentração final de 4 ug / mL. Aquecer o meio a 37 ° C.
- Adicionar laminina a uma concentração final de 0,2 ug / ml para o meio. Filtra-se o meio resultante. Aspirar o meio consumido a partir dos poços de MEAs de 6 poços e a placa de 24 poços e substituí-lo com a forma preparada. Incubar as MEAs de 6 poços e 24 bem placa S / N numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2.
- Adicionar astrócitos de rato (dia 3)
NOTA: Os volumes que são mencionados neste protocolo assumem que os astrócitos de rato são cultivadas em frascos de cultura T75. É crítico que os astrócitos de rato que são adicionadas às culturas são de boa qualidade. Utilizamos dois critérios para verificar se os astrócitos de rato são de boa qualidade. Em primeiro lugar, a cultura de astrócitos de rato deve ser capaz de crescer confluente dentro de dez dias após o isolamento a partir de cérebros embrionários de rato. Em segundo lugar, depois de dividir a cultura de astrócitos de rato, os astrócitos de rato deve ser capaz de formar um confluentes, tesselada monocamada (Figura 1C). Se a cultura de astrócitos de rato não cumpre estes dois critérios, aconselhamos a não utilizar esta cultura para experimentos de diferenciação.- Quente 0,05% de tripsina-EDTA à temperatura ambiente. Aquecer a DPBS e DMEM / F12 com 1% (v / v) de penicilina / estreptomicina a 37 ° C.
- Aspirar o meio gasto da cultura de astrócitos de rato. Lave a cultura através da adição de 5 mL de DPBS e swish ao redor delicadamente.
- ASPIRcomeu o DPBS e adicionar 5 mL de 0,05% de tripsina-EDTA. Swish a tripsina-EDTA em torno gentilmente. Incubar numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2 durante 5 - 10 min.
- Verifique sob o microscópio se as células são separadas. Retire as últimas células por bater o balão algumas vezes.
- Adicionar 5 mL de DMEM / F12 para o balão. Tritura-se as células cuidadosamente dentro do frasco com uma pipeta de 10 mL. Recolha a suspensão celular num tubo de 15 mL. Girar o tubo a 200 xg durante 8 min.
- Aspirar o sobrenadante e ressuspender as células em 1 mL de DMEM / F12. Determinar o número de células (células / mL) utilizando uma câmara de hemocitómetro.
- Adicionar 7,5 x 10 4 por po de astrócitos as MEAs de 6 poços. Adicionar 2,0 x 10 4 astrócitos por poço da placa de 24 poços. Incubar as MEAs e a placa de 24 cavidades O / N numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2.
- Alterar o meio (dia 4)
- Preparar meio Neurobasal com 2% (v / v) de B-27, suplemento, 1% (v / v) de L-alanil-L-glutamina e 1% (v / v) de penicilina / estreptomicina. Adicionar NT-3 a uma concentração final de 10 ng / mL, o BDNF a uma concentração final de 10 ng / mL, e doxiciclina para uma concentração final de 4 ug / mL. Além disso, adicionar citosina β-D-arabinofuranósido a uma concentração de 2 uM.
NOTA: Citosina β-D-arabinofuranósido é adicionado ao meio para inibir a proliferação dos astrócitos e para matar os restantes hiPSCs que não são se diferenciar em neurónios. - Filtrar a médio e quente a 37 ° C. Aspirar o meio consumido a partir dos poços de MEAs de 6 poços e a placa de 24 poços e substituí-lo com a forma preparada. Manter as MEAs de 6 poços e a placa de 24 poços numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2.
- Preparar meio Neurobasal com 2% (v / v) de B-27, suplemento, 1% (v / v) de L-alanil-L-glutamina e 1% (v / v) de penicilina / estreptomicina. Adicionar NT-3 a uma concentração final de 10 ng / mL, o BDNF a uma concentração final de 10 ng / mL, e doxiciclina para uma concentração final de 4 ug / mL. Além disso, adicionar citosina β-D-arabinofuranósido a uma concentração de 2 uM.
- Refrescar o meio (dia 6-28)
NOTA: A partir do dia 6, atualizar metadedo meio a cada dois dias. A partir do dia 10 em diante, o meio é suplementado com FBS para suportar a viabilidade dos astrócitos.- Prepare meio Neurobasal com 2% (v / v) de B-27, suplemento, 1% (v / v) de L-alanil-L-glutamina e 1% (v / v) de penicilina / estreptomicina. Adicionar NT-3 a uma concentração final de 10 ng / mL, o BDNF a uma concentração final de 10 ng / mL, e doxiciclina para uma concentração final de 4 ug / mL. A partir do dia 10 em diante, também suplementar o meio com 2,5% (v / v) de FBS. Filtra-se o meio resultante e aquecido a 37 ° C.
- Remover metade do meio gasto a partir dos poços de MEAs de 6 poços e a placa de 24 poços utilizando uma pipeta de 1000 mL e substituí-lo com a forma preparada. Manter as MEAs de 6 poços e a placa de 24 poços numa atmosfera humidificada a 37 ° C incubadora com uma atmosfera de 5% de CO 2.
4. Estabelecer o perfil neurofisiológica de Neurônios hiPSC derivadas
NOTA: Duas a três semanas após a emdução de diferenciação, os neurónios hiPSC-derivados pode ser usada para diferentes análises a jusante. Nesta secção, exemplos de algumas análises são dados a jusante que pode ser realizado para estabelecer o perfil neurofisiológicos dos neurónios hiPSC-derivados.
- Caracterizar a atividade de rede neuronal usando AAM
- Ficha 20 min de atividade eletrofisiológica de neurônios hiPSC derivados cultivadas na AAM. Durante a gravação, manter a temperatura a 37 ° C, e evitar a evaporação e pH mudanças do meio de insuflação de um fluxo constante e lento de gás humidificada (5% de CO2, 20% O2, 75% N2) para o MEA .
- Depois de 1200X de amplificação (MEA 1060, MCS), a amostragem do sinal a 10 kHz usando o cartão de aquisição de dados MCS. Analisar os dados (pico e detecção estourar) usando um pacote de software personalizado 23.
- Caracterizar a atividade eletrofisiológica de uma única célula
- Transferir as lamelas contendo as culturas neuronais hiPSC derivados para a câmara de gravação de platina fixa submersa em um microscópio vertical. Ficha 20 min da evocadas por potenciais de ação espontânea correntes pós-sináptico (sEPSC) 24. Detectar o evento sináptica usando o programa de neurociência.
- Caracterizar a morfologia neuronal e expressão de sinapsina
- Fix e manchar os neurônios hiPSC-derivados para MAP2, sinapsina-1/2 e PSD-95 22, 24, 25. Quantificar o número de sinapsina-1/2 e PSD-95 pontos lacrimais usando software de análise de imagem.
Resultados
Aqui temos modificada com êxito um protocolo em que hiPSCs são diferenciados directamente em neurónios corticais por sobre-expressar o factor de transcrição neurogenin-2 12 e que se adaptaram-lo para o uso de AAM. Esta abordagem é rápida e eficiente que nos permite obter neurônios funcionais e atividade de rede já durante a terceira semana após a indução de diferenciação.
Durante o curso do protocolo de diferenciação, as células morfologicamente começou a assemelhar-se neurónios: processos pequenas foram formados e neurónios começou ligar uns aos outros (Figura 1A). Nós estabelecemos um perfil neuro-fisiológica dos neurônios derivados de uma linha hiPSC-controle saudável, medindo sua morfologia neuronal e propriedades sinápticas durante o desenvolvimento. hiPSC neurónios derivados foram coradas para MAP2 e sinapsina-1/2 em diferentes dias após o iníciode diferenciação (Figura 2A). Os neurónios derivados mostram morfologia neuronal maduro já 3 semanas após a indução de diferenciação. O número de sinapsina-1/2 pontos lacrimais (uma medida para o número de sinapses) foi quantificada com base na sinapsina-1/2 colorações imunocitoquímica. O número de pontos lacrimais sinapsina-1/2 aumentou com o tempo, sugerindo que o nível de conectividade neuronal está também a aumentar (Figura 2B). O número de sinapsina-1/2 pontos lacrimais 23 dias após a indução de diferenciação foi similar em duas linhas independentes de IPS (Figura 2C). Aos 23 DIV mais synapsin1 / 2 pontos lacrimais foram justapostos a PSD-95 pontos lacrimais, o que é indicativo de sinapses funcionais (Figura 2D).
Consistente com os resultados descritos por Zhang et al., Foi gerada uma população de neurónios corticais da camada superior excitatórios, confirmada por m-cortical específica de subtipo e pan-neuronal arkers tais como BRN2 e SATB2 (camada II / III). Nós não observamos neurônios que eram positivos para os neurônios da camada profunda CTIP2 (camada V) ou FOXP2 (camada VI) (Figura 2E e F)
Para caracterizar a atividade eletrofisiológica dos neurônios hiPSC derivados, utilizamos corrente de célula inteira e gravações braçadeira de tensão, ou seja, as propriedades intrínsecas e entrada excitatória sobre esses neurônios foram medidos durante o desenvolvimento. Os neurónios foram capazes de gerar potenciais de acção já uma semana após a da diferenciação e a percentagem de células que crava foi aumentando ao longo do tempo (Figura 2G e H). Além disso, os neurónios de entrada recebido sináptica excitatória já uma semana após a indução de diferenciação: tanto a frequência ea amplitude da entrada sináptica excitatória aumentada durante o desenvolvimento (Figura 2I - K).
nt "fo: manter-together.within-page =" 1 "> Para entender melhor como a atividade de uma única célula combina para formar funções de nível de rede, é essencial estudar como os neurônios trabalham em conjunto in vitro redes neuronais cultivadas em AAM. constituem um modelo experimental para o estudo da dinâmica neuronais. Foram registradas 20 min de atividade da rede eletrofisiológico dos neurônios derivados de uma linha hiPSC-controle saudáveis cultivados em 6 poços AAM (Figura 2 M). Poucas semanas após a indução de diferenciação, os neurônios derivado de hiPSCs-controlo saudável formado redes neuronais funcionalmente activas, mostrando eventos espontâneos (0,62 ± 0,05 pico / s; a Figura 2N). Nesta fase de desenvolvimento (isto é, 16 d após o início da diferenciação) há eventos síncronos envolvendo todos os canais . dos MEAs são detectados (Figura 2O) o nível de atividade da rede aumentaram durante o desenvolvimento: durante a quarta semana após t ele indução de diferenciação, as redes neuronais mostraram elevado nível de actividade espontânea (2,5 ± 0,1 pico / s; A Figura 2N) em todos os poços de o dispositivo. As redes também exibiu rajadas síncronos de rede (4,1 ± 0,1 explosão / min, Figura 2O) com longa duração (2.100 ± 500 ms).
Figura 1: hiPSC diferenciação em neurônios. A. Três pontos de tempo de diferenciação hiPSCs em neurônios em lamelas. B. chapeamento de hiPSCs na AAM. C. Os astrócitos em 100% de confluência no frasco T75 (note que as células formam uma monocamada tessellated). Barras de escala: 150? m. Por favor clique aqui para ver uma versão maior desta figura.
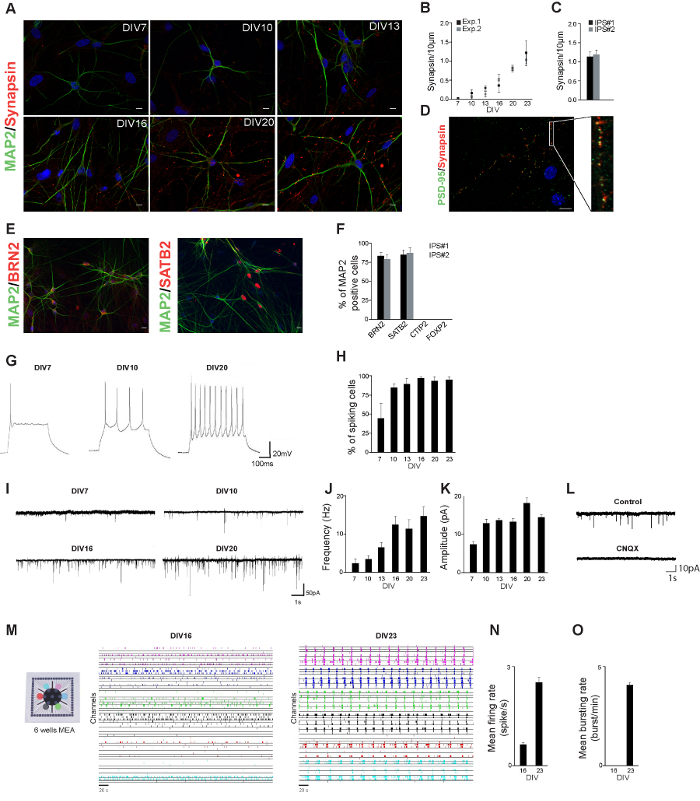
Figura 2. hiPSC derivado Caracterização Os neurónios. Neurónios A. hiPSC derivados foram coradas para MAP2 (verde) e sinapsina-1/2 (vermelho) em diferentes dias após o início da diferenciação. Barra de escala: 10 ^ m. B. Quantificação de pontos lacrimais sinapsina em duas experiências independentes. Em cada experiências foram analisadas pelo menos 10 células. C. Quantificação de puncta sinapsina em DIV23 em neurônios derivados de duas linhas IPS independentes. Neurónios D. hiPSC derivados foram coradas para PSD-95 (verde) e sinapsina-1/2 (vermelho) 23 dias após o início da diferenciação. pontos lacrimais sinapsina são justapostos ao PSD-95 pontos lacrimais. Neurónios E. hiPSC derivados foram coradas para MAP2 (verde) e BRN2 (vermelho) ou SATB2 (vermelho) 23 d após o início da diferenciação. F. percentagem de células positivas MAP2 queforam positivas para os marcadores indicados. G. representativos gravações braçadeira atual demonstra que os potenciais de ação podem ser gerados a partir de 7 d após o início da diferenciação. H. percentagem de células em diferentes dias após a indução de diferenciação que mostram uma ou mais potenciais de acção. I. traços representativos de correntes pós-sinápticos excitatórios (EPSCS) recebidos pelos neurônios hiPSC derivados em diferentes dias após a diferenciação. J. Frequência de correntes pós-sinápticos excitatórios durante o desenvolvimento. K. A amplitude das correntes excitatório pós-sináptico durante o desenvolvimento. L. Representante vestígios de gravações EPSC sem (controle) e com CNQX (CNQX). M. Os neurónios derivados de uma linha hiPSC foram cultivadas em um MEA de 6 poços e actividade de rede é mostrado para as redes neuronais hiPSC derivados 16 e 23 d após a indução de diferenciação. A actividade registada decada (taxa de amostragem de 10 kHz) está bem indicado com uma cor diferente (5 min a 20 min de de gravação são mostradas). N. taxa de disparo 16 e 23 d após a indução de diferenciação. O. taxa Estourando 16 e 23 d após a indução de diferenciação. Por favor clique aqui para ver uma versão maior desta figura.
Tendo em conta os resultados, a qualidade dos neurónios derivados de hiPSC resultantes pode ser avaliada fazendo um perfil neurofisiológicos das células. Isto é, três a quatro semanas após o início da diferenciação, a morfologia, sinapsina-1/2 e electrofisiologia expressão dos neurónios pode ser avaliada. Nesse ponto de tempo, espera-se que os neurónios hiPSC-derivadas para mostrar uma morfologia neuronal semelhante, para ser MAP2, sinapsina / PSD-95 imunocitoquímica positiva quando se realiza, e para exposiT espontânea actividade electrofisiológica (em ambas as de uma única célula e nível de rede).
Discussão
Aqui temos implementado um protocolo hiPSC diferenciação eficiente publicada por Zhang et al. (2013) 12 para medir a atividade de rede de redes neuronais hiPSC derivados na AAM. Nós adaptamos o protocolo original, criando uma linha hiPSC -positivo rtTA / NGN2 antes de induzir a diferenciação neuronal. Este passo adicional permite controlar a densidade de células neuronais no MEA. O controle sobre a densidade neuronal foi um importante pré-requisito para adaptar o protocolo a AAM e para garantir a coerência. Para medir a actividade das redes neuronais usando AAM, os neurônios precisam formar densas redes diretamente em cima dos eletrodos MEA 17,18. Isso requer, necessariamente, um controlo apertado sobre a densidade de revestimento dos neurônios. A linha de hiPSC -positivo rtTA / NGN2 permite o controle de neurônio densidade porque essa tática não depende de transduções lentivirais agudos de hiPSCs antes da diferenciação;a linha hiPSC -positivo rtTA / NGN2, portanto, quase elimina qualquer variação no rendimento final devido a, por exemplo, toxicidade lentiviral e eficiência de infecção variável.
Outro passo importante do procedimento experimental é o número dos astrócitos de rato que são co-cultivadas com as hiPSCs diferenciadores. Astrócitos contribuir activamente para o refinamento de desenvolvimento de circuitos neurais, controlando a formação de sinapses, manutenção e eliminação, todos os quais são processos importantes para o funcionamento neuronal. O protocolo apresentado neste trabalho é altamente dependente astrócitos: a plena maturidade e formam sinapses funcionais, os neurônios exigem o apoio dos astrócitos. Nós experiente que o número de astrócitos deve ser aproximadamente igual ao número de neurónios hiPSC-derivadas para suportar a maturação dos neurónios e a formação de redes neuronais exibindo actividade espontânea. Desde que os nossos astrócitos protocolo rendimentos cul célula primáriaturas com um tempo de vida limitado, o isolamento de astrócitos de rato tem que ser executada regularmente.
A nossa adaptação do protocolo de publicada por Zhang et ai. (2013) 12 para uso com a tecnologia MEA provavelmente vai melhorar significativamente a nossa capacidade de estudar a atividade de rede de redes hiPSC derivados. Anteriormente, os protocolos utilizados para o estudo de redes neuronais hiPSC derivados com AAM invocado procedimentos de diferenciação demoradas 13-16. O protocolo de Zhang et al. (2013) proporciona uma alternativa rápida, e a modificação elimina uma fonte de variabilidade, que torna-se agora mais viável a utilização de neurónios hiPSC derivados em combinação com a tecnologia de MEA, especialmente em high-throughput ou estudos farmacológicos. Além disso, porque o método de publicada por Zhang et ai. (2013) 12 produz uma população homogénea de camada superior neurônios corticais, o nosso protocolo adaptado possibilita estudos focados na rede umctivity deste subconjunto neuronal particular.
No entanto, esta abordagem tem também várias limitações. Em primeiro lugar, a homogeneidade das culturas pode também ser considerada uma desvantagem, porque as culturas são menos propensos a se assemelhar em redes in vivo, em que diferentes classes de neurónios (isto é, neurónios inibitórios e excitatórios) constituem uma rede heterogénea. Para melhorar ainda mais a utilização dos neurónios hiPSC derivados com tecnologia MEA, será importante para desenvolver protocolos rápidos de diferenciação (à base de transgene) para outras populações de células neuronais. Se protocolos tornam-se disponíveis, as in vitro redes imitaria em redes in vivo mais de perto. Em segundo lugar, em astrócitos de rato presentes devem ser adicionados aos neurónios hiPSC-derivadas para suportar o crescimento, e, por conseguinte, a rede neuronal resultante não é uma neuronal sensu stricto rede humana. protocolos confiáveis para diferenciar hiPSCs em astrócitos pode no futuro solve 26 este problema. Em terceiro lugar, as redes neuronais bidimensionais, conforme descrito aqui, são um modelo limitado para estudar complexos tridimensionais in vivo redes neuronais. Felizmente, descrevem protocolos culturas tridimensionais de neurónios primários de rato em combinação com a tecnologia de MEA já estão disponíveis 27,28. Prospectivamente, a combinação de protocolos rápidos para a obtenção de diferenciação de neurónios e astrócitos hiPSC derivados com técnicas de cultura tridimensionais e tecnologia MEA deve proporcionar novos visão sobre os mecanismos biológicos perturbações neurológicas subjacentes.
Divulgações
The authors have nothing to disclose.
Agradecimentos
The authors thank Jessica Classen for performing the whole-cell patch-clamp experiments. The hiPSCs used in our experiments were kindly provided by Huiqing Zhou and Willem van den Akker from the Radboud University Nijmegen. The transfer vectors used in this protocol were kindly provided by Oliver Brüstle, Philipp Koch and Julia Ladewig from the University of Bonn Medical Centre.
Materiais
| Name | Company | Catalog Number | Comments |
| Lebovitz's L-15 medium | Gibco | 11415-064 | |
| B-27 supplement | Gibco | 0080085SA | |
| Poly-D-Lysine (PDL) | Sigma-Aldrich | P6407 | |
| Ca2+/Mg2+-free HBSS | Gibco | 14175-095 | |
| 0.05% Trypsin-EDTA | Gibco | 25300-054 | |
| 2.5% Trypsin | Gibco | 15090-046 | |
| High-glucose DMEM | Gibco | 11965-092 | |
| FBS (Fetal Bovine Serum) | Sigma-Aldrich | F2442-500ML | |
| Penicillin/Streptomycin | Sigma-Aldrich | P4333 | |
| 70 µm cell strainer | BD Falcon | 352350 | |
| DPBS | Gibco | 14190-094 | |
| psPAX2 lentiviral packaging vector | Addgene | Plasmid #12260 | |
| pMD2.G lentiviral packaging vector | Addgene | Plasmid #12259 | |
| Basement membrane matrix | Gibco | A1413201 | |
| DMEM/F12 | Gibco | 11320-074 | |
| Cell detachment solution | Sigma-Aldrich | A6964 | |
| E8 medium | Gibco | A1517001 | |
| ROCK inhibitor | Gibco | A2644501 | Alternatively, ROCK inhibitors like thiazovivin can be used. |
| Polybrene | Sigma-Aldrich | H9268-5G | |
| G418 | Sigma-Aldrich | G8168-10ML | |
| Puromycin | Sigma-Aldrich | P9620-10ML | |
| Vitronectin | Gibco | A14700 | |
| 6-well MEAs | Multi Channel Systems | 60-6wellMEA200/30iR-Ti-tcr | |
| Glass coverslips | VWR | 631-0899 | |
| Poly-L-Ornithine (PLO) | Sigma-Aldrich | P3655-10MG | |
| Laminin | Sigma-Aldrich | L2020-1MG | |
| Doxycyclin | Sigma-Aldrich | D9891-5G | |
| N-2 supplement | Gibco | 17502-048 | |
| Non-essential amino acids | Sigma-Aldrich | M7145 | |
| NT-3, human recombinant | Promokine | C66425 | |
| BDNF, human recombinant | Promokine | C66212 | |
| Trypsin-EDTA | Gibco | 25300-054 | |
| L-alanyl-L-glutamine | Gibco | 35050-038 | |
| Neurobasal medium | Gibco | 21103-049 | |
| Cytosine β-D-arabinofuranoside | Sigma-Aldrich | C1768-100MG | |
| Straight fine-tipped forceps | Fine Science Tools | 11251 | |
| Fine-tipped spring scissors | Fine Science Tools | 91500-09 |
Referências
- Ahfeldt, T., Litterman, N. K., Rubin, L. Studying human disease using human neurons. Brain Res. , (2016).
- Zhang, S. C., Wernig, M., Duncan, I. D., Brustle, O., Thomson, J. A. In vitro differentiation of transplantable neural precursors from human embryonic stem cells. Nat Biotechnol. 19 (12), 1129-1133 (2001).
- Perrier, A. L., et al. Derivation of midbrain dopamine neurons from human embryonic stem cells. Proc Natl Acad Sci USA. 101 (34), 12543-12548 (2004).
- Wu, H., et al. Integrative genomic and functional analyses reveal neuronal subtype differentiation bias in human embryonic stem cell lines. Proc Natl Acad Sci USA. 104 (34), 13821-13826 (2007).
- Chambers, S. M., Fasano, C. A., Papapetrou, E. P., Tomishima, M., Sadelain, M., Studer, L. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Shi, Y., Kirwan, P., Livesey, F. J. Directed differentiation of human pluripotent stem cells to cerebral cortex neurons and neural networks. Nat Protoc. 7, 1836-1846 (2012).
- Espuny-Camacho, I., et al. Pyramidal neurons derived from human pluripotent stem cells integrate efficiently into mouse brain circuits in vivo. Neuron. 77 (3), 440-456 (2013).
- Maroof, A. M., et al. Directed differentiation and functional maturation of cortical interneurons from human embryonic stem cells. Cell Stem Cell. 12 (5), 559-572 (2013).
- Johnson, M. A., Weick, J. P., Pearce, R. A., Zhang, S. C. Functional neural development from human embryonic stem cells: accelerated synaptic activity via astrocyte coculture. J Neurosci. 27 (12), 3069-3077 (2007).
- Hu, B. Y., et al. Neural differentiation of human induced pluripotent stem cells follows developmental principles but with variable potency. Proc Natl Acad Sci USA. 107 (9), 4335-4340 (2010).
- Kim, H., et al. miR-371-3 expression predicts neural differentiation propensity in human pluripotent stem cells. Cell Stem Cell. 8 (6), 695-706 (2011).
- Zhang, Y., et al. Rapid single-step induction of functional neurons from human pluripotent stem cells. Neuron. 78 (5), 785-798 (2013).
- Odawara, A., Saitoh, Y., Alhebshi, A. H., Gotoh, M., Suzuki, I. Long-term electrophysiological activity and pharmacological response of a human induced pluripotent stem cell-derived neuron and astrocyte co-culture. Biochem Biophys Res Commun. 443 (4), 1176-1181 (2014).
- Odawara, A., Katoh, H., Matsuda, N., Suzuki, I. Physiological maturation and drug responses of human induced pluripotent stem cell-derived cortical neuronal networks in long-term culture. Sci Rep. 6, 26181 (2016).
- Amin, H., Maccione, A., Marinaro, F., Zordan, S., Nieus, T., Berdondini, L. Electrical Responses and Spontaneous Activity of Human iPS-Derived Neuronal Networks Characterized for 3-month Culture with 4096-Electrode Arrays. Front Neurosci. 10, (2016).
- Heikkila, T. J., et al. Human embryonic stem cell-derived neuronal cells form spontaneously active neuronal networks in vitro. Exp Neurol. 218 (1), 109-116 (2009).
- Massobrio, P., Massobrio, G., Martinoia, S. Multi-program approach for simulating recorded extracellular signals generated by neurons coupled to microelectrode arrays. Neurocomputing. 70, 2467-2476 (2007).
- Wang, L., Riss, M., Buitrago, J. O., Claverol-Tinturé, E. Biophysics of microchannel-enabled neuron-electrode interfaces. J Neural Eng. 9 (2), (2012).
- McCarthy, K. D., de Vellis, J. Preparation of separate astroglial and oligodendroglial cell cultures from rat cerebral tissue. J Cell Biol. 85, 890-902 (1980).
- Schildge, S., Bohrer, C., Beck, K., Schachtrup, C. Isolation and culture of mouse cortical astrocytes. J. Vis. Exp. (71), e50079 (2013).
- Pacifici, M., Peruzzi, F. Isolation and culture of rat embryonic neural cells: a quick protocol. J Vis Exp. (63), e3965 (2012).
- Ba, W., et al. ARHGAP12 functions as a developmental brake on excitatory synapse function. Cell Rep. 14 (6), 1355-1368 (2016).
- Bologna, L. L., et al. Investigating neuronal activity by SPYCODE multi-channel data analyzer. Neural Netw. 23 (6), 685-697 (2010).
- Ba, W., et al. TRIO loss of function is associated with mild intellectual disability and affects dendritic branching and synapse function. Hum Mol Genet. 25 (5), 892-902 (2016).
- Benevento, M., et al. Histone methylation by the Kleefstra syndrome protein EHMT1 mediates homeostatic synaptic scaling. Neuron. 91 (2), 341-355 (2016).
- Krencik, R., Weick, J. P., Liu, Y., Zhang, Z. -. J., Zhang, S. -. C. Specification of transplantable astroglial subtypes from human pluripotent stem cells. Nature biotechnology. 29 (6), 528-534 (2011).
- Frega, M., Tedesco, M., Massobrio, P., Pesce, M., Martinoia, S. Network dynamics of 3D engineered neuronal cultures: a new experimental model for in-vitro electrophysiology. Sci Rep. 4, 5489 (2014).
- Tedesco, M., Frega, M., Martinoia, S., Pesce, M., Massobrio, P. Interfacing 3D engineered neuronal cultures to micro-electrode arrays: an innovative in vitro experimental model. J Vis Exp. (104), e53080 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados