Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un dosage Simple journaliste Fluorescence pour identifier les composants cellulaires nécessaires à la ricine toxine une chaîne (RTA) traite des levures
Dans cet article
Résumé
Dans le manuscrit, nous décrivons l’utilisation d’un essai de journaliste de fluorescence à base de levure pour identifier les composants cellulaires impliqués dans le trafic et de tuer les processus de la cytotoxique une sous-unité de la ricine de toxine végétale (RTA).
Résumé
Bactérienne et centrale A / toxines B exploitent les voies naturelles de trafic dans les cellules eucaryotes pour atteindre leurs cibles intracellulaires dans le cytosol et finalement tuer. Tel A / toxines B sont généralement consistant d’un enzymatiquement actif Asubunit (p. ex., la toxine A (RTA) de la ricine) et une ou plusieurs cellule lie Bsubunit(s), qui est responsables de toxine contraignantes spécifiques des récepteurs de surface de cellules. Nos connaissances actuelles de la façon dont A / B toxines sont capables d’enivrantes efficacement aidés les cellules scientifiques pour comprendre les mécanismes cellulaires fondamentaux, comme l’endocytose et intracellulaire des protéines dans les cellules eucaryotes supérieures de tri. D’un point de vue médical, il est également important d’identifier les voies du trafic toxine majeure à trouver des solutions de traitement approprié pour les patients ou à finissent par développer des applications thérapeutiques pour le traitement du cancer à base de toxines.
Depuis les analyses du génome de A / la toxine B traite des cellules de mammifères est complexe, fastidieux et coûteux, plusieurs études sur A / transport de toxine B ont été réalisées dans l’organisme modèle de levure Saccharomyces cerevisiae. Bien qu’il soit moins complexes, fondamentaux des processus cellulaires chez les levures et les cellules eucaryotes supérieures sont semblables et très souvent les résultats obtenus chez les levures peuvent être transférées à la situation chez les mammifères.
Nous décrivons ici un essai de journaliste rapide et facile à utiliser pour analyser le trafic intracellulaire de RTA dans la levure. Un avantage essentiel de la nouvelle méthode de dosage est l’occasion d’étudier non seulement la RTA rétro-translocation du réticulum endoplasmique (ER) dans le cytosol, mais plutôt l’endocytose et toxine rétrograde de transport de la membrane plasmique dans le re. Le test utilise un plasmide de journaliste qui permet une mesure indirecte de la toxicité de la RTA à travers l’émission de fluorescence de la protéine fluorescente verte (GFP) après in vivo de la traduction. Comme RTA empêche efficacement l’initiation de la biosynthèse des protéines par dépurination ARNr 28 s, ce test permet l’identification des protéines de cellules hôtes impliqués dans le transport intracellulaire de RTA par le biais de la détection de changements dans l’émission de fluorescence.
Introduction
Patients souffrant d’infections par des bactéries productrices de toxine représentent un fardeau médical et financier grave pour chaque système de soins de santé social, en particulier les traitements thérapeutiques efficaces étant encore largement défaut. À élaborer de nouvelles stratégies thérapeutiques, les mécanismes de l’intoxication complexe de médicalement pertinente A / toxines B tels que la toxine cholérique, Shiga toxine ou ricine doivent être entièrement comprises au niveau moléculaire issu des nouveaux dosages puissants qui doivent être mises en œuvre.
Ces dernières années, plusieurs études ont tenté d’analyser A / transport de toxine B chez les levures et les cellules de mammifères en utilisant des méthodes fastidieuses et coûteuses telles que les toxines radioactives étiquetage1,2 mais aussi de dépistage basé sur siRNA 3les approches. Dans certains cas, traite de la toxine a été visualisée in vivo par microscopie de fluorescence après couplage chimique ou génétique des sous-unités de la toxine individuelle avec des fluorophores, points quantiques ou protéines fluorescentes4,5. Malheureusement, ces modifications entraînent souvent inactives et/ou altération des propriétés naturelles des toxines. Une autre façon élégante de répondre indirectement à une grande variété de questions scientifiques est l’utilisation de systèmes de journaliste issu des enzymes comme lacZ, luciférase, ou protéines fluorescentes (p. ex. GFP ou Acropora sp. (protéine fluorescente rouge dsRed)).
Dans ce manuscrit, un protocole simple est décrite qui identifie les composants cellulaires requis pour le transport intracellulaire des RTA extracellulaire appliquée chez S. cerevisiae. Ainsi, un plasmide de fluorescence-journaliste contenant un signal de N-borne ER-import suivi de GFP agit comme un capteur de biosynthèse de protéine, qui mesure indirectement l’inhibition de la protéine induite par la RTA traduction par GFP émission de fluorescence après in vivo traduction6. En cas que l’endocytose RTA et/ou trafic intracellulaire est négativement (ou positivement) affectés dans un mutant de délétion de levure particulière par rapport au type sauvage, cela peut être détectée par une augmentation (ou diminution) dans GFP fluorescence d’émission6.
Jusqu’ici, toutes les méthodes d’analyse transport RTA dans les cellules de levure sont limitaient au processus de rétro-translocation ER-à-cytosol de RTA. Dans un tel système artificiel, RTA contenant un signal d’importation ER s’exprime d’un promoteur inductible, ce qui entraîne un phénotype suicidaire1,7. Bien que la cellule liant la sous-unité B de la ricine est également absent dans le dispositif expérimental décrit dans ce manuscrit et, ainsi, ne pas entièrement représente la situation naturelle de ricine holotoxine intoxication8, transport de la toxine du plasma membrane à travers l’appareil de Golgi à la salle d’urgence peut être imité étroitement avec ce nouveau dosage. Fait intéressant, les résultats préliminaires obtenus dans l’étude pilote indiquent que les voies de trafic utilisés par RTA révèlent des ressemblances frappantes avec la route de l’intoxication de ricine holotoxine.
En résumé, la méthode décrite peut être utilisée pour déterminer le rôle spécifique des protéines cellulaires sélectionnés dans l’endocytose de la RTA et traite des levures. En outre, ce dispositif expérimental pourrait être facilement adapté aux autres ribosomes inactivation des toxines produites et sécrétées par les différentes levures et espèces bactériennes telles que zymocin ou la toxine de Shiga.
Protocole
Remarque : Une vue d’ensemble du flux de travail expérimental général est représenté dans la Figure 1.
ATTENTION : RTA est hautement toxique pour les humains. L’autorisation de laboratoire de sécurité S2 (niveau de biosécurité 2 équivalent) est nécessaire. Veuillez porter des gants pendant l’expérience entière.
1. hétérologue Expression de son étiquette RTA chez Escherichia coli
- Transformer des cellules d’e. coli avec l’expression plasmide pET24-RTA(sa) 6 ou sur le pET24a de vecteur vide(+) à l’aide d’électroporation standard protocoles9,10. Les cellules contenant le plasmide vide servent de témoin négatif.
- Après sélection des clones positifs sur plaques de LB (100 µg/mL) contenant de kanamycine, ensemencer les cellules contenant le plasmide d’expression RTA ou le vecteur vide dans un milieukan mL 5 LB (milieu LB avec 100 µg/mL de kanamycine) et incuber à 37 ° C et 220 tr/mn pendant 24 h.
- Supplément 1 LB Lkan moyen avec pré-culture 5 mL et incuber les cellules à 37 ° C et 220 tr/mn jusqu'à ce que les cellules ont atteint une OD600 de 0,8 à 1,0 (environ 3-4 h). Par la suite, réduire la température de culture à 28 ° C et préparer un 1 M de la solution mère de l’isopropyl-β-D-1-thiogalactopyranoside (IPTG) dans H2O.
- Induisent l’expression RTA de la e. coli en ajoutant IPTG à une concentration finale de 1 mM.
- Après 3,5 h à 28 ° C et 220 tr/mn, récolte des cellules par centrifugation à 10 000 x g et 4 ° C pendant 10 min, laver le culot deux fois avec 5 mL de tampon de liaison (500 mM NaCl, imidazole de 10 mM et 20 mM KH2PO4 pH = 7,4) à 10 000 x g et 4 ° C pendant 10 min et remettre pellet dans 5 mL de tampon de liaison.
Remarque : Le protocole peut être suspendue à ce stade et cellules peuvent être stockées à 80 ° C pendant plusieurs jours.
2. purification du RTA son marqués par chromatographie d’affinité
- Laisser agir de cellules sur la glace en utilisant le protocole suivant : 15 s pulse (20 microns), pause de s 30. Répétez cette opération cinq fois.
- Centrifuger la cellule lysat à 21 000 x g et 4 ° C pendant 15 minutes et filtrer le surnageant à l’aide d’un système de filtre de seringue stérile (taille des pores 0,2 µm).
NOTE : Granules cellulaires d’échantillons avec succès aux ultrasons montrent des bordures transparentes. - Utiliser un système de purification automatique équipé d’un 5 mL Ni2 +-base colonne d’affinité pour purifier la fraction RTA His-tag de la stérile filtré par e. coli surnageant. En général, utiliser une vitesse d’élution de 1 mL/min et laisser refroidir le système entier de purification afin d’empêcher la liaison de la toxine non efficace et perte d’activité de la toxine.
NOTE : Les paramètres pour la purification efficace de RTA sont répertoriés dans le tableau 1. Voir aussi Becker et al. Pour plus amples information sur9.- En bref, équilibrer la colonne d’affinité avec 20 mL de tampon de liaison pour retirer le tampon de stockage. Appliquer le surnageant stérile filtrée dans la colonne d’affinité à l’aide d’une seringue.
- Laver la colonne avec 25-35 mL de tampon de liaison pour éliminer les protéines non liées de la colonne. Exécutez les étapes de lavage jusqu'à l’absorbance UV à 280 nm est proche de la valeur initiale des UV.
- Éluer la fraction liée de RTA dans 20 à 35 mL de tampon d’élution (imidazole de 500 mM, 500 mM NaCl, 20 mM KH2PO4, pH = 7,2) et conserver l’échantillon sur la glace (Figure 2 a et 2 b de la Figure).
Remarque : L’élution de la fraction de la RTA est marquée par une augmentation de l’absorption UV. Veuillez noter pour collecter exclusivement cette fraction pour éviter la contamination avec des protéines non spécifiques liés. - Utilisez 20 µL d’échantillons éluées pour effectuer11 bleu de Coomassie ou Western blot analyse12,13 (facultatif). Utilisez les primaires contre son anticorps (1:1, 000) et secondaire anti-mouse-IgG-HRP (01:10, 000) pour détecter la RTA et de vérifier la pureté de l’échantillon (Figure 3).
- Desalt éluée fraction de RTA sur une colonne de dessalement de 5 mL et équilibrer échantillon à 0,8 M sorbitol.
- Remplacer la colonne d’affinité du système de purification par la colonne de dessalement et d’abord équilibrer la colonne avec 20 mL de 0,8 M sorbitol.
- S’applique à la fraction éluée de RTA pour la colonne en utilisant une seringue. Laver la colonne avec 100 mL de 0,8 M sorbitol et Éluer la morue dessalée fraction de RTA dans un tube de 15 mL dès l’absorption UV commence à augmenter (Figure 2).
- Fraction de magasin la LLUH éluée à 4 ° C.
Remarque : Arrêter directement d’échantillonnage de la RTA fraction lorsque la conductance augmente pour éviter toute contamination du sel. Paramètres pour la procédure de dessalement sont répertoriés dans le tableau 1.
- Éluées fraction de RTA à 10 000 x g et 4 ° C pendant 30 à 180 minutes en utilisant une colonne de spin de coupure 10 kDa pour un volume final de 1 à 2 mL de concentré et conserver l’échantillon à 4 ° C pendant 3 à 4 semaines.
ATTENTION : Ne pas congeler l’échantillon depuis gel conduit à une perte complète de l’activité de la RTA. - Déterminer la concentration de protéines à l’aide d’un kit de dosage de protéines classiques. La concentration de protéines doit être de l’ordre de 1 à 1,5 mg/mL.
Remarque : Le RTA a tendance à précipiter si la concentration en protéines est trop élevée (> 5 mg/mL).
3. Transformation et l’élimination de la paroi cellulaire de levure
- Transformer les mutants de délétion de levure sauvage ou sélectionné avec la GFP journaliste plasmide pRS315-K28SPGFP -6 à l’aide de lithium standard acétate transformation méthodes14. Incuber les cellules sur la leucine abandon (d/o) plaques de glucose (2 % de glucose, 1,5 % d’agar, sulfate d’ammonium 0,5 %, 0,17 % d’azote de levure Base (YNB) et 0,087 % d/o mix sans leucine) à 30 ° C pendant 2-3 jours pour la sélection positive clone.
- Sélectionner les 3 levure différents clones de chaque plaque et ensemencer les clones dans 100 mL de leucine d/o raffinose moyenne (2 % raffinose, sulfate d’ammonium 0,5 %, 0,17 % YNB et 0,087 % d/o mélanger sans leucine) à 220 tr/mn et 30 ° C à OD600 = 1,0 à 2,0 (2-4 x 107 cellules/mL).
NOTE : La croissance des cellules des différentes souches de levures suppression est différente. Moniteur OD600 via un spectrophotomètre. - Pour calculer les valeurs de600 OD, diluer les échantillons à OD600 = 0,1-0,3 (1:5 à 01:10 dilutions) et mesurer OD600 dans un spectrophotomètre. Comme référence, utilisez H2O additionné de la quantité correspondante de milieu de leucine d/o raffionose.
- Après finition étape 3.5, mélanger 4 µL de la culture de spheroplasted 50 mL avec 496 µL de tampon de sphéroplastes (environ 2 × 106 sphéroplastes) et centrifuger pendant 10 min à 400 g.
- Culot de remettre dans 10 mL H2O distillée, échantillon de vortex pour 30 s et plaque à 10 µL de l’échantillon sur des plaques de glucose d/o leucine (2 % de glucose, 1,5 % d’agar, sulfate d’ammonium 0,5 %, 0,17 % YNB et 0,087 % d/o mix sans leucine).
- Incuber les cellules pendant 3 jours à 30 ° C. Compter le nombre total de colonies de cellules cultivées sur la plaque. Pour l’évaluation des données, utilisez uniquement des échantillons avec une efficacité supérieure à 98 % (total nombre de colonies de cellules < 40 colonies/plaque).
4. GFP Reporter Assay mesure en plaques 96 puits
- Graine sur les sphéroplastes de cellules de levure obtenues à l’étape 3,7 en plaques 96 puits (200 µL/puits).
- Ajouter 70 µL leucine stabilisée d/o raffinose milieu contenant du contrôle négatif (éluat d’a Ni2 +-affinité purifié lysat cellulaire induite par l’IPTG e. coli cellules exprimant le vecteur vide) ou purifiée RTA dans une concentration finale de RTA de 5 µM ( correspond à 160 g/L RTA) dans chaque puits.
- Immédiatement ajouter 30 µL stabilisé galactose solution (galactose de 30 %, 0,8 M sorbitol) pour induire l’expression de la GFP et ensuite lancer la mesure.
Remarque : Effectuer au moins 3 répétitions techniques par expérience et biologiques 3 répétitions par souche de levure. - Après avoir terminé la préparation de l’échantillon (mesures 4.1-4.3), mettre la plaque de 96 puits en un lecteur de fluorescence et démarrer la mesure. Utilisez le filtre de nm de 475/509 requis pour la détection par fluorescence GFP. Effectuer des mesures à 30 ° C, 120 t/mn et avec un secousse diamètre de 1 mm sur une fenêtre de temps de 20 h (10 min d’intervalle de mesure).
Remarque : Le jeu de filtres GFP est normalement disponible dans tous les systèmes de lecteur. Température, la fenêtre de temps, mesure des intervalles et la concentration de RTA peut être ajustée pour les besoins propres. Résultats représentatifs sont indiquées à la Figure 4. - En option : Utiliser des contrôles internes supplémentaires dans la mesure pour le contrôle qualité. Préparer un contrôle négatif en ajoutant 30 µL leucine stabilisée d/o raffinose de milieu au lieu de 30 % de galactose (aucune induction GFP). En outre, ajouter 70 µL de solution de G418 sorbitol stabilisé de 0,8 M (300 µg/mL). L’inhibiteur de la protéine traduction G418 sert de témoin positif pour l’inhibition de la protéine car il empêche l’expression de la GFP dans la levure.
- Calculer la fluorescence GFP relative en pourcentage pour le point de temps 20 h selon l’équation suivante (voir Figure 4 a) :
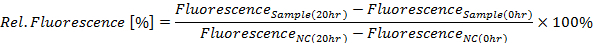 NC où est le contrôle négatif
NC où est le contrôle négatif
- Vous pouvez également déterminer la fluorescence GFP relative pour chaque point de mesure (voir aussi dans ce cas 10 min, l’étape 4.4) en utilisant l’équation ci-dessus. Comme le montre la Figure 4 b, créer un graphique en tamponnant les intensités de fluorescence GFP (axe y) au fil du temps (axe des x).
Résultats
Le déroulement général du protocole décrit dans ce manuscrit est illustré à la Figure 1, environ résumant les étapes simples pour succès RTA purification et l’expérience de dosage de journaliste GFP ultérieure. On trouvera une description plus détaillée de chaque étape individuelle dans le protocole. La figure 2 illustre le résultat attendu d’une purification de RTA réussie par chromatographie d’affinité (<...
Discussion
Lorsque vous effectuez le protocole ci-dessus, nous vous recommandons les suggestions suivantes pour parvenir à une issue positive de l’expérience.
Pour l’expression de la protéine hétérologue, il est important pour ne pas dépasser la concentration de l’IPTG de 1 mM. Concentrations de l’IPTG > 1 mM inhibent l’expression de RTA induite par le promoteur et le plomb pour abaisser les rendements de la toxine. En outre, les cellules ne soient pas cultivées à des températures ...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Parties de cette étude ont été gracieusement pris en charge par une bourse de la Deutsche Forschungsgemeinschaft (SFB 1027, A6).
matériels
| Name | Company | Catalog Number | Comments |
| Bacterial and yeast strains | |||
| E. coli BL21 DE3(Gold) | Aligent Technologies | 230130 | |
| S. cerevisiae BY4742 | Euroscarf | Y10000 | |
| S. cerevisiae BY4742 deletion mutants | Dharmacon | YSC1054 | whole collection |
| Name | Company | Catalog Number | Comments |
| Plasmids used in this protocol | |||
| pET24a(+) (Novagen) | Millipore | 69772-3 | |
| pET-RTA(His6) | Becker et al. (2016)3 | ||
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Zymolyase 20T | USBio | Z1000.250 | lytic enzyme for cell wall removal |
| LB broth medium | Thermo Scientific | 10855021 | 15 g agar was added for plate production |
| YNB | Thermo Scientific | DF0335-15-9 | |
| Ammonium sulfate | Sigma-Aldrich | A4418-100G | |
| Yeast drop-out mix supplemts without leucine | Sigma-Aldrich | Y1376-20G | |
| Agar | Sigma-Aldrich | 05040-100G | |
| D-glucose | Sigma-Aldrich | G8270-100G | |
| DTT | Sigma-Aldrich | 10197777001 | |
| D-raffinose | Sigma-Aldrich | 83400-25G | |
| D-sorbitol | Sigma-Aldrich | S1876-1KG | |
| D-galactose | Sigma-Aldrich | G0750-10MG | |
| G418 | Thermo Scientific | 11811031 | |
| IPTG | Sigma-Aldrich | I6758-1G | |
| Imidazole | Roth | 3899.1 | |
| PAGE ruler prestained | Fermentas | 26616 | protein ladder used for Western analysis |
| Name | Company | Catalog Number | Comments |
| Material for RTA purification, desalting and reader measurements | |||
| Spectrophotometer Ultrospec 2100 pro | Amersham | ||
| Soniprep 150 | MSE | old model, other models available | |
| Fluoroskan Ascent | Thermo Scientific | 5210470 | old model, not available anymore |
| ÄKTAPurifier | Thermo Scientific | 28406266 | Product is discontinued and replaced |
| HisTRAP HP column | GE Healthcare | 17-5248-02 | |
| HiTRAP desalting column | GE Healthcare | 11-0003-29 | |
| Midisart sterile filter | Sartorius | 16534K | 0.2 µm pore size |
| BCA protein assay kit | Pierce | 23225 | |
| 660 nm assay kit | Thermo Scientific | 22660 | |
| 96 well plates | Thermo Scientific | 260860 | |
| Name | Company | Catalog Number | Comments |
| Antibodies (optional) | |||
| Anti-Tetra-His | Qiagen | 34670 | primary antibody; 1:1,000 dilution |
| Anti-mouse-HRP | Sigma-Aldrich | A9044-2ML | secondary antibody, 1:10,000 dilution |
Références
- Li, S., et al. Folding-competent and folding-defective forms of ricin A chain have different fates after retrotranslocation from the endoplasmic reticulum. Mol Biol Cell. 21 (15), 2543-2554 (2010).
- Li, S., Spooner, R. A., Hampton, R. Y., Lord, J. M., Roberts, L. M. Cytosolic entry of Shiga-like toxin a chain from the yeast endoplasmic reticulum requires catalytically active Hrd1p. PLoS One. 7 (7), e41119 (2012).
- Moreau, D., et al. Genome-wide RNAi screens identify genes required for Ricin and PE intoxications. Dev Cell. 21 (2), 231-244 (2011).
- Giepmans, B. N., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312 (5771), 217-224 (2006).
- Majoul, I. V., Bastiaens, P. I., Soling, H. D. Transport of an external Lys-Asp-Glu-Leu (KDEL) protein from the plasma membrane to the endoplasmic reticulum: studies with cholera toxin in Vero cells. J Cell Biol. 133 (4), 777-789 (1996).
- Becker, B., Schnoder, T., Schmitt, M. J. Yeast Reporter Assay to Identify Cellular Components of Ricin Toxin A Chain Trafficking. Toxins (Basel). 8 (12), (2016).
- Li, X. P., Baricevic, M., Saidasan, H., Tumer, N. E. Ribosome depurination is not sufficient for ricin-mediated cell death in Saccharomyces cerevisiae. Infect Immun. 75 (1), 417-428 (2007).
- Lord, J. M., Roberts, L. M., Robertus, J. D. Ricin: structure, mode of action, and some current applications. Faseb J. 8 (2), 201-208 (1994).
- Becker, B., Schmitt, M. J. Adapting yeast as model to study ricin toxin a uptake and trafficking. Toxins (Basel). 3 (7), 834-847 (2011).
- Seidman, C. E., Struhl, K., Sheen, J., Jessen, T. Introduction of plasmid DNA into cells. Curr Protoc Mol Biol. , (2001).
- Brunelle, J. L., Green, R. Coomassie blue staining. Methods Enzymol. 541, 161-167 (2014).
- Shapiro, A. L., Vinuela, E., Maizel, J. V. Molecular weight estimation of polypeptide chains by electrophoresis in SDS-polyacrylamide gels. Biochem Biophys Res Commun. 28 (5), 815-820 (1967).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. 1979. Biotechnology. 24, 145-149 (1992).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J Bacteriol. 153 (1), 163-168 (1983).
- Vitetta, E. S., Yen, N. Expression and functional properties of genetically engineered ricin B chain lacking galactose-binding activity. Biochim Biophys Acta. 1049 (2), 151-157 (1990).
- Wales, R., Roberts, L. M., Lord, J. M. Addition of an endoplasmic reticulum retrieval sequence to ricin A chain significantly increases its cytotoxicity to mammalian cells. J Biol Chem. 268 (32), 23986-23990 (1993).
- Breslow, D. K., et al. A comprehensive strategy enabling high-resolution functional analysis of the yeast genome. Nat Methods. 5 (8), 711-718 (2008).
- Jablonowski, D., Schaffrath, R. Zymocin, a composite chitinase and tRNase killer toxin from yeast. Biochem Soc Trans. 35 (Pt 6), 1533-1537 (2007).
- Jablonowski, D., Schaffrath, R. Saccharomyces cerevisiae RNA polymerase II is affected by Kluyveromyces lactis zymocin. J Biol Chem. 277 (29), 26276-26280 (2002).
- Jablonowski, D., Frohloff, F., Fichtner, L., Stark, M. J., Schaffrath, R. Kluyveromyces lactis zymocin mode of action is linked to RNA polymerase II function via Elongator. Mol Microbiol. 42 (4), 1095-1105 (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon