È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un'analisi del Reporter di fluorescenza-basata semplice per identificare i componenti cellulari necessari per tossina ricina una catena (RTA) tratta di lievito
In questo articolo
Riepilogo
Nel manoscritto, descriviamo l'uso di un'analisi del reporter di fluorescenza basati su lievito per identificare componenti cellulari coinvolti nel traffico e uccidere i processi dei citotossici una subunità della ricina di tossina della pianta (RTA).
Abstract
Batterica e centrale un / tossine B sfruttano le naturali vie di traffico nelle cellule eucariotiche per raggiungere i loro target intracellulare nel citosol e infine uccidere. Un / tossine B consistono generalmente di un enzimaticamente attivo Asubunit (ad es., tossina ricina un (RTA)) e uno o più celle, associazione Bsubunit(s), che sono responsabili di tossina vincolante a specifici recettori di superficie di cella. La nostra attuale conoscenza di come A / B tossine sono in grado di efficientemente inebrianti cellule ha aiutate gli scienziati per comprendere i meccanismi cellulari fondamentali, come endocitosi e proteina intracellulare in cellule eucariotiche superiori. Da un punto di vista medico, allo stesso modo è importante identificare le rotte del narcotraffico tossina principali per trovare soluzioni adeguate di trattamento per i pazienti o per eventualmente sviluppare applicazioni basate su tossina terapeutiche per la terapia del cancro.
Dall'analisi del genoma della A / tossina B traffico in cellule di mammifero è complesso, lungo e costoso, parecchi studi sulla A / trasporto di tossina B sono stati effettuati nell'organismo modello lievito Saccharomyces cerevisiae. Pur essendo meno complessi, fondamentali processi cellulari in lievito e cellule eucariotiche superiori sono simile e molto spesso i risultati ottenuti in lievito possono essere trasferiti alla situazione dei mammiferi.
Qui, descriviamo un'analisi del reporter di veloce e facile da usare per analizzare il traffico intracellulare di RTA in lievito. Un vantaggio essenziale del nuovo test è la possibilità di indagare non solo RTA retrò-traslocazione dal reticolo endoplasmatico (ER) nel citosol, ma piuttosto di endocitosi e retrograda tossina trasporto dalla membrana plasmatica in ER. Il test si avvale di un plasmide del reporter che permette la misura indiretta della tossicità di RTA attraverso l'emissione di fluorescenza della proteina fluorescente verde (GFP) dopo la traduzione in vivo . Poiché RTA previene efficacemente l'inizio della biosintesi della proteina da depurinazione rRNA 28S, questo test consente l'identificazione delle proteine della cellula ospite coinvolti nel trasporto intracellulare di RTA attraverso la rilevazione dei cambiamenti nell'emissione di fluorescenza.
Introduzione
Pazienti affetti da infezioni da batteri che producono la tossina rappresentano un grave onere medico e finanziario per ogni sistema di assistenza sanitaria sociale, in particolare quanto efficaci trattamenti terapeutici sono ancora in gran parte mancante. Per sviluppare nuove strategie terapeutiche, i meccanismi complessi intossicazione della medicamente relativi A / tossine B come la tossina del colera, produttore della tossina Shiga o ricina ha bisogno di essere compresa a livello molecolare, basato sul romanzo saggi potente che devono essere implementati.
Negli ultimi anni, diversi studi ha tentato di analizzare A / trasporto di tossina B in lievito e cellule di mammifero usando metodi che richiede tempo e costosa come tossina radioattivo etichettatura1,2 , nonché a screening basato su siRNA si avvicina a3. In alcuni casi, traffico di tossina è stata visualizzata in vivo mediante microscopia a fluorescenza dopo accoppiamento chimico e/o genetico delle subunità di tossina individuali con fluorofori, punti quantici o proteine fluorescenti4,5. Purtroppo, tali modifiche spesso portano a inattivo e/o alterata proprietà naturali delle tossine. Un altro modo elegante per rispondere indirettamente a una vasta gamma di domande scientifiche è l'uso di sistemi reporter basato sugli enzimi come lacZ, luciferasi, o proteine fluorescenti (ad es. GFP o Discosoma sp. (proteina fluorescente rosso dsRed)).
In questo manoscritto, un semplice protocollo è descritto che identifica i componenti cellulari necessari per il trasporto intracellulare di RTA extracellularly applicata in S. cerevisiae. In tal modo, un plasmide di fluorescenza-reporter contenente un segnale N-terminale ER-importazione seguito da GFP agisce come un sensore di biosintesi della proteina, che misura indirettamente l'inibizione di traduzione di RTA-mediato della proteina GFP emissione di fluorescenza dopo in vivo traduzione6. Nel caso che endocitosi RTA e/o traffico intracellulare è influenzata negativamente (o positivamente) in un mutante di delezione di lievito particolare rispetto al wild type, questo può essere rilevato attraverso un aumento (o diminuzione) GFP fluorescenza emissione6.
Finora, tutti i metodi di analisi trasporto RTA in cellule di lievito sono stati limitati al processo retrò-traslocazione ER-a-citosol di RTA. In un sistema così artificiale, RTA contenente un segnale di importazione di ER è espresso da un promotore inducibile, risultante in un fenotipo suicida1,7. Anche se la cella associazione subunità B della ricina similarmente è manca nel setup sperimentale descritto in questo manoscritto e, quindi, non rappresenti perfettamente la situazione naturale di ricina holotoxin intossicazione8, tossina trasporti dal plasma membrana attraverso l'apparato del Golgi al pronto soccorso può essere imitata molto attentamente con questa analisi Novella. Interessante, i risultati preliminari ottenuti nello studio pilota indicano che le vie di traffico utilizzate da RTA rivelano sorprendenti analogie con l'itinerario di intossicazione di ricina holotoxin.
In sintesi, il metodo descritto può essere utilizzato per determinare il ruolo specifico di proteine cellulari selezionati nell'endocitosi RTA e traffico in lievito. Inoltre, questa messa a punto sperimentale potrebbe essere facilmente adattato a altri ribosoma inattivanti tossine prodotto e secreto da diverse specie batteriche come zymocin o tossina Shiga e lievito.
Protocollo
Nota: Una panoramica del flusso di lavoro sperimentale generale è raffigurata nella Figura 1.
Attenzione: RTA è altamente tossico per gli esseri umani. È necessaria l'autorizzazione del laboratorio di sicurezza S2 (livello di biosicurezza 2 equivalente). Si prega di indossare guanti durante l'intero esperimento.
1. eterologhi di sua etichetta RTA in Escherichia coli
- Trasformare le cellule di Escherichia coli con l'espressione del plasmide pET24-RTA(sua) 6 o il vettore vuoto pET24a(+) utilizzando protocolli standard elettroporazione9,10. Celle contenenti il plasmide vuoto servono come controllo negativo.
- Dopo la selezione dei cloni positivi sulle piastre di LB (100 µ g/mL) contenenti kanamicina, inoculare le cellule contenenti il plasmide di espressione di RTA o il vettore vuoto nel mezzo di 5ml LBkan (LB medium con 100 µ g/mL di kanamicina) e incubare a 37 ° C e 220 giri/min per 24 h.
- Supplemento 1 L LBkan medio con la pre-cultura 5ml e incubare le cellule a 37 ° C e 220 giri/min fino a quando le cellule hanno raggiunto un OD600 di 0.8-1.0 (circa 3-4 h). Da allora in poi, ridurre la temperatura di cultura a 28 ° C e preparare un 1m della soluzione di riserva (IPTG) isopropilico-β-D-1-thiogalactopyranoside in H2O.
- Inducono l'espressione di RTA di e. coli con l'aggiunta di IPTG ad una concentrazione finale di 1 mM.
- Dopo 3,5 h a 28 ° C e 220 giri/min, raccolto cellule mediante centrifugazione a 10.000 x g e 4 ° C per 10 minuti, lavare la pallina due volte con 5 mL di tampone di associazione (500 mM NaCl, imidazolo di 10 mM e 20 mM KH2PO4 pH = 7,4) a 10.000 x g e a 4 ° C per 10 min e risospendere il pellet in 5 mL di tampone di associazione.
Nota: Il protocollo può essere sospesa in questa fase e le cellule possono essere memorizzate a 80 ° C per diversi giorni.
2. la purificazione della sua etichetta RTA tramite cromatografia di affinità
- Sottoporre ad ultrasuoni cellule sul ghiaccio con il seguente protocollo: 15 s pulse (20 micron), 30 s pausa. Ripetere questo passaggio cinque volte.
- Centrifugare a 21.000 x g e a 4 ° C per 15 min lysate delle cellule e filtrare il surnatante utilizzando un sistema di filtri siringa sterile (dimensione dei pori di 0,2 µm).
Nota: Il pellet cellulare dei campioni lisati con successo mediante mostrano bordi trasparenti. - Utilizzare un sistema di purificazione automatizzata con 5ml Ni2 +-base colonna di affinità per purificare la frazione di His-Tag RTA dalla sterile filtrata e. coli surnatante. In generale, utilizzare una velocità di eluizione di 1 mL/min e raffreddare il sistema di purificazione di tutto per prevenire la perdita di attività di tossina e associazione tossina non efficiente.
Nota: I parametri per purificazione efficiente RTA sono elencati nella tabella 1. Vedi anche Becker et al. Per ulteriori informazioni9.- Brevemente, equilibrare la colonna di affinità con 20 mL di buffer obbligatorio rimuovere il buffer di memoria. Applicare il surnatante filtrato sterile sulla colonna di affinità usando una siringa.
- Lavare la colonna con 25-35 mL di buffer di associazione per rimuovere le proteine non associate dalla colonna. Eseguire la fase di lavaggio fino a capacità di assorbimento UV a 280 nm è vicino al valore iniziale di UV.
- Eluire la frazione legata RTA in 20-35 mL di tampone di eluizione (imidazolo di 500 mM, 500 mM NaCl, 20 mM KH2PO4, pH = 7.2) e tenere il campione sul ghiaccio (Figura 2A e 2B di figura).
Nota: Eluizione della frazione RTA è contrassegnata da un aumento nell'assorbimento di UV. Si prega di notare per raccogliere esclusivamente questa frazione per evitare la contaminazione con proteine rilegate non specifici. - Utilizzare 20 µ l di campioni eluiti per eseguire Blue di Coomassie macchiatura11 o Western blot analisi12,13 (facoltativo). Utilizzare primario anti-sua anticorpi (1:1, 000) e secondario anti-mouse-IgG-HRP (01:10, 000) per rilevare RTA e verificare la purezza del campione (Figura 3).
- Desalificare eluiti frazione RTA su una colonna di dissalazione 5ml ed equilibrare il campione in sorbitolo di 0,8 M.
- Sostituire la colonna di affinità del sistema di purificazione dalla colonna dissalazione ed equilibrare in primo luogo la colonna con 20 mL di sorbitolo di 0,8 M.
- Applicare la frazione di RTA eluita alla colonna tramite una siringa. Lavare la colonna con 100 mL di sorbitolo 0,8 M ed eluire la frazione di RTA dissalata in una provetta da 15 mL, non appena l'assorbimento di UV inizia ad aumentare (Figura 2).
- Memorizzare la frazione eluita RTA a 4 ° C.
Nota: Interrompere direttamente campionamento frazione RTA quando aumenta la conduttanza per evitare contaminazione salina. Parametri per la procedura di dissalazione sono elencati nella tabella 1.
- Eluite frazione RTA a 10.000 x g e a 4 ° C per 30-180 min utilizzando una colonna di spin 10 kDa cut-off ad un volume finale di 1-2 mL di concentrato e conservare il campione a 4 ° C per 3-4 settimane.
Attenzione: Non congelare il campione dal congelamento conduce ad una perdita completa di attività di RTA. - Calcolare la concentrazione di proteina mediante un kit di determinazione della proteina convenzionali. Concentrazione nella proteina dovrebbe essere nel range di 1-1.5 mg/mL.
Nota: RTA tende a precipitare se la concentrazione nella proteina è troppo alta (> 5 mg/mL).
3. trasformazione e rimozione della parete cellulare del lievito
- Trasformare mutanti di delezione di lievito wild-type o selezionato con la GFP reporter plasmide pRS315-K28SP- GFP6 utilizzando standard litio acetato trasformazione metodi14. Incubare le cellule su leucina abbandono (d/o) piastre di glucosio (glucosio 2%, 1,5% agar, 0,5% di solfato di ammonio, 0,17% lievito azoto Base (YNB) e 0,087% d/o mix senza leucina) a 30 ° C per 2-3 giorni per selezione clonale positivo.
- Scegli 3 cloni di lievito diverso da ogni piatto e inoculare i cloni in 100 mL di leucina d/o raffinosio medio (2% raffinosio, 0,5% di solfato di ammonio, 0,17% YNB e mescolare senza leucina 0,087% dallavalle) a 220 giri/min e 30 ° C a OD600 = 1.0-2.0 (2-4 x 107 cellule/mL).
Nota: La crescita delle cellule i diverso l'eliminazione di ceppi di lievito è diverso. Monitor OD600 tramite uno spettrofotometro. - Per calcolare i valori di OD600 , diluire i campioni a OD600 = 0.1-0.3 (1:5 a 01:10 diluizioni) e misurare OD600 con uno spettrofotometro. Come riferimento, utilizzare H2O completato con la corrispondente quantità di leucina d/o raffionose mezzo di.
- Dopo finitura passo 3.5, mescolare 4 µ l della cultura spheroplasted 50ml con 496 µ l di tampone di spheroplasting (circa 2 × 106 Sferoplasti) e centrifugare per 10 min a 400 x g.
- Risospendere il pellet in 10 mL di H2O distillata, campione di vortice per 30 s e piastra fuori 10 µ l del campione su piastre di glucosio di d/o di leucina (2% di glucosio, agar 1,5%, 0,5% di solfato di ammonio, 0,17% YNB e 0,087% d/o mix senza leucina).
- Incubare le cellule per 3 giorni a 30 ° C. Contare il numero totale delle colonie delle cellule coltivate sulla piastra. Per la valutazione dei dati, utilizzare solo i campioni con efficienza supera al 98% (totale numero di Colonia di cellule < 40 colonie/piastra).
4. GFP Reporter Assay misura in piastre da 96 pozzetti
- Seme fuori gli sferoplasti di cellule di lievito ottenute al passaggio 3,7 in piastre da 96 pozzetti (200 µ l/pozzetto).
- Aggiungere 70 µ l leucina stabilizzato d/o raffinosio terreno contenente il controllo negativo (eluato di una Ni2 +-affinità purificato lysate delle cellule dalle cellule di IPTG-indotta e. coli che esprimono il vettore vuoto) o purificata RTA a una concentrazione finale di RTA di 5 µM ( corrispondenti a 160 g/L RTA) in ciascun pozzetto.
- Immediatamente aggiungere 30 µ l stabilizzato galattosio soluzione (30% galattosio, sorbitolo 0,8 M) per indurre l'espressione di GFP e successivamente avviare la misurazione.
Nota: Eseguire la tecniche almeno 3 ripetizioni per esperimento e 3 biologici replica al ceppo di lievito. - Dopo aver completato la preparazione del campione (punti 4.1-4.3), mettere la piastra a 96 pozzetti in un lettore di fluorescenza e avvia la misurazione. Utilizzare il 475/509 nm set di filtri necessari per la rilevazione della fluorescenza di GFP. Eseguire misurazioni a 30 ° C, 120 giri/min e con un diametro d'agitazione di 1 mm sopra una finestra di tempo di 20 h (gli intervalli di 10 min di misurazione).
Nota: Il set di filtri GFP è normalmente disponibile in tutti i sistemi di lettore. Temperatura, intervallo di tempo, misurare intervalli e RTA concentrazione può essere regolata per le proprie esigenze. Risultati rappresentativi sono mostrati in Figura 4. - Facoltativo: Utilizzare ulteriori controlli interni nella misura per il controllo qualità. Preparare un controllo negativo aggiungendo 30 µ l leucina stabilizzato d/o raffinosio medium invece 30% galattosio (nessuna induzione di GFP). Inoltre, aggiungere 70 µ l di soluzione di 0,8 M sorbitolo stabilizzato G418 (300 µ g/mL). L'inibitore di traduzione proteica G418 serve come controllo positivo per l'inibizione della proteina impedisce espressione di GFP in lievito.
- Calcolare in per cento per il punto di tempo di 20 h secondo la seguente equazione relativa fluorescenza GFP (Vedi Figura 4A):
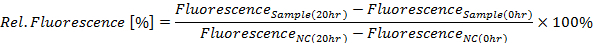 dove NC è il controllo negativo
dove NC è il controllo negativo
- In alternativa, determinare la fluorescenza di GFP relativa per ciascun punto di misura (in questo caso 10 min, vedi anche punto 4.4) utilizzando l'equazione di cui sopra. Come mostrato in Figura 4B, è possibile creare un grafico di macchiare le intensità di fluorescenza di GFP (asse y) nel corso del tempo (asse x).
Risultati
Il flusso di lavoro generale del protocollo descritto in questo manoscritto è illustrata nella Figura 1, approssimativamente che riassume i singoli passaggi per successo RTA purificazione e il successivo esperimento di analisi del reporter GFP. Una descrizione più dettagliata di ogni singolo passaggio può essere trovata nel protocollo. La figura 2 illustra il risultato previsto di una purificazione di RTA successo da cromatogr...
Discussione
Quando si esegue il protocollo di cui sopra, si consiglia i seguenti suggerimenti per ottenere un risultato di successo dell'esperimento.
Per l'espressione della proteina eterologa, è importante non superare la concentrazione di IPTG di 1 mM. Concentrazioni di IPTG > 1 mM inibire indotta da promotore espressione di RTA e piombo per abbassare i rendimenti di tossina. Inoltre, le cellule non vanno coltivate a temperature superiori a 28 ° C per impedire la formazione del corpo di inclusione...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Parti di questo studio sono state gentilmente sostenuta da una sovvenzione del Deutsche Forschungsgemeinschaft (SFB 1027, A6).
Materiali
| Name | Company | Catalog Number | Comments |
| Bacterial and yeast strains | |||
| E. coli BL21 DE3(Gold) | Aligent Technologies | 230130 | |
| S. cerevisiae BY4742 | Euroscarf | Y10000 | |
| S. cerevisiae BY4742 deletion mutants | Dharmacon | YSC1054 | whole collection |
| Name | Company | Catalog Number | Comments |
| Plasmids used in this protocol | |||
| pET24a(+) (Novagen) | Millipore | 69772-3 | |
| pET-RTA(His6) | Becker et al. (2016)3 | ||
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Zymolyase 20T | USBio | Z1000.250 | lytic enzyme for cell wall removal |
| LB broth medium | Thermo Scientific | 10855021 | 15 g agar was added for plate production |
| YNB | Thermo Scientific | DF0335-15-9 | |
| Ammonium sulfate | Sigma-Aldrich | A4418-100G | |
| Yeast drop-out mix supplemts without leucine | Sigma-Aldrich | Y1376-20G | |
| Agar | Sigma-Aldrich | 05040-100G | |
| D-glucose | Sigma-Aldrich | G8270-100G | |
| DTT | Sigma-Aldrich | 10197777001 | |
| D-raffinose | Sigma-Aldrich | 83400-25G | |
| D-sorbitol | Sigma-Aldrich | S1876-1KG | |
| D-galactose | Sigma-Aldrich | G0750-10MG | |
| G418 | Thermo Scientific | 11811031 | |
| IPTG | Sigma-Aldrich | I6758-1G | |
| Imidazole | Roth | 3899.1 | |
| PAGE ruler prestained | Fermentas | 26616 | protein ladder used for Western analysis |
| Name | Company | Catalog Number | Comments |
| Material for RTA purification, desalting and reader measurements | |||
| Spectrophotometer Ultrospec 2100 pro | Amersham | ||
| Soniprep 150 | MSE | old model, other models available | |
| Fluoroskan Ascent | Thermo Scientific | 5210470 | old model, not available anymore |
| ÄKTAPurifier | Thermo Scientific | 28406266 | Product is discontinued and replaced |
| HisTRAP HP column | GE Healthcare | 17-5248-02 | |
| HiTRAP desalting column | GE Healthcare | 11-0003-29 | |
| Midisart sterile filter | Sartorius | 16534K | 0.2 µm pore size |
| BCA protein assay kit | Pierce | 23225 | |
| 660 nm assay kit | Thermo Scientific | 22660 | |
| 96 well plates | Thermo Scientific | 260860 | |
| Name | Company | Catalog Number | Comments |
| Antibodies (optional) | |||
| Anti-Tetra-His | Qiagen | 34670 | primary antibody; 1:1,000 dilution |
| Anti-mouse-HRP | Sigma-Aldrich | A9044-2ML | secondary antibody, 1:10,000 dilution |
Riferimenti
- Li, S., et al. Folding-competent and folding-defective forms of ricin A chain have different fates after retrotranslocation from the endoplasmic reticulum. Mol Biol Cell. 21 (15), 2543-2554 (2010).
- Li, S., Spooner, R. A., Hampton, R. Y., Lord, J. M., Roberts, L. M. Cytosolic entry of Shiga-like toxin a chain from the yeast endoplasmic reticulum requires catalytically active Hrd1p. PLoS One. 7 (7), e41119 (2012).
- Moreau, D., et al. Genome-wide RNAi screens identify genes required for Ricin and PE intoxications. Dev Cell. 21 (2), 231-244 (2011).
- Giepmans, B. N., Adams, S. R., Ellisman, M. H., Tsien, R. Y. The fluorescent toolbox for assessing protein location and function. Science. 312 (5771), 217-224 (2006).
- Majoul, I. V., Bastiaens, P. I., Soling, H. D. Transport of an external Lys-Asp-Glu-Leu (KDEL) protein from the plasma membrane to the endoplasmic reticulum: studies with cholera toxin in Vero cells. J Cell Biol. 133 (4), 777-789 (1996).
- Becker, B., Schnoder, T., Schmitt, M. J. Yeast Reporter Assay to Identify Cellular Components of Ricin Toxin A Chain Trafficking. Toxins (Basel). 8 (12), (2016).
- Li, X. P., Baricevic, M., Saidasan, H., Tumer, N. E. Ribosome depurination is not sufficient for ricin-mediated cell death in Saccharomyces cerevisiae. Infect Immun. 75 (1), 417-428 (2007).
- Lord, J. M., Roberts, L. M., Robertus, J. D. Ricin: structure, mode of action, and some current applications. Faseb J. 8 (2), 201-208 (1994).
- Becker, B., Schmitt, M. J. Adapting yeast as model to study ricin toxin a uptake and trafficking. Toxins (Basel). 3 (7), 834-847 (2011).
- Seidman, C. E., Struhl, K., Sheen, J., Jessen, T. Introduction of plasmid DNA into cells. Curr Protoc Mol Biol. , (2001).
- Brunelle, J. L., Green, R. Coomassie blue staining. Methods Enzymol. 541, 161-167 (2014).
- Shapiro, A. L., Vinuela, E., Maizel, J. V. Molecular weight estimation of polypeptide chains by electrophoresis in SDS-polyacrylamide gels. Biochem Biophys Res Commun. 28 (5), 815-820 (1967).
- Towbin, H., Staehelin, T., Gordon, J. Electrophoretic transfer of proteins from polyacrylamide gels to nitrocellulose sheets: procedure and some applications. 1979. Biotechnology. 24, 145-149 (1992).
- Ito, H., Fukuda, Y., Murata, K., Kimura, A. Transformation of intact yeast cells treated with alkali cations. J Bacteriol. 153 (1), 163-168 (1983).
- Vitetta, E. S., Yen, N. Expression and functional properties of genetically engineered ricin B chain lacking galactose-binding activity. Biochim Biophys Acta. 1049 (2), 151-157 (1990).
- Wales, R., Roberts, L. M., Lord, J. M. Addition of an endoplasmic reticulum retrieval sequence to ricin A chain significantly increases its cytotoxicity to mammalian cells. J Biol Chem. 268 (32), 23986-23990 (1993).
- Breslow, D. K., et al. A comprehensive strategy enabling high-resolution functional analysis of the yeast genome. Nat Methods. 5 (8), 711-718 (2008).
- Jablonowski, D., Schaffrath, R. Zymocin, a composite chitinase and tRNase killer toxin from yeast. Biochem Soc Trans. 35 (Pt 6), 1533-1537 (2007).
- Jablonowski, D., Schaffrath, R. Saccharomyces cerevisiae RNA polymerase II is affected by Kluyveromyces lactis zymocin. J Biol Chem. 277 (29), 26276-26280 (2002).
- Jablonowski, D., Frohloff, F., Fichtner, L., Stark, M. J., Schaffrath, R. Kluyveromyces lactis zymocin mode of action is linked to RNA polymerase II function via Elongator. Mol Microbiol. 42 (4), 1095-1105 (2001).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon