Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Microscopie spectrale réflectométrique sur les axones myélinisés In Situ
Dans cet article
Résumé
Nous présentons ici un protocole étape par étape pour l’imagerie des axones myélinisés dans une tranche de cerveau fixe à l’aide d’une échelle nanométrique exempte d’étiquette d’imagerie technique basée sur la réflectométrie spectrale.
Résumé
Dans un système nerveux des mammifères, la myéline fournit une isolation électrique par Envellopants les fibres axonales dans une spirale de multicouche. Inspiré par son architecture sous-cellulaire très organisés, nous avons récemment développé une nouvelle modalité d’imagerie, nommée réflectométrie spectrale (diffuseurs), qui permet l’imagerie nanométriques de label-free sans précédent de l' axones myélinisés live in situ. Le principe sous-jacent est d’obtenir des informations de la nanostructure en analysant le spectre de réflectance de la structure multicouche subcellulaire. Dans cet article, nous décrivons un protocole détaillé étape par étape pour réaliser une imagerie de diffuseurs de base des tissus nerveux à l’aide d’un système microscopique confocal commercial, équipé d’un laser de lumière blanche et d’un filtre accordable. Nous couvrons les procédures de préparation des échantillons, acquisition de données spectrales et traitement d’image pour obtenir des informations de nanostructure.
Introduction
Dans le système nerveux des mammifères, la myéline fournit la conduction nerveuse rapide et intégrité axonale par Envellopants les fibres axonales avec gaines membraneuses multicouches. Sa structure multicouche est composé d’une alternance de minces nanométriques composées de membranes plasmiques (~ 5 nm), cytosol (environ 3 nm) et les espaces extracellulaires (~ 7 nm)1,2. La microscopie optique, y compris la microscopie de Super-résolution récente, ne conviennent pas pour l’observation de la dynamique de la myéline échelle nanométrique en raison de leur résolution insuffisante en raison de la diffraction optique3,4,5. Bien que la microscopie électronique peut fournir des détails de la nanostructure de myéline, ce n’est pas compatible avec les systèmes biologiques vivants en raison des préparations très envahissante impliquant la fixation chimique et ultrasectioning6,7 . Jusqu'à tout récemment, il n’y a eu aucune technique il y a lieu d’observer nanométriques dynamique des axones myélinisés in situ.
Schain et coll. a déjà signalé que les axones myélinisés présentent8de la réflexion de la lumière colorée. En adoptant l’analyse spectroscopique de la lumière réfléchie, nous avons mis au point une nouvelle modalité d’imagerie pour l’imagerie nanométrique des axones myélinisés, nommé réflectométrie spectrale (diffuseurs)9. Diffuseurs repose sur l’ingérence de minces qui se produisent dans la structure multicouche de la gaine de myéline (Figure 1). Par simulation optique sur les axones différents, nous avons révélé que le spectre de réflectance est une fonction périodique de nombre d’onde et sa périodicité (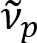 ) est inversement proportionnelle au diamètre de l’axone (d). Cette relation simple (
) est inversement proportionnelle au diamètre de l’axone (d). Cette relation simple ( ) propose une quantification du diamètre de l’axone d’après les données de diffuseurs. Grâce à cela, nous avons révélé l’axone répandue renflement sous léger traumatisme crânien dans notre rapport précédent.
) propose une quantification du diamètre de l’axone d’après les données de diffuseurs. Grâce à cela, nous avons révélé l’axone répandue renflement sous léger traumatisme crânien dans notre rapport précédent.
Le système de diffuseurs repose sur la microscopie confocale et se compose d’une source laser spécialisés et filtre (Figure 2). La source d’entrée est un laser de lumière blanche, fournissant une sortie à large bande spectrale visible à l’infrarouge. Pour l’analyse spectrale, le système est muni de deux dispositifs acousto-optiques : un filtre accordable acousto-optique (AOTF) pour fournir une longueur d’onde sélectionnée de la source à large bande d’entrée et un séparateur de faisceau d’acousto-optique (AOBS) pour guider les sélectionnés reflétés longueur d’onde du détecteur. Le logiciel pour la microscopie confocal hyperspectrales (voir Table des matières) fournit une option de balayage spectral personnalisable d’acquérir dans l’ordre les images de réflectance à différentes longueurs d’onde d’entrée. Par ailleurs, l’aberration chromatique peut gravement interférer dans la mesure spectrale ; par conséquent, l’utilisation d’une lentille d’objectif apochromat est recommandée.
À noter, lumière blanche lasers produisent une sortie spectrale inégale et les composants optiques affectent également le profil spectral. Par conséquent, les spectres acquis doivent être calibrées pour l’analyse quantitative ultérieur. Un miroir argent protégé est généralement utilisé comme référence, qui offre une réflexion presque constante (> 97 %) au-dessus de la région complètement visible. Les spectres acquis sont ensuite divisés par les spectres de référence par le miroir.
La taille de palier spectrale pour l’analyse spectrale détermine la vitesse d’acquisition ; ainsi, il doit être optimisé. Comme un axone plus grande et dispose d’un délai spectral plus élevé, il faut plus fine de l’échantillonnage spectral. Par exemple, un axone d’un diamètre de 10 µm, une des axones physiologiques plus grands, dispose d’un délai spectral de ~ 8 nm. En appliquant les critères d’échantillonnage de Nyquist, nous avons utilisé l’intervalle d’échantillonnage spectral de 4 nm pour couvrir tous les axones physiologiques dans les tissus nerveux de la souris. Cette approche prend généralement plus de quelques secondes pour un scan complet spectral et donc n’est pas adaptée pour des applications in vivo , où le mouvement physiologique (p. ex. respiration et rythme cardiaque) s’ingère acquisition spectrale stable. Précédemment, nous avons résolu ce problème en instrumentant un microscope droit sur mesure, conçu pour acquérir le spectre complet pour chaque point à l’aide d’un spectromètre de tableau (acquisition Vitesse ≈ 30 ms par pixel).
Dans ce rapport, nous décrivons un protocole détaillé sur l’imagerie de diffuseurs sur une tranche fixe de cerveau, qui peut être effectuée dans un microscope hyperspectral commerciale (voir Table des matières). Ainsi, le protocole peut être complété par les expérimentateurs sans expertise en instrumentation optique. Nous couvrons également les problèmes potentiels et dépannage pour l’acquisition et l’analyse des données de diffuseurs.
Protocole
Toutes les interventions chirurgicales ont été approuvées par l’animalier institutionnel et utilisation Comité (IACUC) de l’Université Sungkyunkwan.
1. préparation de l’échantillon
Remarque : Stériliser tous les instruments chirurgicaux avant toute manipulation animale. Effectuer toutes les performances chirurgicales dans une salle dédiée aux interventions chirurgicales. Gants et blouses stériles doivent être portées par tout le personnel dans la salle de chirurgie en permanence.
-
Fixation du tissu
- Préparer les deux seringues de 10 mL que chacun rempli de solution saline tamponnée au phosphate (PBS) et 4 % de paraformaldéhyde (PFA) dans du PBS.
- Anesthésier une souris âgée de 7-12 semaine, (C57BL/6J ou des lignées de souris ou l’autre sexe) par l’administration d’un mélange de zoletil et de xylazine (1:1, 20 mg/kg chacun) ou d’un régime alternatif anesthésie appropriée (p. ex., mélange de kétamine 80 mg/kg et 10 mg/kg xylazine) avec une injection intrapéritonéale.
- Lorsque la souris atteint un plan chirurgical de l’anesthésie (utilisez la méthode de pincement ou de non-réponse orteil), exposer le cœur en coupant de la peau et la cage thoracique avec des ciseaux.
- Découper l’oreillette droite avec petits ciseaux pour drainer le sang et perfuse la solution PBS et PFA séquentiellement dans le ventricule gauche avec une aiguille (taux de perfusion = 4 mL/min, volume = 10 mL de chaque solution).
Remarque : La souris se raide si la procédure est menée à bien. L’utilisation de pommade ophtalmique ou de stérilisation n’est pas nécessaire car la procédure est en phase terminale. La procédure de fixation peut-être être ignorée. Toutefois, cette étape est recommandée afin de minimiser les déformations structurelles, y compris les dommages de tissu mécanique et un gonflement axonal ischémique. Pour plus de détails sur la fixation du tissu, reportez-vous à l’article par Gage, G. J. et al. 10
-
Préparation de la tranche de cerveau
- Décapiter la souris avec des ciseaux chirurgicaux à travers le ventre thorax et l’abdomen.
- Enlever le cuir chevelu et le périoste en utilisant une paire de ciseaux convenablement petite jusqu'à exposer entièrement le crâne.
- Briser l’os frontal et couper le crâne le long de la suture sagittale de l’os occipital, à l’aide de petits ciseaux avec soin ne pas d’endommager le cerveau.
- Retirez doucement le crâne et la dure-mère successivement à l’aide de pinces fines.
- Extraire le cerveau à l’aide d’une cuillère en plastique souple, en prenant soin de ne pas pour endommager les tissus.
- Rincer et faire tremper le cerveau extrait dans une solution PFA de 4 % pendant 30 min à 4 ° C.
- Tranchez le cerveau fixe en 100 – 150 µm sections à l’aide d’un vibratome.
Remarque : Pour plus de détails sur la préparation du tissu, reportez-vous à l’article de Segev et al. 11. pour les procédures suivantes, le tissu doit être découpé en sections plus mince que l’épaisseur de la cale d’espacement.
-
Montage de la tranche de cerveau
- Préparer la lame de 1 verre et 2 verres à couvercle carrés (22 × 22 × 0,17 mm) pour chaque tranche de tissu.
- Un des verres couvercle carrés coupé en moitié (deux morceaux rectangulaires) à l’aide d’un coupe-verre.
- Fixez les deux des lunettes couverture coupées en deux à l’aide d’une colle super sur la vitre de la diapositive.
Remarque : L’espace entre les deux moitiés verres devrait être légèrement supérieure à la taille de la tranche de tissu. - Récolter la tranche de cerveau à l’aide d’une brosse ou une pipette et poser le tissu sur le verre de glissement entre l’entretoise. Prendre soin de ne pas pour plier le tissu.
- Déposer 100 μl de PBS sur la surface du tissu.
- Placez le couvercle en autre carré verre sur le dessus du tissu. Éviter l’inclusion de bulles d’air lors de cette étape.
- Joint autour du verre de couverture avec un vernis à ongles (ou adhésifs alternativement appropriés) pour empêcher évaporation de PBS et de contamination par la poussière lors de la session d’imagerie ultérieure.
Remarque : L’expérimentateur peut faire une pause à ce stade.
2. étalonnage
- Allumer le microscope au moins 1 h avant l’imagerie pour permettre la stabilisation thermique du laser source (voir la Table des matières). Ensuite, activer le logiciel pour l’analyse spectrale (voir Table des matières) et cliquez sur le bouton acquérir sur le dessus de GUI (Figure 3 a).
- Allumez l’obturateur de logiciel pour le laser de lumière blanche (WLL) et un tube photomultiplicateur (PMT) (Figure 3 a).
- Sélectionnez le mode de xyΛ sur la liste déroulante en Mode d’Acquisition, puis, le mode d’entrée du laser est automatiquement changé de Constant pour cent à Puissance constante. Décochez la case à cocher du Mouvement à remontage automatique SP. Puis, affectez la fenêtre spectrale du laser d’entrée 470 – 670 nm et la taille de palier spectrale à 4 nm sur Λ-Excitation Lambda Scan paramètres (Figure 3 b).
- DÉFINIR la gamme spectrale de PMT à 450-690 en double-cliquant ou en déplaçant la barre de réglage pour la gamme spectrale (Figure 3C).
Remarque : Cette gamme spectrale devrait inclure la largeur de bande complète de la source d’entrée. - Sélectionnez un objectif immersion dans l’eau, convenablement avec une grande ouverture numérique (NA > 0,7) et faire n’importe quelle valeur pouvoir PMT et laser pour activer le bouton configuration AOBS et Live Scan . Passer le chemin optique en vérifiant la réflexion dans la Configuration AOBS (Figure 3d).
- Monter un miroir de renvoi (voir Table des matières) sur la platine du microscope. La surface du miroir doit être face contre l’objectif. S’il n’est pas facile de mettre le miroir sur la platine du microscope, coller un miroir sur la plaque plate (p. ex. verre de diapositive).
- Contrôler la platine du microscope pour aligner le plan focal à la surface du miroir.
- Ajuster le gain de la PMT et la puissance du laser compte tenu de la gamme dynamique du détecteur et puis changer le mode de saisie de laser de Constant pour cent à Puissance constante.
Remarque : Comme il est typique, un PMT gain de 500 (V) et une puissance laser relative de 0,1 % servent à 570 nm. - Confirmer en mode pseudo-couleur pour vérifier qu’il n’y a aucune saturation sur toute la gamme de longueur d’onde. Si la saturation est observée, baisser la puissance du laser (Figure 3 a).
- Exécutez l’acquisition Lambda Scan .
- Retirer le miroir de la scène et répétez l’acquisition même sans un échantillon afin d’obtenir la référence sombre (par exemple, décalage sombre).
- Enregistrer les données dans un format TIF de base .
3. Acquisition d’images de diffuseurs
- Placez le tissu monté sur la platine du microscope. Pour à peu près aligner les tissus et le plan focal de l’objectif, utilisez le mode grand-angulaire fluorescence par un oculaire.
- Avec l’option Live Scan sur contrôler la platine du microscope pour aligner le plan focal de la région d’intérêt dans le tissu. Pour éviter le bruit de fond de la lamelle couvre-objet, sélectionnez la région cible d’au moins 15 μm profondeur dans l’interface de verre-tissu.
- Acquérir la pile d’image spectrale pour la région de cible en utilisant la même procédure comme décrit aux points 2.1 à 2.10.
- Enregistrer les données pour le tissu et le décalage sombre en format TIF de base .
Remarque : L’expérimentateur peut faire une pause à ce stade.
4. traitement et analyse d’images
- Ouvrez les données spectrales (base TIF) pour le miroir de renvoi et le tissu cérébral dans ImageJ.
- Sélectionnez la ROIs (régions d’intérêt) pour les piles d’image ouverte — la zone centrale pour le miroir de renvoi et le segment d’une fibre de l’axone pour le tissu cérébral.
- Acquérir les spectres brutes pour la ROIs sélectionnée par l’Image en cours d’exécution, piles → → axe z à tracer le profil dans le menu ImageJ.
- Ouvrir les données décalage sombres, une prise pour le miroir de renvoi et les autres prises pour le tissu cérébral.
- Acquérir les spectres pour les offsets sombres par l’Image en cours d’exécution, piles → → axe z à tracer le profil dans le menu ImageJ.
- Enregistrez tous les spectres acquis en utilisant les options de copier / coller .
Remarque : Pour l’imagerie de diffuseurs, de centre d’imagerie du champ afin de minimiser les aberrations optiques hors-axe est recommandé. Les fibres de l’axone peuvent être structurellement hétérogènes sur toute leur longueur. Par conséquent, en sélectionnant le retour sur investissement sur un segment de petit axon, typiquement < 5 µm, pour minimiser l’artefact de volume partiel est recommandé.
5. données initiales Correction et analyse des signaux de diffuseurs
- Soustraire les décalage spectres des spectres du miroir de renvoi et les tissus cérébraux.
- Normaliser chaque spectre en divisant l’intensité maximale du spectre.
- Diviser le spectre normalisé de l’axone par le spectre normalisé du miroir référence et soustraire le décalage CC parmi les fréquences normalisées.
- Mesurer la fréquence de l’onde en ajustant le spectre acquis à une sinusoïdale courbe (voir la Table des matières) et puis convertir le nombre d’onde acquise à périodicité par prise réciproque.
- Convertir le diamètre de l’axone à l’aide de l’équation dans la Figure 4 cde la périodicité de nombre d’onde.
Résultats
Selon le protocole, une tranche fixe de cerveau a été préparée avec la coloration exogène, un fluorophore de ciblage de la myéline (voir Table des matières). Diffuseurs imagerie a été réalisée sur la tranche du cerveau en utilisant un microscope confocal hyperspectral commercial conjointement avec confocal fluorescence imaging (Figure 4 a). Pour diffuseurs, intensité optique d’entrée a été fixée à 5 µW/µm2 avec...
Discussion
Diffuseurs est une nouvelle modalité d’imagerie exempte d’étiquette basée sur l’interférométrie spectrale, qui pour la première fois, offre l’information de l’échelle nanométrique dans les axones myélinisés direct. Dans le protocole actuel de l’acquisition, la résolution spatiale pour le diamètre de l’axone est de l’ordre de 10 nm. En outre, diffuseurs utilise des ordres de grandeur plus faible dose légère par rapport aux autres microscopies de Super-résolution ; Il est donc exempt de phot...
Déclarations de divulgation
Les auteurs déclarent des intérêts financiers divergents : Kwon J. et M. Choi sont les inventeurs de la technologie en instance de brevet décrit dans cet Article.
Remerciements
Ce travail a été soutenu par l’Institut de sciences fondamentales (IBS-R015-D1) et de base programme de recherche scientifique grâce à la Fondation de la recherche nationale de Corée (NRF) financé par le ministère de l’éducation (2017R1A6A1A03015642).
matériels
| Name | Company | Catalog Number | Comments |
| Glass cutter | - | - | Can be purchased in a local convenience store or online stores. |
| Nail polish | - | - | Can be purchased in a local convenience store or online stores. |
| Apochromat objective 40×, NA 1.1 | Leica Microsystems | 15506357 | Water-immersion type |
| Fluoromyelin Green | Thermo Fisher | F34651 | Alternatively, Fluoromyelin Red (F34652) can be used. |
| Leica SP8 TCS microscope | Leica Microsystems | SP8 | Refer to the "Configuration of microscope" in Introduction Section for details. |
| Imaging software | Leica Microsystems | LAS-X | - |
| Matlab | MathWorks | - | - |
| Mirror | Thorlabs | PF10-03-P01 | Coated with protected silver. |
| Phosphate-buffered saline (PBS) | Life technologies | 14190-136 | - |
| Paraformaldehyde | Biosolution | BP031a | 4% v/v in PBS |
| Cover slip | Thermo Fisher | 3306 | Thickness: #1 (0.13 to 0.17 mm) |
| Slide glass | Muto Pure Chemicals | 5116-20F | Thickness: ~1 mm |
| Super glue | Henkel | Loctite 406 | Use a dispensing equipment to avoid skin or eye contact. |
| Syringe pump | Brainetree Scientific | BS-8000 DUAL | - |
| Vibratome | Leica Biosystems | VT1200S | - |
| White-light laser | NKT photonics | EXB-6 | EXB-6 was discontinued and replaced by EXU-6. |
Références
- Fernandez-Moran, H., Finean, J. B. Electron microscope and low-angle x-ray diffraction studies of the nerve myelin sheath. Journal of Cell Biology. 3, (1957).
- Blaurock, A. E. The spaces between membrane bilayers within PNS myelin as characterized by X-ray diffraction. Brain Research. 210, 383-387 (1981).
- Shim, S. -. H., et al. Super-resolution fluorescence imaging of organelles in live cells with photoswitchable membrane probes. Proceedings of the National Academy of Sciences. , 13978-13983 (2012).
- Urban, N. T., Willig, K. I., Hell, S. W., Nägerl, U. V. STED nanoscopy of actin dynamics in synapses deep inside living brain slices. Biophysics Journal. 101, 1277-1284 (2011).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nature Methods. 5, 155-157 (2008).
- Peters, A., Sethares, C. Is there remyelination during aging of the primate central nervous system?. Journal of Comparative Neurology. 460, 238-254 (2003).
- De Campos Vidal, B., Silveira Mello, M. L., Caseiro-Filho, A. C., Godo, C. Anisotropic properties of the myelin sheath. Acta Histochemica. 66, 32-39 (1980).
- Schain, A. J., Hill, R. A., Grutzendler, J. Label-free in vivo imaging of myelinated axons in health and disease with spectral confocal reflectance microscopy. Nature Medicine. 20, 443-449 (2014).
- Kwon, J., et al. Label-free nanoscale optical metrology on myelinated axons in vivo. Nature Communication. 8, 1832 (2017).
- Gage, G. J., Kipke, D. R., Shain, W. Whole Animal Perfusion Fixation for Rodents. JoVE. (65), e3564 (2012).
- Segev, A., Garcia-Oscos, F., Kourrich, S. Whole-cell Patch-clamp Recordings in Brain Slices. JoVE. (112), e54024 (2016).
- Waldchen, S., Lehmann, J., Klein, T., Van De Linde, S., Sauer, M. Light-induced cell damage in live-cell super-resolution microscopy. Scientific Reports. 5, 15348 (2015).
- Chang, J. B., et al. Iterative expansion microscopy. Nature Methods. 14, 593-599 (2017).
- Polishchuk, R. S., et al. Correlative light-electron microscopy reveals the tubular-saccular ultrastructure of carriers operating between Golgi apparatus and plasma membrane. Journal of Cell Biology. 148, 45-58 (2000).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon