È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Microscopia spettrale riflettometrica su assoni Myelinated In Situ
In questo articolo
Riepilogo
Qui, presentiamo un protocollo passo-passo per l'imaging di assoni myelinated in una fetta di cervello fisso utilizzando una scala nanometrica privo di etichetta tecnica basato su riflettometria spettrale di imaging.
Abstract
In un sistema nervoso dei mammiferi, mielina fornisce un isolamento elettrico da avviluppando le fibre dell'assone in una spirale multistrata. Ispirato dalla sua architettura subcellulare altamente organizzato, abbiamo recentemente sviluppato una nuova modalità di imaging, denominata riflettometria spettrale (isotropi), che permette l'imaging senza precedenti su scala nanometrica privo di etichetta degli assoni myelinated live in situ. Il principio di fondo è quello di ottenere informazioni nanostrutturale analizzando lo spettro di riflettanza della struttura subcellulare multistrato. In questo articolo, descriviamo un protocollo dettagliato passo-passo per l'esecuzione di un imaging di SpeRe base dei tessuti nervosi utilizzando un sistema al microscopio confocale commerciale, dotato di un laser a luce bianca e un filtro sintonizzabile. Copriamo le procedure di preparazione del campione, acquisizione di dati spettrali e di elaborazione delle immagini per ottenere informazioni nanostrutturale.
Introduzione
Nel sistema nervoso dei mammiferi, mielina fornisce integrità assonale e conduzione nervosa rapida avviluppando le fibre dell'assone con guaine membranose multistrate. La sua struttura multistrato è composto da alternando film sottili su scala nanometrica composto da membrane plasmatiche (~ 5 nm), cytosol (~ 3 nm) e spazi extracellulari (7 ~ nm)1,2. Microscopia ottica, compreso il recente microscopia di Super-risoluzione, non sono adatti per l'osservazione delle dinamiche di mielina su scala nanometrica a causa della loro risoluzione insufficiente a causa della diffrazione ottica3,4,5. Sebbene la microscopia elettronica può fornire dettagli della nanostruttura mielina, non è compatibile con i sistemi biologici viventi a causa della preparazione del campione altamente invasive che coinvolgono la fissazione chimica e ultrasectioning6,7 . Fino a poco tempo, c' non è stata nessuna tecnica applicabile per osservare la dinamica su scala nanometrica di assoni myelinated in situ.
Schain et al ha segnalato in precedenza che gli assoni myelinated esibiscono la riflessione della luce colorata8. Adottando l'analisi spettroscopica sulla luce riflessa, abbiamo messo a punto una nuova modalità di imaging per l'imaging su scala nanometrica di assoni myelinated, denominata riflettometria spettrale (isotropi)9. Bullonerie si basa sull'interferenza di film sottile che si verificano nella struttura multistrata della guaina mielinica (Figura 1). Di simulazione ottica su vari assoni, abbiamo rivelato che lo spettro di riflettanza è una funzione periodica del numero d'onda e la sua periodicità (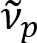 ) è inversamente proporzionale al diametro dell'assone (d). Questa semplice relazione (
) è inversamente proporzionale al diametro dell'assone (d). Questa semplice relazione ( ) offre facile quantificazione del diametro dell'assone dai dati isotropi. Utilizzando questo, abbiamo rivelato l'assone prevalente sporgenti sotto la ferita traumatica delicata del cervello nella nostra precedente relazione.
) offre facile quantificazione del diametro dell'assone dai dati isotropi. Utilizzando questo, abbiamo rivelato l'assone prevalente sporgenti sotto la ferita traumatica delicata del cervello nella nostra precedente relazione.
Il sistema di SpeRe si basa su microscopia confocal e consiste di una sorgente laser specializzate e filtri (Figura 2). La sorgente di ingresso è un laser a luce bianca, fornendo a banda larga output spettrale visibile alle regioni a infrarossi. Per l'analisi spettrale, il sistema è dotato di due dispositivi acusto-ottico: un filtro sintonizzabile acusto-ottico (AOTF) per la distribuzione una lunghezza d'onda selezionata la sorgente di ingresso a banda larga e un divisore di fascio acusto-ottico (AOBS) per guidare il selezionato riflette lunghezza d'onda al rivelatore. Il software per microscopia confocale iperspettrale (Vedi Tabella materiali) offre un'opzione di analisi spettrale personalizzabile di acquisire in sequenza le immagini di riflettanza a varie lunghezze d'onda di input. Inoltre, l'aberrazione cromatica criticamente possono interferire nella misurazione spettrale; Pertanto, l'uso di un obiettivo apocromatico è raccomandato.
Della nota, bianco-luce laser produce un output spettrale irregolare e i componenti ottici influiscono anche sul profilo spettrale. Di conseguenza, gli spettri acquisiti devono essere calibrate per la successiva analisi quantitativa. Uno specchio d'argento protetto viene in genere utilizzato come riferimento, che fornisce un grado di riflessione quasi costante (> 97%) sopra la regione piena e visibile. Gli spettri acquisiti sono poi diviso per gli spettri di riferimento dallo specchio.
Le dimensioni di passaggio spettrale per l'analisi spettrale determinano la velocità di acquisizione; così, ha bisogno di essere ottimizzato. Come un assone più grande ha un maggiore periodo spettrale, richiede più fine spettrale campionamento. Ad esempio, un assone con un diametro di 10 µm, uno dei più grandi assoni fisiologici, ha un periodo spettrale di ~ 8 nm. Applicando criteri di campionamento di Nyquist, abbiamo impiegato l'intervallo di campionamento spettrale di 4 nm a coprire tutti gli assoni fisiologici nei tessuti nervosi del mouse. Questo approccio assume in genere alcuni secondi per una scansione completa spettrale e pertanto non è adatto per applicazioni in vivo , dove il movimento fisiologico (ad es. respirazione e battito cardiaco) interferisce stabile acquisizione spettrale. In precedenza abbiamo risolto questo problema tramite la strumentazione di un microscopio dritto su misura, progettato per acquisire l'intero spettro per ogni punto utilizzando uno spettrometro di matrice (acquisizione velocità ≈ 30 ms per ogni pixel).
In questo rapporto, descriviamo un protocollo dettagliato sull'imaging di SpeRe su una fetta di cervello fisso, che può essere eseguita in un microscopio commerciale iperspettrale (Vedi Tabella materiali). Così, il protocollo può essere completato da sperimentatori senza esperienza nella strumentazione ottica. Copriamo anche i potenziali problemi e risoluzione dei problemi per acquisizione e analisi dei dati di Bullonerie.
Protocollo
Tutte le procedure chirurgiche sono state approvate dal istituzionale Animal Care e uso Committee (IACUC) di Sungkyunkwan University.
1. preparazione del campione
Nota: Autoclave tutti gli strumenti chirurgici prima di movimentazione degli animali. Condurre tutte le prestazioni chirurgiche in una sala dedicata alle procedure chirurgiche. Guanti e camici chirurgici sterili devono essere indossati per tutto il personale nella sala chirurgica in ogni momento.
-
Fissazione del tessuto
- Preparare due siringhe da 10 mL che ciascuno riempito con tampone fosfato salino (PBS) e paraformaldeide al 4% (PFA) in PBS.
- Anestetizzare un mouse week-old 7-12, (C57BL/6J o qualsiasi mouse linee con entrambi i sessi) con la somministrazione di una miscela di zoletil e xilazina (1:1, 20 mg/kg ogni) o un regime di anestesia adatto alternativo (ad es., miscela di 80 mg/kg ketamina e 10 mg/kg xilazina) con un'iniezione intraperitoneale.
- Una volta che il mouse ha raggiunto un aereo chirurgico di anestesia (uso il metodo di pizzico-risposta di punta), è possibile esporre il cuore, tagliando la pelle e la gabbia toracica con le forbici.
- Praticare l'atrio destro con piccole forbici per drenare il sangue e irrorare la soluzione PBS e PFA in sequenza attraverso il ventricolo sinistro con un ago (tasso di perfusione = 4 mL/min, volume = 10 mL per ogni soluzione).
Nota: Il mouse si irrigidisce se la procedura è completata. L'uso dell'unguento oculare o la sterilizzazione non è necessaria in quanto la procedura è terminale. La procedura di fissazione può essere ignorata. Tuttavia, questo passaggio è consigliato per minimizzare le deformazioni strutturali, compresi i danni di meccanica del tessuto e gonfiore axonal ischemico. Per ulteriori dettagli sulla fissazione del tessuto, fare riferimento all'articolo di Gage, G. J. et al. 10
-
Preparazione della fetta del cervello
- Decapita il mouse con forbici chirurgiche attraverso il ventrale torace e l'addome.
- Rimuovere il cuoio capelluto e il periostio utilizzando un opportunamente piccole forbici fino ad esporre completamente il cranio.
- Rompere l'osso frontale e tagliare il cranio lungo la sutura sagittale dall'osso occipitale utilizzando piccole forbici con cura di non per danneggiare il cervello.
- Rimuovere delicatamente il cranio e il mater di dura in modo sequenziale utilizzando una pinzetta.
- Estrarre il cervello utilizzando un cucchiaio di plastica morbido, facendo attenzione a non per danneggiare il tessuto.
- Sciacquare e mettere a bagno il cervello Estratto in una soluzione PFA 4% per 30 min a 4 ° C.
- Affettare il cervello fisso in 100 – 150 µm sezioni utilizzando un vibratomo.
Nota: Per ulteriori dettagli sulla preparazione del tessuto, vedere l'articolo di Segev et al. 11. per le procedure successive, il tessuto deve essere affettato in sezioni più sottile rispetto allo spessore del distanziale.
-
Montaggio della fetta del cervello
- Preparare 1 vetrino e 2 quadrati vetrini coprioggetto (22 × 22 × 0,17 mm) per ogni sezione del tessuto.
- Uno dei bicchieri coperchio quadrati tagliato a metà (due pezzi rettangolari) utilizzando un tagliavetro.
- Collegare i due degli occhiali copertina dimezzati utilizzando una colla super sul vetro dello scivolo.
Nota: Lo spazio tra i due cristalli dimezzati dovrebbe essere leggermente più grande rispetto alla dimensione della fetta del tessuto. - Raccogliere la fetta di cervello con una spazzola o una pipetta e posare il tessuto sul vetro scorrevole tra il distanziale. Fare attenzione a non per piegare il tessuto.
- Pipettare 100 μL di PBS sulla superficie del tessuto.
- Posizionare il vetro di copertura altri quadrato sopra il tessuto. Evitare l'inclusione di bolle d'aria durante questa fase.
- Guarnizione intorno al vetro di copertura usando uno smalto per unghie (o in alternativa apposito collante) per evitare l'evaporazione di PBS e la contaminazione da polvere durante la successiva sessione di imaging.
Nota: Lo sperimentatore può mettere in pausa in questa fase.
2. taratura
- Accendere il microscopio almeno 1 h prima l'imaging per consentire la stabilizzazione termica del laser source (Vedi Tabella materiali). Quindi, attivare il software per l'analisi spettrale (Vedi Tabella materiali) e fare clic sul pulsante Acquisisci sulla cima di GUI (Figura 3a).
- Attivare l'otturatore di software per il bianco-luce laser (WLL) e tubo di fotomoltiplicatore (PMT) (Figura 3a).
- Selezionare la modalità di xyΛ nell'elenco a discesa in Modalità di acquisizione, quindi, la modalità di input del laser viene automaticamente modificata da costante per cento a Potenza costante. Deselezionare la casella di controllo di Movimento automatico SP. E quindi impostare la finestra spettrale del laser ingresso 470 – 670 nm e le dimensioni di passaggio spettrale a 4 nm su Λ-eccitazione Lambda Scan Impostazioni (Figura 3b).
- IMPOSTARE l'intervallo spettrale di PMT a 450-690 facendo doppio clic o spostando la barra di regolazione per la gamma spettrale (Figura 3C).
Nota: Questo intervallo spettrale dovrebbe includere la larghezza di banda completa sorgente di ingresso. - Selezionare un obiettivo di immersione in acqua, opportunamente con un'apertura numerica elevata (NA > 0.7) e dare qualsiasi valore alla potenza PMT e laser per attivare il pulsante di configurazione AOBS e Live Scan . Passare il percorso ottico controllando riflesso nella Configurazione AOBS (figura 3d).
- Montare uno specchio di riferimento (Vedi Tabella materiali) sulla fase di microscopio. La superficie a specchio dovrebbe essere affrontata contro la lente dell'obiettivo. Se non è facile mettere lo specchio sopra il tavolino del microscopio, legame uno specchio sul piatto piatto (es. vetro diapositiva).
- Controllo il tavolino del microscopio per allineare il piano focale per la superficie a specchio.
- Regolare il guadagno PMT e la potenza del laser considerando la gamma dinamica del sensore e quindi modificare la modalità di input di laser da costante per cento a Potenza costante.
Nota: Come è tipico, un PMT guadagno di 500 (V) e una potenza del laser relativa dello 0,1% sono utilizzati a 570 nm. - Confermare in modo pseudo-colore per verificare che non esiste nessuna saturazione su tutta la gamma di lunghezza d'onda. Se si osserva la saturazione, abbassare la potenza del laser (Figura 3a).
- Eseguire l'acquisizione Lambda Scan .
- Rimuovere lo specchio dal palco e ripetere l'acquisizione stessa senza un campione al fine di ottenere il riferimento scuro (cioè, offset scuro).
- Salvare i dati in un formato TIF Multistacked .
3. SpeRe Image Acquisition
- Posizionare il tessuto montato sul tavolino del microscopio. Per allineare approssimativamente il tessuto per il piano focale della lente dell'obiettivo, è necessario utilizzare la modalità di fluorescenza grandangolari attraverso un oculare.
- Con il Live Scan , controllo il tavolino del microscopio per allineare il piano focale nella regione di interesse per il tessuto. Per evitare il rumore di fondo dal vetrino coprioggetto, seleziona la regione di destinazione di almeno 15 μm profondità dall'interfaccia vetro-tessuto.
- Acquisire lo stack di immagine spettrale per la regione di destinazione utilizzando la stessa procedura come descritto ai punti 2.1 – 2,10.
- Salvare i dati per il tessuto e l'offset scuro in formato TIF Multistacked .
Nota: Lo sperimentatore può mettere in pausa in questa fase.
4. elaborazione ed analisi di immagini
- Aprire i dati spettrali (multistacked TIF) per lo specchio di riferimento e il tessuto cerebrale in ImageJ.
- Selezionare il ROIs (regioni di interesse) per gli stack di immagine aperto — zona centrale per lo specchio di riferimento e il segmento di una fibra di assone per il tessuto cerebrale.
- Acquisire gli spettri raw per il ROIs selezionati da in esecuzione immagine → pile → trama asse z profilo dal menu ImageJ.
- Aprire i dati offset scuri, una presa per lo specchio di riferimento e le altre scattate per il tessuto cerebrale.
- Acquisire gli spettri per gli offset scuri di esecuzione immagine → pile → trama asse z profilo dal menu ImageJ.
- Salvare tutti gli spettri acquisiti utilizzando le opzioni di copia e incolla .
Nota: Per l'imaging di SpeRe, uso del campo di formazione immagine centrale per ridurre al minimo le aberrazioni ottiche fuori asse è consigliabile. Le fibre dell'assone possono essere strutturalmente eterogenee lungo la loro lunghezza. Quindi, selezionando il ROI su un segmento piccolo assone, tipicamente < 5 µm, per ridurre al minimo il volume parziale artefatto raccomanda.
5. baseline correzione e analisi del segnale di SpeRe
- Sottrarre gli spettri offset da spettri dello specchio di riferimento e i tessuti del cervello.
- Normalizzare ogni spettro dividendo l'intensità massima dello spettro.
- Dividono lo spettro normalizzato dell'assone dallo spettro normalizzato dello specchio di riferimento e sottrarre offset DC lo spettro normalizzato.
- Misurare la frequenza di wavenumber inserendo lo spettro acquisito a un sinusoidale curva (Vedi Tabella materiali) e poi convertire il numero d'onda acquisito alla periodicità per prendendo reciproco.
- Convertire la periodicità wavenumber al diametro dell'assone usando l'equazione in Figura 4c.
Risultati
Secondo il protocollo, una fetta di cervello fisso è stata preparata con un fluoroforo mielina-targeting la colorazione esogena (Vedi Tabella materiali). Bullonerie imaging è stata eseguita sulla fetta di cervello utilizzando un microscopio confocale commerciale iperspettrali in combinazione con fluorescenza confocale imaging (Figura 4a). Bullonerie, ingresso ottico intensità è stato impostato per 5 µW/µm2 con un tempo di sos...
Discussione
Bullonerie è una nuova modalità imaging privo di etichetta basata su interferometria spettrale, che per la prima volta, offre le informazioni su scala nanometrica in assoni myelinated dal vivo. L'attuale protocollo di acquisizione, la risoluzione spaziale per il diametro dell'assone è dell'ordine di 10 nm. Inoltre, SpeRe utilizza gli ordini di grandezza più bassa dose di luce rispetto ad altri microscopie di Super-risoluzione; così, è esente da fototossicità e photobleaching. Bullonerie fornirebbe una nuova strada...
Divulgazioni
Gli autori dichiarano concorrenti interessi finanziari: Kwon J. e M. Choi sono gli inventori della tecnologia in attesa di brevetto descritto in questo articolo.
Riconoscimenti
Questo lavoro è stato supportato dall'Istituto di scienze fondamentali (IBS-R015-D1) e dal programma di base scienza ricerca attraverso la nazionale Ricerca Fondazione della Corea (NRF) finanziato dal Ministero dell'istruzione (2017R1A6A1A03015642).
Materiali
| Name | Company | Catalog Number | Comments |
| Glass cutter | - | - | Can be purchased in a local convenience store or online stores. |
| Nail polish | - | - | Can be purchased in a local convenience store or online stores. |
| Apochromat objective 40×, NA 1.1 | Leica Microsystems | 15506357 | Water-immersion type |
| Fluoromyelin Green | Thermo Fisher | F34651 | Alternatively, Fluoromyelin Red (F34652) can be used. |
| Leica SP8 TCS microscope | Leica Microsystems | SP8 | Refer to the "Configuration of microscope" in Introduction Section for details. |
| Imaging software | Leica Microsystems | LAS-X | - |
| Matlab | MathWorks | - | - |
| Mirror | Thorlabs | PF10-03-P01 | Coated with protected silver. |
| Phosphate-buffered saline (PBS) | Life technologies | 14190-136 | - |
| Paraformaldehyde | Biosolution | BP031a | 4% v/v in PBS |
| Cover slip | Thermo Fisher | 3306 | Thickness: #1 (0.13 to 0.17 mm) |
| Slide glass | Muto Pure Chemicals | 5116-20F | Thickness: ~1 mm |
| Super glue | Henkel | Loctite 406 | Use a dispensing equipment to avoid skin or eye contact. |
| Syringe pump | Brainetree Scientific | BS-8000 DUAL | - |
| Vibratome | Leica Biosystems | VT1200S | - |
| White-light laser | NKT photonics | EXB-6 | EXB-6 was discontinued and replaced by EXU-6. |
Riferimenti
- Fernandez-Moran, H., Finean, J. B. Electron microscope and low-angle x-ray diffraction studies of the nerve myelin sheath. Journal of Cell Biology. 3, (1957).
- Blaurock, A. E. The spaces between membrane bilayers within PNS myelin as characterized by X-ray diffraction. Brain Research. 210, 383-387 (1981).
- Shim, S. -. H., et al. Super-resolution fluorescence imaging of organelles in live cells with photoswitchable membrane probes. Proceedings of the National Academy of Sciences. , 13978-13983 (2012).
- Urban, N. T., Willig, K. I., Hell, S. W., Nägerl, U. V. STED nanoscopy of actin dynamics in synapses deep inside living brain slices. Biophysics Journal. 101, 1277-1284 (2011).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nature Methods. 5, 155-157 (2008).
- Peters, A., Sethares, C. Is there remyelination during aging of the primate central nervous system?. Journal of Comparative Neurology. 460, 238-254 (2003).
- De Campos Vidal, B., Silveira Mello, M. L., Caseiro-Filho, A. C., Godo, C. Anisotropic properties of the myelin sheath. Acta Histochemica. 66, 32-39 (1980).
- Schain, A. J., Hill, R. A., Grutzendler, J. Label-free in vivo imaging of myelinated axons in health and disease with spectral confocal reflectance microscopy. Nature Medicine. 20, 443-449 (2014).
- Kwon, J., et al. Label-free nanoscale optical metrology on myelinated axons in vivo. Nature Communication. 8, 1832 (2017).
- Gage, G. J., Kipke, D. R., Shain, W. Whole Animal Perfusion Fixation for Rodents. JoVE. (65), e3564 (2012).
- Segev, A., Garcia-Oscos, F., Kourrich, S. Whole-cell Patch-clamp Recordings in Brain Slices. JoVE. (112), e54024 (2016).
- Waldchen, S., Lehmann, J., Klein, T., Van De Linde, S., Sauer, M. Light-induced cell damage in live-cell super-resolution microscopy. Scientific Reports. 5, 15348 (2015).
- Chang, J. B., et al. Iterative expansion microscopy. Nature Methods. 14, 593-599 (2017).
- Polishchuk, R. S., et al. Correlative light-electron microscopy reveals the tubular-saccular ultrastructure of carriers operating between Golgi apparatus and plasma membrane. Journal of Cell Biology. 148, 45-58 (2000).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon