Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Étude simultanée du recrutement des sous-populations de Monocyte sous flux In Vitro
Dans cet article
Résumé
Nous présentons ici un protocole intégré qui mesure la sous-population de monocyte traite sous flux in vitro par l’utilisation de marqueurs de surface spécifiques et la microscopie confocal fluorescence. Ce protocole peut être utilisé pour explorer les étapes séquentielles de recrutement aussi bien quant à profil autres sous-types de leucocytes à l’aide d’autres marqueurs de surface spécifiques.
Résumé
Le recrutement des monocytes du sang vers les tissus périphériques ciblés est essentiels pour le processus inflammatoire au cours de la lésion tissulaire, le développement de tumeurs et les maladies auto-immunes. Ceci est facilité grâce à un processus de capture de libre circulation sur la surface luminale de cellules endothéliales activées, suivie de leur migration transendothéliale et de l’adhérence (transmigration) dans le tissu sous-jacent affecté. Cependant, les mécanismes qui prennent en charge le recrutement préférentiel et contextuelle des sous-populations de monocytes ne sont pas encore pleinement compris. Par conséquent, nous avons développé une méthode qui permet le recrutement des sous-populations de monocyte différents en même temps visualisé et calculées sous flux. Cette méthode basée sur l’imagerie confocale Time-lapse, permettant de distinguer sans ambiguïté les monocytes adhérentes et réincarnés. Ici, nous décrivons comment cette méthode peut être utilisée pour étudier simultanément la cascade de recrutement des monocytes pro-angiogénique et non-angiogéniques in vitro. En outre, cette méthode peut être étendue afin d’étudier les différentes étapes du recrutement de trois populations de monocytes.
Introduction
Monocytes constituent une composante phagocytaire de l’immunité innée qui est essentielle pour la lutte contre les agents pathogènes, nettoyer les tissus endommagés, l’angiogenèse et la pathophysiologie de nombreuses maladies dont les cancers1,2,3 . Les monocytes sont des cellules dérivées de la moelle osseuse composées de populations hétérogènes qui circulent dans le sang, mais peuvent être recrutées sur le site de l’inflammation dans les tissus périphériques par le biais de mécanismes moléculaires spécifiques. Les cascades de recrutement des monocytes, en ce qui concerne les leucocytes en général, implique différentes étapes, y compris la capture, rouler, ramper, arrestation, la migration transendothéliale (transmigration) et migration à travers la paroi des vaisseaux (membrane basale et peinture murale 4de cellules). Ces mesures concernent principalement induite par l’inflammation des molécules sur la surface endothéliale luminale comme sélectines, ligands glycoprotéine, chimiokines, molécules d’adhésion intercellulaire et jonctionnel et leurs récepteurs sur leucocytes comme sélectine ligands et les intégrines. Voies de trafic à travers les jonctions de cellules endothéliales (paracellulaires) ou par l’intermédiaire de l’organisme de cellules endothélial (transcellulaire) peuvent être utilisés par les leucocytes de franchir la barrière endothéliale5. Tandis que les monocytes ont historiquement été documentés à transmigrer la voie transcellulaire, divergences potentielles dans leur voie migratoire ont été proposés comme monocytes ne sont plus considérés comme une population homogène de cellules. Il est maintenant évident que la diversité des monocytes peuvent être définies par chacun de leurs différences et points communs, en ce qui concerne leur extravasation distinctive cascades3,6. Par conséquent, afin de distinguer sans ambiguïté les sous-populations de monocytes, il est crucial de visualiser, et le processus de phénotype le comportement de chacun de ces différentes sous-populations lors du recrutement.
Monocytes d’être humain, porc, rat et souris ont été subdivisées en sous-populations phénotypiques avec certaines fonctions attribuées et les comportements migratoires spécifiques7,8,9. Par exemple, chez les humains, monocytes se divisent en trois sous-ensembles basés sur leur expression superficielle de CD14, un corécepteur pour lipopolysaccharide bactérien et CD16, les récepteurs Fc-gamma III. Sous-populations de monocyte humain incluent CD14 classique+CD16-, les intermédiaires CD14+CD16+ et non-classique CD14dimCD16+ cellules6,9. Le CD14 classique+CD16– monocytes se sont avérées être principalement inflammatoire alors que la piscine du CD16+ monocytes trouvées collectivement de présenter TIE2 expression et on fonction10. Régulièrement, la stimulation des cellules endothéliales par des cytokines inflammatoires telles que la nécrose de tumeur humaine facteur α (TNF) ou l’interleukine (il-1) beta (inflammation conventionnelle) est suffisante pour déclencher le recrutement complet de CD14 classique+CD16 – monocytes. Cependant, des actions simultanées de facteur de croissance endothélial vasculaire (VEGF) A et de TNFα (inflammation d’axée sur les facteurs angiogéniques) sont nécessaires pour provoquer la transmigration de le CD16+ on piscine de monocytes3. Historiquement, le système traditionnel de Transwell sous des conditions statiques, la chambre de flux de plaques parallèles et les chambres de débit µ-diapositive ont été utilisées pour analyser quantitativement le recrutement de la population à un moment en vitro11 un leucocyte ,12,13. Tandis que ces protocoles ont été validés, une méthode plus robuste permettant l’analyse simultanée de plusieurs sous-populations de monocyte serait considérée comme plus perspicace. Ces méthodes doivent tenir compte des interactions multiples et les différentes fréquences de chaque population respective et également fournir une interprétation mécaniste des similarités et des spécificités pour les cascades de recrutement qui définissent chaque monocyte sous-ensemble.
Nous présentons ici une méthode basée sur l’imagerie Time-lapse de recrutement de monocytes sous flux qui permet les cascades migrateurs des sous-populations de monocyte différents à étudier simultanément à l’aide de la microscopie confocale. Cette méthode intègre certaines caractéristiques essentielles qui imitent l’inflammation des cellules endothéliales, ainsi que l’hémodynamique des monocytes dans les veinules post capillaires, en circulation l’emplacement principal du recrutement des leucocytes in vivo. La méthode proposée utilise des cellules endothéliales de veine ombilicale humaine (HUVEC), qui sont générés par un protocole bien établi d’isolement du humaine de cordons ombilicaux. Cette ressource clinique a l’avantage d’être facilement disponible comme sous-produit biologique, tout en fournissant également un rendement raisonnable des cellules endothéliales qui peuvent être isolés dans la veine ombilicale. Nous avons également utilisé les colorants fluorescents et immunofluorescence d’établir une distinction entre les différents composants cellulaires et microscopie confocale de définir sans ambiguïté le monocyte positionnement (luminal vs abluminal) au fil du temps. Le protocole présenté ici a été développé pour mesurer simultanément les niveaux de la transmigration des sous-populations de monocytes. En outre, il convient de noter que cette méthode peut être étendue afin d’étudier les autres sous-populations de leucocytes et le processus de recrutement par l’utilisation de différents biomarqueurs et l’étiquetage.
Protocole
Matériel humain ont été utilisés avec le consentement éclairé des donneurs volontaires et conformément à la Suisse les comités d’éthique sur la recherche clinique.
1. isolement et gel des ombilical humain de la veine des cellules endothéliales (HUVEC)
- Ajouter 5 mL de solution d’enrobage dans une fiole de T75 (0,1 mg/mL collagène G et 0,2 % de gélatine dans du PBS solution saline tamponnée au phosphate à pH 7,4) pendant 30 min à 37 ° C avant de lancer l’isolement HUVEC.
- Nettoyer le cordon avec du PBS, essuyer avec une compresse stérile et placez-le dans une boîte de Pétri stérile de 20 cm. Couper les extrémités du cordon avec des ciseaux stériles.
- Identifier la grande veine unique et les deux petites artères. Insérez doucement une canule avec un robinet à trois voies en annexe dans les extrémités de la veine à l’extrémité de la corde.
- Serrez le cordon et la connexion de la canule solidement avec une longueur de fil.
- Perfuse le cordon deux fois avec 20 mL de milieu RPMI contenant 100 U/mL de pénicilline, 100 U/mL de streptomycine et 250 ng/mL d’amphotéricine B pour laver des veines du cordon. Ce processus rend l’aspect du cordon, plus blanches et plus claire. Vider la veine avant l’ajout de collagénase en recueillant le RPMI avec une seringue à une extrémité.
- Perfuse la veine avec 12 mL de 1 mg/mL collagénase type j’ai (0,22 µm filtré).
- Fermez le robinet d’arrêt sur le câble se termine et incuber le cordon à 37 ° C pendant 12 min.
- Massez délicatement le cordon pour détacher des cellules endothéliales de la lumière de la veine.
- Prenez 30 mL de RPMI contenant 10 % de sérum de veau foetal avec une seringue de 50 mL et connectez-le à une extrémité du cordon ombilical.
- Connecter une seringue vide de 50 mL à l’autre extrémité du cordon ombilical
- Ouvrir le robinet et perfuse la veine d’un bout tout en recueillant réciproquement de l’autre extrémité.
Remarque : La suspension recueillie contient des cellules endothéliales. - Centrifuger cette suspension cellulaire à 200 x g pendant 5 min.
- Jeter le surnageant et remettre en suspension le culot cellulaire avec 10 mL de milieu M199 complet (M199 suppléments contenant 20 % FCS, la croissance de cellules endothéliales 15 µg/mL, 100 µg/mL Héparine sodique, hydrocortisone 0,5 de µM, 10 µg/mL d’acide L-ascorbique, 100 U/mL de pénicilline, 100 U/mL streptomycine et 250 ng/mL amphotéricine B).
- Enlever la solution de revêtement de la fiole T75 et rincez une fois avec du PBS.
- Graines les cellules récoltées étape 1.13 dans le ballon T75 et placez-le dans l’incubateur à 37 ° C, avec 5 % de CO2.
- Le lendemain, rincer le ballon 3 fois avec le milieu M199 complet pour enlever résiduelle des culots globulaires et puis changer le milieu tous les 2 jours jusqu’au confluent.
- Au confluent de 80 à 90 %, rincer la monocouche HUVEC fois avec 5 mL de PBS et détacher les cellules avec 5 mL de trypsine de 0,05 % à 1 mM EDTA à 37 ° C pendant 5 min. Ajouter 4 mL de M199 et 1 mL de FCS pour arrêter l’action de la trypsine. Rincer le ballon pour détacher tous HUVEC.
- Prélever une partie aliquote de 50 µL d’être utilisés pour la coloration de la VE-cadhérine, PECAM-1 et gp38 et d’analyser par cytométrie de flux pour vérifier la pureté HUVEC.
- Recueillir le reste des HUVEC étape 1.18 dans un tube de 15 mL et centrifuger à 200 x g pendant 5 min à température ambiante.
- Jeter le surnageant de l’étape 1.19, resuspendre le culot dans de la solution (FCS contenant 10 % DMSO) à une densité de 5 x 105 cellules/mL dans des cryotubes et congeler à-80 ° C ou dans l’azote liquide jusqu'à utilisation.
-
Pour vérifier la pureté HUVEC :
- Ajouter 1 µL d’anticorps de VE-cadhérine-FITC anti-humain, 1 µL d’anticorps PECAM1-PE anti-humain et 1 µL d’anticorps Podoplanin-APC anti-humain à l’aliquote de 50 µL de HUVEC recueillie à l’étape 1.18.
- Incuber à température ambiante pendant 10 min.
- Ajouter 100 µL de PBS et centrifuger à 400 x g pendant 30 s.
- Jeter le surnageant et remettre en suspension dans 100 µL de PBS. Données peuvent maintenant être acquis par des techniques de cytométrie en flux.
Remarque : HUVEC sont positives pour la VE-cadhérine et PECAM-1 et négatif pour les Podoplanin.
2. HUVEC dégivrage
Remarque : Utilisez des HUVEC à passage bas pour les expériences (maximum 5 passages).
- Enduire une fiole T75 avec 1 mL de la solution de revêtement à 37 ° C pendant 30 min.
- Defreeze HUVEC à 37 ° C pendant 2 min et remettre en suspension les cellules dans 10 mL de M199 complet rapidement.
- Centrifuger les cellules à 200 x g à température ambiante pendant 5 min et éliminer le surnageant.
- Resuspendre le culot cellulaire dans 10 mL de M199 complet.
- Transférer la suspension cellulaire dans la fiole revêtue. Placer la fiole dans l’incubateur à 37 ° C, avec 5 % de CO2. Changer le milieu de culture cellulaire tous les 2 jours.
3. HUVEC Culture à 0,4 µ-Slide chambre
- Cinq jours avant le début de l’expérience de flux, enduire préalablement les chambres d’un Dia 0,4 µ avec 30 µL de PBS contenant du collagène de 0,1 mg/mL G, 0,2 % de gélatine à 37 ° C pendant 30 min.
- Laver les chambres avec 100 µL de PBS.
- Détacher les cellules d’un HUVEC confluentes de 80 à 90 % d’une fiole de T75.
- Rincez HUVEC avec 5 mL de PBS et détacher avec 5 mL de trypsine de 0,05 % à 37 ° C pendant 5 min.
- Rincer et recueillent la suspension cellulaire dans M199 complet et compter les cellules de la méthode la plus pratique. Centrifuger à 200 x g pendant 5 min à température ambiante.
- Resuspendre le culot cellulaire à 106 cellules/mL et distribuer 30 µL (30 000 cellules) par chambre.
- Incuber les cellules dans un incubateur à 37 ° C, avec 5 % de CO2 pendant 1 h.
- Ajouter 150 µL de M199 complète dans chaque chambre et la culture des cellules pendant 5 jours dans l’incubateur à 37 ° C et 5 % de CO2. Changer le milieu tous les 2 jours.
4. HUVEC coloration pour dosage de recrutement de monocytes sous flux
- Préparer le milieu étiquetage fait de M199 et 1 µM de CMFDA (5-chloromethylfluorescein diacétate) et le réchauffer à 37 ° C pendant 5 min avant marquage cellulaire.
- HUVEC laver deux fois avec le milieu M199 chauffé à 37 ° C.
- Remettez le milieu 30 µL de réchauffé milieu étiquetage contenant 1 µM de CMFDA et placez dans l’incubateur à 37 ° C et 5 % de CO2 pendant 10 min.
- Laver une fois avec M199 complet et incuber les cellules avec M199 complète dans l’incubateur à 37 ° C et 5 % de CO2 pendant 30 min.
Remarque : Il est important d’éliminer toutes traces de sérum avant l’addition de la solution d’étiquetage, dans le cas contraire, vous pourrez modifier HUVEC coloration. - Remplacer le support avec M199 complet contenant soit TNFα humain (500 U/mL) ou un mélange de TNFα humain (500 U/mL) avec VEGFA humaine (1 µg/mL) pendant 6 h dans un incubateur à 37 ° C et 5 % de CO2.
5. isolement des Monocytes humains Pan et la coloration des sous-populations
- Utiliser une couche leuco-plaquettaire du concentré de sang humain, ou 20 mL de sang fraîchement isolée, recueilli le jour de l’expérience dans les tubes vacutainer EDTA.
- Diluer le sang en PBS-1 mM EDTA (1:1) et Pipeter doucement 20 mL du sang dilué sur le dessus de la 20 mL de média gradient de densité. Centrifuger à 400 g pendant 30 min à température ambiante avec accélération lente et sans frein.
- Recueillir les cellules mononucléées du sang périphérique (PBMC)-couche de plaquettes (entre médias gradient de densité et de couches de plasma) dans un nouveau tube de 50 mL contenant 40 mL de PBS - 1 mM EDTA. Supérieure à 50 mL de PBS - 1 mM EDTA.
- Centrifuger à 200 x g à température ambiante pendant 5 min. éliminer le surnageant.
- Resuspendre le culot cellulaire avec 10 mL de tampon de coloration (PBS - 1 mM EDTA contenant 0,5 % d’albumine sérique bovine BSA).
- Centrifuger à 200 x g à température ambiante pendant 5 min. éliminer le surnageant.
- Répétez les étapes 5.5 et 5.6.
- Resuspendre le culot cellulaire avec 10 mL de tampon de coloration. Prélever une partie aliquote de 10 µL pour un nombre de cellules.
- Contrôler les populations de PBMC et compter les cellules rapidement avec un cytomètre en flux.
Remarque : Les populations de lymphocytes et monocytes caractéristiques peuvent être observées (Figure 1 a). 50 mL de sang humain frais attendons sur 50-100 x 106 PBMC. -
Pour le recrutement de CD14 + versus CD14-PBMC sous flux:
- Laver le culot trois fois avec le tampon de flux (M199 contenant 0,5 % de BSA) et remettre en suspension les cellules mononucléaires dans le tampon courant à 6 x 106 cellules / mL.
- Faire des aliquots de 200 µL pour chaque dosage. Incuber à 37 ° C jusqu'à 20 min avant le dosage.
- Ajouter 5 µL d’anti-CD14-PE et Hoechst 33342 à une concentration finale de 2 µM à chaque aliquote. Mélanger et laisser incuber à 37 ° C pendant 10 min.
- Centrifuger l’aliquote à 400 x g pendant 30 s.
- Jeter le surnageant et Resuspendre le culot avec 200 µL de tampon de flux.
-
Pour le recrutement des sous-populations de monocyte sous flux:
- Isoler les monocytes avec un kit d’isolation de monocyte pan conformément aux instructions du fabricant.
Remarque : Le protocole suivant est de 50 x 106 cellules. Il peut évoluer vers le haut ou vers le bas aussi longtemps que c’est dans les recommandations du fabricant. - Centrifuger la suspension PBMC à 200 x g à température ambiante pendant 5 min.
- Jeter le surnageant et Resuspendre le culot avec 400 µL de tampon de coloration.
- Ajouter 50 µL de réactif de blocage des récepteurs Fc et 50 µL d’anticorps Pan Monocyte cocktail.
- Incuber à température ambiante pendant 10 min.
- Ajouter 400 µL de tampon et 100 µL d’anticorps anti-biotine conjugué de billes magnétiques de coloration. Incuber à température ambiante pendant 15 minutes.
- Ajouter 2 mL de tampon de coloration et d’utiliser une colonne de MACS LS couplée avec un aimant.
- Placer la colonne LS sur l’aimant et ajouter 1 mL de tampon de coloration. Jeter le cheminement.
- Passer la suspension PBMC dans la colonne et rassembler l’écoulement clair, bien que contenant des monocytes pan dans un nouveau tube de 15 mL.
- Ajouter de la mémoire tampon de marquage vers le haut à 5 mL.
- Prélever une partie aliquote et vérifier la qualité de l’isolement de monocytes avec un cytomètre en flux.
- Déterminer le nombre de monocytes de pan.
Remarque : Seulement la population monocytes peut être observée (Figure 1 b). - Centrifuger le reste des monocytes à l’étape 5.11.11 à 200 x g pendant 5 min.
- Jeter le surnageant.
- Resuspendre le culot dans 5 mL de tampon de flux (M199 contenant 0,5 % de BSA).
- Répétez les 5.11.13 à 5.11.14 deux fois pour éliminer toute trace d’EDTA.
- Isoler les monocytes avec un kit d’isolation de monocyte pan conformément aux instructions du fabricant.
- Faire suspension de monocytes dans le flux de la mémoire tampon (M199 avec 0,5 % de BSA) 6 x 106 cellules/ml.
- Faire des aliquots de 200 µL de monocytes pour chaque série de tests de recrutement.
- Conservez l’aliquote à 37 ° C dans l’incubateur jusqu'à 20 min avant l’injection.
- Ajouter 5 µL de l’anticorps anti-CD16-PE et Hoechst 33342 (finale de 2 µM) à chaque aliquote.
- Mélanger et laisser incuber à 37 ° C pendant 10 min.
- Centrifuger l’aliquote à 400 x g pendant 30 s.
- Jeter le surnageant et Resuspendre le culot avec 250 µL de tampon de flux.
- Ajouter 30 µL de la suspension de monocytes dans une chambre de la diapositive à servir pour définir les paramètres d’acquisition sur la microscopie confocale.
- Empêcher les aliquotes de suspension monocyte étape 5.18 à 37 ° C.
Remarque : Cette suspension est prête à être injecté dans le système d’écoulement.
6. préparation du système fluidique
- Veiller à ce que l’incubateur de cellules pour l’imagerie à 37 ° C.
Remarque : Un schéma du système de débit est donné à la Figure 2. - Assembler la partie de tube i : insérer un raccord Luer connecteur mâle à une extrémité d’un morceau de tube de silicone (8 cm long et 3 mm d’épaisseur) et branchez l’autre extrémité à un ensemble d’injection de Luer en ligne. Connectez le connecteur Luer ce dernier à un morceau de tube de silicone (40 cm et 3 mm d’épaisseur) à une extrémité.
Remarque : Éventuellement, un robinet 3 voies relié à une seringue de 5 mL peut être inséré entre le jeu d’injection de Luer en ligne et le tuyau pour l’enlèvement de bulle air éventuelle de silicone. - Assembler la partie tube II : Connectez une seringue de 20 mL à une extrémité d’une longueur de tuyauterie de silicone (1 m de long et 3 mm d’épaisseur). Insérer un raccord Luer connecteur mâle à l’autre extrémité du tuyau.
- Branchez la partie I et partie tube II en insérant les mâles de connecteur Luer à un manchon de verrouillage Luer femelle (Figure 2 a).
- Mettre l’extrémité libre du tuyau silicone dans le réservoir contenant le tampon de flux (M199 + 0,5 % de BSA) chauffé à 37 ° C.
- Tirez sur le piston de la seringue de 20 mL pour remplir le tube avec le tampon du flux.
- Placer la seringue sur la pompe et le fixer.
- Mettre la pompe en retirer mode (par opposition à infuser) et spécifier le débit.
- Déterminer le débit selon la diapositive IBIDI utilisé en utilisant la formule suivante :
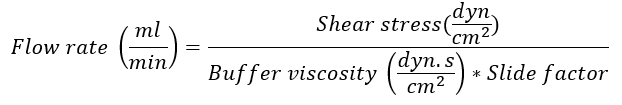
Remarque : Le facteur de la diapositive est tributaire de la diapositive IBIDI utilisée pour l’expérience. Pour le µ-slide j’ai0,4 Luer lock utilisé dans cet exemple, le facteur de la diapositive est 131,6. Pour facteurs de diapositive spécifique, consultez le site Web de compagnie14. La viscosité de tampon de flux est 0,0072 dyn.s/cm2. Contrainte de cisaillement dans les veinules post capillaires est environ 0,5 dynes/cm2. -
Connecter la diapositive (Figure 2 b) :
- Serrer le tube de silicone autour le manchon de verrouillage Luer femelle et débrancher les deux mâles de connecteur Luer le coupleur.
- Connectez-les à des réservoirs de la diapositive contenant stimulée HUVEC et remplissez-le de médium. Éviter les bulles d’air au cours de cette étape.
- Enlever les pinces, puis assurez-vous que la connexion n’est pas une fuite.
- Placez la lame au microscope pour l’imagerie Time-lapse et démarrer la pompe.
7. Time-lapse imagerie du recrutement de monocytes sous flux par microscopie confocale
- Utiliser un objectif 40 x (voir Table des matières) pour l’imagerie.
- Activer la 405 nm (noyaux de monocyte bleu), 488 nm (cellules endothéliales vertes) et 561 nm (rouge CD16 + sous-ensemble) lasers.
- Utilisez la chambre qui contient les monocytes pour définir les paramètres d’acquisition.
Remarque : Pour détecter les monocytes non-transmigré et réincarnés, le sténopé et l’intensité du laser 405 nm sont attachées hautes. Ainsi, non-transmigré monocytes sont légèrement visibles dans le plan basal. Monocytes réincarnés mais peut présentent une superficie non colorée autour du noyau correspondant à l’espace de nouveau occupé sous les cellules endothéliales. - Placez la chambre à acquérir sous le microscope.
- Choisissez 3 champs de vues au sein de 1 cm de rayon pour l’imagerie confocale multi-positions.
- Définir la basale et les parties apicales des cellules endothéliales
- Mettre une z-pile sur la plage de µm de 10 – 12 (étape de 0,5 µm). Exécuter une Time-lapse acquisition toutes les 1 min.
- Après 3 min de l’imagerie, injecter 200 µL de la suspension de monocyte (6 x 106 cellules/mL) par l’orifice d’injection en ligne Luer.
Remarque : Rapidement les monocytes apparaissent dans le plan focal apical, respecter et commencer la transmigration (transit entre l’apicale et le plan basal). - Image pendant au moins 30 min. Une fois terminé, arrêtez d’imagerie et arrêter l’écoulement. Clamper la tubulure pour Débranchez-les de la diapositive.
- Difficulté la diapositive avec 4 % de paraformaldéhyde à 4 ° C pendant 10 min.
- Laver la lame avec du PBS et stocker la diapositive à 4 ° C pour une analyse ultérieure si nécessaire.
8. analyser les données avec ImageJ
- Compter le nombre de totales monocytes adhérentes dans chaque domaine. Déterminer le nombre de cellules par mm2.
- Compter les monocytes réincarnés qui sont présents dans le plan basal sous les cellules endothéliales et identifiés par la présence d’un trou noir (dans le canal vert) autour du noyau.
- Séparer le nombre de leucocytes réincarnés par le nombre total de leucocytes adhérentes. Le taux de la transmigration est présenté sous forme de pourcentage des monocytes adhérentes.
- À titre d’illustration, l’apicale et les côtés basales peuvent être affichées simultanément pour illustrer les événements qui se produisent dans chacun de ces compartiments endothéliales.
Résultats
Détermination de l’état d’activation de HUVEC induite par le TNFα
La bio-activité de la cytokine inflammatoire TNFα peut être varier selon le lot et la réplétion du cycle gel-dégel. Il est important de vérifier l’état d’activation des HUVEC avec traitement de TNFα. Cela pourrait être interprété par coloration en parallèle quelques échantillons de HUVEC confluente pour l’induction inflammatoire des sélect...
Discussion
Nous rapportons ici une méthode décrivant une étude de comment les sous-populations de monocyte transmigrer par le biais de la monocouche endothéliale enflammée. La méthode discutée utilisé la microscopie confocale au lieu de la microscopie à contraste de phase, qui est également utilisée pour étudier le recrutement des monocytes sous flux3,11,19. Un des principaux avantages de l’utilisation de la microscopie confo...
Déclarations de divulgation
Les auteurs n’ont aucun intérêt financier concurrentes.
Remerciements
Nous remercions le Dr Paul Bradfield pour lecture de manuscrit et des rétroactions. A. S. a reçu un soutien financier de la Sir Jules Thorn Charitable Overseas Trust Reg.,
matériels
| Name | Company | Catalog Number | Comments |
| Tissue Culture Flasks 75 cm2 | TPP | 90076 | Routine culture of isolated HUVEC |
| µ-Slide VI 0.4 | IBIDI | 80606 | |
| Centrifuge Tubes 15 mL | TPP | 191015 | |
| Centrifuge Tubes 50 mL | TPP | 191050 | |
| Collagen G | Biochrom | L 7213 | For coating of cell culture flasks |
| Gelatin | Sigma-Aldrich | 1393 | For coating of cell culture flasks |
| Dulbecco’s Phosphate Buffered Saline (without MgCl2 and CaCl2) | Sigma-Aldrich | D8537 | |
| Dulbecco’s Phosphate Buffered Saline (with MgCl2 and CaCl2) | Sigma-Aldrich | D8662 | |
| RPMI-1640 Medium | Sigma-Aldrich | R8758 | |
| 3-Way Stopcocks | BIO-RAD | 7328103 | |
| penicillin 10,000 μ/mL streptomycine 10,000 μg/mL fungizone 25 μ/mL | AMIMED | 4-02F00-H | |
| Collagenase type 1 | Worthington | LS004216 | |
| Medium 199 1x avec Earle's salts, L-Glutamine, 25 mM Hepes | GIBCO | 22340020 | |
| Bovine Albumin Fraction V | ThermoFisher | 15260037 | |
| Endothelial Cell Growth Supplement, 150 mg | Millipore | 02-102 | |
| Heparin Sodium | Sigma-Aldrich | H3149RT | |
| Hydrocortisone | Sigma-Aldrich | H6909 | |
| L-Ascorbic acid | Sigma-Aldrich | A 4544 | |
| EDTA disodium salt dihydrate C10H14N2Na2O8 · 2H2O | APPLICHEM | A2937.0500 | |
| CD144 (VE-Cadherin), human recombinant clone: REA199, FITC | Miltenyi Biotech | 130-100-713 | AB_2655150 |
| CD31-PE antibody, human recombinant clone: REA730, PE | Miltenyi Biotech | 130-110-807 | AB_2657280 |
| Anti-Podoplanin-APC, human recombinantclone: REA446, APC | Miltenyi Biotech | 130-107-016 | AB_2653263 |
| BD Accuri C6 Plus | BD Bioscience | ||
| µ-Slide I Luer | IBIDI | 80176 | |
| CMFDA (5-chloromethylfluorescein diacetate) | ThermoFisher | C2925 | |
| Recombinant human TNFα | Peprotech | 300-01A | |
| Recombinant human VEGFA | Peprotech | 100-20 | |
| NE-1000 Programmable Syringe Pump | KF Technology | NE-1000 | |
| Ficoll Paque Plus | GE Healthcare | 17-1440-02 | |
| Anti-human CD14-PE, human recombinant clone: REA599, PE | Miltenyi Biotech | 130-110-519 | AB_2655051 |
| Pan Monocyte Isolation Kit, human | Miltenyi Biotech | 130-096-537 | |
| Anti-human CD16-PE, human recombinant clone: REA423, PE | Miltenyi Biotech | 130-106-762 | AB_2655403 |
| LS columns | Miltenyi Biotech | 130-042-401 | |
| QuadroMACS Separator | Miltenyi Biotech | 130-090-976 | |
| Hoechst 33342, Trihydrochloride, Trihydrate | ThermoFisher | H1399 | |
| Silicone tubing | IBIDI | 10841 | |
| Elbow Luer Connector | IBIDI | 10802 | |
| Female Luer Lock Coupler | IBIDI | 10823 | |
| Luer Lock Connector Female | IBIDI | 10825 | |
| In-line Luer Injection Port | IBIDI | 10820 | |
| Ar1 confocal microscope | Nikon | ||
| 40x objective | Nikon | 40x 0.6 CFI ELWD S Plane Fluor WD:3.6-2.8mm correction 0-2mm | |
| ImageJ Software | NIH |
Références
- Auffray, C., Sieweke, M. H., Geissmann, F. Blood Monocytes: Development, Heterogeneity, and Relationship with Dendritic Cells. Annual Review of Immunology. 27 (1), 669-692 (2009).
- De Palma, M., Venneri, M. A., Roca, C., Naldini, L. Targeting exogenous genes to tumor angiogenesis by transplantation of genetically modified hematopoietic stem cells. Nature Medicine. 9 (6), 789-795 (2003).
- Sidibe, A., et al. Angiogenic factor-driven inflammation promotes extravasation of human proangiogenic monocytes to tumours. Nature Communications. 9 (1), 355 (2018).
- Ley, K., Laudanna, C., Cybulsky, M. I., Nourshargh, S. Getting to the site of inflammation: the leukocyte adhesion cascade updated. Nature Review Immunology. 7 (9), 678-689 (2007).
- Nourshargh, S., Alon, R. Leukocyte Migration into Inflamed Tissues. Immunity. 41 (5), 694-707 (2014).
- Cros, J., et al. Human CD14dim monocytes patrol and sense nucleic acids and viruses via TLR7 and TLR8 receptors. Immunity. 33 (3), 375-386 (2010).
- Geissmann, F., Jung, S., Littman, D. R. Blood monocytes consist of two principal subsets with distinct migratory properties. Immunity. 19 (1), 71-82 (2003).
- Chamorro, S., et al. In vitro differentiation of porcine blood CD163− and CD163+ monocytes into functional dendritic cells. Immunobiology. 209 (1-2), 57-65 (2004).
- Passlick, B., Flieger, D., Ziegler-Heitbrock, H. Identification and characterization of a novel monocyte subpopulation in human peripheral blood. Blood. 74 (7), (1989).
- Venneri, M. A., et al. Identification of proangiogenic TIE2-expressing monocytes (TEMs) in human peripheral blood and cancer. Blood. 109 (12), 5276-5285 (2007).
- Bradfield, P. F., et al. JAM-C regulates unidirectional monocyte transendothelial migration in inflammation. Blood. 110 (7), 2545-2555 (2007).
- Schenkel, A. R., Mamdouh, Z., Muller, W. A. Locomotion of monocytes on endothelium is a critical step during extravasation. Nature Immunology. 5 (4), 393-400 (2004).
- Luu, N. T., Rainger, G. E., Nash, G. B. Kinetics of the different steps during neutrophil migration through cultured endothelial monolayers treated with tumour necrosis factor-alpha. Journal Vascular Research. 36 (6), 477-485 (1999).
- ibidi GmbH. . Shear Stress and Shear Rates for ibidi µ-Slides - Based on Numerical Calculations. , (2014).
- Yang, L., Froio, R. M., Sciuto, T. E., Dvorak, A. M., Alon, R., Luscinskas, F. W. ICAM-1 regulates neutrophil adhesion and transcellular migration of TNF-alpha-activated vascular endothelium under flow. Blood. 106 (2), 584-592 (2005).
- Yang, C. -. R., Hsieh, S. -. L., Ho, F. -. M., Lin, W. -. W. Decoy receptor 3 increases monocyte adhesion to endothelial cells via NF-kappa B-dependent up-regulation of intercellular adhesion molecule-1, VCAM-1, and IL-8 expression. Journal of Immunology. 174 (3), 1647-1656 (2005).
- Wong, D., Dorovini-Zis, K. Expression of vascular cell adhesion molecule-1 (VCAM-1) by human brain microvessel endothelial cells in primary culture. Microvascular Research. 49 (3), 325-339 (1995).
- Bradfield, P. F., Nourshargh, S., Aurrand-Lions, M., Imhof, B. A. JAM family and related proteins in leukocyte migration (Vestweber series). Arteriosclerosis Thrombosis and Vascular Biology. 27 (10), 2104-2112 (2007).
- Bradfield, P. F., et al. Divergent JAM-C Expression Accelerates Monocyte-Derived Cell Exit from Atherosclerotic Plaques. PLoS One. 11 (7), e0159679 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon