È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Studio simultaneo del reclutamento di sottopopolazioni monocito sotto flusso In Vitro
In questo articolo
Riepilogo
Qui, presentiamo un protocollo integrato che misura sottopopolazione del monocito traffico sotto flusso in vitro mediante uso di specifici marcatori di superficie e microscopia a fluorescenza confocale. Questo protocollo consente di esplorare reclutamento sequenziale passaggi anche per quanto riguarda il profilo di altri sottotipi del leucocita utilizzando altri specifici marcatori di superficie.
Abstract
Il reclutamento dei monociti dal sangue ai tessuti periferici mirati è fondamentale per il processo infiammatorio durante la lesione del tessuto, lo sviluppo del tumore e le malattie autoimmuni. Ciò è facilitato attraverso un processo di acquisizione da flusso libero sulla superficie luminale delle cellule endoteliali attivate, seguita da loro adesione e transendoteliale migrazione (Trasmigrazione) nel tessuto interessato sottostante. Tuttavia, i meccanismi che supportano l'assunzione preferenziale e dipendente dal contesto delle sottopopolazioni monocito ancora completamente non sono capiti. Di conseguenza, abbiamo sviluppato un metodo che permette il reclutamento di sottopopolazioni monocito differenti per essere visualizzati e misurata sotto flusso contemporaneamente. Questo metodo, basato sull'imaging confocale time-lapse, consente la distinzione inequivocabile tra aderente e transmigrated monociti. Qui, descriviamo come questo metodo può essere utilizzato per studiare simultaneamente la cascata di reclutamento dei monociti pro-angiogenici e non-angiogenici in vitro. Inoltre, questo metodo può essere esteso per studiare le diverse fasi del reclutamento di fino a tre popolazioni di monociti.
Introduzione
Monociti costituiscono una componente fagocitica dell'immunità innata che è essenziale per combattere gli agenti patogeni, pulizia di tessuti danneggiati, l'angiogenesi e la fisiopatologia di molte malattie compreso i cancri1,2,3 . I monociti sono cellule derivate da midollo osseo composte di sottopopolazioni eterogenee che circolano nel sangue, ma possono essere reclutate nel sito di infiammazione nel tessuto periferico attraverso specifici meccanismi molecolari. Le cascate di reclutamento dei monociti, per quanto riguarda i leucociti in generale, implica diversi passaggi tra cui cattura, rotolamento, strisciando, arresto, migrazione transendoteliale (Trasmigrazione) e migrazione attraverso la parete del vaso (membrana dello scantinato e murale di cellule)4. Questi passaggi comportano principalmente indotta da infiammazione molecole sulla superficie luminal endoteliale come selectine, glicoproteina ligandi, chemochine, molecole di adesione intercellulare e giunzionale e dei loro recettori sui leucociti quali selectina ligandi e le integrine. Vie di traffico attraverso giunzioni delle cellule endoteliali (paracellular) o attraverso il corpo delle cellule endoteliale (transcellular) possono essere utilizzate dai leucociti di attraversare la barriera endoteliale5. Mentre i monociti sono stati documentati storicamente trasmigrano attraverso la via transcellulare, potenziali divergenze nel loro percorso migratorio sono stati proposti come monociti non siano più considerati una popolazione omogenea delle cellule. Ora sta diventando chiaro che la diversità del monocito può essere definito da ciascuna delle loro differenze e somiglianze, per quanto riguarda il loro distintivo stravaso cascate3,6. Pertanto, al fine di discriminare in modo inequivocabile le sottopopolazioni monocito, è fondamentale per visualizzare ed elaborare di fenotipo il comportamento di ciascuna di queste sottopopolazioni differenti durante il reclutamento.
Monociti da umano, maiale, ratti e topi sono stati suddivisi in sottopopolazioni fenotipiche con determinate funzioni ascritte e specifici comportamenti migratori7,8,9. Ad esempio, in esseri umani, monociti possono suddividere in tre sottogruppi basati sulla loro superficie espressione di CD14, corecettore per lipopolisaccaride batterico e CD16, il ricevitore di Fc-gamma III. Sottopopolazioni monocito umano includono CD14 classica+CD16-, intermedio CD14+CD16+ e tronchetti CD14dimCD16 cellule+ 6,9. Il CD14 classica+CD16– monociti sono stati indicati per essere principalmente infiammatorie mentre la piscina di CD16+ monociti collettivamente sono stati trovati per presentare TIE2 espressione e proangiogenic funzione10. Coerentemente, la stimolazione di cellule endoteliali con citochine infiammatorie come il fattore di necrosi tumorale umano fattore α (TNF) o interleuchina (il-1) beta (convenzionale infiammazione) è sufficiente a far scattare la completa assunzione di CD14 classica+CD16 – monociti. Tuttavia, sono necessari per provocare la trasmigrazione del CD16 azioni simultanee di A di fattore di crescita endoteliale vascolare (VEGF) e TNFα (infiammazione guidata da fattori angiogenici)+ proangiogenic piscina di monociti3. Storicamente, il sistema tradizionale di Transwell in condizioni statiche, la camera di flusso di piatti paralleli e gli alloggiamenti di flusso µ-diapositiva sono stati usati per analizzare quantitativamente il reclutamento della popolazione uno leucocitaria a un tempo in vitro11 ,12,13. Mentre questi protocolli sono stati convalidati, un metodo più robusto che ha permesso l'analisi simultanea di più sottopopolazioni monocito sarebbe considerato più penetrante. Tali metodologie devono rappresentano interazioni multiple e le diverse frequenze di ogni rispettiva popolazione e anche fornire una comprensione meccanicistica delle affinità e specificità per le cascate di reclutamento che definiscono ogni monocito sottoinsieme.
Qui, presentiamo un metodo basato sull'imaging time-lapse del reclutamento di monociti sotto flusso che permette le cascate migratori delle sottopopolazioni monocito diverso da studiare simultaneamente mediante microscopia confocale. Questo metodo integra alcune caratteristiche critiche che imitano l'infiammazione delle cellule endoteliali, come pure l'emodinamica di fare circolare i monociti in venule post-capillari, la sede principale del reclutamento dei leucociti in vivo. Il metodo proposto utilizza cellule endoteliali di vena ombelicale umana (HUVEC), che vengono generate attraverso un protocollo ben consolidato di isolamento dal cordone ombelicale umani. Questa risorsa clinica ha il vantaggio di essere facilmente disponibile come sottoprodotto biologico, fornendo inoltre un rendimento ragionevole delle cellule endoteliali che può essere isolato dalla vena ombelicale. Abbiamo anche usato tinture fluorescenti e immunofluorescenza per distinguere tra i diversi componenti cellulari e microscopia confocale per definire inequivocabilmente monocito posizionamento (luminal vs abluminale) nel corso del tempo. Il protocollo qui presentato è stato sviluppato simultaneamente misurare i livelli di trasmigrazione delle sottopopolazioni monocito. Inoltre, si deve osservare che questa metodologia può essere esteso per studiare altre sottopopolazioni di leucociti e processi di reclutamento mediante uso di diversi biomarcatori ed etichettatura.
Protocollo
Materiali umani sono stati utilizzati con il consenso informato dei donatori volontari e in conformità con i comitati di etica svizzero sulla ricerca clinica.
1. isolamento e congelamento del cordone ombelicale umano cellule endoteliali (HUVEC) della vena
- Aggiungere 5 mL di soluzione di rivestimento un matraccio T75 (0,1 mg/mL collagene G e gelatina di 0,2% in tampone fosfato salino PBS a pH 7.4) per 30 min a 37 ° C prima dell'inizio di isolamento HUVEC.
- Pulire il cavo con PBS, pulirlo con compresse sterili e posizionarlo in una sterile 20cm di Petri. Tagliare le estremità del cavo con forbici sterili.
- Identificare la singola vena grande e due piccole arterie. Inserire delicatamente una cannula con un rubinetto a tre vie collegato ad esso in vena estremità alle estremità del cavo.
- Stringere il cavo e la connessione di cannula saldamente con una lunghezza di filo.
- Irrorare il cavo due volte con 20 mL di terreno RPMI contenente 100 U/mL di penicillina, 100 U/mL di streptomicina e 250 ng/mL anfotericina B per lavare le vene del cavo. Questo processo rende l'aspetto del midollo più bianca e più chiara. Svuotare la vena prima dell'aggiunta della collagenosi raccogliendo il RPMI con una siringa ad una estremità.
- Irrorare la vena con 12 mL di 1 mg/mL collagenasi tipo ho (0,22 µm-filtrati).
- Chiudi il rubinetto il cavo termina e incubare il cavo a 37 ° C per 12 min.
- Massaggiare delicatamente il cavo per staccare le cellule endoteliali dal lume della vena.
- Prendere 30 mL di RPMI contenente 10% di siero fetale di vitello con una siringa da 50 mL e collegarlo a un'estremità del cavo ombelicale.
- Collegare una siringa vuota 50ml per l'altra estremità del cavo ombelicale
- Aprire il rubinetto e irrorare la vena da un'estremità, mentre la raccolta reciprocamente da altra estremità.
Nota: La sospensione raccolta contiene cellule endoteliali. - Centrifugare la sospensione cellulare a 200 x g per 5 min.
- Eliminare il supernatante e risospendere il pellet cellulare con 10 mL di terreno completo M199 (M199 integratori contenenti 20% FCS, la crescita delle cellule endoteliali di 15 µ g/mL, 100 µ g/mL eparina sodica, idrocortisone di 0,5 µM, 10 µ g/mL dell'acido L-ascorbico, 100 U/mL di penicillina, 100 U/mL streptomicina e 250 ng/mL anfotericina B).
- Rimuovere la soluzione di rivestimento dal pallone T75 e sciacquare una volta con PBS.
- Seme le cellule raccolte dal punto 1.13 nel pallone T75 e posizionarlo nell'incubatore a 37 ° C con 5% CO2.
- Il giorno successivo, risciacquare il matraccio 3 volte con la sostanza M199 completa per rimuovere residui globuli rossi e quindi modificare il mezzo ogni 2 giorni fino alla confluenza.
- Confluenza di 80 – 90%, sciacquare il monostrato HUVEC una volta con 5 mL di PBS e staccare le cellule con 5 mL di tripsina 0.05% in 1 mM EDTA a 37 ° C per 5 min. aggiungere 4 mL di M199 e 1 mL di FCS per fermare l'azione della tripsina. Lavare la beuta per staccare tutte le HUVEC.
- Raccogliere un'aliquota di 50 µ l di essere utilizzato per la colorazione di VE-caderina, PECAM-1 e gp38 e analizzare tramite flusso cytometry per verificare la purezza HUVEC.
- Raccogliere il resto di HUVEC dal passaggio 1.18 in una provetta da 15 mL e centrifugare a 200 x g per 5 min a temperatura ambiente.
- Scartare il surnatante dal passaggio 1.19, risospendere il pellet cellulare in soluzione (FCS contenente 10% DMSO) di congelamento ad una densità di 5 x 105 cellule/mL in provette per criogenia e congelare a-80 ° C o in azoto liquido fino all'utilizzo.
-
Per verificare la purezza HUVEC:
- Aggiungere 1 µ l di anticorpo anti-umano VE-caderina-FITC, 1 µ l di anticorpo anti-umano PECAM1-PE e 1 µ l di anticorpo anti-umano Podoplanin-APC l'aliquota di 50 µ l di HUVEC raccolti al passaggio 1.18.
- Incubare a temperatura ambiente per 10 min.
- Aggiungere 100 µ l di PBS e centrifuga a 400 x g per 30 s.
- Scartare il surnatante e risospendere in 100 µ l di PBS. Dati possono ora essere acquisiti da tecniche di citometria a flusso.
Nota: HUVEC sono positivi per VE-caderina e PECAM-1 e la negazione per Podoplanin.
2. HUVEC sbrinamento
Nota: Utilizzare HUVEC a basso passaggio per esperimenti (massimi 5 passaggi).
- Rivestire un pallone T75 con 1 mL di soluzione di rivestimento a 37 ° C per 30 min.
- Scongelare HUVEC a 37 ° C per 2 min e risospendere le cellule in 10 mL di M199 completa rapidamente.
- Centrifugare le cellule a 200 x g a temperatura ambiente per 5 minuti e scartare il surnatante.
- Risospendere il pellet cellulare in 10 mL di M199 completa.
- Trasferire la sospensione di eritrociti nel matraccio prepatinato. Posto il pallone in incubatore a 37 ° C con 5% CO2. Cambiare il mezzo di coltura cellulare ogni 2 giorni.
3. HUVEC cultura nella camera di 0,4 µ-Slide
- Cinque giorni prima di iniziare l'esperimento di flusso, pre-cappotto alloggiamenti di un 0,4 µ-scivolo con 30 µ l di PBS contenente collagene di 0,1 mg/mL G, gelatina di 0.2% a 37 ° C per 30 min.
- Lavare gli alloggiamenti con 100 µ l di PBS.
- Staccare le cellule da un HUVEC confluenti di 80 – 90% di un pallone da T75.
- Sciacquare HUVEC con 5 mL di PBS e staccarli con 5 mL di tripsina 0.05% a 37 ° C per 5 min.
- Sciacquare e raccogliere la sospensione cellulare in completa M199 e contare le celle con il metodo più conveniente. Centrifugare a 200 x g per 5 min a temperatura ambiente.
- Risospendere il pellet cellulare a 106 cellule/mL e distribuire 30 µ l (30.000 cellule) in ogni camera.
- Incubare le cellule in un incubatore a 37 ° C con 5% CO2 per 1 h.
- Aggiungere 150 µ l di M199 completa per ogni camera e cultura le cellule per 5 giorni nell'incubatore a 37 ° C e 5% CO2. Modificare il mezzo ogni 2 giorni.
4. HUVEC macchiatura per dosaggio di reclutamento di monociti sotto flusso
- Preparare il supporto di etichettatura costituito M199 e 1 µM di CMFDA (5-chloromethylfluorescein diacetato) e riscaldarla a 37 ° C per 5 min prima cella etichettatura.
- HUVEC lavare due volte con medium M199 riscaldato a 37 ° C.
- Sostituire il mezzo con 30 µ l di riscaldato mezzo d'etichettatura contenente 1 µM di CMFDA e inserire in incubatore a 37 ° C e 5% CO2 per 10 min.
- Lavare una volta con M199 completa e incubare le cellule con M199 completa nell'incubatore a 37 ° C e 5% CO2 per 30 min.
Nota: È importante rimuovere tutte le tracce di siero prima dell'aggiunta della soluzione di etichettatura, in caso contrario può alterare HUVEC macchiatura. - Sostituire il supporto con M199 completa che contiene entrambi TNFα umano (500 U/mL) o un mix di TNFα umano (500 U/mL) con VEGFA umana (1 µ g/mL) per 6 h in un incubatore a 37 ° C e 5% CO2.
5. isolamento dei monociti umani Pan e colorazione delle sottopopolazioni
- Utilizzare un cappotto buffy di sangue umano concentrato, oppure 20 mL di sangue umano fresco isolato, raccolti il giorno dell'esperimento in provette vacutainer con EDTA.
- Diluire il sangue in PBS-1 mM EDTA (1:1) e delicatamente pipetta 20 mL di sangue diluito in cima i 20 mL di media di gradienti di densità. Centrifugare a 400 x g per 30 min a temperatura ambiente con accelerazione lenta e senza freno.
- Raccogliere le cellule mononucleate del sangue periferico (PBMC)-strato della piastrina (tra media gradienti di densità e livelli del plasma) in una nuova provetta da 50 mL contenente 40 mL di PBS - 1 mM EDTA. Superiore a 50 mL con PBS - 1 mM EDTA.
- Centrifuga a 200 x g a temperatura ambiente per 5 min, scartare il surnatante.
- Risospendere il pellet cellulare con 10 mL di buffer di colorazione (PBS - 1 mM EDTA contenente albumina di siero bovino 0,5% BSA).
- Centrifuga a 200 x g a temperatura ambiente per 5 min, scartare il surnatante.
- Ripetere i passaggi da 5.5 e 5.6.
- Risospendere il pellet cellulare con 10 mL di tampone di colorazione. Prendere un'aliquota di 10 µ l per un conteggio delle cellule.
- Controllare le popolazioni di PBMC e contare le celle rapidamente con un citometro a flusso.
Nota: Le popolazioni di linfociti e monociti caratteristiche possono essere osservate (Figura 1A). Da 50 mL di sangue umano fresco si aspettano circa 50-100 x 106 PBMC. -
Per l'assunzione di CD14 + contro CD14-PBMC sotto flusso:
- Lavare la pallina tre volte con tampone di flusso (M199 contenente 0.5% BSA) e risospendere le cellule mononucleari nel buffer di flusso a 6 x 106 cellule per mL.
- Rendere le aliquote di 200 µ l per ogni dosaggio. Incubare a 37 ° C fino a 20 min prima del dosaggio.
- Aggiungere 5 µ l di anti-CD14-PE e Hoechst 33342 ad una concentrazione finale di 2 µM ad ogni aliquota. Mescolare e incubare a 37 ° C per 10 min.
- Centrifugare l'aliquota a 400 x g per 30 s.
- Eliminare il supernatante e risospendere il sedimento con 200 µ l di tampone di flusso.
-
Per il reclutamento delle sottopopolazioni monocito sotto flusso:
- Isolare i monociti con un kit di isolamento del monocito padella secondo le istruzioni del produttore.
Nota: Il seguente protocollo di isolamento è per 50 x 106 cellule. Può essere scalata verso l'alto o verso il basso fino a quando è all'interno, le raccomandazioni del produttore. - Centrifugare la sospensione PBMC a 200 x g a temperatura ambiente per 5 min.
- Eliminare il supernatante e risospendere il sedimento con 400 µ l di tampone di colorazione.
- Aggiungere 50 µ l di reagente bloccante del recettore Fc e 50 µ l di anticorpo Pan monocito cocktail.
- Incubare a temperatura ambiente per 10 min.
- Aggiungere 400 µ l di macchiatura buffer e 100 µ l di anticorpo anti-biotina coniugati di biglie magnetiche. Incubare a temperatura ambiente per 15 min.
- Aggiungere 2 mL di buffer di colorazione e utilizzare una colonna di MACS LS accoppiata con un magnete.
- Posizionare la colonna LS sul magnete e aggiungere 1 mL di tampone di colorazione. Scartare il flusso continuo.
- Passare la sospensione PBMC nella colonna e raccogliere il flusso chiaro anche se contenenti i monociti di pan in una nuova provetta da 15 mL.
- Aggiungere il buffer di colorazione per di più fino a 5 mL.
- Prendere un'aliquota e controllare la qualità dell'isolamento del monocito con un citometro a flusso.
- Determinare il numero di monociti di pan.
Nota: Solo della popolazione monocito può essere osservata (Figura 1B). - Centrifugare il resto dei monociti dal passaggio 5.11.11 a 200 x g per 5 min.
- Scartare il surnatante.
- Risospendere il pellet cellulare in 5 mL di buffer di flusso (M199 contenente 0.5% BSA).
- Ripetere 5.11.13 a 5.11.14 due volte per eliminare ogni traccia di EDTA.
- Isolare i monociti con un kit di isolamento del monocito padella secondo le istruzioni del produttore.
- Rendere il monocito sospensione nel flusso del buffer (M199 con 0,5% BSA) 6 x 106 cellule/ml.
- Rendere le aliquote di 200 µ l di monociti per ciascun test di reclutamento.
- Mantenere l'aliquota a 37 ° C nell'incubatore fino 20 min prima dell'iniezione.
- Aggiungere 5 µ l di anticorpo anti-CD16-PE e Hoechst 33342 (2 µM finali) ad ogni aliquota.
- Mescolare e incubare a 37 ° C per 10 min.
- Centrifugare l'aliquota a 400 x g per 30 s.
- Eliminare il supernatante e risospendere il sedimento con 250 µ l di tampone di flusso.
- Aggiungere 30 µ l della sospensione del monocito in un alloggiamento della diapositiva a servire per l'impostazione dei parametri di acquisizione il microscopio confocale.
- Mantenere le aliquote delle sospensioni del monocito dal passaggio 5,18 a 37 ° C.
Nota: Questa sospensione è pronta per essere iniettato nel sistema di flusso.
6. preparazione del sistema fluidico
- Garantire che l'incubatrice di cella per l'imaging a 37 ° C.
Nota: Nella Figura 2è riportato un diagramma del sistema di flusso. - Assemblare la parte di tubazione i: inserire un connettore Luer maschio ad una estremità di un pezzo di tubo in silicone (8 cm lunghezza e 3 mm di spessore) e collegare l'altra estremità a un set di iniezione Luer in linea. Collegare il connettore Luer quest'ultimo a un pezzo di cordone di silicone (40 cm e 3 mm di spessore) ad una estremità.
Nota: Facoltativamente, un rubinetto 3 vie collegato ad una siringa da 5 mL può essere inserito tra il set di iniezione Luer in linea e il tubo per la rimozione di eventuale aria bolla in silicone. - Montare la tubazione parte II: collegare una siringa da 20 mL a un'estremità di una lunghezza del cordone di silicone (1 m di lunghezza e 3 mm di spessore). Inserire un connettore Luer maschio a altra estremità del tubo.
- Collegare la parte I e parte della tubazione II inserendo i maschi di connettore Luer per un accoppiatore di blocco Luer femmina (Figura 2A).
- Mettere l'estremità libera del tubo di silicone nel serbatoio contenente il buffer di flusso (M199 + 0,5% BSA) riscaldato a 37 ° C.
- Tirare lo stantuffo della siringa da 20 mL per riempire il tubo con buffer di flusso.
- Posizionare la siringa sulla pompa e fissarlo.
- Impostare la pompa nel prelevare modalità (in contrapposizione a infondere) e specificare la portata.
- Determinare la portata secondo la diapositiva IBIDI utilizzata, utilizzando la seguente formula:
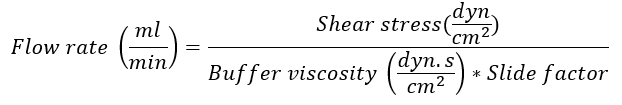
Nota: Il fattore di scorrimento dipende dalla diapositiva IBIDI utilizzata per l'esperimento. Per µ-slide ho0,4 Luer lock utilizzato in questo esempio, il fattore di diapositiva è 131,6. Per fattori di diapositiva specifica, vedere il sito Web di società14. La viscosità di buffer del flusso è 0,0072 dyn.s/cm2. Sollecitazione di taglio presso le venule post-capillari è circa 0,5 dyn/cm2. -
Collegare il vetrino (Figura 2B):
- Assicurare il tubo flessibile del silicone intorno il raccordo di blocco Luer femmina e staccare i due maschi di connettore Luer l'accoppiatore.
- Collegarli ai serbatoi della diapositiva contenente stimolato HUVEC e riempire con il mezzo. Evitare le bolle d'aria durante questo passaggio.
- Togliere i morsetti e assicurarsi che la connessione non è perdita.
- Porre il vetrino al microscopio per l'imaging di time-lapse e avviare la pompa.
7. Time-lapse Imaging del monocito reclutamento sotto flusso mediante microscopia confocale
- Utilizzare un obiettivo 40x (Vedi Tabella materiali) per l'imaging.
- Attivare il 405 nm (blu monocito nuclei), 488 nm (cellule endoteliali verde) e 561 nm (sottoinsieme di rosso CD16 +) laser.
- Utilizzare la camera contenente i monociti per impostare i parametri di acquisizione.
Nota: Per rilevare non trasmigrato e transmigrated monociti, il foro stenopeico e l'intensità del laser 405 nm sono impostati alti. Così, non trasmigrato i monociti sono leggermente visibili nel piano basale. Tuttavia soltanto transmigrated monociti presentano un'area non colorata intorno al nucleo corrispondente al nuovo spazio occupato sotto le cellule endoteliali. - Posizionare la camera per essere acquisita sotto il microscopio.
- Scegliere 3 campi di vista nel raggio di 1 cm per l'imaging confocale multiposizione.
- Definire il basale e i lati apicali delle cellule endoteliali
- Impostare un z-stack per la gamma di 10 – 12 µm (passo 0,5 µm). Eseguire un'acquisizione di time-lapse ogni 1 min.
- Dopo 3 min di imaging, è possibile iniettare 200 µ l di sospensione del monocito (6 x 106 cellule/mL) attraverso il foro di iniezione Luer in linea.
Nota: Rapidamente i monociti appaiono nel piano focale apicale, aderire e iniziare la trasmigrazione (transito da apicale al piano basale). - Immagine per almeno 30 min. Una volta finito, interrompere la formazione immagine e fermare il flusso. Assicurare il tubo flessibile per scollegarli dalla diapositiva.
- Difficoltà la diapositiva con paraformaldeide al 4% a 4 ° C per 10 min.
- Lavare il vetrino con PBS e conservare il vetrino a 4 ° C per un'ulteriore analisi se necessario.
8. analizzare i dati con ImageJ
- Contare il numero dei monociti aderenti totali in ogni campo. Determinare il conteggio delle cellule per mm2.
- Contare transmigrated monociti che sono presenti nel piano basale sotto le cellule endoteliali e identificati dalla presenza di un buco nero (nel canale verde) intorno al nucleo.
- Dividere il conteggio dei leucociti transmigrated dal numero totale di leucociti aderenti. Il tasso di trasmigrazione è presentato come una percentuale dei monociti aderenti.
- Per l'illustrazione, l'apicale e i lati basali possono essere visualizzati contemporaneamente per illustrare gli eventi che si verificano in ciascuno di questi comparti endoteliali.
Risultati
Determinazione dello stato di attivazione di HUVEC indotta dal TNFα
La bio-attività della citochina infiammatoria TNFα può variare secondo il batch e la replezione del ciclo di congelamento-scongelamento. È importante verificare lo stato di attivazione di HUVEC con trattamento TNFα. Questa potrebbe essere eseguita dalla macchiatura in parallelo alcuni campioni di HUVEC confluenti per l'induzione infiammatoria selectins, ICAM-...
Discussione
Qui, segnaliamo un metodo dettagliare uno studio di come le sottopopolazioni monocito trasmigrano attraverso il monostrato endoteliale infiammato. Il metodo discusso 4x0 microscopia confocale microscopia di contrasto di fase, che è anche usata per studiare il reclutamento di monociti sotto flusso3,11,19. Uno dei principali vantaggi dell'utilizzo di microscopia confocale per l'imaging di time-lapse è la capacità di discriminar...
Divulgazioni
Gli autori non hanno nessun concorrenti interessi finanziari.
Riconoscimenti
Si ringrazia il Dr. Paul Bradfield per manoscritto leggendo e feedback. A. S. ha ricevuto il sostegno finanziario il Signore Jules Thorn caritatevole Overseas Trust reg.,
Materiali
| Name | Company | Catalog Number | Comments |
| Tissue Culture Flasks 75 cm2 | TPP | 90076 | Routine culture of isolated HUVEC |
| µ-Slide VI 0.4 | IBIDI | 80606 | |
| Centrifuge Tubes 15 mL | TPP | 191015 | |
| Centrifuge Tubes 50 mL | TPP | 191050 | |
| Collagen G | Biochrom | L 7213 | For coating of cell culture flasks |
| Gelatin | Sigma-Aldrich | 1393 | For coating of cell culture flasks |
| Dulbecco’s Phosphate Buffered Saline (without MgCl2 and CaCl2) | Sigma-Aldrich | D8537 | |
| Dulbecco’s Phosphate Buffered Saline (with MgCl2 and CaCl2) | Sigma-Aldrich | D8662 | |
| RPMI-1640 Medium | Sigma-Aldrich | R8758 | |
| 3-Way Stopcocks | BIO-RAD | 7328103 | |
| penicillin 10,000 μ/mL streptomycine 10,000 μg/mL fungizone 25 μ/mL | AMIMED | 4-02F00-H | |
| Collagenase type 1 | Worthington | LS004216 | |
| Medium 199 1x avec Earle's salts, L-Glutamine, 25 mM Hepes | GIBCO | 22340020 | |
| Bovine Albumin Fraction V | ThermoFisher | 15260037 | |
| Endothelial Cell Growth Supplement, 150 mg | Millipore | 02-102 | |
| Heparin Sodium | Sigma-Aldrich | H3149RT | |
| Hydrocortisone | Sigma-Aldrich | H6909 | |
| L-Ascorbic acid | Sigma-Aldrich | A 4544 | |
| EDTA disodium salt dihydrate C10H14N2Na2O8 · 2H2O | APPLICHEM | A2937.0500 | |
| CD144 (VE-Cadherin), human recombinant clone: REA199, FITC | Miltenyi Biotech | 130-100-713 | AB_2655150 |
| CD31-PE antibody, human recombinant clone: REA730, PE | Miltenyi Biotech | 130-110-807 | AB_2657280 |
| Anti-Podoplanin-APC, human recombinantclone: REA446, APC | Miltenyi Biotech | 130-107-016 | AB_2653263 |
| BD Accuri C6 Plus | BD Bioscience | ||
| µ-Slide I Luer | IBIDI | 80176 | |
| CMFDA (5-chloromethylfluorescein diacetate) | ThermoFisher | C2925 | |
| Recombinant human TNFα | Peprotech | 300-01A | |
| Recombinant human VEGFA | Peprotech | 100-20 | |
| NE-1000 Programmable Syringe Pump | KF Technology | NE-1000 | |
| Ficoll Paque Plus | GE Healthcare | 17-1440-02 | |
| Anti-human CD14-PE, human recombinant clone: REA599, PE | Miltenyi Biotech | 130-110-519 | AB_2655051 |
| Pan Monocyte Isolation Kit, human | Miltenyi Biotech | 130-096-537 | |
| Anti-human CD16-PE, human recombinant clone: REA423, PE | Miltenyi Biotech | 130-106-762 | AB_2655403 |
| LS columns | Miltenyi Biotech | 130-042-401 | |
| QuadroMACS Separator | Miltenyi Biotech | 130-090-976 | |
| Hoechst 33342, Trihydrochloride, Trihydrate | ThermoFisher | H1399 | |
| Silicone tubing | IBIDI | 10841 | |
| Elbow Luer Connector | IBIDI | 10802 | |
| Female Luer Lock Coupler | IBIDI | 10823 | |
| Luer Lock Connector Female | IBIDI | 10825 | |
| In-line Luer Injection Port | IBIDI | 10820 | |
| Ar1 confocal microscope | Nikon | ||
| 40x objective | Nikon | 40x 0.6 CFI ELWD S Plane Fluor WD:3.6-2.8mm correction 0-2mm | |
| ImageJ Software | NIH |
Riferimenti
- Auffray, C., Sieweke, M. H., Geissmann, F. Blood Monocytes: Development, Heterogeneity, and Relationship with Dendritic Cells. Annual Review of Immunology. 27 (1), 669-692 (2009).
- De Palma, M., Venneri, M. A., Roca, C., Naldini, L. Targeting exogenous genes to tumor angiogenesis by transplantation of genetically modified hematopoietic stem cells. Nature Medicine. 9 (6), 789-795 (2003).
- Sidibe, A., et al. Angiogenic factor-driven inflammation promotes extravasation of human proangiogenic monocytes to tumours. Nature Communications. 9 (1), 355 (2018).
- Ley, K., Laudanna, C., Cybulsky, M. I., Nourshargh, S. Getting to the site of inflammation: the leukocyte adhesion cascade updated. Nature Review Immunology. 7 (9), 678-689 (2007).
- Nourshargh, S., Alon, R. Leukocyte Migration into Inflamed Tissues. Immunity. 41 (5), 694-707 (2014).
- Cros, J., et al. Human CD14dim monocytes patrol and sense nucleic acids and viruses via TLR7 and TLR8 receptors. Immunity. 33 (3), 375-386 (2010).
- Geissmann, F., Jung, S., Littman, D. R. Blood monocytes consist of two principal subsets with distinct migratory properties. Immunity. 19 (1), 71-82 (2003).
- Chamorro, S., et al. In vitro differentiation of porcine blood CD163− and CD163+ monocytes into functional dendritic cells. Immunobiology. 209 (1-2), 57-65 (2004).
- Passlick, B., Flieger, D., Ziegler-Heitbrock, H. Identification and characterization of a novel monocyte subpopulation in human peripheral blood. Blood. 74 (7), (1989).
- Venneri, M. A., et al. Identification of proangiogenic TIE2-expressing monocytes (TEMs) in human peripheral blood and cancer. Blood. 109 (12), 5276-5285 (2007).
- Bradfield, P. F., et al. JAM-C regulates unidirectional monocyte transendothelial migration in inflammation. Blood. 110 (7), 2545-2555 (2007).
- Schenkel, A. R., Mamdouh, Z., Muller, W. A. Locomotion of monocytes on endothelium is a critical step during extravasation. Nature Immunology. 5 (4), 393-400 (2004).
- Luu, N. T., Rainger, G. E., Nash, G. B. Kinetics of the different steps during neutrophil migration through cultured endothelial monolayers treated with tumour necrosis factor-alpha. Journal Vascular Research. 36 (6), 477-485 (1999).
- ibidi GmbH. . Shear Stress and Shear Rates for ibidi µ-Slides - Based on Numerical Calculations. , (2014).
- Yang, L., Froio, R. M., Sciuto, T. E., Dvorak, A. M., Alon, R., Luscinskas, F. W. ICAM-1 regulates neutrophil adhesion and transcellular migration of TNF-alpha-activated vascular endothelium under flow. Blood. 106 (2), 584-592 (2005).
- Yang, C. -. R., Hsieh, S. -. L., Ho, F. -. M., Lin, W. -. W. Decoy receptor 3 increases monocyte adhesion to endothelial cells via NF-kappa B-dependent up-regulation of intercellular adhesion molecule-1, VCAM-1, and IL-8 expression. Journal of Immunology. 174 (3), 1647-1656 (2005).
- Wong, D., Dorovini-Zis, K. Expression of vascular cell adhesion molecule-1 (VCAM-1) by human brain microvessel endothelial cells in primary culture. Microvascular Research. 49 (3), 325-339 (1995).
- Bradfield, P. F., Nourshargh, S., Aurrand-Lions, M., Imhof, B. A. JAM family and related proteins in leukocyte migration (Vestweber series). Arteriosclerosis Thrombosis and Vascular Biology. 27 (10), 2104-2112 (2007).
- Bradfield, P. F., et al. Divergent JAM-C Expression Accelerates Monocyte-Derived Cell Exit from Atherosclerotic Plaques. PLoS One. 11 (7), e0159679 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon