Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Quantification automatisée basée sur l'image des pièges extracellulaires neutrophiles à l'aide de NETQUANT
Dans cet article
Résumé
Ici, nous présentons un protocole pour générer des pièges extracellulaires neutrophiles (NET) et l'exploitation de NETQUANT, une option logicielle entièrement automatique pour la quantification des NET dans les images d'immunofluorescence.
Résumé
Les pièges extracellulaires de Neutrophile (NET) sont des structures antimicrobiennes web-like se composant de l'ADN et des protéines antimicrobiennes dérivées de granule. La microscopie d'immunofluorescence et les méthodes de quantification basées sur l'image demeurent des outils importants pour quantifier la formation de pièges extracellulaires neutrophiles. Cependant, il y a des limites clés aux méthodes basées sur l'immunofluorescence qui sont actuellement disponibles pour quantifier les ENI. Les méthodes manuelles de quantification NET basée sur l'image sont souvent subjectives, sujettes à l'erreur et fastidieuses pour les utilisateurs, en particulier les utilisateurs non expérimentés. En outre, les options logicielles actuellement disponibles pour la quantification sont soit semi-automatiques ou nécessitent une formation avant l'opération. Ici, nous démontrons la mise en œuvre d'une méthode automatisée de quantification d'image basée sur l'immunofluorescence pour évaluer la formation net appelée NETQUANT. Le logiciel est facile à utiliser et dispose d'une interface utilisateur graphique conviviale (GUI). Il tient compte de paramètres biologiquement pertinents tels qu'une augmentation de la surface et du rapport protéine marqueur DNA:NET, et la déformation nucléaire pour définir la formation de l'EI. En outre, cet outil est conçu comme une application librement disponible, et permet de quantifier et d'analyser la résolution d'une seule cellule.
Introduction
Les neutrophiles sont des médiateurs cruciaux des réponses innées de défense d'hôte contre une grande variété d'agents pathogènes microbiens1. Ils exécutent leurs fonctions antimicrobiennes en libérant leurs granules contenant un large éventail de protéines antimicrobiennes2, produisant des espèces réactives d'oxygène (ROS) et de l'hypochlorite1, et par phagocytose3. En outre, Brinkmann et coll. 4 ont décrit les pièges extracellulaires de neutrophile (NETs) comme mécanisme nouveau par lequel les neutrophiles piègent et éliminent les pathogènes envahissants. Depuis leur découverte il y a un peu plus d'une décennie4, les TN ont été impliqués dans une grande variété de morbidités infectieusesde 5,6 et non infectieuses7. La formation de NET est un processus actif et entraîne l'extrusion de l'ADN de chromatine enduit de protéines antimicrobiennes dérivées du granule8. Certains des changements clés dans la morphologie cellulaire et nucléaire associée à la formation net comprennent la perte de morphologie nucléaire, la décondensation de chromatine, la mobilisation des protéines granules du cytoplasme au noyau et une augmentation du diamètre nucléaire et cellulaire8,9.
Les NET sain, qui peuvent apparaître comme des structures diffuses légèrement plus grandes que la cellule ou comme des structures plusieurs fois plus grandes qu'un seul neutrophile sont considérées comme des indicateurs de NETosis5,10. À l'aide de la microscopie à fluorescence, les TNE peuvent être détectés en sondant l'ADN à l'aide d'une sonde fluorescente comme l'élastase de 4',6-diamidino-2-phenylindole (DAPI) et par la coloration immunofluorescence contre les protéines liées au NET comme l'élastase de neutrophile. La quantification des zones de coloration qui se chevauchent pour l'ADN et les protéines liées à l'EI détermine la superficie totale sous les EE dans une image11.
Un certain nombre d'options d'analyse d'image sont disponibles pour effectuer la quantification basée sur l'image de fluorescence des NETs11,12. Mais ces options logicielles présentent des limites en n'étant pas convivialet et/ou entièrement automatisé. Dans cet article, nous démontrons le fonctionnement de NETQUANT13, une application librement disponible qui peut effectuer une microscopie immunofluorescence entièrement automatisée impartiale sur l'image de la quantification NET. L'application dispose d'une interface graphique conviviale (GUI) et peut effectuer une analyse monocellulaire. Le logiciel quantifie neTosis dans une image en détectant les changements morphologiques dans le domaine du marqueur lié à l'ADN-NET, la décondensation de chromatine a associé la déformation du noyau et l'augmentation du rapport protéine LIÉ à l'ADN: NET. Pris ensemble, les multiples critères de définition NET permettent une quantification NET rigoureuse sur plusieurs ensembles de données de manière impartiale.
Protocole
Le comité d'éthique de l'Université de Lund a approuvé la collecte de sang veineux auprès de volontaires en bonne santé conformément à la Déclaration d'Helsinki (2013/728). Tous les bénévoles ont donné leur consentement éclairé écrit.
1. Isolement des neutrophiles périphériques de sang utilisant la centrifugation de densité-gradient
- Recueillir le sang veineux humain dans des tubes contenant de l'héparine et permettre aux tubes d'atteindre la température ambiante.
Remarque : Un minimum de 16 ml de sang provenant d'un donneur en bonne santé est nécessaire pour produire une pastille cellulaire suffisamment grande. - Mélanger le sang avec un volume de 2% de dextran dans saline (0,9% NaCl) et laisser les sédiments à température ambiante pendant 30 min dans un tube stérile de centrifugeuse conique de 50 ml.
- Aspirez le supernatant dans un tube de centrifugeuse conique stérile de 50 ml et une centrifugeuse à 200 x g pendant 10 min à 4 oC.
- À partir de cette étape, continuer l'isolement à 4 oC ou sur la glace.
- Resuspendre la pastille dans 5 ml de saline glacée et la couche au-dessus de 5 ml de gradient d'isolement des leucocytes (9,1 % de diatrizoat de sodium avec 5,7 % de dextran, w/v) dans un tube stérile de centrifugeulation conique de 15 mll.
- Centrifugeuse de 30 min à 400 x g à 4 oC.
- Aspirez le supernatant et jetez-le.
- Lyser les globules rouges en rependant les granulés dans 3 ml d'eau glacée pendant 30 s. Ajouter immédiatement 1 ml de 3,6 % de NaCl, puis remplir de 10 ml de salin glacé.
- Centrifugelant la suspension cellulaire pendant 10 min à 350 x g.
- Retirez le supernatant, collectez le granule cellulaire et suspendez-le en 1 ml de salin. Réserver 10 L dans un tube de microcentrifuge pour l'évaluation du nombre de cellules et de la viabilité à l'aide du bleu trypan dans une chambre de Bûrker.
- Ajouter 10 l de suspension cellulaire à 90 'L de 0,4% de solution bleu trypan. Prendre 10 ll de la suspension de la cellule dans une chambre de Bûrker. Comptez les cellules dans les 4 carrés liés par 3 lignes à chaque coin de la chambre. Les cellules qui semblent bleu foncé à l'utilisation de colorant ne sont pas viables, les excluent du nombre total de cellules.
- Exprimez le nombre de cellules en tant que cellules/mL tel que défini par l'équation ci-dessous.
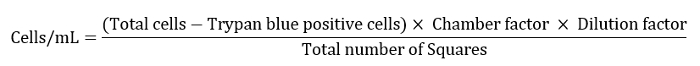
Note : Ici, le facteur de chambre était de 10 000, le facteur de dilution était de 10 et le nombre total de carrés était de 4. - Diluer la suspension de la cellule restante à 10 ml pour une dernière étape de lavage.
- Centrifugeuse de 5 min à 200 x g.
- Resuspendre les neutrophiles dans RPMI-1640 avec 2 mg/mL d'albumine de sérum humain inactivée par la chaleur (HSA) à une concentration de 5 x 105 cellules/mL.
2. Préparation des fiches de couverture et stimulation des neutrophiles
- Placez un bordereau (10 mm, #1) dans chaque puits d'une assiette de 12 puits et enrobez le bordereau en ajoutant 200 ll de la solution poly-L-lysine de 0,01 % et laissez-le à 37 oC pendant la nuit.
- Laver les feuilles de couverture avec une saline tampon de phosphate de 300 l (PBS) une fois et laisser sécher.
- Ajouter 400 oL de 5 x 105 neutrophiles/mL à chaque puits et incuber à température ambiante pendant 15 min.
- Déplacer la plaque contenant des neutrophiles vers un incubateur à 37 oC avec 5 % de CO2 pendant 15 min.
- Retirez le supernatant. Ajouter 400 ll de rpMI-1640 moyen préréchauffé avec 2 mg/mL HSA pour les contrôles. Ajouter 300 L de RPMI préréchauffé avec 20 nM phorbol 12-myristate 13-acétate (PMA) pour la stimulation.
- Stimuler les neutrophiles pendant 150 min à 37 oC avec 5 % de CO2.
3. Visualisation des EI
- Retirer le supernatant et laver les échantillons 2x avec 200 L de PBS.
- Fixer les échantillons en ajoutant 200 oL de 4 % de paraformaldéhyde (PFA) en PBS pendant 20 min à 37 oC.
Remarque : L'APF est toxique et doit être manipuléavec soin. - Laver les échantillons 3x avec 200 l de PBS.
- Perméabilisez les échantillons en ajoutant 50 L de 0,5 % Triton X-100 pour 30 s.
- Laver les échantillons 3x avec 200 l de PBS.
- Bloquer les échantillons avec 5% de sérum de chèvre en PBS pendant 1 h à 37 oC.
- Ajouter 300 l d'élastase de neutrophile anti-humain primaire de lapin dans la solution de blocage à une dilution de 1:500 pendant 90 min à 37 oC.
- Laver les échantillons 3x avec 300 l l de PBS.
- Ajouter 300 l d'anticorps fluorescents anti-lapin de chèvre secondaire à une dilution de 1:1000 pendant 90 min à 37 oC.
- Laver les couvertures 3x avec 300 L de PBS.
- Retirez les couvertures des puits et montez la glissière de couverture avec un support de montage de 10 l contenant du DAPI. Conserver toute la nuit à température ambiante dans l'obscurité pour sécher les échantillons.
Note: La coloration de l'ADN avec DAPI est certainement une étape critique dans la méthode. Les utilisateurs peuvent également dépanner en ajoutant une solution DAPI exogène à une plage de concentration finale de 0,1 'u20120.5 'g/mL pour 2'u20123 min, suivie de 3 étapes de lavage avec 300 'L PBS. - Acquérir des images avec un microscope à fluorescence à large champ à l'aide d'un objectif 20X.
4. Analyse et quantification des NETÀ à l'aide de NETQUANT
Remarque : NETQUANT peut être téléchargé en cliquant sur le fichier d'installation trouvé sur les archives Zenodo Github ou sur le site Web du Nordenfelt Lab(https://nordlab.med.lu.se/?page_id=34).
- Importation de jeux de données pour l'analyse, la dénomination des canaux et la conversion d'images
- Ouvrez l'onglet Setup dans NETQUANT.
- Choisissez le dossier source pour l'analyse en cliquant sur l'option Get path dans le menu source et sélectionnez le dossier contenant les séquences d'images à analyser.
- Cliquez sur l'option Get Path dans le menu cible et sélectionnez le dossier pour enregistrer les données suivant l'analyse d'image.
- Nommez les canaux de sorte que le « canal d'ADN » correspond à la coloration de l'ADN(par exemple,ADN ou DAPI) et « NET-canal » représente la coloration de protéine LIÉE à NET(par exemple,NET, elastase de neutrophile) dans les images. Pour le bon fonctionnement du logiciel, (recommandé) nom du dossier contenant les fichiers d'image de contrôle comme «contrôle».
Remarque : Le canal NET fait référence à la coloration du marqueur de protéine granule lié à NET seulement. - Alimentez les métadonnées d'image dans le logiciel en cliquant sur le bouton d'information d'image de charge sur le sous-menu d'informations d'image.
- Sélectionnez l'ordre de canal correct contenu dans les images dans le sous-menu de l'ordre Channel. Cette option a été incluse comme un coffre-fort pour prévenir les inadéquations accidentelles.
- Acquérir des propriétés d'image primaire à partir des données brutes et convertir les images en cliquant sur le bouton De données Préparer. Les images converties apparaissent dans le sous-menu de type Exemple. Cliquez sur le menu type Exemple pour afficher et sélectionner tous les jeux de données acquis pour analyse.
- Sélectionnez une image du menu sous-modèle de type Exemple et cliquez sur le bouton de données d'image Display pour afficher les images divisées en ADN et net respectivement.
- Segmentation des cellules dans le canal de l'ADN et le canal NET
- Sélectionnez la méthode de segmentation en cliquant sur le sous-menu Méthode dans le canal DNA et le canal NET.
Remarque : La méthode de segmentation par défaut est adaptée et constitue le paramètre recommandé. D'autres options sont également disponibles, y compris global, edge et Chan-Vese. Une option de bassin versant est également incluse pour aider à distinguer les cellules étroitement placées ou les NET. - Entrez l'onglet Segmentation pour segmenter les cellules de contrôle d'abord dans les deux canaux en cliquant sur l'option Échantillons de contrôle segment.
- Sélectionnez PMA dans le sous-menu de type d'échantillon et cliquez sur l'option Batch (recommandé) pour commencer la segmentation de toutes les images incluses dans l'ensemble de données. Sélectionnez les images dans le menu de type échantillon et cliquez sur le bouton de données d'image Display pour visualiser et valider les masques d'image binaires (masque d'ADN et masque NET) générés post-segmentation.
- Sélectionnez la méthode de segmentation en cliquant sur le sous-menu Méthode dans le canal DNA et le canal NET.
- Analyse unicellulaire des propriétés identifiables
- Entrez l'onglet d'analyse et analysez les échantillons de contrôle en cliquant sur le bouton Déterminer le seuil.
- Changez le type d'échantillon en PMA et cliquez sur le bouton Obtenir les propriétés cellulaires pour compléter l'analyse des échantillons stimulés.
- Sélectionnez une image du sous-menu de type Exemple et cliquez sur le bouton de données d'image Display pour afficher la superpose et le nombre de cellules et de cellules de formation NET dans l'image.
- Comparaison des propriétés cellulaires pour identifier les cellules qui forment net
- Sélectionnez l'échantillon du sous-menu de type d'échantillon et cliquez sur le bouton Analyse NETs pour compléter l'analyse. Les images individuelles peuvent être sélectionnées à partir du sous-menu de type d'échantillon pour analyse ou l'ensemble du lot d'images peut être analysé en sélectionnant l'option de lot (recommandé).
- Ajuster manuellement les critères NET pour obtenir des résultats optimaux pour un échantillon donné. Comparez les ENE identifiés avec les images originales afin d'évaluer la qualité de l'identification.
Remarque : Les critères NET peuvent ensuite être utilisés sur toutes les images de l'ensemble de données. Toute modification des critères NET est appliquée simultanément dans tous les échantillons témoins. Cela limite la possibilité de toute différence potentielle qui peut survenir en raison du sur-ajustement des paramètres NET. Les paramètres des critères NET peuvent être ajustés en fonction des exigences de l'utilisateur. La relation entre les taux de fausses découvertes et NETQUANT a été explorée précédemment13. Les plages typiques pour l'augmentation de la superficie sont de 2 u20124, la circularité à 0,7 u20120.9 et le ratio ADN/NET à 0,6-2,0. - Inspectez le résumé des données dans le sous-menu de données cellulaires où le nombre d'images, le nombre de cellules par image et le pourcentage de NETs par image sont affichés.
Remarque : Le pourcentage total d'EE dans l'ensemble des données est affiché par la « jauge NET ». Nombre total d'images, nombre de cellules, pourcentage d'EE dans l'échantillon (NET%) et les NET% de l'échantillon témoin sont affichés dans le tableau des statistiques sommaires sous la jauge NET. Nous recommandons que les données de contrôle soient communiquées parallèlement aux données obtenues à partir d'échantillons stimulés.
- Résultats Sorties
- Entrez l'onglet Sortie pour sélectionner et afficher les sorties de résultats.
- Explorez et comparez les différentes sorties de données générées par l'analyse du contrôle et de la PMA en sélectionnant la forme de la sortie et en cliquant sur le bouton Résultats de sortie.
Remarque : Toutes les données générées après l'analyse pour les contrôles et les stimulations sont enregistrées dans le dossier d'analyse choisi dans le sous-menu cible. Les données sont enregistrées dans des formats .csv ou .pdf. - Lancer le fichier Méthode pour obtenir la version du logiciel et des critères NET utilisés pour l'analyse (à inclure dans la section méthodes à des fins de publication).
- Cliquez sur le tableau des données Résultats pour visualiser les points de données individuels d'un échantillon donné.
- Visualisez la distribution de la zone NET et le rapport ADN:NET dans les échantillons. La ligne rouge indique la valeur seuil dans les graphiques.
- Déterminez la zone NET par rapport à la forme de l'ADN en cliquant sur le fichier de distribution Bivariate.
- Chargement de l'analyse précédente et de la production de toutes les étapes
- Chargez les paramètres d'analyse précédemment réussis dans NETQUANT à l'aide du bouton d'analyse précédente Charge.
- Utilisez le bouton Batch toutes les étapes inclus dans le menu de configuration pour exécuter les étapes 5-u201212 (Figure 1, Figure 2, Figure 3, Figure 4, Figure 5) directement pour obtenir la sortie finale.
Résultats
5 x 105 neutrophiles/mL ont été ensepépins sur des feuilles de couverture placées dans une plaque de 12 puits et stimulées avec 20 nM PMA ou laissées non stimulées pendant 150 min. Les échantillons ont ensuite été tachés à l'aide d'anticorps primaires d'élastase de neutrophiles de lapin, d'anticorps conjugués secondaires de fluorophore anti-lapin de chèvre et d'anticorps conjugués d'API - un colorant fluorescent étiqueté qui tache l'ADN (voir le tablea...
Discussion
La formation de NET est un ajout relativement récent à l'armamentarium neutrophile diversifié4 et il y a eu un regain d'intérêt notable pour étudier l'implication des NET dans un large éventail de domaines de recherche5,7,14,15. L'acquisition d'images à l'aide de la microscopie immunofluorescence et de la quantification à base d'images subséquente est une méth...
Déclarations de divulgation
TM et PN ont un brevet en instance lié aux algorithmes utilisés dans NETQUANT.
Remerciements
Les travaux ont été financés par la Fondation Crafoord (TM et PN), la subvention du gouvernement suédois pour la recherche (PN, TM), le Conseil suédois de la recherche (PN) et la Fondation Groschinsky (TM, PN).
matériels
| Name | Company | Catalog Number | Comments |
| BD Vacutainer Heparinised plastic tubes | BD Biosciences | 367885 | |
| Lymphoprep | Axis-Shield | 114547 | |
| RPMI-1640 with L-Glutamine | Gibco | 11835-030 | |
| 50mL conical flasks | Sarstedt | 62.547.004 | |
| 15mL conical flasks | Sarstedt | 62.554.002 | |
| 12-well Tissue culture plates | Falcon | 10626491 | |
| #1 Coverslips 10mm | Menzel Glaser | CS10100 | |
| Glass slides | Menzel Glaser | 631-0098 | |
| Primary anti-human elastase | DAKO | DAKO rabbit 1373, contract immunization | |
| Secondary fluorophore conjugated goat anti-rabbit | Life technologies | A-11072, A-11070 | |
| PROLONG-Gold Antifade reagent with DAPI | Life technologies | P36930 | Mounting medium |
| Goat serum | Sigma-Aldrich | G9023 | |
| Phorbol 12-myristate 13-acetate (PMA) | Sigma-Aldrich | 79346 | |
| Paraformaldehyde | Sigma-Aldrich | 158127 | |
| Triton X-100 | Sigma-Aldrich | T8787 | |
| Nikon Ti-E Epifluorescence microscope | Nikon | ||
| CCD camera | Andor Zyla | ||
| Plan Apochromat 20x, 40x objectives | Nikon | ||
| Windows 10 | Microsoft | Operating system | |
| macOS Sierra 10.12 | Apple | Operating system | |
| MATLAB | Mathworks |
Références
- Nauseef, W. M., Borregaard, N. Neutrophils at work. Nature Immunology. 15 (7), 602-611 (2014).
- Borregaard, N., Cowland, J. B. Granules of the human neutrophilic polymorphonuclear leukocyte. Blood. 89 (10), 3503-3521 (1997).
- Nordenfelt, P., Tapper, H. Phagosome dynamics during phagocytosis by neutrophils. Journal of Leukocyte Biology. 90 (2), 271-284 (2011).
- Brinkmann, V., et al. Neutrophil extracellular traps kill bacteria. Science. 303 (5663), 1532-1535 (2004).
- Sorensen, O. E., Borregaard, N. Neutrophil extracellular traps - the dark side of neutrophils. Journal of Clinical Investigation. 126 (5), 1612-1620 (2016).
- Yipp, B. G., Kubes, P. NETosis: how vital is it?. Blood. 122 (16), 2784-2794 (2013).
- Jorch, S. K., Kubes, P. An emerging role for neutrophil extracellular traps in noninfectious disease. Nature Medicine. 23 (3), 279-287 (2017).
- Fuchs, T. A., et al. Novel cell death program leads to neutrophil extracellular traps. J Cell Biol. 176 (2), 231-241 (2007).
- Mohanty, T., et al. A novel mechanism for NETosis provides antimicrobial defense at the oral mucosa. Blood. 126 (18), 2128-2137 (2015).
- Hakkim, A., et al. Activation of the Raf-MEK-ERK pathway is required for neutrophil extracellular trap formation. Nature Chemical Biology. 7 (2), 75-77 (2011).
- Brinkmann, V., Goosmann, C., Kuhn, L. I., Zychlinsky, A. Automatic quantification of in vitro NET formation. Frontiers in Immunology. 3, 413 (2012).
- Coelho, L. P., et al. Automatic determination of NET (neutrophil extracellular traps) coverage in fluorescent microscopy images. Bioinformatics. 31 (14), 2364-2370 (2015).
- Mohanty, T., Sorensen, O. E., Nordenfelt, P. NETQUANT: Automated Quantification of Neutrophil Extracellular Traps. Frontiers in Immunology. 8, 1999 (2017).
- Kaplan, M. J., Radic, M. Neutrophil extracellular traps: double-edged swords of innate immunity. Journal of Immunology. 189 (6), 2689-2695 (2012).
- Cedervall, J., Olsson, A. K. Immunity Gone Astray - NETs in Cancer. Trends in Cancer. 2 (11), 633-634 (2016).
- Ginley, B. G., et al. Computational detection and quantification of human and mouse neutrophil extracellular traps in flow cytometry and confocal microscopy. Scientific Reports. 7 (1), 17755 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon