Method Article
Évaluer la capacité de différenciation des cellules épithéliales de la prostate de la souris à l'aide de la culture organoïde
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Les organoïdes de prostate de souris représentent un contexte prometteur pour évaluer des mécanismes qui règlent la différenciation. Cet article décrit une approche améliorée pour établir des organoïdes de prostate, et introduit des méthodes pour (1) recueillir le lysate de protéine des organoïdes, et (2) fixer et tacher des organoïdes pour la microscopie confocale de monture entière.
Résumé
L'épithélium de la prostate est composé principalement de cellules basales et lumineuses. Le tracé in vivo de lignée a été utilisé pour définir la capacité de différenciation des cellules basales et lumineuses de prostate de souris pendant le développement, la régénération de tissu et la transformation. Cependant, l'évaluation des régulateurs cellulaires et extrinsèques de la capacité de différenciation épithéliale de la prostate à l'aide d'une approche de traçage de lignée nécessite souvent une reproduction intensive et peut être prohibitive sur le plan des coûts. Dans l'analyse organoïde de prostate, les cellules basales et lumineuses génèrent l'épithélium prostatique ex vivo. Fait important, les cellules épithéliales primaires peuvent être isolées de souris de tout milieu génétique ou de souris traitées avec un certain nombre de petites molécules avant ou après, le placage dans la culture tridimensionnelle (3D). Le matériel suffisant pour l'évaluation de la capacité de différenciation est produit après 7-10 jours. La collecte d'organoïdes basaux et d'organoïdes d'origine luminale pour (1) l'analyse des protéines par western blot et (2) l'analyse immunohistochimique des organoïdes intacts par microscopie confocale à monture entière permet aux chercheurs d'évaluer la différenciation ex vivo capacité des cellules épithéliales de prostate. Lorsqu'elles sont utilisées en combinaison, ces deux approches fournissent des informations complémentaires sur la capacité de différenciation des cellules basales et lumineuses de la prostate en réponse à la manipulation génétique ou pharmacologique.
Introduction
Les cellules basiques et lumineuses constituent la majorité de l'épithélium de la prostate1. Les études de traçage de lignée ont indiqué que ces types de cellules sont principalement auto-soutenus par des progéniteurs distincts dans la souris adulte2; cependant, la différenciation luminale des progéniteurs basaux a été observée dans plusieurs contextes comprenant le développement3,4, régénération de tissu5, inflammation6,7 et initiation de cancer de la prostate2,8. En outre, les données émergentes soutiennent l'existence d'ancêtres luminaires multipotents ainsi que d'ancêtres luminairesengagés 9. Dans le cancer de la prostate métastatique, la différenciation d'une lignée luminal AR-dépendante à une lignée AR-indifférente avec des dispositifs basal et neuroendocrines représente un mécanisme de plus en plus apprécié de résistance aux inhibiteurs de voie d'androgène10,11,12. Par conséquent, comme la différenciation est impliquée dans la physiologie normale, l'initiation du cancer et la résistance à la thérapie, il est essentiel d'élucider les principaux régulateurs moléculaires de la différenciation épithéliale des cellules de la prostate.
Le modèle organoïde de prostate de souris a émergé comme contexte ex vivo élégant pour étudier la différenciation épithéliale de cellulesdeprostate 9,13,14. Dans cet analyse, les cellules épithéliales individuelles sont plaquées dans une matrice 3D où elles génèrent des structures glandulaires contenant des cellules basales et luminales dans un délai de 1 semaine. Bien que les approches existantes pour placage des cellules dans la culture organoïde puissent être utilisées pour générer efficacement des organoïdes, ces approches nécessitent une optimisation plus poussée14. Les défis notables liés à la culture des organoïdes de la prostate comprennent (1) l'exclusion des colonies bidimensionnelles (2D) qui se forment sous le Matrigel (gel de matrice) de l'analyse, (2) le maintien de l'intégrité du gel de matrice pendant les changements de médias, et (3) le comptage des organoïdes avec précision. Cet article décrit une approche pour produire des organoïdes à partir de cellules épithéliales isolées de la prostate de souris. L'approche décrite consiste en plaques de revêtement avec du méthacrylate poly(2-hydroxyethyl) (Poly-HEMA) pour empêcher l'apparition de colonies 2D. En outre, les cellules sont plaquées dans un anneau de gel de matrice, plutôt qu'un disque de gel de matrice, qui rend le changement du support et le comptage des organoïdes moins provocants. Ces techniques permettent aux chercheurs d'étudier plus facilement comment les altérations génétiques ou les petites molécules introduites avant ou pendant la formation d'organoïdes modifient les processus clés tels que la différenciation.
La récolte des organoïdes de prostate pour l'analyse occidentale de tache ou immunohistochemical par la microscopie confocale de monture entière peut fournir la perspicacité mécaniste valable dans la différenciation13,pourtant des protocoles bien établis pour préparer des organoïdes pour de telles techniques manquent. Ce manuscrit décrit les approches de la récolte des organoïdes pour (1) la collecte de lysate protéique, ou (2) la fixation et la coloration pour la microscopie confocale. Fait important, l'approche décrite pour fixer et tacher les organoïdes de la prostate est considérablement améliorée par rapport aux méthodes existantes. Bien que ceux-ci reposent sur la section des organoïdes15, la méthode décrite dans ce manuscrit utilise des organoïdes intacts, ce qui aide à protéger contre les dommages organoïdes pendant la préparation de l'échantillon. Lorsqu'il est utilisé en combinaison, western blot et microscopie confocale peut fournir un aperçu précieux dans les régulateurs moléculaires de la différenciation. Alternativement, ces approches peuvent être utilisées pour modéliser d'autres processus tels que le développement et la transformation.
Protocole
Toutes les méthodes décrites ici ont été approuvées par l'Institutional Review Board de l'Université de Californie à Los Angeles.
REMARQUE: Un schéma illustrant les approches décrites dans l'article est fourni à la figure 1.
1. Isoler la souris Basal et Luminal Prostate Epithelial Cells Using Fluorescence-activated Cell Sorting (FACS) — TIMING: 30 Min
REMARQUE: Effectuer des étapes 1.3-1.5 dans l'obscurité.
- Après avoir dissocié les cellules de la prostate totale de la souris, tel que décrit dans Lawson etal. 16, transférer les cellules aux tubes FACS et resuspendre 0,1-5 x 106 cellules dans 100 l de médias de dissociation (Tableau 1).
- Ajoutez le volume approprié des anticorps primaires directement conjugués suivants : CD45, CD31, Ter-119, EpCAM et CD49f.
- Incuber sur glace, à l'abri de la lumière, pendant 20 min.
REMARQUE: Il est recommandé d'utiliser 10% du total des cellules dissociées pour les contrôles non tachés et non tachés. Ces contrôles sont nécessaires pour définir la compensation correcte et la tension pour le tri. - Désincer le cocktail d'anticorps en ajoutant 1 ml de support de dissociation à chaque échantillon. Pelleter les cellules par centrifugation à 800 x g pendant 5 min à température ambiante (RT) et retirer le supernatant en aspirant.
- Resuspendre les cellules en volume approprié (250 l par 1 x 106 cellules) de la dissociation du support contenant 1 g/mL 4',6-diamidino-2-phenylindole (DAPI). Passez à FACS. Les parcelles de cytométrie de flux démontrant l'isolement des cellules épithéliales de la prostate basale et luminale de souris sont illustrées dans la figure 2.
2. Plating Sorted Prostate Epithelial Cells into Primary Mouse Organoid Culture — TIMING: 2-3 H (à l'exclusion de la préparation des plaques enduites de poly-HEMA)
REMARQUE: Les plaques sont recouvertes de Poly-HEMA pour empêcher la formation de colonies 2D à la surface du puits sous le gel de matrice. Préparer des plaques enduites de Poly-HEMA 1 jour avant de placage rentrent les cellules épithéliales de la prostate basales ou luminales dans la culture organoïde de souris. Décongeler 1 mL d'aliquots de gel de matrice de facteur de croissance réduit, ci-après appelé gel de matrice, sur la glace 2 h avant l'étape 2.1. Y-27632 (inhibiteur de ROCK) doit être ajouté au support organoïde de souris immédiatement avant l'étape 2.1. Effectuer des étapes 2.1-2.8 sur la glace.
- Pelleter les cellules dans des tubes à fond rond de 5 ml par centrifugation à 800 x g pendant 5 min à 4 oC et aspirer le supernatant.
- Laver la pastille cellulaire dans 500 oL de la souris organoïde (tableau 2)14.
- Pelleter les cellules par centrifugation à 800 x g pendant 5 min à 4 oC et aspirer le supernatant.
- Resuspendre dans les médias organoïdes de souris à une densité cellulaire de 1 000 cellules/L.
- Pour préparer les mélanges principaux, mélanger les cellules épithéliales suspendues dans les organes organoïdes de souris avec le gel de matrice pour générer un mélange final qui contient 25% de cellules/médias et 75% de gel de matrice. Les cellules basales sont généralement plaquées à une concentration de 100 à 2 000 cellules/80 L, tandis que les cellules luminales sont généralement plaquées à une concentration de 2 000 à 10 000 cellules/80 l. La densité des cellules plaquées varie selon le jour de la collecte prévue des matériaux et l'application en aval souhaitée.
REMARQUE: Refroidir les tubes de taille appropriée pour le volume de mélange maître prévu 5 min avant la préparation du mélange maître. Pour s'assurer que le gel de matrice ne durcit pas pendant la manipulation, il est essentiel de refroidir la pointe de pipette en pipetting le gel de matrice 3-4 fois avant de le transférer à un nouveau tube. - Ajouter 80 l de gel de matrice/mélange cellulaire par puits d'une plaque de 24 puits. Il est recommandé d'acheminer une gouttelette sur la moitié inférieure du mur du puits, tout en évitant tout contact direct avec le revêtement Poly-HEMA. Après l'ajout du gel de matrice, faites tourbillonner la plaque pour permettre au gel de matrice/mélange cellulaire de former un anneau autour du bord du puits.
- Placer la plaque de 24 puits dans un incubateur co2 de 37 oC 5 % vers le haut pendant 10 min pour permettre au gel de matrice de durcir partiellement.
REMARQUE: Commencer à réchauffer les organes de la souris à 37 oC immédiatement après avoir placé la plaque de 24 puits dans l'incubateur. - Après avoir incubé pendant 10 min, retourner la plaque de 24 puits à l'envers et incuber pendant 50 min supplémentaires pour permettre au gel de matrice de durcir complètement.
- Ajouter 350 l de milieux organoïdes de souris préréchauffés dans le sens de la chute au centre de chaque puits.
REMARQUE: Pour maintenir l'intégrité du gel de matrice, il est essentiel d'éviter l'anneau de gel de matrice tout en ajoutant le média. - Après l'ajout du support, retournez la plaque de 24 puits à l'incubateur de CO2 de 37 oC 5 %.
3. Reconstitution des médias organoïdes de souris — TIMING : 10-15 min par plaque de 24 puits
REMARQUE: Les médias existants devraient être remplacés par des médias frais tous les 48 heures. Avant chaque changement de média, les médias organoïdes de souris pré-chauds. Il n'est pas nécessaire d'ajouter l'inhibiteur ROCK aux supports utilisés pour le réapprovisionnement.
- Inclinez la plaque de 24 puits à un angle de 45 degrés et retirez délicatement les milieux existants du centre de chaque puits à l'aide d'une pipette p1000, tout en évitant l'anneau de gel de matrice.
- Ajouter 350 l de supports organoïdes de souris préréchauffés comme à l'étape 2.9. Il est recommandé d'ajouter un plus grand volume de médias (jusqu'à 1 ml) aux organoïdes cultivés pendant plus de 5 jours afin d'éviter l'épuisement rapide des nutriments clés et des facteurs de croissance.
4. Extraction de lysate protéique à partir d'organoïdes de la prostate pour l'analyse western Blot — TIMING: 2.5-4 H
REMARQUE: Avant de recueillir des organoïdes pour l'extraction du lysate protéique, préparez et pré-réchauffez les supports contenant des dispase (tableau 1).
- Retirez les médias de chaque puits ainsi que dans l'étape 3.1.
- Pour recueillir les organoïdes, soufflez à plusieurs reprises le gel de matrice en pipetting 1 ml de médias contenant de la dispase directement sur l'anneau de gel de matrice jusqu'à ce que l'anneau entier soit délogé, et transférer à un tube de microcentrifuge de 1,5 ml.
REMARQUE: Il est essentiel d'éviter tout contact direct avec les puits enduits poly-HEMA. Le contact direct peut causer la contamination du matériel recueilli avec Poly-HEMA, ce qui pourrait avoir un impact négatif sur la survie des cellules. - Placer le tube microcentrifuge de 1,5 ml dans un incubateur de CO2 de 37 oC de 5 % pendant 30 min à 1 h pour permettre une digestion complète du gel matriciel par dispase.
- Pellet organoïdes par centrifugation à 800 x g pendant 5 min à RT et retirer le supernatant à l'aide d'une micropipette.
- Ajouter la saline tamponnée de phosphate (PBS) à la pastille organoïde et la resuspendre en clignotant doucement.
REMARQUE: Le défaut de resuspendre suffisamment le granule organoïde peut entraîner la contamination du matériel organoïde avec le dispase résiduel ou le gel de matrice. - Pelleter les organoïdes par centrifugation à 800 x g pendant 5 min à RT et retirer le supernatant à l'aide d'une micropipette.
- Congelez rapidement les granulés d'organoïdes en plaçant chaque tube dans une solution contenant de la glace sèche et du méthanol. Conserver le tube jusqu'à l'utilisation future à -80 oC. Alternativement, extraire le lysate de protéine immédiatement après l'étape 4.6.
- Resuspendre les granulés d'organoïdes dans 100 l de tampon de lyse protéique (tableau 1) par 10 l de volume de cellules emballées. Flick pour resuspendre.
REMARQUE: Si vous reprenez après la congélation rapide, assurez-vous que le tampon de lyse protéique est décongelé avant de retirer les échantillons de -80 oC, car le tampon de lyse doit être ajouté aux échantillons immédiatement afin de prévenir l'activité de la phosphatase et de la protéase. - Incuber les échantillons dans un tampon de lyse protéique sur glace pendant au moins 45 minutes.
REMARQUE: Il est recommandé de sonicate avant l'incubation sur la glace pour augmenter l'efficacité de la récupération des protéines nucléaires; cependant, la sonication n'est pas nécessaire. Si la sonication n'est pas effectuée, passez à l'étape 4.10.- Pour sonicate, immerger les tubes dans la glace humide et appliquer délicatement la pointe du dismembrar sonique à l'extérieur du tube microcentrifuge. Sonicate pour 40 s à 20 kHz.
- Procédez à Western blot en suivant les protocoles établis.
5. Fixation et coloration des organoïdes de la prostate pour l'analyse immunohistochimique par microscopie Confocal à monture entière
-
Collecte d'organoïdes de la prostate à partir de plaques de 24 puits — TIMING : 45-60 min
REMARQUE: Lors de la collecte des organoïdes de la prostate pour traiter la microscopie confocale, il est essentiel de les manipuler avec soin afin de maintenir leur structure. Le protocole de collecte ci-dessous est conçu pour réduire la perturbation de la structure organoïde pendant l'isolement.- Retirez les médias de chaque puits ainsi que dans l'étape 3.1.
- Diglez le gel de matrice en couvant avec 500 l de supports contenant de la dispase (tableau 1) pendant 30 min dans un incubateur de CO2 de 37 oC à 5 %.
- Recueillir la suspension organoïde digérée dans un tube de microcentrifuge et granuler les organoïdes par centrifugation à 800 x g pendant 3 min à RT. Retirez le supernatant.
-
Coloration immunofluorescente à monture entière des organoïdes de la prostate — TIMING : 3-4 jours (1-5 h/jour)
- Ajouter 500 l de paraformaldéhyde de 4 % dans pbS et incuber pendant 2 h à RT avec des secousses douces.
- Pelleter les organoïdes par centrifugation à 800 x g pendant 3 min à RT, enlever le supernatant, et laver la pastille avec 1 ml de PBS pendant 15 min avec des secousses douces.
- Laver la pastille comme à l'étape 5.2.2 pour deux fois supplémentaires.
- Pelleter les organoïdes par centrifugation à 800 x g pendant 3 min à RT et enlever le supernatant. Ajouter 1 DAPI g/mL dans la solution de blocage (tableau 1). Incuber pendant 2 h à RT ou alternativement toute la nuit à 4 oC avec des secousses douces.
- Pelleter les organoïdes par centrifugation à 800 x g pendant 3 min à RT et enlever le supernatant. Ajouter l'anticorps primaire (lapin anti-p63, souris anti-cytokératine 8) dans la solution de blocage et incuber toute la nuit à 4 oC avec des secousses douces.
- Pelleter les organoïdes par centrifugation à 800 x g pendant 3 min à RT et enlever le supernatant. Laver la pastille avec 1 ml de PBS pendant 15 min avec des secousses douces.
- Laver la pastille comme à l'étape 5.2.6 pour deux fois supplémentaires.
- Pelleter les organoïdes par centrifugation à 800 x g pendant 3 min à RT et enlever le supernatant. Ajouter l'anticorps secondaire (chèvre anti-lapin IgG-Alexa Fluor 594, chèvre anti-souris IgG-Alexa Fluor 488) dans la solution de blocage et couver toute la nuit à 4 oC avec des secousses douces.
- Pelleter les organoïdes par centrifugation à 800 x g pendant 3 min à RT, enlever le supernatant, et laver la pastille avec 1 ml de PBS pendant 15 min avec des secousses douces.
- Laver la pastille comme à l'étape 5.2.9 pour deux fois supplémentaires.
6. Défrichage et montage des organoïdes de la prostate tachés pour la microscopie Confocal à monture entière — TIMING : 7 H
- Pelleter les organoïdes par centrifugation à 800 x g pendant 3 min à RT et enlever le supernatant.
- Ajouter 1 mL de 30% de saccharose en PBS avec 1% Triton X-100 et incuber pendant 2 h à RT avec des secousses douces.
- Pelleter les organoïdes par centrifugation à 800 x g pendant 3 min à RT et enlever le supernatant.
- Ajouter 1 mL de 45% de saccharose en PBS avec 1% Triton X-100 et incuber pendant 2 h à RT avec des secousses douces.
- Pelleter les organoïdes par centrifugation à 800 x g pendant 3 min à RT et enlever le supernatant.
- Ajouter 1 mL de 60% de saccharose en PBS avec 1% Triton X-100 et incuber pendant 2 h à RT avec des secousses douces.
- Pelleter les organoïdes par centrifugation à 800 x g pendant 3 min à RT et enlever 95% du supernatant.
REMARQUE: La pastille devient plus lâche à mesure que la concentration de saccharose devient plus élevée. Il est recommandé d'observer les organoïdes tachés de DAPI sous la lumière UV pour confirmer qu'ils n'ont pas été perdus lors de l'ablation du supernatant. - Transférer une gouttelette de 10 à 20 l de la suspension restante sur un bordereau chambré et procéder à une microscopie confocale.
REMARQUE: Les fragments de glissement de couverture peuvent être placés de chaque côté de la gouttelette pour être utilisés comme espaceurs (Figure 4C). Ceux-ci empêchent les organoïdes de s'effondrer lorsqu'une feuille de couverture est placée au-dessus de la gouttelette.
Résultats
Les cellules épithéliales de la prostate sont plaquées dans la culture organoïde de souris où elles forment des organoïdes, qui sont récoltés avant la préparation à l'analyse en aval (figure 1).
Les cellules épithéliales basales et luminaires sont isolées à l'aide de FACS. Après avoir exclu lescellules DAPI et appauvri les cellules Lin (CD45, CD31, Ter119), les cellules basales et lumineuses se distinguent en fonction de l'expression différentielle de l'EpCAM et du CD49f(figure 2). L'approche décrite pour plaquer les cellules basales et lumineuses de prostate dans la culture organoïde implique : (1) placage des cellules dans des anneaux de gel de matrice, et (2) puits de revêtement avec Poly-HEMA. Le plaquage dans les anneaux a été précédemment décrit dans Agarwal et autres9. L'utilisation de cette approche (Figure 3A) permet aux chercheurs d'éviter plus facilement le gel matriciel tout en réapprovisionnant les médias (étape 3), et de compter plus facilement les organoïdes en suivant la circonférence du puits. Il a été démontré que les puits de revêtement avec Poly-HEMA empêchent la formation de colonies 2D dans les organoïdes rétiniens17; cependant, cette approche n'a pas été employée dans le modèle organoïde de prostate. Fait important, le revêtement des puits avec Poly-HEMA (tableau 3) élimine l'apparition de colonies 2D sans interférer avec la formation d'organoïdes (Figure 3B). Ces modifications augmentent les capacités de l'organe de la prostate.
Les cellules basales et lumineuses forment des organoïdes avec des morphologies distinctes (Figure 4A). Alors que la plupart des organoïdes basaux sont de taille similaire (100-300 m de diamètre) après 7 jours de culture, les organoïdes d'origine lumineuse présentent une hétérogénéité significative (30-450 m de diamètre). De plus, la plupart des organoïdes basaux contiennent des lumens entourés d'épithélium à plusieurs couches(figure 4A, en haut),tandis que les organoïdes d'origine luminaire varient en morphologie du creux, avec épithélium à couches uniques au solide, avec des cordons multicouches de cellules qui ne canalisent pas (Figure 4A, en bas). Les approches décrites ci-dessus pour préparer les organoïdes à l'analyse en aval (étapes 4, 5) ont été utilisées pour déterminer si ces différences phénotypiques reflètent les différences dans l'expression des marqueurs de lignée. L'analyse occidentale de tache a indiqué que les organoïdes basal et luminal-dérivés conservent des dispositifs liés aux cellules primaires basales et lumineuses. Les organoïdes basoïdes expriment des niveaux plus élevés de la cytokératine de marqueur basal 5 (K5), tandis que les organoïdes d'origine luminaire expriment des niveaux plus élevés de la cytokératine de marqueur luminal 8 (K8)(figure 4B). Des marqueurs basaux et luminal ont été détectés chez des organoïdes basaux et d'origine lumineuse dans la population en vrac, ce qui suggère peut-être une différenciation (figure 4B).
Nous avons cherché à caractériser l'expression des marqueurs de lignée chez les organoïdes d'origine basale et à déterminer si les organoïdes dérivés de la sommité se distinguent morphologiquement présentent des différences dans l'expression des marqueurs en tachant des organoïdes intacts et en effectuant une microscopie confocale (Figure 4D). Les organoïdes basoïdes ont contenu l'épithélium multi-couches avec des couches extérieures exprimant des niveaux élevés du marqueur basal p63 et des niveaux modérés du marqueur luminal K8 (p63salut, K8milieu),et des couches intérieures sans niveaux détectables de p63 et des niveaux élevés de K8 (p63lo, K8salut) (Figure 4D, haut). Alors que toutes les cellules des organoïdes d'origine luminaire à couches uniques souillaient positivement pour le K8, seules certaines cellules contenaient des cellules nucléaires p63(figure 4D, en bas). Ces données valident les approches de récolte et de préparation des organoïdes pour l'analyse par la microscopie occidentale de tache ou confocale et augmentent ainsi la capacité de l'analyse organoïde pour étudier les processus cellulaires principaux, y compris la différenciation.
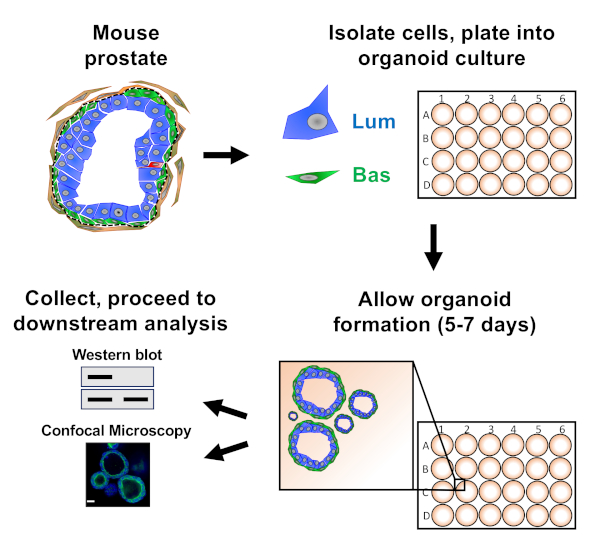
Figure 1 : Flux de travail schématique d'illustration pour produire des organoïdes de prostate pour la collecte et l'analyse. La prostate totale de souris est dissociée et les cellules épithéliales de prostate basale et luminale sont isolées par le tri fluorescence-activé de cellules par l'intermédiaire des protocoles établis18,19. Les cellules basales ou lumineuses suspendues dans un mélange de médias organoïdes de souris et de gel de matrice sont plaquées dans des anneaux de gel de matrice. Après 5 à 7 jours de culture, les organoïdes sont récoltés pour analyse par la tache occidentale ou la microscopie confocale. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
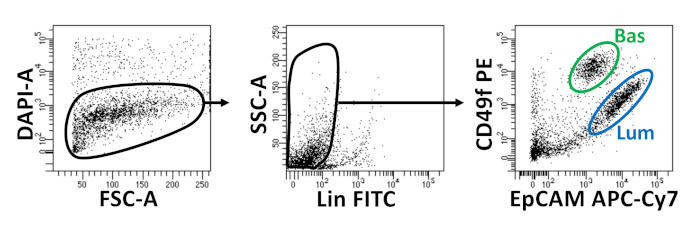
Figure 2 : Isolement des cellules épithéliales de la prostate basale et luminale à l'aide du tri cellulaire activé par la fluorescence (FACS). Les cellules dissociées de la prostate de souris sont tachées avec DAPI, pour distinguer vivant des cellules mortes, et des anticorps de surface, pour distinguer les cellules basales des cellules lumineuses, avant FACS. Gauche - Gated sur DAPI- cellules. FSC-A - diffusion vers l'avant. Centre - Gated sur Lin- cellules (CD45lo, CD31lo, Ter119lo). SSC-A - diffusion latérale. Droite - Cellules basales (Bas) (EpCAMsalut, CD49fsalut), cellules luminales (Lum) (EpCAMsalut, CD49fmi). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
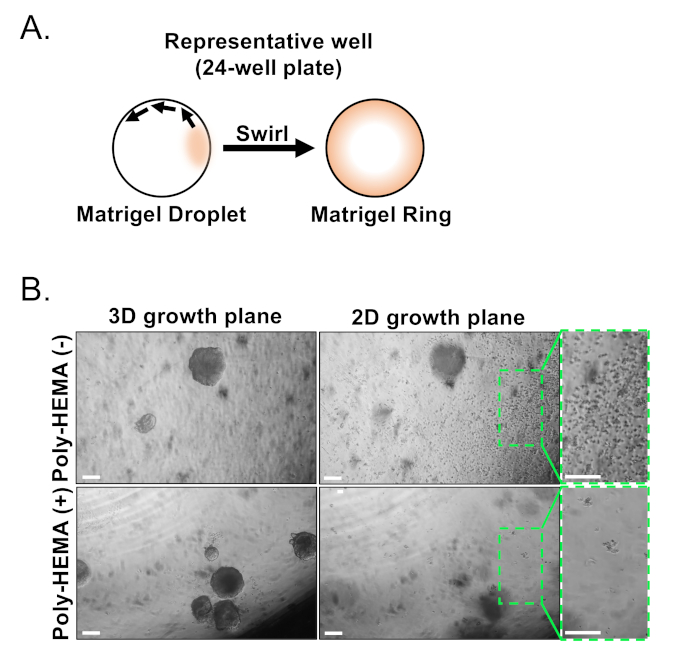
Figure 3 : Établissement d'organoïdes de la prostate de souris. (A) Approche d'illustration schématique pour générer un anneau de gel de matrice dans un puits d'une plaque de 24 puits. (B) Images de contraste de phase représentative des organoïdes (plan de croissance 3D) et des colonies bidimensionnelles (plan de croissance 2D) formées 7 jours après placage des cellules épithéliales de prostate dans des plaques non enduites (Poly-HEMA (-)), ou enduites (Poly-HEMA () 24-puits. Les régions en boîte dans le plan de croissance 2D sont amplifiées sur la droite. Barres d'échelle de 200 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
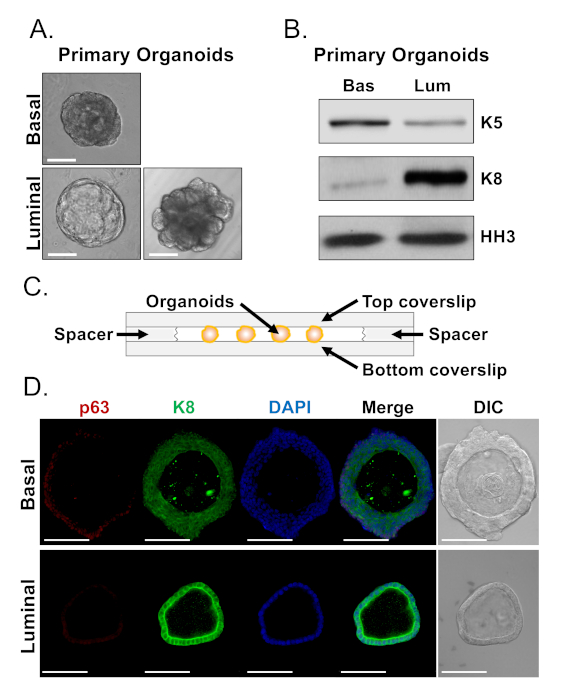
Figure 4 : Analyse de l'expression des marqueurs de lignée chez les organoïdes de la prostate par la tache occidentale et la microscopie confocale à monture entière. (A) Images de contraste de phase représentative des organoïdes basal-dérivés (en haut) et luminal-dérivés (bas) après 7 jours de culture. Barre à l'échelle de 100 m. (B) Analyse de tache occidentale des organoïdes basal-dérivés (Bas) et luminal-dérivés (Lum) après 5 jours de culture. Staining pour le marqueur basal, cytokératine 5 (K5), et le marqueur luminaire, cytokératine 8 (K8), et un contrôle de chargement, histone H3 (HH3). (C) Schematic illustrant la couverture chambrée avec des espaceurs. (D) Contraste différentiel représentatif d'interférence (DIC) et images immunofluorescentes des organoïdes basal-dérivés (en haut) et luminal -en bas après 7 jours de culture. Coloration pour p63 (rouge), K8 (vert) et DAPI (bleu) individuellement et fusionné. Barres d'échelle de 100 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| Recettes | |
| Médias contenant des dispase | 1 mg/mL de dispase à 10 M ROCK dans le DMEM F12 avancé. Filtrer la stérilisation à l'aide d'un filtre de 0,22 m. |
| Médias de dissociation | 10% FBS 1x Pénicilline-Streptomycine dans RPMI 1640. Filtrer la stérilisation à l'aide d'un filtre de 0,22 m. |
| Tampon de lyse protéique | Tampon RIPA et inhibiteurs de la phosphatase et inhibiteurs de la protéase |
| Solution de blocage | 10% FBS en PBS avec 0,2% Triton X-100 |
Tableau 1 : Instructions pour la préparation des solutions clés.
| Composant | Concentration |
| B-27 (en) | 1x (dilué à partir de concentré 50x) |
| GlutaMAX (glutaMAX) | 1x (dilué à partir de concentré 100x) |
| N-acétyl-L-cystéine | 1,25 mM |
| Normocin Normocin | 50 g/mL |
| EGF humain recombinant, sans animaux | 50 ng/mL |
| Noggin humain recombinant | 100 ng/mL |
| R-spondin 1-conditionné médias | 10% de supports conditionnés |
| A83-01 | 200 nM |
| Dht | 1 nM |
| Y-27632 dihydrochlorure (inhibiteur ROCK) | 10 M |
| DMEM/F-12 avancé | Médias de base |
| Les médias conditionnés R-spondin 1 sont générés comme décrit dans Drost, et al.13. Après l'ajout de tous les composants, filtrez stériliser les supports organoïdes de souris à l'aide d'un filtre de 0,22 m. L'inhibiteur ROCK n'est ajouté que lors de l'établissement de la culture et de la passaging des organoïdes. | |
Tableau 2 : Instructions pour la préparation des supports organoïdes de souris.
| Protocole pour la préparation des plaques enduites de Poly-HEMA | |
| 1 | Ajouter 0,25 g De Poly-HEMA à 50 ml 98% EtOH. Dissoudre Poly-HEMA à 37 oC sur un shaker. Ce processus prend au moins 4 h. |
| 2 | Filtrer la stérilisation Poly-HEMA à l'aide d'un filtre de 0,22 m. |
| 3 | Ajouter 200 l de solution Poly-HEMA par puits d'une plaque de 24 puits. |
| 4 | Retirer le couvercle (s) des plaques de 24 puits après l'ajout de Poly-HEMA et permettre à la solution de s'évaporer pendant la nuit. |
| 5 | Laver chaque puits deux fois avec du PBS et s'assurer que les puits sont complètement secs avant d'être entreposés après le lavage final. REMARQUE : Perturber le revêtement de Poly-HEMA pendant le lavage pourrait contribuer à la croissance bidimensionnelle en placage des cellules épithéliales dans la culture organoïde. Pour éviter d'endommager les puits recouverts de poly-HEMA, évitez tout contact direct avec la pointe de la pipette pendant le lavage. L'intégrité des puits enduits de Poly-HEMA restera intacte à moins que le Poly-HEMA ne soit gratté par la pointe de la pipette. |
| 6 | Les plaques enduites de poly-HEMA peuvent être conservées à 4 oC pendant une durée pouvant aller jusqu'à deux semaines. REMARQUE : L'emballage des plaques dans le parafilm avant l'entreposage réduira le risque de contamination. |
Tableau 3 : Protocole de préparation des plaques enduites de Poly-HEMA.
Discussion
La différenciation épithéliale de cellules de prostate a été impliquée dans la biologienormale de prostate2,3,4,5,6,7 et la biologie de la maladie 8,10,11,12; toutefois, les principaux régulateurs de ce processus ne sont toujours pas définis. L'identification des régulateurs clés de la différenciation épithéliale de cellules de prostate a été difficile en partie due à l'absence de contextes bien établis pour la modeler. Tandis que la culture 2D de monocouche peut être employée pour modéliser la différenciation11,12,ce contexte ne récapitule pas le microenvironnement complexe de prostate. En outre, les contextes in vivo pour modéliser la différenciation ne se prêtent pas aux études mécanistes, car ils sont difficiles à manipuler. Par conséquent, l'identification d'un contexte facile à manipuler, mais physiologiquement pertinent, pour étudier la différenciation est essentielle.
Le modèle organoïde de prostate représente un contexte ex vivo élégant où la différenciation basale à luminal est rapportée pour se produire. Les méthodes d'établissement des organoïdes de la prostate sont bien établies14; cependant, une optimisation plus poussée de ces méthodes est nécessaire. En outre, les approches de récolte et de préparation des organoïdes de la prostate pour l'analyse ne sont pas clairement décrites. Cet article décrit une approche aux cellules épithéliales de prostate de plaque isolées de la prostate de souris dans la culture organoïde. Cette approche permet aux chercheurs (1) de prévenir l'apparition de colonies 2D pendant la formation d'organoïdes, (2) de réduire le risque de perturbation du gel de matrice pendant le réapprovisionnement des médias, et (3) de compter plus efficacement les organoïdes. En outre, ce manuscrit décrit les approches de la récolte des organoïdes pour la préparation à l'analyse de tache occidentale, ou microscopie confocale de montage entier. Fait important, l'approche utilisée pour préparer les organoïdes pour la microscopie confocale maintient la structure intacte des organoïdes tout au long de sa durée, ce qui réduit les dommages organoïdes avant l'acquisition de l'image. Au total, les approches décrites augmentent les capacités de l'organe de la prostate.
Notamment, la capacité de formation d'organoïdes des cellules basales et lumineuses peut être modifiée à la fois par des méthodes utilisées pour isoler les populations respectives, et par les conditions de culture. Les conditions de culture organoïde utilisées dans cet essais ont d'abord été décrites par Karthaus et al.13. Alors que Karthaus et coll. ont signalé que les cellules basales ont une capacité de formation organoïde plus élevée (15 %) que les cellules luminales (1 %)13, Chua et coll., en utilisant des méthodes d'isolement distinctes et des conditions de culture, ont signalé que les cellules luminales (0,2-0,3 %) ont une capacité de formation d'organoïdes plus élevée que les cellules basales (0,03 %)20. Dans l'ensemble, les méthodes décrites par Karthaus et coll. conduisent à des taux de formation d'organoïdes plus élevés pour les cellules basales et lumineuses, reflétant probablement les différences dans l'approche utilisée pour isoler les cellules basales et lumineuses13, par opposition aux conditions de culture qui biaisent la formation d'organoïdes à partir de cellules lumineuses. Il n'est pas clair si le protocole décrit dans ce manuscrit favorise la formation d'organoïdes luminal à partir d'ancêtres sommaux multipotents, ou d'ancêtres lumineux commis9. Bien que opportune et coût-prohibitive, les études in vivo de traçage de lignée peuvent être employées pour valider des dispositifs d'ancêtre liés aux lignées épithéliales distinctes de prostate élucidées dans l'analyse organoïde.
Des processus tels que le développement, la différenciation et la transformation sont non seulement pertinents pour la biologie de la prostate, mais aussi pour la biologie d'autres tissus, y compris le cerveau, les poumons, l'intestin, le pancréas et le foie. Les méthodes décrites facilitent l'utilisation du modèle organoïde pour étudier ces processus non seulement dans la prostate, mais aussi dans un large éventail de tissus.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
PDC et JMG sont soutenus par le Ruth L. Kirschstein National Research Service Award GM007185. JAD est soutenu par l'Institut national des sciences médicales générales des National Institutes of Health (R25GM055052) décerné à T. Hasson et la bourse Saul Martinez. ASG est soutenu par le Spitzer Family Foundation Fund et le Gill Endowment. Ce travail a été soutenu par l'American Cancer Society (RSG-17-068-01-TBG), le ministère de la Défense (W81XWH-13-1-0470), Margaret E. Early Medical Research Trust, NIH/NCI (P50CA092131/UCLA SPORE in Prostate Cancer), Rose Hills Foundation, et le soutien du Jonsson Comprehensive Cancer Center de l'UCLA, du Broad Stem Cell Research Center, du Clinical and Translational Science Institute et de l'Institute of Urologic Oncology.
matériels
| Name | Company | Catalog Number | Comments |
| µ-Dish 35 mm, high | ibidi | 81156 | |
| 16% Paraformaldehyde | Thermo Fisher Scientific | 50-980-487 | |
| 4’,6-diamidino-2-phenylindole (DAPI) | Thermo Fisher Scientific | D1306 | |
| A83-01 | Tocris | 2939 | |
| Advanced DMEM/F-12 | Thermo Fisher Scientific | 12634010 | |
| APC/Cy7 anti-mouse CD326 (Ep-CAM) Antibody, 100 μg | BioLegend | 118218 | |
| B-27 Supplement (50x), Serum Free | Thermo Fisher Scientific | 17504044 | |
| Complete Protease Inhibitor Cocktail | Sigma | 11836145001 | |
| (DiHydro)testosterone (5α-Androstan-17β-ol-3-one) | Sigma | A-8380 | |
| Dispase II, Powder | Thermo Fisher Scientific | 17-105-041 | |
| Fetal Bovine Serum (FBS) | Sigma | F8667 | |
| FITC anti-mouse CD31 Antibody (0.5 mg/mL, 50 μg) | BioLegend | 102405 | |
| FITC anti-mouse CD45 Antibody (0.5 mg/mL, 50 μg) | BioLegend | 103107 | |
| FITC anti-mouse TER-119/Erythroid Cells Antibody (0.5 mg/mL, 50 μg) | BioLegend | 116205 | |
| Goat anti-mouse IgG-Alexa Fluor 488 | Invitrogen | A28175 | |
| Goat anti-rabbit IgG-Alexa Fluor 594 | Invitrogen | A11012 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Halt Phosphatase Inhibitor | Thermo Fisher Scientific | 78428 | |
| Matrigel GFR Membrane Matrix | Corning | CB-40230C | |
| Mouse anti-cytokeratin 8 | BioLegend | 904804 | |
| N-acetyl-L-cysteine | Sigma | A9165 | |
| Normocin | Thermo Fisher Scientific | ant-nr-1 | |
| PE anti-human/mouse CD49f Antibody | BioLegend | 313612 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | 15-140-122 | |
| Poly(2-hydroxyethyl methacrylate) (Poly-HEMA) | Sigma | P3932-25G | |
| Rabbit anti-p63 | BioLegend | 619002 | |
| Radioimmunoprecipitation assay (RIPA) | Thermo Fisher Scientific | PI89901 | |
| Recombinant Human EGF, Animal-Free | PeproTech | AF-100-15 | |
| Recombinant Human Noggin | PeproTech | 120-10C | |
| RPMI 1640 Medium, HEPES (cs of 10) | Thermo Fisher Scientific | 22400105 | |
| Sonic Dismembrator | Thermo Fisher Scientific | FB120 | |
| Sucrose | Sigma | S0389-500G | |
| Triton X-100 | Sigma | X100-5ML | |
| Y-27632 dihydrochloride (ROCK inhibitor) | Selleck Chemical | S1049-50MG |
Références
- Kwon, O. J., Xin, L. Prostate epithelial stem and progenitor cells. American Journal of Clinical and Experimental Urology. 2 (3), 209-218 (2014).
- Choi, N., Zhang, B., Zhang, L., Ittmann, M., Xin, L. Adult Murine Prostate Basal and Luminal Cells Are Self-Sustained Lineages that Can Both Serve as Targets for Prostate Cancer Initiation. Cancer Cell. 21 (2), 253-265 (2012).
- Ousset, M., Van Keymeulen, A., et al. Multipotent and unipotent progenitors contribute to prostate postnatal development. Nature Cell Biology. 14 (11), 1131-1138 (2012).
- Wang, J., et al. Symmetrical and asymmetrical division analysis provides evidence for a hierarchy of prostate epithelial cell lineages. Nature Communications. 5, 1-13 (2014).
- Wang, Z. A., Mitrofanova, A., et al. Lineage analysis of basal epithelial cells reveals their unexpected plasticity and supports a cell-of-origin model for prostate cancer heterogeneity. Nature Cell Biology. 15 (3), 274-283 (2013).
- Kwon, O. J., Zhang, B., Zhang, L., Xin, L. High fat diet promotes prostatic basal-to-luminal differentiation and accelerates initiation of prostate epithelial hyperplasia originated from basal cells. Stem Cell Research. 16 (3), 682-691 (2016).
- Kwon, O. J., Zhang, L., Ittmann, M. M., Xin, L. Prostatic inflammation enhances basal-to-luminal differentiation and accelerates initiation of prostate cancer with a basal cell origin. Proceedings of the National Academy of Sciences. 192 (3), 997-999 (2014).
- Stoyanova, T., et al. Prostate cancer originating in basal cells progresses to adenocarcinoma propagated by luminal-like cells. Proceedings of the National Academy of Sciences. 110 (50), 20111-20116 (2013).
- Agarwal, S., Hynes, P. G., et al. Identification of Different Classes of Luminal Progenitor Cells within Prostate Tumors. Cell Reports. 13 (10), 2147-2158 (2015).
- Ku, S. Y., et al. Rb1 and Trp53 cooperate to suppress prostate cancer lineage plasticity, metastasis, and antiandrogen resistance. Science. 355 (6320), 78-83 (2017).
- Mu, P., et al. SOX2 promotes lineage plasticity and antiandrogen resistance in TP53- and RB1-deficient prostate cancer. Science. 355 (6320), 84-88 (2017).
- Bishop, J. L., et al. The Master Neural Transcription Factor BRN2 Is an Androgen Receptor-Suppressed Driver of Neuroendocrine Differentiation in Prostate Cancer. Cancer Discovery. 7 (1), 54-71 (2016).
- Karthaus, W. R., et al. Identification of multipotent luminal progenitor cells in human prostate organoid cultures. Cell. 159 (1), 163-175 (2014).
- Drost, J., Karthaus, W. R., et al. Organoid culture systems for prostate epithelial and cancer tissue. Nature Protocols. 11 (2), 347-358 (2016).
- McCray, T., Richards, Z., Marsili, J., Prins, G. S., Nonn, L. Handling and Assessment of Human Primary Prostate Organoid Culture. Journal of Visualized Experiments. (143), e59051(2019).
- Lawson, D. A., Xin, L., Lukacs, R. U., Cheng, D., Witte, O. N. Isolation and functional characterization of murine prostate stem cells. Proceedings of the National Academy of Sciences of the United States of America. 104 (1), 181-186 (2007).
- Chen, H. Y., Kaya, K. D., Dong, L., Swaroop, A. Three-dimensional retinal organoids from mouse pluripotent stem cells mimic in vivo development with enhanced stratification and rod photoreceptor differentiation. Molecular vision. 22, 1077-1094 (2016).
- Liu, X., et al. Low CD38 Identifies Progenitor-like Inflammation-Associated Luminal Cells that Can Initiate Human Prostate Cancer and Predict Poor Outcome. Cell Reports. 17 (10), 2596-2606 (2016).
- Lukacs, R. U., Goldstein, A. S., Lawson, D. A., Cheng, D., Witte, O. N. Isolation, cultivation and characterization of adult murine prostate stem cells. Nature protocols. 5 (4), 702-713 (2010).
- Chua, C. W., Shibata, M., et al. Single luminal epithelial progenitors can generate prostate organoids in culture. Nature Cell Biology. 16 (10), 951-961 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon