Method Article
PeptiQuick, une incorporation en une seule étape des protéines de membrane dans les Peptidiscs biotinylated pour les essais de liaison de protéine rationalisés
Dans cet article
Résumé
Nous présentons une méthode qui combine la purification et la reconstitution de protéine de membrane en peptidiscs en une seule étape chromatographique. Les échafaudages biotinylated sont employés pour l'attachement direct de surface et la mesure des interactions protéine-ligand par l'intermédiaire de l'interférométrie de biocouche.
Résumé
Les protéines membranaires, y compris les transporteurs, les canaux et les récepteurs, constituent près d'un quart du protéome cellulaire et plus de la moitié des cibles actuelles des médicaments. Pourtant, un obstacle majeur à leur caractérisation et leur exploitation dans les milieux académiques ou industriels est que la plupart des stratégies biochimiques, biophysiques et de dépistage des médicaments exigent que ces protéines soient dans un état soluble dans l'eau. Notre laboratoire a récemment mis au point le peptidisc, un mimétique membranaire offrant une approche « universelle » au problème de la solubilité des protéines membranaires. Nous présentons ici un protocole simplifié qui combine la purification des protéines et la reconstitution peptidisc en une seule étape chromatographique. Ce flux de travail, appelé PeptiQuick, permet de contourner la dialyse et l'incubation avec des perles de polystyrène, réduisant ainsi considérablement l'exposition au détergent, la dénaturation des protéines et la perte d'échantillons. Lorsque PeptiQuick est effectué avec des échafaudages biotinylated, la préparation peut être directement attachée aux surfaces enduites de streptavidin. Il n'est pas nécessaire de biotinylate ou de modifier la cible de protéines membranaires. PeptiQuick est présenté ici avec le récepteur membranaire FhuA et la colicine ligand antimicrobienne M, en utilisant l'interférométrie biocouche pour déterminer la cinétique précise de leur interaction. Il est conclu que PeptiQuick est un moyen pratique de préparer et d'analyser les interactions protéines-ligand membrane en un jour dans un environnement sans détergent.
Introduction
Les protéines membranaires sont souvent exclues des programmes de recherche sur la découverte de médicaments ou d'anticorps en raison de la propension des protéines membranaires à s'agréger à l'extérieur de l'environnement bicouche lipidique, en particulier en présence de détergents1. Par conséquent, ces dernières années, plusieurs mimétiques membranaires (appelés échafaudages) ont été développés pour faciliter l'isolement et l'interrogatoire des protéines membranaires dans un environnement complètement sans détergent (c.-à-d. nanodisques, SMALPs, amphipols, etc.) 2,3,4,5,6. Cependant, la reconstitution des protéines membranaires dans ces mimétiques nécessite souvent une optimisation étendue, qui prend beaucoup de temps et s'accompagne généralement d'une perte de récupération des protéines7,8. Pour surmonter ces limites, notre laboratoire a récemment mis au point une formulation « universelle » connue sous le nom de peptidisc9. Le peptidisc se forme lorsque plusieurs copies d'un peptide biphélical amphipathique de 4,5 kDa se lient à la surface hydrophobe d'une protéine membranaire cible. La reconstitution stable dans le peptidisc se produit sur l'enlèvement du détergent, attachant des lipides endogènes et des protéines solubilisées de membrane dans les particules solubles dans l'eau. Ces particules stabilisées sont maintenant propices à de nombreuses applications en aval.
La méthode peptidisc offre plusieurs avantages; par exemple, la reconstitution est simple, puisque la liaison de l'échafaudage peptidique sur la cible est guidée par le modèle de protéine lui-même9,10. La stoichiométrie peptidique est également autodéterminée, et l'ajout de lipides exogènes n'est pas nécessaire. La formation de peptidisc se produit par la dilution simple de détergent, un avantage important sur la dialyse ou l'adsorption sur des perles de polystyrène, qui ont souvent comme conséquence le bas rendement de protéine dû à l'association et à l'agrégation de surface non spécifiques11,12 ,13. L'assemblage final du peptidisque est très thermostable et invariablement soluble dans différents tampons ou en présence de cations divalentes (p. ex., Ni2). La pureté et l'homogénéité structurelle de l'échafaudage sont également élevées (p. ex., sans endotoxine), et le peptide peut être personnalisé avec des groupes fonctionnels placés à des positions différentes.
Nous présentons ici un workflow de laboratoire appelé PeptiQuick, également connu sous le nom de reconstitution sur les perles9. Ce protocole combine la purification des protéines membranaires et la reconstitution peptidisc en une seule étape et sur le même support chromatographique. Comme son nom l'indique, PeptiQuick est rapide par rapport à d'autres méthodes de reconstitution, et il réduit également sérieusement le temps d'exposition au détergent. Les effets détergents négatifs tels que le développement et l'agrégation des protéines se produisent souvent d'une manière dépendante du temps; par conséquent, il est essentiel de minimiser l'exposition aux détergents pour maintenir la conformation protéique indigène14,15. Ceci est crucial pour l'exactitude des méthodes qui rendent compte des interactions protéiques et des affinités liant ligand.
Tout en développant ce protocole, nous présentons la nouvelle version biotinylated de l'échafaudage peptidisque, appelé Bio-Peptidisc. Les groupes fonctionnels de biotine permettent l'attachement de la protéine de membrane cible sur les surfaces streptavidin-enduites. Étant donné que l'étiquetage de la biotine est limité à l'échafaudage, les sites de liaison sur la protéine membranaire cible demeurent inchangés. À l'aide du Bio-Peptidisc, la cinétique de liaison du récepteur de membrane bactérienne FhuA et de la colicine de peptide antimicrobienne M (ColM) sont déterminées16. Cette affinité est mesurée par interférométrie biocouche (BLI), qui analyse les interactions en temps réel en fonction des modèles d'interférence de la lumière blanche réfléchis à partir d'une pointe du capteur.
En utilisant ce protocole, le besoin de détergent lors de l'analyse BLI est éliminé, ce qui est un développement important, car les détergents peuvent perturber les interactions. Les affinités de liaison peuvent être mesurées rapidement avec cette méthode, et les résultats sont comparables à ceux rapportés plus tôt en utilisant des nanodisques et la calorimétrie de titration isothermale (ITC)16. Les étapes critiques du flux de travail PeptiQuick sont montrées et discutées, telles que la préparation des protéines, la dilution des détergents, l'addition de peptide et la reconstitution, ainsi que des conseils pour dépanner le ligand et la liaison analyte dans l'analyse BLI. En utilisant le flux de travail PeptiQuick, il est constaté que les protéines membranaires peuvent être capturées dans les peptidisques et leurs interactions mesurées dans une journée.
Protocole
1. Préparation et solubilisation du récepteur membranaire FhuA
- Express Son6-étiquetéFhuA dans Escherichia coli souche AW740. Cultiver des cellules pendant 18 h à 37 oC dans les médias M9 (tableau 1). Voir Mills et coll.16 pour un protocole d'expression détaillé.
- Récolte des cellules par centrifugation (5 000 x g, 10 min, 4 oC) et les resuspend dans 50 ml de tampon Tris-salt-glycerol (TSG) (tableau 1). Dounce les cellules en suspension et ajouter phenylmethanesulfonylfluoride (PMSF) à une concentration finale de 1 mM juste avant la lyse.
CAUTION: PMSF est toxique et corrosif. - Lyser les cellules à l'aide d'un microfluidifiant (trois passages) à 15 000 psi ou d'une presse Français (trois passages) à 8 000 psi. Pelleter les cellules non lysées et d'autres matériaux insolubles par centrifugation à basse vitesse (5 000 x g, 10 min, 4 oC).
- Ultracentrifuge le supernatant (200 000 x g, 40 min, 4 oC) pour isoler la fraction de membrane brute. Jeter le supernatant, resuspendre la pastille membranaire dans un minimum de tampon TSG, et dounce à l'aide d'un appareil de verre ou de métal douncer pour assurer l'homogénéité.
- Effectuez un astodonte de Bradford pour vérifier la concentration en protéines de la membrane brute en suspension. Diluer la membrane brute à une concentration finale de 3 mg/mL à l'aide d'un tampon TSG avant la solubilité.
- Ajouter le détergent Triton X-100 à une concentration finale de 1 % (v/v) pour solubiliser sélectivement la membrane interne bactérienne pendant 1 h à 4 oC avec un basculement doux. Pelleter le matériau insoluble (fraction de membrane externe) par ultracentrifugation (200 000 x g, 40 min, 4 oC).
- Jeter le supernatant contenant la membrane solubilisée et resuspendre la pastille dans TSG à une concentration finale de 3 mg/mL. Ajouter l'oxyde de lauryldimethylamine (LDAO) à une concentration de 1 % (v/v) à la fraction de membrane externe en suspension et solubilité pendant 1 h à 4 oC avec un basculement doux.
- Effectuer une dernière étape de centrifugation pour granuler tous les matériaux insolubles (200 000 x g,40 min, 4 oC).
REMARQUE : Le supernatant résultant contient les protéines solubilisées de membrane externe, y compris la cible son FhuA marqué.
2. Purification et reconstitution de FhuA à l'aide du flux de travail PeptiQuick
- Pré-équilibre réchauffement d'une colonne Ni-NTA préemballée (volume de résine de 5 mL) avec deux volumes de colonnes de chromatographie d'affinité métallique immobilisée (IMAC) tampon de lavage (tableau 1).
- Diluer la membrane externe solubilisée de 1% LDAO à 0.04% LDAO utilisant TSG. Ensuite, ajouter l'imidazole à une concentration finale de 5 mM.
L'imidazole est toxique et corrosif. - Chargez les protéines de la membrane externe solubilisée sur la résine Ni-NTA et collectez le débit. Rechargez le flux sur la résine pour augmenter la liaison de résine de FhuA et de recueillir le débit secondaire.
REMARQUE : Conserver un aliquot de 20 L de matière solubilisée comme référence pour l'analyse ultérieure du polyacrylamide de gel de sulfate de dodecyl de sodium (SDS-PAGE). - Laver la résine avec 250 ml de tampon de lavage IMAC et recueillir les 50 premiers ml d'éluate. Égoutter le tampon de lavage à la hauteur du volume de lit de résine et fermer le robinet d'arrêt sur la colonne.
- Ajouter 1 mL de solution de peptide bio-peptidisc concentrée de 10 mg/mL (tableau 1) à la colonne. Ajouter 50 ml de solution de peptide bio-peptidisc diluée de 1 mg/mL dans TSG et remuer la résine avec une tige de verre pour resuspendre les perles dans TSG.
- Après le piégeage peptidisque, laisser la résine se déposer et égoutter l'excédent de 1 mg/mL de solution bio-peptidisc par la résine.
- Laver les particules peptdisciques fixées par Ni-NTA avec 50 ml de TSG. Élifier les particules peptidiques avec 15 ml de tampon d'élution IMAC (tableau 1) contenant 600 mM d'imidazole dans TSG. Recueillir 1 ml de fractions et ajouter immédiatement 10 'L de 0,5 M EDTA pour les ions de nickel lixiviés.
CAUTION: EDTA est un irritant.
3. Évaluation de la reconstitution PeptiQuick
- Analyse SDS-PAGE
- Chargez 10 aliquots ll du matériau de démarrage, flowthrough, wash(es), et des fractions élucées de la reconstitution PeptiQuick sur un gel et une électrophorese dénaturant SDS de 12 % pendant 30 min à un courant constant de 60 mA.
- Tainrisez le gel avec du colorant bleu Coomassie, detinez le gel et visualisez-le sur un scanner.
- Chromatographie d'exclusion de taille (SEC)
- Basé sur l'analyse SDS-PAGE de 12 % de la reconstitution PeptiQuick, isolez et mettez en commun les fractions pertinentes et concentrez-les à l'aide d'un concentrateur centrifuge de 30 kDa.
REMARQUE : Dans la vidéo d'accompagnement, les fractions F3-F7 sont regroupées et concentrées en dessous de 1 ml (le volume de boucle d'injection sur l'instrument SEC). - Injecter 1 ml des fractions d'élution IMAC mises en commun sur une colonne de filtration de gel S200 (300/10) à un débit de 0,25 ml/min dans le tampon TSG. Recueillir 1 mL fractions et les exécuter sur un gel SDS 12% pour déterminer quelles fractions SEC à mettre en commun et à se concentrer.
REMARQUE : Les fractions PeptiQuick élunées peuvent être mises en commun sans concentration, et un aliquot peut être injecté sur la SEC pour vérifier la qualité de la reconstitution. Il s'agit d'un contrôle important pour les protéines membranaires qui sont potentiellement sensibles à l'agrégation pendant la concentration centrifuge.
- Basé sur l'analyse SDS-PAGE de 12 % de la reconstitution PeptiQuick, isolez et mettez en commun les fractions pertinentes et concentrez-les à l'aide d'un concentrateur centrifuge de 30 kDa.
4. Interférométrie Biolayer
- Configuration expérimentale BLI
- Configurez la plaque de puits 96 à la main.
REMARQUE : Tous les puits sont remplis jusqu'à un volume final de 200 L. - Dans la colonne 1, les lignes A-E, chargez 200 l de tampon cinétique pour permettre aux pointes d'équilibrer et de former un signal de base.
- Diluer le ligand (FhuA dans Bio-Peptidisc) à une concentration de 2,5 g/mL dans le tampon cinétique (tableau 1). Chargez 200 l de cette dilution en rangées A-D dans la colonne 2.
- Ajouter 200 l de tampon cinétique uniquement à E2 (le capteur de référence).
- Ajouter 200 l de tampon cinétique à la colonne 3, lignes A-E pour laver l'excès de FhuA de la pointe.
- Dans la colonne 4, effectuer des dilutions en série doubles de l'analyte (ColM) en bas de la plaque de A4 à D4. Commencez par 28 nM (8 Kd) en A4 à 3,5 nM (1 Kd) en D4.
- Ajouter 28 nM ColM à E4 pour mesurer la liaison non spécifique (la concentration de ColM la plus élevée utilisée).
- Ajouter 200 l de tampon cinétique à la colonne 5, lignes A-E.
REMARQUE : Ici, le ColM se dissocie de la pointe, et la dissociation est mesurée. - Placez la plaque d'configuration dans l'instrument BLI.
- Placez le plateau de pointe du capteur dans l'instrument BLI.
- Ouvrez le logiciel d'acquisition de données BLI et sélectionnez New Kinetics Experiment sur l'assistant logiciel.
- Utilisez l'onglet de définition de la plaque pour entrer la disposition de la plaque de puits 96 dans le logiciel.
REMARQUE: Les puits contenant le ligand, FhuA sont saisis comme le "Load". Les puits contenant uniquement du tampon sont saisis sous le titre de « tampon ». Les puits contenant l'analyte, ColM, sont entrés comme « échantillon ». - Utilisez l'onglet de définition de l'analyse pour définir la durée et la vitesse de rotation des plaques pour chaque étape de l'expérience.
REMARQUE: Les étapes expérimentales sont définies comme: 1) ligne de base: 60 s; 2) chargement : 250 s ; 3) baseline2 : 300 s ; 4) association : 450 s ; et 5) dissociation : 900 s. Laissez la vitesse de secousse par défaut (1 000 tr/min). Les étapes ci-dessus sont attribuées individuellement à chaque colonne dans la plaque de puits 96 en sélectionnant l'étape désirée et cliquez à droite Ajouter l'étape d'assay sur chaque colonne. - Attribuez la première étape en cliquant à droite sur la première colonne et sélectionnez Démarrer de nouveaux résultats. Assurez-vous que l'étape de base est correctement assignée à l'aide de la fenêtre du bas à droite et changez avec le menu déroulant au besoin.
- Attribuez les étapes suivantes à chaque colonne en cliquant à droite le long de la plaque et en sélectionnant Add Assay Step. Attribuez-les à la colonne appropriée, telle qu'elle est fixée à l'étape 4.1.13.
- Utilisez l'onglet d'affectation du capteur pour vous assurer que l'instrument d'octet prend des broches BLI à partir de l'emplacement correct dans le plateau du capteur.
REMARQUE : Dans l'onglet d'affectation du capteur, seul A1-E1 doit être mis en surbrillance en bleu. Assurez-vous qu'une pointe du capteur est présente à ces endroits dans le bac du capteur et assurez-vous que le F1-H1 est vide. - Mettez en surbrillance F1-H1 à l'écran, puis cliquez à droite et sélectionnez Supprimer pour les marquer comme vides. Remplacer les capteurs de tic-tac dans le plateau après utilisation pour conserver les capteurs utilisés.
- Dans l'onglet expérience d'examen, qui fournit un aperçu final de l'expérience avant l'exécution, passez en revue les étapes expérimentales pour vous assurer que la configuration entière est correcte.
- Dans l'onglet expérience en cours d'exécution, sélectionnez un emplacement de fichier pour enregistrer les fichiers de méthode.
- Changer la température de la plaque à la température ambiante.
- Sélectionnez GO pour exécuter l'expérience.
- Configurez la plaque de puits 96 à la main.
- Analyse des données BLI
- Ouvrez le logiciel d'analyse de données Octet BLI.
- Utilisez l'onglet de sélection de données pour localiser l'expérience et vérifier le résumé expérimental. Importer le fichier de projet à partir de son emplacement d'enregistrement défini au cours de l'étape 4.1.19.
- Entrez les concentrations de l'analyte (ici, ColM) en sélectionnant les informations du capteur pour chaque expérience.
- Attribuez la pointe dans E1 comme pointe de référence.
- Utilisez l'onglet de traitement pour soustraire le signal de référence (E1) des données expérimentales. Vérifiez les données brutes de la pointe de référence pour la liaison analyte non spécifique. Si aucun n'est observé, procéder.
REMARQUE : Il s'agit d'un contrôle négatif important. Si l'on observe une liaison non spécifique, elle sera comptabilisée à l'étape 4.2.7. - Alignez l'axe y à la deuxième étape de base. Ne cochez pas la connexion interstep. Sélectionnez filtrage Savitzky-Golay. Sélectionnez les données de processus!.
- Dans l'onglet analyse, sélectionnez les données expérimentales dans le tableau sous la parcelle pour voir les courbes d'association et de dissociation avec le signal du capteur de référence soustrait.
- Sélectionnez le modèle de liaison 1:1 et sélectionnez un ajustement de courbe partielle. Sélectionnez Courbes Fit!.
- Analyser la parcelle de vue résiduelle pour vérifier le raccord de courbe.
- Faites défiler la table pour voir le Kdcalculé .
- Sélectionnez Enregistrer Le rapport... pour enregistrer un document de feuille de calcul détaillant la configuration, les parcelles des données brutes et analysées, et les constantes de dissociation calculées.
Résultats
Le récepteur membranaire FhuA est exprimé dans une souche de laboratoire E. coli. La fraction de membrane externe est récoltée par centrifugation, et les protéines sont solubilisées à l'aide d'une extraction de détergent en deux étapes. Les protéines de membrane solubilized sont chargées sur les perles d'agarose de Ni-NTA emballées dans une colonne en plastique, suivies du flux de travail peptiQuick tel que présenté dans le protocole. Après un contrôle de qualité utilisant la chromatographie d'exclusion de taille, les particules de Bio-Peptidisc sont immobilisées sur des goupilles streptavidin-enduites. L'analyse BLI est effectuée pour mesurer la cinétique des interactions entre FhuA et ColM. L'aperçu schématique de cette expérience est présenté à la figure 1.
Un gel SDS est exécuté pour déterminer la qualité de la reconstitution après l'élution des particules FhuA Bio-Peptidisc de la colonne IMAC (Figure 2). Les aliquots des fractions correspondant au démarrage, au débit, aux lavages et aux fractions d'élution sont chargés sur le gel SDS pour évaluer le succès de la méthode PeptiQuick. L'épuisement de FhuA dans la fraction de flux est corrélé avec un enrichissement dans les fractions d'élution, ce qui indique que la purification de la protéine a été efficace. Des bandes de protéines contaminantes mineures sont observées dans les fractions élucées. L'analyse du gel SDS est utilisée pour déterminer quelles fractions doivent être mises en commun avant l'analyse de la SEC. Un gel indigène des particules FhuA Bio-Peptidisc éluestaires est également exécuté pour confirmer la solubilité FhuA (Figure supplémentaire 1). La migration de la protéine reconstituée dans le gel indigène indique la solubilité des protéines dans un environnement sans détergent.
L'analyse SEC est effectuée pour évaluer la solubilité des particules peptidiques. Le chromatogramme présenté dans la figure 3A montre une seule période de pointe symétrique à environ 14 ml (6 ml au-delà du volume vide de 8,0 ml sur la colonne de filtration de gel S200 10/300). La position de ce pic, au-delà du volume vide, confirme la solubilité des particules FhuA Bio-Peptidisc. À titre de référence, la figure 3B illustre une préparation théorique au peptidisque sous-optimale. Dans ce cas, le chromatogramme montre un pic de protéine plus grand élitant au volume vide, indiquant la présence des agrégats de protéine. Le pic plus petit qui vient juste après le pic peptidisque principal correspond à l'excès de peptides.
L'interaction de FhuA avec l'analyte ColM est déterminée après l'attachement des peptidisques aux capteurs enduits de streptavidin. Les pointes du capteur sont d'abord libéées dans un tampon cinétique, puis transférées dans un tampon contenant les Bio-Peptidiscs FhuA. La concentration des Bio-Peptidiscs FhuA et la durée pendant laquelle ils sont incubés avec la pointe sont optimisées avant cette expérience(figure supplémentaire 1). Après les étapes de chargement et de lavage, l'étape d'association mesure les interactions entre les pointes chargées et quatre concentrations différentes de colicine M. Le mouvement ultérieur des pointes de nouveau dans le tampon a comme conséquence la dissociation, qui est mesurée par le décalage de longueur d'onde de la lumière blanche se reflétant outre de la pointe.
Figure 4A affiche la sortie de sensorgram de données brutes de cette expérience. Toutes les traces semblent uniformes dans les étapes de chargement et de ligne de base, à l'exception de la trace de référence en jaune. La pointe de référence n'a pas de ligand chargé, mais est exposée à l'analyte pour l'essayer pour la liaison non spécifique, qui est un contrôle négatif important. Pour une analyse plus détaillée des résultats, le signal de la pointe de référence est soustrait des traces pour les conseils expérimentaux pour tenir compte de la liaison non spécifique. Les données sont alignées sur le début de l'étape d'association pour permettre une comparaison visuelle directe, comme le montre la figure 4B. Cette comparaison montre une augmentation du décalage de longueur d'onde pour augmenter les concentrations de colicine M. La courbe est adaptée aux données et présente un modèle de liaison classique 1:1.
La parcelle de vue résiduelle(figure 4B, en bas), qui décrit les différences entre les données d'ajustement et expérimentales, montre que l'ajustement pour la concentration la plus élevée de ColM (28 nM) est faible par rapport aux trois concentrations plus faibles. Le profil de la courbe elle-même n'a pas le plateau de courbe de liaison, qui est caractéristique de la saturation du site de liaison dans une expérience de liaison typique. L'absence de plateau suggère une liaison hétérogène, qui est probablement un artefact de la forte concentration de ColM. Cette concentration de ColM la plus élevée est donc écartée de l'analyse, et les trois autres concentrations sont utilisées pour déterminer la constante de dissociation (figure 4C,D). Le test t-testd'un étudiant à deux queues, à un niveau de confiance de 95 %, a révélé que la constante de dissociation observée de cette expérience n'est pas significativement différente des valeurs obtenues à l'aide de différentes techniques (figure 4E)16.

Figure 1 : Aperçu du flux de travail PeptiQuick à l'aide de Bio-Peptidiscs et de l'analyse BLI. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 2 : Analyse du gel SDS (12 %) fractions de démarrage, de débit, de lavage et d'élution collectées par le flux de travail PeptiQuick. Environ 10 l'échantillon ont été chargés dans chaque voie et ont couru pendant 30 min à un courant constant de 60 mA. Le gel a été taché avec le bleu de Coomassie, destained, et visualisé sur un scanner de gel. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
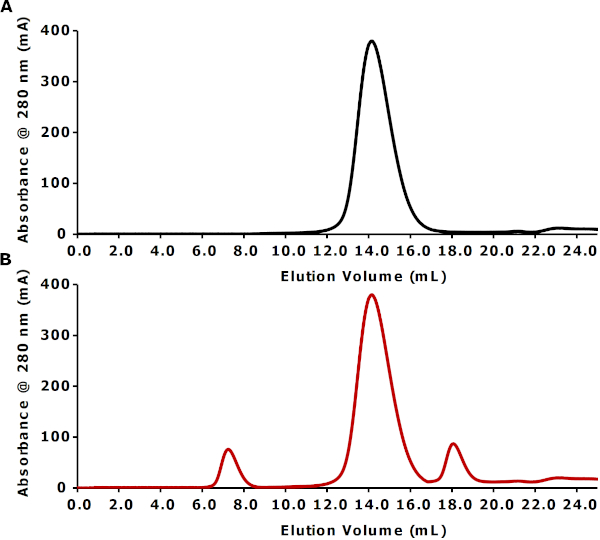
Figure 3 : Profils SEC expérimentaux et théoriques représentant des reconstitutions optimales et sous-optimales, respectivement. (A) Profil SEC expérimental de PeptiQuick reconstitué FhuA (82 kDa) en Bio-Peptidiscs. Les fractions d'élution IMAC F3-F7 ont été mises en commun et concentrées à l'aide d'un concentrateur centrifuge de 30 kDa à 1 ml. Cet échantillon concentré a été injecté à 0,25 ml/min sur une colonne de filtration de gel S200 (10/300) dans le tampon TSG. (B) Profil théorique de la SEC d'une reconstitution PeptiQuick sous-optimale. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
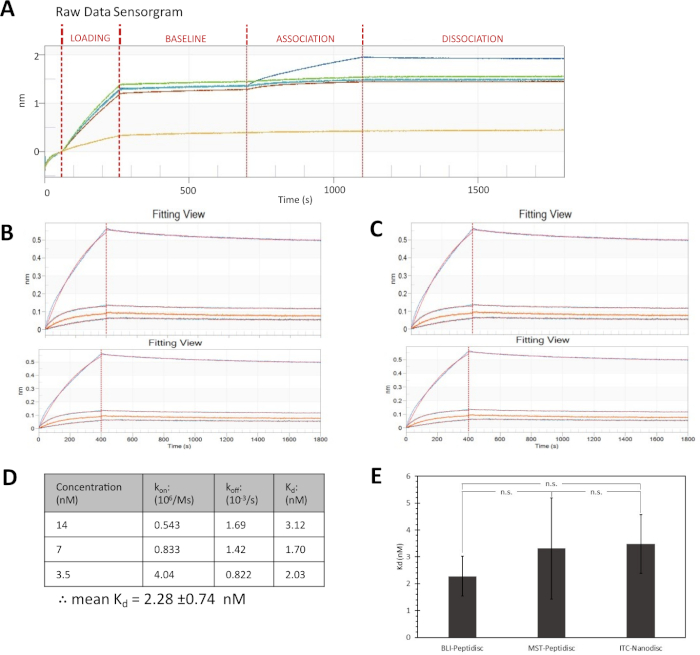
Figure 4 : Étudier les interactions colM avec FhuA reconstituées dans les Bio-Peptidiscs. (A) Capteur de données brutes avec trace de capteur de référence en jaune. (B,C) Haut : étapes d'association et de dissociation avec raccord partiel de courbe de fixation 1:1. En bas : vues résiduelles illustrant la différence entre les données expérimentales et l'ajustement computationnel. (C) Replotting de (B) avec l'exclusion de la plus forte concentration de ColM. (D) Taux d'association et de dissociation observés (kon et koff, respectivement) et les constantes de dissociation (Kd). (E) Comparaison des constantes de dissociation FhuA-ColM obtenues par peptidisc et BLI (dans cette expérience), le peptidisque et la thermophoresis micro-échelle (Saville, données non publiées), et la calorimétrie de titration nanodisc et isothermique16. Les barres d'erreur représentent un SD d'incertitude, tandis que n.s. ne dénote aucune différence significative au niveau de confiance de 95%. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| M9 Media (par litre) |
| 200 ml de sel M9 |
| 800 mL dH2O |
| 10 mL 40% de glucose |
| 80 L 1 M CaCl2 |
| 2,5 mL 1 M MgSO4 |
| 300 l 15 mg/mL de thiamine |
| Tampon TSG |
| 50 mM Tris-HCl, pH 7,8 |
| 50 mM NaCl |
| 10% de glycérol |
| Tampon de lavage IMAC |
| 50 mM Tris-HCl, pH 7,8 |
| 50 mM NaCl |
| 10% de glycérol |
| 0,04 % LDAO |
| 5 mM DM Imidazole |
| Bio-Peptidisc Solution |
| Dissoudre le Bio-Peptidisc prêt à l'emploi (la pureté de 95 %) dans dH2O stérile. La concentration et le volume de la solution peptide dépendent de l'étape du processus de reconstitution. |
| 50 mM Tris-HCl, pH 7,8 |
| 50 mM NaCl |
| REMARQUE : Ce stock de peptides bio-peptidiscest est stable à 4 oC pendant plus d'un mois. |
| Tampon d'élution IMAC |
| 50 mM Tris-HCl, pH 7,8 |
| 50 mM NaCl |
| 10% de glycérol |
| 600 mM Imidazole |
| Tampon cinétique |
| 50 mM Tris-HCl, pH 7,8 |
| 50 mM NaCl |
| 0,002% Entre-20 |
| 0,1 % BSA |
Tableau 1 : Recettes pour la préparation des médias et des solutions.
Figure supplémentaire 1 : analyse claire du gel indigène de 4 % à 16 % des fractions d'élution recueillies dans le flux de travail PeptiQuick. Environ 10 l'échantillon ont été chargés dans chaque voie et ont couru pendant 40 min à un courant constant de 30 mA. Le gel a été taché avec le bleu de Coomassie, destained, et visualisé sur un scanner. S'il vous plaît cliquez ici pour télécharger ce chiffre.
Figure supplémentaire 2 : Optimisation de la concentration de Ligand à l'aide d'une seule broche. (A) Les six concentrations différentes de FhuA dans Bio-Peptidisc testé pour la liaison à une seule goupille enduite de streptavidin. Ceci a été mis en place dans une plaque de puits 96 avec tampon dans les puits entre les concentrations de ligands (voir Le fichier supplémentaire 1 pour le protocole d'configuration). (B) Sensorgram de données brutes pour l'expérience d'optimisation de ligand. La broche donne la plus grande réponse et atteint en outre la saturation au numéro de concentration 4 (ou 2,5 g/mL). S'il vous plaît cliquez ici pour télécharger ce chiffre.
Fichier supplémentaire 1. S'il vous plaît cliquez ici pour voir ce fichier (Clic droit pour télécharger).
Discussion
Tandis que les détergents restent la méthode la plus simple pour extraire et purifier les protéines membranaires, ces surfactants peuvent avoir beaucoup d'effets indésirables sur la stabilité de protéine, la fonction, et les analyses en aval1,2,3, 4,5,6,7,8,9. Ces difficultés ont motivé le développement de mimétiques membranaires, qui s'efforcent de minimiser la présence de détergents et de reproduire l'environnement membranaire indigène autant que possible2,3,4 ,5,6. La majorité des méthodes de reconstitution, cependant, nécessitent une optimisation significative des conditions de reconstitution et nécessitent souvent des étapes de purification supplémentaires, qui diminuent le rendement final7,8. Le peptidisque s'adapte spontanément à la protéine membranaire cible et, comparativement, nécessite peu d'optimisation et de purification en aval9,10. Dans ce protocole, PeptiQuick est présenté comme un moyen simple de rationaliser le protocole de reconstitution pour l'analyse d'interaction protéine-protéine en aval.
Bien que simples, il existe plusieurs mises en garde expérimentales qui peuvent conduire à une reconstitution infructueuse. Parmi ceux-ci, le plus commun est dû à l'agrégation de protéine. Il est donc essentiel d'effectuer une chromatographie d'exclusion de taille pour surveiller le processus de reconstitution. Par exemple, un pic d'exclusion de taille élutant au volume vide est indicatif des agrégats de protéines (Figure 3B)17,18. Les agrégats de protéines membranaires se forment généralement lors d'une exposition prolongée à des conditions détergentes sous-optimales avant la reconstitution. En particulier, il a été constaté que les protéines membranaires dans le détergent ont tendance à former des agrégats lorsqu'elles sont concentrées par ultrafiltration sur des dispositifs centrifuges. Dans ce cas, les protéines membranaires sensibles peuvent être concentrées à l'aide de l'ultra-filtration sous vide, une méthode de concentration plus douce et plus homogène puisque l'adsorption des protéines au filtre est diminuée.
En général, pour éviter la concentration de protéines, les fractions IMAC éluées doivent être mises en commun, et un aliquot injecté sur une colonne d'exclusion de taille pour vérifier sa qualité de reconstitution. Il convient de noter que les élutes de peptide libre non incorporées juste après le pic peptidisque principal (figure 3B). L'échafaudage libre n'entrave pas nécessairement les expériences en aval. Cependant, si nécessaire, cet excès peut être enlevé par la chromatographie d'exclusion de taille. Alternativement, l'augmentation du volume de lavage pendant la reconstitution, et avant l'élution de la résine IMAC, est suffisante pour enlever efficacement la plupart du peptide libre. Par conséquent, la chromatographie d'exclusion de taille est recommandée comme moyen rapide et simple de vérifier la qualité de reconstitution.
L'expérience BLI nécessite une optimisation minutieuse des concentrations de ligands et d'analytes. La liaison Ligand doit être suffisante pour obtenir un signal clair, mais la surcharge entraînera la saturation du signal, ce qui entraîne des artefacts de données du surpeuplement et de l'obstacle stérilisé sur la surface de pointe. Par conséquent, la concentration du ligand et la durée du temps que la pointe passe dans la solution ligand doivent être optimisées pour chaque échantillon de protéines(figure supplémentaire 2). La concentration d'analytes doit également être optimisée. Si la constante de dissociation est connue, cette étape devient plus facile, puisque la plage de concentration peut être approximative. Un bon point de départ pour cette analyse est d'utiliser des concentrations de protéines entre 0,1 et 20 fois le Kdprévu 19.
Après l'acquisition de données BLI, une analyse minutieuse des données doit être effectuée pour éviter toute mauvaise interprétation. Le calcul de la constante de dissociation dépend de l'ajustement d'une courbe de liaison. À moins que la stoichiométrie de liaison soit déjà connue, un modèle classique d'interaction bimoléculaire 1:1 devrait être employé pour l'ajustement initial. Il convient de noter qu'une courbe de liaison hétérogène est souvent le résultat d'artefacts et d'un comportement non idéal causé par une forte concentration d'analytes, qui peut être mal interprétée comme un modèle de liaison complexe. Par conséquent, l'abaissement de la concentration d'analytes jusqu'à ce que le profil sensorgram affiche 1:1 stoichiométrie de liaison peut aider à différencier la liaison hétérogène des interactions plus complexes. Toutes les données de liaison hétérogènes résiduelles sont ensuite actualisées comme le montre la figure 420.
Dans ce rapport, une constante de dissociation de 2,28 à 0,74 nM pour l'interaction FhuA-ColM est mesurée. Cette valeur est compatible avec la constante de dissociation précédemment déterminée dans notre groupe avec nanodisque ou peptidisque utilisant ITC ou MST, respectivement (Figure 4E)16. Cette cohérence donne confiance sur la reconstitution peptidisc et l'analyse BLI comme un moyen de déterminer la cinétique interaction. Fait important, il convient de noter que les protéines sont généralement immobilisées sur les biocapteurs de streptavidin soit par biotine chimique cross-linking ou l'ajout spécifique au site en utilisant la transtin e. coli ligaseBirA 21. De toute évidence, la biotinylating de l'échafaudage peptidisque, au lieu de la protéine de membrane cible, a beaucoup d'avantages. La biotinylation des échafaudages permet de gagner du temps et de réduire au minimum le risque de perturber d'importants sites de liaison protéique. Nous avons également constaté que PeptiQuick s'applique à un large éventail de classes cibles protéiques, y compris les récepteurs couplés aux protéines G (GPCR), les canaux ioniques et les protéines membranaires des barils d'Ile-de-France. En général, il convient de noter que l'extrait détergent initial des protéines membranaires dans un état exempt d'agrégats est essentiel, et que la reconstitution immédiate en peptidisc diminue les problèmes d'agrégation en aval. Compte tenu de la simplicité, il est prévu que PeptiQuick sera étendu à d'autres essais de liaison à base de streptavidin, tels que la résonance plasmon de surface (SPR), essais ELISA, et l'affinité pull-downs en utilisant des perles de streptavidin.
Déclarations de divulgation
FD est le fondateur de Peptidisc Biotech pour distribuer des peptides peptidisques à la communauté universitaire. Peptidisc Biotech s'associe également à des sociétés biotechnologiques et pharmaceutiques industrielles pour mettre en œuvre le peptidisc dans leurs workflows de découverte. La publication Open Access de cet article a été parrainée par FortéBio.
Remerciements
Nous remercions le Conseil de recherches en sciences naturelles et en génie du Canada. JS détient une bourse d'études des IRSC de la SCG-M. LT a reçu l'appui du Partenariat de formation doctorale en biosciences du Sud-Ouest financé par le Conseil des biotechnologies et des sciences biologiques [référence de la subvention de formation BB/M009122/1]. FD est titulaire d'une chaire de recherche du Canada de niveau II.
matériels
| Name | Company | Catalog Number | Comments |
| 30 kDa cut-off centrifugal concentrator | Millipore Sigma | C7715 | - |
| Ampicillin | BioShop | 69-52-3 | Sodium Salt |
| Bio-Peptidisc Peptide | Peptidisc Biotech | https://peptidisc.com | - |
| Bovine Serum Albumin (BSA) | Sigma | 9048-46-8 | Lyophilized powder |
| CaCl2 | Fisher Chemical | 10035-04-8 | Certified ACS |
| EDTA | BioShop | 6381-92-6 | Biotechnology Grade |
| Ettan LC (AKTA) | Amersham Pharmacia Biotech | 18-1145-58 | - |
| Glucose | BioShop | 50-99-7 | Anhydrous, Reagent Grade |
| Glycerol | Fisher Chemical | 56-81-5 | Certified ACS |
| Imidazole | BioShop | 288-32-4 | Biotechnology Grade |
| Lauryldimethylamine oxide (LDAO) | Sigma | 101822204 | ~30% in H2O |
| MgSO4 | Fisher Chemical | 10034-99-8 | Certified ACS |
| Microfluidizer | Microfluidics | M-110L | Fit with F20Y 75µ diruption chamber |
| Ni-NTA Resin | Gold Biotechnology | H-320-50 | - |
| Non-binding 96well BLI Plate | Greiner Bio-one | 655076 | Microplate, PS, 96 well, F-Bottom (chimney well), Black, Fluotrac, Med. Binding |
| Octet Red96 | FortéBio | OCTET RED96E-GxP | Octet RED96e instrument, Octet CFR software, desktop computer, LC monitor, accessory kit, IQ/OQ kit, PQ Kits and one-year warranty |
| Phenylmethanesulfonylfluoride (PMSF) | Sigma | P-7626 | - |
| Protein Assay Dye - Reagent Concentrate | Bio-Rad | 5000006 | Used in the bradford assay to determine protein concentration - 5x concentration |
| Sodium Chloride (NaCl) | BioShop | 7647-14-5 | Biotechnology Grade |
| Streptavidin (SA) Biosensors | ForteBio | 18-5019 | One tray of 96 biosensors coated with streptavidin for quantitation, screening, or kinetic applications. |
| Superdex S200 (300/10) | Amersham Pharmacia Biotech | 175175-01 | - |
| Thiamine | Merck | 69271 | - |
| Tris-HCl | BioShop | 77-86-1 | Reagent Grade |
| Triton X-100 | BioShop | 900998-1 | Biotechnology Grade |
| Tween-20 | BioShop | 56-40-6 | Reagent Grade |
| Ultra centrifuge | Beckman Coulter | 365668 | - |
Références
- Rawlings, A. E. Membrane proteins: always an insoluble problem. Biochemical Society Transactions. 44 (3), 790-795 (2016).
- Popot, J. L. Amphipols, Nanodiscs, and Fluorinated Surfactants: Three Non Conventional Approaches to Studying Membrane Proteins in Aqueous Solutions. Annual Review of Biochemistry. 79, 737-775 (2010).
- Lee, S. C., et al. A Method for Detergent-Free Isolation of Membrane Proteins in their Local Lipid Environment. Nature Protocols. 11, 1149-1162 (2016).
- Frauenfeld, J., et al. A Saposin-Lipoprotein Nanoparticle System for Membrane Proteins. Nature Methods. 13, 345-351 (2016).
- Bayburt, T. H., Grinkova, Y. V., Sligar, S. G. Assembly of Single Bacteriorhodopsin Trimers in Bilayer Nanodiscs. Archives of Biochemistry and Biophysics. 450, 215-222 (2006).
- Denisov, I. G., Grinkova, Y. V., Lazarides, A. A., Sligar, S. G. Directed Self-Assembly of Monodisperse Phospholipid Bilayer Nanodiscs with Controlled Size. Journal of the American Chemical Society. 126, 3477-3487 (2004).
- Denisov, I. G., Sligar, S. G. Nanodiscs for Structural and Functional Studies of Membrane Proteins. Nature Structural, Molecular Biology. 23, 481-486 (2016).
- Hagn, F., Etzkorn, M., Raschle, T., Wagner, G. Optimized Phospholipid Bilayer Nanodiscs Facilitate High-Resolution Structure Determination of Membrane Proteins. Journal of the American Chemical Society. 135, 1919-1925 (2013).
- Carlson, M. L., et al. The Peptidisc, A Simple Method for Stabilizing Membrane Proteins in Detergent-Free Solution. eLife. 7, 34085 (2018).
- Carlson, M. L., et al. Profiling the E. coli Membrane Interactome Captured in Peptidisc Libraries. eLife. 8, 46615 (2019).
- Serdakowski, A., et al. A novel method to determine residual detergent in biological samples post endotoxin reduction treatment and evaluation of strategies for subsequent detergent removal. International Immunopharmacology. 37, 16-22 (2016).
- Smith, S. M. Strategies for the Purification of Membrane Proteins. Protein Chromatography. 681, 485-496 (2011).
- Lin, S. H., Guidotti, G. Purification of Membrane Proteins. Methods in Enzymology. 463, 619-629 (2009).
- Wolfe, A. J., Gugel, J. F., Chen, M., Movileanu, L. Kinetics of Membrane Protein-Detergent Interactions Depend on Protein Electrostatics. Journal of Physical Chemistry B. 122 (41), 9471-9481 (2018).
- Montigny, C., et al. Slow Phospholipid Exchange between a Detergent-Solubilized Membrane Protein and Lipid-Detergent Mixed Micelles: Brominated Phospholipids as Tools to Follow Its Kinetics. PLoS ONE. 12 (1), 0170481 (2017).
- Mills, A. T., Le H, T., Coulton, J. W., Duong, F. FhuA Interactions in a Detergent-Free Nanodisc Environment. Biochimica et Biophysica Acta - Biomembranes. 1838 (1), 364-371 (2014).
- Hong, P., Koza, S., Bouvier, E. S. P. Size-Exclusion Chromatography for the Analysis of Protein Biotherapeutics and their Aggregates. Journal of Liquid Chromatography Related Technologies. 35 (20), 2923-2950 (2012).
- Mahler, H. C., Friess, W., Grauschopf, U., Kiese, S. Protein aggregation: Pathways, induction factors and analysis. Journal of Pharmaceutical Sciences. 98 (9), 2909-2934 (2009).
- Trinkle-Mulcahy, L. Recent advances in proximity-based labeling methods for interactome mapping. F1000Research. 8, 135 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon