Method Article
PeptiQuick, uma incorporação de um passo de proteínas da membrana em Peptidiscs biotinylated para ensaios obrigatórios de proteína aerodinâmica
Neste Artigo
Resumo
Apresentamos um método que combina purificação de proteína de membrana e reconstituição em peptidiscs em um único passo cromatográfico. Andaimes biotinylados são usados para fixação direta da superfície e medição de interações proteína-ligand através da interferometria da biocamada.
Resumo
As proteínas da membrana, incluindo transportadores, canais e receptores, constituem quase um quarto do proteoma celular e mais da metade dos alvos atuais de medicamentos. No entanto, uma grande barreira à sua caracterização e exploração em ambientes acadêmicos ou industriais é que a maioria das estratégias de triagem bioquímica, biofísica e de drogas exigem que essas proteínas estejam em um estado solúvel em água. Nosso laboratório desenvolveu recentemente o peptidisc, um mimético da membrana que oferece "one-size-fits-all" aproximação ao problema da solubilidade da proteína da membrana. Apresentamos aqui um protocolo simplificado que combina purificação de proteínas e reconstituição peptidisc em um único passo cromatográfico. Este fluxo de trabalho, denominado PeptiQuick, permite contornar a diálise e a incubação com grânulos de poliestireno, reduzindo assim extremamente a exposição ao detergente, à desnaturação da proteína, e à perda da amostra. Quando PeptiQuick é realizado com andaimes biotinylated, a preparação pode ser diretamente ligada a superfícies revestidas de streptavidina. Não há necessidade de biotinylate ou modificar o alvo da proteína da membrana. PeptiQuick é apresentado aqui com o receptor de membrana FhuA e antimicrobiana ligand colicin M, usando interferometria biocamada para determinar a cinética precisa de sua interação. Conclui-se que peptiquick é uma maneira conveniente de preparar e analisar membrana proteína-ligand interações dentro de um dia em um ambiente livre de detergentes.
Introdução
As proteínas da membrana são muitas vezes excluídas dos programas de pesquisa de descoberta de medicamentos ou anticorpos devido à propensão de proteínas de membrana para agregar fora do ambiente de bicamada lipídica, especialmente na presença de detergentes1. Portanto, nos últimos anos, vários miméticos de membrana (andaimes denominados) foram desenvolvidos para facilitar o isolamento e interrogatório de proteínas de membrana em um ambiente completamente livre de detergente (ou seja, nanodiscos, SMALPs, anfídios, etc.) 2,3,4,5,6. No entanto, a reconstituição das proteínas de membrana nestas mímicas muitas vezes requer otimização extensiva, que é demorada e geralmente acompanhada de perda de recuperação deproteínas7,8. Para superar essas limitações, nosso laboratório desenvolveu recentemente uma formulação "one-size-fits-all" conhecida como peptidisc9. O peptidisc é formado quando várias cópias de um designer 4.5 kDa pítico píático peptídeo biedático ligar à superfície hidrofóbica de uma proteína de membrana alvo. Reconstituição estável em peptidisc ocorre após a remoção de detergente, prendendo lipídios endógenos e proteínas de membrana solubilizadas em partículas solúveis em água. Estas partículas estabilizadas são agora passíveis para inúmeras aplicações a jusante.
O método peptidisc oferece várias vantagens; por exemplo, a reconstituição é simples, uma vez que a ligação do andaime peptidisc sobre o alvo é guiada pelo modelo de proteína em si9,10. A stoichiometria peptídea também é autodeterminada, e a adição de lipídios exógenos não é necessária. A formação Peptidisc ocorre por diluição detergente simples, uma vantagem importante sobre diálise ou adsorção em contas de poliestireno, que muitas vezes resultam em baixo rendimento de proteína devido à associação de superfície não específica e agregação11,12 13depessoas. A montagem final peptidisc é altamente termostável e invariavelmente solúvel em diferentes buffers ou na presença de cations divalentes (por exemplo, Ni2+). A pureza e a homogeneidade estrutural do andaime também são altas (por exemplo, livres de endotoxinas), e o peptídeo pode ser personalizado com grupos funcionais colocados em diferentes posições.
Apresentamos aqui um fluxo de trabalho de laboratório chamado PeptiQuick, também conhecido como reconstituição em contas9. Este protocolo combina purificação de proteína de membrana e reconstituição peptidisc em um único passo e no mesmo suporte cromatográfico. Como o nome indica, PeptiQuick é rápido em comparação com outros métodos de reconstituição, e também reduz seriamente o tempo de exposição ao detergente. Efeitos negativos de detergente, como desdobramento proteico e agregação, muitas vezes ocorrem de forma dependente do tempo; Portanto, minimizar a exposição ao detergente é fundamental para manter a conformação de proteínas nativas14,15. Isto é crucial para a precisão dos métodos que relatam sobre interações proteicas e afinidades de ligação.
Ao desenvolver este protocolo, apresentamos a nova versão biotinylated do andaime peptidisc, denominado Bio-Peptidisc. Os grupos funcionais da biotina permitem o acessório da proteína da membrana do alvo em superfícies streptavidin-revestidas. Como a rotulagem de biotina é limitada ao andaime, os locais de ligação na proteína da membrana alvo permanecem inalterados. Usando Bio-Peptidisc, a cinética de ligação do receptor de membrana bacteriana FhuA e o peptídeo antimicrobiano cólicina M (ColM) são determinados16. Essa afinidade é medida por meio de interferometria biocamada (BLI), que analisa interações em tempo real com base em padrões de interferência de luz branca refletidos a partir de uma ponta do sensor.
Usando este protocolo, a necessidade de detergente durante a análise bli é eliminada, que é um desenvolvimento importante, como detergentes podem interromper as interações. As afinidades de ligação podem ser medidas rapidamente com este método, e os resultados são comparáveis aos relatados anteriormente usando nanodiscos e calómetria de titria isomal (ITC)16. Os passos críticos no fluxo de trabalho PeptiQuick são mostrados e discutidos, como preparação de proteínas, diluição de detergente, adição de peptídeos e reconstituição, juntamente com dicas para solucionar problemas de ligantes e analitos no ensaio bli. Usando o fluxo de trabalho PeptiQuick, verificou-se que as proteínas da membrana podem ser capturadas em peptidiscs e suas interações medidas dentro de um dia.
Protocolo
1. Preparação e solubilização do receptor de membrana FhuA
- Expresse seuFhuA6-etiquetado na tensão AW740 do coli de Escherichia. Crescer as células por 18 h em 37 °C em m9 mídia (Tabela 1). Veja Mills et al.16 para um protocolo de expressão detalhado.
- Colheas células por centrífuga (5.000 x g,10 min, 4 °C) e as resuspendem em 50 mL de tampão tris-sal-glicerol (TSG) (Tabela 1). Dounce as células resuspendidas e adicionar fenilmetanosulfonylfluoreto (PMSF) a uma concentração final de 1 mM pouco antes da lyse.
CUIDADO: Pmsf é tóxico e corrosivo. - Lyse as células usando um microfluidizer (três passagens) em 15.000 psi ou uma imprensa francesa (três passagens) em 8.000 psi. Pelotas as células não lysed e outros materiais insolúveis por centrífuga de baixa velocidade (5.000 x g,10 min, 4 °C).
- Ultracentrifuge o supernatant (200.000 x g,40 min, 4 °C) para isolar a fração da membrana bruta. Descarte o supernatant, resuspenda a pelota da membrana em uma quantidade mínima de tampão de TSG, e dounce usando um instrumento do uncer do vidro ou do metal para assegurar a homogeneidade.
- Realize um ensaio de Bradford para verificar a concentração de proteína da membrana bruta resuspendida. Diluir a membrana bruta para uma concentração final de 3 mg/mL usando tampão TSG antes da solubilização.
- Adicione o detergente Triton X-100 a uma concentração final de 1% (v/v) para solubilizar seletivamente a membrana interna bacteriana por 1 h a 4 °C com balanço suave. Pelotas o material insolúvel (fração de membrana externa) por ultracentrífugation (200.000 x g,40 min, 4 °C).
- Descarte o supernatant contendo a membrana solubilizada e resuspenda a pelota no TSG para uma concentração final de 3 mg/mL. Adicione o óxido de laurildimamina (LDAO) a uma concentração de 1% (v/v) à fração de membrana externa resuspendida e solubilize por 1 h a 4 °C com balanço suave.
- Realize um passo de centrifugação final para pelota todo o material insolúvel (200.000 x g,40 min, 4 °C).
NOTA: O supernatant resultante contém as proteínas solubilizadas da membrana externa, incluindo o alvo seu FhuA marcado.
2. Purificação e reconstituição da FhuA usando o fluxo de trabalho PeptiQuick
- Pré-equilibra uma coluna Ni-NTA pré-embalada (volume de resina de 5 mL) com dois volumes de coluna de tampão de lavagem de cromatografia de metal imobilizado (IMAC)(Tabela 1).
- Diluir a membrana exterior solubilizada de 1% LDAO para 0,04% LDAO usando TSG. Em seguida, adicione o imidazol a uma concentração final de 5 mM.
CUIDADO: Imidazolé é tóxico e corrosivo. - Carregue as proteínas solubilizadas da membrana externa na resina de Ni-NTA e colete o fluxo. Recarregue o fluxo através da resina para aumentar a ligação de resina de FhuA e recolher o fluxo secundário.
NOTA: Mantenha um alibquot de 20 μL de material solubilizado como referência para uma análise mais tarde de sulfato dofedlo de sódio poliacrilamida gel (SDS-PAGE) análise. - Lave a resina com 250 mL de tampão de lavagem IMAC e recolher os primeiros 50 mL de eluate. Escorra o tampão da lavagem à altura do volume da cama da resina e feche o stopcock na coluna.
- Adicione 1 mL de soluçõe concentrada de peptídeos 10 mg/mL Bio-Peptidisc(Tabela 1)à coluna. Adicione 50 mL de diluído 1 mg/mL Bio-Peptidisc peptídeo solução em TSG e mexa a resina com uma haste de vidro para resuspender as contas em TSG.
- Após a captura peptidisc, permitir que a resina para resolver e drenar o excesso de 1 mg / mL solução Bio-Peptidisc através da resina.
- Lave as partículas peptidisc anexadas ni-NTA com 50 mL de TSG. Elute as partículas peptidisc com 15 mL de tampão de elução IMAC (Tabela 1) contendo 600 mM imidazole no TSG. Colete 1 mL frações e adicione imediatamente 10 μL de 0,5 M EDTA para quelam íons de níquel lixiviados.
CUIDADO: EDTA é um irritante.
3. Avaliação da reconstituição PeptiQuick
- Análise do SDS-PAGE
- Carregue 10 μL alíquotas do material inicial, flowthrough, wash (es) e frações de eluted da reconstituição PeptiQuick em um gel e eletroforrese de 12% que desemnizam SDS por 30 min a uma corrente constante de 60 mA.
- Manchar o gel com tinaria azul Coomassie, destain o gel, e visualizar em um scanner.
- Cromatografia de exclusão de tamanho (SEC)
- Com base na análise de 12% da Reconstituição PeptiQuick, isolá-lo e reunir as frações relevantes, e concentrá-los usando um concentrador centrífuga de corte de 30 kDa.
NOTA: No vídeo que acompanha, frações F3-F7 são agrupados e concentrados abaixo de 1 mL (o volume de loop de injeção no instrumento SEC). - Injetar ~ 1 mL das frações de elução IMAC agrupadas em uma filtragem de gel S200 (300/10) coluna a uma taxa de fluxo de 0,25 mL / min em buffer TSG. Colete 1 mL frações e executá-los em um gel SDS de 12% para determinar quais frações SEC para piscina e concentração.
NOTA: As frações peptiQuick eluted podem ser agrupadas sem concentração, e um alibamento pode ser injetado na SEC para verificar a qualidade da reconstituição. Este é um controle importante para proteínas de membrana que são potencialmente sensíveis à agregação durante a concentração centrífuga.
- Com base na análise de 12% da Reconstituição PeptiQuick, isolá-lo e reunir as frações relevantes, e concentrá-los usando um concentrador centrífuga de corte de 30 kDa.
4. Interferometria biocamada
- Configuração experimental bli
- Configure o prato de 96 poços à mão.
NOTA: Todos os poços são preenchidos a um volume final de 200 μL. - Na coluna 1, as linhas A-E, carregam 200 μL de cinética tampão para permitir que as dicas para equilibrar e formar um sinal de base.
- Diluir o ligand (FhuA em Bio-Peptidisc) a uma concentração de 2.5 μg/mL no amortecedor da cinética (tabela 1). Load 200 μL desta diluição nas linhas A-D na coluna 2.
- Adicione 200 μL de cinética tampão apenas para E2 (o sensor de referência).
- Adicione 200 μL de cinética tampão para coluna 3, linhas A-E para lavar o excesso de FhuA da ponta.
- Na coluna 4, conduza duas vezes diluições em série do anályte (ColM) abaixo da placa de A4 a D4. Comece com 28 nM (8*Kd) em A4 a 3.5 nM (1*Kd) em D4.
- Adicionar 28 nM ColM à E4 para medir a ligação não específica (a maior concentração de ColM utilizada).
- Adicione 200 μL de cinética tampão para coluna 5, linhas A-E.
NOTA: Aqui, o ColM irá dissociar a partir da ponta, ea dissociação é medida. - Coloque a placa de configuração no instrumento BLI.
- Coloque a bandeja de ponta do sensor no instrumento BLI.
- Abra o software de aquisição de dados BLI e selecione O Novo Experimento de Kinética no assistente de software.
- Use a guia de definição de placa para inserir o layout da placa de 96 poços no software.
NOTA: Os poços que contêm o ligand, FhuA são introduzidos como a "carga". Poços contendo buffer só são introduzidos como o "Buffer". Poços contendo o anályte, ColM, são introduzidos como a "Amostra". - Use a guia de definição de ensaio para definir o período de tempo e a velocidade de rotação da placa para cada etapa do experimento.
NOTA: As etapas experimentais são configuradas como: 1) linha de base: 60 s; 2) carregamento: 250 s; 3) linha de base2: 300 s; 4) associação: 450 s; e 5) dissociação: 900 s. Deixe a velocidade de shake como padrão (1.000 rpm). As etapas acima são atribuídas individualmente a cada coluna na placa de 96 poços, selecionando a etapa desejada e clique direito Adicionar Passo de Ensaio em cada coluna. - Atribua o primeiro passo clicando na primeira coluna e selecione Iniciar Novo Ensaio. Certifique-se de que a etapa de base seja corretamente atribuída usando a janela inferior do lado direito e altere com o menu suspenso conforme necessário.
- Atribuir as etapas subsequentes para cada coluna, clicando ao longo da placa e selecionando Adicionar Passo de Ensaio. Atribuí-los à coluna apropriada, conforme definido na etapa 4.1.13.
- Use a guia de atribuição do sensor para garantir que o instrumento de octeto esteja tirando os pinos BLI do local correto na bandeja do sensor.
NOTA: Na guia de atribuição do sensor, apenas A1-E1 deve ser destacado azul. Certifique-se de que uma ponta do sensor está presente nesses locais na bandeja do sensor e certifique-se de que o F1-H1 esteja vazio. - Destaque F1-H1 na tela, em seguida, clique direito e selecione Remover para marcar estes como vazios. Tick Substituir sensores na bandeja após o uso para reter os sensores usados.
- Na guia experimento de revisão, que fornece uma visão final do experimento antes da execução, passar por cima das etapas experimentais para garantir que toda a configuração está correta.
- Na guia de experimentos de execução, selecione um local de arquivo para salvar os arquivos do método.
- Mude a temperatura da placa para a temperatura ambiente.
- Selecione GO para executar o experimento.
- Configure o prato de 96 poços à mão.
- Análise de dados bli
- Abra o software de análise de dados Octet BLI.
- Use a guia de seleção de dados para localizar o experimento e verificar o resumo experimental. Importe o arquivo do projeto de sua localização de salvamento definido durante a etapa 4.1.19.
- Insume as concentrações do anályte (aqui, ColM) selecionando as informações do sensor para cada experimento.
- Atribuir a ponta na E1 como dica de referência.
- Use a guia de processamento para subtrair o sinal de referência (E1) dos dados experimentais. Verifique os dados brutos da ponta de referência para a ligação de anrólinha inespecífica. Se nenhum é observado, prossiga.
NOTA: Este é um controle negativo importante. Se for observada ligação inespecífica, isso será contabilizado na etapa 4.2.7. - Alinhe o eixo y à segunda etapa de linha de base. Não marque a conexão entre passos. Selecione a filtragem de Savitzky-Golay. Selecione dados de processo!. .
- Na guia de análise, selecione os dados experimentais na tabela abaixo do enredo para ver as curvas de associação e dissociação com o sinal do sensor de referência subtraído.
- Selecione o modelo de ligação 1:1 e selecione um ajuste parcial da curva. Selecione Fit Curves!.
- Analise o enredo de visualização residual para verificar o encaixe da curva.
- Percorra a mesa para ver o Calculado Kd.
- Selecione O Relatório salve... para salvar um documento de planilha detalhando a configuração, as parcelas dos dados brutos e analisados e as constantes de dissociação calculadas.
Resultados
O receptor de membrana FhuA é expresso em uma cepa de laboratório E. coli. A fração da membrana externa é colhida por centrífuga, e as proteínas são solubilizadas usando uma extração de detergente em duas etapas. As proteínas de membrana solubilizadas são carregadas em contas de agarose Ni-NTA embaladas em uma coluna de plástico, seguida pelo fluxo de trabalho PeptiQuick, conforme apresentado no protocolo. Depois de um controle de qualidade usando cromatografia de exclusão de tamanho, as partículas Bio-Peptidisc são imobilizadas em pinos revestidos de streptavidina. A análise bli é realizada para medir a cinética das interações entre FhuA e ColM. A visão geral esquemática deste experimento é apresentada na Figura 1.
Um gel SDS é executado para determinar a qualidade da reconstituição após a elução de partículas FhuA Bio-Peptidisc da coluna IMAC (Figura 2). Aliquots das frações correspondentes ao início, fluxo, lave, e frações de elução são carregados para o SDS-gel para avaliar o sucesso do método PeptiQuick. O esgotamento de FhuA na fração flowthrough é correlacionado com um enriquecimento nas frações de elução, o que indica que a purificação da proteína tem sido eficaz. Pequenas faixas de proteína contaminante são observadas nas frações eluted. A análise do gel SDS é usada para determinar quais frações devem ser agrupadas antes da análise da SEC. Um gel nativo das partículas fhua bio-peptidisc eluted também é executado para confirmar fhua solubilidade (Figura Suplementar 1). A migração da proteína reconstituída para o gel nativo indica a solubilidade proteica em um ambiente livre de detergentes.
A análise da SEC é realizada para avaliar a solubilidade das partículas peptidisc. O cromatograma apresentado na Figura 3A mostra um pico único e simétrico que se aluta a cerca de 14 mL (6 mL após o volume vazio de 8,0 mL na coluna de filtragem de gel S200 10/300). A posição deste pico, além do volume de vazio, confirma a solubilidade das partículas FhuA Bio-Peptidisc. Para referência, a Figura 3B ilustra uma preparação teórica de peptidisc suboptimal. Neste caso, o cromatograma mostra um pico de proteína maior que se aluta no volume nulo, indicando a presença de agregados proteicos. O pico menor que eluting apenas após o pico principal do peptidisc corresponde aos peptides adicionais do peptidisc.
A interação de FhuA com o análito ColM é determinada após o acessório dos peptidiscs aos sensores revestidos do streptavidin. As pontas do sensor são primeiramente equilibradas no amortecedor da cinética, a seguir transferidas no amortecedor que contem os Bio-Peptidiscs de FhuA. A concentração dos Bio-Peptidiscs fhua e o período de tempo para o qual são incubados com a ponta são otimizados antes deste experimento(Figura Suplementar 1). Seguindo as etapas de carregamento e lavagem, a etapa da associação mede as interações entre as pontas carregadas e quatro concentrações diferentes de cólicina M. O movimento subseqüente das pontas de volta no amortecedor conduz à dissociação, que é medida pelo deslocamento do comprimento de onda da luz branca que reflete fora da ponta.
A Figura 4A exibe a saída de sensorgramas de dados brutos deste experimento. Todos os traços aparecem uniformes nas etapas de carregamento e linha de base, além do traço de referência em amarelo. A ponta de referência não tem ligand carregado mas é expor ao analyte ao ensaio para o encaderamento inespecífico, que é um controle negativo importante. Para uma análise mais detalhada dos resultados, o sinal da ponta de referência é subtraído dos traços para que as pontas experimentais respondam para o encadernador inespecífico. Os dados estão alinhados ao início da etapa da associação para permitir uma comparação visual direta, como mostrado na Figura 4B. Esta comparação mostra um aumento na mudança de comprimento de onda para o aumento das concentrações de cólicina M. A curva está apta aos dados e exibe um modelo clássico de ligação 1:1.
O enredo de visão residual (Figura 4B,inferior), que descreve as diferenças entre os dados de montagem e experimental, mostra que a adequação para a maior concentração de ColM (28 nM) é pobre em comparação com as três concentrações mais baixas. O perfil da curva em si não tem o platô curva de ligação, que é característico da saturação do local de ligação em um experimento típico de ligação. A falta de planalto sugere ligação heterogênea, que é provavelmente um artefato da alta concentração de ColM. Essa maior concentração de ColM é, portanto, descontada da análise, e as outras três concentrações são usadas para determinar a constante de dissociação(Figura 4C,D). O teste tde um aluno de duas caudas, em um nível de confiança de 95%, constatou que a constante de dissociação observada deste experimento não é significativamente diferente dos valores obtidos usando diferentes técnicas (Figura 4E)16.

Figura 1: Visão geral para o fluxo de trabalho PeptiQuick usando bio-Peptidiscs e análise BLI. Clique aqui para ver uma versão maior deste número.

Figura 2: Análise de gel SDS (12%) de início, fluxo, lavagem e elução frações coletadas através do fluxo de trabalho PeptiQuick. Cerca de 10 μL de amostra foi carregado em cada pista e executado por 30 min em uma corrente constante de 60 mA. O gel foi manchado com Coomassie azul, desdetido, e visualizado em um scanner de gel. Clique aqui para ver uma versão maior deste número.
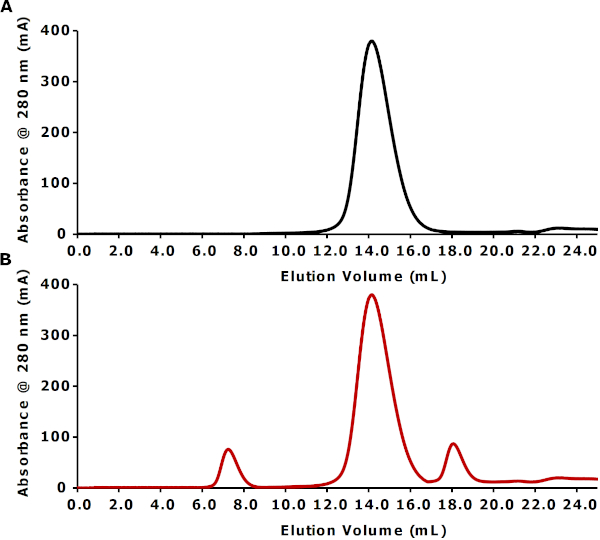
Figura 3: Perfis experimentais e teóricos da SEC que representam reconstituições ideais e abaixo do ideal, respectivamente. (A)Perfil experimental da SEC do PeptiQuick reconstituído FhuA (~82 kDa) em Bio-Peptidiscs. As frações de elução do IMAC F3-F7 foram agrupadas e concentradas usando um concentrador centrífuga de corte de 30 kDa para ~1 mL. Esta amostra concentrada foi injetada em 0,25 mL/min em uma coluna De filtração de gel S200 (10/300) no buffer TSG. (B) Perfil teórico da SEC de uma reconstituição peptiQuick suboptimal. Clique aqui para ver uma versão maior deste número.
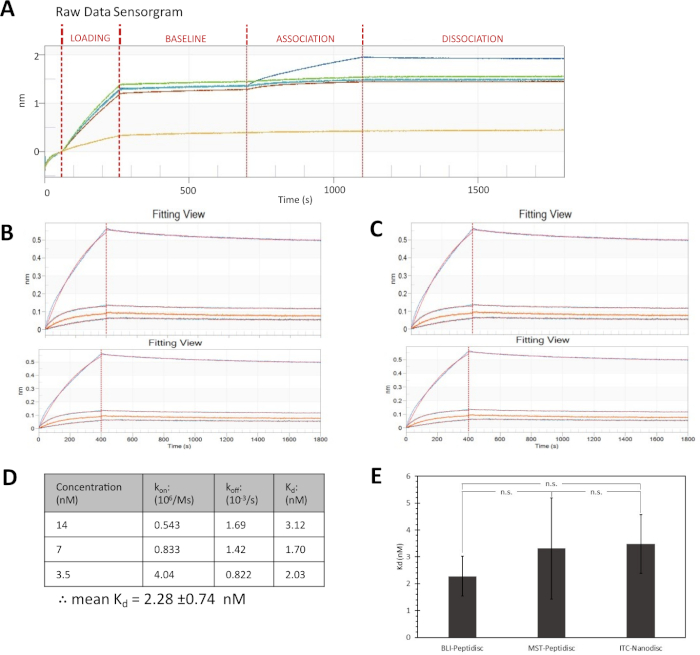
Figura 4: Investigar as interações da ColM com a FhuA reconstituídas em Bio-Peptidiscs. (A)Sensorgrama de dados brutos com sensor de referência rastreamento em amarelo. (B,C) ( B,C) Topo: etapas de associação e dissociação com encaixe parcial da curva de ligação de 1:1. Inferior: visões residuais que descrevem a diferença entre dados experimentais e encaixe computacional. (C) Reploting de (B)com a exclusão da maior concentração de ColM. (D)Taxas observadas de associação e dissociação (kon e koff,respectivamente) e as constantes de dissociação (Kd). (E) Comparação das constantes de dissociação fhua-ColM obtidas por peptidisc e BLI (neste experimento), peptidisc e termoforrese microescala (Saville, dados inéditos) e calómetria de titeração nanonível e isothermal16. As barras de erro representam um SD de incerteza, enquanto n.s. não denota diferença significativa no nível de confiança de 95%. Clique aqui para ver uma versão maior deste número.
| M9 Mídia (por litro) |
| 200 mL M9 sal |
| 800 mL dH 2 O 800 mL dH2O |
| 10 mL 40% glicose |
| 80 μl 1 M Cacl2 |
| 2,5 mL 1 M MgSO4 |
| 300 μL 15 mg/mL Tiamina |
| Tampão de TSG |
| 50 mM Tris-HCl, pH 7.8 |
| 50 mM NaCl |
| 10% glicerol |
| Tampão da lavagem de IMAC |
| 50 mM Tris-HCl, pH 7.8 |
| 50 mM NaCl |
| 10% glicerol |
| 0,04% LDAO |
| 5 mM Imidazole |
| Solução Bio-Peptidisc |
| Dissolva o Bio-Peptidisc pronto para uso (>95% de pureza) em dH estéril2O. A concentração e o volume da solução peptídea dependem do passo no processo de reconstituição. |
| 50 mM Tris-HCl, pH 7.8 |
| 50 mM NaCl |
| NOTA: Este estoque de peptídeos Bio-Peptidisc é estável em 4 °C por mais de um mês. |
| Tampão de elução do IMAC |
| 50 mM Tris-HCl, pH 7.8 |
| 50 mM NaCl |
| 10% glicerol |
| 600 m Imidazole |
| Tampão da cinética |
| 50 mM Tris-HCl, pH 7.8 |
| 50 mM NaCl |
| 0,002% Tween-20 |
| 0,1% BSA |
Tabela 1: Receitas para a preparação de mídia e soluções.
Figura suplementar 1: 4%-16% clara análise de gel nativo das frações de elução coletadas a partir do fluxo de trabalho PeptiQuick. Cerca de 10 μL de amostra foi carregado em cada pista e executado por 40 min em uma corrente constante de 30 mA. O gel foi manchado com Coomassie azul, desdetido e visualizado em um scanner. Por favor, clique aqui para baixar este número.
Figura suplementar 2: Otimização de concentração de ligand usando um único pino. (A)As seis concentrações diferentes de FhuA em Bio-Peptidisc testados para a ligação a um único pino revestido streptavidin. Isto foi criado em uma placa de 96 poços com tampão nos poços entre concentrações de ligand (ver Arquivo Suplementar 1 para protocolo de configuração). (B) Grama de sensor de dados brutos para o experimento de otimização de ligand. O pino dá a maior resposta e, adicionalmente, atinge a saturação na concentração número 4 (ou 2,5 μg/mL). Por favor, clique aqui para baixar este número.
Arquivo Suplementar 1. Clique aqui para ver este arquivo (Clique certo para baixar).
Discussão
Enquanto os detergentes continuam a ser o método mais simples para extrair e purificar proteínas de membrana, estes surfactantes podem ter muitos efeitos indesejados sobre a estabilidade de proteínas, função e análises a jusante1,2,3, 4,5,6,7,8,9. Essas dificuldades motivaram o desenvolvimento da mimética da membrana, que se esforça para minimizar a presença de detergentes e replicar o ambiente de membrana nativa, tanto quanto possível2,3,4 5,6. A maioria dos métodos de reconstituição, no entanto, exigem otimização significativa das condições de reconstituição e muitas vezes exigem etapas adicionais de purificação, que diminuem o rendimento final7,8. O peptidisc se adapta espontaneamente à proteína da membrana alvo e comparativamente, requer pouca otimização e purificação a jusante9,10. Neste protocolo, peptiquick é apresentado como um meio simples para simplificar o protocolo de reconstituição para a análise de interação proteína-proteína a jusante.
Embora simples, existem várias ressalvas experimentais que podem levar a uma reconstituição mal sucedida. Entre estes, o mais comum é devido à agregação de proteínas. Por conseguinte, é fundamental realizar cromatografia de exclusão de tamanho para acompanhar o processo de reconstituição. Por exemplo, um pico de exclusão de tamanho que se adiancüe no volume nulo é indicativo de agregados proteicos (Figura 3B)17,18. Os agregados da proteína da membrana dão forma tipicamente em cima da exposição prolongada às circunstâncias sub-optimal do detergente antes do reconstitution. Em particular, verificou-se que as proteínas de membrana em detergente tendem a formar agregados quando concentradas por ultrafiltração em dispositivos centrífugas. Neste caso, proteínas de membrana sensíveis podem ser concentradas usando ultra-filtração de vácuo, um método de concentração mais suave e homogêneo, uma vez que a adsorção de proteínas para o filtro é diminuída.
Em geral, para evitar a concentração de proteínas, as frações imac eluted devem ser agrupadas, e um alibamento injetado em uma coluna de exclusão de tamanho para verificar a sua qualidade de reconstituição. Note-se que elutes peptídeos livres não incorporados logo após o pico peptidisc principal(Figura 3B). O andaime livre não impede necessariamente experimentos a jusante. No entanto, se necessário, esse excesso pode ser removido pela cromatografia de exclusão de tamanho. Alternativamente, aumentar o volume de lavagem durante a reconstituição, e antes da elução da resina IMAC, é suficiente para remover efetivamente a maior parte do peptidisc peptidisc livre. Portanto, a cromatografia de exclusão de tamanho é recomendada como um meio rápido e simples para verificar a qualidade da reconstituição.
O experimento BLI requer otimização cuidadosa das concentrações de ligantes e análises. A ligação da ligand deve ser suficiente para obter um sinal claro, mas a sobrecarga causará saturação de sinal, o que resulta em artefatos de dados de superlotação e obstáculo stérico na superfície da ponta. Portanto, tanto a concentração do ligante quanto o período de tempo que a ponta gasta na solução de ligand devem ser otimizados para cada amostra de proteína(Figura Suplementar 2). A concentração de anrótes também deve ser otimizada. Se a constante de dissociação é conhecida, este passo torna-se mais fácil, uma vez que o alcance de concentração pode ser aproximado. Um bom ponto de partida para esta análise é o uso de concentrações de proteína entre 0,1 e 20 vezes o esperado Kd19.
Após a aquisição de dados bli, análise de dados cuidadosa deve ser realizada para evitar interpretações errôneas. O cálculo da constante de dissociação depende do ajuste de uma curva de ligação. A menos que a stoichiometria obrigatória já seja conhecida, um modelo clássico de interação bimolecular 1:1 deve ser usado para a montagem inicial. Note-se que uma curva de ligação heterogênea é muitas vezes o resultado de artefactos e comportamentonão ideal causado pela elevada concentração de anrólicos, que pode ser mal interpretada como um modelo de ligação complexo. Portanto, reduzir a concentração de anróste até que o perfil do sensorgrama exije a stoichiometria de ligação 1:1 pode ajudar a diferenciar a ligação heterogênea de interações mais complexas. Quaisquer dados de ligação heterogêneos residuais são então descontados, como mostrado na Figura 420.
Neste relatório, uma constante de dissociação de 2,28 ± 0,74 nM para a interação FhuA-ColM é medida. Esse valor é consistente com a constante de dissociação previamente determinada em nosso grupo com nanodisc ou peptidisc usando ITC ou MST, respectivamente (Figura 4E)16. Essa consistência fornece confiança sobre a reconstituição peptidisc e a análise do BLI como um meio de determinar a cinética de interação. Importante, deve-se notar que as proteínas são geralmente imobilizadas em biossensores de streptavidin, seja através de biotina química cross-linking ou adição site-specific usando o E. coli biotin ligase BirA21. Evidentemente, biotinylating o andaime peptidisc, em vez da proteína da membrana alvo, tem muitas vantagens. A biotinylação do andaime poupa tempo e minimiza o potencial de interromper importantes sítios de ligação proteica. Também descobrimos que o PeptiQuick é aplicável a uma ampla gama de classes-alvo de proteínas, incluindo receptores acoplados à proteína G (GPCRs), canais de íons e proteínas de membrana β-barris. Em geral, deve-se notar que o extrato de detergente inicial de proteínas de membrana em um estado livre de agregados é fundamental, e que a reconstituição imediata em peptidisc diminui os problemas de agregação a jusante. Dada a simplicidade, prevê-se que PeptiQuick será estendido a outros ensaios de ligação baseados em streptavidin, como ressonância plasmon superfície (SPR), ensaios ELISA, e afinidade pull-downs usando contas streptavidin.
Divulgações
FD é fundador da Peptidisc Biotech para distribuir peptidisc peptips para a comunidade acadêmica. A Peptidisc Biotech também está fazendo parcerias com empresas industriais de biotecnologia e farmacêuticas para implementar o peptidisc em seus fluxos de trabalho de descoberta. A publicação de Acesso Aberto deste artigo foi patrocinada pela FortéBio.
Agradecimentos
Agradecemos ao Conselho de Pesquisa em Ciências Naturais e Engenharia do Canadá. JS possui uma bolsa CGS-M CIHR. A LT foi apoiada pela Parceria de Formação de Doutorado em Biociências do Sudoeste financiada pelo Conselho de Biociências de Biotecnologia e Ciências Biológicas [referência de concessão de treinamento BB/M009122/1]. FD é uma cadeira de pesquisa de Nível II do Canadá.
Materiais
| Name | Company | Catalog Number | Comments |
| 30 kDa cut-off centrifugal concentrator | Millipore Sigma | C7715 | - |
| Ampicillin | BioShop | 69-52-3 | Sodium Salt |
| Bio-Peptidisc Peptide | Peptidisc Biotech | https://peptidisc.com | - |
| Bovine Serum Albumin (BSA) | Sigma | 9048-46-8 | Lyophilized powder |
| CaCl2 | Fisher Chemical | 10035-04-8 | Certified ACS |
| EDTA | BioShop | 6381-92-6 | Biotechnology Grade |
| Ettan LC (AKTA) | Amersham Pharmacia Biotech | 18-1145-58 | - |
| Glucose | BioShop | 50-99-7 | Anhydrous, Reagent Grade |
| Glycerol | Fisher Chemical | 56-81-5 | Certified ACS |
| Imidazole | BioShop | 288-32-4 | Biotechnology Grade |
| Lauryldimethylamine oxide (LDAO) | Sigma | 101822204 | ~30% in H2O |
| MgSO4 | Fisher Chemical | 10034-99-8 | Certified ACS |
| Microfluidizer | Microfluidics | M-110L | Fit with F20Y 75µ diruption chamber |
| Ni-NTA Resin | Gold Biotechnology | H-320-50 | - |
| Non-binding 96well BLI Plate | Greiner Bio-one | 655076 | Microplate, PS, 96 well, F-Bottom (chimney well), Black, Fluotrac, Med. Binding |
| Octet Red96 | FortéBio | OCTET RED96E-GxP | Octet RED96e instrument, Octet CFR software, desktop computer, LC monitor, accessory kit, IQ/OQ kit, PQ Kits and one-year warranty |
| Phenylmethanesulfonylfluoride (PMSF) | Sigma | P-7626 | - |
| Protein Assay Dye - Reagent Concentrate | Bio-Rad | 5000006 | Used in the bradford assay to determine protein concentration - 5x concentration |
| Sodium Chloride (NaCl) | BioShop | 7647-14-5 | Biotechnology Grade |
| Streptavidin (SA) Biosensors | ForteBio | 18-5019 | One tray of 96 biosensors coated with streptavidin for quantitation, screening, or kinetic applications. |
| Superdex S200 (300/10) | Amersham Pharmacia Biotech | 175175-01 | - |
| Thiamine | Merck | 69271 | - |
| Tris-HCl | BioShop | 77-86-1 | Reagent Grade |
| Triton X-100 | BioShop | 900998-1 | Biotechnology Grade |
| Tween-20 | BioShop | 56-40-6 | Reagent Grade |
| Ultra centrifuge | Beckman Coulter | 365668 | - |
Referências
- Rawlings, A. E. Membrane proteins: always an insoluble problem. Biochemical Society Transactions. 44 (3), 790-795 (2016).
- Popot, J. L. Amphipols, Nanodiscs, and Fluorinated Surfactants: Three Non Conventional Approaches to Studying Membrane Proteins in Aqueous Solutions. Annual Review of Biochemistry. 79, 737-775 (2010).
- Lee, S. C., et al. A Method for Detergent-Free Isolation of Membrane Proteins in their Local Lipid Environment. Nature Protocols. 11, 1149-1162 (2016).
- Frauenfeld, J., et al. A Saposin-Lipoprotein Nanoparticle System for Membrane Proteins. Nature Methods. 13, 345-351 (2016).
- Bayburt, T. H., Grinkova, Y. V., Sligar, S. G. Assembly of Single Bacteriorhodopsin Trimers in Bilayer Nanodiscs. Archives of Biochemistry and Biophysics. 450, 215-222 (2006).
- Denisov, I. G., Grinkova, Y. V., Lazarides, A. A., Sligar, S. G. Directed Self-Assembly of Monodisperse Phospholipid Bilayer Nanodiscs with Controlled Size. Journal of the American Chemical Society. 126, 3477-3487 (2004).
- Denisov, I. G., Sligar, S. G. Nanodiscs for Structural and Functional Studies of Membrane Proteins. Nature Structural, Molecular Biology. 23, 481-486 (2016).
- Hagn, F., Etzkorn, M., Raschle, T., Wagner, G. Optimized Phospholipid Bilayer Nanodiscs Facilitate High-Resolution Structure Determination of Membrane Proteins. Journal of the American Chemical Society. 135, 1919-1925 (2013).
- Carlson, M. L., et al. The Peptidisc, A Simple Method for Stabilizing Membrane Proteins in Detergent-Free Solution. eLife. 7, 34085 (2018).
- Carlson, M. L., et al. Profiling the E. coli Membrane Interactome Captured in Peptidisc Libraries. eLife. 8, 46615 (2019).
- Serdakowski, A., et al. A novel method to determine residual detergent in biological samples post endotoxin reduction treatment and evaluation of strategies for subsequent detergent removal. International Immunopharmacology. 37, 16-22 (2016).
- Smith, S. M. Strategies for the Purification of Membrane Proteins. Protein Chromatography. 681, 485-496 (2011).
- Lin, S. H., Guidotti, G. Purification of Membrane Proteins. Methods in Enzymology. 463, 619-629 (2009).
- Wolfe, A. J., Gugel, J. F., Chen, M., Movileanu, L. Kinetics of Membrane Protein-Detergent Interactions Depend on Protein Electrostatics. Journal of Physical Chemistry B. 122 (41), 9471-9481 (2018).
- Montigny, C., et al. Slow Phospholipid Exchange between a Detergent-Solubilized Membrane Protein and Lipid-Detergent Mixed Micelles: Brominated Phospholipids as Tools to Follow Its Kinetics. PLoS ONE. 12 (1), 0170481 (2017).
- Mills, A. T., Le H, T., Coulton, J. W., Duong, F. FhuA Interactions in a Detergent-Free Nanodisc Environment. Biochimica et Biophysica Acta - Biomembranes. 1838 (1), 364-371 (2014).
- Hong, P., Koza, S., Bouvier, E. S. P. Size-Exclusion Chromatography for the Analysis of Protein Biotherapeutics and their Aggregates. Journal of Liquid Chromatography Related Technologies. 35 (20), 2923-2950 (2012).
- Mahler, H. C., Friess, W., Grauschopf, U., Kiese, S. Protein aggregation: Pathways, induction factors and analysis. Journal of Pharmaceutical Sciences. 98 (9), 2909-2934 (2009).
- Trinkle-Mulcahy, L. Recent advances in proximity-based labeling methods for interactome mapping. F1000Research. 8, 135 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados