Method Article
Fixation et isolement rapides in vivo de complexes translationnels à partir de cellules eucaryotes
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous présentons une technique pour stabiliser rapidement les complexes translationnels (biosynthèse des protéines) avec réticulation du formaldéhyde dans les cellules de levures et de mammifères vivantes. L’approche permet de disséquer les intermédiaires transitoires et les interactions dynamiques ARN:protéine. Les complexes réticulés peuvent être utilisés dans de multiples applications en aval telles que les méthodes de profilage basées sur le séquençage en profondeur, la microscopie et la spectrométrie de masse.
Résumé
Les réponses rapides impliquant une redistribution rapide de l’ARN messager (m) et des altérations de la traduction de l’ARNm sont pertinentes pour les ajustements homéostatiques en cours des cellules. Ces ajustements sont essentiels à la survie des cellules eucaryotes et au « contrôle des dommages » lors de la fluctuation des niveaux de nutriments et de salinité, de la température et de divers stress chimiques et radiologiques. En raison de la nature très dynamique des réponses au niveau de l’ARN et de l’instabilité de nombreux intermédiaires ARN:ARN et ARN:protéine, l’obtention d’un instantané significatif de l’état de l’ARN cytoplasmique n’est possible qu’avec un nombre limité de méthodes. Les expériences de profilage de ribosomes à l’échelle du transcriptome et basées sur l’ARN sont parmi les sources de données les plus informatives pour le contrôle de la traduction. Cependant, l’absence d’une stabilisation intermédiaire uniforme de l’ARN et de l’ARN:protéine peut entraîner différents biais, en particulier dans les voies de réponse cellulaire au rythme rapide. Dans cet article, nous fournissons un protocole détaillé de fixation rapide applicable aux cellules eucaryotes de perméabilité différente, pour aider à la stabilisation intermédiaire de l’ARN et de l’ARN: protéine. Nous fournissons également des exemples d’isolement des complexes ARN:protéine stabilisés en fonction de leur co-sédimentation avec des fractions ribosomiques et poly(ribo)somales. Le matériau stabilisé séparé peut ensuite être utilisé dans le cadre d’expériences de type profilage de ribosomes, telles que l’approche de séquençage de profils complexes de traduction (TCP-seq) et ses dérivés. La polyvalence des méthodes de style TCP-seq a maintenant été démontrée par les applications dans une variété d’organismes et de types de cellules. Les complexes stabilisés peuvent également être purifiés par affinité et imagés à l’aide de la microscopie électronique, séparés en différentes fractions poly(ribo)somales et soumis au séquençage de l’ARN, en raison de la facilité de l’inversion de la réticulation. Par conséquent, les méthodes basées sur le refroidissement instantané et la fixation du formaldéhyde, suivies de l’enrichissement à base de sédimentation ou d’un autre type d’enrichissement du complexe ARN:protéine, peuvent être particulièrement intéressantes pour étudier les détails plus fins de la dynamique rapide du complexe ARN:protéine dans les cellules vivantes.
Introduction
Les organismes vivants sont soumis à des changements intra- et extracellulaires dynamiques tout au long de leur vie, qui nécessitent des réponses rapides pour maintenir l’homéostasie et assurer leur survie. Pour permettre l’adaptation environnementale, les cellules eucaryotes ajustent leur métabolisme via le contrôle de l’expression des gènes. Le contrôle de l’expression génique peut être exercé pendant la transcription et/ou la traduction; avec des réponses translationnelles se produisant généralement plus rapidement1,2,3,4. Par exemple, les changements translationnels surviennent généralement dans les 1 à 30 minutes suivant l’apparition du stress, tandis que les altérations au niveau de la transcription suivent des heures aprèsl’expositionau stress3,4,5. Les altérations de la production de traduction sont obtenues plus rapidement en raison de la disponibilité persistante de molécules d’ARN messager (m) dans le cytoplasme. Inversement, au niveau de la transcription, de nouvelles molécules d’ARNm doivent être synthétisées, et chez les eucaryotes, traitées et exportées à partir du noyau, produisant des retards importants dans le temps de réponse2,4,6,7,8.
La réponse translationnelle aiguë au stress est généralement caractérisée par une diminution globale de la production de traduction, avec la régulation sélective à la hausse des protéines nécessaires à la survie cellulaire1,3,4,9. La diminution de la production de protéines est considérée comme cruciale en raison de la dépense énergétique élevée du processus3,7. Pour faciliter l’inhibition sélective et la régulation à la hausse, les réponses translationnelles sont servies par une gamme de mécanismes de régulation complexes. La régulation peut être exercée à travers toutes les phases de la traduction : initiation, allongement, fin de la biosynthèse polypeptidique et recyclage ribosomique10,11,12,13, mais se manifeste le plus fortement à la phase d’initiation5,7,9,10,13. Au cours de l’initiation, la petite sous-unité ribosomique (SSU), assistée par des facteurs d’initiation eucaryotes (eIF), se lie et scanne la région non traduite (UTR) de 5' de l’ARNm jusqu’à ce qu’un codon de départ soit reconnu2,5,6,8,11,12,13. Les mécanismes de réglementation ciblent souvent les eIF affectant la reconnaissance des joints, la numérisation et le démarrage. Par exemple, le facteur d’initiation eIF2, un facteur de traduction essentiel qui aide au recrutement d’un initiateur Met-ARNtiMet à la SSU, est souvent ciblé chez les eucaryotes dans des conditions de stress4,6,11. Chez la levure, la phosphorylation de ce facteur peut être induite sous privation de nutriments et stress osmotique1,4,11,14,15, et dans les cellules de mammifères, la famine en acides aminés, le stress du réticulum endoplasmique (RE), le stress UV, l’infection virale et les niveaux d’oxygène altérés peuvent déclencher cette réponse8,9,11. La régulation rapide de la traduction spécifique de l’ARNm est évidente dans la réponse des cellules de mammifères à l’hypoxie, qui présente une inhibition globale de la traduction rapide et une régulation sélective à la hausse de la biosynthèse des facteurs inductibles par l’hypoxie (HIF). Les HIF sont des facteurs de transcription, qui provoquent ensuite une reprogrammation cellulaire à plus long terme au niveau de la transcription de l’ADN.8,9,16. Des réponses similaires ont été observées chez les levures soumises à un stress thermique, avec une expression translationnelle rapide des protéines de choc thermique (HSP) suivie de réponses retardées au niveau de la transcription17,18. En plus de la privation de nutriments et du choc thermique, les réponses translationnelles chez la levure ont été étudiées sous différents modes d’oxygène.8,19salinité5, phosphate, soufre20,21 et l’azote22,23 Niveaux. Cette recherche a des implications généralisées pour les utilisations industrielles de la levure, telles que la cuisson et la fermentation24,25. Les réponses translationnelles peuvent également contribuer à mieux comprendre des maladies telles que les troubles neurodégénératifs et les maladies cardiaques, qui se caractérisent par des stress intracellulaires tels que le stress oxydatif. Dans l’ensemble, les réponses translationnelles font partie intégrante du contrôle de l’expression génique et facilitent l’adaptation rapide à un large éventail de conditions de stress chez les organismes eucaryotes.
Afin d’étudier les réponses translationnelles, il est nécessaire de disposer de méthodes qui fournissent des instantanés du paysage de la traduction les moins déformés. Le profilage des polysomes est une approche classique utilisée dans l’étude de la traduction à travers l’ARNm, impliquant la séparation des fractions poly(ribo)somales de l’ARNm par ultracentrifugation par gradients de saccharose26,27. L’approche peut être utilisée pour explorer les niveaux de traduction pour les ARNm individuels (avec les méthodes de détection telles que la transcription inverse et la réaction en chaîne par polymérase, RT-PCR26),ou globalement en conjonction avec des techniques à haut débit (microréseau ou ARN-seq28,29). Une approche plus évoluée est le profilage des ribosomes, qui permet d’étudier les positions des ribosomes allongés le long d’une molécule d’ARNm à l’échelle du génome, ainsi que l’inférence de l’efficacité de la traduction à travers le transcriptome et l’utilisation des sites de départ principaux et alternatifs30,31. Le profilage des ribosomes implique l’isolement et le séquençage de fragments d’ARNm protégés par la présence ribosomique sur eux. Le profilage des ribosomes a fourni des informations considérables sur la dynamique de la traduction dans un certain nombre de conditions, y compris le stress hypoxique, le choc thermique et le stress oxydatif31,32. La technique a été adaptée à plusieurs types de matériaux sources, y compris les cellules de levure et de mammifères.
Alors que le profilage des polysomes et des ribosomes a été fondamental dans l’extension des capacités de la recherche en traduction, le processus de traduction comprend divers intermédiaires et complexes translationnels qui sont difficiles à capturer avec ces méthodes11,13. Une limitation supplémentaire provient du manque de capacité à étudier les types de réponse rapide, car les complexes translationnels sont soit stabilisés in vivo par l’ajout d’inhibiteurs de traduction spécifiques (antibiotiques), conduisant à certains artefacts de distribution des ribosomes, soit ex vivo lors de la lyse cellulaire spécifiquement (antibiotiques) ou de manière non spécifique (ions sel ou magnésium élevés), conduisant à la privation des intermédiaires à vie plus courte ou moins stables33, 34,35.
Le formaldéhyde est largement utilisé pour réticuler les acides nucléiques et les protéines, comme dans les études d’immunoprécipitation de la chromatine (ChIP) et d’immunoprécipitation réticulative (CLIP). Sa petite taille et son excellente perméabilité cellulaire permettent une action in vivo rapide36. Sur la base de la réticulation rapide du formaldéhyde, l’approche de profilage des ribosomes a été étendue avec le séquençage de profil complexe de traduction (TCP-seq)10,36,37,38,39,40. TCP-seq, d’abord développé en levure, permet la capture de tous les intermédiaires de traduction, y compris les complexes SSU à balayage ou post-terminaison et les configurations ribosomiquesmultiples37,38,41,42. La méthode a été utilisée dans plusieurs études10,38,39,41,42, dont certaines utilisent une approche combinatoire des inhibiteurs de traduction et de la réticulation du formaldéhyde pour faciliter l’arrêt de la traduction. Une autre version modifiée de la technique, tcp-seq39sélectif, a récemment été utilisée pour inclure l’immunopurification des complexes réticulés, élargissant ainsi la portée des applications TCP-seq. La nature rapide, efficace et réversible de la réticulation du formaldéhyde rend ces approches adaptées à l’étude des interactions complexes transitoires ARNm:traduction, en particulier dans le contexte de voies de réponse très dynamiques au niveau de la traduction.
Nous détaillons ici les processus de réticulation in vivo du formaldéhyde dans le but d’une stabilisation et d’un isolement complexes de traduction complète. Nous fournissons des protocoles distincts nuancés pour les cellules de levure et de mammifère(Figure 1). Nous décrivons également des exemples d’utilisation ultérieure du matériau stabilisé par réticulation(figure 1),par exemple pour la détection du facteur protéique co-purifié à l’aide de l’immunobuvardage (Western-blotting), de la purification immuno-assistée (ou « immunoprécipitation »; IP) et l’enrichissement de complexes translationnels contenant des facteurs d’intérêt spécifiques, la microscopie électronique et le séquençage de l’ARN.
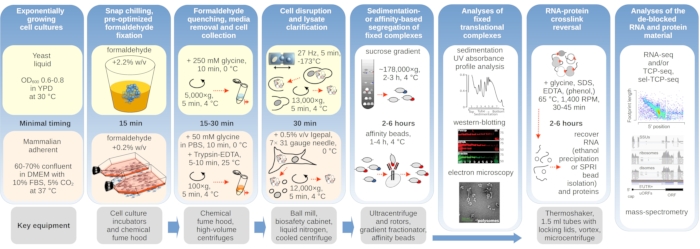
Figure 1: Schéma illustrant une vue d’ensemble de la configuration expérimentale typique. Les principales étapes de la stabilisation in vivo du formaldéhyde des complexes translationnels sont représentées sous la forme d’un organigramme, complété par des informations sur les principaux instruments nécessaires. Les applications potentielles en aval du matériau réticulé sont décrites, y compris des exemples qui ont été utilisés avec succès mais qui ne sont pas directement couverts par ce protocole, tels que la purification de l’ARN par billes SPRI, le séquençage de l’ARN et la spectrométrie de masse. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Protocole de cellule de levure
- Culture et fixation de cellules de levure
REMARQUE: La fixation et la récolte des cellules sont adaptées de10,38avec des modifications.- Mettre en place une culture cellulaire de levure de 1 L (type sauvage (WT) BY4741 sont donnés à titre d’exemple) dans un agitateur orbital avec une densité optique de départ ne dépassant pas 0,05 UA à 600 nm (OD600) dans des milieux appropriés (1 % p/v d’extrait de levure, 2 % p/v de peptone, 2 % p/v de dextrose (glucose), 40 mg/L de sulfate d’adénine (YPD) utilisé à titre d’exemple) dans les conditions souhaitées (30 °C utilisé dans ce expérience).
- Mettre en place une centrifugeuse préparative avec des bouteilles de rotor et de centrifugeuse compatibles pour granuler la culture en suspension liquide des cellules de levure. Pour les expériences de privation de glucose, pastillez les cellules une fois que la densité optique de 0,6-0,8 UA à 600 nm (OD600)est atteinte, en utilisant une brève centrifugation à 30 °C, 5 000 x g pendant 1 min.
REMARQUE: Gardez une trace de la DO des cellules en croissance et laissez les cellules croître jusqu’à ce que l’OD600 atteigne 0,6-0,8 UA, si la phase de croissance exponentielle est d’un intérêt. - Remettre immédiatement la pastille dans un milieu YP chaud (30 °C) ne contenant pas ou peu (0,25 % p/v) de glucose ajouté et incuber la culture pendant 10 min supplémentaires à 30 °C dans un agitateur-incubateur orbital.
REMARQUE : La composition du média peut affecter l’efficacité de la réticulation ultérieure. Ce protocole a été testé en utilisant YPD uniquement. Lors de la réalisation d’expériences de famine, il est essentiel de respecter le calendrier et de minimiser les délais entre les procédures. - Une fois les cellules prêtes, installez une glacière à l’intérieur de la hotte avec un bécher contenant 250 g de glace d’eau pilée propre. S’assurer que les bandelettes de 25 mL et la solution de formaldéhyde stabilisée au méthanol fraîchement achetée sont accessibles à l’intérieur de la hotte. Verser la culture de 1 L dans le bécher contenant 25 % p/v de glace d’eau pilée.
REMARQUE: Gardez les cellules sur la glace tout au long de toutes les opérations ultérieures jusqu’à ce que les cellules soient gelées, sauf indication contraire. - Ajouter 75 mL de solution de formaldéhyde à 37 % p/v à une concentration finale de 2,2 % p/v et remuer intensément le mélange jusqu’à ce que la glace fonde.
- Une fois la glace fondue, installez une minuterie pendant 10 min.
REMARQUE: Respectez les délais et le régime de température recommandés pour obtenir des résultats de fixation reproductibles. - Après incubation pendant 10 min, transférer la culture dans les bouteilles de centrifugeuse prérefroidies et granuler les cellules par centrifugation à 4 °C, 5 000 x g pendant 5 min. Pendant que ce spin est activé, prérefroidissez un tube de 50 mL et gardez le tampon A fraîchement préparé (contenant de la glycine pour neutraliser tout formaldéhyde restant) sur la glace.
REMARQUE : Reportez-vous au tableau fourni pour connaître les compositions exactes de la mémoire tampon. - Après la centrifugation, placez les tubes de centrifugeuse sur de la glace avec le côté granulé en contact avec la glace. Apportez les tubes dans la hotte et jetez le surnageant dans un conteneur à déchets de formaldéhyde.
- Remettre en suspension la pastille de la cellule de tous les tubes dans 20 mL de tampon A à l’aide d’une bandelette de 25 mL et transférer dans un tube de 50 mL.
REMARQUE: Ce lavage est essentiel pour éviter la réticulation irreproductible et l’ajout de tampon ne doit pas dépasser 20 minutes de temps à partir de la récolte des cellules. - Porter le volume à 40 mL avec le tampon A et prélever les cellules lavées par centrifugation à 4 °C, 5 000 x g pendant 5 min.
- Jeter le surnageant et remettre en suspension la pastille cellulaire dans 40 mL de tampon A1, qui est le tampon A ne contenant pas de glycine, pour éliminer toute contamination par la glycine.
- Cellules à granulés à nouveau par centrifugation à 4 °C, 5 000 x g pendant 5 min.
- Répétez les lavages avec le tampon A1 une fois de plus. Jetez le surnageant et placez la pastille de cellule sur de la glace. Peser le tube avec la pastille (la masse cellulaire humide doit être d’environ 1 g pour 1 L de culture cellulaire).
- Perturbation des cellules de levure et collecte de cytosols
- Remplissez une boîte en mousse de polystyrène recouverte de papier d’aluminium avec de l’azote liquide jusqu’à une profondeur d’environ 3 cm. Placez un tube de 50 mL à la verticale dans la boîte.
- Remettre en suspension la pastille (~1 g de masse cellulaire humide) dans 550 μL de tampon A2 par pipetage et vortex pendant 10 s. Ajouter 10 μL de 40 U/μL d’inhibiteur de la RNase et vortex à nouveau pendant 10 s.
ATTENTION : Portez un équipement de protection approprié, comme des gants isolés thermiquement, lorsque vous manipulez de l’azote liquide. Assurez-vous que tout récipient utilisé pour contenir de l’azote liquide ne fuit pas et que le porte-tubes à l’intérieur ne flottera pas ou ne tombera pas sur le côté. Travaillez dans un endroit bien ventilé pour éviter l’épuisement de l’oxygène. - À l’aide d’une pipette de 1 mL, égoutter la suspension cellulaire dans le tube de 50 mL contenant l’azote liquide.
REMARQUE: Le goutte-à-goutte doit être effectué lentement et soigneusement pour éviter l’agrégation des gouttelettes. Assurez-vous que les gouttelettes gèlent avant d’introduire de nouvelles gouttelettes. - Transférer le tube de 50 mL avec les gouttelettes de suspension cellulaire congelées à température ambiante et attendre que l’azote liquide s’évapore complètement. Scellez le tube avec son bouchon et stockez les pastilles de cellule à -80 °C ou continuez immédiatement.
ATTENTION : Assurez-vous que l’azote liquide est complètement évaporé avant de sceller le tube. Les restes d’azote liquide dans un tube scellé peuvent provoquer une accumulation de pression dangereuse. - Pour vous préparer à l’étape suivante, prérefroidissez des tubes sans nucléase de 1,5 mL et des pots de broyage en acier inoxydable de 10 mL sur de la glace carbonique.
- Transférez les gouttelettes de suspension cellulaire congelées dans les bocaux à l’aide d’une spatule propre et stérile.
ATTENTION : Assurez-vous que les bocaux de broyage sont hermétiquement fermés. - Immergez les pots de broyage dans l’azote liquide pendant 1 min en vous assurant que la phase liquide reste sous la jonction. Installez un broyeur cryo mélangeur à 27 Hz pour l’agitation pendant 1 min.
REMARQUE: Équilibrez toujours la cartouche de broyage avec une autre du même modèle, même si l’échantillon ne nécessite qu’une seule cartouche pour le traitement. - Agiter les pots de broyage scellés à 27 Hz pendant 1 min dans le broyeur mélangeur.
- Refroidir à nouveau les pots de broyage dans de l’azote liquide comme précédemment et agiter à 27 Hz pendant 1 min plus loin dans le broyeur mélangeur.
- Transférez les pots dans la glacière contenant de la glace carbonique avec les tubes sans nucléase de 1,5 mL. À l’aide d’une petite spatule en acier, transférer l’échantillon en poudre résultant dans les tubes dans des aliquotes d’environ 100 mg et stocker les tubes à -80 °C.
REMARQUE: Il est recommandé d’utiliser environ 600 mg de l’échantillon par expérience comprenant l’analyse du profil de sédimentation des polysomes, la séparation du cytosol en fractions traduites et non traduites, et la séparation supplémentaire de la fraction traduite en fractions SSU, ribosome et disome lors de la digestion de la RNase.
- Séparation des complexes (poly)ribosomiques fixes des fractions non traduites du cytosol
REMARQUE: La procédure établie précédemment10,38est généralement suivi pour enrichir l’ARN traduit en fonction de sa co-sédimentation avec des (poly)ribosomes. Une approche plus raffinée pour séparer les fractions de cytosol traduites et non traduites est introduite ici, éliminant le besoin de précipiter et de re-solubiliser ensuite le matériau.- Préparer des gradients de saccharose linéaires de 2,5 mL à 10 % à 20 % p/v avec le tampon B à l’aide de la méthode de congélation-décongélation43 dans des tubes ultracentrifuges à paroi mince (5 mL, 13 x 51 mm).
REMARQUE: La méthode de congélation-décongélation est réalisée par l’addition séquentielle et la congélation de couches de saccharose tamponnées avec des concentrations régressives linéaires les unes sur les autres. Voir le tableau supplémentaire 1 pour plus de détails. - Pour créer un coussin de saccharose discontinu de 50 % p/v, lors de la décongélation et de la stabilisation des gradients linéaires, distribuer lentement 0,5 mL de saccharose à 50 % dans le tampon B directement sur le fond des tubes à l’aide d’une seringue de 1 mL attachée à une aiguille de 19 G x 1,5 po ou d’un capillaire en verre de dimensions similaires/appropriées. Avant de distribuer, enfoncez soigneusement et lentement la pointe de l’aiguille ou du capillaire de haut en bas des gradients de saccharose préformés, en évitant toute perturbation, jusqu’à ce qu’elle atteigne le fond du tube.
NOTE: Voir le tableau supplémentaire 1 pour les instructions sur la préparation du tampon B. - Équilibrez soigneusement les gradients en retirant les parties supérieures ou en superposant plus de 10 w/v de saccharose dans le tampon B et en les maintenant au froid ou à 4 °C.
REMARQUE: Le gradient discontinu avec la couche inférieure de saccharose à 50% est nécessaire pour recueillir des matériaux avec une vitesse de sédimentation plus élevée sans les précipiter sur la paroi du tube. - Décongeler environ 100 mg de l’échantillon congelé en poudre de cellules à température ambiante et les placer immédiatement sur de la glace. Mélanger 150 μL de tampon A2 par pipetage, ajouter l’inhibiteur de RNase à 1 U/μL et mélanger par vortex (éviter la mousse excessive et le mélange avec la phase gazeuse) pendant 10 s.
REMARQUE : Poursuivre toutes les opérations tout en gardant le matériau sur la glace, sauf indication contraire. - Abattez les débris cellulaires en centrifugant les tubes à 4 °C, 13 000 x g pendant 5 min et récupérez le surnageant clarifié (~150 μL) dans un nouveau tube de liaison à faible teneur en protéines de 1,5 mL.
- Charger le mélange clarifié résultant sur les tubes à gradient de saccharose discontinus de l’étape 1.3.3 et les équilibrer soigneusement.
- Ultracentrifugez les tubes dans un rotor à godet oscillant de volume moyen à 4 °C, avec une force g moyenne de 287 980 x g (facteur k 49) pendant 1 h 30 min.
REMARQUE: Ces conditions ont été pré-optimisées (à l’aide de l’analyse des traces d’absorbance UV post-gradient d’ultracentrifugation) pour conserver les SSU libres (non (poly)ribosomiques) et les LSU (grande sous-unité ribosomique) dans la partie supérieure (10%-20% saccharose) du gradient tout en concentrant la fraction (poly)ribosomique dans le coussin de saccharose inférieur (50%) sans granuler le matériau. - Utilisez une nouvelle seringue stérile de 1 mL équipée d’une aiguille de 19 G x 1,5 po pour recueillir la fraction de cytosol traduite. Placez le gradient de 5 mL sur un rack stable en veillant à ce que le fond du tube soit visible.
- Du haut du tube, collez l’aiguille directement dans le bas du gradient (sans perforer le tube) et doucement, sans créer de bulles, dessinez exactement 0,5 mL de la solution inférieure contenant le pool d’ARN traduit.
REMARQUE: Assurez-vous que cette étape est effectuée dans une chambre froide et que le tube est maintenu fermement. Il est recommandé de dessiner la totalité des 0,5 mL en un seul mouvement ascendant pour éviter toute perturbation du gradient. - Confirmer la présence (poly)ribosomique et l’épuisement de l’USU, de l’UGB et des fractions plus légères dans le mélange résultant par lecture d’absorbance du gradient de saccharose lors de l’ultracentrifugation.
- Concentrer le pool d’ARN traduit collecté de l’étape précédente à 100 μL en utilisant l’ultrafiltration dans un dispositif de micro-concentration avec une membrane de cellulose régénérée coupée de 10 kDa.
REMARQUE : Prélavez la membrane du dispositif de microconcentration avec 0,5 mL de tampon 1 (voir la figure 2a) et utilisez les conditions d’essorage(g)recommandées par le fabricant. - Diluer davantage le matériau de l’étape précédente cinq fois (ajouter 400 μL) avec le tampon 1 et concentrer à nouveau à 200 μL, pour permettre un volume plus petit ainsi que l’élimination partielle du saccharose.
REMARQUE: Il est recommandé de stocker les mélanges résultants à -80 ° C jusqu’à 6 mois et de les utiliser comme matériau d’entrée pour la construction de la bibliothèque d’ARN-seq « ARN traduit total » ou l’étape de digestion de la RNase de la construction de la bibliothèque TCP-seq. La fraction de cytosol « non traduite » peut être récupérée à partir du haut du gradient en utilisant une procédure similaire et stockée à -80 ° C.
- Préparer des gradients de saccharose linéaires de 2,5 mL à 10 % à 20 % p/v avec le tampon B à l’aide de la méthode de congélation-décongélation43 dans des tubes ultracentrifuges à paroi mince (5 mL, 13 x 51 mm).
- Digestion par RNase des complexes (poly)ribosomiques fixes et séparation du matériel digéré en petites sous-unités ribosomiques (SSU), monoribosomales (ribosomes, RS) et diribonomiques (disomes, DS) fractions
REMARQUE : La procédure suit généralement une approche décrite précédemment10,38mais un type de gradient modifié, le temps de séparation, l’accélération et les conditions de digestion de la RNase sont utilisés, pour obtenir la meilleure résolution sur les trois fractions isolées.- Préparer des gradients de saccharose linéaires soigneusement équilibrés de 12,5 mL à 10 % à 40 % p/v fabriqués avec des tubes en polypropylène tampon de 1 dans 13 mL à paroi mince, 14 x 89 mm, en utilisant la méthode de congélation-décongélation43 décrite à l’étape 1.3.1 et notez-y.
- Décongeler à température ambiante et transférer immédiatement les échantillons sur de la glace ou prélever la fraction de cytosol traduite concentrée et appauvrie en saccharose à partir de l’étape 1.3.12.
REMARQUE : Poursuivre toutes les procédures sur la glace, sauf indication contraire. - Digérer la fraction de cytosol traduite en mélangeant 4,5 U d’E. coli RNase I pour 1 od260 unité de la fraction pendant 30 min à 23 °C. Ajouter et mélanger immédiatement en pipetant l’inhibiteur de la RNase capable d’inactiver la RNase I à 0,25 U/μL au mélange, pour inactiver la RNase I.
REMARQUE: Utilisez un inhibiteur de la RNase capable d’inhiber la RNase I. Dérivez AU260 en utilisant AU260 = (Absorbance à 260 nm normalisée en unités de densité optique équivalentes à 1 cm de chemin optique x volume du lysat en μL) / 1 000. - Transférer immédiatement les échantillons sur la glace.
ATTENTION: Il est essentiel de respecter les conditions recommandées de digestion et de mesurer soigneusement la quantité de RNase I ajoutée. L’unité RNase I mentionnée ici est définie comme la quantité de l’enzyme nécessaire pour produire 1 μg de matière soluble dans l’acide à partir de l’ARN du foie de souris en 30 min à 37 °C. Les lots de RNase I peuvent présenter des variations d’activité non documentées et nécessiter des expérimentations pour obtenir des conditions de digestion optimales. Si le stock enzymatique est trop concentré, il est recommandé de le diluer avec le tampon 1 pour éviter de pipeter de très petits volumes de la solution. - Charger les mélanges réactionnels sur les gradients de saccharose de 10 % à 40 % p/v de l’étape 1.4.1.
REMARQUE: Utilisez des volumes finaux compris entre 150 et 300 μL par gradient. Chaque purification nécessite au minimum deux gradients. Utilisez différents volumes d’entrée du matériau (AU260inférieur, 10-11 UA260, pour DS et comparativement plus élevé AU260, 13-14 AU260, pour SSU ou RS) pour obtenir une séparation optimale. - Ultracentrifugez les tubes dans un rotor à godet oscillant de volume moyen à 4 °C avec une force g moyenne de 178 305 x g (facteur k 143,9) pendant 3 h 30 min.
ATTENTION : Si des tubes d’équilibrage de rechange sont nécessaires, égaliser leur masse et leur répartition massique avec les tubes contenant de l’échantillon. Utilisez des gradients de saccharose de rechange recouverts d’une quantité de tampon équivalente à celle de la superposition de l’échantillon et non des tubes à concentration uniforme de saccharose. - Installez un dispositif de fractionnement de gradient au moins 30 minutes avant la fin du spin d’ultracentrifugation, y compris le remplissage de la solution de poursuite lourde filtrée de 0,2 μm (par exemple, 60% de saccharose dans de l’eau désionisée telle qu’utilisée ici) dans la pompe de déplacement.
REMARQUE: Il est recommandé de décontaminer les conduites et les tubes du fractionneur à l’aide d’eau désionisée, suivie d’une solution de FDS à 1% à 2% dans de l’eau désionisée, de l’eau désionisée et enfin de 80% d’éthanol dans une solution d’eau désionisée avant et après les cycles. - Ajustez la ligne de base de lecture de l’absorbance en remplissant d’abord le système d’eau désionisée et en mettant à zéro l’optique conformément aux recommandations du fabricant, puis en compensant le décalage de la ligne de base à l’aide d’un gradient de saccharose de rechange déchargé de 14 x 89 mm fabriqué avec un tampon identique aux tubes d’échantillonnage (par exemple, tampon 1).
REMARQUE: Utilisez la même vitesse de déplacement pour effectuer les ajustements que pour la lecture de l’échantillon, par exemple 1,5 mL / min. - Mesurez le volume mort du système de déplacement en comptant avec précision le temps entre la première entrée de la solution dans le chemin optique du détecteur et sa première apparition à la sortie du collecteur de fractions.
REMARQUE: Avec la vitesse recommandée de 1,5 mL / min, le fractionnement peut être effectué à température ambiante. Il est recommandé de transférer immédiatement les fractions collectées sur la glace. - Effectuer le fractionnement à l’aide d’une lecture d’absorbance sous tension à 254 nm, d’une vitesse de déplacement de 1,5 mL/min et d’une détection de fraction en ligne en fonction de la position de sédimentation et du profil d’absorbance attendus des échantillons. Utilisez la commutation du tube collecteur avec un délai correspondant au volume mort mesuré précédemment.
- Isoler les fractions correspondant aux positions et à la mobilité des complexes SSU, RS et DS et les collecter dans de nouveaux tubes de microcentrifugation de 1,5 mL à faible liaison aux protéines; transférer immédiatement les fractions isolées sur de la glace et les congeler si elles ne sont pas traitées immédiatement.
REMARQUE: Il est recommandé de congeler immédiatement les fractions collectées dans de la glace carbonique ou de l’azote liquide et de les conserver à -80 ° C ou moins pendant 6 mois.
- Dé-réticulation des complexes ribosomiques et isolement de l’ARN pour construire des bibliothèques d’ARN-seq
- Pour débloquer/inverser les réticulations et isoler l’ARN des protéines associées, transférer environ la moitié de l’ensemble des fractions du gradient de saccharose dans de nouveaux tubes de microcentrifuge en polypropylène de 1,5 mL à faible liaison aux acides nucléiques (350 μL par tube) avec des dispositifs de sécurité et de verrouillage des couvercles.
- Compléter les mélanges avec 40 μL de solution d’arrêt à 100 % (10 % de SDS p/v et 100 mM d’EDTA), 4 μL de 1 M de Tris-HCl pH2 à 25 °C (à 10 mM), 1,6 μL de glycine 2,5 M (à 10 mM) et de l’eau sans nucléase désionisée pour obtenir le volume final de 400 μL.
- Mélanger le contenu des tubes par pipetage et transférer les tubes à température ambiante.
- Ajouter un volume égal du mélange acide phénol:chloroforme:alcool isoamylique 125:24:1 (pH 4,0-5,0) dans chaque tube. Agiter vigoureusement les mélanges pendant 2 min à l’aide d’un mélangeur vortex réglé à la vitesse maximale.
ATTENTION : Le phénol et le chloroforme sont corrosifs et toxiques. Évitez tout contact physique avec les liquides et travaillez dans un endroit bien ventilé ou sous une hotte aspirante. Utilisez toujours des gants, une blouse de laboratoire et des lunettes de protection ou un écran facial lorsque vous travaillez avec du phénol ou du chloroforme. - Placez les tubes dans un thermochaîneur et secouez continuellement à 65 °C, 1 400 tr/min pendant 30 min.
- Faciliter l’agrégation de phase en centrifugant le mélange à 12 000 x g pendant 10 min à température ambiante.
- Recueillir les phases aqueuses supérieures et les transférer dans des tubes frais de 1,5 mL liant les acides nucléiques faibles.
REMARQUE: Pour éviter la contamination croisée, n’essayez pas de récupérer complètement les phases aqueuses. Un volume de récupération raisonnable est de 300-350 μL. - Compléter les phases aqueuses collectées avec 0,1 volume d’acétate de sodium 3 M (pH 5 à 25 °C), 20 μg de glycogène (en utilisant du stock de 5 μg/μL) et 2,5 volumes d’éthanol absolu. Mélangez soigneusement les solutions en vortexant les tubes pendant 1 min.
- Précipiter l’ARN en incubant les échantillons à -20 °C pendant au moins 2 h (recommandé pendant la nuit).
- Réchauffer les tubes à température ambiante et mélanger par vortex.
REMARQUE: Le préchauffage des tubes et la centrifugation ultérieure à température ambiante (sans refroidissement forcé) aident à réduire la coprécipitation et le report du sel et du phénol. Ces conditions ne doivent pas entraîner de perte matérielle ou d’inefficacité de la collecte d’ARN si elle est effectuée comme décrit et en utilisant de l’éthanol suffisamment pur. - Coupez l’ARN précipité en centrifugant les tubes à 12 000 x g pendant 30 min à température ambiante.
- Jeter le surnageant et laver la pastille deux fois avec de l’éthanol à 80 % v/v, en le recueillant à chaque fois par centrifugation à 12 000 x g pendant 10 min à température ambiante.
- Séchez les pastilles d’ARN en ouvrant les couvercles des tubes et en plaçant les tubes ouverts dans un réchauffeur à bloc sec réglé à 45 °C pendant 10 min. Dissoudre la pastille séchée résultante dans 20 μL de tampon 1x HE.
- Estimer la concentration d’ARN résultante à l’aide de la mesure du spectre d’absorbance UV.
REMARQUE: La longueur et la quantité totale du fragment d’ARN peuvent être évaluées plus en détail à l’aide de l’électrophorèse sur gel dénaturant, par exemple dans un appareil automatisé d’électrophorèse sur gel capillaire à fluorescence.
- Co-immunopurification sélective des SSU par les eIF balisés et analyse par transfert western de l’enrichissement sélectif des SSU
REMARQUE: Utilisez ~15 UA (260 nm) de la fraction SSU digérée et séparée par sédimentation de l’étape 1.4.11 pour effectuer la purification d’affinité à l’aide de billes d’IgG magnétiques. Économisez ~5% de la fraction SSU en tant que contrôle d’entrée (fraction d’entrée, I). eIF4A-tagged (TIF1-TAP; Tandem Affinity Purification tag) souche de levure a été utilisée qui permet également de détecter eIF4A en sondant le TAP-tag à l’aide d’anticorps anti-TAP.- Transférer 100 μL de suspension de billes d’IgG magnétiques (1 mg des billes a été utilisé pour chaque 15 UA (260 nm) du lysat ou de la fraction) dans un nouveau tube de 1,5 mL à faible liaison aux protéines; recueillir les perles à l’aide d’un rack magnétique et les aspirer.
- Lavez les billes magnétiques deux fois avec 1 mL de tampon 1 en utilisant la remise en suspension séquentielle par pipetage et collecte à l’aide du rack magnétique.
- Après le lavage, collectez et décantez les perles, tout en les gardant sur la grille magnétique.
- Ajouter la fraction SSU aux billes lavées et incuber le mélange pendant 4 h avec rotation à 4 °C dans un cyclomixeur réglé à ~20 tr/min.
- Collectez les billes à l’aide du rack magnétique à 4 °C et économisez le surnageant (fraction Flow-through, FT).
- Lavez les billes deux fois à 4 °C avec le tampon 1 complété par 4 mM de TNT, en tournant à chaque fois pendant 10 min dans le cyclomixeur et en collectant et décantant les perles sur le rack magnétique. Enregistrez les lavages (fractions W1 et W2).
- Pour une application analytique telle que le western blotting, éluez le matériau lié dans des conditions de dénaturation et de réduction en ajoutant un tampon d’échantillon d’électrophorèse sur gel de polyacrylamide (PAGE) LDS (sulfate de dodécyle de lithium) avec un pH de 8,5 à 1x et un DTT à 2 mM.
- Chauffer le mélange à 95 °C pendant 5 min dans un bloc thermique pour finaliser l’élution.
- Recueillir les billes à l’aide de la grille magnétique et récupérer l’éluat dénaturé (fraction E) dans un tube de microcentrifugation frais à faible teneur en protéines de 1,5 mL.
- Utilisez la fraction E de l’étape précédente pour exécuter immédiatement une PAGE de dodécylsulfate de sodium (FDS) dénaturante, ou stockez la fraction E à -20 °C.
REMARQUE: Pour une collection préparative des complexes translationnels enrichis en étiquettes TAP pour toute application ultérieure, utilisez une autre approche d’élution utilisant la protéase du virus de la gravure du tabac (TEV). Reportez-vous au tableau supplémentaire 1 pour plus de détails. - Pour concentrer les fractions DILUÉES FT, W1 et W2, précipitez leur matériau en ajoutant 3x volumes d’acétone glacée. Incuber le mélange échantillon-acétone à -20 °C pendant 3 h.
- Pastillez le précipité en centrifugant les tubes à 13 000 x g pendant 10 min à 4 °C.
- Jeter le surnageant et sécher à l’air libre la pastille dans les tubes ouverts à température ambiante pendant 30 min.
- Dissoudre la pastille dans 7 μL de 1x tampon de chargement LDS complété par 2 mM de TNT. Chauffer les échantillons dans un bloc thermique réglé à 95 °C pendant 5 min.
- Chargez tous les échantillons I, FT, W1, W2 et E sur un gradient d’acrylamide de 4 % à 12 % p/v, gel de dénaturation du polyacrylamide Bis-Tris. Faites fonctionner le gel à l’aide de 1x MES SDS (acide 2-[N-mopholino]éthanesulfonique, dodécylsulfate de sodium) tampon à 80 V, jusqu’à ce que le marqueur protéique (10-250 kDa) se résolve bien et que le colorant principal atteigne le fond du gel.
REMARQUE: Il est recommandé de charger des dilutions en série du WCL (lysat de cellules entières) (2-10 μg) sur le gel comme témoin. Il peut falloir plusieurs tentatives pour obtenir une charge comparable du gel à travers le matériau de la fraction. - Transférer la teneur en protéines du gel sur une membrane de difluorure de polyvinylidène (PVDF) par méthode de transfert humide à 100 V pendant 1 h dans une chambre froide comme recommandé par le fabricant d’équipement de transfert western.
- Bloquez la membrane à l’aide d’un tampon bloquant approprié (à base de solution saline tamponnée au phosphate) à température ambiante pendant 1 h sous agitation constante.
- En suivant les instructions du fabricant pour la dilution des anticorps, sondez la membrane avec un anticorps anti-TAP pour détecter la protéine eIF4A marquée, l’anticorps anti-Pab1p ou l’anticorps anti-β-actine (ou toute autre cible souhaitable) par incubation nocturne de la membrane avec un anticorps dilué pbS (dilution 1:1 000) dans un cyclomixeur dans une chambre froide.
REMARQUE: La dilution des anticorps 1:1 000 est un bon point de départ. - Lavez la membrane trois fois avec 1x solution saline tamponnée au phosphate, 0,2% v/v Tween 20 (PBST) pendant 10 min chacun.
- Sondez la membrane avec des anticorps secondaires marqués par fluorescence en suivant les instructions du fabricant en incubant dans un cyclomixeur à température ambiante pendant 1 h.
REMARQUE: La dilution des anticorps à 1:20 000 est un bon point de départ. - Lavez la membrane trois fois avec 1x PBST pendant 10 minutes chacun. Rincez brièvement la membrane à l’eau désionisée, puis au méthanol absolu. Séchez et visualisez la membrane dans un système d’imagerie fluorescente selon les instructions du fabricant.
REMARQUE: La coloration d’autres protéines peut être obtenue en utilisant des anticorps secondaires avec des colorants correspondant à différents canaux fluorescents (comme dans la paire eIF4A-TAP vs β-actine utilisée ici), en colorant ou en décapant séquentiel et en colorant la même membrane ou en coupant la membrane d’un gel chargé de fractions répétitives et en sondant séparément chaque pièce avec des anticorps respectifs (comme dans l’exemple Pab1p utilisé ici).
2. Protocole des cellules de mammifères
- Culture et fixation de cellules de mammifères
- Dans 2 flacons T-175, faire croître les cellules HEK293 jusqu’à une confluence de 60 % à 70 % dans le milieu Eagle modifié de Dulbecco et 10 % v/v de sérum fœtal bovin à 37 °C et 5 % v/v de dioxyde de carbone.
REMARQUE: Le milieu complet est fabriqué en ajoutant 55 mL de FBS commercial dans un 500 mL de DMEM acheté commercialement avec un taux élevé de glucose, contenant de la L-glutamine, du rouge de phénol et du bicarbonate de sodium, mais pas de HEPES ou de pyruvate de sodium. Le nombre de cellules par fiole T-175 à 70 % de confluence doit être compris entre 1,7 et 2,0 x 107. - Au moins 3 h avant le temps de fixation souhaité, remplacez le support des flacons T-175 par précisément 30 mL de milieu complet préchauffé et remplacez les flacons dans un incubateur cellulaire.
REMARQUE: Assurez-vous que le média frais est pipeté sur le côté opposé de la fiole à la monocouche de la cellule pour éviter le détachement de la cellule. Essayez d’effectuer l’échange de médias le plus rapidement possible, en introduisant une perturbation minimale de l’équilibre des gaz et de la température. - Une fois le milieu cellulaire remplacé, préparez les tampons et les produits chimiques nécessaires à la fixation. Préparez la solution saline tamponnée au phosphate (DPBS) de Dulbecco avec 50 mM de glycine en ajoutant 10,2 mL de glycine de 2,5 M à une bouteille de 500 mL de DPBS et en mélangeant.
- Préparer un flacon de DMEM complété par 10% de FBS comme à l’étape 2.1.1 à utiliser dans des conditions non stériles et une aliquote de 100 mL de 0,25% de trypsine-EDTA. Procurez-vous une bouteille supplémentaire de DPBS commercial pré-formulé avec du chlorure de calcium (CaCl2)et du chlorure de magnésium (MgCl2).
REMARQUE: Les solutions peuvent être conservées à 4 °C jusqu’à 2 semaines. - Préparez une glacière à ras bord avec de la glace d’eau pilée de sorte qu’une fiole T-175 puisse s’adapter uniformément sur le dessus et la conserver dans la hotte avec les tampons préparés, également sur la glace.
REMARQUE: En raison des réponses rapides de la traduction à tout changement environnemental, tous les délais entre le retrait des flacons cellulaires de l’incubateur et l’ajout de la solution de formaldéhyde doivent être minimisés. - Pour refroidir les cellules, retirez la fiole T-175 de l’incubateur et appuyez fermement contre la glace pour assurer un contact maximal avec la surface. À l’intérieur de la hotte chimique, inclinez la fiole sur le côté afin que le milieu s’accumule sur le côté opposé aux cellules. Pipette 168 μL de formaldéhyde à 37 % p/v directement dans le milieu groupé (jusqu’à une concentration finale de 0,2 % p/v). Mélangez immédiatement en berçant doucement la fiole d’avant en arrière, fermez et repositionnez la fiole sur la glace, en vous assurant qu’elle est horizontale et que les cellules sont recouvertes uniformément.
ATTENTION: Le formaldéhyde est une substance nocive avec des effets indésirables potentiels à long terme et aussi un irritant pour le système respiratoire et la peau. Il ne doit être manipulé que dans une hotte chimique appropriée. Les contenants de formaldéhyde doivent toujours être scellés à l’extérieur de la hotte.
REMARQUE: Assurez-vous que le formaldéhyde est ajouté directement dans le milieu cellulaire et non à la paroi du flacon. L’étape 2.1.6 devrait prendre moins de 1 min. - Incuber les flacons sur de la glace pendant 10 minutes supplémentaires. Versez le milieu dans un récipient à déchets approprié à travers le côté de la fiole opposé aux cellules.
- À l’aide d’une stripette, pipetter dans 30 mL de solution saline tamponnée au phosphate de Dulbecco sans ions calcium et magnésium et contenant en outre 50 mM de glycine, doucement sur le côté opposé aux cellules. Mélanger en berçant la fiole; remettre la fiole en position horizontale et incuber pendant 10 minutes de plus sur de la glace.
- Verser la solution à travers le côté de la fiole opposé aux cellules et ajouter doucement 7 mL de la solution standard de Trypsine-EDTA à 0,25 % p/v pour détacher et remettre en suspension les cellules. Incuber la fiole à température ambiante pendant 5-10 min.
REMARQUE: Assurez-vous que la solution de trypsine-EDTA couvre toutes les cellules uniformément. Utilisez une inclinaison et un balancement doux périodiques pour favoriser le détachement cellulaire. - Déplacez la fiole verticalement et, à l’aide d’une stripette, collectez les cellules détachées en lavant doucement les cellules restantes des parois de la fiole. Transférer la suspension dans un tube de 50 mL posé sur de la glace.
REMARQUE: Les cellules fixes peuvent devenir plus fragiles; ne pipettez pas intensément ou plus que ce qui est nécessaire pour détacher les cellules de la paroi du ballon. - Complétez immédiatement la suspension cellulaire collectée avec 20 mL de milieu complet (le milieu glacé non stérile avec 10 % de FBS) et mélangez en retournant doucement le tube.
REMARQUE: Le milieu de culture cellulaire complet (y compris 10% FBS) est ajouté pour neutraliser la trypsine, empêchant ainsi d’autres dommages aux membranes cellulaires et la désintégration cellulaire. - Abattez les cellules en centrifugant le tube à 100 x g pendant 5 min et 4 °C. La pastille de cellule doit être clairement visible.
- Versez le support et remettez doucement en suspension la pastille cellulaire dans 10 mL de DPBS glacé avec Du Ca2+,Mg2+et sans glycine.
- Répétez l’étape 2.1.12.
- Versez le tampon de lavage et remettez doucement la pastille de cellule dans 800 μL de DPBS glacé avec Du Ca2+,Mg2+,sans glycine, sur de la glace. Transférer les cellules remises en suspension dans un nouveau tube de microcentrifugation de 1,5 mL à faible teneur en protéines.
- Centrifuger le tube à 100 x g pendant 3 min et 4 °C. Jetez soigneusement le surnageant à l’aide d’un pipetteur de 1 mL. À ce stade, la pastille cellulaire peut être congelée à -80 °C ou passer à l’étape de lyse cellulaire.
REMARQUE: Les granulés de cellules congelés peuvent être conservés à -80 ° C jusqu’à 1 an. Nous avons constaté que la congélation des granulés cellulaires facilite la lyse ultérieure et recommandons la congélation même si un stockage à plus long terme n’est pas prévu.
- Dans 2 flacons T-175, faire croître les cellules HEK293 jusqu’à une confluence de 60 % à 70 % dans le milieu Eagle modifié de Dulbecco et 10 % v/v de sérum fœtal bovin à 37 °C et 5 % v/v de dioxyde de carbone.
- Perturbation des cellules de mammifères et collecte de cytosols
- Dans une armoire de biosécurité, ajouter 300 μL de tampon de lyse à base de détergent non ionique et non dénaturant et 7 μL d’inhibiteur de 40 U/μL de RNase. Bien mélanger en pipetant à l’aide d’une pointe de 1 mL.
- Fixez soigneusement une aiguille de 25 G à une seringue de 1 à 3 mL et pipetez vigoureusement le mélange, en utilisant au moins sept prises lentes vers le haut et des coups d’échappement rapides vers le bas.
- Jetez la seringue et l’aiguille dans un bac à objets tranchants et répétez la procédure à l’aide d’une seringue de 0,3 mL équipée d’une aiguille de 31 G.
- Jetez la seringue et l’aiguille dans un bac à objets tranchants. Centrifugez les tubes à 4 °C, 12 000 x g pendant 5 min pour granuler les débris cellulaires.
- Transférer le surnageant dans un nouveau tube de microcentrifugation de 1,5 mL à faible teneur en protéines. Stocker à la fois les débris cellulaires (à des fins de contrôle) et le lysat cellulaire clarifié qui en résulte à -80 °C.
REMARQUE: La densité optique du lysat varie entre 25-30 AU260 lorsque deux flacons T-175 sont combinés et que les volumes recommandés sont suivis. Les lysats et les débris cellulaires peuvent être stockés à -80 °C jusqu’à 1 an.
- Séparation des complexes (poly)ribosomiques fixes des fractions non traduites du cytosol
- Préparer des gradients linéaires de saccharose de 15 % à 45 % p/v dans des tubes de polypropylène à paroi mince de 13 mL, de 14 x 89 mm, en utilisant la méthode de congélation-décongélation généralement décrite à l’étape 1.3.1 du protocole de levure, mais en utilisant le tampon 2(Figure 2a).
REMARQUE: Décongeler les gradients pendant la nuit dans une chambre froide à 4 ° C la nuit précédant le fractionnement. - Chargez 150-250 (maximum 300) μL du lysat cellulaire de l’étape précédente 2.2.5 sur les gradients équilibrés. Conserver le lysat restant à -80 °C et l’utiliser à des fins de contrôle.
REMARQUE: Ici, un exemple de ségrégation basée sur la sédimentation en fractions polysomiques, ribosomiques et « libres » SSU est fourni. Reportez-vous au tableau supplémentaire 1 fourni pour une autre approche. - Ultracentrifugez les tubes dans un rotor à godet oscillant de volume moyen à 4 °C, force g moyenne de 178 305 x g (facteur k 143,9) pendant 1 h 45 min.
- 30 minutes avant la fin du spin, configurez et définissez la ligne de base du fractionneur de gradient, comme décrit dans les étapes 1.4.7-1.4.9 du protocole de levure.
- Fractionner les gradients généralement comme décrit dans les étapes 1.4.10-1.4.11 du protocole de levure.
REMARQUE: Cette étape séparera les fractions SSU polysomiques, ribosomiques et « libres ». Les fractions polysomiques peuvent être utilisées dans les expériences de profilage des polysomes. - Transférer immédiatement les fractions collectées sur de la glace et, si elles ne sont pas traitées ultérieurement, conserver à -80 ° C jusqu’à 6 mois.
REMARQUE: Si le changement de tube collecteur de fraction est synchronisé avec l’identification et la ségrégation de fraction en ligne, nous recommandons d’utiliser jusqu’à 800 μL de fractions (temps de collecte de 32 s par fraction à 1,5 mL / min). Si le fractionnement est effectué sans utiliser la lecture d’absorbance en ligne, il est recommandé d’utiliser des fractions de 250 à 500 μL (10 à 20 s par fraction à 1,5 mL / min). Après séparation, les fractions peuvent être utilisées pour l’immunopurification, la microscopie électronique, la dénaturation immédiate de PAGE et le western blotting, ou soumises à une inversion de réticulation pour des analyses ultérieures d’ARN et / ou de protéomique.
- Préparer des gradients linéaires de saccharose de 15 % à 45 % p/v dans des tubes de polypropylène à paroi mince de 13 mL, de 14 x 89 mm, en utilisant la méthode de congélation-décongélation généralement décrite à l’étape 1.3.1 du protocole de levure, mais en utilisant le tampon 2(Figure 2a).
Résultats
Les complexes translationnels sont sensibles à la composition ionique des tampons, ce qui est particulièrement important lors de l’ultracentrifugation où les propriétés de sédimentation sont évaluées. Nous avons donc testé plusieurs tampons de sédimentation à l’aide de lysat clarifié extrait de matériaux de levure non fixes broyés, afin de sélectionner les conditions les mieux adaptées pour résoudre les complexes translationnels et séparer les sous-unités ribosomiques (SSU, LSU), les monosomes (RS) et les polysomes à travers le gradient. Tous les tampons étaient basés sur la composition du noyau contenant 25 mM HEPES-KOH pH 7,6 et 2 mM TNT. Les concentrations de KCl, MgCl2,CaCl2et EDTA ont été modifiées dans les tampons(figure 2a),et ces composants ont été ajoutés aux lysats avant le chargement du gradient et aux tampons du gradient de saccharose avant la coulée en gradient, en conséquence.
Dans les tampons, 1 et 2 complexes translationnels bien résolus ont été obtenus. Le tampon 1 a permis une séparation un peu meilleure des petites sous-unités ribosomiques (USS)(Figure 2a). L’omission de MgCl2 et l’ajout d’EDTA (tampons 3,4) ont entraîné la perte des propriétés de sédimentation élevées pour la plupart des polysomes et probablement leur désassemblage partiel(Figure 2a). Bien que l’ajout de 2,5 mM de CaCl2 ait donné des pics polysomiques un peu plus homogènes, l’amélioration a été marginale et la quantité globale de matière polysomique a diminué dans ce cas(figure 2a)par rapport aux tampons 1 et 2. Nous avons donc choisi le tampon 1 comme tampon de travail de choix.
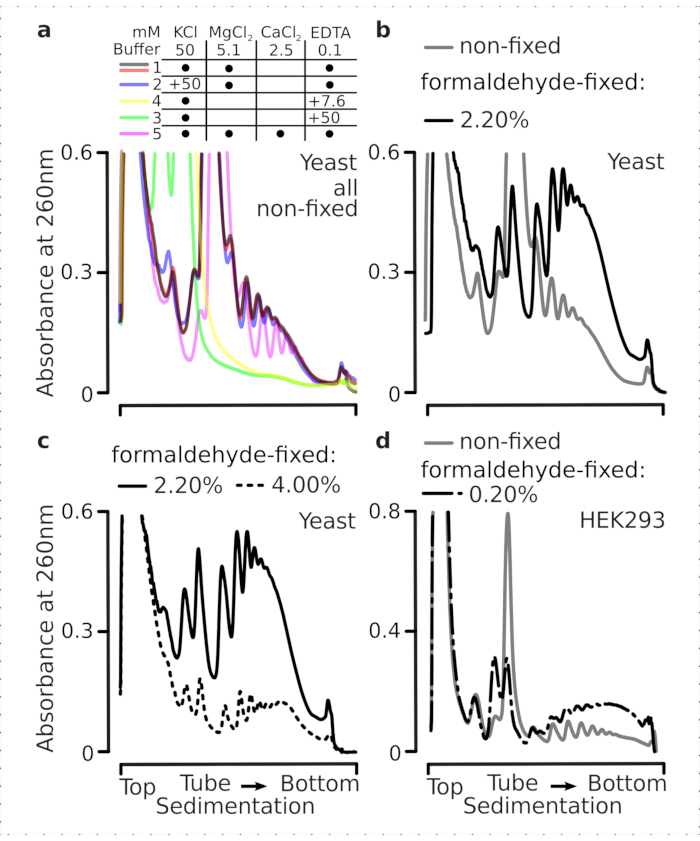
Figure 2: Conditions tampons pour l’extraction translationnelle complexe et évaluation de l’effet stabilisateur de la fixation. Les profils d’absorbance UV collectés à 260 nm pour le lysat total de cellules de levure sont séparés en gradients de saccharose de 10% à 40% p / v. a) Effets des sels mono- et divalents et de la séquestration des ions magnésium sur la sédimentation des matériaux extraits de cellules de levure non fixes. Les lignes rouges et grises représentent une réplique typique. (b,c) Comparaison des lysats dérivés de cellules de levure non fixes (ligne grise), 2,2 % (ligne noire) et 4,4 % (ligne pointillée noire) avec v de cellules de levure fixées au formaldéhyde. (d) Stabilisation des polysomes par l’optimisation de 0,2 % p/v de fixation du formaldéhyde (ligne pointillée et pointillée noire) des cellules HEK 293T, par rapport au matériau provenant des mêmes cellules non fixes (ligne grise). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Nous avons ensuite vérifié l’effet de la stabilisation polysomique par fixation à différentes concentrations de formaldéhyde. En utilisant le même matériau cellulaire, les tampons, la manipulation cellulaire et les approches de synchronisation, nous avons comparé le matériau extrait de cellules non fixes et de cellules fixées avec 2,2% et 4% p / v de formaldéhyde (Figure 2b, c). Nous avons constaté que 2,2 % p/v de formaldéhyde convenaient mieux à la fixation car, bien qu’il préserve parfaitement les polysomes, comme en témoigne le rapport polysome-monosome(figure 2b),il ne réduit pas le rendement global du matériau ribosomique par rapport à 4 % p/v de formaldéhyde, qui présentait des signes évidents de surfixation (figure 2c).
Pour le matériel dérivé de cellules de mammifères, en raison du rapport plus grand entre le volume tampon de lyse et le volume cellulaire requis par l’extraction à base de détergent, le tampon 2(figure 2a)a été utilisé. Cela a produit des complexes translationnels bien résolus lors de la sédimentation dans des gradients de saccharose(Figure 2d). Notamment, une concentration beaucoup plus faible de formaldéhyde de 0,2 % p/v a été utilisée, car des concentrations plus élevées ont entraîné une perte importante de matière polysomique et ribosomique (données non présentées). En similitude avec les résultats obtenus avec les cellules de levure, le matériau stabilisé par réticulation a démontré une meilleure conservation des polysomes et un rapport polysome-monosome plus élevé (Figure 2d).
Nous avons ensuite testé si les conditions de fixation du formaldéhyde sélectionnées sont suffisamment efficaces pour stabiliser l’ARNm activement traduit dans les fractions polysomiques à la suite de la réticulation, et l’amélioration du rendement polysomique n’est pas seulement une conséquence de l’inhibition de la fonction enzymatique et de la progression de l’allongement de la traduction. Nous avons utilisé de l’EDTA et du sel monovalent élevé (KCl) pour déstabiliser les polysomes et les ribosomes. Ces réactifs ont été ajoutés aux lysats de cellules de levure clarifiés et inclus dans tous les tampons et gradients de saccharose ultérieurs au-dessus de la composition du tampon 1, respectivement.
En effet, l’EDTA 15 mM a montré un effet déstabilisateur moindre sur les fractions polysomiques dérivées des cellules fixes(Figure 3a),confirmant que les complexes réticulés sont plus robustes. Les effets déstabilisateurs de l’EDTA peuvent être quelque peu surmontés en augmentant la concentration de formaldéhyde, car le matériau des 4% p/v des cellules fixées au formaldéhyde résiste mieux au déploiement(Figure 3a). Cependant, l’augmentation de la concentration d’EDTA à 50 mM a entraîné la déstabilisation de la plupart des complexes translationnels dans des conditions fixes et non fixes, comme on peut le déduire de la sédimentation plus lente du matériau et de l’absence de pics bien formés(figure 3b). Cela peut s’expliquer par le déploiement partiel des structures et la perte globale de compacité, plutôt que par la dissociation complète des composants polysomiques de l’ARNm. Même dans ce cas, le matériau réticulé a démontré une sédimentation plus rapide(figure 3b).
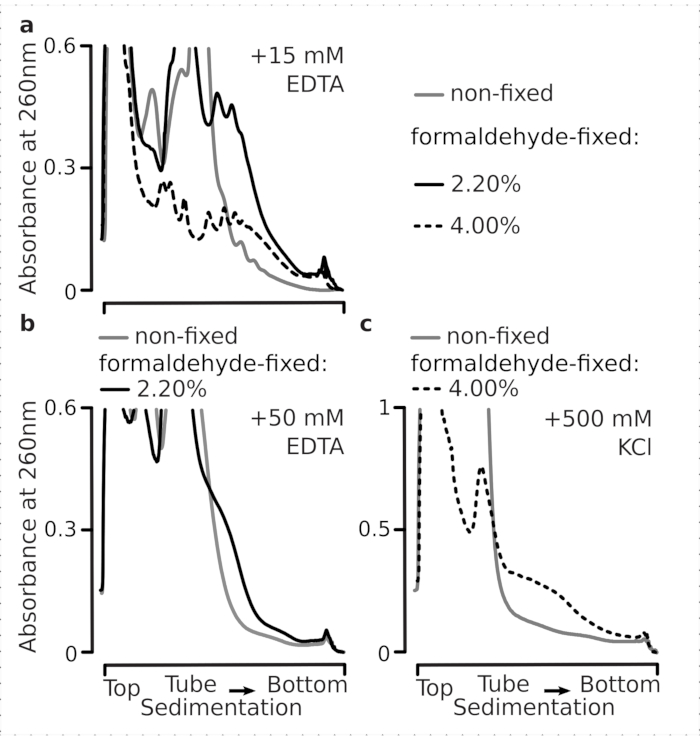
Figure 3: Effets de la fixation in vivo du formaldéhyde de levure sur la stabilité des polysomes. La mémoire tampon 1 (voir texte et figure 2a)a été utilisée dans toutes les expériences. Type de données et traçage comme décrit dans la légende de la figure 2. (a) Comparaison de l’ajout de 15 mM d’EDTA aux lysats cellulaires et aux tampons ultérieurs sur la stabilité des polysomes dérivés de cellules non fixes (ligne grise), 2,2 % (ligne noire) et 4 % (ligne pointillée noire) p/v de cellules fixées au formaldéhyde. b) identique àa), mais pour l’addition de 50 mM d’EDTA et à l’exclusion de 4 % p/v de cellules fixées au formaldéhyde. c) identique à (a), mais pour l’addition de 500 mM KCl et à l’exclusion de 2,2 % p/v de cellules fixées au formaldéhyde. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Semblable aux effets de l’EDTA, à 500 mM KCl, nous avons constaté une amélioration majeure de la stabilité avec 4% p/v de fixation du formaldéhyde(Figure 3c). La perte apparente de compacité dans ce cas peut également s’expliquer par un détachement partiel des constituants des complexes ribosomiques, plutôt que par leur dissociation complète de l’ARN. Dans l’ensemble, les polysomes dérivés de cellules fixées au formaldéhyde ont montré une résistance plus élevée au dépliage et à la déstabilisation structurelle, ce qui correspond à la formation de liaisons covalentes supplémentaires au sein de ces complexes.
Au cours de conditions de croissance stimulantes, les ARNm peuvent être rapidement initiés, ce qui entraîne l’accumulation de plusieurs ribosomes sur les mêmes molécules d’ARNm, qui forment des structures appelées polyribosomes ou polysomes. Les polysomes peuvent être séparés par ultracentrifugation dans des gradients de saccharose, où ils sédimentent en fonction de leur ordre (nombre de ribosomes simultanément attachés sur l’ARNm). Lorsque la traduction est supprimée, les ribosomes ne parviennent pas à s’engager dans un autre cycle de traduction assez tôt, ce qui entraîne un « désassemblage » (partiel) des polysomes, qui se présente comme un transfert modal vers les polysomes d’un ordre inférieur et une accumulation de monosomes4,26.
Un modèle de réponse translationnelle qui peut être visualisé au niveau de la distribution de l’ordre des polysomes peut être fourni par la famine de glucose. L’épuisement du glucose provoque l’un des effets inhibiteurs translationnels les plus spectaculaires et les plus rapides sur la levure1,3,40. Des études antérieures ont montré que dans les 1 min suivant l’épuisement du glucose, la perte de polysomes, l’accumulation de monosomes et l’inhibition de l’initiation de la traduction peuvent se produire4. Dans les 5 minutes suivant le supplément de glucose, la traduction est rapidement rétablie avec une augmentation évidente des polysomes3,4. Il a également été observé que la traduction était inhibée lorsque les cellules étaient exposées à des milieux contenant du glucose de 0,5 % (p/v) ou moins et qu’aucun effet n’était observé chez les taux de glucose de 0,6 % (p/v) ou plus.
Nous avons donc voulu déterminer si nos conditions de fixation sont adaptées à la préservation des différences translationnelles au sein de la dynamique de la réponse au stress glycémique, comme le permet le rapport polysome-monosome. Nous avons comparé le matériau des cellules cultivées en phase moyenne exponentielle sur un taux de glucose élevé (2,00 % p/v ajouté) avec ceux transférés pendant 10 min dans un milieu sans ou faiblement ajouté (0,00 % ou 0,25 % p/v, respectivement). La fixation a été réalisée à l’aide de 2,2 % p/v de formaldéhyde en parallèle dans le témoin (non affamé ; remplacement rapide du milieu par le même milieu standard contenant 2 % p/v de glucose ajouté, suivi d’une incubation de 10 min et d’une fixation) et de 10 min affamé (remplacement rapide du milieu par le même milieu mais faible 0,25 p/v ou sans glucose ajouté, suivie d’une incubation de 10 min et d’une fixation) cellules.
Conformément aux résultats précédents, nous avons observé que les cellules de levure suppriment fortement la traduction lors du stress de la famine de glucose (Figure 4a). Les conditions sans ajout et à faible teneur en glucose ont induit le désassemblage des polysomes, avec légèrement mais évidemment plus de polysomes retenus dans le cas d’un faible taux de glucose ajouté. Ainsi, la réponse d’élimination de la levure de glucose peut ne pas être de type all-on ou all-off et est progressivement réglée. Affirmant les attentes quant à l’action stabilisatrice de la réticulation du formaldéhyde, le matériau polysomique des cellules fixes a démontré une distinction plus élevée entre les cellules affamées et non affamées, préservant sans doute une plage dynamique plus élevée de la réponse(Figure 4b). Curieusement, dans le cas du matériel provenant des cellules fixes, une faible concentration de glucose ajouté a entraîné une abondance polysomique spécifique qui est beaucoup mieux différenciée de la condition de glucose sans ajout, par rapport aux cellules non fixes (Figure 4a). Il s’agit d’une indication forte de la pertinence de l’approche de fixation du formaldéhyde pour préserver et capturer des différences relativement infimes et transitoires dans l’équilibre de processus hautement dynamiques, par exemple lors de réponses translationnelles.

Figure 4: Capture des changements rapides dans la traduction des levures lors de la famine de glucose. La mémoire tampon 1 (voir texte et figure 2a)a été utilisée dans toutes les expériences. Type de données et traçage comme décrit dans la légende de la figure 2. (a) Lysats cellulaires obtenus à partir de cellules de levure non affamées (ligne grise), pauvres en glucose restreint (0,25 % p/v de glucose ajouté pendant 10 min; ligne brune) et appauvries en glucose (pas de glucose ajouté pendant 10 min; ligne rouge). b) identique à (a), mais pour les cellules fixées au formaldéhyde à 2,2 % p/v. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La surveillance de l’état translationnel par les ribosomes associés à la traduction active de l’ARNm à l’aide de la sédimentation par gradient de saccharose (« profilage des polysomes ») est une technique largement appliquée26,27,28. En combinaison avec l’analyse quantitative des microréseaux et plus récemment avec le séquençage à haut débit28,44, le profilage des polysomes fournit des informations sur les ARNm associés aux ribosomes à l’échelle du transcriptome. Avec plusieurs hypothèses, il a été traditionnellement soutenu dans le domaine de la biosynthèse des protéines que la présence polysomique est une indication d’implication active dans la traduction des ARNm respectifs. Une autre conclusion est souvent (mais pas toujours) justifiée, que plus les ribosomes sont présents sur un ARNm d’une longueur donnée (plus l’ordre des polysomes est élevé), plus l’ARNm est activement impliqué dans la traduction. Ainsi, séparer la fraction polysomique du reste du matériel peut être utile du point de vue de l’isolement de l’ARN activement traduit. Dans les approches de profilage de l’empreinte, et en particulier TCP-seq10,38,39 qui génère une population distincte des SSU libérés dérivées des complexes de codons de balayage, de démarrage et d’arrêt, il peut être également judicieux d’éliminer les sous-unités ribosomiques qui ne co-sédimentent pas avec les monosomes ou polysomes complets.
Nous avons donc utilisé la séparation des ARNm « non traduits » tels que les SSU libres (ARNm liés à un seul SSU ou SSU sans ARNm attaché) du pool « traduisant activement » des ARNm. Pour ce faire, nous avons supposé que les ARNm impliqués dans les interactions avec un (mono-) ou plusieurs ribosomes (polysomes) peuvent être activement traduits. De tels complexes peuvent être séparés des autres par leur coefficient de sédimentation plus élevé. Nous avons également suggéré de séparer le pool d’ARNm « activement traduit » en un coussin de saccharose (50% p / v de saccharose) au lieu de granuler directement le matériau sur la paroi du tube. La centrifugation des complexes à sédimentation rapide dans le coussin nous a permis de surveiller la séparation à l’aide de la lecture du profil d’absorbance et d’obtenir un rendement plus élevé du matériau solubilisé, non agrégé et non dénaturé, par rapport à la granulation et à la re-solubilisation10,38.
Dans l’ensemble, pour purifier les SSU individuels, les ribosomes, les disomes et les polysomes potentiellement compacts d’un ordre supérieur, les lysats clarifiés fixes ont été soumis à un processus d’ultracentrifugation en deux étapes(Figure 5). Dans le premier gradient de saccharose, l’ultracentrifugation a entraîné la séparation des USS libres et des ULS dans la partie supérieure (10 % à 20 % p/v de saccharose) du gradient, tandis que le pool traduit réticulé comprenant des polysomes et des ARNm associés à un ribosome complet était concentré au bas (50 % p/v de saccharose) du gradient (Figure 5a ). Les 50 % inférieurs p/v de la couche de saccharose contenant le pool d’ARNm traduit ont ensuite été concentrés et son ARN digéré avec la RNase I, suivie d’une deuxième ultracentrifugation du gradient de saccharose pour obtenir des disomes résistants aux SSU, LSU, RS, RNase (DS) séparés et une fraction mineure de polysomes résistants aux nucléases d’ordre supérieur(Figure 5b). La coloration négative à l’acétate d’uranyle et l’imagerie au microscope électronique à transmission ont confirmé l’identité des complexes isolés à chaque étape de sédimentation (Figure 5).

Figure 5: Isolement des fractions totales d’ARN traduites par rapport à l’ARN non traduit. (a,c) Schéma (à gauche) et résultats représentatifs respectifs (à droite; type de données et tracé comme décrit dans la légende de la figure 2) de (a) première séparation discontinue du gradient de saccharose des fractions de cytosol non traduites, y compris les SSU libres et le pool d’ARNm traduit identifié par co-sédimentation avec des ribosomes et des polysomes, et (c) la séparation des complexes ribosomiques individuels libérés du pool d’ARNm traduit par digestion contrôlée de la RNase I et ultracentrifugation par un deuxième gradient linéaire de saccharose en fractions SSU, LSU, ribosomiques (RS) et disomales résistantes aux nucléases (DS). Des quantités élevées (15 UA260)et faibles(8 UA 260)de matière digérée non affamée ont été incluses pour démontrer la possibilité d’augmenter les charges d’ultracentrifugation lorsque des fractions mineures présentent un intérêt. Des polysomes résistants aux nucléases d’ordre supérieur peuvent également être identifiés (p. ex., les trisomes dans les exemples fournis). (b,d) Images TEM représentatives de fractions contrastées par l’acétate d’uranyle de (a, c), respectivement telles qu’étiquetées. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Afin de vérifier l’adéquation du schéma de fixation pour la rétention des protéines transitoires associées aux ribosomes (en particulier les eIF), nous avons testé la co-sédimentation d’eIF4A, un eIF labile lié dynamiquement au ribosome, à travers les fractions ribosomales. Nous avons tiré parti de la souche de levure marquée eIF4A Tandem Affinity Purification (TAP) (TIF1-TAP) et étudié la présence d’eIF4A dans le matériel dérivé des cellules fixes vs non fixes en utilisant des anticorps anti-TAP, par rapport à l’abondance de Pab1p comme contrôle supplémentaire de liaison à l’ARN, en utilisant SDS-PAGE suivi du western blotting (Figure 6).
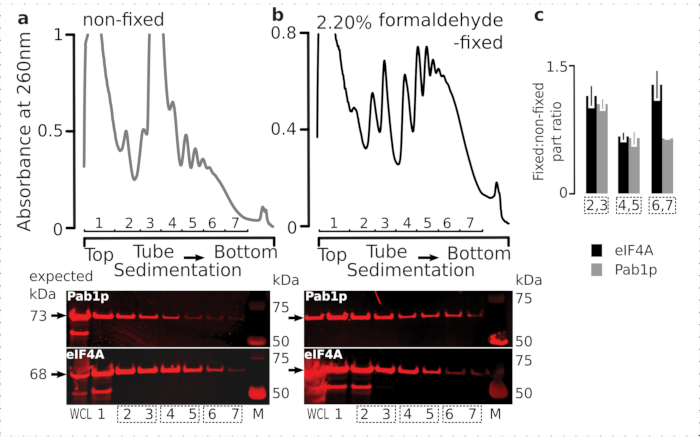
Figure 6: Stabilisation des protéines transitoires dans les complexes translationnels lors de la fixation in vivo du formaldéhyde. (a,b) (top plots) Lysat de cellules entières (WCL) de (a) cellules de levure eIF4A-TAP non fixes et (b) fixées à 2,2% de formaldéhyde séparées par ultracentrifugation et visualisées comme décrit dans la légende de la figure 2. (parcelles du bas) Imagerie par transfert western des fractions de gradient de saccharose respectives lors de la séparation du matériau analysé dans les gradients correspondants (graphiques supérieurs) et WCL comme témoin. c)Rapport moyen entre l’abondance eIF4A ou Pab1p en fractions de matière fixe et non fixe. Les proportions relatives (normalisées au signal de toutes les 2 à 7 fractions) d’eIF4A (barres noires) et de Pab1p (barres grises) ont été calculées sur 2,3 (SSU, LSU), 4,5 (RS, polysomes légers) et 6,7 (polysomes lourds) à partir des données de (a,b) (graphiques du bas), et leur rapport fixe à non fixe a été pris. Les barres d’erreur indiquent l’écart-type du rapport par rapport à la moyenne, les fractions regroupées (cases en pointillés) étant traitées comme des répétitions. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Conformément à leur grande abondance dans les cellules, nous avons observé une intensité élevée du signal provenant à la fois des protéines du lysat cellulaire entier (WCL) et des fractions à sédimentation plus lente dérivées de cellules non fixes(Figure 6a,panneau inférieur). Nous avons également détecté des quantités substantielles de ces protéines dans le WCL dérivées des cellules fixes et rassurant l’efficacité de l’extraction de matériaux réticulés et l’absence de pertes inattendues(Figure 6b,panneau du bas). Cependant, contrairement aux cellules non fixes, le matériau des cellules fixes a montré une présence relative élevée d’eIF4A dans les fractions ribosomiques à sédimentation plus rapide, par rapport à Pab1p(Figure 6c). Ce résultat suggère que eIF4A reste plus fermement associé aux polysomes dans les matériaux réticulés au formaldéhyde.
Après avoir confirmé l’effet stabilisateur positif et spécifique de la réticulation sur la présence d’eIF4A dans les fractions ribosomiques, nous avons utilisé le matériau fixe de la souche de levure marquée eIF4A (TIF1-TAP) pour capturer et enrichir les complexes contenant de l’eIF4A par purification d’affinité avec des billes d’IgG magnétiques. Nous avons des fractions WCL enrichies en affinité, SSU libres et polysomiques (pool d’ARNm traduit) après la première sédimentation par gradient de saccharose (par exemple, section 1.3 du protocole de levure), ainsi que des fractions SSU, LSU et RS de la deuxième sédimentation lors du désassemblage du pool traduit en complexes individuels avec la RNase I (par exemple, section 1.4 du protocole de levure) (Figure 7 ). Dans tous les cas, à l’exception de la fraction LSU, nous avons pu observer un enrichissement sélectif de l’eIF4A dans les fractions purifiées (éluat, E), par rapport à la présence de β-actine dans la matière première (input, I)(Figure 7).
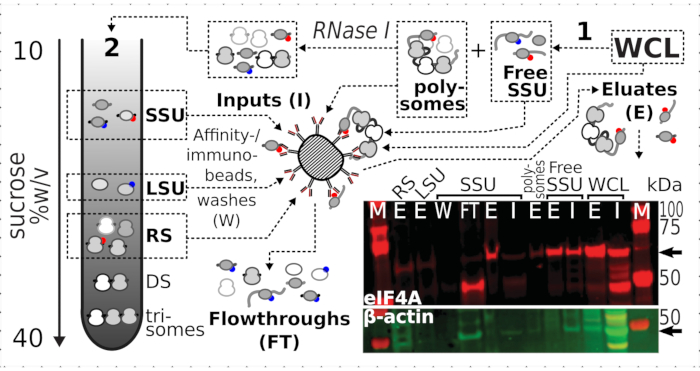
Figure 7: Immunopurification sélective de complexes translationnels stabilisés au formaldéhyde in vivo par eIF4A transitoirement associés. Le schéma illustre la source de différents complexes translationnels et de l’épitope eIF4A, y compris le WCL clarifié non fractionné des cellules de levure eIF4A-TAP; SSU libres et pool d’ARN traduit (polysomes) séparés lors de la première ultracentrifugation; Fractions SSU, LSU et RS libérées de l’ARN traduit par digestion de la RNase I et séparées par deuxième ultracentrifugation (voir texte). L’image de western blot fournit une visualisation de l’abondance d’eIF4A dans les fractions par rapport à l’abondance du contrôle de l’β-actine simultanément coloré. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau supplémentaire 1. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
La fixation du formaldéhyde est une méthode pratique et populaire pour obtenir une réticulation rapide in vivo des biomolécules10,36,45,46,47,48. Par rapport aux autres cibles potentielles de biomolécules, la capture réussie de complexes translationnels nécessite une fixation immédiate lors du refroidissement instantané des cellules ou d’un autre matériau. Sans la stabilisation non retardée, il est possible que différents processus liés à la traduction se poursuivent, déplaçant la distribution complexe loin de l’état in vivo non perturbé49. Par rapport aux autres méthodes d’arrêt translationnel et de stabilisation du complexe ribosomique, la rapidité de l’action du formaldéhyde à travers les membranes cellulaires et la nature aveugle des réticulations promettent la préservation de la diversité maximale des intermédiaires complexes de traduction plus proches de leurs états distribués nativement50.
L’approche présentée ici a été établie et optimisée dans les cellules de levure et de mammifère, et des méthodes ont maintenant été dérivées par d’autres groupes pour une utilisation dans du matériel biologique plus diversifié, comme chez les vertébrés entiers (par exemple, les embryons de poisson zèbre)10,38,39,49,51,52 . Bien que ces travaux rassurent collectivement la polyvalence et la large applicabilité de l’approche, la réticulation rapide du formaldéhyde des complexes translationnels peut être considérée comme quelque peu difficile à transposer à de nouveaux types de matériel biologique en raison de la nécessité d’optimisations et d’ajustements.
Une exigence primordiale pour le succès de la méthode est la ré-optimisation de la concentration du formaldéhyde et la technique de collecte et de perturbation cellulaire. Les cellules de levure moins perméables, petites et rondes nécessitent une concentration de formaldéhyde beaucoup plus élevée (au moins 10 fois) et une perturbation physique des cellules fixes. En revanche, les cellules de mammifères adhérentes grandes et aplaties en culture peuvent être facilement surfixées et nécessitent une manipulation douce lors de la fixation, tandis que l’extraction des complexes fixes peut être effectuée chimiquement avec perturbation de la membrane à l’aide de détergents. La sous-réticulation peut permettre à des intermédiaires moins stables ou à plus courte durée de vie de se dissocier ou de s’infiltrer dans un état ultérieur. La surréticulation peut affecter négativement la capacité d’isoler et d’étudier les fractions ribosomiques et peut créer des biais sélectifs tels que l’épuisement plus profond des complexes lourds. Dans notre observation, même des altérations mineures, telles que le type de cellules humaines adhérentes utilisées, peuvent affecter le rendement des complexes réticulés récupérés et peuvent nécessiter une réoptimisation du schéma de réticulation. Nous pouvons également prévoir que les cellules ayant des propriétés de perméabilité sensiblement différentes, telles que les cellules végétales, nécessiteront une optimisation supplémentaire étendue des conditions de fixation52. Pourtant, il est difficile d’imaginer un type de matériel biologique qui serait totalement incompatible avec l’approche.
Une considération pertinente pour le protocole de fixation des mammifères est la densité et la quantité de matériel cellulaire utilisé comme intrant. Il est recommandé de faire croître les cellules en continu sans réensemencement ou autres perturbations pendant au moins 2 jours pour éviter les influences externes sur la dynamique de la traduction cellulaire. Applicable pour la plupart des types de cellules, mais pour la majorité des cellules adhérentes, des niveaux de confluence ne dépassant pas 70% garantiront l’absence d’effets majeurs d’inhibition de contact pouvant affecter négativement et de manière imprévisible les taux de traduction.
Une autre caractéristique intéressante, et potentiellement particulièrement pratique, de la fixation du formaldéhyde découlant de sa réactivité aveugle est l’effet de stabilisation sur les complexes translationnels dans les systèmes de taxonomie mixte. Les complexes bactériens, et plus encore translationnels de mitochondries, de chloroplastes et de différents parasites intracellulaires, ont été notoirement difficiles à cibler avec des inhibiteurs de traduction spécifiques. En revanche, dans les données TCP-seq, les empreintes mappées au mitotranscriptome sont facilement observables dans les données38,39,50. Un développement ultérieur intéressant pourrait être l’utilisation de l’approche pour étudier la traduction dans des microcommunautés entières, telles que dans des échantillons de sol, d’eau ou d’intestin, où un arrêt translationnel rapide fiable et une stabilisation complexe avec tout autre moyen seraient problématiques.
Il convient également de mentionner que pour les matériaux les plus compliqués (tels que les tissus durs et / ou volumineux), rien n’empêche l’utilisation de la stabilisation du formaldéhyde immédiatement après la perturbation cellulaire et l’homogénéisation du matériau. Cette approche est déjà fréquemment utilisée pour éliminer le délai d’entrée cellulaire lors de la stabilisation de complexes translationnels avec des inhibiteurs spécifiques de petites molécules33,53,54,55. Étant donné que la fixation du formaldéhyde a été traditionnellement utilisée avec d’excellents résultats pour la stabilisation d’échantillons ex vivo / in vitro dans des applications telles que la microscopie électronique45,56 , 57,58, nous pouvons nous attendre à des effets encore moins négatifs dans ce cas, en particulier ceux associés à la mauvaise extraction des complexes translationnels des cellules complètement fixées.
Nos résultats confirment la facilité d’utilisation de la fixation rapide du formaldéhyde pour stabiliser les complexes hautement transitoires, tels que ceux qui incluent eIF4A. Il est à noter que, contrairement aux mammifères, la levure eIF4A est beaucoup plus faiblement associée au complexe de liaison au capuchon eIF4F et, par conséquent, aux complexes translationnels en général. eIF4A est généralement perdu lors d’une purification étendue du matériel ribosomique dans la levure29,59,60,61,62,63. Pourtant, dans le matériau de levure fixe in vivo,il est possible d’obtenir un enrichissement fiable d’eIF4A dans toutes les fractions de complexes translationnels où sa présence serait anticipée. Les données Sel-TCP-seq publiées précédemment ont démontré l’enrichissement d’eIF2 et d’eIF3 qui s’associent plus fortement aux ribosomes (mais ont également révélé un assemblage transitoire de complexe protéique co-translationnel)39. Ainsi, la méthode convient à la détection des constituants attachés plus forts et plus faibles des complexes translationnels.
Pour résumer, nous avons présenté une approche utile pour mieux comprendre principalement les changements qui se produisent à la phase d’initiation de la traduction et lorsque la distribution ribosomique sur l’ARNm est minimalement perturbée. Il est important de noter que l’approche convient à la stabilisation de composants relativement labiles et dynamiques de complexes translationnels, tels que eIF4A, et peut être largement utilisée sous réserve des optimisations nécessaires. Nous avons également fourni des preuves de l’utilité de la fixation du formaldéhyde dans les scénarios de changement dynamique rapide de la traduction, ouvrant des domaines d’investigation tels que les réponses cellulaires rapides aux changements environnementaux ou aux conditions de stress.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail a été soutenu par une subvention du projet de découverte du Conseil australien de la recherche (DP180100111 à T.P. et N.E.S.), une subvention de chercheur du Conseil national de la santé et de la recherche médicale (GNT1175388 à N.E.S.) et une bourse de recherche (APP1135928 à T.P.). Les auteurs reconnaissent les installations de Microscopy Australia au Centre for Advanced Microscopy de l’Université nationale australienne, une installation financée par l’Université et le gouvernement fédéral.
matériels
| Name | Company | Catalog Number | Comments |
| Yeast extract | Merck, Sigma-Aldrich | 70161 | |
| Peptone | Merck, Sigma-Aldrich | 70178 | |
| D-Glucose (Dextrose) | Merck, Sigma-Aldrich | 49139 | |
| Adenine sulphate | Amresco | 0607-50G | |
| Formaldehyde solution | Merck Sigma-Aldrich | F11635-500ML | ACS reagent, 37 wt. % in H2O, contains 10-15% Methanol as stabiliser (to prevent polymerisation) |
| RNaseOUT™ Recombinant Ribonuclease Inhibitor | Invitrogen™ byThermo Fischer Scientific | 10777019 | |
| cOmplete™, EDTA-free Protease Inhibitor Cocktail | COEDTAF-RO Roche by Merck | 11873580001 | |
| Magnesium chloride solution | (Merck/Sigma-Aldrich) | M1028 | |
| Ethylenediaminetetraacetic acid solution | (Merck/Sigma-Aldrich) | E7889 | |
| Ambion™ RNase I, cloned, 100 U/µL | Ambion | AM2294 | |
| SUPERase•In™ RNase Inhibitor (20 U/μL) | Invitrogen™ by Thermo Fisher Scientific | AM2694 | |
| Acidic phenol:chlorophorm:isoamyl alcohol 125:24:1 (pH 4.0-5.0) | (Merck/Sigma-Aldrich) | P1944-100ML | |
| Dynabeads™ Goat Anti-Mouse IgG | Invitrogen™ by Thermo Fisher Scientific) | 11033 | |
| Sodium Acetate (3 M), pH 5.5 | Invitrogen™ by Thermo Fisher Scientific) | AM9740 | |
| Glycogen (5 mg/ml) | Invitrogen™ by Thermo Fisher Scientific) | AM9510 | |
| Ethyl alcohol, Pure | Merck; Sigma Aldrich | E7023 | |
| Amersham™ Hybond® P Western blotting membranes, PVDF | Merck | GE10600023 | PVDF membrane for western blotting |
| Bolt™ 4 to 12%, Bis-Tris, 1.0 mm, Mini Protein Gel | Invitrogen™ by ThermoFischer Sientific | NW04120BOX | Protein gel |
| 4X Bolt™ LDS Sample Buffer | Invitrogen™ by ThermoFischer Sientific | B0007 | LDS sample loading buffer |
| Precision Plus Protein™ Kaleidoscope™ Prestained Protein Standards | BioRad | 1610375 | Protein ladder |
| 20X Bolt™ MES SDS Running Buffer | ThermoFischer Scientific | B0002 | PAGE runninjg buffer |
| Intercept® (PBS) Blocking Buffer | LI-COR | 927-70001 | Odyssey Blcoking buffer (PBS) |
| IRDye® 800CW Goat anti-Mouse IgG Secondary Antibody | LI-COR | 92632210 | |
| IRDye® 800CW Goat anti-Rabbit IgG Secondary Antibody | LI-COR | 92632211 | |
| TAP Tag Polyclonal Antibody | Invitrogen™ by ThermoFischer Sientific | CAB1001 | |
| Anti-beta Actin antibody | Abcam | ab8227 | |
| Sucrose | (Merck/Sigma-Aldrich) | 84097 | BioUltra, for molecular biology, ≥99.5% (HPLC) |
| DL-Dithiothreitol solution | (Merck/Sigma-Aldrich) | 43816 | BioUltra, for molecular biology, ~1 M in H2O |
| Terumo Syringe 1CC/mL | Terumo Syringe | 878499 | |
| Potassium chloride | (Merck/Sigma-Aldrich) | 60128 | |
| HEPES | (Merck/Sigma-Aldrich) | H3375 | |
| Dulbecco's Modified Eagle's Medium - high glucose | Sigma Aldrich | D5796 | |
| Fetal Bovine Serum | Sigma Aldrich | 12003C | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300062 | |
| Dulbecco's Phosphate Buffered Saline with Calcium and magnesium | Sigma-Aldrich | D8662 | |
| Glycine | Sigma-Aldrich | G7126 | |
| Tris hydrochloride | Merck/Sigma-Aldrich | 10812846001 | |
| Sodium dodecyl sulfate | Merck/Sigma-Aldrich | 436143 | |
| IGEPAL CA-630 | Merck/Sigma-Aldrich | I3021 | |
| Rnasin Ribonuclease Inhibitor | Promega | N2111 | |
| Stainless steel grinding jar | Retsch | 02.462.0059 | |
| MM400 mixer mill | Retsch | 20.745.0001 | |
| Gradient Fractionator | Brandel | BRN-BR-188 | |
| Thermomixer R | Eppendorf | Z605271 | |
| Nanodrop spectrophotometer | Thermo Fisher Scientific | ND-2000 | |
| 0.5-ml microcentrifuge tubes with locking devices | Eppendorf Safe-Lock | 30121023 | |
| Mini Gel Tank | (Thermo Fisher Scientific) | A25977 | PAGE running tank |
| 5 mL, Open-Top Thinwall Ultra-Clear Tube, 13 x 51mm | Beckman-Coulter | 344057 | |
| 13.2 mL, Certified Free Open-Top Thinwall Polypropylene, 14 x 89mm - 50Pk | Beckman-Coulter | 331372 | |
| Amicon Ultra-0.5 ultrafiltration devices | Merck | UFC5030 | Ultracel-30 regenerated cellulose membrane, 0.5 mL sample volume |
| Thermo Sorvall Evolution RC Floor Super Speed Centrifuge | Cambridge Scientific | 15566 | |
| Beckman Coulter Optima L-90K | GMI | 8043-30-1191 | |
| Nunc EasYFlask 175cm2 | Thermofisher Scientific | 159910 | |
| Falcon 50 mL Conical Centrifuge Tubes | Thermofisher Scientific | 14-432-22 | |
| 25 mL Serological Pipette | Sigma-Aldrich | SIAL1250 | |
| 10 mL Serological Pipette | Sigma-Aldrich | SIAL1100 | |
| DNA lobind tubes | Eppendorf | 30108051 | |
| Cold Centrifuge 5810 R | Eppendorf | EP022628188 | for 50 mL tubes |
| Orbital Shaking Incubator | Ratek | OM11 | |
| Frezco 17 Microcentrifuge | Thermofisher Scientific | 75002402 | |
| Eppendorf DNA lo-bind tubes | Merck/Sigma-Aldrich | EP0030108051 | |
| Eppendorf® Protein LoBind tubes | Merck/Sigma-Aldrich | EP0030108116 | |
| SW 41 Ti Swinging bucket rotor | Beckman-Coulter | 331362 | |
| Heracell™ 150i CO2 Incubator, 150 L | Thermofisher Scientific | 51026282 | |
| 0,3 mL ultra-fine II short insulin syringe | BD Medical | 328822 | |
| 3 mL syringe with Luer Lok tip | BD Medical | 302113 | |
| 25 G x 16 mm Hypodermic Needle | Terumo | TUAN2516R1 |
Références
- Janapala, Y., Preiss, T., Shirokikh, N. E. Control of translation at the initiation phase during glucose starvation in yeast. International Journal of Molecular Sciences. 20 (16), 4043 (2019).
- Masvidal, L., Hulea, L., Furic, L., Topisirovic, I., Larsson, O. mTOR-sensitive translation: Cleared fog reveals more trees. RNA Biology. 14 (10), 1299-1305 (2017).
- Ashe, M. P., De Long, S. K., Sachs, A. B. Glucose depletion rapidly inhibits translation initiation in yeast. Molecular Biology of the Cell. 11 (3), 833-848 (2000).
- Crawford, R. A., Pavitt, G. D. Translational regulation in response to stress in Saccharomyces cerevisiae. Yeast. 36 (1), 5-21 (2019).
- Melamed, D., Pnueli, L., Arava, Y. Yeast translational response to high salinity: global analysis reveals regulation at multiple levels. RNA. 14 (7), 1337-1351 (2008).
- Hershey, J. W., Sonenberg, N., Mathews, M. B. Principles of translational control: An overview. Cold Spring Harbor Perspectives in Biology. 4 (12), 011528 (2012).
- Mata, J., Marguerat, S., Bähler, J. Post-transcriptional control of gene expression: a genome-wide perspective. Trends in Biochemical Sciences. 30 (9), 506-514 (2005).
- Spriggs, K. A., Bushell, M., Willis, A. E. Translational regulation of gene expression during conditions of cell stress. Molecular Cell. 40 (2), 228-237 (2010).
- Liu, B., Qian, S. B. Translational reprogramming in cellular stress response. Wiley Interdisciplinary Reviews RNA. 5 (3), 301-315 (2014).
- Archer, S. K., Shirokikh, N. E., Beilharz, T. H., Preiss, T. Dynamics of ribosome scanning and recycling revealed by translation complex profiling. Nature. 535 (7613), 570-574 (2016).
- Hinnebusch, A. G., Ivanov, I. P., Sonenberg, N. Translational control by 5'-untranslated regions of eukaryotic mRNAs. Science. 352 (6292), 1413-1416 (2016).
- Dever, T. E., Green, R. The elongation, termination, and recycling phases of translation in eukaryotes. Cold Spring Harbor Perspectives in Biology. 4 (7), 013706 (2012).
- Shirokikh, N. E., Preiss, T. Translation initiation by cap-dependent ribosome recruitment: Recent insights and open questions. Wiley Interdisciplinary Reviews RNA. 9 (4), 1473 (2018).
- Jiménez-Díaz, A., Remacha, M., Ballesta, J. P., Berlanga, J. J. Phosphorylation of initiation factor eIF2 in response to stress conditions is mediated by acidic ribosomal P1/P2 proteins in Saccharomyces cerevisiae. PLoS One. 8 (12), 84219 (2013).
- Sonenberg, N., Hinnebusch, A. G. Regulation of translation initiation in eukaryotes: mechanisms and biological targets. Cell. 136 (4), 731-745 (2009).
- Majmundar, A. J., Wong, W. J., Simon, M. C. Hypoxia-inducible factors and the response to hypoxic stress. Molecular Cell. 40 (2), 294-309 (2010).
- Barraza, C. E., et al. The role of PKA in the translational response to heat stress in Saccharomyces cerevisiae. PLoS One. 12 (10), 0185416 (2017).
- Richter, K., Haslbeck, M., Buchner, J. The heat shock response: Life on the verge of death. Molecular Cell. 40 (2), 253-266 (2010).
- Jamar, N. H., Kritsiligkou, P., Grant, C. M. The non-stop decay mRNA surveillance pathway is required for oxidative stress tolerance. Nucleic Acids Research. 45 (11), 6881-6893 (2017).
- Chen, Z., et al. The complete pathway for thiosulfate utilization in Saccharomyces cerevisiae. Applied and Environmental Microbiology. 84 (22), (2018).
- Marzluf, G. A. Molecular genetics of sulfur assimilation in filamentous fungi and yeast. Annual Review of Microbiology. 51, 73-96 (1997).
- Miller, D., Brandt, N., Gresham, D. Systematic identification of factors mediating accelerated mRNA degradation in response to changes in environmental nitrogen. PLoS Genetics. 14 (5), 1007406 (2018).
- Zhang, W., Du, G., Zhou, J., Chen, J. Regulation of sensing, transportation, and catabolism of nitrogen sources in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 82 (1), (2018).
- Tokpohozin, S. E., Fischer, S., Becker, T. Selection of a new Saccharomyces yeast to enhance relevant sorghum beer aroma components, higher alcohols, and esters. Food Microbiology. 83, 181-186 (2019).
- Walker, G. M., Stewart, G. G. Saccharomyces cerevisiae in the production of fermented beverages. Beverages. 2 (4), 30 (2016).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Jin, H. Y., Xiao, C. An integrated polysome profiling and ribosome profiling method to investigate in vivo translatome. Methods in Molecular Biology. 1712, 1-18 (2018).
- Arava, Y., et al. Genome-wide analysis of mRNA translation profiles in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences of the United States of America. 100 (7), 3889-3894 (2003).
- Lackner, D. H., et al. A network of multiple regulatory layers shapes gene expression in fission yeast. Molecular Cell. 26 (1), 145-155 (2007).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-Wide Analysis in Vivo of Translation with Nucleotide Resolution Using Ribosome Profiling. Science. 324 (5924), 218-223 (2009).
- Ingolia, N. T., Hussmann, J. A., Weissman, J. S. Ribosome Profiling: Global Views of Translation. Cold Spring Harbor Perspectives in Biology. 11 (5), (2019).
- Gerashchenko, M. V., Lobanov, A. V., Gladyshev, V. N. Genome-wide ribosome profiling reveals complex translational regulation in response to oxidative stress. Proceedings of the National Academy of Sciences of the United States of America. 109 (43), 17394-17399 (2012).
- Hussmann, J. A., Patchett, S., Johnson, A., Sawyer, S., Press, W. H. Understanding biases in ribosome profiling experiments reveals signatures of translation dynamics in yeast. Proceedings of the National Academy of Sciences Genetics. 11 (12), 1005732 (2015).
- Santos, D. A., Shi, L., Tu, B. P., Weissman, J. S. Cycloheximide can distort measurements of mRNA levels and translation efficiency. Nucleic Acids Research. 47 (10), 4974-4985 (2019).
- Schneider-Poetsch, T., et al. Inhibition of eukaryotic translation elongation by cycloheximide and lactimidomycin. Nature Chemical Biology. 6 (3), 209-217 (2010).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: A tool for the study of chromatin complexes. Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
- Kage, U., Powell, J. J., Gardiner, D. M., Kazan, K. Ribosome profiling in plants: What is not lost in translation. Journal of Experimental Botany. 71 (18), 5323-5332 (2020).
- Shirokikh, N. E., Archer, S. K., Beilharz, T. H., Powell, D., Preiss, T. Translation complex profile sequencing to study the in vivo dynamics of mRNA-ribosome interactions during translation initiation, elongation and termination. Nature Protocols. 12 (4), 697-731 (2017).
- Wagner, S., et al. Selective translation complex profiling reveals staged initiation and co-translational assembly of initiation factor complexes. Molecular Cell. 79 (4), 546-560 (2020).
- Zlotorynski, E. Profiling ribosome dynamics. Nature Reviews Molecular Cell Biology. 17 (9), 535-535 (2016).
- Sen, N. D., Gupta, N., S, K. A., Preiss, T., Lorsch, J. R., Hinnebusch, A. G. Functional interplay between DEAD-box RNA helicases Ded1 and Dbp1 in preinitiation complex attachment and scanning on structured mRNAs in vivo. Nucleic Acids Research. 47 (16), 8785-8806 (2019).
- Zhao, J., Qin, B., Nikolay, R., Spahn, C. M. T., Zhang, G. Translatomics: The global view of translation. International Journal of Molecular Sciences. 20 (1), 20010212 (2019).
- Luthe, D. S. A simple technique for the preparation and storage of sucrose gradients. Analytical Biochemistry. 135 (1), 230-232 (1983).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Review Genetics. 10 (1), 57-63 (2009).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trends in Biochemical Sciences. 25 (3), 99-104 (2000).
- Schmiedeberg, L., Skene, P., Deaton, A., Bird, A. A Temporal Threshold for Formaldehyde Crosslinking and Fixation. PLoS One. 4 (2), 4636 (2009).
- Solomon, M. J., Varshavsky, A. Formaldehyde-mediated DNA-protein crosslinking: A probe for in vivo chromatin structures. Proceedings of the National Academy of Sciences. 82 (19), 6470-6474 (1985).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping proteinDNA interactions in vivo with formaldehyde: Evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Bohlen, J., Fenzl, K., Kramer, G., Bukau, B., Teleman, A. A. Selective 40S footprinting reveals cap-tethered ribosome scanning in human cells. Molecular Cell. 79 (4), 561-574 (2020).
- Shirokikh, N. E. Translation complex stabilization on messenger RNA and footprint profiling to study the RNA responses and dynamics of protein biosynthesis in the cells. Critical Reviews in Biochemistry and Molecular Biology. , (2021).
- Giess, A., et al. Profiling of small ribosomal subunits reveals modes and regulation of translation initiation. Cell Reports. 31 (3), 107534 (2020).
- Firmino, A. A. P., et al. Separation and paired proteome profiling of plant chloroplast and cytoplasmic ribosomes. Plants (Basel). 9 (7), (2020).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic Acids Research. 42 (17), 134 (2014).
- Santos, D. A., Shi, L., Tu, B. P., Weissman, J. S. Cycloheximide can distort measurements of mRNA levels and translation efficiency. Nucleic Acids Research. 47 (10), 4974-4985 (2019).
- Schneider-Poetsch, T., et al. Inhibition of eukaryotic translation elongation by cycloheximide and lactimidomycin. Nature Chemical Biology. 6 (3), 209-217 (2010).
- Plénat, F., et al. Formaldehyde fixation in the third millennium. Annales De Pathologie. 21 (1), 29-47 (2001).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proceedings of the National Academy of Sciences. 105 (7), 2415-2420 (2008).
- Wang, N. S., Minassian, H. The formaldehyde-fixed and paraffin-embedded tissues for diagnostic transmission electron microscopy: A retrospective and prospective study. Human Pathology. 18 (7), 715-727 (1987).
- Grifo, J. A., et al. Characterization of eukaryotic initiation factor 4A, a protein involved in ATP-dependent binding of globin mRNA. Journal of Biological Chemistry. 257 (9), 5246-5252 (1982).
- Li, Y. Commonly used tag combinations for tandem affinity purification. Biotechnology and Applied Biochemistry. 55 (2), 73-83 (2010).
- Blum, S., et al. ATP hydrolysis by initiation factor 4A is required for translation initiation in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences. 89 (16), 7664-7668 (1992).
- Merrick, W. C. eIF4F: A Retrospective. Journal of Biological Chemistry. 290 (40), 24091-24099 (2015).
- Rogers, G. W., Komar, A. A., Merrick, W. C. eIF4A: The godfather of the DEAD box helicases. Progress in Nucleic Acid Research and Molecular Biology. 72, 307-331 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon