Method Article
Correção rápida in Vivo e isolamento de complexos translacionais de células eucarióticas
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Apresentamos uma técnica para estabilizar rapidamente complexos translacionais (biossíntese de proteínas) com formaldeído interligando em leveduras vivas e células mamíferas. A abordagem permite dissecar intermediários transitórios e interações dinâmicas de RNA:protein. Os complexos interligados podem ser usados em vários aplicativos a jusante, como em métodos de perfil baseados em sequenciamento profundo, microscopia e espectrometria de massa.
Resumo
Respostas rápidas envolvendo rápida redistribuição do mensageiro(m)RNA e alterações da tradução de mRNA são pertinentes aos ajustes homeostáticos contínuos das células. Esses ajustes são fundamentais para a sobrevivência das células eucarióticas e o "controle de danos" durante os níveis flutuantes de nutrientes e salinidade, temperatura e vários estresses químicos e de radiação. Devido à natureza altamente dinâmica das respostas de nível RNA, e à instabilidade de muitos dos intermediários RNA:RNA e RNA:protein, obter um instantâneo significativo do estado rna citoplasmado só é possível com um número limitado de métodos. Experimentos do tipo de perfil ribossomo baseados em RNA e RNA estão entre as fontes de dados mais informativas para o controle da tradução. No entanto, a ausência de um RNA uniforme e estabilização intermediária de RNA:proteínas pode levar a diferentes vieses, particularmente nas vias de resposta celular em ritmo acelerado. Neste artigo, fornecemos um protocolo detalhado de fixação rápida aplicável às células eucarióticas de diferentes permeabilidade, para auxiliar na estabilização intermediária do RNA e RNA:proteína. Fornecemos ainda exemplos de isolamento dos complexos estabilizados de RNA:proteínas com base em sua co-sedimentação com frações ribossômicas e poli(ribo)somal. O material estabilizado separado pode ser posteriormente usado como parte de experimentos do tipo de perfil ribossomo, como na abordagem de sequenciamento do perfil do complexo de tradução (TCP-seq) e seus derivados. A versatilidade dos métodos estilo TCP-seq foi agora demonstrada pelas aplicações em uma variedade de organismos e tipos de células. Os complexos estabilizados também podem ser adicionalmente purificados e imageados usando microscopia eletrônica, separados em diferentes frações poli(ribo)somal e submetidos ao sequenciamento de RNA, devido à facilidade da reversão do crosslink. Portanto, métodos baseados na fixação de snap-chilling e formaldeído, seguidos pelo tipo de enriquecimento complexo de RNA ou outro tipo de RNA:protein complex, podem ser de particular interesse em investigar detalhes mais finos da dinâmica complexa rápida do RNA:protein complex em células vivas.
Introdução
Os organismos vivos estão sujeitos a mudanças dinâmicas intra e extracelulares ao longo de suas vidas, que requerem respostas rápidas para manter a homeostase e garantir a sobrevivência. Para permitir a adaptação ambiental, as células eucarióticas ajustam seu metabolismo através do controle da expressão genética. O controle da expressão genética pode ser exercido durante a transcrição e/ou tradução; com respostas translacionais geralmente ocorrendo mais rapidamente1,2,3,4. Por exemplo, as alterações translacionais geralmente surgem dentro de 1-30 minutos do início do estresse, enquanto alterações no nível de transcrição seguem horas após a exposição ao estresse3,4,5. Alterações na produção de tradução são obtidas mais rapidamente devido à persistente disponibilidade de moléculas de mensageiro (m)RNA no citoplasma. Por outro lado, no nível de transcrição, novas moléculas de mRNA devem ser sintetizadas, e em eucariotes, processadas e exportadas do núcleo, produzindo extensos atrasos no tempo de resposta2,4,6,7,8.
A resposta translacional aguda ao estresse é geralmente caracterizada por uma diminuição geral na produção de tradução, com a regulação seletiva das proteínas necessárias para a sobrevivência celular1,3,4,9. A diminuição da produção de proteínas é considerada crucial devido ao alto gasto energético do processo3,7. Para facilitar a inibição seletiva e a regulação, as respostas translacionais são atendidas por uma série de mecanismos regulatórios complexos. A regulação pode ser exercida em todas as fases de tradução: iniciação, alongamento, término da biossíntese de polipeptídeos e reciclagem ribossômica10,11,12,13, mas é exibido mais fortemente na fase de iniciação5,7,9,10,13. Durante a iniciação, a pequena subunidade ribossômica (SSU), auxiliada por fatores de iniciação eucariótica (eIFs), liga-se e escaneia a região não traduzida (UTR) de 5' de mRNA até que um códon inicial seja reconhecido2,5,6,8,11,12,13. Os mecanismos regulatórios geralmente visam os EIFs que afetam o anexo, a digitalização e o início do reconhecimento de codon. Por exemplo, o fator de iniciação eIF2, um fator de tradução essencial que auxilia no recrutamento de um iniciador Met-tRNAiMet para a SSU, é muitas vezes alvo em eucariotes sob condições de estresse4,6,11. Na levedura, a fosforilação desse fator pode ser induzida sob privação de nutrientes e estresse osmótico1,4,11,14,15, e em células de mamíferos, fome de aminoácidos, estresse órticulo endoplasmático (ER), estresse UV, infecção viral e níveis alterados de oxigênio podem desencadear essa resposta8,9,11. A rápida regulação da tradução específica de mRNA é evidente na resposta das células mamíferas à hipóxia, que exibe uma inibição global de tradução rápida e a regulação seletiva da biossíntese de fatores indutores de hipóxia (HIFs). HIFs são fatores de transcrição, que então provocam reprogramação celular a longo prazo no nível de transcrição de DNA8,9,16. Respostas semelhantes têm sido observadas na levedura sob estresse térmico, com rápida expressão translacional de Proteínas de Choque térmico (HSPs) seguidas por respostas atrasadas do nível de transcrição17,18. Além da privação de nutrientes e choque térmico, as respostas translacionais na levedura têm sido estudadas sob oxigênio variado8,19salinidade5, fosfato, enxofre20,21 e nitrogênio22,23 Níveis. Esta pesquisa tem implicações generalizadas para os usos industriais da levedura, como cozimento e fermentação24,25. As respostas translacionais também podem ser fundamentais para promover a compreensão de doenças como distúrbios neurodegenerativos e doenças cardíacas, que são caracterizadas por estresse intracelular, como o estresse oxidativo. No geral, as respostas translacionais são parte integrante do controle da expressão genética e facilitam a rápida adaptação a uma ampla gama de condições de estresse em organismos eucarióticos.
Para estudar respostas translacionais, são necessários métodos que forneçam instantâneos minimamente distorcidos do cenário de tradução. O perfil polimento é uma abordagem clássica utilizada no estudo da tradução através do mRNA, envolvendo a separação de frações de poli(ribo)somal de mRNA via ultracentrifugação através de gradientes de sacarose26,27. A abordagem pode ser usada para explorar níveis de tradução para mRNAs individuais (com os métodos de detecção, como transcrição reversa e reação em cadeia de polimerase, RT-PCR26), ou globalmente em conjunto com técnicas de alta throughput (microarray ou RNA-seq28,29). Uma abordagem mais evoluída é o perfil ribossomo, que permite o estudo de posições de ribossomos alongados ao longo de uma molécula de mRNA em escala de genoma, bem como a inferência da eficiência da tradução através do transcriptome e utilização dos principais e alternativos locais de partida30,31. O perfil ribossomo envolve o isolamento e sequenciamento de fragmentos de mRNA protegidos pela presença ribossômica sobre eles. O perfil ribossomo forneceu considerável visão da dinâmica de tradução em várias condições, incluindo estresse hipóxico, choque térmico e estresse oxidativo31,32. A técnica foi adaptada a vários tipos de material de origem, incluindo leveduras e células mamíferas.
Embora o perfil polimento e ribossomo tenham sido fundamentais na ampliação das capacidades de pesquisa na tradução, o processo de tradução inclui vários intermediários translacionais e complexos que são difíceis de capturar com esses métodos11,13. Uma limitação adicional decorre da falta de capacidade de estudar tipos de resposta rápida, uma vez que os complexos translacionais são estabilizados in vivo pela adição de inibidores específicos de tradução (antibióticos), levando a certos artefatos de distribuição ribossosome, ou ex vivo sobre a lise celular especificamente (antibióticos) ou não (íons de sal ou magnésio elevados), levando à privação dos intermediários de menor duração ou menos estáveis33, 34,35.
O formaldeído é amplamente utilizado para cruzar ácidos e proteínas nucleicas, como em estudos de imunoprecipitação de cromatina (ChIP) e imunoprecipitação transligada (CLIP). Seu pequeno tamanho e excelente permeabilidade celular permitem uma rápida ação in vivo 36. Com base no cruzamento rápido do formaldeído, a abordagem de perfil ribossomo foi estendida com o Sequenciamento de Perfil do Complexo de Tradução (TCP-seq)10,36,37,38,39,40. O TCP-seq, desenvolvido pela primeira vez em levedura, permite a captura de todos os intermediários de tradução, incluindo complexos SSU de digitalização ou pós-término e múltiplas configurações ribossômicas37,38,41,42. O método tem sido utilizado em diversos estudos10,38,39,41,42, alguns dos quais utilizam uma abordagem combinatória tanto dos inibidores de tradução quanto do crosslinking formaldeído para facilitar a prisão da tradução. Uma versão mais modificada da técnica, a seletiva TCP-seq39,foi recentemente empregada para incluir a imunopurificação dos complexos interligados, ampliando o escopo das aplicações TCP-seq. A natureza rápida, eficiente e reversível do crosslinking formaldeído torna essas abordagens adequadas para estudar interações complexas transitórias de mRNA:tradução, particularmente no contexto de caminhos de resposta em nível de tradução altamente dinâmicos.
Aqui detalhamos os processos de crosslinking in vivo formaldeído com o propósito de estabilização e isolamento complexos de tradução abrangentes. Fornecemos protocolos separados nuances para leveduras e células mamíferas(Figura 1). Delineamos ainda exemplos do uso subsequente do material estabilizado por crosslink(Figura 1),como para detecção de fatores proteicos co-purificados usando imunoblotting (mancha ocidental), purificação assistida por imuno-assistuo (ou 'imunoprecipitação'; IP) e enriquecimento de complexos translacionais contendo fatores específicos de interesse, microscopia eletrônica e sequenciamento de RNA.
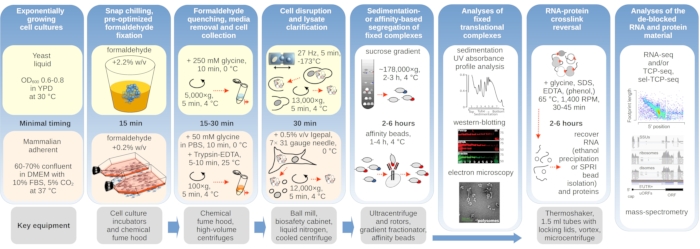
Figura 1: Esquema representando uma visão geral da configuração experimental típica. Os principais passos da estabilização in vivo formaldeído dos complexos translacionais são retratados como um fluxograma, complementado por informações sobre os principais instrumentos necessários. Potenciais aplicações a jusante do material interligado são delineadas, incluindo exemplos que foram empregados com sucesso, mas não diretamente cobertos neste protocolo, como a purificação de contas SPRI do RNA, sequenciamento de RNA e espectrometria em massa. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. Protocolo de célula de levedura
- Cultura e fixação celular de levedura
NOTA: A fixação e a colheita celular são adaptadas a partir de10,38com modificações.- Configuração de 1 L cultura celular de levedura (tipo selvagem (WT) BY4741 são dadas como exemplo) em um agitador orbital com a densidade óptica inicial de não mais do que 0,05 UA a 600 nm (OD600) em mídia adequada (1% w/v de extrato de levedura, 2% c/v de peptone, 2% w/v de dextrose (glicose), 40 mg/L de sulfato de adenina (YPD) usado como exemplo) nas condições desejadas (30 °C usado neste experimento).
- Configure uma centrífuga preparatória com rotor compatível e garrafas de centrífuga para a pelleting da cultura de suspensão líquida das células de levedura. Para experimentos de fome de glicose, pelota as células uma vez que a densidade óptica de 0,6-0,8 UA a 600 nm (OD600) é atingida, usando uma breve centrifugação a 30 °C, 5.000 x g por 1 min.
NOTA: Mantenha registro do OD das células em crescimento e deixe as células crescerem até que o OD600 atinja 0,6-0,8 UA, se a fase de crescimento exponencial for de interesse. - Resuspenque a pelota imediatamente em mídia YP quente (30 °C) contendo nenhuma ou baixa (0,25% c/v) adicionada glicose e incubar a cultura por mais 10 minutos a 30 °C em uma incubadora orbital.
NOTA: A composição da mídia pode afetar a eficiência subsequente de crosslinking. Este protocolo foi testado apenas usando YPD. Ao realizar experimentos de fome, aderir ao tempo e minimizar os atrasos entre os procedimentos é fundamental. - Uma vez que as células estejam prontas, configure uma caixa de gelo dentro do capô da fumaça com um béquer contendo 250 g de gelo de água limpa esmagada. Certifique-se de que as listras de 25 mL e a solução de formaldeído estabilizada de metanol recém-adquirida de 37% c/v sejam acessíveis dentro do capô. Despeje a cultura 1 L no béquer contendo 25% de c/v de gelo de água esmagada.
NOTA: Mantenha as células no gelo durante todas as operações subsequentes até que as células estejam congeladas, a menos que seja indicada o contrário. - Adicione 75 mL de 37% c/v de solução de formaldeído a uma concentração final de 2,2% c/v e mexa intensamente a mistura até que o gelo derreta.
- Uma vez que o gelo é derretido, configure um temporizador por 10 minutos.
NOTA: Adere aos tempos e regimes de temperatura recomendados para alcançar resultados de fixação reprodutíveis. - Depois de incubar por 10 minutos, transfira a cultura para as garrafas de centrífugas pré-transformadas e pelota as células por centrifugação a 4 °C, 5.000 x g por 5 min. Enquanto este giro está ligado, pré-basta um tubo de 50 mL e mantenha o tampão A recém-preparado (contendo glicina para neutralizar qualquer formaldeído restante) no gelo.
NOTA: Consulte a tabela fornecida para obter as composições exatas do buffer. - Após a centrifugação, coloque os tubos de centrífugas no gelo com o lado da pelota em contato com o gelo. Leve os tubos para dentro do capô da fumaça e descarte o sobrenatante em um recipiente de resíduos de formaldeído.
- Resuspenja a pelota celular de todos os tubos em 20 mL de tampão A usando uma stripette de 25 mL e transferindo para um tubo de 50 mL.
NOTA: Esta lavagem é fundamental para evitar a ligação cruzada irreprodutível e a adição do buffer não deve exceder 20 minutos de tempo da colheita das células. - Faça o volume até 40 mL com tampão A e colete as células lavadas por centrifugação a 4 °C, 5.000 x g por 5 min.
- Descarte o supernasciente e resuspenque a pelota de célula em 40 mL de tampão A1, que é tampão A não contendo glicina, para remover qualquer contaminação glicina.
- Células de pelota novamente por centrifugação a 4 °C, 5.000 x g por 5 min.
- Repita as lavagens com tampão A1 mais uma vez. Descarte o supernasce e coloque a pelota no gelo. Pesar o tubo com a pelota (massa celular molhada deve ser ~1 g por 1 L da cultura celular).
- Rompimento de célula de levedura e coleta de citoso
- Encha uma caixa de espuma de poliestireno forrada com papel alumínio com nitrogênio líquido a uma profundidade de aproximadamente 3 cm. Coloque um tubo de 50 mL na caixa.
- Resuspende a pelota (~1 g de massa celular molhada) em 550 μL de tampão A2 por pipetação e vórtice para 10 s. Adicione 10 μL de 40 U/μL de inibidor de RNase e vórtice novamente por 10 s.
ATENÇÃO: Use equipamentos de proteção adequados, como luvas termicamente isoladas, ao manusear nitrogênio líquido. Certifique-se de que qualquer recipiente usado para conter nitrogênio líquido não vaze, e que o rack de tubo dentro não flutuará para cima ou caia de lado. Trabalhe em uma área bem ventilada para evitar o esgotamento do oxigênio. - Usando uma pipeta de 1 mL, escorra a suspensão celular no tubo de 50 mL contendo o nitrogênio líquido.
NOTA: O gotejamento deve ser realizado de forma lenta e cuidadosa para evitar a agregação das gotículas. Certifique-se de que as gotículas congelem antes de introduzir novas gotículas. - Transfira o tubo de 50 mL com as gotículas de suspensão celular congelada para temperatura ambiente e espere até que o nitrogênio líquido evapore completamente. Sele o tubo com a tampa e armazene as pelotas da célula a -80 °C ou prossiga imediatamente.
ATENÇÃO: Certifique-se de que o nitrogênio líquido está completamente evaporado antes de selar o tubo. O nitrogênio líquido que sobrou em um tubo selado pode causar um acúmulo de pressão perigosa. - Para se preparar para o próximo passo, tubos pré-finos de 1,5 mL sem nuclease e potes de moagem de aço inoxidável de 10 mL em gelo seco.
- Transfira as gotículas de suspensão da célula congelada para os frascos usando uma espátula limpa e estéril.
ATENÇÃO: Certifique-se de que os frascos de moagem estão bem selados. - Submergir os frascos de moagem no nitrogênio líquido por 1 min, garantindo que a fase líquida permaneça abaixo da junção. Configure um moinho de mistura criogenia a 27 Hz para agitação por 1 min.
NOTA: Equilibre sempre o recipiente de moagem com outro do mesmo modelo, mesmo que a amostra exija apenas um recipiente para processamento. - Agitar os frascos de moagem selados a 27 Hz por 1 min no moinho de mistura.
- Resfrie os frascos de moagem em nitrogênio líquido como antes e agite a 27 Hz por 1 min a mais no moinho de mistura.
- Transfira os frascos para a caixa de gelo contendo gelo seco junto com os tubos sem nuclease de 1,5 mL. Usando uma espátula de aço pequena, transfira a amostra em pó resultante para os tubos em alíquotas de ~100 mg e armazene os tubos a -80 °C.
NOTA: Recomenda-se o uso de ~600 mg da amostra por experimento que compreende a análise do perfil de sedimentação polisso, a separação do citosol em frações traduzidas e não traduzidas e a separação adicional da fração traduzida em SSU, ribossomo e frações difusas na digestão RNase.
- Separação dos complexos fixos (poli)ribossômicos das frações não traduzidas do citosol
NOTA: O procedimento estabelecido anteriormente10,38é geralmente seguido para enriquecer RNA traduzido com base em sua co-sedimentação com (poli)ribossomos. Uma abordagem mais refinada para separar as frações de citoso traduzidas e não traduzidas é introduzida aqui, eliminando a necessidade de precipitar e, posteriormente, ressusolubilizar o material.- Prepare 2,5 mL linear 10%-20% c/v gradientes de sacarose com tampão B usando o método de degelo congelante43 em tubos de ultracentrifuge de parede fina (5 mL, 13 x 51 mm).
NOTA: O método de congelamento é realizado pela adição sequencial e congelamento de camadas de sacarose tamponadas com concentrações de regressão linear em cima umas das outras. Consulte a Tabela Suplementar 1 para obter detalhes. - Para criar uma almofada de sacarose de 50% w/v, sobre os gradientes lineares descongelando e estabilizando, dispense lentamente 0,5 mL de 50% de sacarose no tampão B diretamente na parte inferior dos tubos usando uma seringa de 1 mL presa a agulha de 19 G x 1,5" ou um capilar de vidro de dimensões semelhantes/adequadas. Antes de dispensar, dirija com cuidado e lentamente a ponta da agulha ou capilar do topo para baixo dos gradientes de sacarose pré-formados, evitando qualquer perturbação, até chegar ao fundo do tubo.
NOTA: Consulte a Tabela Suplementar 1 para obter instruções sobre a preparação do buffer B. - Equilibre cuidadosamente os gradientes removendo as porções superiores ou colocando mais 10 w/v de sacarose no buffer B e mantenha-os gelados ou a 4 °C.
NOTA: O gradiente descontínuo com a camada inferior de 50% de sacarose é necessário para coletar material com maior taxa de sedimentação sem precipitar-o na parede do tubo. - Descongele ~100 mg da amostra em pó de célula congelada à temperatura ambiente e coloque imediatamente no gelo. Misture em 150 μL de tampão A2 por pipetação, adicione o inibidor de RNase a 1 U/μL e misture por vórtice (evite espuma excessiva e mistura com a fase gasosa) por 10 s.
NOTA: Continue todas as operações mantendo o material no gelo, salvo indicação em contrário. - Pelota os detritos celulares centrifugando os tubos a 4 °C, 13.000 x g por 5 min e recupere o sobrenante esclarecido (~150 μL) em um novo tubo de ligação de baixa proteína de 1,5 mL.
- Carregue a mistura clarificada resultante nos tubos de gradiente de sacarose descontínuos a partir da etapa 1.3.3 e equilibre-os cuidadosamente.
- Ultracentrificar os tubos em um rotor de balde de oscilação de volume médio a 4 °C, com força G média 287.980 x g (k-factor 49) por 1 h 30 min.
NOTA: Estas condições foram pré-otimizadas (usando análise de traço de absorção UV de gradiente de gradiente pós-ultracentrifugação) para reter as SSUs e LSUs (não-(poli)ribossômicas) e LSUs (riboso grande subunidademal) na parte superior (10%-20% sacarose) da porção do gradiente, concentrando a (poli)fração ribossômica na almofada de sacarose inferior (50%) sem pelotas do material. - Use uma nova seringa estéril de 1 mL equipada com uma agulha de 19 G x 1,5" para coletar a fração de citosol traduzido. Coloque o gradiente de 5 mL em um rack estável, garantindo que a parte inferior do tubo esteja visível.
- Do topo do tubo, coloque a agulha diretamente na parte inferior do gradiente (sem perfurar o tubo) e, suavemente, sem criar bolhas, retire exatamente 0,5 mL da solução inferior contendo a piscina de RNA traduzida.
NOTA: Certifique-se de que esta etapa é realizada em uma sala fria e o tubo é mantido firmemente. Recomenda-se desenhar todo o 0,5 mL em um único movimento de upstroke para evitar perturbação do gradiente. - Confirme a presença (poli)ribossômica e o esgotamento das frações SSU, LSU e mais leves na mistura resultante pela leitura de absorvência do gradiente de sacarose após a execução da ultracentrifugação.
- Concentre o pool de RNA traduzido coletado da etapa anterior para 100 μL usando ultrafiltração em um dispositivo de micro-concentração com membrana de celulose regenerada de 10 kDa.
NOTA: Pré-lave a membrana do dispositivo de micro-concentração com 0,5 mL de tampão 1 (ver Figura 2a) e use condições de giro(g)recomendadas pelo fabricante. - Diluir ainda mais o material da etapa anterior cinco vezes (adicionar 400 μL) com tampão 1 e concentrar-se de volta a 200 μL, para permitir um volume menor, bem como a remoção parcial da sacarose.
NOTA: Recomenda-se armazenar as misturas resultantes a -80 °C por até 6 meses e usar como material de entrada para a construção da biblioteca RNA-seq 'total traduzida' ou a etapa de digestão RNase da construção da biblioteca TCP-seq. A fração de citosol 'não traduzida' pode ser recuperada da parte superior do gradiente usando um procedimento semelhante e armazenada a -80 °C.
- Prepare 2,5 mL linear 10%-20% c/v gradientes de sacarose com tampão B usando o método de degelo congelante43 em tubos de ultracentrifuge de parede fina (5 mL, 13 x 51 mm).
- Digestão RNase dos complexos fixos (poli)ribossômicos e separação do material digerido em pequenas frações ribossômicas (SSU), monoribosomal (ribossomos, RS) e diribosomal (disós, DS)
NOTA: O procedimento geralmente segue uma abordagem descrita anteriormente10,38mas um tipo de gradiente modificado, tempo de separação, aceleração e condições de digestão RNase são empregados, para obter a melhor resolução em todas as três frações isoladas.- Prepare cuidadosamente equilibrados 12,5 mL linear 10%-40% c/v gradientes de sacarose feitos com tampão 1 em tubos de polipropileno de parede fina de 13 mL, 14 x 89 mm, usando o método de degelo congelante43 conforme descrito na etapa 1.3.1 e note-o.
- Descongele à temperatura ambiente e transfira imediatamente as amostras no gelo ou pegue a fração de citosol traduzida concentrada e esgotada por sacarose a partir da etapa 1.3.12.
NOTA: Continue todos os procedimentos no gelo, a menos que seja indicado em contrário. - Digerir a fração de citosol traduzido misturando-se em 4,5 U de E. coli RNase I por 1 OD260 unidade da fração por 30 min a 23 °C. Adicione e misture imediatamente ao pipetar o inibidor RNase capaz de inativar RNase I a 0,25 U/μL à mistura, para inativar RNase I.
NOTA: Use inibidor RNase capaz de inibir RNase I. DerivaR AU260 usando AU260 = (Absorvância a 260 nm padronizado a unidades de densidade óptica equivalente a 1 cm de caminho óptico x volume do lysate em μL) / 1.000. - Transfira imediatamente as amostras para o gelo.
ATENÇÃO: É fundamental aderir às condições recomendadas de digestão e medir cuidadosamente a quantidade do RNase I adicionado. A unidade RNase I referida aqui é definida como a quantidade da enzima necessária para produzir 1 μg de material solúvel ácido do RNA do fígado de camundongo em 30 min a 37 °C. Os lotes RNase I podem ter variações não documentadas na atividade e podem exigir experimentação para alcançar condições ideais de digestão. Se o estoque de enzimas estiver muito concentrado, recomenda-se diluí-lo com tampão 1 para evitar a tubulação de volumes muito pequenos da solução. - Coloque as misturas de reação nos gradientes de sacarose de 10%-40% w/v a partir da etapa 1.4.1.
NOTA: Use volumes finais na faixa de 150-300 μL por gradiente. Cada purificação requer minimamente dois gradientes. Utilize diferentes volumes de entrada do material (menor AU260, 10-11 AU260, para DS e AU260, 13-14 AU260, para SSU ou RS) para alcançar a separação ideal. - Ultracentrificar os tubos em um rotor de balde de oscilação de volume médio a 4 °C com força G média 178.305 x g (fator k 143,9) por 3 h 30 min.
ATENÇÃO: Se forem necessários tubos de equilíbrio sobressalentes, equalize sua massa e distribuição de massa com os tubos contendo amostras. Use gradientes de sacarose sobrepostos com uma quantidade de tampão equivalente à da sobreposição amostral e não tubos com concentração uniforme de sacarose. - Configure um dispositivo fracionador de gradiente pelo menos 30 minutos antes da conclusão do giro de ultracentrifugação, incluindo o preenchimento da solução de perseguição pesada filtrada de 0,2 μm (por exemplo, 60% de sacarose em água desionizada como usada aqui) na bomba de deslocamento.
NOTA: Recomenda-se des contaminar as linhas e tubos do fracionado utilizando água deionizada, seguido de solução de 1%-2% de SDS em água deionizada, água deionizada e, finalmente, 80% de etanol em solução de água deionizada antes e depois das corridas. - Ajuste a linha de base de leitura de absorvência, primeiro preenchendo o sistema com água desionizada e zerando a óptica de acordo com as recomendações do fabricante e, em seguida, compensando o turno da linha de base usando um gradiente de sacarose de 14 x 89 mm feito com um tampão idêntico aos tubos de amostra (por exemplo, tampão 1).
NOTA: Use a mesma velocidade de deslocamento para fazer os ajustes quanto à leitura da amostra, como 1,5 mL/min. - Meça o volume morto do sistema de deslocamento contando com precisão o tempo entre a solução entrando primeiro no caminho óptico do detector e aparecendo pela primeira vez na saída do coletor de frações.
NOTA: Com a velocidade recomendada de 1,5 mL/min, o fracionamento pode ser realizado à temperatura ambiente. Recomenda-se transferir imediatamente as frações coletadas no gelo. - Realize o fracionamento utilizando leitura de absorvância ao vivo a 254 nm, velocidade de deslocamento de 1,5 mL/min e detecção de frações em linha com base na posição de sedimentação esperada e no perfil de absorção das amostras. Use a comutação do tubo coletor com um atraso de tempo correspondente ao volume morto, conforme medido anteriormente.
- Isole frações correspondentes às posições e mobilidade dos complexos SSU, RS e DS e colete-as em novos tubos de microcentrifuuge de baixa proteína de 1,5 mL; transfira imediatamente as frações isoladas no gelo e congele se não for processado imediatamente.
NOTA: Recomenda-se congelar imediatamente as frações coletadas em gelo seco ou nitrogênio líquido e armazenar a -80 °C ou abaixo por até 6 meses.
- Desvinculação dos complexos ribossômicos e isolamento do RNA para construir bibliotecas RNA-seq
- Para destrancar/reverter os crosslinks e isolar o RNA das proteínas associadas, transfira aproximadamente metade de todo o gradiente de sacarose em novos tubos de polipropileno de ligação de ácido nuclease baixo 1,5 mL (350 μL por tubo) com dispositivos de segurança/travamento da tampa.
- Suplemente as misturas com 40 μL de solução de parada de 100% (10% SDS w/v e 100 mM EDTA), 4 μL de 1 M Tris-HCl pH 2 a 2 5 °C (até 10 mM), 1,6 μL de 4,5 M de gliccina (até 10 mM) e água sem nuclease deionizada para obter o volume final de 400 μL.
- Misture o conteúdo dos tubos por pipetação e transfira os tubos à temperatura ambiente.
- Adicione o volume igual da mistura ácida fenol:clorofórmio:isoamyl álcool 125:24:1 (pH 4.0-5.0) mistura a cada tubo. Agite vigorosamente as misturas por 2 minutos usando uma batedeira de vórtice definida em velocidade máxima.
ATENÇÃO: O fenol e o clorofórmio são corrosivos e tóxicos. Evite o contato físico com os líquidos e trabalhe em uma área bem ventilada ou sob um capô de fumaça. Use sempre luvas, jaleco e óculos de proteção ou um escudo facial ao trabalhar com fenol ou clorofórmio. - Coloque os tubos em um termoshaker e agite continuamente a 65 °C, 1.400 rpm por 30 min.
- Facilite a agregação de fases centrifugando a mistura a 12.000 x g por 10 min a temperatura ambiente.
- Colete as fases aquosas superiores e transfira-as para tubos de ligação de ácido nucleico fresco de baixa ligação de 1,5 mL.
NOTA: Para evitar contaminação cruzada, não tente recuperar completamente as fases aquosas. Um volume de recuperação razoável é de 300-350 μL. - Suplemente as fases aquosas coletadas com 0,1 volumes de acetato de sódio de 3 M (pH 5 a 25 °C), 20 μg de glicogênio (utilizando 5 μg/μL de estoque) e 2,5 volumes de etanol absoluto. Misture cuidadosamente as soluções vórtices dos tubos por 1 min.
- Precipitar o RNA incubando as amostras a -20 °C por pelo menos 2 h (recomendado durante a noite).
- Aqueça os tubos à temperatura ambiente e misture por vórtice.
NOTA: O pré-aquecimento dos tubos e a centrifugação subsequente à temperatura ambiente (sem refrigeração forçada) ajudam a reduzir a co-precipitação e a transferência de sal e fenol. Essas condições não devem resultar em perda material ou ineficiência da coleta de RNA se realizadas conforme descrito e utilizando etanol suficientemente puro. - Pelota o RNA precipitar-se centrifugando os tubos a 12.000 x g por 30 min à temperatura ambiente.
- Descarte o supernasce e lave a pelota duas vezes com 80% de v/v de etanol, coletando-a cada vez por centrifugação a 12.000 x g por 10 minutos em temperatura ambiente.
- Seque as pelotas de RNA abrindo as tampas do tubo e colocando os tubos abertos em um aquecedor de bloco seco definido a 45 °C por 10 minutos. Dissolva a pelota seca resultante em 20 μL de tampão HE 1x.
- Estime a concentração de RNA resultante usando a medição do espectro de absorvência UV.
NOTA: O comprimento do fragmento de RNA e a quantidade total podem ser avaliados ainda mais usando gel-eletroforese desnaturing, como em um aparelho automatizado de eletroforese de gel capilar baseado em fluorescência.
- Co-imunopurificação seletiva das SSUs pelos EIFs marcados e análise de manchas ocidentais do enriquecimento seletivo da SSU
NOTA: Use ~15 UA (260 nm) da fração SSU digerida e segregada por sedimentação a partir da etapa 1.4.11 para realizar a purificação de afinidade usando contas magnéticas de IgG. Economize ~5% da fração SSU como controle de entrada (Fração de entrada, I). etiquetado eIF4A (TIF1-TAP; Tandem Affinity Purification tag) cepa de leveduras que também permite detectar eIF4A sondando para a tag TAP usando anticorpo anti-TAP.- Transfira 100 μL de suspensão de contas magnéticas IgG (1 mg das contas foram utilizadas para cada 15 UA (260 nm) do liseto ou fração) em um novo tubo de ligação de baixa proteína de 1,5 mL; coletar as contas usando rack magnético e aspirar-as.
- Lave as contas magnéticas duas vezes com 1 mL de tampão 1 usando ressuspensão sequencial por pipetação e coleta usando o rack magnético.
- Depois de lavar, coletar e decantar as contas, mantendo-as no rack magnético.
- Adicione a fração SSU às contas lavadas e incubar a mistura por 4h com rotação a 4 °C em um conjunto de ciclomixer a ~20 rpm.
- Colete as contas usando o rack magnético a 4 °C e salve o supernaspe (Fração flow-through, FT).
- Lave as contas duas vezes a 4 °C com tampão 1 complementado com 4 mM DTT, cada vez girando por 10 minutos no ciclomixer e coletando e decantando as contas no rack magnético. Guarde as lavagens (frações W1 e W2).
- Para uma aplicação analítica, como a mancha ocidental, elute o material vinculado sob desnaturação e condições de redução adicionando LDS (sulfato de lítio dodecila) tampão de amostra de gel de poliacrilamida (PAGE) com pH 8,5 a 1x e DTT a 2 mM.
- Aqueça a mistura a 95 °C por 5 min em um bloco térmico para finalizar a eluição.
- Colete as contas usando o rack magnético e recupere o eluato desnaturado (fração E) em um tubo de microcentrifuuge de baixa proteína fresco de 1,5 mL.
- Use a fração E da etapa anterior para executar imediatamente uma PÁGINA de sulfato de dodecyl de sódio desnaturação (SDS) ou armazene a fração E a -20 °C.
NOTA: Para uma coleção preparatória dos complexos translacionais enriquecidos com etiquetas TAP para qualquer aplicação subsequente, use uma abordagem alternativa de elução empregando protease Tobacco Etch Virus (TEV). Consulte a Tabela Suplementar 1 para mais detalhes. - Para concentrar as frações ft diluídas, W1 e W2, precipitar seu material adicionando volumes de 3x de acetona gelada. Incubar a mistura amostra-acetona a -20 °C por 3 h.
- Pellte o precipitado centrifugando os tubos a 13.000 x g por 10 min a 4 °C.
- Descarte o sobrenaspeente e seque a pelota nos tubos abertos à temperatura ambiente por 30 minutos.
- Dissolva a pelota em 7 μL de 1x LDS tampão de carga complementado com 2 mM DTT. Aqueça as amostras em um bloco térmico definido a 95 °C por 5 minutos.
- Carregue todas as amostras de I, FT, W1, W2 e E em um gradiente de acrilamida de 4%-12% w/v de acrilamida, gel de denaturamento de poliacrilamida Bis-Tris. Execute o gel usando 1x MES SDS (2- [N-mopholino]ácido esulfônico, sulfato de dodecila de sódio) em 80 V, até que o marcador de proteína (10-250 kDa) se resolva bem e o corante de chumbo atinja a parte inferior do gel.
NOTA: Recomenda-se carregar diluições seriais do WCL (lysate de células inteiras) (2-10 μg) no gel como controle. Pode levar várias tentativas para obter o carregamento comparável do gel através do material fracionado. - Transfira o teor proteico do gel para uma membrana de difluoreto de polivinida (PVDF) por método de transferência molhada a 100 V por 1h em uma sala fria, conforme recomendado pelo fabricante de equipamentos de mancha ocidental.
- Bloqueie a membrana usando um tampão de bloqueio apropriado (à base de salina tampão fosfato) à temperatura ambiente por 1h sob agitação constante.
- Seguindo as instruções do fabricante para diluição de anticorpos, teste a membrana com anticorpo anti-TAP para detectar a proteína eIF4A marcada, anticorpo anti-Pab1p ou anticorpo anti-β-actin (ou qualquer outro alvo desejável) por incubação durante a noite da membrana com tampão de bloqueio (PBS) anticorpo diluído (1:1.000 diluição) em um ciclomíbrio em uma sala fria.
NOTA: 1:1.000 diluição de anticorpos é um bom ponto de partida. - Lave a membrana três vezes com 1x Salina Tamponada fosfato, 0,2% v/v Tween 20 (PBST) por 10 min cada.
- Teste a membrana com anticorpos secundários fluorescentes rotulados seguindo as instruções do fabricante incubando em um ciclomixer à temperatura ambiente por 1h.
NOTA: 1:20.000 diluição de anticorpos é um bom ponto de partida. - Lave a membrana três vezes com 1x PBST por 10 min cada. Enxágue brevemente a membrana com água deionizada e, em seguida, com metanol absoluto. Seque e visualize a membrana em um sistema de imagem fluorescente de acordo com as instruções do fabricante.
NOTA: A coloração para outras proteínas pode ser obtida usando anticorpos secundários com corantes que correspondem a diferentes canais fluorescentes (como no eIF4A-TAP vs. β-actin par usado aqui), por coloração sequencial ou descascamento e coloração da mesma membrana ou corte da membrana de um gel carregado com repetição de padrão de frações e sondando separadamente cada peça com respectivos anticorpos (como no exemplo Pab1p usado aqui).
2. Protocolo celular de mamíferos
- Cultura e fixação de células mamíferas
- Em 2 frascos T-175, cresça células HEK293 a 60%-70% de confluência no Meio Águia Modificada de Dulbecco e 10% v/v Soro Bovino Fetal a 37 °C e 5% v/v de dióxido de carbono.
NOTA: A mídia completa é feita adicionando 55 mL de FBS comerciais em um DMEM de 500 mL comprado comercialmente com alta glicose, contendo L-glutamina, vermelho fenol e bicarbonato de sódio, mas sem HEPES ou piruvato de sódio. As contagens de células por frasco T-175 a 70% de confluência devem estar na faixa de 1,7-2,0 x 107. - Pelo menos 3 h antes do tempo de fixação desejado, substitua a mídia dos frascos T-175 por precisamente 30 mL de mídia completa pré-aquecida e substitua os frascos em uma incubadora celular.
NOTA: Certifique-se de que a mídia fresca está espetada no lado oposto do frasco para a monocamada celular para evitar o descolamento celular. Tente conduzir a troca de mídia o mais rápido possível, introduzindo um mínimo de perturbação do equilíbrio de temperatura e gás. - Uma vez que a mídia celular tenha sido substituída, prepare buffers e produtos químicos necessários para fixação. Prepare o Phosphate Buffered-Saline (DPBS) da Dulbecco com 50 mM de glicato adicionando 10,2 mL de 2,5 M de glicerina a uma garrafa de 500 mL de DPBS e mistura.
- Prepare uma garrafa de DMEM suplementada com 10% de FBS como na etapa 2.1.1 para ser usada em condições não estéreis e uma alíquota de 100 mL de 0,25% Trypsin-EDTA. Fonte de uma garrafa adicional de DPBS comercial pré-formulada com cloreto de cálcio (CaCl2) e cloreto de magnésio (TML2).
NOTA: As soluções podem ser armazenadas a 4 °C por até 2 semanas. - Prepare uma caixa de gelo até a borda com gelo de água esmagada de tal forma que um frasco T-175 possa caber uniformemente em cima e manter no capô da fumaça junto com os tampões preparados, também no gelo.
NOTA: Devido às rápidas respostas da tradução para qualquer mudança ambiental, todos os tempos entre a remoção dos frascos celulares da incubadora e a adição da solução de formaldeído devem ser minimizados. - Para esfriar as células, remova o frasco T-175 da incubadora e pressione-o firmemente contra o gelo, garantindo o contato máximo da superfície. Dentro da capa de fumaça química, incline o frasco para o lado para que a mídia se colete ao lado oposto das células. Pipeta 168 μL de 37% w/v formaldeído diretamente na mídia agrupada (para uma concentração final de 0,2% c/v). Misture imediatamente balançando suavemente o frasco para frente e para trás, feche e reposicione o frasco no gelo, garantindo que seja horizontal e as células estejam cobertas uniformemente.
ATENÇÃO: O formaldeído é uma substância prejudicial com potenciais efeitos adversos a longo prazo e também um irritante tanto para o sistema respiratório quanto para a pele. Só deve ser manuseado em um capô de fumaça química adequado. Os recipientes de formaldeído devem ser sempre selados quando fora do capô da fumaça.
NOTA: Certifique-se de que o formaldeído seja adicionado diretamente na mídia celular e não na parede do frasco. O passo 2.1.6 deve levar menos de 1 min. - Incubar os frascos no gelo por mais 10 minutos. Despeje a mídia em um recipiente de resíduos apropriado através do lado do frasco em frente às células.
- Usando uma stripette, pipeta em 30 mL de Salina Tamponada fosfato de Dulbecco sem íons de cálcio e magnésio e adicionalmente contendo 50 mM de glicina, suavemente ao lado oposto das células. Misture balançando o frasco; retornar o frasco para a posição horizontal e incubar por mais 10 minutos no gelo.
- Despeje a solução através do lado do frasco em frente às células e adicione suavemente 7 mL da solução padrão de 0,25% w/v Trypsin-EDTA para desapegar e resuspensar as células. Incubar o frasco à temperatura ambiente por 5-10 min.
NOTA: Certifique-se de que a solução Trypsin-EDTA cubra todas as células uniformemente. Use inclinação suave periódica e balanço para promover o descolamento celular. - Realoque o frasco verticalmente e usando uma stripette colete as células separadas lavando suavemente todas as células restantes das paredes do frasco. Transfira a suspensão para um tubo de 50 mL no gelo.
NOTA: As células fixas podem ficar mais frágeis; não pipeta intensamente ou mais do que o necessário para separar as células da parede do frasco. - Suplemente imediatamente a suspensão celular coletada com 20 mL de mídia completa (a mídia gelada não estéril com 10% de FBS) e misture girando suavemente o tubo.
NOTA: A mídia completa de cultura celular (incluindo 10% FBS) é adicionada para neutralizar a trippsina, evitando danos adicionais às membranas celulares e à desintegração celular. - Pelota as células centrifugando o tubo a 100 x g por 5 min e 4 °C. A pelota de célula deve ser claramente visível.
- Despeje a mídia e resuspenque suavemente a pelota de célula em 10 mL de DPBS gelado com Ca2+, Mg2+e sem glicina.
- Repita o passo 2.1.12.
- Despeje o tampão de lavagem e resuspenque suavemente a pelota da célula em 800 μL de DPBS gelado com Ca2+, Mg2+, sem glicina, no gelo. Transfira as células resuspended em um novo tubo de microcentrifuge de baixa proteína de 1,5 mL.
- Centrifugar o tubo a 100 x g por 3 min e 4 °C. Descarte cuidadosamente o supernatante usando um pipettor de 1 mL. Nesta fase, a pelota celular pode ser congelada a -80 °C ou seguir para a etapa de lise celular.
NOTA: As pelotas de células congeladas podem ser armazenadas a -80 °C até 1 ano. Descobrimos que o congelamento de pelotas de células facilita a lise subsequente e recomenda congelar mesmo que o armazenamento a longo prazo não seja planejado.
- Em 2 frascos T-175, cresça células HEK293 a 60%-70% de confluência no Meio Águia Modificada de Dulbecco e 10% v/v Soro Bovino Fetal a 37 °C e 5% v/v de dióxido de carbono.
- Rompimento de células de mamíferos e coleta de citos
- Em um gabinete de biossegurança, adicione 300 μL do tampão de lise à base de detergente noniônico, não desndenatura e 7 μL de 40 U/μL RNase inibidor. Misture bem com a pipetação usando uma ponta de 1 mL.
- Conecte cuidadosamente uma agulha de 25 G a uma seringa de 1-3 mL e enconte vigorosamente a mistura, usando pelo menos sete ingestão lenta para cima e traços rápidos de escape para baixo.
- Descarte a seringa e a agulha em uma caixa afiada e repita o procedimento usando uma seringa de 0,3 mL equipada com uma agulha de 31 G.
- Descarte a seringa e a agulha em uma caixa afiada. Centrifugar os tubos a 4 °C, 12.000 x g por 5 min para pelotar os detritos celulares.
- Transfira o supernatante para um novo tubo de microcentrifuge de baixa proteína de 1,5 mL. Armazene ambos, os detritos celulares (para fins de controle) e o lise celular esclarecido resultante a -80 °C.
NOTA: A densidade óptica do lysate varia entre 25-30 AU260 quando dois frascos T-175 são combinados e os volumes recomendados são seguidos. Os lises e detritos celulares podem ser armazenados a -80 °C até 1 ano.
- Separação dos complexos fixos (poli)ribossômicos das frações não traduzidas do citosol
- Prepare gradientes lineares de 15%-45% c/v de sacarose em tubos de polipropileno de parede fina de 13 mL, 14 x 89 mm, utilizando método de degelo congelante geralmente conforme descrito na etapa 1.3.1 do protocolo de levedura, mas utilizando tampão 2(Figura 2a).
NOTA: Descongele os gradientes durante a noite em uma sala fria a 4 °C na noite anterior ao fracionamento. - Carregar 150-250 (máxima de 300) μL da célula lysate da etapa anterior 2.2.5 para os gradientes equilibrados. Armazene o restante em -80 °C e use para fins de controle.
NOTA: Aqui é fornecido um exemplo de segregação baseada em sedimentação em frações SSU polissômicas, ribossômicas e "livres". Consulte a Tabela Suplementar 1 fornecida para uma abordagem alternativa. - Ultracentrificar os tubos em um rotor de balde de oscilação de volume médio a 4 °C, força G média 178.305 x g (fator k 143,9) por 1 h 45 min.
- 30 minutos antes da conclusão do giro, configuração e linha de base do fracionado gradiente, conforme descrito nas etapas do protocolo de levedura 1.4.7-1.4.9.
- Fracionar os gradientes geralmente conforme descrito nas etapas do protocolo de levedura 1.4.10-1.4.11.
NOTA: Esta etapa separará frações de SSU polissômicos, ribossômicos e 'livres'. Frações polissômicas podem ser usadas em experimentos de perfil de polisso. - Transfira imediatamente as frações coletadas no gelo e, se não for mais processada, armazene a -80°C até 6 meses.
NOTA: Se a troca do tubo coletor de frações estiver sincronizada com a identificação e segregação da fração on-line, recomendamos o uso de até 800 frações de μL (tempo de coleta de 32 s por fração a 1,5 mL/min). Se o fracionamento for realizado sem o uso da leitura de absorção em linha, é recomendado o uso de frações de 250-500 μL (10-20 s por fração a 1,5 mL/min). Após a separação, as frações podem ser usadas para imunopurificação, microscopia eletrônica, desnaturação PAGE e mancha ocidental imediatamente, ou submetidas à reversão de crosslink para análises subsequentes de RNA e/ou proteômica.
- Prepare gradientes lineares de 15%-45% c/v de sacarose em tubos de polipropileno de parede fina de 13 mL, 14 x 89 mm, utilizando método de degelo congelante geralmente conforme descrito na etapa 1.3.1 do protocolo de levedura, mas utilizando tampão 2(Figura 2a).
Resultados
Os complexos translacionais são sensíveis à composição iônica dos buffers, o que é particularmente importante durante a ultracentrifugação onde as propriedades de sedimentação são avaliadas. Testamos, assim, vários tampões de sedimentação utilizando lisetos esclarecidos extraídos de material de levedura não fixa terrestre, a fim de selecionar condições mais adequadas para resolver complexos translacionais e subunidades ribossômicas separadas (SSU, LSU), monossomos (RS) e polissomos em todo o gradiente. Todos os buffers foram baseados na composição do núcleo contendo 25 mM HEPES-KOH pH 7.6 e 2 mM DTT. As concentrações de KCl, MgCl2, CaCl2e EDTA foram ainda modificadas entre os buffers (Figura 2a), e esses componentes foram adicionados aos lises antes do carregamento gradiente e aos buffers gradientes de sacarose antes da fundição gradiente, em conformidade.
Nos buffers foram obtidos 1 e 2 complexos translacionais bem resolvidos. O buffer 1 resultou em uma separação um pouco melhor das pequenas subunidades ribossômicas (SSUs)(Figura 2a). A omissão de MgCl2 e a adição de EDTA (tampões 3,4) causaram perda das altas propriedades de sedimentação para a maioria dos polimentos e provavelmente sua desmontagem parcial(Figura 2a). Enquanto a adição de 2,5 mM CaCl2 resultou em picos polissômicos um pouco mais homogêneos, a melhoria foi marginal e a quantidade global do material polissomal diminuiu neste caso (Figura 2a) em comparação com os buffers 1 e 2. Assim, selecionamos o buffer 1 como o buffer de trabalho de escolha.
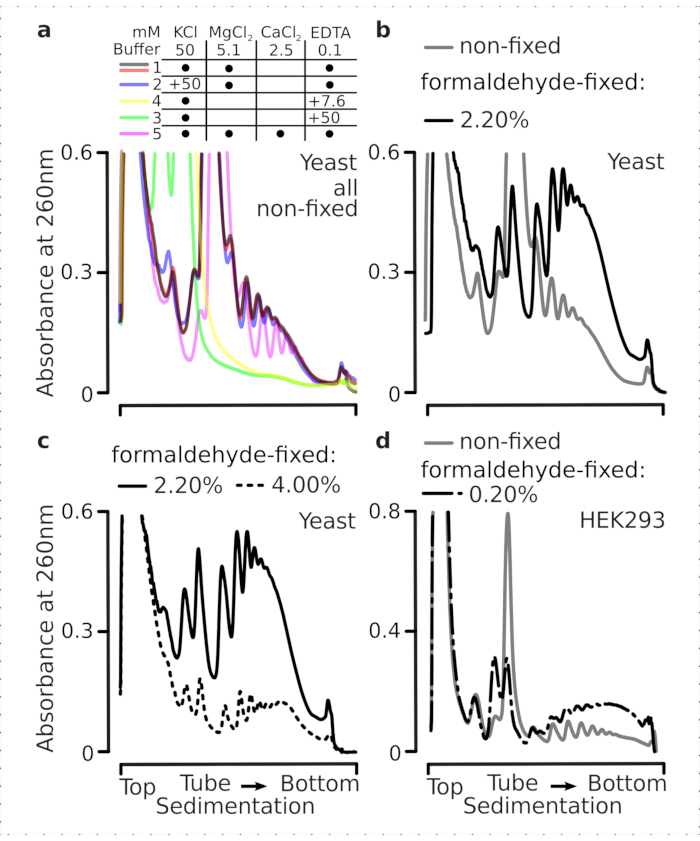
Figura 2: Condições tampão para extração e avaliação do efeito estabilizador da fixação. São mostrados perfis de absorvência UV coletados a 260 nm para o total de células de levedura separadas em gradientes de sucrose de 10%-40% w/v. (a) Efeitos de sais mono e divalentes e sequestro de íons de magnésio na sedimentação de material extraído de células de levedura não fixas. Linhas vermelhas e cinzas representam uma réplica típica. (b,c) Comparação de lises derivados de células de levedura não fixas (linha cinza), 2,2% (linha preta) e 4,4% (linha pontilhada preta) c/v de células de levedura fixas por formaldeído. (d) Estabilização de polísmos pelo otimizado 0,2% w/v de fixação de formaldeído (linha tracejada e pontilhada preta) das células HEK 293T, em comparação com o material de mesmas células não fixas (linha cinza). Clique aqui para ver uma versão maior desta figura.
Em seguida, verificamos o efeito da estabilização polissômica por fixação com diferentes concentrações de formaldeído. Utilizando o mesmo material celular, tampões, manuseio celular e abordagens de tempo, comparamos material extraído de células e células não fixas com 2,2% e 4% c/v de formaldeído(Figura 2b,c). Verificou-se que 2,2% c/v de formaldeído foi mais adequado para fixação, pois enquanto preservava excelentemente os polimentos como pode ser julgado pela relação polisome-monosome(Figura 2b),não reduziu o rendimento global do material ribossômico em comparação com 4% w/v de formaldeído, que apresentava sinais claros de super-fixação(Figura 2c).
Para o material derivado de células mamíferas, devido à maior razão de volume tampão-célula exigida pela extração baseada em detergente, foi utilizado buffer 2 (Figura 2a). Isso produziu complexos translacionais bem resolvidos após a sedimentação em gradientes de sacarose(Figura 2d). Notavelmente, foi utilizada uma concentração muito menor de formaldeído de 0,2% c/v, uma vez que concentrações mais elevadas resultaram em perda substancial de material polissomal e ribossômico (dados não apresentados). Em semelhança com os resultados obtidos com células de levedura, o material estabilizado por crosslink demonstrou melhor preservação dos polimentos e maior relação polisome-monosome(Figura 2d).
Em seguida, testamos se as condições selecionadas de fixação do formaldeído são eficientes o suficiente para estabilizar o mRNA traduzido ativamente dentro das frações polissômicas como resultado da ligação cruzada, e o rendimento polissomal melhorado não é apenas uma consequência da inibição da função enzimática e da progressão do alongamento da tradução. Usamos EDTA e sal monovalente alto (KCl) para desestabilizar polissomos e ribossomos. Estes reagentes foram adicionados aos lises de células de levedura clarificadas, e incluídos em todos os buffers subsequentes e gradientes de sacarose em cima da composição do buffer 1, respectivamente.
De fato, 15 mM EDTA apresentaram um efeito de desestabilização menor nas frações polissômicas derivadas das células fixas(Figura 3a),confirmando que os complexos transligados são mais robustos. Os efeitos desestabilizadores do EDTA podem ser um pouco superados pelo aumento da concentração de formaldeído, uma vez que o material a partir de 4% c/v de células fixas de formaldeído resistia a se desdobrar melhor(Figura 3a). No entanto, o aumento da concentração de EDTA para 50 mM resultou na desestabilização da maioria dos complexos translacionais em condições fixas e não fixas, como pode ser deduzido da sedimentação mais lenta do material e ausência de picos bem moldados(Figura 3b). Isso pode ser explicado pelo desdobramento parcial das estruturas e perda global de compactação, e não pela dissociação completa dos componentes polissômicos do mRNA. Mesmo neste caso, o material transligado demonstrou sedimentação mais rápida(Figura 3b).
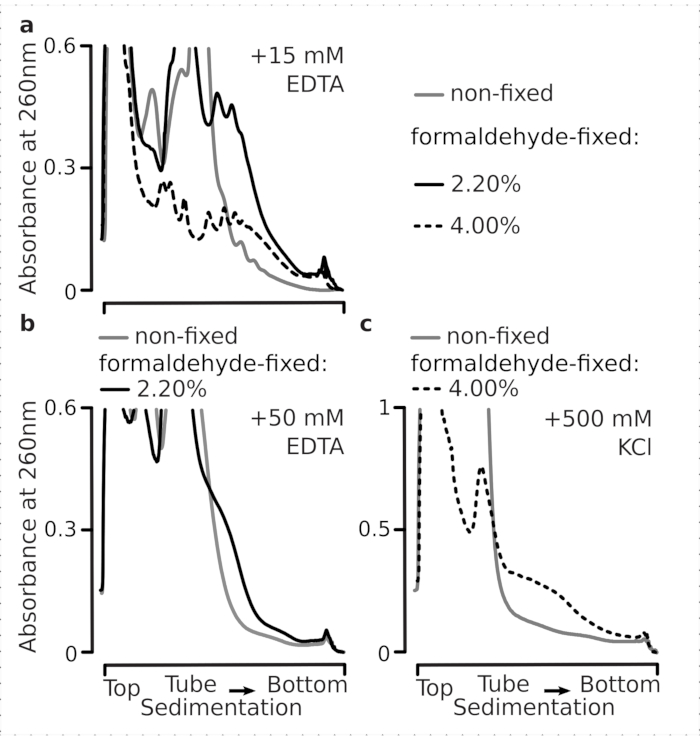
Figura 3: Efeitos da fixação de formaldeído de levedura in vivo na estabilidade dos polissomos. O buffer 1 (ver texto e Figura 2a) foi usado em todos os experimentos. Tipo de dados e plotagem conforme descrito na legenda da Figura 2. (a) Comparação da adição de 15 mM EDTA aos lises celulares e buffers subsequentes sobre a estabilidade dos polissóis derivados de células não fixas (linha cinza), 2,2% (linha preta) e 4% (linha pontilhada preta) c/v de células fixas de formaldeído. (b) mesmoque( a ), mas para a adição de 50 mM EDTA e excluindo 4% c/v de células fixas de formaldeído. (c) mesmoque( a ), mas para a adição de 500 mM KCl e excluindo 2,2% c/v de células fixas de formaldeído. Clique aqui para ver uma versão maior desta figura.
Semelhante aos efeitos EDTA, a 500 mM KCl, encontramos grande melhora da estabilidade com 4% c/v de fixação de formaldeído(Figura 3c). A aparente perda de compactação neste caso também pode ser explicada pelo desprendimento parcial dos constituintes dos complexos ribossômicos, em vez de sua completa dissociação do RNA. No geral, os polissomos derivados de células fixas de formaldeído demonstraram maior resistência ao desdobramento e à desestabilização estrutural, consistente com a formação de ligações covalentes adicionais dentro desses complexos.
Durante as condições de crescimento estimulantes, os mRNAs podem ser rapidamente iniciados resultando no acúmulo de múltiplos ribossomos nas mesmas moléculas de mRNA, que formam estruturas conhecidas como poliribosos, ou polissomos. Os polimentos podem ser separados por ultracentrifugação em gradientes de sacarose, onde sedimentam com base em sua ordem (número de ribossomos concomitantemente ligados ao mRNA). Quando a tradução é suprimida, os ribossomos não conseguem se envolver em outra rodada de tradução em breve, resultando em "desmontagem parcial) de polissomos, que é exibido como uma mudança modal em direção aos polissomos de uma ordem inferior e acúmulo de monossomos4,26.
Um modelo de resposta translacional que pode ser visualizado no nível de distribuição de pedidos polissários pode ser fornecido pela fome de glicose. O esgotamento da glicose provoca um dos efeitos inibitórios translacionais mais dramáticos e rápidos na levedura1,3,40. Estudos anteriores evidenciaram que dentro de 1 min de esgotamento da glicose, perda de polissomos, acúmulo de monossomos e inibição da iniciação da tradução podem ocorrer4. Dentro de 5 min de reprosso de glicose, a tradução é rapidamente restaurada com evidente aumento em polissomas3,4. Observou-se também que a tradução foi inibida quando as células foram expostas a meios contendo glicose de 0,5% (c/v) ou inferior e não houve efeito visto nos níveis de glicose de 0,6% (w/v) ou superior.
Por isso, queríamos determinar se nossas condições de fixação são adequadas para a preservação das diferenças translacionais dentro da dinâmica da resposta ao estresse glicário, como pode ser avaliado pela razão polisome-monosome. Comparamos o material das células cultivadas em fase exponencial média em alta glicose (2,00% c/v adicionado) com aqueles transferidos por 10 minutos para mídia sem ou baixo adicionado (0,00% ou 0,25% w/v, respectivamente) glicose. A fixação foi realizada utilizando 2,2% c/v de formaldeído em paralelo no controle (não-fome; substituição rápida de mídia com a mesma mídia padrão contendo 2% de glicose adicionada w/v, seguida de incubação por 10 min e fixação) e 10 min de fome (substituição rápida de mídia com a mesma mídia, mas baixa de 0,25 w/v ou sem glicose adicionada, seguido de incubação para células de 10 min e fixação).
Consistente com os achados anteriores, observamos que as células de levedura suprimem fortemente a tradução após o estresse da fome de glicose(Figura 4a). Ambas, nenhuma condição de glicose adicionada e baixa induziu a desmontagem polissós, com um pouco, mas evidentemente, mais polissomos retidos no caso de baixa glicose adicionada. Assim, a resposta de remoção de glicose de levedura pode não ser de um tipo all-on ou all-off e é gradualmente sintonizada. As expectativas de estabilização do material polissalo e polissomo do formaldeído das células fixas demonstraram uma maior distinção entre as células famintas e não famintas, preservando, sem dúvida, uma maior amplitude dinâmica da resposta(Figura 4b). Curiosamente, no caso do material das células fixas, a baixa concentração de glicose adicionada resultou na abundância polissômica específica que é muito melhor diferenciada da condição de glicose não adicionada, em comparação com as células não fixas(Figura 4a). Trata-se de uma forte indicação da adequação da abordagem de fixação do formaldeído na preservação e captura de diferenças relativamente minuciosas e transitórias no equilíbrio de processos altamente dinâmicos, como durante as respostas translacionais.

Figura 4: Captura de rápidas mudanças na tradução de leveduras após a fome de glicose. O buffer 1 (ver texto e Figura 2a) foi usado em todos os experimentos. Tipo de dados e plotagem conforme descrito na legenda da Figura 2. (a) Lises celulares obtidas de células não famintas (linha cinza), células de levedura restritas (0,25% w/v adicionadas por 10 min; linha marrom) e glicose esgotada (sem adição de glicose por 10 minutos; linha vermelha) células de levedura não fixas. (b) mesmo que (a), mas para células fixas de formaldeído de 2,2% w/v. Clique aqui para ver uma versão maior desta figura.
Monitorar o status translacional pelos ribossomos associados à tradução ativa do mRNA usando sedimentação gradiente de sacarose ('perfil de polimento') é uma técnica amplamente aplicada26,27,28. Em combinação com a análise quantitativa de microarray e, mais recentemente, com o sequenciamento de alto rendimento28,44, o perfil de polissóbico fornece informações sobre mRNAs associados ao ribossomo em toda a transcrição. Com várias suposições, tem sido tradicionalmente argumentado no campo da pesquisa de biossíntese proteica que a presença polissômica é uma indicação de envolvimento ativo na tradução dos respectivos mRNAs. Uma conclusão adicional é muitas vezes (mas nem sempre) justificada, que quanto mais ribossomos estão presentes em um mRNA de um determinado comprimento (quanto maior a ordem dos polísmos), mais ativamente que o mRNA está envolvido na tradução. Assim, separar a fração polissômica do resto do material pode ser útil do ponto de vista de isolar o RNA ativamente traduzido. Dentro da pegada, o perfil se aproxima, e particularmente o TCP-seq10,38,39 que gera uma população separada das SSUs liberadas derivadas dos complexos de digitalização, início e parada de códon, pode ser adicionalmente perspicaz remover subunidades ribossômicas que não co-sedimentam com os monossómos completos ou polissóis.
Por isso, empregamos a separação dos mRNPs 'não traduzidos', como SSUs gratuitos (mRNA vinculado a SSU ou SSUs únicos sem mRNA anexado) longe do pool de mRNAs "ativamente traduzidas". Para isso, assumimos que os mRNAs envolvidos em interações com um (mono-) ou vários ribossomos (polissomos) podem ser ativamente traduzidos. Tais complexos podem ser separados dos outros por seu coeficiente de sedimentação mais elevado. Sugerimos também separar a piscina "ativamente traduzida" de mRNAs em uma almofada de sacarose (50% c/v de sacarose) em vez de pelotização direta do material na parede do tubo. A centrifugação dos complexos de sedimentação rápida na almofada nos permitiu monitorar a separação usando leitura de perfil de absorvência e alcançar uma maior produção do material solubilizado, não agregado e não desnaturado, em comparação com a pelotização e re-solubilização10,38.
No geral, para purificar as SSUs individuais, ribossomos, disacrosos e polissomos potencialmente compactamente embalados de uma ordem superior, os lysates esclarecidos fixos foram submetidos a um processo de ultracentrifugação em duas etapas(Figura 5). No primeiro gradiente de sacarose, a ultracentrifugação resultou em SSUs e LSUs livres separados na parte superior (10%-20% w/v de sacarose) do gradiente, enquanto a piscina traduzida cruzada, incluindo polisomos e mRNAs associados a um ribossomo completo, estavam concentrados na parte inferior (50% w/v de sacarose) do gradiente (Figura 5a ). A parte inferior de 50% c/v de camada de sacarose contendo a piscina mRNA traduzida foi então concentrada e seu RNA digerido com RNase I, seguido por uma segunda ultracentrifugação gradiente de sacarose para obter SSU separado, LSU, RS, dise resistente à RNase (DS) e pequena fração de polimentos resistentes a nuclease de alta ordem(Figura 5b). A coloração negativa com acetato de urânio e imagem com microscópio eletrônico de transmissão confirmou a identidade dos complexos isolados em cada estágio de sedimentação(Figura 5).

Figura 5: Isolamento do total traduzido frações de RNA longe do RNA não traduzido. (a,c) Esquemático (esquerda) e os respectivos resultados representativos (à direita; tipo de dados e plotagem conforme descrito na legenda da Figura 2) da (a)primeira separação de gradiente de sacarose descontínua das frações de citosol não traduzidas, incluindo SSUs livres e o pool de mRNA traduzido identificado por co-sedimentação com ribossomos e polissomos, e (c)separação dos complexos ribossômicos individuais liberados da piscina de mRNA traduzida pela digestão e ultracentrização RNase I controladas através de um segundo gradiente linear de sacarose em frações SSU, LSU, ribossômicas (RS) e resistentes a nuclease (DS). Altas (15UA 260) e baixas (8AU 260) quantidades do material digerido não faminto foram incluídas para demonstrar a possibilidade de aumentar as cargas de ultracentrifugação quando pequenas frações são de interesse. Polissomos resistentes a nuclease de ordem superior também podem ser identificados (por exemplo, trissóis nos exemplos fornecidos). (b,d) Imagens tem representativas de frações contrastadas com acetatos de urano de (a,c), respectivamente como rotulado. Clique aqui para ver uma versão maior desta figura.
A fim de verificar a adequação do regime de fixação para a retenção de proteínas transitórias ribossômicas associadas (particularmente, eIFs), testamos para a co-sedimentação do eIF4A, um eIF labile dinamicamente ligado ao ribossomo, através das frações ribossômicas. Aproveitamos a cepa de levedura marcada eIF4A Tandem Affinity (TAP) (TIF1-TAP) e investigamos a presença eIF4A em material derivado das células fixas vs. não fixas usando anticorpo anti-TAP, em comparação com a abundância de Pab1p como um controle adicional de ligação de RNA, usando SDS-PAGE seguido de mancha ocidental(Figura 6).
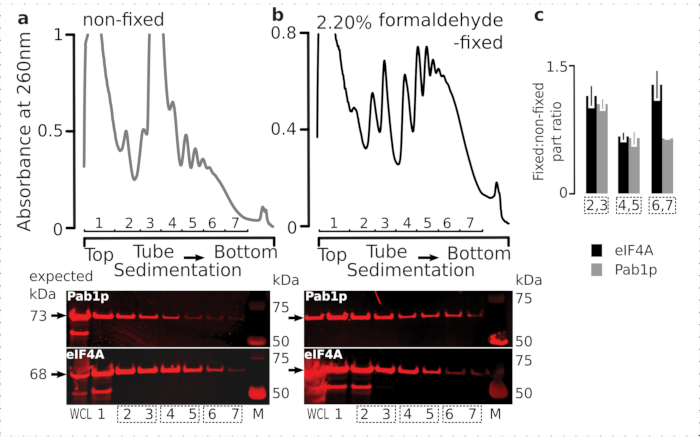
Figura 6: Estabilização de proteínas transitórias nos complexos translacionais após fixação in vivo formaldeído. (a,b)(parcelas superiores) Liseto celular inteiro (WCL) de (a)células não fixas e (b)2,2% células de leveduras eIF4A-TAP corrigidas por ultracentrifugação e visualizadas conforme descrito na legenda da Figura 2. (parcelas inferiores) Imagem ocidental das respectivas frações de gradiente de sacarose após a separação do material analisado nos gradientes correspondentes (parcelas superiores) e WCL como controle. (c) Razão média entre a abundância eIF4A ou Pab1p nas frações de material fixo e não fixo. As proporções relativas (normalizadas ao sinal de todas as 2-7 frações) de eIF4A (barras pretas) e Pab1p (barras cinzas) foram calculadas em 2,3 (SSU, LSU), 4,5 (RS, polimentos leves) e 6,7 (polimentos pesados) a partir dos dados de (a,b) (parcelas inferiores) e sua razão fixa para não fixa. As barras de erro indicam o desvio padrão da razão da média com as frações agrupadas (caixas pontilhadas) tratadas como réplicas. Clique aqui para ver uma versão maior desta figura.
Consistente com sua alta abundância nas células, observamos uma alta intensidade do sinal de ambas as proteínas em toda a célula lysate (LCA) e frações de sedimentação mais lenta derivadas de células não fixas(Figura 6a, painel inferior). Também detectamos quantidades substanciais dessas proteínas na LCA derivadas das células fixas e tranquilizando a eficiência da extração de material interligado e ausência de perdas inesperadas(Figura 6b, painel inferior). Entretanto, em contraste com as células não fixas, o material das células fixas demonstrou elevada presença relativa de eIF4A nas frações ribossômicas de sedimentação mais rápida, em comparação com Pab1p(Figura 6c). Este resultado sugere que o eIF4A permanece mais firmemente associado com os polissomos em material cruzado de formaldeído.
Tendo confirmado o efeito de estabilização positivo e específico da interligação na presença eIF4A nas frações ribossômicas, utilizamos o material fixo da linha de levedura eIF4A marcada (TIF1-TAP) para capturar e enriquecer complexos contendo eIF4A por purificação de afinidade com contas de IgG magnético. Temos frações de WCL enriquecidas por afinidade, SSU livre e polissomal (piscina mRNA traduzida) após a primeira sedimentação através de gradiente de sacarose (por exemplo, seção 1.3 do protocolo de levedura), bem como frações SSU, LSU e RS da segunda sedimentação após a desmontagem da piscina traduzida em complexos individuais com RNase I (por exemplo, seção 1.4 do protocolo de levedura) (Figura 7 ). Em todos os casos, com exceção da fração LSU, pudemos observar o enriquecimento seletivo do eIF4A nas frações purificadas (elunato, E), em comparação com a presença de β-actin no material de origem (entrada, I) (Figura 7).
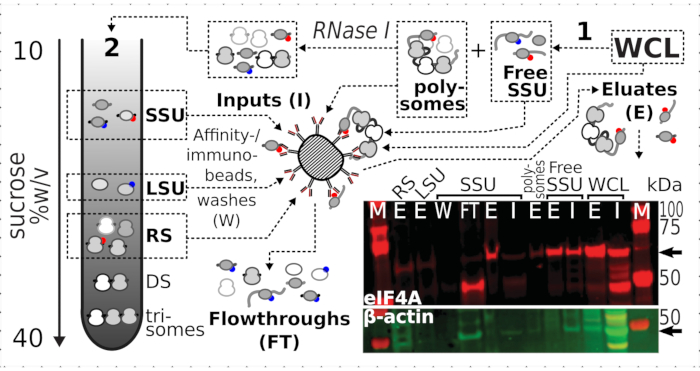
Figura 7: Imunopurificação seletiva de complexos translacionais estabilizados in vivo formaldeído por eIF4A associado transitoriamente. O esquema ilustra a fonte de diferentes complexos translacionais e epítope eIF4A, incluindo a WCL clarificada não fracionada das células de levedura eIF4A-TAP; SSUs gratuitos e piscina de RNA traduzida (polissomas) segregadas na primeira ultracentrifugação; Frações SSU, LSU e RS liberadas do RNA traduzido pela digestão RNase I e segregadas usando segunda ultracentrifugação (ver texto). A imagem da mancha ocidental fornece uma visualização da abundância eIF4A nas frações em comparação com a abundância de controle de β-actin simultaneamente manchado. Clique aqui para ver uma versão maior desta figura.
Tabela suplementar 1. Clique aqui para baixar esta Tabela.
Discussão
A fixação do formaldeído é um método conveniente e popular de alcançar o cruzamento rápido in vivo de biomoléculas10,36,45,46,47,48. Em comparação com os outros potenciais alvos de biomolécula, a captura bem sucedida de complexos translacionais requer uma fixação imediata durante o snap chilling das células ou outro material. Sem a estabilização inexperiente, há um potencial para que diferentes processos relacionados à tradução continuem, afastando a complexa distribuição do estado in vivo 49não perturbado . Em comparação com os outros métodos de apreensão translacional e estabilização do complexo ribossômico, a rapidez da ação formaldeída entre as membranas celulares e a natureza indiscriminada dos crosslinks prometem a preservação da diversidade máxima dos intermediários do complexo de tradução mais próximos de seus estados nativamente distribuídos50.
A abordagem aqui apresentada foi estabelecida e otimizada em células de levedura e mamíferos, e os métodos foram agora derivados por outros grupos para uso em materiais biológicos mais diversos, como em vertebrados inteiros (por exemplo, embriões de zebrafish)10,38,39,49,51,52 . Embora esses trabalhos tranquilizem coletivamente a versatilidade e a ampla aplicabilidade da abordagem, o cruzamento rápido de formaldeídos de complexos translacionais pode ser considerado um pouco difícil de transpor para novos tipos de material biológico devido à necessidade de otimizações e ajustes.
Um dos principais requisitos para o sucesso do método é a ree otimização da concentração do formaldeído e da técnica de coleta e ruptura celular. Células de leveduras menos permeáveis, pequenas e redondas requerem concentração de formaldeído muito maior (pelo menos, 10 vezes) e interrupção física das células fixas. Em contraste, grandes e achatadas células de mamíferos adeptos na cultura podem ser facilmente superfixadas e requerem manuseio suave após a fixação, enquanto a extração dos complexos fixos pode ser realizada quimicamente com a interrupção da membrana usando detergentes. A sub-ligação pode permitir que intermediários menos estáveis ou mais de curta duração se dissociem ou vazem para um estado posterior. O excesso de ligação pode afetar negativamente a capacidade de isolar e estudar frações ribossômicas e pode criar vieses seletivos, como o esgotamento mais profundo de complexos pesados. Em nossa observação, mesmo pequenas alterações, como o tipo de células humanas aderentes utilizadas, podem afetar o rendimento dos complexos cruzados recuperados e podem exigir a ree otimização do regime de crosslinking. Também podemos prever que células com propriedades de permeabilidade substancialmente diferentes, como células vegetais, exigirão otimização extensiva adicional das condições de fixação52. No entanto, é difícil imaginar um tipo de material biológico que seria totalmente incompatível com a abordagem.
Uma consideração pertinente ao protocolo de fixação de mamíferos é a densidade e quantidade de material celular usado como insumo. Recomenda-se que as células cresçam continuamente sem re-semeadura ou outras perturbações por pelo menos 2 dias para evitar influências externas na dinâmica de tradução celular. Aplicável para a maioria dos tipos de células, mas para a maioria das células aderentes consistentemente alcançou níveis de confluência de não mais de 70% garantirá a ausência de grandes efeitos de inibição de contato que podem afetar negativamente e imprevisívelmente as taxas de tradução.
Outra característica interessante, e potencialmente conveniente, da fixação do formaldeído decorrente de sua reatividade indiscriminada é o efeito de estabilização sobre complexos translacionais em sistemas de taxonomia mista. Os complexos bacterianos, e ainda mais translacionais de mitocôndrias, cloroplastos e diferentes parasitas intracelulares, têm sido notoriamente difíceis de atingir com inibidores específicos de tradução. Em contraste, nos dados TCP-seq, as pegadas mapeando o mitotranscriptome são prontamente observáveis nos dados38,39,50. Um desenvolvimento subsequente interessante poderia ser o uso da abordagem para investigar a tradução em microcommunidades inteiras, como no solo, água ou amostras de intestino, onde a prisão rápida e estabilização complexa confiável com qualquer outro meio seria problemática.
Deve-se mencionar também que para o material mais complicado (como tecidos duros e/ou volumosos), nada impede o uso de estabilização de formaldeído imediatamente após interrupção celular e homogeneização de materiais. Esta abordagem já é frequentemente empregada para remover o atraso de entrada celular ao estabilizar complexos translacionais com inibidores específicos de pequenas moléculas33,53,54,55. Dado que a fixação do formaldeído tem sido tradicionalmente utilizada com excelentes resultados para estabilização de amostras ex vivo/in vitro em aplicações como microscopia eletrônica45,56,57,58, podemos esperar efeitos ainda menos negativos neste caso, particularmente aqueles associados à má extração dos complexos translacionais das células completamente fixas.
Nossos achados confirmam a usabilidade da fixação rápida de formaldeído para estabilizar complexos altamente transitórios, como os que incluem o eIF4A. Vale ressaltar que, ao contrário dos mamíferos, a levedura eIF4A está muito mais fracamente associada com o complexo de ligação de tampa eIF4F e, como resultado, complexos translacionais em geral. eIF4A é geralmente perdido durante qualquer purificação extensiva do material ribossômico na levedura29,59,60,61,62,63. No entanto, no material de levedura in vivofixo, é possível obter enriquecimento confiável do eIF4A em todas as frações de complexos translacionais onde sua presença seria antecipada. Os dados sel-TCP-seq publicados anteriormente demonstraram o enriquecimento do eIF2 e eIF3 que associam mais fortemente com os ribossomos (mas também revelaram o conjunto complexo de proteína co-translacional que ocorre transitoriamente)39. Assim, o método é adequado para a detecção de ambos, constituintes mais fortes e mais fracos ligados dos complexos translacionais.
Resumindo, apresentamos uma abordagem útil para obter insights principalmente sobre as mudanças que ocorrem na fase de início da tradução e quando a distribuição ribossômica minimamente perturbada sobre o mRNA é necessária. É importante ressaltar que a abordagem é adequada para a estabilização de componentes relativamente labíteis e dinâmicos de complexos translacionais, como o eIF4A, e pode ser amplamente submetida às otimizações necessárias. Também fornecemos evidências da utilidade da fixação do formaldeído nos cenários de rápida mudança dinâmica de tradução, abrindo áreas de investigação, como respostas celulares rápidas a mudanças ambientais ou condições de estresse.
Divulgações
Os autores não declaram conflitos de interesse.
Agradecimentos
Este trabalho foi apoiado pela bolsa do Australian Research Council Discovery Project (DP18010111 a T.P. e N.E.S), National Health and Medical Research Council Investigator Grant (GNT1175388 para N.E.S.) e Research Fellowship (APP1135928 a T.P.). Os autores reconhecem as instalações da Microscopia Austrália no Centro de Microscopia Avançada, Universidade Nacional Australiana, uma instalação que é financiada pela Universidade e pelo Governo Federal.
Materiais
| Name | Company | Catalog Number | Comments |
| Yeast extract | Merck, Sigma-Aldrich | 70161 | |
| Peptone | Merck, Sigma-Aldrich | 70178 | |
| D-Glucose (Dextrose) | Merck, Sigma-Aldrich | 49139 | |
| Adenine sulphate | Amresco | 0607-50G | |
| Formaldehyde solution | Merck Sigma-Aldrich | F11635-500ML | ACS reagent, 37 wt. % in H2O, contains 10-15% Methanol as stabiliser (to prevent polymerisation) |
| RNaseOUT™ Recombinant Ribonuclease Inhibitor | Invitrogen™ byThermo Fischer Scientific | 10777019 | |
| cOmplete™, EDTA-free Protease Inhibitor Cocktail | COEDTAF-RO Roche by Merck | 11873580001 | |
| Magnesium chloride solution | (Merck/Sigma-Aldrich) | M1028 | |
| Ethylenediaminetetraacetic acid solution | (Merck/Sigma-Aldrich) | E7889 | |
| Ambion™ RNase I, cloned, 100 U/µL | Ambion | AM2294 | |
| SUPERase•In™ RNase Inhibitor (20 U/μL) | Invitrogen™ by Thermo Fisher Scientific | AM2694 | |
| Acidic phenol:chlorophorm:isoamyl alcohol 125:24:1 (pH 4.0-5.0) | (Merck/Sigma-Aldrich) | P1944-100ML | |
| Dynabeads™ Goat Anti-Mouse IgG | Invitrogen™ by Thermo Fisher Scientific) | 11033 | |
| Sodium Acetate (3 M), pH 5.5 | Invitrogen™ by Thermo Fisher Scientific) | AM9740 | |
| Glycogen (5 mg/ml) | Invitrogen™ by Thermo Fisher Scientific) | AM9510 | |
| Ethyl alcohol, Pure | Merck; Sigma Aldrich | E7023 | |
| Amersham™ Hybond® P Western blotting membranes, PVDF | Merck | GE10600023 | PVDF membrane for western blotting |
| Bolt™ 4 to 12%, Bis-Tris, 1.0 mm, Mini Protein Gel | Invitrogen™ by ThermoFischer Sientific | NW04120BOX | Protein gel |
| 4X Bolt™ LDS Sample Buffer | Invitrogen™ by ThermoFischer Sientific | B0007 | LDS sample loading buffer |
| Precision Plus Protein™ Kaleidoscope™ Prestained Protein Standards | BioRad | 1610375 | Protein ladder |
| 20X Bolt™ MES SDS Running Buffer | ThermoFischer Scientific | B0002 | PAGE runninjg buffer |
| Intercept® (PBS) Blocking Buffer | LI-COR | 927-70001 | Odyssey Blcoking buffer (PBS) |
| IRDye® 800CW Goat anti-Mouse IgG Secondary Antibody | LI-COR | 92632210 | |
| IRDye® 800CW Goat anti-Rabbit IgG Secondary Antibody | LI-COR | 92632211 | |
| TAP Tag Polyclonal Antibody | Invitrogen™ by ThermoFischer Sientific | CAB1001 | |
| Anti-beta Actin antibody | Abcam | ab8227 | |
| Sucrose | (Merck/Sigma-Aldrich) | 84097 | BioUltra, for molecular biology, ≥99.5% (HPLC) |
| DL-Dithiothreitol solution | (Merck/Sigma-Aldrich) | 43816 | BioUltra, for molecular biology, ~1 M in H2O |
| Terumo Syringe 1CC/mL | Terumo Syringe | 878499 | |
| Potassium chloride | (Merck/Sigma-Aldrich) | 60128 | |
| HEPES | (Merck/Sigma-Aldrich) | H3375 | |
| Dulbecco's Modified Eagle's Medium - high glucose | Sigma Aldrich | D5796 | |
| Fetal Bovine Serum | Sigma Aldrich | 12003C | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300062 | |
| Dulbecco's Phosphate Buffered Saline with Calcium and magnesium | Sigma-Aldrich | D8662 | |
| Glycine | Sigma-Aldrich | G7126 | |
| Tris hydrochloride | Merck/Sigma-Aldrich | 10812846001 | |
| Sodium dodecyl sulfate | Merck/Sigma-Aldrich | 436143 | |
| IGEPAL CA-630 | Merck/Sigma-Aldrich | I3021 | |
| Rnasin Ribonuclease Inhibitor | Promega | N2111 | |
| Stainless steel grinding jar | Retsch | 02.462.0059 | |
| MM400 mixer mill | Retsch | 20.745.0001 | |
| Gradient Fractionator | Brandel | BRN-BR-188 | |
| Thermomixer R | Eppendorf | Z605271 | |
| Nanodrop spectrophotometer | Thermo Fisher Scientific | ND-2000 | |
| 0.5-ml microcentrifuge tubes with locking devices | Eppendorf Safe-Lock | 30121023 | |
| Mini Gel Tank | (Thermo Fisher Scientific) | A25977 | PAGE running tank |
| 5 mL, Open-Top Thinwall Ultra-Clear Tube, 13 x 51mm | Beckman-Coulter | 344057 | |
| 13.2 mL, Certified Free Open-Top Thinwall Polypropylene, 14 x 89mm - 50Pk | Beckman-Coulter | 331372 | |
| Amicon Ultra-0.5 ultrafiltration devices | Merck | UFC5030 | Ultracel-30 regenerated cellulose membrane, 0.5 mL sample volume |
| Thermo Sorvall Evolution RC Floor Super Speed Centrifuge | Cambridge Scientific | 15566 | |
| Beckman Coulter Optima L-90K | GMI | 8043-30-1191 | |
| Nunc EasYFlask 175cm2 | Thermofisher Scientific | 159910 | |
| Falcon 50 mL Conical Centrifuge Tubes | Thermofisher Scientific | 14-432-22 | |
| 25 mL Serological Pipette | Sigma-Aldrich | SIAL1250 | |
| 10 mL Serological Pipette | Sigma-Aldrich | SIAL1100 | |
| DNA lobind tubes | Eppendorf | 30108051 | |
| Cold Centrifuge 5810 R | Eppendorf | EP022628188 | for 50 mL tubes |
| Orbital Shaking Incubator | Ratek | OM11 | |
| Frezco 17 Microcentrifuge | Thermofisher Scientific | 75002402 | |
| Eppendorf DNA lo-bind tubes | Merck/Sigma-Aldrich | EP0030108051 | |
| Eppendorf® Protein LoBind tubes | Merck/Sigma-Aldrich | EP0030108116 | |
| SW 41 Ti Swinging bucket rotor | Beckman-Coulter | 331362 | |
| Heracell™ 150i CO2 Incubator, 150 L | Thermofisher Scientific | 51026282 | |
| 0,3 mL ultra-fine II short insulin syringe | BD Medical | 328822 | |
| 3 mL syringe with Luer Lok tip | BD Medical | 302113 | |
| 25 G x 16 mm Hypodermic Needle | Terumo | TUAN2516R1 |
Referências
- Janapala, Y., Preiss, T., Shirokikh, N. E. Control of translation at the initiation phase during glucose starvation in yeast. International Journal of Molecular Sciences. 20 (16), 4043 (2019).
- Masvidal, L., Hulea, L., Furic, L., Topisirovic, I., Larsson, O. mTOR-sensitive translation: Cleared fog reveals more trees. RNA Biology. 14 (10), 1299-1305 (2017).
- Ashe, M. P., De Long, S. K., Sachs, A. B. Glucose depletion rapidly inhibits translation initiation in yeast. Molecular Biology of the Cell. 11 (3), 833-848 (2000).
- Crawford, R. A., Pavitt, G. D. Translational regulation in response to stress in Saccharomyces cerevisiae. Yeast. 36 (1), 5-21 (2019).
- Melamed, D., Pnueli, L., Arava, Y. Yeast translational response to high salinity: global analysis reveals regulation at multiple levels. RNA. 14 (7), 1337-1351 (2008).
- Hershey, J. W., Sonenberg, N., Mathews, M. B. Principles of translational control: An overview. Cold Spring Harbor Perspectives in Biology. 4 (12), 011528 (2012).
- Mata, J., Marguerat, S., Bähler, J. Post-transcriptional control of gene expression: a genome-wide perspective. Trends in Biochemical Sciences. 30 (9), 506-514 (2005).
- Spriggs, K. A., Bushell, M., Willis, A. E. Translational regulation of gene expression during conditions of cell stress. Molecular Cell. 40 (2), 228-237 (2010).
- Liu, B., Qian, S. B. Translational reprogramming in cellular stress response. Wiley Interdisciplinary Reviews RNA. 5 (3), 301-315 (2014).
- Archer, S. K., Shirokikh, N. E., Beilharz, T. H., Preiss, T. Dynamics of ribosome scanning and recycling revealed by translation complex profiling. Nature. 535 (7613), 570-574 (2016).
- Hinnebusch, A. G., Ivanov, I. P., Sonenberg, N. Translational control by 5'-untranslated regions of eukaryotic mRNAs. Science. 352 (6292), 1413-1416 (2016).
- Dever, T. E., Green, R. The elongation, termination, and recycling phases of translation in eukaryotes. Cold Spring Harbor Perspectives in Biology. 4 (7), 013706 (2012).
- Shirokikh, N. E., Preiss, T. Translation initiation by cap-dependent ribosome recruitment: Recent insights and open questions. Wiley Interdisciplinary Reviews RNA. 9 (4), 1473 (2018).
- Jiménez-Díaz, A., Remacha, M., Ballesta, J. P., Berlanga, J. J. Phosphorylation of initiation factor eIF2 in response to stress conditions is mediated by acidic ribosomal P1/P2 proteins in Saccharomyces cerevisiae. PLoS One. 8 (12), 84219 (2013).
- Sonenberg, N., Hinnebusch, A. G. Regulation of translation initiation in eukaryotes: mechanisms and biological targets. Cell. 136 (4), 731-745 (2009).
- Majmundar, A. J., Wong, W. J., Simon, M. C. Hypoxia-inducible factors and the response to hypoxic stress. Molecular Cell. 40 (2), 294-309 (2010).
- Barraza, C. E., et al. The role of PKA in the translational response to heat stress in Saccharomyces cerevisiae. PLoS One. 12 (10), 0185416 (2017).
- Richter, K., Haslbeck, M., Buchner, J. The heat shock response: Life on the verge of death. Molecular Cell. 40 (2), 253-266 (2010).
- Jamar, N. H., Kritsiligkou, P., Grant, C. M. The non-stop decay mRNA surveillance pathway is required for oxidative stress tolerance. Nucleic Acids Research. 45 (11), 6881-6893 (2017).
- Chen, Z., et al. The complete pathway for thiosulfate utilization in Saccharomyces cerevisiae. Applied and Environmental Microbiology. 84 (22), (2018).
- Marzluf, G. A. Molecular genetics of sulfur assimilation in filamentous fungi and yeast. Annual Review of Microbiology. 51, 73-96 (1997).
- Miller, D., Brandt, N., Gresham, D. Systematic identification of factors mediating accelerated mRNA degradation in response to changes in environmental nitrogen. PLoS Genetics. 14 (5), 1007406 (2018).
- Zhang, W., Du, G., Zhou, J., Chen, J. Regulation of sensing, transportation, and catabolism of nitrogen sources in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 82 (1), (2018).
- Tokpohozin, S. E., Fischer, S., Becker, T. Selection of a new Saccharomyces yeast to enhance relevant sorghum beer aroma components, higher alcohols, and esters. Food Microbiology. 83, 181-186 (2019).
- Walker, G. M., Stewart, G. G. Saccharomyces cerevisiae in the production of fermented beverages. Beverages. 2 (4), 30 (2016).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Jin, H. Y., Xiao, C. An integrated polysome profiling and ribosome profiling method to investigate in vivo translatome. Methods in Molecular Biology. 1712, 1-18 (2018).
- Arava, Y., et al. Genome-wide analysis of mRNA translation profiles in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences of the United States of America. 100 (7), 3889-3894 (2003).
- Lackner, D. H., et al. A network of multiple regulatory layers shapes gene expression in fission yeast. Molecular Cell. 26 (1), 145-155 (2007).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-Wide Analysis in Vivo of Translation with Nucleotide Resolution Using Ribosome Profiling. Science. 324 (5924), 218-223 (2009).
- Ingolia, N. T., Hussmann, J. A., Weissman, J. S. Ribosome Profiling: Global Views of Translation. Cold Spring Harbor Perspectives in Biology. 11 (5), (2019).
- Gerashchenko, M. V., Lobanov, A. V., Gladyshev, V. N. Genome-wide ribosome profiling reveals complex translational regulation in response to oxidative stress. Proceedings of the National Academy of Sciences of the United States of America. 109 (43), 17394-17399 (2012).
- Hussmann, J. A., Patchett, S., Johnson, A., Sawyer, S., Press, W. H. Understanding biases in ribosome profiling experiments reveals signatures of translation dynamics in yeast. Proceedings of the National Academy of Sciences Genetics. 11 (12), 1005732 (2015).
- Santos, D. A., Shi, L., Tu, B. P., Weissman, J. S. Cycloheximide can distort measurements of mRNA levels and translation efficiency. Nucleic Acids Research. 47 (10), 4974-4985 (2019).
- Schneider-Poetsch, T., et al. Inhibition of eukaryotic translation elongation by cycloheximide and lactimidomycin. Nature Chemical Biology. 6 (3), 209-217 (2010).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: A tool for the study of chromatin complexes. Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
- Kage, U., Powell, J. J., Gardiner, D. M., Kazan, K. Ribosome profiling in plants: What is not lost in translation. Journal of Experimental Botany. 71 (18), 5323-5332 (2020).
- Shirokikh, N. E., Archer, S. K., Beilharz, T. H., Powell, D., Preiss, T. Translation complex profile sequencing to study the in vivo dynamics of mRNA-ribosome interactions during translation initiation, elongation and termination. Nature Protocols. 12 (4), 697-731 (2017).
- Wagner, S., et al. Selective translation complex profiling reveals staged initiation and co-translational assembly of initiation factor complexes. Molecular Cell. 79 (4), 546-560 (2020).
- Zlotorynski, E. Profiling ribosome dynamics. Nature Reviews Molecular Cell Biology. 17 (9), 535-535 (2016).
- Sen, N. D., Gupta, N., S, K. A., Preiss, T., Lorsch, J. R., Hinnebusch, A. G. Functional interplay between DEAD-box RNA helicases Ded1 and Dbp1 in preinitiation complex attachment and scanning on structured mRNAs in vivo. Nucleic Acids Research. 47 (16), 8785-8806 (2019).
- Zhao, J., Qin, B., Nikolay, R., Spahn, C. M. T., Zhang, G. Translatomics: The global view of translation. International Journal of Molecular Sciences. 20 (1), 20010212 (2019).
- Luthe, D. S. A simple technique for the preparation and storage of sucrose gradients. Analytical Biochemistry. 135 (1), 230-232 (1983).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Review Genetics. 10 (1), 57-63 (2009).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trends in Biochemical Sciences. 25 (3), 99-104 (2000).
- Schmiedeberg, L., Skene, P., Deaton, A., Bird, A. A Temporal Threshold for Formaldehyde Crosslinking and Fixation. PLoS One. 4 (2), 4636 (2009).
- Solomon, M. J., Varshavsky, A. Formaldehyde-mediated DNA-protein crosslinking: A probe for in vivo chromatin structures. Proceedings of the National Academy of Sciences. 82 (19), 6470-6474 (1985).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping proteinDNA interactions in vivo with formaldehyde: Evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Bohlen, J., Fenzl, K., Kramer, G., Bukau, B., Teleman, A. A. Selective 40S footprinting reveals cap-tethered ribosome scanning in human cells. Molecular Cell. 79 (4), 561-574 (2020).
- Shirokikh, N. E. Translation complex stabilization on messenger RNA and footprint profiling to study the RNA responses and dynamics of protein biosynthesis in the cells. Critical Reviews in Biochemistry and Molecular Biology. , (2021).
- Giess, A., et al. Profiling of small ribosomal subunits reveals modes and regulation of translation initiation. Cell Reports. 31 (3), 107534 (2020).
- Firmino, A. A. P., et al. Separation and paired proteome profiling of plant chloroplast and cytoplasmic ribosomes. Plants (Basel). 9 (7), (2020).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic Acids Research. 42 (17), 134 (2014).
- Santos, D. A., Shi, L., Tu, B. P., Weissman, J. S. Cycloheximide can distort measurements of mRNA levels and translation efficiency. Nucleic Acids Research. 47 (10), 4974-4985 (2019).
- Schneider-Poetsch, T., et al. Inhibition of eukaryotic translation elongation by cycloheximide and lactimidomycin. Nature Chemical Biology. 6 (3), 209-217 (2010).
- Plénat, F., et al. Formaldehyde fixation in the third millennium. Annales De Pathologie. 21 (1), 29-47 (2001).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proceedings of the National Academy of Sciences. 105 (7), 2415-2420 (2008).
- Wang, N. S., Minassian, H. The formaldehyde-fixed and paraffin-embedded tissues for diagnostic transmission electron microscopy: A retrospective and prospective study. Human Pathology. 18 (7), 715-727 (1987).
- Grifo, J. A., et al. Characterization of eukaryotic initiation factor 4A, a protein involved in ATP-dependent binding of globin mRNA. Journal of Biological Chemistry. 257 (9), 5246-5252 (1982).
- Li, Y. Commonly used tag combinations for tandem affinity purification. Biotechnology and Applied Biochemistry. 55 (2), 73-83 (2010).
- Blum, S., et al. ATP hydrolysis by initiation factor 4A is required for translation initiation in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences. 89 (16), 7664-7668 (1992).
- Merrick, W. C. eIF4F: A Retrospective. Journal of Biological Chemistry. 290 (40), 24091-24099 (2015).
- Rogers, G. W., Komar, A. A., Merrick, W. C. eIF4A: The godfather of the DEAD box helicases. Progress in Nucleic Acid Research and Molecular Biology. 72, 307-331 (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados