Method Article
Rapida fissazione in vivo e isolamento dei complessi traslazionali dalle cellule eucariotiche
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Presentiamo una tecnica per stabilizzare rapidamente complessi traslazionali (biosintesi proteica) con reticolazione di formaldeide in cellule di lievito vivo e di mammifero. L'approccio consente di sezionare intermedi transitori e interazioni dinamiche RNA:proteina. I complessi reticolati possono essere utilizzati in molteplici applicazioni a valle come nei metodi di profilazione basati sul sequenziamento profondo, nella microscopia e nella spettrometria di massa.
Abstract
Le risposte rapide che coinvolgono una rapida ridistribuzione del messaggero (m) RNA e le alterazioni della traduzione dell'mRNA sono pertinenti agli aggiustamenti omeostatici in corso delle cellule. Questi aggiustamenti sono fondamentali per la sopravvivenza delle cellule eucariotiche e il "controllo dei danni" durante i livelli fluttuanti di nutrienti e salinità, la temperatura e vari stress chimici e di radiazione. A causa della natura altamente dinamica delle risposte a livello di RNA e dell'instabilità di molti degli intermedi RNA:RNA e RNA:proteina, ottenere un'istantanea significativa dello stato dell'RNA citoplasmatico è possibile solo con un numero limitato di metodi. Gli esperimenti di tipo ribosoma a livello di trascrittoma e basati su RNA-seq sono tra le fonti di dati più informative per il controllo della traduzione. Tuttavia, l'assenza di una stabilizzazione intermedia uniforme di RNA e RNA:proteina può portare a diversi pregiudizi, in particolare nelle vie di risposta cellulare frenetiche. In questo articolo, forniamo un protocollo dettagliato di fissazione rapida applicabile alle cellule eucariotiche di diversa permeabilità, per aiutare nella stabilizzazione intermedia di RNA e RNA: proteina. Forniamo inoltre esempi di isolamento dei complessi RNA:proteina stabilizzati in base alla loro co-sedimentazione con frazioni ribosomiali e poli(ribo)somali. Il materiale stabilizzato separato può essere successivamente utilizzato come parte di esperimenti di tipo ribosoma, come nell'approccio tcp-seq (Translation Complex Profile sequencing) e nei suoi derivati. La versatilità dei metodi in stile TCP-seq è stata ora dimostrata dalle applicazioni in una varietà di organismi e tipi di cellule. I complessi stabilizzati possono anche essere ulteriormente purificati per affinità e ripresi utilizzando la microscopia elettronica, separati in diverse frazioni poli(ribo)somali e sottoposti a sequenziamento dell'RNA, grazie alla facilità dell'inversione del reticolo. Pertanto, i metodi basati sullo snap-chilling e sulla fissazione della formaldeide, seguiti dall'arricchimento del complesso RNA:protein basato sulla sedimentazione o su un altro tipo di arricchimento del complesso RNA:protein, possono essere di particolare interesse nello studio di dettagli più fini della dinamica rapida del complesso RNA:protein nelle cellule vive.
Introduzione
Gli organismi viventi sono soggetti a cambiamenti dinamici intra ed extracellulari nel corso della loro vita, che richiedono risposte rapide per mantenere l'omeostasi e garantire la sopravvivenza. Per consentire l'adattamento ambientale, le cellule eucariotiche regolano il loro metabolismo attraverso il controllo dell'espressione genica. Il controllo dell'espressione genica può essere esercitato durante la trascrizione e/o la traduzione; con risposte traslazionali che si verificano generalmente più rapidamente1,2,3,4. Ad esempio, i cambiamenti traslazionali si verificano tipicamente entro 1-30 minuti dall'inizio dello stress, mentre le alterazioni a livello di trascrizione seguono ore dopo l'esposizione allo stress3,4,5. Le alterazioni della produzione di traduzione si ottengono più rapidamente a causa della persistente disponibilità di molecole di (m)RNA messaggero nel citoplasma. Viceversa, a livello di trascrizione, nuove molecole di mRNA devono essere sintetizzate, e negli eucarioti, elaborate ed esportate dal nucleo, producendo ampi ritardi nel tempo di risposta2,4,6,7,8.
La risposta traslazionale acuta allo stress è generalmente caratterizzata da una diminuzione complessiva della produzione di traduzione, con l'upregulation selettiva delle proteine necessarie per la sopravvivenza cellulare1,3,4,9. Si ritiene che la riduzione della produzione di proteine sia cruciale a causa dell'elevato costo energetico del processo3,7. Per facilitare l'inibizione selettiva e l'upregulation, le risposte traslazionali sono servite da una serie di complessi meccanismi di regolazione. La regolazione può essere esercitata in tutte le fasi della traduzione: inizio, allungamento, cessazione della biosintesi polipeptidica e riciclaggio ribosomiale10,11,12,13, ma è esposto più fortemente nella fase di iniziazione5,7,9,10,13. Durante l'inizio, la piccola subunità ribosomiale (SSU), assistita da fattori di iniziazione eucariotici (eIF), si lega e scansiona la regione 5' non tradotta (UTR) dell'mRNA fino a quando non viene riconosciuto un codone iniziale2,5,6,8,11,12,13. I meccanismi di regolamentazione spesso prendono di mira gli eIF che influiscono sull'allegato, la scansione e il riconoscimento dei codoni di avvio. Ad esempio, il fattore di iniziazione eIF2, un fattore di traduzione essenziale che aiuta nel reclutamento di un Met-tRNA iniziatoreiMet alla SSU, è spesso mirato negli eucarioti in condizioni di stress4,6,11. Nel lievito, la fosforilazione di questo fattore può essere indotta sotto deprivazione di nutrienti e stress osmotico1,4,11,14,15, e nelle cellule di mammifero, la fame di aminoacidi, lo stress del reticolo endoplasmatico (ER), lo stress UV, l'infezione virale e i livelli di ossigeno alterati possono innescare questa risposta8,9,11. La rapida sovraregolazione della traduzione specifica dell'mRNA è evidente nella risposta delle cellule di mammifero all'ipossia, che presenta un'inibizione globale della traduzione rapida e una sovraregolazione selettiva della biosintesi dei fattori inducibili dall'ipossia (HIF). Gli HIF sono fattori di trascrizione, che poi suscitano una riprogrammazione cellulare a lungo termine a livello di trascrizione del DNA8,9,16. Risposte simili sono state osservate nel lievito sotto stress termico, con rapida espressione traslazionale di heat shock proteins (HSP) seguita da risposte ritardate a livello di trascrizione17,18. Oltre alla privazione di nutrienti e allo shock termico, le risposte traslazionali nel lievito sono state studiate sotto ossigeno variabile.8,19salinità5, fosfato, zolfo20,21 e azoto22,23 Livelli. Questa ricerca ha implicazioni diffuse per gli usi industriali del lievito, come la cottura e la fermentazione24,25. Le risposte traslazionali possono anche essere strumentali nel promuovere la comprensione di malattie come i disturbi neurodegenerativi e le malattie cardiache, che sono caratterizzate da stress intracellulari come lo stress ossidativo. Nel complesso, le risposte traslazionali sono parte integrante del controllo dell'espressione genica e facilitano un rapido adattamento a una vasta gamma di condizioni di stress negli organismi eucariotici.
Per studiare le risposte traslazionali, sono necessari metodi che forniscano istantanee minimamente distorte del panorama della traduzione. La profilazione dei polisomi è un approccio classico utilizzato nello studio della traslazione attraverso l'mRNA, che coinvolge la separazione delle frazioni poli(ribo)somali dell'mRNA tramite ultracentrifugazione attraverso gradienti di saccarosio26,27. L'approccio può essere utilizzato per esplorare i livelli di traduzione per i singoli mRNA (con i metodi di rilevamento come la trascrizione inversa e la reazione a catena della polimerasi, RT-PCR26), o globalmente in combinazione con tecniche ad alto rendimento (microarray o RNA-seq28,29). Un approccio più evoluto è la profilazione dei ribosomi, che consente lo studio delle posizioni dei ribosomi allunganti lungo una molecola di mRNA su scala genomica, nonché l'inferenza dell'efficienza della traduzione attraverso il trascrittoma e l'utilizzo dei siti di partenza principali e alternativi30,31. La profilazione dei ribosomi comporta l'isolamento e il sequenziamento di frammenti di mRNA protetti dalla presenza ribosomiale su di essi. La profilazione dei ribosomi ha fornito una notevole comprensione delle dinamiche di traduzione in una serie di condizioni, tra cui stress ipossico, shock termico e stress ossidativo31,32. La tecnica è stata adattata a più tipi di materiale di origine, tra cui lievito e cellule di mammifero.
Mentre la profilazione di polisomi e ribosomi è stata fondamentale per estendere le capacità di ricerca nella traduzione, il processo di traduzione include vari intermedi e complessi traslazionali difficili da catturare con questi metodi11,13. Un'ulteriore limitazione deriva dalla mancanza di capacità di studiare i tipi di risposta rapida, poiché i complessi traslazionali sono stabilizzati in vivo dall'aggiunta di specifici inibitori della traduzione (antibiotici), portando a determinati artefatti di distribuzione dei ribosomi, o ex vivo su lisi cellulare specificamente (antibiotici) o in modo non specifico (ioni salini o magnesio elevati), portando alla privazione degli intermedi di vita più breve o meno stabili33, 34,35.
La formaldeide è ampiamente utilizzata per reticolare acidi nucleici e proteine, come negli studi di immunoprecipitazione della cromatina (ChIP) e immunoprecipitazione reticolante (CLIP). Le sue piccole dimensioni e l'eccellente permeabilità cellulare consentono una rapida azione in vivo 36. Basato sulla rapida reticolazione della formaldeide, l'approccio di profilazione dei ribosomi è stato esteso con il Translation Complex Profile Sequencing (TCP-seq)10,36,37,38,39,40. TCP-seq, sviluppato per la prima volta nel lievito, consente la cattura di tutti gli intermedi di traduzione, compresi i complessi SSU di scansione o post-terminazione e le configurazioni ribosomiali multiple37,38,41,42. Il metodo è stato utilizzato in diversi studi10,38,39,41,42,alcuni dei quali utilizzano un approccio combinatorio sia di inibitori della traduzione che di reticolazione della formaldeide per facilitare l'arresto della traduzione. Un'ulteriore versione modificata della tecnica, TCP-seq39selettivo, è stata recentemente impiegata per includere l'immunopurazione dei complessi reticolati, ampliando la portata delle applicazioni TCP-seq. La natura rapida, efficiente e reversibile della reticolazione della formaldeide rende questi approcci adatti allo studio di interazioni complesse transitorie tra mRNA:traduzione, in particolare nel contesto di percorsi di risposta a livello di traduzione altamente dinamici.
Qui descriviamo in dettaglio i processi di reticolazione della formaldeide in vivo ai fini della stabilizzazione e dell'isolamento completi del complesso di traduzione. Forniamo protocolli separati sfumati per le cellule di lievito e mammifero (Figura 1). Descriviamo inoltre esempi del successivo utilizzo del materiale stabilizzato con reticolazione (Figura 1), ad esempio per il rilevamento di fattori proteici co-purificati mediante immunoblotting (western-blotting), purificazione immuno-assistita (o "immunoprecipitazione"; IP) e arricchimento di complessi traslazionali contenenti specifici fattori di interesse, microscopia elettronica e sequenziamento dell'RNA.
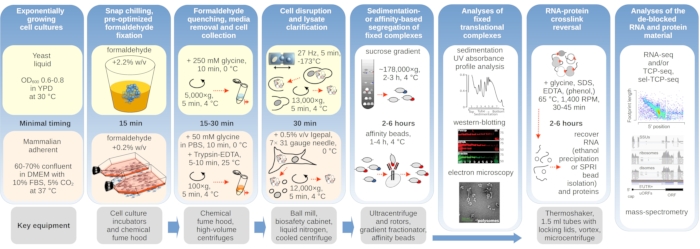
Figura 1: Schema che illustra una panoramica della configurazione sperimentale tipica. Le fasi principali della stabilizzazione in vivo della formaldeide dei complessi traslazionali sono rappresentate come un diagramma di flusso, integrato da informazioni sugli strumenti chiave necessari. Vengono delineate le potenziali applicazioni a valle del materiale reticolato, inclusi esempi che sono stati impiegati con successo ma non direttamente coperti in questo protocollo, come la purificazione del tallone SPRI dell'RNA, il sequenziamento dell'RNA e la spettrometria di massa. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
1. Protocollo delle cellule di lievito
- Coltura e fissazione delle cellule di lievito
NOTA: la fissazione e la raccolta delle cellule sono adattate da10,38con modifiche.- Impostare una coltura cellulare di lievito da 1 L (wild-type (WT) BY4741 sono forniti come esempio) in uno shaker orbitale con densità ottica iniziale non superiore a 0,05 UA a 600 nm (OD600) in mezzi adatti (1% p /v di estratto di lievito, 2% p/v di peptone, 2% p/v di destrosio (glucosio), 40 mg/L di solfato di adenina (YPD) usati come esempio) nelle condizioni desiderate (30 °C utilizzati in questo esperimento).
- Impostare una centrifuga preparativa con rotore compatibile e bottiglie centrifughe per la pellettizzazione della coltura in sospensione liquida delle cellule di lievito. Per gli esperimenti di fame di glucosio, pellet le cellule una volta raggiunta la densità ottica di 0,6-0,8 UA a 600 nm (OD600), utilizzando una breve centrifugazione a 30 °C, 5.000 x g per 1 min.
NOTA: Tenere traccia dell'OD delle cellule in crescita e lasciare che le cellule crescano fino a quando l'OD600 raggiunge 0,6-0,8 UA, se la fase di crescita esponenziale è di interesse. - Sospendere immediatamente il pellet in un mezzo YP caldo (30 °C) contenente glucosio aggiunto nullo o basso (0,25% p/v) e incubare la coltura per altri 10 minuti a 30 °C in uno shaker-incubatore orbitale.
NOTA: la composizione dei supporti potrebbe influire sull'efficienza della reticolazione successiva. Questo protocollo è stato testato utilizzando solo YPD. Quando si eseguono esperimenti di fame, è fondamentale rispettare i tempi e ridurre al minimo i ritardi tra le procedure. - Una volta che le celle sono pronte, allestire una ghiacciaia all'interno della cappa aspirante con un becher contenente 250 g di ghiaccio d'acqua frantumato pulito. Assicurarsi che 25 mL di stripette e la soluzione di formaldeide al 37% p/v stabilizzata al metanolo appena acquistata siano accessibili all'interno della cappa. Versare la coltura da 1 L nel becher contenente il 25% p/v di ghiaccio d'acqua frantumato.
NOTA: Mantenere le cellule su ghiaccio durante tutte le operazioni successive fino a quando le cellule non sono congelate, salvo diversa indicazione. - Aggiungere 75 ml di 37% p/v di soluzione di formaldeide ad una concentrazione finale del 2,2% p/v e mescolare intensamente la miscela fino a quando il ghiaccio si scioglie.
- Una volta sciolto il ghiaccio, impostare un timer per 10 minuti.
NOTA: Attenersi ai tempi e al regime di temperatura raccomandati per ottenere risultati di fissazione riproducibili. - Dopo l'incubazione per 10 minuti, trasferire la coltura nelle bottiglie della centrifuga preraffreddata e pellettizzare le celle mediante centrifugazione a 4 °C, 5.000 x g per 5 minuti. Mentre questo giro è acceso, preraffreddare un tubo da 50 ml e mantenere il tampone A appena preparato (contenente glicina per neutralizzare l'eventuale formaldeide rimanente) sul ghiaccio.
NOTA: fare riferimento alla tabella fornita per le composizioni esatte del buffer. - Dopo la centrifugazione, posizionare i tubi della centrifuga sul ghiaccio con il lato pellet a contatto con il ghiaccio. Portare i tubi nella cappa aspirante e gettare il surnatante in un contenitore per rifiuti di formaldeide.
- Risospendare il pellet di cella da tutti i tubi in 20 mL di tampone A utilizzando una stripette da 25 mL e trasferendolo in un tubo da 50 mL.
NOTA: questo lavaggio è fondamentale per evitare reticolazioni irriproducibili e l'aggiunta del tampone non deve superare i 20 minuti di tempo dalla raccolta delle cellule. - Portare il volume a 40 ml con tampone A e raccogliere le celle lavate mediante centrifugazione a 4 °C, 5.000 x g per 5 min.
- Scartare il surnatante e risospesciare il pellet cellulare in 40 ml di tampone A1, che è il tampone A non contenente glicina, per rimuovere qualsiasi contaminazione da glicina.
- Celle a pellet di nuovo per centrifugazione a 4 °C, 5.000 x g per 5 min.
- Ripetere i lavaggi con il buffer A1 ancora una volta. Scartare il surnatante e posizionare il pellet cellulare sul ghiaccio. Pesare il tubo con il pellet (la massa delle cellule umide dovrebbe essere di ~ 1 g per 1 L della coltura cellulare).
- Interruzione delle cellule di lievito e raccolta del citosol
- Riempire una scatola di polistirene espanso rivestita con un foglio di alluminio con azoto liquido ad una profondità di circa 3 cm. Posizionare un tubo da 50 ml in posizione verticale nella scatola.
- Risospesso il pellet (~ 1 g di massa cellulare umida) in 550 μL di tampone A2 mediante pipettaggio e vortice per 10 s. Aggiungere 10 μL di 40 U/μL di inibitore della RNasi e vortice di nuovo per 10 s.
ATTENZIONE: Indossare dispositivi di protezione appropriati, come guanti isolati termicamente, quando si maneggia azoto liquido. Assicurarsi che qualsiasi contenitore utilizzato per contenere azoto liquido non perda e che il rack del tubo all'interno non galleggi o cada su un lato. Lavorare in un'area ben ventilata per evitare l'esaurimento di ossigeno. - Utilizzando una pipetta da 1 mL, gocciolare la sospensione cellulare nel tubo da 50 mL contenente l'azoto liquido.
NOTA: Il gocciolamento deve essere eseguito lentamente e con attenzione per evitare l'aggregazione delle goccioline. Assicurarsi che le goccioline si congelino prima di introdurre nuove goccioline. - Trasferire il tubo da 50 ml con le goccioline di sospensione cellulare congelate a temperatura ambiente e attendere che l'azoto liquido evapori completamente. Sigillare il tubo con il suo tappo e conservare il pellet cellulare a -80 °C o procedere immediatamente oltre.
ATTENZIONE: Assicurarsi che l'azoto liquido sia completamente evaporato prima di sigillare il tubo. L'azoto liquido residuo in un tubo sigillato può causare un pericoloso accumulo di pressione. - Per prepararsi al passo successivo, preraffreddare tubi senza nucleasi da 1,5 mL e barattoli di macinazione in acciaio inossidabile da 10 mL su ghiaccio secco.
- Trasferire le goccioline di sospensione cellulare congelate nei barattoli usando una spatola pulita e sterile.
ATTENZIONE: Assicurarsi che i barattoli di macinazione siano ben sigillati. - Immergere i barattoli di macinazione nell'azoto liquido per 1 minuto assicurandosi che la fase liquida rimanga al di sotto della giunzione. Impostare un mulino criocondatore a 27 Hz per l'agitazione per 1 minuto.
NOTA: bilanciare sempre il contenitore di rettifica con un altro dello stesso modello, anche se il campione richiede un solo contenitore per l'elaborazione. - Agitare i barattoli di macinazione sigillati a 27 Hz per 1 minuto nel mulino miscelatore.
- Raffreddare nuovamente i barattoli di macinazione in azoto liquido come prima e agitare a 27 Hz per 1 minuto ulteriormente nel mulino miscelatore.
- Trasferire i barattoli nella ghiacciaia contenente ghiaccio secco insieme ai tubi da 1,5 mL privi di nucleasi. Utilizzando una piccola spatola di acciaio, trasferire il campione in polvere risultante nelle provette in aliquote ~100 mg e conservare le provette a -80 °C.
NOTA: Si raccomanda di utilizzare ~ 600 mg del campione per esperimento comprendente l'analisi del profilo di sedimentazione dei polisomi, la separazione del citosol in frazioni tradotte e non tradotte e un'ulteriore separazione della frazione tradotta in SSU, ribosoma e frazioni disomi dopo la digestione della RNasi.
- Separazione dei complessi (poli)ribosomiali fissi dalle frazioni non tradotte del citosol
NOTA: la procedura stabilita in precedenza10,38è generalmente seguito per arricchire l'RNA tradotto in base alla sua co-sedimentazione con (poli)ribosomi. Qui viene introdotto un approccio più raffinato per separare le frazioni di citosol tradotte e non tradotte, eliminando la necessità di precipitare e successivamente risoluzionare il materiale.- Preparare gradienti lineari di saccarosio da 2,5 mL 10%-20% p/v con tampone B utilizzando il metodo freeze-thaw43 in tubi ultracentrifuga a parete sottile (5 mL, 13 x 51 mm).
NOTA: Il metodo di congelamento-scongelamento viene eseguito mediante l'aggiunta sequenziale e il congelamento di strati tamponati di saccarosio con concentrazioni linearmente regressive l'uno sull'altro. Cfr. tabella supplementare 1 per i dettagli. - Per creare un cuscino discontinuo di saccarosio al 50% p/v, dopo lo scongelamento e la stabilizzazione dei gradienti lineari, erogare lentamente 0,5 mL di saccarosio al 50% nel tampone B direttamente sul fondo dei tubi utilizzando una siringa da 1 mL attaccata ad un ago da 19 G x 1,5" o un capillare di vetro di dimensioni simili/adatte. Prima di erogare, guidare con attenzione e lentamente la punta dell'ago o del capillare dall'alto verso il basso dei gradienti di saccarosio preformati, evitando qualsiasi disturbo, fino a raggiungere il fondo del tubo.
NOTA: vedere la tabella supplementare 1 per le istruzioni sulla preparazione del tampone B. - Bilanciare accuratamente i gradienti rimuovendo le porzioni superiori o stratificando più 10 p/v di saccarosio nel tampone B e mantenerli ghiacciati o a 4 °C.
NOTA: Il gradiente discontinuo con lo strato inferiore di saccarosio al 50% è necessario per raccogliere materiale con una maggiore velocità di sedimentazione senza precipitarlo sulla parete del tubo. - Scongelare ~ 100 mg del campione in polvere di cellule congelate a temperatura ambiente e mettere immediatamente sul ghiaccio. Miscelare in 150 μL di tampone A2 mediante pipettaggio, aggiungere l'inibitore della RNasi a 1 U/μL e miscelare mediante vortice (evitare un'eccessiva formazione di schiuma e miscelazione con la fase gassosa) per 10 s.
NOTA: Continuare tutte le operazioni mantenendo il materiale su ghiaccio, se non diversamente indicato. - Pellet i detriti cellulari centrifugando i tubi a 4 °C, 13.000 x g per 5 min e recuperando il surnatante chiarificato (~150 μL) in un nuovo tubo a bassa legante proteica da 1,5 mL.
- Caricare la miscela chiarificata risultante sui tubi a gradiente di saccarosio discontinuo dal punto 1.3.3 e bilanciarli accuratamente.
- Ultracentrifugare i tubi in un rotore a benna oscillante di medio volume a 4 °C, con forza g media 287.980 x g (fattore k 49) per 1 ora e 30 min.
NOTA: Queste condizioni sono state pre-ottimizzate (utilizzando l'analisi delle tracce di assorbanza UV con gradiente post-ultracentrifugazione) per mantenere le SSU libere (non (poli)ribosomiali) e le LSU (grande subunità ribosomiale) nella parte superiore (10%-20% di saccarosio) del gradiente mentre si concentra la frazione (poli)ribosomiale nella parte inferiore (50%) cuscino di saccarosio senza pellettare il materiale. - Utilizzare una nuova siringa sterile da 1 mL dotata di un ago da 19 G x 1,5" per raccogliere la frazione di citosol tradotta. Posizionare la pendenza di 5 ml su un rack stabile assicurandosi che il fondo del tubo sia visibile.
- Dalla parte superiore del tubo, infilare l'ago direttamente nella parte inferiore del gradiente (senza forare il tubo) e delicatamente, senza creare bolle, aspirare esattamente 0,5 ml della soluzione inferiore contenente il pool di RNA tradotto.
NOTA: Assicurarsi che questo passaggio venga eseguito in una cella frigorifera e che il tubo sia tenuto saldamente. Si consiglia di disegnare l'intero 0,5 mL in un singolo movimento verso l'alto per evitare disturbi della pendenza. - Confermare la presenza (poli)ribosomiale e l'esaurimento della SSU, LSU e frazioni più leggere nella miscela risultante mediante lettura dell'assorbanza del gradiente di saccarosio al momento dell'esecuzione ultracentrifugazione.
- Concentrare il pool di RNA tradotto raccolto dalla fase precedente a 100 μL utilizzando l'ultrafiltrazione in un dispositivo di micro-concentrazione con membrana di cellulosa rigenerata cut-off da 10 kDa.
NOTA: prelavare la membrana del dispositivo di microconcentrazione con 0,5 mL di tampone 1 (vedere Figura 2a)e utilizzare le condizioni di centrifuga (g) raccomandate dal produttore. - Diluire ulteriormente il materiale dalla fase precedente cinque volte (aggiungere 400 μL) con tampone 1 e concentrare di nuovo a 200 μL, per consentire un volume più piccolo e una rimozione parziale del saccarosio.
NOTA: Si consiglia di conservare le miscele risultanti a -80 °C per un massimo di 6 mesi e di utilizzarle come materiale di input per la costruzione della libreria RNA-seq "RNA totale tradotto" o la fase di digestione della RNasi della costruzione della libreria TCP-seq. La frazione di citosol "non tradotta" può essere recuperata dalla parte superiore del gradiente utilizzando una procedura simile e conservata a -80 °C.
- Preparare gradienti lineari di saccarosio da 2,5 mL 10%-20% p/v con tampone B utilizzando il metodo freeze-thaw43 in tubi ultracentrifuga a parete sottile (5 mL, 13 x 51 mm).
- Digestione della RNasi dei complessi fissi (poli)ribosomiali e separazione del materiale digerito in piccole subunità ribosomiali (SSU), monoricobosomiche (ribosomi, RS) e frazioni diribosoma (disomi, DS)
NOTA: la procedura segue generalmente un approccio descritto in precedenza10,38ma vengono impiegati un tipo di gradiente modificato, tempo di separazione, accelerazione e condizioni di digestione della RNasi, per ottenere la migliore risoluzione in tutte e tre le frazioni isolate.- Preparare gradienti di saccarosio lineari 12,5 mL 10%-40% p/v accuratamente bilanciati realizzati con tampone 1 in tubi di polipropilene a parete sottile da 13 mL, 14 x 89 mm, utilizzando il metodo di congelamento-scongelamento43 come descritto al punto 1.3.1 e annotarlo.
- Scongelare a temperatura ambiente e trasferire immediatamente i campioni su ghiaccio o prelevare la frazione di citosol tradotto concentrata e impoverita di saccarosio dal punto 1.3.12.
NOTA: Continuare tutte le procedure sul ghiaccio se non diversamente indicato. - Digerire la frazione di citosol tradotta mescolando in 4,5 U di E. coli RNasi I per 1 unità OD260 della frazione per 30 minuti a 23 °C. Aggiungere e miscelare immediatamente pipettando l'inibitore della RNasi in grado di inattivare la RNasi I a 0,25 U/μL alla miscela, per inattivare la RNasi I.
NOTA: Utilizzare L'inibitore della RNasi in grado di inibire la RNasi I. Derivare AU260 utilizzando AU260 = (Assorbanza a 260 nm standardizzata a unità di densità ottica equivalenti a 1 cm di percorso ottico x volume del lisato in μL) / 1.000. - Trasferire immediatamente i campioni sul ghiaccio.
ATTENZIONE: È fondamentale rispettare le condizioni raccomandate di digestione e misurare attentamente la quantità di RNasi I aggiunta. L'unità RNasi I a cui si fa riferimento qui è definita come la quantità dell'enzima necessaria per produrre 1 μg di materiale acidosolubile dall'RNA del fegato di topo in 30 minuti a 37 °C. I lotti di RNasi I possono avere variazioni non documentate nell'attività e possono richiedere la sperimentazione per ottenere condizioni di digestione ottimali. Se lo stock enzimatico è troppo concentrato, si consiglia di diluirlo con tampone 1 per evitare di pipettare volumi molto piccoli della soluzione. - Caricare le miscele di reazione sui gradienti di saccarosio 10%-40% p/v dal punto 1.4.1.
NOTA: utilizzare volumi finali nell'intervallo 150-300 μL per gradiente. Ogni purificazione richiede almeno due gradienti. Utilizzare diversi volumi di input del materiale (AU260inferiore, 10-11 AU260,per DS e AU260relativamente superiore, 13-14 AU260,per SSU o RS) per ottenere una separazione ottimale. - Ultracentrifugare i tubi in un rotore a benna oscillante di medio volume a 4 °C con forza g media 178.305 x g (fattore k 143,9) per 3 ore e 30 minuti.
ATTENZIONE: Se sono necessari tubi di bilanciamento di riserva, equalizzare la loro massa e distribuzione di massa con i tubi contenenti il campione. Utilizzare gradienti di saccarosio di riserva sovrapposti a una quantità di tampone equivalente a quella della sovrapposizione del campione e non tubi con concentrazione uniforme di saccarosio. - Installare un dispositivo di frazionamento a gradiente almeno 30 minuti prima del completamento dello spin di ultracentrifugazione, compreso il riempimento della soluzione filtrata per inseguimento pesante filtrata da 0,2 μm (ad esempio, il 60% di saccarosio in acqua deionizzata come usato qui) nella pompa volumetrica.
NOTA: Si raccomanda di de-contaminare le linee e i tubi del frazionatore utilizzando acqua deionizzata, seguita da soluzione SDS all'1%-2% in acqua deionizzata, acqua deionizzata e infine etanolo all'80% in soluzione acquosa deionizzata prima e dopo le corse. - Regolare la linea di base della lettura dell'assorbanza riempiendo prima il sistema con acqua deionizzata e azzerando l'ottica secondo le raccomandazioni del produttore, quindi compensando lo spostamento della linea di base utilizzando un gradiente di saccarosio di riserva scaricato 14 x 89 mm realizzato con un buffer identico alle provette del campione (ad esempio, tampone 1).
NOTA: utilizzare la stessa velocità di spostamento per effettuare le regolazioni della lettura del campione, ad esempio 1,5 ml/min. - Misurare il volume morto del sistema di spostamento contando con precisione il tempo tra la soluzione che entra per la prima volta nel percorso ottico del rivelatore e appare per la prima volta all'uscita del collettore di frazioni.
NOTA: Con la velocità consigliata di 1,5 ml/min, il frazionamento può essere eseguito a temperatura ambiente. Si raccomanda di trasferire immediatamente le frazioni raccolte sul ghiaccio. - Eseguire il frazionamento utilizzando la lettura dell'assorbanza dal vivo a 254 nm, velocità di spostamento di 1,5 ml/min e rilevamento della frazione in linea in base alla posizione di sedimentazione prevista e al profilo di assorbanza dei campioni. Utilizzare la commutazione del tubo del collettore con un ritardo temporale corrispondente al volume morto misurato in precedenza.
- Isolare le frazioni corrispondenti alle posizioni e alla mobilità dei complessi SSU, RS e DS e raccoglierle in nuovi tubi microcentrifuga da 1,5 mL a basso legame proteico; trasferire immediatamente le frazioni isolate su ghiaccio e congelare se non lavorate ulteriormente subito.
NOTA: Si raccomanda di congelare immediatamente le frazioni raccolte in ghiaccio secco o azoto liquido e di conservarle a -80 °C o meno per un massimo di 6 mesi.
- De-crosslinking dei complessi ribosomiali e isolamento dell'RNA per costruire librerie RNA-seq
- Per de-bloccare/invertire i legami incrociati e isolare l'RNA lontano dalle proteine associate, trasferire circa la metà dell'intera frazione di gradiente di saccarosio in nuovi tubi di microcentrifuga da 1,5 mL privi di nucleasi a bassa leganza degli acidi nucleici (350 μL per tubo) con dispositivi di sicurezza/bloccaggio del coperchio.
- Integrare le miscele con 40 μL di soluzione 100% stop (10% SDS p/v e 100 mM EDTA), 4 μL di 1 M Tris-HCl pH 2 a 25 °C (a 10 mM), 1,6 μL di 2,5 M di glicina (a 10 mM) e acqua priva di nucleasi deionizzata per ottenere il volume finale di 400 μL.
- Mescolare il contenuto dei tubi mediante pipettaggio e trasferire i tubi a temperatura ambiente.
- Aggiungere un volume uguale della miscela acida di fenolo:cloroformio:alcool isoamilico 125:24:1 (pH 4,0-5,0) a ciascun tubo. Agitare vigorosamente le miscele per 2 minuti utilizzando un miscelatore a vortice impostato alla massima velocità.
ATTENZIONE: Il fenolo e il cloroformio sono corrosivi e tossici. Evitare il contatto fisico con i liquidi e lavorare in un'area ben ventilata o sotto una cappa aspirante. Utilizzare sempre guanti, camice da laboratorio e occhiali protettivi o una visiera quando si lavora con fenolo o cloroformio. - Posizionare i tubi in un termoscuolatore e agitare continuamente a 65 °C, 1.400 giri/min per 30 minuti.
- Facilitare l'aggregazione di fase centrifugando la miscela a 12.000 x g per 10 minuti a temperatura ambiente.
- Raccogliere le fasi acquose superiori e trasferirle in tubi freschi da 1,5 mL leganti acidi nucleici bassi.
NOTA: Per evitare la contaminazione incrociata, non tentare di recuperare completamente le fasi acquose. Un volume di recupero ragionevole è di 300-350 μL. - Integrare le fasi acquose raccolte con 0,1 volumi di 3 M di acetato di sodio (pH 5 a 25 °C), 20 μg di glicogeno (utilizzando 5 μg/μL di stock) e 2,5 volumi di etanolo assoluto. Mescolare accuratamente le soluzioni ruotando i tubi per 1 minuto.
- Precipitare l'RNA incubando i campioni a -20 °C per almeno 2 ore (consigliato durante la notte).
- Riscaldare i tubi a temperatura ambiente e mescolarli vorticosamente.
NOTA: Il preriscaldamento dei tubi e la successiva centrifugazione a temperatura ambiente (senza refrigerazione forzata) aiutano a ridurre la co-precipitazione e il riporto di sale e fenolo. Queste condizioni non dovrebbero comportare la perdita di materiale o l'inefficienza della raccolta di RNA se eseguite come descritto e utilizzando etanolo sufficientemente puro. - Pellet l'RNA precipitato centrifugando i tubi a 12.000 x g per 30 minuti a temperatura ambiente.
- Scartare il surnatante e lavare il pellet due volte con etanolo all'80% v/v, raccogliendolo ogni volta per centrifugazione a 12.000 x g per 10 minuti a temperatura ambiente.
- Asciugare i pellet di RNA aprendo i coperchi dei tubi e posizionando i tubi aperti in un riscaldatore a blocco secco impostato a 45 °C per 10 minuti. Sciogliere il pellet essiccato risultante in 20 μL di 1x tampone HE.
- Stimare la concentrazione di RNA risultante utilizzando la misurazione dello spettro di assorbanza UV.
NOTA: la lunghezza del frammento di RNA e la quantità totale possono essere ulteriormente valutate utilizzando l'elettroforesi su gel denaturante, ad esempio in un apparato automatizzato di elettroforesi su gel capillare a base di fluorescenza.
- Co-immunopurificazione selettiva delle SSU mediante eIF con tag e eifs e western blot dell'arricchimento selettivo delle SSU
NOTA: Utilizzare ~ 15 UA (260 nm) della frazione SSU digerita e segregata per sedimentazione dal passaggio 1.4.11 per eseguire la purificazione dell'affinità utilizzando perline magnetiche IgG. Salvare circa il 5% della frazione SSU come controllo di input (Frazione di input, I). Tag eIF4A (TIF1-TAP; Tandem Affinity Purification tag) ceppo di lievito è stato utilizzato che consente anche di rilevare eIF4A sondando il TAP-tag utilizzando l'anticorpo anti-TAP.- Trasferire 100 μL di sospensione magnetica di perline IgG (1 mg delle perline sono state utilizzate per ogni 15 UA (260 nm) del lisato o della frazione) in un nuovo tubo da 1,5 mL a basso legame proteico; raccogliere le perline utilizzando rack magnetico e aspirarle.
- Lavare le perline magnetiche due volte con 1 mL di tampone 1 utilizzando la risospensione sequenziale mediante pipettaggio e raccolta utilizzando il rack magnetico.
- Dopo il lavaggio, raccogliere e decantare le perline, tenendole sul rack magnetico.
- Aggiungere la frazione SSU alle perline lavate e incubare la miscela per 4 ore con rotazione a 4 °C in un ciclomissatore impostato a ~ 20 rpm.
- Raccogliere le perline utilizzando il rack magnetico a 4 °C e salvare il surnatante (Frazione flow-through, FT).
- Lavare le perline due volte a 4 °C con tampone 1 integrato con 4 mM DTT, ruotando ogni volta per 10 minuti nel ciclomissatore e raccogliendo e decantando le perline sul rack magnetico. Salvare i lavaggi (frazioni W1 e W2).
- Per un'applicazione analitica come il western blotting, eluire il materiale legato in condizioni di denaturazione e riduzione aggiungendo il tampone campione di elettroforesi su gel di poliacrilammide (PAGE) LDS (litio dodecilsalfato) con pH da 8,5 a 1x e DTT a 2 mM.
- Riscaldare la miscela a 95 °C per 5 minuti in un blocco termico per finalizzare l'eluizione.
- Raccogliere le perline utilizzando il rack magnetico e recuperare l'eluato denaturato (frazione E) in un tubo microcentrifuga da 1,5 mL a basso legame proteico fresco.
- Utilizzare immediatamente la frazione E della fase precedente per eseguire immediatamente una PAGINA di dodecilsolfato di sodio (SDS) denaturante o conservare la frazione E a -20 °C.
NOTA: per una raccolta preparativa dei complessi traslazionali arricchiti con TAG TAP per qualsiasi applicazione successiva, utilizzare un approccio alternativo di eluizione che utilizza la proteasi del virus dell'incisione del tabacco (TEV). Fare riferimento alla tabella supplementare 1 per ulteriori dettagli. - Per concentrare le frazioni FT, W1 e W2 diluite, precipitare il loro materiale aggiungendo 3x volumi di acetone ghiacciato. Incubare la miscela campione-acetone a -20 °C per 3 ore.
- Pellet il precipitato centrifugando i tubi a 13.000 x g per 10 min a 4 °C.
- Scartare il surnatante e asciugare all'aria il pellet nei tubi aperti a temperatura ambiente per 30 minuti.
- Sciogliere il pellet in 7 μL di 1x buffer di carico LDS integrato con 2 mM DTT. Riscaldare i campioni in un blocco termico impostato a 95 °C per 5 min.
- Caricare tutti i campioni I, FT, W1, W2 ed E su un 4%-12% p/v di gradiente di acrilammide, gel denaturante di poliacrilammide Bis-Tris. Eseguire il gel utilizzando 1x MES SDS (acido 2- [N-mofolino]etansolfonico, sodio dodecil solfato) con tampone a 80 V, fino a quando il marcatore proteico (10-250 kDa) si risolve bene e il colorante al piombo raggiunge il fondo del gel.
NOTA: Si consiglia di caricare le diluizioni seriali del WCL (lisato a cellule intere) (2-10 μg) sul gel come controllo. Potrebbero essere necessari diversi tentativi per ottenere un carico comparabile del gel attraverso il materiale della frazione. - Trasferire il contenuto proteico del gel su una membrana di polivinilidene difluoruro (PVDF) con metodo di trasferimento a umido a 100 V per 1 ora in una cella frigorifera come raccomandato dal produttore di apparecchiature western-blotting.
- Bloccare la membrana utilizzando un apposito tampone bloccante (a base di fosfato tamponato con soluzione salina) a temperatura ambiente per 1 ora sotto scuotimento costante.
- Seguendo le istruzioni del produttore per la diluizione degli anticorpi, sondare la membrana con anticorpo anti-TAP per rilevare la proteina eIF4A marcata, l'anticorpo anti-Pab1p o l'anticorpo anti-β-actina (o qualsiasi altro bersaglio desiderabile) mediante incubazione notturna della membrana con anticorpo diluito con tampone bloccante (PBS) (diluizione 1:1.000) in un ciclomissatore in una cella frigorifera.
NOTA: la diluizione anticorpale 1:1.000 è un buon punto di partenza. - Lavare la membrana tre volte con 1x Phosphate Buffered Saline, 0.2% v/v Tween 20 (PBST) per 10 minuti ciascuno.
- Sondare la membrana con anticorpi secondari marcati fluorescentemente seguendo le istruzioni del produttore incubando in un ciclomixer a temperatura ambiente per 1 ora.
NOTA: la diluizione degli anticorpi 1:20.000 è un buon punto di partenza. - Lavare la membrana tre volte con 1x PBST per 10 minuti ciascuno. Risciacquare brevemente la membrana con acqua deionizzata e quindi con metanolo assoluto. Asciugare e visualizzare la membrana in un sistema di imaging fluorescente secondo le istruzioni del produttore.
NOTA: La colorazione per altre proteine può essere ottenuta utilizzando anticorpi secondari con coloranti che corrispondono a diversi canali fluorescenti (come nella coppia eIF4A-TAP vs. β-actina qui utilizzata), colorando o spogliando e macchiando la stessa membrana sequenziale o tagliando la membrana da un gel caricato con un modello ripetuto di frazioni e sondando separatamente ogni pezzo con i rispettivi anticorpi (come nell'esempio Pab1p usato qui).
2. Protocollo cellulare di mammifero
- Coltura e fissazione delle cellule di mammifero
- In 2 palloni T-175, far crescere le cellule HEK293 fino al 60%-70% di confluenza nel Modified Eagle Medium di Dulbecco e al 10% v/v fetale bovino siero a 37 °C e 5% v/v anidride carbonica.
NOTA: Il supporto completo è realizzato aggiungendo 55 ml di FBS commerciale in un DMEM acquistato commercialmente con glucosio elevato, contenente L-glutammina, rosso fenolo e bicarbonato di sodio, ma senza HEPES o piruvato di sodio. La conta cellulare per pallone T-175 al 70% di confluenza dovrebbe essere compresa tra 1,7 e 2,0 x 107. - Almeno 3 ore prima del tempo di fissazione desiderato, sostituire il supporto dei palloni T-175 con esattamente 30 ml di supporti completi preriscaldati e sostituire i palloni in un incubatore cellulare.
NOTA: assicurarsi che il fluido fresco sia pipettato sul lato opposto del pallone al monostrato cellulare per evitare il distacco della cella. Tentare di condurre lo scambio di fluidi il più rapidamente possibile, introducendo un disturbo minimo del gas e dell'equilibrio della temperatura. - Una volta che il supporto cellulare è stato sostituito, preparare i tamponi e le sostanze chimiche necessarie per la fissazione. Preparare la soluzione salina tamponata con fosfato (DPBS) di Dulbecco con 50 mM di glicina aggiungendo 10,2 mL di 2,5 M di glicina a una bottiglia da 500 mL di DPBS e mescolando.
- Preparare una bottiglia di DMEM integrata con FBS al 10% come al punto 2.1.1 da utilizzare in condizioni non sterili e un'aliquota di 100 ml di tripsina-EDTA allo 0,25%. Procuratevi una bottiglia aggiuntiva di DPBS commerciale pre-formulata con cloruro di calcio (CaCl2) e cloruro di magnesio (MgCl2).
NOTA: Le soluzioni possono essere conservate a 4 °C per un massimo di 2 settimane. - Preparare una ghiacciaia fino all'orlo con ghiaccio d'acqua frantumato in modo tale che un pallone T-175 possa adattarsi uniformemente sulla parte superiore e tenere nella cappa aspirante insieme ai tamponi preparati, anche su ghiaccio.
NOTA: A causa delle rapide risposte della traduzione a qualsiasi cambiamento ambientale, tutti i tempi tra la rimozione dei palloni cellulari dall'incubatore e l'aggiunta della soluzione di formaldeide devono essere ridotti al minimo. - Per raffreddare a scatto le celle, rimuovere il pallone T-175 dall'incubatore e premerlo saldamente contro il ghiaccio assicurando il massimo contatto superficiale. All'interno della cappa chimica, inclinare il pallone su un lato in modo che il fluido si raccolga sul lato opposto alle celle. Pipetta 168 μL di formaldeide al 37% p/v direttamente nel mezzo aggregato (fino ad una concentrazione finale dello 0,2% p/v). Mescolare immediatamente facendo oscillare delicatamente il pallone avanti e indietro, chiudere e riposizionare il pallone sul ghiaccio, assicurandosi che sia orizzontale e che le celle siano coperte uniformemente.
ATTENZIONE: La formaldeide è una sostanza nociva con potenziali effetti avversi a lungo termine e anche irritante sia per il sistema respiratorio che per la pelle. Dovrebbe essere maneggiato solo in una cappa aspirante chimica adatta. I contenitori di formaldeide devono essere sempre sigillati all'esterno della cappa aspirante.
NOTA: Assicurarsi che la formaldeide venga aggiunta direttamente nel supporto cellulare e non alla parete del pallone. Il passaggio 2.1.6 dovrebbe richiedere meno di 1 minuto. - Incubare i palloni sul ghiaccio per altri 10 minuti. Versare il supporto in un contenitore di rifiuti appropriato attraverso il lato del pallone opposto alle celle.
- Utilizzando una stripette, pipettare in 30 ml di Fosfato Tamponato Soluzione Salina di Dulbecco senza ioni calcio e magnesio e contenente inoltre 50 mM di glicina, delicatamente sul lato opposto alle cellule. Mescolare dondolando il pallone; riportare il pallone in posizione orizzontale e incubare per altri 10 minuti sul ghiaccio.
- Versare la soluzione attraverso il lato del matraccio opposto alle celle e aggiungere delicatamente 7 mL della soluzione standard di tripsina-EDTA allo 0,25% p/v per staccare e risospesso le cellule. Incubare il matraccio a temperatura ambiente per 5-10 min.
NOTA: Assicurarsi che la soluzione di tripsina-EDTA copra tutte le cellule in modo uniforme. Utilizzare l'inclinazione e il dondolo delicati periodici per promuovere il distacco delle cellule. - Riposizionare il pallone verticalmente e con una stripette raccogliere le celle staccate lavando delicatamente le celle rimanenti dalle pareti del pallone. Trasferire la sospensione in un tubo da 50 ml posto sul ghiaccio.
NOTA: le celle fisse possono diventare più fragili; non pipettare intensamente o più di quanto è necessario per staccare le cellule dalla parete del pallone. - Integrare immediatamente la sospensione cellulare raccolta con 20 ml di fluido completo (il mezzo ghiacciato non sterile con il 10% di FBS) e mescolare capovolgendo delicatamente il tubo.
NOTA: Il terreno di coltura cellulare completo (incluso il 10% FBS) viene aggiunto per neutralizzare la tripsina, prevenendo ulteriori danni alle membrane cellulari e la disintegrazione cellulare. - Pellet le celle centrifugando il tubo a 100 x g per 5 min e 4 °C. Il pellet cellulare deve essere chiaramente visibile.
- Versare il fluido e risospesciare delicatamente il pellet cellulare in 10 ml di DPBS ghiacciato con Ca2+,Mg2+e senza glicina.
- Ripetere il passaggio 2.1.12.
- Versare il tampone di lavaggio e risospesciare delicatamente il pellet cellulare in 800 μL di DPBS ghiacciato con Ca2+,Mg2+,senza glicina, su ghiaccio. Trasferire le cellule risospese in un nuovo tubo microcentrifuga da 1,5 mL a basso legame proteico.
- Centrifugare il tubo a 100 x g per 3 min e 4 °C. Scartare con attenzione il surnatante usando un pipettor da 1 mL. In questa fase, il pellet cellulare può essere congelato a -80 °C o procedere alla fase di lisi cellulare.
NOTA: i pellet di celle congelati possono essere conservati a -80 °C fino a 1 anno. Abbiamo scoperto che il congelamento del pellet cellulare facilita la successiva lisi e raccomandiamo il congelamento anche se non è prevista una conservazione a lungo termine.
- In 2 palloni T-175, far crescere le cellule HEK293 fino al 60%-70% di confluenza nel Modified Eagle Medium di Dulbecco e al 10% v/v fetale bovino siero a 37 °C e 5% v/v anidride carbonica.
- Interruzione delle cellule di mammifero e raccolta di citosol
- In un armadio di biosicurezza, aggiungere 300 μL del tampone di lisi a base di detergente non ionico e non in dentaturante e 7 μL di inibitore della RNasi da 40 U/μL. Mescolare bene pipettando usando una punta da 1 mL.
- Attaccare con attenzione un ago da 25 G a una siringa da 1-3 ml e pipettare vigorosamente la miscela, utilizzando almeno sette prese lente verso l'alto e corse di scarico veloci verso il basso.
- Gettare la siringa e l'ago in un contenitore per oggetti taglienti e ripetere la procedura utilizzando una siringa da 0,3 mL dotata di un ago da 31 G.
- Gettare la siringa e l'ago in un contenitore per taglienti. Centrifugare i tubi a 4 °C, 12.000 x g per 5 minuti per pellettizzare i detriti cellulari.
- Trasferire il surnatante in un nuovo tubo microcentrifuga da 1,5 mL a basso legame proteico. Conservare sia i detriti cellulari (a scopo di controllo) che il lisato cellulare chiarificato risultante a -80 °C.
NOTA: la densità ottica del lisato varia tra 25-30 AU260 quando vengono combinati due palloni T-175 e seguono i volumi raccomandati. I lisati e i detriti cellulari possono essere conservati a -80 °C fino a 1 anno.
- Separazione dei complessi (poli)ribosomiali fissi dalle frazioni non tradotte del citosol
- Preparare gradienti lineari di saccarosio 15%-45% p/v in tubi di polipropilene a parete sottile da 13 ml, 14 x 89 mm, utilizzando il metodo di congelamento-disgelo generalmente descritto nella fase 1.3.1 del protocollo di lievito, ma utilizzando il tampone 2 (Figura 2a).
NOTA: Scongelare le pendenze durante la notte in una cella frigorifera a 4 °C la notte prima del frazionamento. - Caricare 150-250 (massimo 300) μL del lisato cellulare dal precedente passaggio 2.2.5 sui gradienti bilanciati. Conservare il lisato rimanente a -80 °C e utilizzarlo a scopo di controllo.
NOTA: Qui viene fornito un esempio di segregazione basata sulla sedimentazione in frazioni SSU polisomiche, ribosomiali e "libere". Fare riferimento alla tabella supplementare 1 fornita per un approccio alternativo. - Ultracentrifugare i tubi in un rotore a benna oscillante di medio volume a 4 °C, forza g media 178.305 x g (fattore k 143,9) per 1 ora e 45 min.
- 30 minuti prima del completamento dello spin, impostare e basare il frazionatore di gradiente, come descritto nei passaggi del protocollo di lievito 1.4.7-1.4.9.
- Frazionare i gradienti generalmente come descritto nelle fasi del protocollo del lievito 1.4.10-1.4.11.
NOTA: questo passaggio separerà le frazioni SSU polisomiali, ribosomiali e "libere". Le frazioni polisomatiche possono essere utilizzate in esperimenti di profilazione dei polisomi. - Trasferire immediatamente le frazioni raccolte su ghiaccio e, se non ulteriormente lavorate, conservare a -80°C fino a 6 mesi.
NOTA: se la modifica del tubo del collettore di frazione è sincronizzata con l'identificazione e la segregazione della frazione in linea, si consiglia di utilizzare frazioni fino a 800 μL (tempo di raccolta di 32 s per frazione a 1,5 ml/min). Se il frazionamento viene eseguito senza utilizzare la lettura dell'assorbanza in linea, si consiglia di utilizzare frazioni da 250-500 μL (10-20 s per frazione a 1,5 ml/min). Dopo la separazione, le frazioni possono essere utilizzate per l'immunopurificazione, la microscopia elettronica, la denaturazione di PAGE e il western blotting immediatamente, oppure sottoposte a inversione di reticolazione per successive analisi di RNA e/o proteomica.
- Preparare gradienti lineari di saccarosio 15%-45% p/v in tubi di polipropilene a parete sottile da 13 ml, 14 x 89 mm, utilizzando il metodo di congelamento-disgelo generalmente descritto nella fase 1.3.1 del protocollo di lievito, ma utilizzando il tampone 2 (Figura 2a).
Risultati
I complessi traslazionali sono sensibili alla composizione ionica dei tamponi, che è particolarmente importante durante l'ultracentrifugazione in cui vengono valutate le proprietà di sedimentazione. Abbiamo quindi testato diversi tamponi di sedimentazione utilizzando lisato chiarificato estratto da materiale di lievito macinato non fisso, al fine di selezionare le condizioni più adatte a risolvere complessi traslazionali e subunità ribosomiali separate (SSU, LSU), monosomi (RS) e polisomi attraverso il gradiente. Tutti i tamponi erano basati sulla composizione del nucleo contenente 25 mM HEPES-KOH pH 7,6 e 2 mM DTT. Le concentrazioni di KCl, MgCl2, CaCl2ed EDTA sono state ulteriormente modificate attraverso i tamponi (Figura 2a), e questi componenti sono stati aggiunti ai lisati prima del carico del gradiente e ai tamponi del gradiente del saccarosio prima della colata del gradiente, di conseguenza.
Nei buffer sono stati ottenuti 1 e 2 complessi traslazionali ben risolti. Il tampone 1 ha determinato una separazione leggermente migliore delle piccole subunità ribosomiali (SSU) (Figura 2a). L'omissione di MgCl2 e l'aggiunta di EDTA (tamponi 3,4) hanno causato la perdita delle elevate proprietà di sedimentazione per la maggior parte dei polisomi e probabilmente il loro parziale disassemblaggio (Figura 2a). Mentre l'aggiunta di 2,5 mM di CaCl2 ha determinato picchi polisomiali leggermente più omogenei, il miglioramento è stato marginale e la quantità complessiva del materiale polisomiale è diminuita in questo caso (Figura 2a) rispetto ai tamponi 1 e 2. Abbiamo quindi selezionato il buffer 1 come buffer di lavoro preferito.
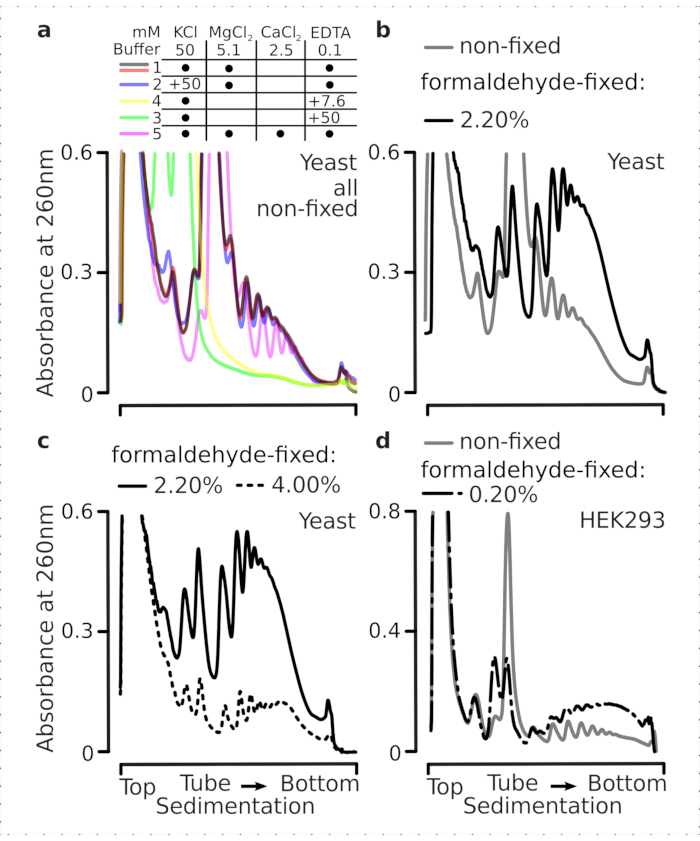
Figura 2: Condizioni tampone per l'estrazione di complessi traslazionali e valutazione dell'effetto stabilizzante della fissazione. Sono mostrati profili di assorbanza UV raccolti a 260 nm per il lisato totale di cellule di lievito separato in gradienti di saccarosio 10%-40% p/v. a) Effetti dei sali mono e bivalenti e del sequestro degli ioni di magnesio sulla sedimentazione di materiale estratto da cellule di lievito non fisse. Le linee rosse e grigie rappresentano una replica tipica. (b,c) Confronto di lisati derivati da cellule di lievito non fisse (linea grigia), 2,2% (linea nera) e 4,4% (linea tratteggiata nera) con cellule di lievito fissate alla formaldeide. (d) Stabilizzazione dei polisomi mediante lo 0,2% p/v ottimizzato di fissazione della formaldeide (linea tratteggiata e tratteggiata nera) delle celle HEK 293T, rispetto al materiale delle stesse celle non fisse (linea grigia). Fare clic qui per visualizzare una versione più grande di questa figura.
Abbiamo quindi verificato l'effetto della stabilizzazione polisomiale mediante fissazione con diverse concentrazioni di formaldeide. Utilizzando lo stesso materiale cellulare, i buffer, la gestione delle cellule e gli approcci di temporizzazione, abbiamo confrontato il materiale estratto da celle non fisse e le celle fissate con il 2,2% e il 4% p/v di formaldeide (Figura 2b,c). Abbiamo scoperto che il 2,2% p/v di formaldeide era più adatto per la fissazione in quanto mentre conservava in modo eccellente i polisomi come si può giudicare dal rapporto polisoma-monosoma (Figura 2b), non riduceva la resa complessiva del materiale ribosomiale rispetto al 4% p/ v di formaldeide, che mostrava chiari segni di sovra-fissazione (Figura 2c).
Per il materiale derivato dalle cellule di mammifero, a causa del maggiore rapporto tampone-volume cellulare di lisi richiesto dall'estrazione a base di detergente, è stato utilizzato il tampone 2 (Figura 2a). Questo ha prodotto complessi traslazionali ben risolti dopo la sedimentazione in gradienti di saccarosio (Figura 2d). In particolare, è stata utilizzata una concentrazione molto più bassa di formaldeide dello 0,2% p/v, poiché concentrazioni più elevate hanno comportato una sostanziale perdita di materiale polisomiale e ribosomiale (dati non mostrati). Analogamente ai risultati ottenuti con le cellule di lievito, il materiale stabilizzato con reticolazione ha dimostrato una migliore conservazione dei polisomi e un rapporto polisoma-monosoma più elevato (Figura 2d).
Abbiamo poi testato se le condizioni di fissazione della formaldeide selezionate sono abbastanza efficienti da stabilizzare attivamente l'mRNA tradotto all'interno delle frazioni polisomiali a seguito della reticolazione, e la migliore resa polisomiale non è solo una conseguenza dell'inibizione della funzione enzimatica e della progressione dell'allungamento della traduzione. Abbiamo usato EDTA e sale monovalente alto (KCl) per destabilizzare polisomi e ribosomi. Questi reagenti sono stati aggiunti ai lisati di cellule di lievito chiarificati e inclusi in tutti i tamponi successivi e gradienti di saccarosio sulla composizione del tampone 1, rispettivamente.
Infatti, 15 mM EDTA hanno mostrato un minore effetto destabilizzante sulle frazioni polisomiali derivate dalle cellule fisse (Figura 3a), confermando che i complessi reticolati sono più robusti. Gli effetti destabilizzanti dell'EDTA possono essere in qualche modo superati aumentando la concentrazione di formaldeide, poiché il materiale del 4% p/v di cellule fissate alla formaldeide resisteva a dispiegarsi meglio (Figura 3a). Tuttavia, l'aumento della concentrazione di EDTA a 50 mM ha comportato la destabilizzazione della maggior parte dei complessi traslazionali in condizioni sia fisse che non fisse, come si può dedurre dalla sedimentazione più lenta del materiale e dall'assenza di picchi ben modellati (Figura 3b). Ciò può essere spiegato dal parziale dispiegamento delle strutture e dalla perdita complessiva di compattezza, piuttosto che dalla completa dissociazione dei componenti polisomiali dall'mRNA. Anche in questo caso, il materiale reticolato ha dimostrato una sedimentazione più rapida (Figura 3b).
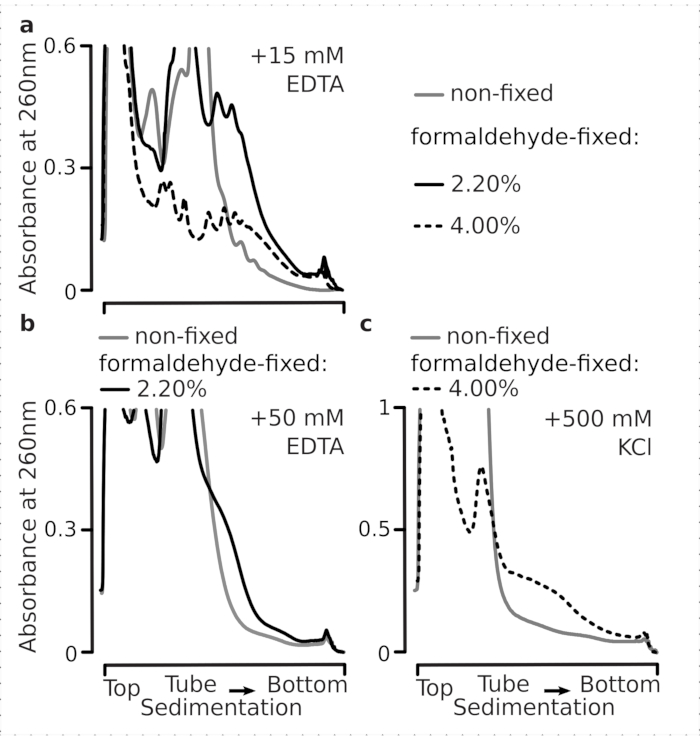
Figura 3: Effetti della fissazione della formaldeide del lievito in vivo sulla stabilità dei polisomi. Il buffer 1 (vedi testo e Figura 2a)è stato utilizzato in tutti gli esperimenti. Tipo di dati e plottaggio come descritto nella legenda della Figura 2. (a) Confronto dell'aggiunta di 15 mM EDTA ai lisati cellulari e successivi tamponi sulla stabilità dei polisomi derivati da cellule non fisse (linea grigia), 2,2% (linea nera) e 4% (linea tratteggiata nera) con/v di cellule fissate alla formaldeide. b) comea), ma per l'aggiunta di 50 mM di EDTA ed escluso il 4% p/v di cellule fissate con formaldeide. c) comea), ma per l'aggiunta di 500 mM KCl ed escluso il 2,2% p/v di cellule fissate alla formaldeide. Fare clic qui per visualizzare una versione più grande di questa figura.
Analogamente agli effetti EDTA, a 500 mM KCl, abbiamo riscontrato un importante miglioramento della stabilità con il 4% p/v di fissazione della formaldeide (Figura 3c). L'apparente perdita di compattezza in questo caso può anche essere spiegata dal distacco parziale dei costituenti dei complessi ribosomiali, piuttosto che dalla loro completa dissociazione dall'RNA. Nel complesso, i polisomi derivati da cellule fissate alla formaldeide hanno dimostrato una maggiore resistenza allo sviluppo e alla destabilizzazione strutturale, coerente con la formazione di ulteriori legami covalenti all'interno di questi complessi.
Durante le condizioni di crescita stimolante, gli mRNA possono essere rapidamente avviati con conseguente accumulo di più ribosomi sulle stesse molecole di mRNA, che formano strutture note come poliribosomi o polisomi. I polisomi possono essere separati mediante ultracentrifugazione in gradienti di saccarosio, dove sedimentano in base al loro ordine (numero di ribosomi attaccati contemporaneamente sull'mRNA). Quando la traduzione viene soppressa, i ribosomi non riescono a impegnarsi in un altro ciclo di traduzione abbastanza presto, con conseguente (parziale) "disassemblaggio" dei polisomi, che viene esibito come un trasferimento modale verso i polisomi di ordine inferiore e l'accumulo di monosomi4,26.
Un modello di risposta traslazionale che può essere visualizzato sul livello di distribuzione dell'ordine dei polisomi può essere fornito dalla fame di glucosio. L'esaurimento del glucosio provoca uno degli effetti inibitori traslazionali più drammatici e rapidi sul lievito1,3,40. Studi precedenti hanno evidenziato che entro 1 minuto dalla deplezione del glucosio, dalla perdita di polisomi, dall'accumulo di monosomi e dall'inibizionedell'iniziodella traduzione possono verificarsi 4 . Entro 5 minuti dal reintegro di glucosio, la traduzione viene rapidamente ripristinata con evidente aumento dei polisomi3,4. È stato anche osservato che la traduzione è stata inibita quando le cellule sono state esposte a mezzi contenenti glucosio dello 0,5% (p / v) o inferiore e non è stato osservato alcun effetto nei livelli di glucosio dello 0,6% (p / v) o superiore.
Abbiamo quindi voluto determinare se le nostre condizioni di fissazione sono adatte alla conservazione delle differenze traslazionali all'interno della dinamica della risposta allo stress glucoso, come può essere valutato dal rapporto polisoma-monosoma. Abbiamo confrontato il materiale delle cellule cresciute in fase media-esponenziale su glucosio alto (2,00% p/v aggiunto) con quelli trasferiti per 10 minuti in media con nessun o basso aggiunto (0,00% o 0,25% p/v, rispettivamente) di glucosio. La fissazione è stata eseguita utilizzando il 2,2% p/v di formaldeide in parallelo nel controllo (non affamato; sostituzione rapida del fluido con lo stesso mezzo standard contenente il 2% p/v di glucosio aggiunto, seguito da incubazione per 10 minuti e fissazione) e 10 minuti affamati (sostituzione rapida del fluido con lo stesso mezzo ma basso 0,25 w/v o senza glucosio aggiunto, seguita da incubazione per 10 minuti e fissazione) cellule.
Coerentemente con i risultati precedenti, abbiamo osservato che le cellule di lievito sopprimono pesantemente la traduzione dopo lo stress da fame di glucosio (Figura 4a). Sia le condizioni di non aggiunta che quelle di basso glucosio hanno indotto lo smontaggio dei polisomi, con leggermente ma evidentemente più polisomi trattenuti nel caso di glucosio a basso contenuto aggiunto. Pertanto, la risposta di rimozione del glucosio del lievito potrebbe non essere di tipo all-on o all-off e viene gradualmente sintonizzata. Affermando le aspettative per l'azione stabilizzante della reticolazione della formaldeide, il materiale polisomiale delle cellule fisse ha dimostrato una maggiore distinzione tra le cellule affamate e non affamate, probabilmente preservando un intervallo dinamico più elevato della risposta (Figura 4b). Curiosamente, nel caso del materiale proveniente dalle cellule fisse, una bassa concentrazione di glucosio aggiunto ha portato all'abbondanza polisomiale specifica che è molto meglio differenziata dalla condizione di glucosio senza aggiunta, rispetto alle cellule non fisse (Figura 4a). Questa è una forte indicazione dell'idoneità dell'approccio di fissazione della formaldeide nel preservare e catturare differenze relativamente piccole e transitorie nell'equilibrio di processi altamente dinamici, come durante le risposte traslazionali.

Figura 4: Catturare rapidi cambiamenti nella traduzione del lievito dopo la fame di glucosio. Il buffer 1 (vedi testo e Figura 2a)è stato utilizzato in tutti gli esperimenti. Tipo di dati e plottaggio come descritto nella legenda della Figura 2. a)Lisati cellulari ottenuti da cellule di lievito non affamate (linea grigia), con glucosio affamato limitato (0,25% p/v di glucosio aggiunto per 10 minuti; linea marrone) e con glucosio impoverito di glucosio (senza glucosio aggiunto per 10 minuti; linea rossa). b) come a ), ma peril2,2% p/v di cellule fissate con formaldeide. Fare clic qui per visualizzare una versione più grande di questa figura.
Il monitoraggio dello stato traslazionale da parte dei ribosomi associati alla traduzione attiva dell'mRNA utilizzando la sedimentazione a gradiente di saccarosio ("profilo dei polisomi") è una tecnica ampiamente applicata26,27,28. In combinazione con l'analisi quantitativa dei microarray e più recentemente con il sequenziamento ad alto rendimento28,44,la profilazione dei polisomi fornisce informazioni sugli mRNA associati ai ribosomi a livello di trascrittoma. Con diverse ipotesi, è stato tradizionalmente sostenuto nel campo della ricerca sulla biosintesi proteica che la presenza polisomiale è un'indicazione di coinvolgimento attivo nella traduzione dei rispettivi mRNA. Un'ulteriore conclusione è spesso (ma non sempre) giustificata, che più ribosomi sono presenti su un mRNA di una data lunghezza (più alto è l'ordine dei polisomi), più attivamente l'mRNA è coinvolto nella traduzione. Pertanto, separare la frazione polisomiale dal resto del materiale può essere utile dal punto di vista dell'isolamento dell'RNA tradotto attivamente. All'interno degli approcci di profilazione dell'impronta, e in particolare TCP-seq10,38,39 che genera una popolazione separata delle SSU liberate derivate dai complessi di codone di scansione, avvio e arresto, può essere inoltre perspicace rimuovere le subunità ribosomiali che non co-sedimentano con i monosomi o polisomi completi.
Abbiamo quindi impiegato la separazione degli mRNP "non tradotti" come le SSU libere (mRNA legato a singoli SSU o SSU senza mRNA collegato) dal pool di mRNA "che traducono attivamente". Per raggiungere questo obiettivo, abbiamo ipotizzato che gli mRNA coinvolti nelle interazioni con uno (mono-) o diversi ribosomi (polisomi) possano essere tradotti attivamente. Tali complessi possono essere separati dagli altri dal loro coefficiente di sedimentazione più elevato. Abbiamo anche suggerito di separare il pool di mRNA "tradotto attivamente" in un cuscino di saccarosio (50% p / v di saccarosio) invece di pellettare direttamente il materiale sulla parete del tubo. La centrifugazione dei complessi a sedimentazione rapida nel cuscino ha permesso di monitorare la separazione utilizzando la lettura del profilo di assorbanza e di ottenere una maggiore resa del materiale solubilizzato, non aggregato e non denaturato, rispetto alla pellettizzazione e alla risisolizzazione10,38.
Nel complesso, per purificare le singole SSU, ribosomi, disomi e polisomi potenzialmente compatti di ordine superiore, i lisati chiarificati fissi sono stati sottoposti a un processo di ultracentrifugazione a due stadi (Figura 5). Nel primo gradiente di saccarosio, l'ultracentrifugazione ha portato a SSU e LSU libere separate nella parte superiore (10%-20% p/v di saccarosio) del gradiente, mentre il pool tradotto reticolato comprendente polisomi e mRNA associati a un ribosoma completo era concentrato nella parte inferiore (50% p/v di saccarosio) del gradiente (Figura 5a ). Il 50% p/v inferiore dello strato di saccarosio contenente il pool di mRNA tradotto è stato quindi concentrato e il suo RNA digerito con RNasi I, seguito da una seconda ultracentrifugazione a gradiente di saccarosio per ottenere disomi resistenti alla SSU, LSU, RS, RNasi (DS) separati e frazione minore di polisomi resistenti alla nucleasi di ordine superiore (Figura 5b). La colorazione negativa con acetato di uranile e l'imaging al microscopio elettronico a trasmissione hanno confermato l'identità dei complessi isolati in ogni fase di sedimentazione (Figura 5).

Figura 5: Isolamento delle frazioni totali di RNA tradotte lontano dall'RNA non tradotto. (a,c) Schematico (a sinistra) e i rispettivi risultati rappresentativi (a destra; tipo di dati e grafico come descritto nella legenda figura 2) di (a) prima separazione discontinua del gradiente di saccarosio delle frazioni di citosol non tradotte, comprese le SSU libere e il pool di mRNA tradotto identificato mediante co-sedimentazione con ribosomi e polisomi, e (c) separazione dei singoli complessi ribosomiali liberati dal pool di mRNA tradotti mediante digestione controllata della RNasi I e ultracentrifugazione attraverso un secondo gradiente lineare di saccarosio in frazioni SSU, LSU, ribosomiali (RS) e disomali resistenti alla nucleasi (DS). Sono state incluse quantità elevate (15 AU260)e basse(8 AU 260) del materiale digerito non affamato per dimostrare la possibilità di aumentare i carichi di ultracentrifugazione quando le frazioni minori sono di interesse. Possono anche essere identificati polisomi resistenti alla nucleasi di ordine superiore (ad esempio, trisomi negli esempi forniti). (b,d) Immagini TEM rappresentative di frazioni in contrasto con acetato di uranile da (a,c), rispettivamente come etichettato. Fare clic qui per visualizzare una versione più grande di questa figura.
Al fine di verificare l'idoneità del regime di fissazione per la ritenzione di proteine transitorie associate ai ribosomi (in particolare, eIF), abbiamo testato la co-sedimentazione di eIF4A, un eIF labile legato dinamicamente al ribosoma, attraverso le frazioni ribosomiali. Abbiamo sfruttato il ceppo di lievito marcato eIF4A Tandem Affinity Purification (TAP) (TIF1-TAP) e studiato la presenza di eIF4A nel materiale derivato dalle cellule fisse rispetto a quelle non fisse utilizzando l'anticorpo anti-TAP, rispetto all'abbondanza di Pab1p come ulteriore controllo di legame all'RNA, utilizzando SDS-PAGE seguito da western blotting (Figura 6).
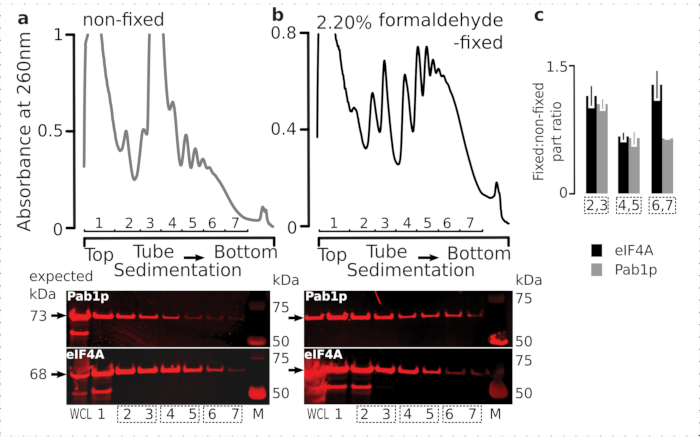
Figura 6: Stabilizzazione di proteine transitorie nei complessi traslazionali su fissazione in vivo di formaldeide. (a,b) ( topplots) Lisato a cellule intere (WCL) di (a) non fisso e (b) 2,2% di cellule di lievito eIF4A-TAP fissate alla formaldeide separate per ultracentrifugazione e visualizzate come descritto nella legenda figura 2. (trame in basso) Imaging Western-blot delle rispettive frazioni di gradiente di saccarosio dopo la separazione del materiale analizzato nei gradienti corrispondenti (top plots) e WCL come controllo. c)Rapporto medio tra l'abbondanza di eIF4A o Pab1p nelle frazioni di materiale fisso e non fisso. Le proporzioni relative (normalizzate al segnale di tutte le 2-7 frazioni) di eIF4A (barre nere) e Pab1p (barre grigie) sono state calcolate su 2,3 (SSU, LSU), 4,5 (RS, polisomi leggeri) e 6,7 (polisomi pesanti) dai dati di (a,b) (grafici inferiori) e il loro rapporto fisso /non fisso preso. Le barre di errore indicano la deviazione standard del rapporto dalla media con le frazioni raggruppate (caselle tratteggiate) trattate come repliche. Fare clic qui per visualizzare una versione più grande di questa figura.
Coerentemente con la loro elevata abbondanza nelle cellule, abbiamo osservato un'alta intensità del segnale sia dalle proteine nel lisato cellulare intero (WCL) che da frazioni a sedimentazione più lenta derivate da cellule non fisse(Figura 6a,pannello inferiore). Abbiamo anche rilevato notevoli quantità di queste proteine nel WCL derivate dalle cellule fisse e rassicurando l'efficienza dell'estrazione del materiale reticolato e l'assenza di perdite impreviste(Figura 6b,pannello inferiore). Tuttavia, a differenza delle cellule non fisse, il materiale delle cellule fisse ha dimostrato un'elevata presenza relativa di eIF4A nelle frazioni ribosomiali a sedimentazione più rapida, rispetto a Pab1p (Figura 6c). Questo risultato suggerisce che eIF4A rimane più saldamente associato ai polisomi nel materiale reticolato alla formaldeide.
Dopo aver confermato l'effetto stabilizzante positivo e specifico della reticolazione sulla presenza di eIF4A nelle frazioni ribosomiali, abbiamo utilizzato il materiale fisso del ceppo di lievito eIF4A-tagged (TIF1-TAP) per catturare e arricchire i complessi contenenti eIF4A mediante purificazione di affinità con perline magnetiche IgG. Abbiamo frazioni WCL arricchite di affinità, SSU libero e polisomiale (pool di mRNA tradotto) dopo la prima sedimentazione attraverso gradiente di saccarosio (ad esempio, sezione 1.3 del protocollo del lievito), nonché frazioni SSU, LSU e RS dalla seconda sedimentazione dopo lo smontaggio del pool tradotto in singoli complessi con RNasi I (ad esempio, sezione 1.4 del protocollo del lievito) (Figura 7 ). In tutti i casi, ad eccezione della frazione LSU, siamo stati in grado di osservare l'arricchimento selettivo dell'eIF4A nelle frazioni purificate (eluato, E), rispetto alla presenza di β-actina nel materiale sorgente (input, I) (Figura 7).
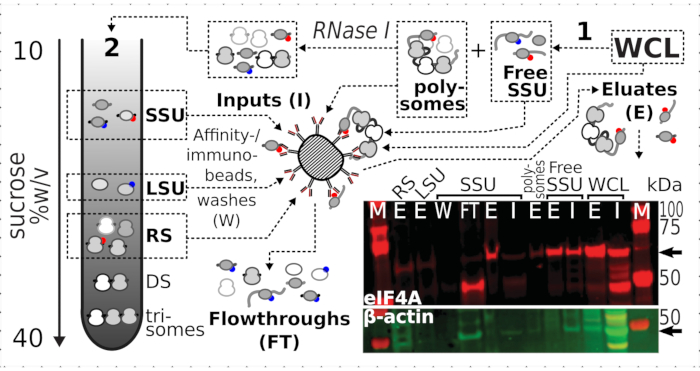
Figura 7: Immunopurificazione selettiva di complessi traslazionali stabilizzati con formaldeide in vivo mediante eIF4A associata transitoriamente. Lo schema illustra la fonte di diversi complessi traslazionali e l'epitopo eIF4A, incluso il WCL chiarificato non frazionato delle cellule di lievito eIF4A-TAP; SSU libere e pool di RNA tradotto (polisomi) segregati nella prima ultracentrifugazione; Frazioni SSU, LSU e RS liberate dall'RNA tradotto dalla digestione della RNasi I e segregate mediante seconda ultracentrifugazione (vedi testo). L'immagine Western blot fornisce una visualizzazione dell'abbondanza di eIF4A nelle frazioni rispetto all'abbondanza di controllo della β-actina macchiata contemporaneamente. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella complementare 1. Fare clic qui per scaricare questa tabella.
Discussione
La fissazione della formaldeide è un metodo conveniente e popolare per ottenere una rapida reticolazione in vivo delle biomolecole10,36,45,46,47,48. Rispetto agli altri potenziali bersagli della biomolecola, la cattura di successo dei complessi traslazionali richiede una fissazione immediata durante il raffreddamento istantaneo delle cellule o di altro materiale. Senza la stabilizzazione non ritardata, c'è un potenziale per i diversi processi relativi alla traduzione di continuare, spostando la distribuzione complessa lontano dallo stato in vivo imperturbabile 49. Rispetto agli altri metodi di arresto traslazionale e stabilizzazione del complesso ribosomiale, la rapidità dell'azione della formaldeide attraverso le membrane cellulari e la natura indiscriminata dei legami incrociati promettono la conservazione della massima diversità degli intermedi complessi di traduzione più vicini ai loro stati nativamente distribuiti50.
L'approccio qui presentato è stato stabilito e ottimizzato sia nel lievito che nelle cellule di mammifero, e i metodi sono stati ora derivati da altri gruppi per l'uso su materiale biologico più diversificato, come in vertebrati interi (ad esempio, embrioni di zebrafish)10,38,39,49,51,52 . Sebbene questi lavori rafforzino collettivamente la versatilità e l'ampia applicabilità dell'approccio, la rapida reticolazione formaldeide dei complessi traslazionali può essere considerata alquanto difficile da trasporre in nuovi tipi di materiale biologico a causa della necessità di ottimizzazioni e aggiustamenti.
Un requisito fondamentale per il successo del metodo è la ri-ottimizzazione della concentrazione della formaldeide e la tecnica di raccolta e distruzione cellulare. Le cellule di lievito meno permeabili, piccole e rotonde richiedono una concentrazione di formaldeide molto più alta (almeno 10 volte) e l'interruzione fisica delle cellule fisse. Al contrario, le cellule di mammifero aderenti grandi e appiattite in coltura possono essere facilmente sovra-fissate e richiedono una manipolazione delicata al momento della fissazione, mentre l'estrazione dei complessi fissi può essere eseguita chimicamente con interruzione della membrana utilizzando detergenti. La reticolazione insufficiente può consentire agli intermedi meno stabili o più di breve durata di dissociarsi o di fuoriuscire in uno stato successivo. L'over-crosslinking può influenzare negativamente la capacità di isolare e studiare le frazioni ribosomiali e può creare pregiudizi selettivi come l'esaurimento più profondo di complessi pesanti. Nella nostra osservazione, anche piccole alterazioni, come il tipo di cellule umane aderenti utilizzate, possono influenzare la resa dei complessi reticolati recuperati e possono richiedere una ri-ottimizzazione del regime di reticolazione. Possiamo anche anticipare che le cellule con proprietà di permeabilità sostanzialmente diverse, come le cellule vegetali, richiederanno un'ulteriore ottimizzazione estesa delle condizioni di fissazione52. Tuttavia, è difficile immaginare un tipo di materiale biologico che sarebbe del tutto incompatibile con l'approccio.
Una considerazione pertinente al protocollo di fissazione dei mammiferi è la densità e la quantità di materiale cellulare utilizzato come input. Si raccomanda di far crescere continuamente le cellule senza riseminare o altre perturbazioni per almeno 2 giorni per evitare influenze esterne sulle dinamiche di traduzione cellulare. Applicabile per la maggior parte dei tipi di cellule, ma per la maggior parte delle cellule aderenti costantemente raggiunti livelli di confluenza non superiori al 70% garantirà l'assenza di importanti effetti di inibizione del contatto che possono influenzare negativamente e imprevedibilmente i tassi di traduzione.
Un'altra caratteristica interessante, e potenzialmente conveniente, della fissazione della formaldeide derivante dalla sua reattività indiscriminata è l'effetto di stabilizzazione sui complessi traslazionali nei sistemi di tassonomia mista. I complessi batterici, e ancor più traslazionali di mitocondri, cloroplasti e diversi parassiti intracellulari, sono stati notoriamente difficili da colpire con specifici inibitori della traduzione. Al contrario, nei dati TCP-seq, le impronte mappate al mitotranscriptome sono facilmente osservabili nei dati38,39,50. Un interessante sviluppo successivo potrebbe essere l'uso dell'approccio per studiare la traduzione in intere microcomunità, come nel suolo, nell'acqua o nei campioni di intestino, dove un arresto traslazionale rapido affidabile e una stabilizzazione complessa con qualsiasi altro mezzo sarebbero problematici.
Va anche detto che per il materiale più complicato (come i tessuti duri e / o voluminosi), nulla impedisce l'uso della stabilizzazione della formaldeide immediatamente dopo la distruzione cellulare e l'omogeneizzazione del materiale. Questo approccio è già frequentemente impiegato per rimuovere il ritardo di ingresso cellulare quando si stabilizzano complessi traslazionali con specifici inibitori di piccole molecole33, 53,54,55. Dato che la fissazione della formaldeide è stata tradizionalmente utilizzata con ottimi risultati per la stabilizzazione di campioni ex vivo/in vitro in applicazioni come la microscopia elettronica45,56,57,58,possiamo aspettarci effetti ancora meno negativi in questo caso, in particolare quelli associati alla scarsa estrazione dei complessi traslazionali dalle cellule accuratamente fissate.
I nostri risultati confermano l'usabilità della rapida fissazione della formaldeide per stabilizzare complessi altamente transitori, come quelli che includono eIF4A. È interessante notare che, a differenza dei mammiferi, il lievito eIF4A è molto più debolmente associato al complesso legante il cappuccio eIF4F e, di conseguenza, ai complessi traslazionali in generale. eIF4A viene solitamente perso durante qualsiasi purificazione estensiva del materiale ribosomiale nel lievito29,59,60,61,62,63. Tuttavia, nel materiale di lievito fisso in vivo,è possibile ottenere un arricchimento affidabile di eIF4A in tutte le frazioni di complessi traslazionali in cui la sua presenza sarebbe prevista. I dati Sel-TCP-seq precedentemente pubblicati hanno dimostrato l'arricchimento di eIF2 ed eIF3 che si associano più fortemente ai ribosomi (ma hanno anche rivelato l'assemblaggio del complesso proteico co-traduzionale che si verifica transitoriamente)39. Pertanto, il metodo è adatto per il rilevamento di entrambi i costituenti attaccati più forti e più deboli dei complessi traslazionali.
Per riassumere, abbiamo presentato un approccio utile per ottenere informazioni principalmente sui cambiamenti che si verificano nella fase di inizio della traduzione e quando è richiesta una distribuzione ribosomiale minimamente perturbata sull'mRNA. È importante sottolineare che l'approccio è adatto per la stabilizzazione di componenti relativamente labili e dinamiche di complessi traslazionali, come eIF4A, e può essere utilizzato ampiamente soggetto alle necessarie ottimizzazioni. Abbiamo anche fornito prove dell'utilità della fissazione della formaldeide negli scenari di rapido cambiamento dinamico della traduzione, aprendo aree di indagine come le risposte cellulari veloci ai cambiamenti ambientali o alle condizioni di stress.
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
Questo lavoro è stato supportato dalla sovvenzione Australian Research Council Discovery Project (DP180100111 a T.P. e N.E.S),National Health and Medical Research Council Investigator Grant (GNT1175388 a N.E.S.) e Research Fellowship (APP1135928 a T.P.). Gli autori riconoscono le strutture di Microscopy Australia presso il Centre for Advanced Microscopy, Australian National University, una struttura finanziata dall'Università e dal governo federale.
Materiali
| Name | Company | Catalog Number | Comments |
| Yeast extract | Merck, Sigma-Aldrich | 70161 | |
| Peptone | Merck, Sigma-Aldrich | 70178 | |
| D-Glucose (Dextrose) | Merck, Sigma-Aldrich | 49139 | |
| Adenine sulphate | Amresco | 0607-50G | |
| Formaldehyde solution | Merck Sigma-Aldrich | F11635-500ML | ACS reagent, 37 wt. % in H2O, contains 10-15% Methanol as stabiliser (to prevent polymerisation) |
| RNaseOUT™ Recombinant Ribonuclease Inhibitor | Invitrogen™ byThermo Fischer Scientific | 10777019 | |
| cOmplete™, EDTA-free Protease Inhibitor Cocktail | COEDTAF-RO Roche by Merck | 11873580001 | |
| Magnesium chloride solution | (Merck/Sigma-Aldrich) | M1028 | |
| Ethylenediaminetetraacetic acid solution | (Merck/Sigma-Aldrich) | E7889 | |
| Ambion™ RNase I, cloned, 100 U/µL | Ambion | AM2294 | |
| SUPERase•In™ RNase Inhibitor (20 U/μL) | Invitrogen™ by Thermo Fisher Scientific | AM2694 | |
| Acidic phenol:chlorophorm:isoamyl alcohol 125:24:1 (pH 4.0-5.0) | (Merck/Sigma-Aldrich) | P1944-100ML | |
| Dynabeads™ Goat Anti-Mouse IgG | Invitrogen™ by Thermo Fisher Scientific) | 11033 | |
| Sodium Acetate (3 M), pH 5.5 | Invitrogen™ by Thermo Fisher Scientific) | AM9740 | |
| Glycogen (5 mg/ml) | Invitrogen™ by Thermo Fisher Scientific) | AM9510 | |
| Ethyl alcohol, Pure | Merck; Sigma Aldrich | E7023 | |
| Amersham™ Hybond® P Western blotting membranes, PVDF | Merck | GE10600023 | PVDF membrane for western blotting |
| Bolt™ 4 to 12%, Bis-Tris, 1.0 mm, Mini Protein Gel | Invitrogen™ by ThermoFischer Sientific | NW04120BOX | Protein gel |
| 4X Bolt™ LDS Sample Buffer | Invitrogen™ by ThermoFischer Sientific | B0007 | LDS sample loading buffer |
| Precision Plus Protein™ Kaleidoscope™ Prestained Protein Standards | BioRad | 1610375 | Protein ladder |
| 20X Bolt™ MES SDS Running Buffer | ThermoFischer Scientific | B0002 | PAGE runninjg buffer |
| Intercept® (PBS) Blocking Buffer | LI-COR | 927-70001 | Odyssey Blcoking buffer (PBS) |
| IRDye® 800CW Goat anti-Mouse IgG Secondary Antibody | LI-COR | 92632210 | |
| IRDye® 800CW Goat anti-Rabbit IgG Secondary Antibody | LI-COR | 92632211 | |
| TAP Tag Polyclonal Antibody | Invitrogen™ by ThermoFischer Sientific | CAB1001 | |
| Anti-beta Actin antibody | Abcam | ab8227 | |
| Sucrose | (Merck/Sigma-Aldrich) | 84097 | BioUltra, for molecular biology, ≥99.5% (HPLC) |
| DL-Dithiothreitol solution | (Merck/Sigma-Aldrich) | 43816 | BioUltra, for molecular biology, ~1 M in H2O |
| Terumo Syringe 1CC/mL | Terumo Syringe | 878499 | |
| Potassium chloride | (Merck/Sigma-Aldrich) | 60128 | |
| HEPES | (Merck/Sigma-Aldrich) | H3375 | |
| Dulbecco's Modified Eagle's Medium - high glucose | Sigma Aldrich | D5796 | |
| Fetal Bovine Serum | Sigma Aldrich | 12003C | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300062 | |
| Dulbecco's Phosphate Buffered Saline with Calcium and magnesium | Sigma-Aldrich | D8662 | |
| Glycine | Sigma-Aldrich | G7126 | |
| Tris hydrochloride | Merck/Sigma-Aldrich | 10812846001 | |
| Sodium dodecyl sulfate | Merck/Sigma-Aldrich | 436143 | |
| IGEPAL CA-630 | Merck/Sigma-Aldrich | I3021 | |
| Rnasin Ribonuclease Inhibitor | Promega | N2111 | |
| Stainless steel grinding jar | Retsch | 02.462.0059 | |
| MM400 mixer mill | Retsch | 20.745.0001 | |
| Gradient Fractionator | Brandel | BRN-BR-188 | |
| Thermomixer R | Eppendorf | Z605271 | |
| Nanodrop spectrophotometer | Thermo Fisher Scientific | ND-2000 | |
| 0.5-ml microcentrifuge tubes with locking devices | Eppendorf Safe-Lock | 30121023 | |
| Mini Gel Tank | (Thermo Fisher Scientific) | A25977 | PAGE running tank |
| 5 mL, Open-Top Thinwall Ultra-Clear Tube, 13 x 51mm | Beckman-Coulter | 344057 | |
| 13.2 mL, Certified Free Open-Top Thinwall Polypropylene, 14 x 89mm - 50Pk | Beckman-Coulter | 331372 | |
| Amicon Ultra-0.5 ultrafiltration devices | Merck | UFC5030 | Ultracel-30 regenerated cellulose membrane, 0.5 mL sample volume |
| Thermo Sorvall Evolution RC Floor Super Speed Centrifuge | Cambridge Scientific | 15566 | |
| Beckman Coulter Optima L-90K | GMI | 8043-30-1191 | |
| Nunc EasYFlask 175cm2 | Thermofisher Scientific | 159910 | |
| Falcon 50 mL Conical Centrifuge Tubes | Thermofisher Scientific | 14-432-22 | |
| 25 mL Serological Pipette | Sigma-Aldrich | SIAL1250 | |
| 10 mL Serological Pipette | Sigma-Aldrich | SIAL1100 | |
| DNA lobind tubes | Eppendorf | 30108051 | |
| Cold Centrifuge 5810 R | Eppendorf | EP022628188 | for 50 mL tubes |
| Orbital Shaking Incubator | Ratek | OM11 | |
| Frezco 17 Microcentrifuge | Thermofisher Scientific | 75002402 | |
| Eppendorf DNA lo-bind tubes | Merck/Sigma-Aldrich | EP0030108051 | |
| Eppendorf® Protein LoBind tubes | Merck/Sigma-Aldrich | EP0030108116 | |
| SW 41 Ti Swinging bucket rotor | Beckman-Coulter | 331362 | |
| Heracell™ 150i CO2 Incubator, 150 L | Thermofisher Scientific | 51026282 | |
| 0,3 mL ultra-fine II short insulin syringe | BD Medical | 328822 | |
| 3 mL syringe with Luer Lok tip | BD Medical | 302113 | |
| 25 G x 16 mm Hypodermic Needle | Terumo | TUAN2516R1 |
Riferimenti
- Janapala, Y., Preiss, T., Shirokikh, N. E. Control of translation at the initiation phase during glucose starvation in yeast. International Journal of Molecular Sciences. 20 (16), 4043 (2019).
- Masvidal, L., Hulea, L., Furic, L., Topisirovic, I., Larsson, O. mTOR-sensitive translation: Cleared fog reveals more trees. RNA Biology. 14 (10), 1299-1305 (2017).
- Ashe, M. P., De Long, S. K., Sachs, A. B. Glucose depletion rapidly inhibits translation initiation in yeast. Molecular Biology of the Cell. 11 (3), 833-848 (2000).
- Crawford, R. A., Pavitt, G. D. Translational regulation in response to stress in Saccharomyces cerevisiae. Yeast. 36 (1), 5-21 (2019).
- Melamed, D., Pnueli, L., Arava, Y. Yeast translational response to high salinity: global analysis reveals regulation at multiple levels. RNA. 14 (7), 1337-1351 (2008).
- Hershey, J. W., Sonenberg, N., Mathews, M. B. Principles of translational control: An overview. Cold Spring Harbor Perspectives in Biology. 4 (12), 011528 (2012).
- Mata, J., Marguerat, S., Bähler, J. Post-transcriptional control of gene expression: a genome-wide perspective. Trends in Biochemical Sciences. 30 (9), 506-514 (2005).
- Spriggs, K. A., Bushell, M., Willis, A. E. Translational regulation of gene expression during conditions of cell stress. Molecular Cell. 40 (2), 228-237 (2010).
- Liu, B., Qian, S. B. Translational reprogramming in cellular stress response. Wiley Interdisciplinary Reviews RNA. 5 (3), 301-315 (2014).
- Archer, S. K., Shirokikh, N. E., Beilharz, T. H., Preiss, T. Dynamics of ribosome scanning and recycling revealed by translation complex profiling. Nature. 535 (7613), 570-574 (2016).
- Hinnebusch, A. G., Ivanov, I. P., Sonenberg, N. Translational control by 5'-untranslated regions of eukaryotic mRNAs. Science. 352 (6292), 1413-1416 (2016).
- Dever, T. E., Green, R. The elongation, termination, and recycling phases of translation in eukaryotes. Cold Spring Harbor Perspectives in Biology. 4 (7), 013706 (2012).
- Shirokikh, N. E., Preiss, T. Translation initiation by cap-dependent ribosome recruitment: Recent insights and open questions. Wiley Interdisciplinary Reviews RNA. 9 (4), 1473 (2018).
- Jiménez-Díaz, A., Remacha, M., Ballesta, J. P., Berlanga, J. J. Phosphorylation of initiation factor eIF2 in response to stress conditions is mediated by acidic ribosomal P1/P2 proteins in Saccharomyces cerevisiae. PLoS One. 8 (12), 84219 (2013).
- Sonenberg, N., Hinnebusch, A. G. Regulation of translation initiation in eukaryotes: mechanisms and biological targets. Cell. 136 (4), 731-745 (2009).
- Majmundar, A. J., Wong, W. J., Simon, M. C. Hypoxia-inducible factors and the response to hypoxic stress. Molecular Cell. 40 (2), 294-309 (2010).
- Barraza, C. E., et al. The role of PKA in the translational response to heat stress in Saccharomyces cerevisiae. PLoS One. 12 (10), 0185416 (2017).
- Richter, K., Haslbeck, M., Buchner, J. The heat shock response: Life on the verge of death. Molecular Cell. 40 (2), 253-266 (2010).
- Jamar, N. H., Kritsiligkou, P., Grant, C. M. The non-stop decay mRNA surveillance pathway is required for oxidative stress tolerance. Nucleic Acids Research. 45 (11), 6881-6893 (2017).
- Chen, Z., et al. The complete pathway for thiosulfate utilization in Saccharomyces cerevisiae. Applied and Environmental Microbiology. 84 (22), (2018).
- Marzluf, G. A. Molecular genetics of sulfur assimilation in filamentous fungi and yeast. Annual Review of Microbiology. 51, 73-96 (1997).
- Miller, D., Brandt, N., Gresham, D. Systematic identification of factors mediating accelerated mRNA degradation in response to changes in environmental nitrogen. PLoS Genetics. 14 (5), 1007406 (2018).
- Zhang, W., Du, G., Zhou, J., Chen, J. Regulation of sensing, transportation, and catabolism of nitrogen sources in Saccharomyces cerevisiae. Microbiology and Molecular Biology Reviews. 82 (1), (2018).
- Tokpohozin, S. E., Fischer, S., Becker, T. Selection of a new Saccharomyces yeast to enhance relevant sorghum beer aroma components, higher alcohols, and esters. Food Microbiology. 83, 181-186 (2019).
- Walker, G. M., Stewart, G. G. Saccharomyces cerevisiae in the production of fermented beverages. Beverages. 2 (4), 30 (2016).
- Chassé, H., Boulben, S., Costache, V., Cormier, P., Morales, J. Analysis of translation using polysome profiling. Nucleic Acids Research. 45 (3), 15 (2017).
- Jin, H. Y., Xiao, C. An integrated polysome profiling and ribosome profiling method to investigate in vivo translatome. Methods in Molecular Biology. 1712, 1-18 (2018).
- Arava, Y., et al. Genome-wide analysis of mRNA translation profiles in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences of the United States of America. 100 (7), 3889-3894 (2003).
- Lackner, D. H., et al. A network of multiple regulatory layers shapes gene expression in fission yeast. Molecular Cell. 26 (1), 145-155 (2007).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-Wide Analysis in Vivo of Translation with Nucleotide Resolution Using Ribosome Profiling. Science. 324 (5924), 218-223 (2009).
- Ingolia, N. T., Hussmann, J. A., Weissman, J. S. Ribosome Profiling: Global Views of Translation. Cold Spring Harbor Perspectives in Biology. 11 (5), (2019).
- Gerashchenko, M. V., Lobanov, A. V., Gladyshev, V. N. Genome-wide ribosome profiling reveals complex translational regulation in response to oxidative stress. Proceedings of the National Academy of Sciences of the United States of America. 109 (43), 17394-17399 (2012).
- Hussmann, J. A., Patchett, S., Johnson, A., Sawyer, S., Press, W. H. Understanding biases in ribosome profiling experiments reveals signatures of translation dynamics in yeast. Proceedings of the National Academy of Sciences Genetics. 11 (12), 1005732 (2015).
- Santos, D. A., Shi, L., Tu, B. P., Weissman, J. S. Cycloheximide can distort measurements of mRNA levels and translation efficiency. Nucleic Acids Research. 47 (10), 4974-4985 (2019).
- Schneider-Poetsch, T., et al. Inhibition of eukaryotic translation elongation by cycloheximide and lactimidomycin. Nature Chemical Biology. 6 (3), 209-217 (2010).
- Hoffman, E. A., Frey, B. L., Smith, L. M., Auble, D. T. Formaldehyde crosslinking: A tool for the study of chromatin complexes. Journal of Biological Chemistry. 290 (44), 26404-26411 (2015).
- Kage, U., Powell, J. J., Gardiner, D. M., Kazan, K. Ribosome profiling in plants: What is not lost in translation. Journal of Experimental Botany. 71 (18), 5323-5332 (2020).
- Shirokikh, N. E., Archer, S. K., Beilharz, T. H., Powell, D., Preiss, T. Translation complex profile sequencing to study the in vivo dynamics of mRNA-ribosome interactions during translation initiation, elongation and termination. Nature Protocols. 12 (4), 697-731 (2017).
- Wagner, S., et al. Selective translation complex profiling reveals staged initiation and co-translational assembly of initiation factor complexes. Molecular Cell. 79 (4), 546-560 (2020).
- Zlotorynski, E. Profiling ribosome dynamics. Nature Reviews Molecular Cell Biology. 17 (9), 535-535 (2016).
- Sen, N. D., Gupta, N., S, K. A., Preiss, T., Lorsch, J. R., Hinnebusch, A. G. Functional interplay between DEAD-box RNA helicases Ded1 and Dbp1 in preinitiation complex attachment and scanning on structured mRNAs in vivo. Nucleic Acids Research. 47 (16), 8785-8806 (2019).
- Zhao, J., Qin, B., Nikolay, R., Spahn, C. M. T., Zhang, G. Translatomics: The global view of translation. International Journal of Molecular Sciences. 20 (1), 20010212 (2019).
- Luthe, D. S. A simple technique for the preparation and storage of sucrose gradients. Analytical Biochemistry. 135 (1), 230-232 (1983).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: A revolutionary tool for transcriptomics. Nature Review Genetics. 10 (1), 57-63 (2009).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trends in Biochemical Sciences. 25 (3), 99-104 (2000).
- Schmiedeberg, L., Skene, P., Deaton, A., Bird, A. A Temporal Threshold for Formaldehyde Crosslinking and Fixation. PLoS One. 4 (2), 4636 (2009).
- Solomon, M. J., Varshavsky, A. Formaldehyde-mediated DNA-protein crosslinking: A probe for in vivo chromatin structures. Proceedings of the National Academy of Sciences. 82 (19), 6470-6474 (1985).
- Solomon, M. J., Larsen, P. L., Varshavsky, A. Mapping proteinDNA interactions in vivo with formaldehyde: Evidence that histone H4 is retained on a highly transcribed gene. Cell. 53 (6), 937-947 (1988).
- Bohlen, J., Fenzl, K., Kramer, G., Bukau, B., Teleman, A. A. Selective 40S footprinting reveals cap-tethered ribosome scanning in human cells. Molecular Cell. 79 (4), 561-574 (2020).
- Shirokikh, N. E. Translation complex stabilization on messenger RNA and footprint profiling to study the RNA responses and dynamics of protein biosynthesis in the cells. Critical Reviews in Biochemistry and Molecular Biology. , (2021).
- Giess, A., et al. Profiling of small ribosomal subunits reveals modes and regulation of translation initiation. Cell Reports. 31 (3), 107534 (2020).
- Firmino, A. A. P., et al. Separation and paired proteome profiling of plant chloroplast and cytoplasmic ribosomes. Plants (Basel). 9 (7), (2020).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic Acids Research. 42 (17), 134 (2014).
- Santos, D. A., Shi, L., Tu, B. P., Weissman, J. S. Cycloheximide can distort measurements of mRNA levels and translation efficiency. Nucleic Acids Research. 47 (10), 4974-4985 (2019).
- Schneider-Poetsch, T., et al. Inhibition of eukaryotic translation elongation by cycloheximide and lactimidomycin. Nature Chemical Biology. 6 (3), 209-217 (2010).
- Plénat, F., et al. Formaldehyde fixation in the third millennium. Annales De Pathologie. 21 (1), 29-47 (2001).
- Salic, A., Mitchison, T. J. A chemical method for fast and sensitive detection of DNA synthesis in vivo. Proceedings of the National Academy of Sciences. 105 (7), 2415-2420 (2008).
- Wang, N. S., Minassian, H. The formaldehyde-fixed and paraffin-embedded tissues for diagnostic transmission electron microscopy: A retrospective and prospective study. Human Pathology. 18 (7), 715-727 (1987).
- Grifo, J. A., et al. Characterization of eukaryotic initiation factor 4A, a protein involved in ATP-dependent binding of globin mRNA. Journal of Biological Chemistry. 257 (9), 5246-5252 (1982).
- Li, Y. Commonly used tag combinations for tandem affinity purification. Biotechnology and Applied Biochemistry. 55 (2), 73-83 (2010).
- Blum, S., et al. ATP hydrolysis by initiation factor 4A is required for translation initiation in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences. 89 (16), 7664-7668 (1992).
- Merrick, W. C. eIF4F: A Retrospective. Journal of Biological Chemistry. 290 (40), 24091-24099 (2015).
- Rogers, G. W., Komar, A. A., Merrick, W. C. eIF4A: The godfather of the DEAD box helicases. Progress in Nucleic Acid Research and Molecular Biology. 72, 307-331 (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon