Method Article
Le séquençage par insertion de transposons comme outil pour élucider les facteurs de colonisation bactérienne dans un symbiote Burkholderia gladioli de coléoptères Lagria villosa
Dans cet article
Résumé
Il s’agit d’une méthode adaptée pour identifier les facteurs de colonisation des insectes candidats dans un symbiote bénéfique Burkholderia. L’hôte du coléoptère est infecté par une bibliothèque mutante aléatoire générée par mutagénèse par transposon, et la complexité de la bibliothèque après la colonisation est comparée à un témoin cultivé in vitro.
Résumé
Déduire la fonction des gènes en manipulant leur activité est un outil essentiel pour comprendre les fondements génétiques de la plupart des processus biologiques. Les progrès de la microbiologie moléculaire ont vu l’émergence de diverses techniques de mutagénèse pour la manipulation des gènes. Parmi eux, le séquençage transposon-insertion (Tn-seq) est un outil précieux pour évaluer simultanément la fonctionnalité de nombreux gènes candidats de manière non ciblée. La technique a été essentielle pour identifier les mécanismes moléculaires de la colonisation des hôtes eucaryotes chez plusieurs microbes pathogènes et quelques symbiotes bénéfiques.
Ici, Tn-seq est établi comme une méthode pour identifier les facteurs de colonisation dans un symbiote mutualiste Burkholderia gladioli du coléoptère Lagria villosa. Par conjugaison, l’insertion par transposon Tn5 d’une cassette de résistance aux antibiotiques est réalisée à des endroits génomiques aléatoires chez B. gladioli. Pour identifier l’effet des perturbations génétiques sur la capacité des bactéries à coloniser l’hôte du coléoptère, la bibliothèque de mutations transposon de B. gladioli générée est inoculée sur les œufs du coléoptère, tandis qu’un témoin est cultivé in vitro dans un milieu de culture liquide. Après avoir prévu suffisamment de temps pour la colonisation, l’ADN est extrait des bibliothèques cultivées in vivo et in vitro. Suivant un protocole de préparation de bibliothèque d’ADN, les échantillons d’ADN sont préparés pour le séquençage d’insertion de transposon. Les fragments d’ADN qui contiennent le bord transposon-insert et l’ADN bactérien flanquant sont sélectionnés, et les sites de mutation sont déterminés par séquençage loin du bord transposon-insert. Enfin, en analysant et en comparant les fréquences de chaque mutant entre les bibliothèques in vivo et in vitro, l’importance de gènes symbiotes spécifiques lors de la colonisation du coléoptère peut être prédite.
Introduction
Burkholderia gladioli peut s’engager dans une association symbiotique avec les coléoptères Lagria villosa, jouant un rôle important dans la défense contre les antagonistes microbiens de l’insecte hôte4,5,6. Les coléoptères femelles abritent plusieurs souches de B. glaïeules dans des glandes spécialisées accessoires au système reproducteur. Lors de la ponte, les femelles étalent les cellules de B. gladioli à la surface de l’œuf où les composés antimicrobiens produits par B. gladioli inhibent les infections par les champignons entomopathogènes4,6. Au cours du développement embryonnaire tardif ou au début de l’éclosion des larves, les bactéries colonisent les invaginations cuticulaires sur la surface dorsale des larves. Malgré cette localisation spécialisée et cette voie de transmission verticale des symbiotes, L. villosa peut vraisemblablement aussi acquérir B. gladioli horizontalement de l’environnement4. De plus, au moins trois souches de B. gladioli ont été trouvées en association avec L. villosa4,6. Parmi ceux-ci, B. gladioli Lv-StA est le seul qui se prête à la culture in vitro.
B. glaïez Lv-StA a une taille de génome de 8,56 Mb6 et contient 7 468 gènes. Lesquels de ces gènes sont importants pour que la bactérie B. gladioli colonise l’hôte du coléoptère? Pour répondre à cette question, nous avons utilisé le séquençage par insertion de transposon (Tn-seq), une méthode exploratoire permettant d’identifier les gènes microbiens conditionnellement essentiels1,2,3. Une bibliothèque mutante de B. gladioli Lv-StA a été créée à l’aide d’un transposon Tn5. Par conjugaison des cellules donneuses d’Escherichia coli à B. gladioli Lv-StA, un plasmide pRL27 portant le transposon Tn5 et une cassette de résistance aux antibiotiques flanquée de répétitions inversées ont été transférés (Figure 1). Ainsi, un ensemble de mutants porteurs individuellement de perturbations de 3 736 gènes symbiotes a été généré (Figure 2).
Le groupe de mutants a été infecté sur des œufs de coléoptères pour identifier les facteurs de colonisation et, comme témoin, a également été cultivé in vitro dans le milieu B de King(KB). Après avoir prévu suffisamment de temps pour la colonisation, les larves écloses ont été collectées et mises en commun pour l’extraction de l’ADN. Des fragments d’ADN contenant l’insert de transposon et la région génomique d’accompagnement de B. gladioli Lv-StA ont été sélectionnés à l’aide d’un protocole de préparation de bibliothèque d’ADN modifié pour le séquençage. Un traitement de qualité de lecture suivi d’une analyse avec DESeq2 a été effectué pour identifier des gènes spécifiques cruciaux pour que B. gladioli Lv-StA colonise les larves de L. villosa lorsqu’elles sont transmises par la surface de l’œuf.
Protocole
1. Préparation des supports et des tampons
- Préparer les supports KB et LB et les plaques de gélose comme indiqué dans le tableau 1, et autoclaver à 121 °C, 15 psi, 20 min.
- Ajouter 50 μg/mL de kanamycine stérilisée par filtre et 300 μM d’acide 2,6-diaminopimélique (DAP) stérilisé par filtre au milieu LB autoclavé avant de cultiver E.coli WM3064 + pRL27.
- Ajouter 50 μg/mL de kanamycine stérilisée par filtre à la gélose KB autoclavée pour verser les plaques nécessaires à la sélection des transconjugants B. gladioli Lv-StA réussis.
- Préparer 1x solution saline tamponnée au phosphate (PBS) en mélangeant les composants suivants : NaCl 8 g/L, KCl 0,201 g/L, Na2HPO4 1,42 g/L et KH2PO4 0,272 g/L. Dissoudre les sels dans de l’eau distillée et autoclaver le mélange à 121 °C, 15 psi, 20 min avant utilisation. Conserver à température ambiante.
- Préparer un tampon de liaison et de lavage 2x en dissolvant les composants suivants : 10 mM de Tris-HCl (pH 7,5), 1 mM d’acide tétraacétique d’éthylènediamine (EDTA) et 2 M de NaCl dans de l’eau distillée. Filtrer-stériliser le mélange avant utilisation. Conserver à température ambiante.
- Préparer 1x Low-TE en dissolvant 10 mM de Tris-HCl (pH 8,0) et 0,1 mM d’EDTA dans de l’eau doublement distillée. Stériliser par autoclavage à 121 °C, 15 psi, 20 min. Conserver à température ambiante.
2. Conjugaison pour générer la bibliothèque de mutants transposons
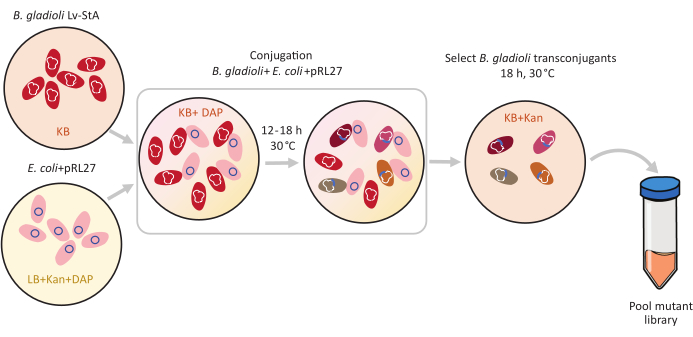
Figure 1: Étapes du protocole de conjugaison. Le receveur de conjugaison Burkholderia gladioli Lv-StA (rouge) et le donneur Escherichia coli contenant le plasmide pRL27 (rose) sont cultivés dans la gélose KB et LB, respectivement, complétées par de la kanamycine et du DAP. Après transfert conjugatif du plasmide pendant 12-18 h à 30 °C, les cellules transconjugantes de B. gladioli sont sélectionnées sur KB contenant de la kanamycine et regroupées. Abréviations : DAP = acide 2,6-diaminopimélique; Kan = kanamycine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Sous un capuchon stérile, inoculer une culture donneuse fraîche d’Escherichia coli WM3064 + pRL27 dans 10 mL de milieu LB complété par de la kanamycine et du DAP. Cellules receveuses inoculées de Burkholderia gladioli Lv-StA dans 5 mL de milieu KB. Incuber les cultures à 30 °C pendant la nuit sur un shaker à 250 tr/min.
- Après une croissance nocturne, centrifuger 4 mL de chacune des cultures à 9 600 × g pendant 6 min pour granuler les cellules. Jetez le surnageant.
- Sous une hotte stérile, laver les cultures cellulaires en granulés dans un milieu KB contenant du DAP et enfin resuspender les cultures séparément dans 4 mL de milieu KB + DAP.
- Dans un tube frais de 15 mL, mélanger 250 μL des cellules donneuses d’E. coli lavées avec 1 mL des cellules receveuses de B. gladioli Lv-StA lavées.
- Repérez 10 μL de ce mélange de cellules de conjugaison sur des plaques de gélose KB contenant du DAP. Laisser reposer la plaque sans être dérangée dans la hotte stérile à température ambiante pendant 1 h. Ensuite, incuber les plaques avec les points de conjugaison à 30 °C pendant 12-18 h.
NOTE: La période de conjugaison peut être ajustée en fonction de l’espèce cible. Cependant, une longue période de conjugaison augmente le risque de double insertion ou d’intégration de plasmides dans le génome. Pour les bactéries à croissance lente, prévoyez des périodes de conjugaison plus longues. - Après l’incubation, ajoutez 2 à 4 mL de 1x PBS dans les plaques sous une hotte stérile et utilisez un grattoir cellulaire pour libérer les taches de conjugaison bactérienne cultivées de la gélose. Pipeter le mélange de cellules conjuguées dans des tubes de microfuge de 2 mL.
- Ar granuler les cellules par centrifugation à 9 600 × g pendant 2 min. Jetez le surnageant et lavez la pastille deux fois dans 1 mL de 1x PBS en pipetant de haut en bas. Resuspendez la pastille finale dans 1200 μL de 1x PBS. Effectuer des dilutions avant le placage si le nombre de cellules dans le mélange est supérieur à 1 × 104.
- Bien mélanger et étaler 200 μL du mélange cellulaire sur de grandes plaques de gélose KB (6 ou plus, si nécessaire) complétées par de la kanamycine et incuber à 30 °C pendant la nuit.
REMARQUE: Les colonies mutantes cibles apparaissent dans les 30 heures sur les plaques de gélose sélectives. En raison du marqueur de résistance aux antibiotiques, seules des colonies mutées apparaissent sur la plaque de gélose sélective. Par conséquent, on s’attend à ce que toutes les colonies soient des transconjugants réussis. - Compter le nombre total de colonies transconjugantes sur trois plaques et extrapoler pour calculer le nombre approximatif de mutants obtenus dans toutes les plaques. Pour augmenter les chances d’obtenir une bibliothèque représentative, assurez-vous que le nombre total de colonies est plusieurs fois plus élevé que le nombre total de gènes dans le génome. Pour confirmer le succès de la conjugaison, effectuez une PCR ciblant la cassette d’insertion en utilisant 10 à 20 colonies d’échantillons, comme décrit à la rubrique 3.
NOTE: L’objectif est de s’assurer que le nombre de colonies est au moins 10 fois le nombre de gènes dans l’ensemble du génome, dans ce cas, > 75 000 mutants. Cependant, il est généralement difficile d’estimer avec précision le nombre de colonies qui correspondrait à une bibliothèque entièrement représentative. Le nombre de gènes uniques mutés n’est pas évident à ce stade, étant donné que les perturbations dans les gènes essentiels ne sont pas capturées, qu’il existe souvent plusieurs sites de mutation différents pour le même gène et que les mutations générées avec les transposons Tn5 ne sont pas entièrement aléatoires. - Sous une hotte stérile, grattez les colonies des plaques en ajoutant 1 à 2 mL de 1x PBS sur la gélose. Regrouper le mélange de cellules gratté des plaques dans des tubes de 50 mL. Vortex la bibliothèque pour bien mélanger, puis diviser 4 mL de la bibliothèque mutante regroupée en plusieurs cryotubes. Ajouter 1 mL de glycérol à 70 % dans les tubes et conserver à -80 °C.
3. PCR et électrophorèse sur gel pour confirmer les insertions réussies dans B. gladioli Lv-StA
- Pour confirmer la présence de l’insertion, choisissez des colonies mutantes individuelles dans les plaques de sélection à l’étape 2.9 et effectuez une PCR ciblant la cassette d’insertion à l’aide des amorces énumérées dans le tableau 2. Préparer le mélange maître PCR conformément au tableau 3 et définir les conditions dans le cycleur thermique comme décrit dans le tableau 4.
- Exécutez les produits PCR sur un gel d’agarose à 1,6% par électrophorèse (250 V, 40 min) pour vérifier si les fragments d’ADN amplifiés sont de la longueur prévue de 1580 bp.
4. Infection mutante de la piscine sur les œufs de coléoptères
- Étapes de lavage de la bibliothèque
- Décongelez une aliquote de la bibliothèque de mutants préparée sur la glace. Centrifuger à 2 683 × g pendant 10 min et retirer le surnageant. Sous une hotte stérile, lavez les cellules avec 4 mL de 1x PBS pour éliminer tout milieu restant des cellules. Resuspendez les cellules dans 4 mL de 1x PBS.
- Comptez le nombre de cellules dans une aliquote de la bibliothèque à l’aide d’une chambre de comptage de cellules. Diluer une partie de la bibliothèque à 2 × 106 cellules/μL dans 1x PBS.
- Vortex la bibliothèque aliquote à fond pour mélanger l’ensemble de la bibliothèque de manière homogène avant de prendre le volume requis.
- Stérilisation de la couvée et infection in vivo
- Sélectionnez une couvée à œufs de L. villosa. Comptez le nombre d’œufs et continuez si la couvée contient plus de 100 œufs.
- Stérilisez toute la couvée d’œufs.
- Ajouter 200 μL d’éthanol à 70 % et laver doucement les œufs pendant 5 min. Retirez l’éthanol et lavez les œufs deux fois avec de l’eau autoclavée.
- Ajouter 200 μL d’eau de Javel à 12 % (NaOCl) et laver doucement les œufs pendant 30 s. Retirez immédiatement l’eau de Javel et lavez à nouveau les œufs trois fois avec 200 μL d’eau autoclavée.
- Infecter 2 × 106 cellules/μL de la bibliothèque mutante lavée sur la couvée stérilisée (2,5 μL par œuf).
- Deux jours après l’éclosion des larves de coléoptères infectées, prélever 100 larves du2 e stade par tube de microfuge de 1,5 mL et les conserver à -80 °C.
-
Contrôle de bibliothèque de mutants in vitro
- Sous une hotte stérile, inoculer 250 μL de 2 × 106 cellules/μL de la bibliothèque mutante lavée dans 10 mL de milieu KB contenant de la kanamycine.
- Incuber la culture mutante in vitro à 30 °C pendant 20 h.
REMARQUE: Calculer la durée de l’incubation pour correspondre au nombre approximatif de générations de WT B. gladioli Lv-StA in vivo pendant la colonisation. - Après 20 h d’incubation, ajouter un volume égal de glycérol à 70 % à la culture mutante in vitro et le stocker à -80 °C.
5. Coléoptères infectés et extraction d’ADN de bibliothèque mutante in vitro
REMARQUE: Les extractions d’ADN ont été effectuées à l’aide d’un kit de purification de l’ADN et de l’ARN conformément au protocole du fabricant brièvement décrit ci-dessous.
- Homogénéiser les larves regroupées (maximum de 4 mg par tube de microfuge) en ajoutant 1 à 2 mL d’azote liquide et en les écrasant avec un pilon.
- Décongeler les cultures mutantes cultivées in vitro à partir de stocks de glycérol sur glace. A granuler les cellules par centrifugation à 9 600 × g pendant 10 min avant la lyse cellulaire.
- Ajouter 300 μL de solution de lyse tissulaire et cellulaire aux échantillons in vitro et in vivo. Ajouter 5 μL de 10 mg/mL de protéinase K, incuber le mélange à 60 °C pendant 15 min, puis placer sur de la glace pendant 3-5 min.
- Ajouter 150 μL de réactif de précipitation de protéines aux lysates et vortex soigneusement. Aruffez les débris protéiques en les centrifugant à 9 600 × g pendant 10 min.
- Transférer le surnageant dans un tube de microfuge de 1,5 mL. Ajouter 500 μL d’isopropanol au surnageant et inverser doucement les tubes au moins 40 fois avant d’incuber à -20 °C pendant 1 h ou pendant la nuit.
- Ar coupez l’ADN précipité par centrifugation à 9 600 × g pendant 10 min. Jetez le surnageant et ajoutez de l’éthanol glacé à 70% à la pastille d’ADN.
- Centrifuger à ≥10 000 × g pendant 5 min. Jeter le surnageant et laisser sécher les échantillons à l’air libre pendant au moins 1 h.
- Resuspendez l’ADN des échantillons in vitro et in vivo dans 100 μL de tampon Low-TE.
- Conserver les échantillons à -20 °C.
6. Préparation de la bibliothèque de séquençage
REMARQUE: Le protocole et les réactifs pour la préparation de la bibliothèque d’ADN sont adaptés et modifiés à partir des instructions fournies par le fabricant du kit de préparation de la bibliothèque d’ADN.
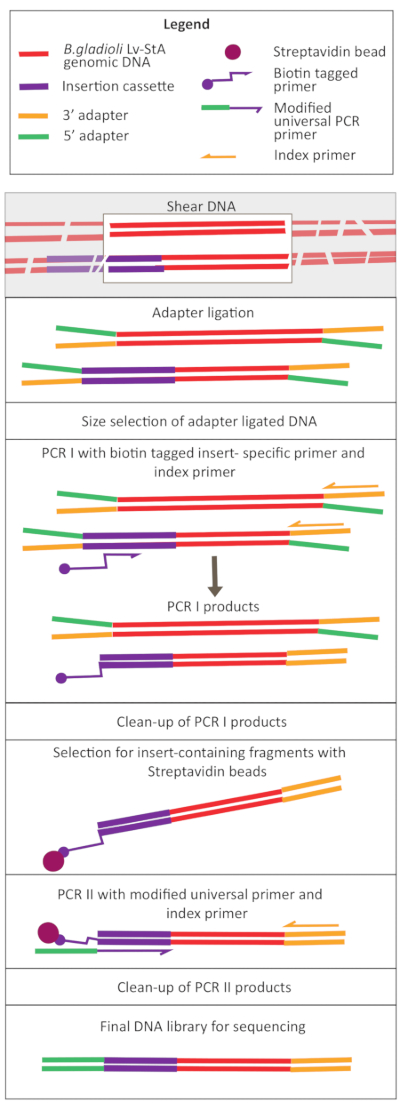
Figure 2: Schéma des étapes de préparation de la bibliothèque d’ADN. Après le cisaillement et la ligature de l’adaptateur, le protocole modifié comprend une étape de sélection des billes de streptavidine pour enrichir les fragments d’ADN contenant la cassette d’insertion. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Diluer les échantillons à une concentration de 20 ng/μL et un volume de 100 μL et les garder sur de la glace.
- Cisaillement in vivo et in vitro de l’ADN à l’aide d’un ultrasonateur. Réglez l’ultrasonateur à 70% de puissance. Vortex les échantillons brièvement et cisaillement pendant 1 min 30 s.
REMARQUE: Les paramètres de l’ultrasonateur diffèrent d’un instrument à l’autre. Dans ce cas, la taille du fragment était de 200 à 400 pb, ce qui est approprié pour cette approche de séquençage de 150 pb, à extrémité appariée (voir étape 9.1). Les paramètres de cisaillement peuvent être ajustés en fonction des exigences de l’expérimentateur. - Vérifiez si l’ADN a été cisaillé à la plage de taille souhaitée (dans ce cas, 200-400 bp). Chargez 5 μL de l’ADN non cisaillement et cisaillé après mélange avec un colorant de chargement de gel dans un rapport de 1:1 sur un gel d’agarose à 1,6 % à 250 V pendant 40 min(Figure 3A,B).
- Préparation des extrémités de fragment requises pour la ligature de l’adaptateur
- A 50 μL de l’ADN cisaillement, ajouter les réactifs de préparation finale donnés dans le kit de préparation de la bibliothèque : 3 μL du mélange enzymatique et 7 μL de tampon réactionnel et bien mélanger par pipetage. Réglez un cycleur thermique avec un couvercle chauffé à ≥ 75 °C et incubez les échantillons pendant 30 min à 20 °C et 30 min à 65 °C. Maintenir à 4 °C.
- Ligature de l’adaptateur
- Pour la ligature de l’adaptateur, ajoutez les réactifs suivants aux produits de l’étape de préparation finale : 30 μL Ligation Master Mix, 1 μL Ligation Enhancer et 2,5 μL dilué Adapter. Bien mélanger par pipetage et incuber l’échantillon pendant 15 min à 20 °C dans le cycleur thermique avec le couvercle chauffé fermé.
- Après 15 min, ajouter 3 μL de l’enzyme (ADN de l’uracile glycosylase + ADN glycosylase-lyase Endonucléase VIII) (voir le Tableau des matériaux). Bien mélanger par pipetage et incuber l’échantillon pendant 15 min à 37 °C dans un cycleur thermique avec le couvercle chauffé à ≥47 °C.
REMARQUE : Le protocole peut être mis en pause à cette étape et les échantillons peuvent être stockés à -20 °C.
- Sélection de la taille de fragments d’ADN ligaturés par adaptateur de 250 pb
- Vortex la solution de billes magnétiques (voir le tableau des matériaux)et placez-la à température ambiante pendant 30 minutes avant utilisation.
- Ajouter 0,3x de billes à 96,5 μL du mélange d’ADN ligaturé et mélanger en pipetant soigneusement. Incuber le mélange de perles pendant 5 min.
REMARQUE: La présence de sels et de polyéthylène glycol dans le mélange de billes facilite la précipitation de fragments d’ADN sur les perles. Un faible rapport entre les perles et les molécules d’ADN conduit à la liaison de fragments d’ADN plus gros aux perles. Dans ce cas, des fragments d’ADN de plus de 250 pb de longueur sont liés aux perles. - Placez les tubes sur un support magnétique pour tirer les perles vers le bas et enlever les fragments d’ADN de taille indésirable. Laissez les billes se déposer pendant 5 min, puis transférez le surnageant clair dans un nouveau tube de microfuge (conservez le surnageant).
- Ajouter 0,15x de perles fraîches au surnageant et mélanger en pipetant bien. Incuber le mélange de billes pendant 5 min, puis placer les tubes sur un support magnétique pour tirer vers le bas les perles liées à l’ADN cible. Attendez 5 min, puis jetez le surnageant (conservez les perles).
REMARQUE: Ce rapport de perles à l’ADN conduit à la liaison de fragments de la taille souhaitée de 250 pb. - Avec les billes sur le support magnétique, ajoutez 200 μL d’éthanol à 80% (fraîchement préparé) et attendez 30 s. Pipette et jeter l’éthanol laver soigneusement sans déranger les perles sur le support magnétique. Répétez cette étape.
- Après le dernier lavage, retirez les traces d’éthanol des perles, puis séchez les perles à l’air libre pendant 2 minutes jusqu’à ce qu’elles apparaissent brillantes mais pas complètement desséchées. Ne séchez pas trop les perles.
- Retirez les tubes du support magnétique et ajoutez 17 μL de Tris-HCl de 10 mM ou 0,1x TE (Low-TE). Mélanger par pipetage ~10 fois et incuber le mélange à température ambiante pendant 2 min.
- Replacez les tubes sur le support magnétique et attendez 5 min. Une fois que les perles se sont déposées, transférez le surnageant d’ADN dans un nouveau tube.
- PCR I pour ajouter une étiquette de biotine aux fragments d’ADN contenant la cassette d’insertion
- Ajouter une étiquette d’amorce biotinylé aux fragments d’ADN contenant la cassette d’insertion Tn5 à l’aide de l’amorce biotinylé spécifique au transposon(tableau 5)et d’une amorce d’index. Préparer le mélange maître pcR conformément au tableau 6 et suivre les conditions PCR pour le cycleur thermique indiqué dans le tableau 7.
- Nettoyage de la PCR I sans sélection de taille
- Vortex 0,9x perles et placez-les à température ambiante pendant au moins 30 minutes avant le nettoyage.
- Ajoutez 0,9x perles aux produits PCR et mélangez bien.
- Placez les perles sur un support magnétique pour tirer les perles vers le bas.
- Retirez le surnageant clair et lavez l’ADN lié aux billes avec 200 μL d’éthanol à 80% fraîchement préparé deux fois.
- Retirez l’éthanol après les étapes de lavage et séchez les perles à l’air libre jusqu’à ce qu’elles aient l’air brillantes mais pas trop sèches.
- Ajouter 32 μL de 10 mM de Tris-HCl ou 0,1X TE (Low-TE) et incuber les billes pendant 5 min. Replacez le mélange sur le support magnétique et transférez le surnageant dans un tube de microfuge frais.
- Liaison de fragments d’ADN biotinylé à des billes de streptavidine
- Resuspendez 32 μL de billes de streptavidine dans 1x tampon Bind-and-wash. Lavez les perles avec le tampon trois fois lorsqu’elles sont placées sur un support magnétique.
- Ajouter 32 μL de 2x tampon bind-and-wash et resuspender les billes. À cela, ajoutez 32 μL des produits PCR 1 nettoyés. Bien mélanger et incuber à température ambiante pendant 30 min.
- Placez le mélange perle-ADN sur un support magnétique pendant 2 min. Pipettez le surnageant car l’ADN marqué à la biotine contenant le bord d’insertion se lie à la streptavidine sur les perles.
- Lavez les billes avec 500 μL de 1x tampon bind-and-wash, puis lavez les perles avec 200 μL de Low-TE. Resuspendez les billes liées à l’ADN dans 17 μL de Low-TE.
- PCR II pour ajouter des adaptateurs aux fragments contenant le bord de la cassette d’insertion
- Préparez un mélange principal, comme indiqué dans le tableau 8, à l’aide des amorces d’index et des amorces PCR universelles modifiées répertoriées dans le tableau 5. Ajouter 15 μL des billes de streptavidine liées à l’ADN de l’étape précédente au mélange PCR. Voir le tableau 7 pour les conditions du cycleur thermique.
- Nettoyez les produits PCR sans sélection de taille comme indiqué à l’étape 6.8 de ce protocole. Éluez les produits d’ADN finaux dans 30 μL d’eau de qualité moléculaire.
- Conservez les échantillons à -20 °C et utilisez-les pour le séquençage.
7. Séquençage et analyse
- Séquencez la bibliothèque à l’aide de la technologie de séquençage à haut débit. Ajustez la profondeur de séquençage en fonction de la taille de la bibliothèque de transposons, comme indiqué ci-dessous. Évaluez la qualité de lecture avec FastQC7. Sélectionnez les lectures contenant l’arête d’insertion Tn5 à l’extrémité 5' de la lecture et supprimez la séquence d’arêtes d’insertion à l’aide de Cutadapt8 et/ou Trimmomatic9.
REMARQUE: Ici, une approche de séquençage par paires a été utilisée pour cibler 150 pb par lecture et un total de 8 millions de lectures. Pour obtenir un ensemble de données représentatif, assurez-vous que le nombre total de lectures séquencées dépasse le nombre maximal possible de mutants dans la bibliothèque, c’est-à-dire le nombre total estimé de colonies à partir de l’étape 2.9. À titre de référence, ce protocole visait 40 fois la taille maximale possible de la bibliothèque. D’autres études réussies utilisant Tn-seq dans un but similaire ont séquencé un nombre total de lectures proche de 25 fois le nombre réel d’insertions uniques dans la bibliothèque de mutants correspondante22,23. - Étant donné que les mutations aux extrémités des gènes ne sont pas fonctionnellement perturbatrices, réduisez de 5% les deux extrémités des annotations géniques du fichier GFF du génome de référence. Mappez les lectures découpées au génome de référence à l’aide de Bowtie210.
- Calculez le nombre d’insertions à partir du nombre de positions uniques de 5' dans le fichier BAM d’alignement.
- À l’aide de FeatureCounts11, obtenez le nombre de gènes atteints pour chaque échantillon répliqué.
- En utilisant le paquet DESeq212 dans RStudio, calculez la différence d’abondance des mutants entre différentes conditions.
Résultats
Les bactéries associées à l’hôte peuvent utiliser plusieurs facteurs pour établir une association, y compris ceux qui médient l’adhésion, la motilité, la chimiotaxie, les réponses au stress ou des transporteurs spécifiques. Alors que des facteurs importants pour les interactions pathogène-hôte ont été rapportés pour plusieursbactéries13,14 , 15,16,17,18, y compris les membres du genre Burkholderia19,20, moins d’études ont exploré les mécanismes moléculaires utilisés par les symbiotes bénéfiques pour la colonisation21,22,23 . En utilisant le séquençage d’insertion de transposons, l’objectif était d’identifier les facteurs moléculaires qui permettent à B. gladioli de coloniser les coléoptères L. villosa.
La mutagénèse médiée par transposon a été réalisée à l’aide du plasmide pRL27, qui porte un transposon Tn5 et une cassette de résistance à la kanamycine flanquée de sites de répétition inversés. Le plasmide a été introduit dans les cellules cibles de B. gladioli Lv-StA par conjugaison avec la souche E. coli WM3064 du donneur de plasmide (comme le montre la figure 1). Après conjugaison, le mélange de conjugaison contenant des cellules donneuses de B. glaïe et d’E. coli a été plaqué sur des plaques de gélose sélectives contenant de la kanamycine. L’absence de DAP sur les plaques a éliminé les cellules d’E. coli donneuses, et la présence de kanamycine sélectionnée pour les transconjugants réussis de B. gladioli Lv-StA. La bibliothèque de mutants B. gladioli Lv-StA obtenue à partir de la récolte des 100 000 colonies transconjuvantes a été préparée pour le séquençage à l’aide d’un kit de préparation de bibliothèque d’ADN modifié et d’amorces personnalisées. La figure 2 met en évidence les étapes de préparation de la bibliothèque d’ADN. Le séquençage a donné 4 millions de lectures appariées; 3 736 gènes sur 7 468 gènes de B. gladioli Lv-StA ont été perturbés.
Pour identifier les mutants qui étaient défectueux de colonisation chez l’hôte, la bibliothèque mutante B. gladioli Lv-StA a été infectée sur les œufs de coléoptères et cultivée in vitro en milieu KB comme témoin. La taille du goulot d’étranglement de la colonisation in vivo a été calculée avant l’expérience. Un nombre connu de cellules de B. gladioli Lv-StA a été infecté sur des œufs de coléoptères, et le nombre de cellules colonisatrices dans les larves du premier stade fraîchement écloses a été obtenu en plaquant une suspension de chaque larve et en comptant les unités formant des colonies par individu. Ces calculs ont été effectués pour s’assurer que le nombre de cellules colonisatrices est suffisant pour évaluer la totalité ou un pourcentage élevé des mutants de la bibliothèque pour leur capacité à coloniser l’hôte. De plus, le temps de croissance entre les conditions in vitro et in vivo a été normalisé en fonction du nombre de générations bactériennes pour rendre ces échantillons comparables.
Après l’éclosion des œufs, 1 296 larves ont été recueillies dans 13 bassins. Les cultures mutantes in vitro correspondantes ont été cultivées et stockées sous forme de stocks de glycérol. L’ADN des bibliothèques de mutants cultivés in vivo et in vitro a été extrait et fragmenté dans un ultrasonateur. La figure 3 montre la distribution granulométrique de l’ADN cisaillé, où la majorité des fragments s’étendent entre 100 et 400 pb, comme prévu. Cette étape a été suivie par le protocole de préparation de la bibliothèque d’ADN modifié pour le séquençage. À chaque étape du protocole, la concentration d’ADN restant a été vérifiée pour s’assurer que les étapes ont été effectuées correctement et pour suivre les pertes d’ADN. Un contrôle de qualité (voir la table des matériaux)avant le séquençage a révélé que les bibliothèques d’ADN contenaient des fragments d’ADN étonnamment grands (>800 pb), ce qui était plus prononcé dans les bibliothèques in vivo. Compte tenu de la difficulté d’optimiser le regroupement de fragments dans les voies de séquençage, il était nécessaire d’augmenter la profondeur de séquençage à 10 Mio de lectures appariées dans les bibliothèques in vivo pour atteindre le nombre de lectures souhaité. L’analyse des résultats du séquençage a révélé qu’une moyenne de 4 Mio de lectures dans les bibliothèques in vivo et de 3,1 Mio de lectures dans les bibliothèques in vitro contenait le bord transposon à l’extrémité 5' de Read-1(tableau 9),ce qui était satisfaisant pour cette expérience. La distribution des 24 224 insertions uniques à travers le génome de B. gladioli dans la bibliothèque originale est illustrée à la figure 4. Une analyse réalisée à l’aide de DESeq2 a révélé que les abondances de 271 mutants étaient significativement différentes entre les conditions in vivo et in vitro.
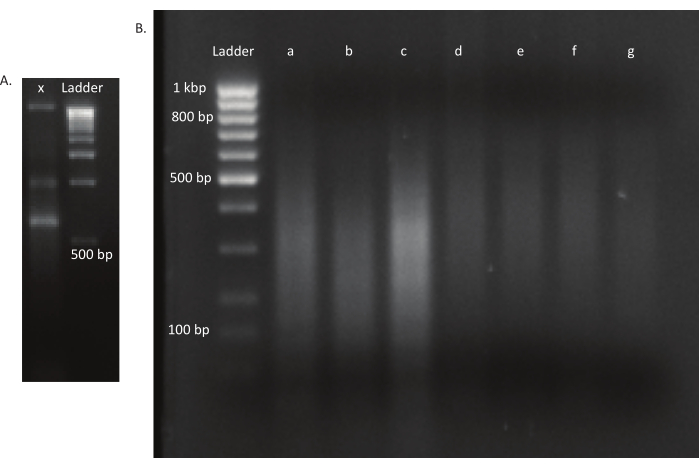
Figure 3: Gels d’agarose d’un mutant et bibliothèques d’ADN. ( A )Geld’agarose avec l’ADN non ciselé d’un mutant dans la voie x et une échelle de 1 kbp pour l’échelle. (B) Gel avec bibliothèque d’ADN cisaillé. Les tailles de bande de l’échelle dans la première voie sont indiquées sur le côté gauche. Les trois premières voies a, b et c contiennent des fragments d’ADN cisaillés des bibliothèques in vivo. Les voies d, e, f et g contiennent des fragments d’ADN cisaillés des bibliothèques in vitro. Veuillez cliquer ici pour voir une version agrandie de cette figure.
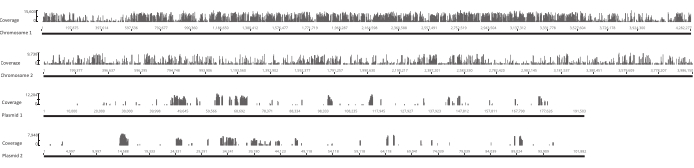
Figure 4: Emplacement des sites d’insertion uniques dans la bibliothèque originale à travers les quatre replicons du génome Lv-StA de Burkholderia gladioli. Chaque barre le long de l’axe des x est située sur un site d’insertion. La hauteur d’une barre le long de l’axe des y correspond au nombre de lectures associées à ce site. Notez que les deux chromosomes et les deux plasmides sont représentés en pleine longueur et ont donc des échelles différentes sur l’axe des x. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| King’s B médium / agar | |
| Peptone (soja) | 20 g/L |
| K2HPO4 | 1,5 g/L |
| MgSO4.7H2O | 1,5 g/L |
| Agar-agar | 15 g/L |
| Dissous dans de l’eau distillée | |
| LB moyen/agar | |
| Tryptone | 10 g/L |
| Extrait de levure | 5 g/L |
| NaCl | 10 g/L |
| Dissous dans de l’eau distillée | |
Tableau 1 : Composantes des médias.
| Non. | Amorces | Séquence | Température de recuit PAR PCR (°C) | |
| 1 | tpnRL17–1RC | 5'-CGTTACATCCCTGGCTTGTT-3' | 58.2 | |
| 2 | tpnRL13–2RC | 5'-TCGTGAAGAAGGTGTTGCTG-3' | ||
Tableau 2 : Amorces pour confirmer le succès de la conjugaison.
| Composant | Volume (μL) |
| Eau purifiée hpLC | 4.92 |
| 10x Buffer S (haute spécificité) | 1 |
| MgCl2 (25 mM) | 0.2 |
| dNTP (2 mM) | 1.2 |
| Amorce 1 (10 pmol/μL) | 0.8 |
| Amorce 2 (10 pmol/μL) | 0.8 |
| Taq (5 U/μL) | 0.08 |
| Total Mastermix | 9 |
| Modèle | 1 |
Tableau 3 : Mélange maître PCR pour confirmer le succès de la conjugaison. Abréviations : CLHP = chromatographie liquide à haute performance; dNTPs = désoxynucléoside triphosphate.
| Escalier | Température °C | Heure | Cycles |
| Dénaturation initiale | 95 | 3 min | 1 |
| Dénaturation | 95 | 40 s | |
| Recuit | 58.2 | 40 s | 30 à 35 |
| Extension | 72 | 1-2 min | |
| Prolongation finale | 72 | 4 min | 1 |
| Tenir | 4 | ∞ | |
Tableau 4 : Conditions de PCR pour confirmer le succès de la conjugaison.
| Amorces | Séquence | Tm °C | Utiliser | Source | |
| Apprêt biotinylé spécifique au transposon | 5'-Biotine-ACAGGAACACTTAACGGCTGACATG -3' | 63.5 | 6.7.1. PCR I | Coutume | |
| Amorce PCR universelle modifiée | 5'- AATGATACGGCGACCACCGAGATC TACACTCTTTCCCTACACGACGCTC TTCCGATCTGAATTCATCGATGAT GGTTGAGATGTGT – 3' | 62 | 6.10.1. PCR II | Coutume | |
| Introduction à l’indice | Reportez-vous au manuel du fabricant | 6.7.1. PCR I & 6.10.1. PCR II | NEBNext Multiplex Oligos for Illumina (Ensemble d’amorces d’indice 1) | ||
| Adaptateur | Reportez-vous au manuel du fabricant | 6.5. Ligature de l’adaptateur | Kit de préparation de la bibliothèque d’ADN NEBNext Ultra II pour Illumina | ||
Tableau 5 : Amorces et adaptateur pour pcR I et II pendant la préparation de la bibliothèque d’ADN.
| Mélange PCR | (μL) |
| Fragments d’ADN ligaturés par adaptateur | 15 |
| Mélange maître NEBNext Ultra II Q5 | 25 |
| Amorce d’indice (10 pmol/μL) | 5 |
| Apprêt biotinylé spécifique au transposon (10 pmol/μL) | 5 |
| Volume total | 50 |
Tableau 6 : Préparation de la bibliothèque d’ADN -Mélange maître PCR I.
| Escalier | Température | Heure | Cycles |
| Dénaturation initiale | 98 °C | 30 s | 1 |
| Dénaturation | 98 °C | 10 s | 6 à 12 |
| Recuit | 65 °C | 30 s | |
| Extension | 72 °C | 30 s | |
| Prolongation finale | 72 °C | 2 min | 1 |
| Tenir | 16 °C | ∞ | |
Tableau 7 : Préparation de la bibliothèque d’ADN -Conditions pcR I et II.
| Mélange PCR | (μL) |
| ADN sélectionné par perles | 15 |
| Mélange maître NEBNext Ultra II Q5 | 25 |
| Introduction à l’indice | 5 |
| Amorce PCR universelle modifiée | 5 |
| Volume total | 50 |
Tableau 8 : Préparation de la bibliothèque d’ADN -Mélange maître PCR II.
| Bibliothèques | Invivo-1 | Invivo-2 | Invivo-3 | Invitro-1 | Invitro-2 | Invitro-3 | Bibliothèque originale | |
| Non. nombre de lectures (PE) | 56,57,710 | 39,19,051 | 30,65,849 | 35,73,494 | 28,83,440 | 36,61,956 | 46,09,410 | |
| Non. de lectures contenant Tn – bord à l’extrémité 5' de Read-1 | 54,15,880 | 37,31,169 | 29,36,247 | 33,00,499 | 27,35,705 | 33,50,402 | 41,53,270 | |
| Taux d’alignement global de Bowtie2 (%) (lecture 1 uniquement) | 95.53% | 83.71% | 89.87% | 80.79% | 78.00% | 73.06% | 74.92% | |
| Nombre d’insertions uniques | 8,539 | 4,134 | 7,183 | 18,930 | 18,421 | 20,438 | 24,224 | |
| Nombre de gènes touchés | 1575 | 993 | 1450 | 2793 | 2597 | 3037 | 3736 | |
Tableau 9 : Résumé de la sortie de séquençage et de la fréquence d’insertion des transposons par bibliothèque. Abréviation : PE = paired-end.
Discussion
Une bibliothèque de mutants transposon de B. gladioli a été générée pour identifier d’importants facteurs de colonisation de l’hôte dans l’interaction symbiotique entre les coléoptères L. villosa et les bactéries B. gladioli. Les principales étapes du protocole étaient la conjugaison, l’infection de l’hôte, la préparation de la bibliothèque d’ADN et le séquençage.
Comme de nombreuses souches de Burkholderia se prêtent à une modification génétique par conjugaison24,25, le plasmide transportant le transposon et la cassette d’insertion d’antibiotiques a été conjugué avec succès dans la souche cible B. gladioli Lv-StA d’E. coli. Les tentatives précédentes de transformation par électroporation ont donné très peu ou presque pas de transformants de B. glaïe oléoli. Il est conseillé d’optimiser la technique de transformation pour que l’organisme cible produise efficacement un grand nombre de transformants.
Un cycle de conjugaison et 40 points de conjugaison ont perturbé 3 736 gènes chez B. gladioli Lv-StA. Avec le recul, plusieurs cycles de conjugaison seraient nécessaires pour perturber la plupart des 7 468 gènes et obtenir une bibliothèque saturée. Notamment, le temps d’incubation pendant la conjugaison n’a pas été autorisé à dépasser 12-18 h, ce qui est la fin de la phase de croissance exponentielle de B. gladioli. Permettre la conjugaison au-delà de la phase de croissance exponentielle des cellules bactériennes réduit les chances de succès d’obtenir des transconjugants26. Par conséquent, la période de conjugaison doit être ajustée en fonction de la croissance des espèces bactériennes.
Pour mener à bien une expérience impliquant l’infection de bibliothèques mutantes chez un hôte, il est important d’évaluer la taille du goulot d’étranglement de la population bactérienne pendant la colonisation et la diversité des mutants dans la bibliothèque avantl’infection1,2,27. En préparation de l’expérience, nous avons estimé que le nombre minimum de coléoptères qui doivent être infectés a de fortes chances que chaque mutant de la bibliothèque soit échantillonné et autorisé à coloniser. Le temps approximatif de génération bactérienne in vivo et le nombre de générations pour la durée de l’expérience ont également été calculés. La culture in vitro a ensuite été cultivée jusqu’à un nombre comparable de générations en ajustant le temps d’incubation. Pour une expérience d’infection similaire chez d’autres hôtes non modèles, la capacité de maintenir une culture de laboratoire et une source constante des organismes hôtes est souhaitable.
Suite à la croissance de la bibliothèque mutante in vivo et in vitro et de la collecte d’échantillons, un protocole de préparation de bibliothèque d’ADN modifié pour le séquençage d’insertion de transposons a été réalisé. La modification du protocole impliquait la conception d’amorces PCR personnalisées et l’ajout d’étapes PCR à sélectionner pour les fragments d’ADN contenant la cassette d’insertion. Étant donné que le protocole a été personnalisé, des cycles PCR supplémentaires dans le protocole ont augmenté le risque de suramplification et d’obtention de fragments d’adaptateur-adaptateur hybridés dans les bibliothèques finales. Par conséquent, une étape de nettoyage finale (sans sélection de taille) après les deux PCR est recommandée, car elle aide à éliminer ces fragments. La distribution de taille des bibliothèques d’ADN était encore plus large que prévu. Cependant, l’augmentation de la profondeur de séquençage a fourni suffisamment de données qui ont été filtrées lors de l’analyse bioinformatique, obtenant des résultats satisfaisants.
Comme la mutagénèse médiée par transposon génère des milliers d’insertions aléatoires en une seule expérience, il est possible de générer une bibliothèque saturée de mutants qui contient tous sauf ceux où les gènes essentiels à la croissance bactérienne ont été perturbés. Nous n’avons probablement pas travaillé avec une bibliothèque de mutants saturés, compte tenu des estimations de gènes essentiels dans d’autres études sur Burkholderia sp. 28,29. Une bibliothèque non saturée aide néanmoins à explorer divers gènes candidats pour d’autres études utilisant la mutagénèse ciblée. Avant les expériences, il est également important de rappeler que certains transposons ont des sites cibles d’insertion spécifiques qui augmentent l’abondance des mutants à certains loci du génome30. Les transposons mariner sont connus pour cibler les sites AT pour l’insertion31, et les transposons Tn5 ont un biais GC32,33. L’inclusion d’étapes au cours de l’analyse bioinformatique pour reconnaître les points chauds pour les insertions de transposons aidera à évaluer tout biais de distribution.
Bien que sujette à des revers, une expérience de séquençage d’insertion de transposon bien conçue peut être un outil puissant pour identifier de nombreux gènes conditionnellement importants chez les bactéries au sein d’une seule expérience. Par exemple, une douzaine de gènes de Burkholderia seminalis importants pour la suppression de la nécrose des feuilles d’orchidée ont été identifiés en combinant la mutagénèse des transposons et lagénomique 34. Au-delà de Burkholderia,plusieurs gènes et transporteurs d’adhésion et de motilité ont été identifiés comme des facteurs de colonisation importants chez les symbiotes Snodgrassella alvi d’Apis mellifera (Abeille)22, et dans les symbiotes Vibrio fischerii d’Euprymna scolopes (calmar bobtail hawaïen)23 en utilisant l’approche de mutagénèse par insertion de transposon.
Comme approche alternative, la mutagénèse transposon peut être suivie d’un dépistage des mutants individuels à l’aide de milieux sélectifs au lieu du séquençage. Un dépistage phénotypique ou des essais biologiques pour identifier les lacunes, telles que la motilité, la production de métabolites secondaires bioactifs ou d’auxotrophies spécifiques, sont réalisables. Par exemple, le criblage d’une bibliothèque de mutants de transposon Burkholderia insecticola (réaffectée au genre Caballeronia35)a été essentiel pour identifier que les symbiotes utilisent des gènes de motilité pour coloniser Riptortus pedestris,leur insecte hôte36. En outre, en utilisant la mutagénèse transposon et le criblage phénotypique, le groupe de gènes biosynthétiques pour le métabolite secondaire bioactif caryoynencine a été identifié dans Burkholderia caryophylli37. Un mutant auxotrophe de Burkholderia pseudomallei a été identifié à la suite d’une mutagénèse et d’un dépistage par transposon et est un candidat vaccin atténué possible contre la mélioïdose, une maladie dangereuse chez l’homme et l’animal38. Ainsi, la mutagénèse et le séquençage des transposons sont une approche précieuse dans l’étude des traits moléculaires des bactéries qui sont importants pour les interactions avec leurs hôtes respectifs dans des associations pathogènes ou mutualistes.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont aucun conflit d’intérêts relatif à l’étude.
Remerciements
Nous sommes reconnaissants à Junbeom Lee d’avoir fourni la souche E. coli WM3064+pRL27 pour la conjugaison et les conseils dans la procédure, à Kathrin Hüffmeier pour avoir aidé au dépannage lors de la génération de bibliothèques mutantes et au professeur André Rodrigues pour soutenir la collecte d’insectes et l’acquisition de permis. Nous remercions également Rebekka Janke et Dagmar Klebsch pour leur soutien dans la collecte et l’élevage des insectes. Nous reconnaissons les autorités brésiliennes pour l’octroi des permis suivants pour l’accès, la collecte et l’exportation de spécimens d’insectes : autorisation SISBIO n° 45742-1, 45742-7 et 45742-10, procédé CNPq nº 01300.004320/2014-21 et 01300.0013848/2017-33, IBAMA Nr. 14BR016151DF et 20BR035212/DF). Cette recherche a été financée par les subventions de recherche FL1051/1-1 et KA2846/6-1 de la Fondation allemande pour la science (DFG).
matériels
| Name | Company | Catalog Number | Comments |
| 2,6- Diaminopimelic Acid | Alfa Aesar | B22391 | For E.coli WM3064+ pRL27 |
| Agar - Agar | Roth | 5210 | |
| Agarose | Biozym | 840004 | |
| AMPure beads XP (magentic beads + polyethylene glycol + salts) | Beckman Coulter | A63880 | Size selection in step 6.6 |
| Bleach (NaOCl) 12% | Roth | 9062 | |
| Bowtie2 v.2.4.2 | Bioinfromatic tool for read mapping. Reference 10 in main manuscript. | ||
| Buffer-S | Peqlab | PEQL01-1020 | For PCRs |
| Cell scraper | Sarstedt | 83.1830 | |
| Cutadapt v.2.10 | Bioinformatic tool for removing specific adapter sequences from the reads. Reference 8 in main manuscript. | ||
| DESeq2 | RStudio package for assessing differential mutant abundance. Usually used for RNAseq analysis. Reference 12 in main manuscript. | ||
| DNA ladder 100 bp | Roth | T834.1 | |
| dNTPs | Life Technology | R0182 | PCR for confirming success of conjugation |
| EDTA, Di-Sodium salt | Roth | 8043 | |
| Epicentre MasterPure Complete DNA and RNA Purification Kit | Lucigen | MC85200 | |
| Ethidium bromide | Roth | 2218.1 | |
| FastQC v.0.11.8 | Bioinformatic tool for assessing the quality of sequencing data. Reference 7 in main manuscript. | ||
| FeatureCounts v.2.0.1 | Bioinformatic tool to obtain read counts per genomic feature. Reference 11 in main manuscript. | ||
| Glycerol | Roth | 7530 | |
| K2HPO4 | Roth | P749 | |
| Kanamycin sulfate | Serva | 26899 | |
| KCl | Merck | 4936 | |
| KH2PO4 | Roth | 3904 | |
| MgSO4.7H2O | Roth | PO27 | |
| Na2HPO4 | Roth | P030 | |
| NaCl | Merck | 6404 | |
| NEBNext Multiplex Oligos for Illumina (Index primers set 1) | New England Biolabs | E7335S | |
| NEBNext Ultra II DNA library prep kit for Illumina | New England Biolabs | E7645S | |
| Peptone (soybean) | Roth | 2365 | For Burkholderia gladioli Lv-StA KB-medium |
| peqGOLD 'Hot' Taq- DNA Polymerase | VWR | PEQL01-1020 | PCR for confirming success of conjugation |
| Petri plates - 145 x 20 mm | Roth | XH90.1 | For selecting transconjugants |
| Petri plates - 90 x 16 mm | Roth | N221.2 | |
| Qiaxcel (StarSEQ GmbH, Germany) | Quality check after DNA library preparation | ||
| Streptavidin beads | Roth | HP57.1 | |
| Taq DNA polymerase | VWR | 01-1020 | |
| Trimmomatic v.0.36 | Bioinformatic tool for trimming low quality reads and also adapter sequences. Reference 9 in main manuscript. | ||
| Tris -HCl | Roth | 9090.1 | |
| Tryptone | Roth | 2366 | For Escherichia coli WM3064+pRL27 LB medium |
| Ultrasonicator | Bandelin | GM 70 HD | For shearing |
| USER enzyme (uracil DNA glycosylase + DNA glycosylase- lyase Endonuclease VIII) | New England Biolabs | E7645S | Ligation step 6.5.2 |
| Yeast extract | Roth | 2363 |
Références
- Cain, A. K., et al. A decade of advances in transposon-insertion sequencing. Nature Reviews Genetics. 21 (9), 526-540 (2020).
- Chao, M. C., Abel, S., Davis, B. M., Waldor, M. K. The design and analysis of transposon insertion sequencing experiments. Nature Reviews Microbiology. 14 (2), 119-128 (2016).
- Barquist, L., Boinett, C. J., Cain, A. K. Approaches to querying bacterial genomes with transposon-insertion sequencing. RNA Biology. 10 (7), 1161-1169 (2013).
- Flórez, L. V., et al. Antibiotic-producing symbionts dynamically transition between plant pathogenicity and insect-defensive mutualism. Nature Communications. 8 (1), 15172(2017).
- Flórez, L. V., Kaltenpoth, M. Symbiont dynamics and strain diversity in the defensive mutualism between Lagria beetles and Burkholderia. Environmental Microbiology. 19 (9), 3674-3688 (2017).
- Flórez, L. V., et al. An antifungal polyketide associated with horizontally acquired genes supports symbiont-mediated defense in Lagria villosa beetles. Nature Communications. 9 (1), 2478(2018).
- Andrews, S. FastQC A quality control tool for high throughput sequence data. , Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2012).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Liao, Y., Smyth, G. K., Shi, W. FeatureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2014).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550(2014).
- Gaytán, M. O., Martínez-Santos, V. I., Soto, E., González-Pedrajo, B. Type three secretion system in attaching and effacing pathogens. Frontiers in Cellular and Infection Microbiology. 6, 129(2016).
- Hachani, A., Wood, T. E., Filloux, A. Type VI secretion and anti-host effectors. Current Opinion in Microbiology. 29, 81-93 (2016).
- Deep, A., Chaudhary, U., Gupta, V. Quorum sensing and bacterial pathogenicity: From molecules to disease. Journal of Laboratory Physicians. 3 (1), 4-11 (2011).
- Silva, A. J., Benitez, J. A. Vibrio cholerae biofilms and cholera pathogenesis. PLOS Neglected Tropical Diseases. 10 (2), 0004330(2016).
- Navarro-Garcia, F., Ruiz-Perez, F., Cataldi, Á, Larzábal, M. Type VI secretion system in pathogenic Escherichia coli: structure, role in virulence, and acquisition. Frontiers in Microbiology. 10, 1965(2019).
- Ribet, D., Cossart, P. How bacterial pathogens colonize their hosts and invade deeper tissues. Microbes and Infection. 17 (3), 173-183 (2015).
- Schwarz, S., et al. Burkholderia Type VI secretion systems have distinct roles in eukaryotic and bacterial cell interactions. PLoS Pathogens. 6 (8), 1001068(2010).
- Jones, C., et al. Kill and cure: genomic phylogeny and bioactivity of Burkholderia gladioli bacteria capable of pathogenic and beneficial lifestyles. Microbial Genomics. 7 (1), 000515(2021).
- Takeshita, K., Kikuchi, Y. Riptortuspedestris and Burkholderia symbiont: an ideal model system for insect-microbe symbiotic associations. Research in Microbiology. 168 (3), 175-187 (2017).
- Powell, J. E., et al. Genome-wide screen identifies host colonization determinants in a bacterial gut symbiont. Proceedings of the National Academy of Sciences of the United States of America. 113 (48), 13887-13892 (2016).
- Brooks, J. F., et al. Global discovery of colonization determinants in the squid symbiont Vibrio fischeri. Proceedings of the National Academy of Sciences of the United States of America. 111 (48), 17284-17289 (2014).
- Somprasong, N., McMillan, I., Karkhoff-Schweizer, R. R., Mongkolsuk, S., Schweizer, H. P. Methods for genetic manipulation of Burkholderia gladioli pathovar cocovenenans. BMC Research Notes. 3 (308), (2010).
- Garcia, E. C. Burkholderia thailandensis: Genetic manipulation. Current Protocols in Microbiology. 45, 1-15 (2017).
- Headd, B., Bradford, S. A. The conjugation window in an Escherichia coli K-12 strain with an IncFII plasmid. Applied and Environmental Microbiology. 86 (17), 00948(2020).
- Van Opijnen, T., Camilli, A. Transposon insertion sequencing: A new tool for systems-level analysis of microorganisms. Nature Reviews Microbiology. 11 (7), 435-442 (2013).
- Gallagher, L. A., Ramage, E., Patrapuvich, R., Weiss, E., Brittnacher, M., Manoil, C. Sequence-defined transposon mutant library of Burkholderia thailandensis. mBio. 4 (6), 00604-00613 (2013).
- Wong, Y. -C., et al. Candidate essential genes in Burkholderia cenocepacia J2315 identified by genome-wide TraDIS. Frontiers in Microbiology. 7, 1288(2016).
- Moule, M. G., et al. Genome-wide saturation mutagenesis of Burkholderia pseudomallei K96243 predicts essential genes and novel targets for antimicrobial development. mBio. 5 (1), 00926(2014).
- Ding, Q., Tan, K. S. Himar1 transposon for efficient random mutagenesis in Aggregatibacter actinomycetemcomitans. Frontiers in Microbiology. 8, 1842(2017).
- Green, B., Bouchier, C., Fairhead, C., Craig, N. L., Cormack, B. P. Insertion site preference of Mu, Tn5, and Tn7 transposons. Mobile DNA. 3, 3(2012).
- Lodge, J. K., Weston-Hafer, K., Berg, D. E. Transposon Tn5 target specificity: Preference for insertion at G/C pairs. Genetics. 120 (3), 645-650 (1988).
- Ará Ujo, W. L., et al. Genome sequencing and transposon mutagenesis of Burkholderia seminalis TC3.4.2R3 identify genes contributing to suppression of orchid necrosis caused by B. gladioli. Molecular Plant-microbe Interactions: MPMI. 29 (6), 435-446 (2016).
- Dobritsa, A. P., Samadpour, M. Reclassification of Burkholderiainsecticola as Caballeroniainsecticola comb. nov. and reliability of conserved signature indels as molecular synapomorphies. International Journal of Systematic and Evolutionary Microbiology. 69 (7), 2057-2063 (2019).
- Ohbayashi, T., et al. Insect's intestinal organ for symbiont sorting. Proceedings of the National Academy of Sciences of the United States of America. 112 (37), 5179-5188 (2015).
- Ross, C., Scherlach, K., Kloss, F., Hertweck, C. The molecular basis of conjugated polyyne biosynthesis in phytopathogenic bacteria. Angewandte Chemie International Edition. 53 (30), 7794-7798 (2014).
- Atkins, T., et al. A mutant of Burkholderia pseudomallei, auxotrophic in the branched chain amino acid biosynthetic pathway, is attenuated and protective in a murine model of melioidosis. Infection and Immunity. 70 (9), 5290-5294 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon