Method Article
Транспозонно-инстиционное секвенирование как инструмент для выяснения факторов бактериальной колонизации у гладиолусов Burkholderia Симбионт Жуков Lagria villosa
В этой статье
Резюме
Это адаптированный метод выявления потенциальных факторов колонизации насекомых в благотворном симбионсе Burkholderia. Жук-хозяин заражен случайной мутантной библиотекой, генерируемой с помощью транспозонного мутагенеза, а сложность библиотеки после колонизации сравнивается с контролем, выращенным in vitro.
Аннотация
Вывод функции генов путем манипулирования их активностью является важным инструментом для понимания генетических основ большинства биологических процессов. Достижения в молекулярной микробиологии привели к появлению различных методов мутагенеза для манипулирования генами. Среди них транспозонно-инжекционное секвенирование (Tn-seq) является ценным инструментом для одновременной оценки функциональности многих генов-кандидатов нецелевым способом. Этот метод был ключевым для выявления молекулярных механизмов колонизации эукариотических хозяев в нескольких патогенных микробах и нескольких полезных симбионтах.
Здесь Tn-seq установлен как метод выявления факторов колонизации у мутуалистического симбиона Burkholderia gladioli жука Lagria villosa. Путем конъюгации транспозонно-опосредованную Tn5 вставку кассеты с устойчивостью к антибиотикам осуществляют в случайных геномных местах у B. гладиолусов. Чтобы выявить влияние нарушений генов на способность бактерий колонизировать жука-хозяина, сгенерированная транспозонно-мутантная библиотека B. gladioli прививается на яйца жуков, в то время как контроль выращивается in vitro в жидкой питательной среде. После того, как у нас будет достаточно времени для колонизации, ДНК извлекается из библиотек, выращенных in vivo и in vitro. Следуя протоколу подготовки библиотеки ДНК, образцы ДНК готовятся для транспозонно-вставного секвенирования. Фрагменты ДНК, которые содержат транспозонно-вставную кромку и фланкировую бактериальную ДНК, выбираются, а места мутации определяются путем секвенирования вдали от края транспозон-вставки. Наконец, анализируя и сравнивая частоты каждого мутанта между библиотеками in vivo и in vitro, можно предсказать важность конкретных генов симбионта во время колонизации жуков.
Введение
Гладиолусы Burkholderia могут вступать в симбиотическую ассоциацию с жуками Lagria villosa, играя важную роль в защите от микробных антагонистов насекомых-хозяев4,5,6. Самки жуков обуславлживают несколько штаммов B. gladioli в специализированных железах, аксессуарах для репродуктивной системы. При яйцекладке самки размазывают клетки гладиолусов B. на поверхности яйца, где антимикробные соединения, продуцируемые B. gladioli, ингибируют инфекции энтомопатогенными грибами4,6. Во время позднего эмбрионального развития или в начале после вылупления личинок бактерии колонизируют кутиккулярные инвагинации на дорсальной поверхности личинок. Несмотря на такую специализированную локализацию и вертикальный маршрут передачи симбионтов, L. villosa предположительно может также приобретать B. гладиолусов горизонтально из окружающей среды4. Кроме того, по крайней мере три штамма B. gladioli были обнаружены в ассоциации с L. villosa4,6. Среди них B. gladioli Lv-StA является единственным, который поддается выращиванию in vitro.
Б. гладиолусы Lv-StA имеет размер генома 8,56 Mb6 и содержит 7 468 генов. Какие из этих генов важны для бактерий B. gladioli для колонизации жука-хозяина? Чтобы ответить на этот вопрос, мы использовали транспозонно-вставное секвенирование (Tn-seq), исследовательский метод идентификации условно необходимых микробных генов1,2,3. Мутантная библиотека B. gladioli Lv-StA была создана с использованием транспозона Tn5. Путем конъюгации из донорских клеток Escherichia coli в B. gladioli Lv-StA была перенесена плазмида pRL27, несущая транспозон Tn5, и кассета устойчивости к антибиотикам, окруженная перевернутыми повторами(рисунок 1). Таким образом, был сгенерирован набор мутантов, которые по отдельности несут нарушения 3 736 симбионтных генов(рисунок 2).
Пул мутантов был заражен яйцами жуков для выявления факторов колонизации и, в качестве контроля, также выращивался in vitro в среде King's B (KB). После того, как у нас было достаточно времени для колонизации, вылупившиеся личинки были собраны и объединены для извлечения ДНК. Фрагменты ДНК, содержащие транспозонную вставку и фланкировую геномную область B. gladioli Lv-StA, были отобраны с использованием модифицированного протокола подготовки библиотеки ДНК для секвенирования. Была проведена обработка качества считывания с последующим анализом с помощью DESeq2 для выявления специфических генов, имеющих решающее значение для B. gladioli Lv-StA для колонизации личинок L. villosa при передаче через поверхность яйца.
протокол
1. Подготовка среды и буфера
- Готовят KB и LB носители и агаровые пластины, как указано в таблице 1,и автоклав при 121 °C, 15 фунтов на кв. дюйм, 20 мин.
- Добавьте 50 мкг/мл стерилизованного с фильтром канамицина и 300 мкМ стерилизованной фильтрующей 2,6-диаминопимеловой кислоты (DAP) в автоклавную среду LB перед культивированием E.coli WM3064 + pRL27.
- Добавьте 50 мкг/мл стерилизованного с фильтром канамицина в автоклавный агар KB для заливки пластин, необходимых для выбора успешных трансконъюгантов B. gladioli Lv-StA.
- Готовят 1x фосфатно-буферный физиологический раствор (PBS), смешивая следующие компоненты: NaCl 8 г/л, KCl 0,201 г/л, Na2HPO4 1,42 г/л и KH2PO4 0,272 г/л. Растворите соли в дистиллированной воде и автоклаве смеси при 121 °C, 15 psi, за 20 мин до использования. Хранить при комнатной температуре.
- Приготовьте 2-кратный буфер для связывания и промывки, растворив следующие компоненты: 10 мМ Tris-HCl (рН 7,5), 1 мМ этилендиамина тетрауксусной кислоты (ЭДТА) и 2 М NaCl в дистиллированной воде. Фильтруйте-стерилизуйте смесь перед использованием. Хранить при комнатной температуре.
- Готовят 1x Low-TE, растворяя 10 мМ Tris-HCl (рН 8,0) и 0,1 мМ ЭДТА в двухдистиллированной воде. Стерилизовать автоклавами при 121 °C, 15 фунтов/усик, 20 мин. Хранить при комнатной температуре.
2. Сопряжение для генерации библиотеки транспозонов мутантов
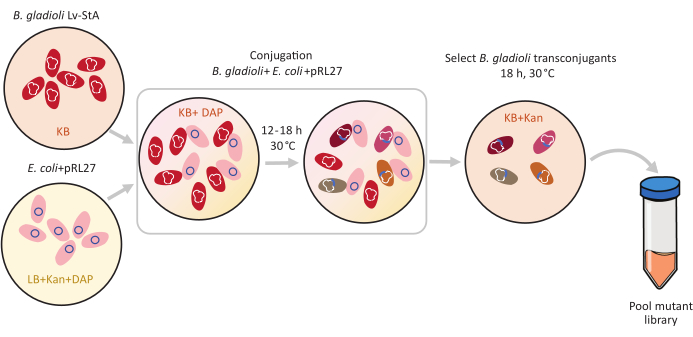
Рисунок 1:Этапы протокола сопряжения. Реципиент конъюгации Burkholderia гладиолусы Lv-StA (красный) и донорская кишечная палочка, содержащие плазмиду pRL27 (розовая), выращивают в агаре KB и LB, соответственно, дополненном канамицином и DAP. После конъюгативного переноса плазмиды в течение 12-18 ч при 30 °С трансконъюгантные клетки гладиолусов B. отбирают на КБ, содержащих канамицин, и объединяют вместе. Сокращения: DAP = 2,6-диаминопимелевая кислота; Кан = канамицин. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
- Под стерильным капюшоном привить свежую донорскую культуру Escherichia coli WM3064 + pRL27 в 10 мл LB-среды, дополненной канамицином и DAP. Инокулируют клетки реципиента Burkholderia gladioli Lv-StA в 5 мл среды КБ. Инкубировать культуры при 30 °C в течение ночи на шейкере при 250 об/мин.
- После ночного роста центрифугируют 4 мл каждой из культур по 9 600 × г в течение 6 мин, чтобы гранулировать клетки. Выбросьте супернатант.
- Под стерильным капюшоном промыть гранулированные клеточные культуры в среде KB, содержащей DAP, и, наконец, повторно суспендовать культуры отдельно в 4 мл среды KB + DAP.
- В свежей пробирке объемом 15 мл смешайте 250 мкл промытых донорских клеток E. coli с 1 мл промытых клеток реципиента Lv-StA B. gladioli.
- Надень 10 мкл этой смеси конъюгационных клеток на пластины агара KB, содержащие DAP. Дайте пластине спокойно отдохнуть в стерильной вытяжке при комнатной температуре в течение 1 ч. Затем инкубируют пластины с пятнами сопряжения при 30 °C в течение 12-18 ч.
ПРИМЕЧАНИЕ: Период спряжения может быть скорректирован в соответствии с целевыми видами. Однако длительный период конъюгации увеличивает риск двойных вставок или интеграции плазмид в геном. Для медленно растущих бактерий допускайте более длительные периоды конъюгации. - После инкубации добавьте 2-4 мл 1x PBS в пластины под стерильным капюшоном и используйте клеточный скребок, чтобы освободить выращенные бактериальные конъюгационные пятна из агара. Пипетка конъюгированной клеточной смеси в 2 мл микрофьюжных пробирок.
- Гранулируют ячейки центрифугированием при 9 600 × г в течение 2 мин. Выбросьте супернатант и промыть гранулу дважды в 1 мл 1x PBS путем пипетки вверх и вниз. Повторное суспендирование конечной гранулы в 1200 мкл 1x PBS. Делают разведения перед покрытием, если количество ячеек в смеси выше 1 × 104.
- Хорошо перемешать и выложить 200 мкл клеточной смеси на большие пластины агара KB (6 или более, если это необходимо), дополненные канамицином, и инкубировать при 30 °C в течение ночи.
ПРИМЕЧАНИЕ: Целевые колонии мутантов появляются в течение 30 ч на селективных агаровых пластинах. Из-за маркера устойчивости к антибиотикам на селективной агартовой пластине появляются только мутировавшие колонии. Поэтому ожидается, что все колонии будут успешными трансконъюгантами. - Подсчитайте общее количество трансконъюгантных колоний на трех пластинах и экстраполируйте, чтобы вычислить приблизительное количество мутантов, полученных во всех пластинах. Чтобы увеличить шансы на получение репрезентативной библиотеки, убедитесь, что общее количество колоний в несколько раз превышает общее количество генов в геноме. Чтобы подтвердить успешность конъюгации, выполните ПЦР, нацеленную на вставную кассету, используя 10-20 колоний образцов, как описано в разделе 3.
ПРИМЕЧАНИЕ: Цель состоит в том, чтобы гарантировать, что количество колоний по крайней мере в 10 раз больше числа генов во всем геноме, в данном случае >75 000 мутантов. Однако, как правило, трудно точно оценить количество колоний, которые соответствовали бы полностью репрезентативной библиотеке. Количество мутировавших уникальных генов на данный момент не очевидно, учитывая, что нарушения в основных генах не улавливаются, часто существует несколько различных участков мутаций для одного и того же гена, а мутации, генерируемые транспозонами Tn5, не являются полностью случайными. - Под стерильным капюшоном соскребают колонии с пластин, добавляя 1-2 мл 1x PBS на агар. Смешанный с пластин клеточной смеси сваливают в пробирки по 50 мл. Вихрь библиотеки, чтобы тщательно смешать, а затем разделить 4 мл объединенной библиотеки мутантов на несколько криотуб. Добавить в пробирки 1 мл 70% глицерина и хранить при -80 °C.
3. ПЦР и гель-электрофорез для подтверждения успешных введений в B. гладиолусы Lv-StA
- Чтобы подтвердить наличие вставки, выберите отдельные колонии мутантов из селекционных пластин на шаге 2.9 и выполните ПЦР, нацеленную на вставную кассету, используя праймеры, перечисленные в таблице 2. Подготовьте мастер-смесь ПЦР в соответствии с таблицей 3 и установите условия в термоциклере, как описано в таблице 4.
- Запустите продукты ПЦР на 1,6% агарозном гелевом путем электрофореза (250 В, 40 мин), чтобы проверить, имеют ли амплифицированные фрагменты ДНК ожидаемую длину 1580 bp.
4. Заражение мутантного пула на яйцах жуков
- Этапы мойки библиотеки
- Разморозить аликвоту подготовленной мутантной библиотеки на льду. Центрифугу при 2,683 × г в течение 10 мин и удаляют супернатант. Под стерильным капюшоном промыть клетки 4 мл 1x PBS, чтобы удалить любую оставшуюся среду из клеток. Повторное суспендирование клеток в 4 мл 1x PBS.
- Подсчитайте количество ячеек в аликвоте библиотеки с помощью камеры подсчета ячеек. Разбавьте часть библиотеки до 2 × 106 ячеек/мкл в 1x PBS.
- Тщательно перемешайте библиотеку, чтобы однородно перемешать всю библиотеку, прежде чем взять требуемый объем.
- Стерилизация кладки яиц и инфекция in vivo
- Выберите кладку яиц L. villosa. Подсчитайте количество яиц и продолжайте, если кладка содержит более 100 яиц.
- Стерилизуйте всю яичную кладку.
- Добавьте 200 мкл 70% этанола и аккуратно вымойте яйца в течение 5 минут. Удалите этанол и дважды вымойте яйца автоклавной водой.
- Добавьте 200 мкл 12% отбеливателя (NaOCl) и осторожно вымойте яйца в течение 30 с. Немедленно удалите отбеливатель и снова вымойте яйца еще три раза 200 мкл автоклавной воды.
- Инфицировать 2 × 106 клеток/мкл промытой мутантной библиотеки на стерилизованной кладке яиц (2,5 мкл на яйцо).
- Через два дня после того, как зараженные личинки жука вылупляются, соберите 1002-х личинок в 1,5 мл микрофьюжной трубки и храните при -80 °C.
-
Управление библиотекой мутантов in vitro
- Под стерильным капюшоном прививкуют 250 мкл 2 × 106 клеток/мкл промытой мутантной библиотеки в 10 мл КБ среды, содержащей канамицин.
- Инкубируют мутантную культуру in vitro при 30 °C в течение 20 ч.
ПРИМЕЧАНИЕ: Рассчитайте продолжительность инкубации, чтобы она соответствовала приблизительное количество поколений WT B. гладиолусов Lv-StA in vivo во время колонизации. - После 20-ч инкубации добавляют равный объем 70% глицерина в мутантную культуру in vitro и хранят ее при -80 °C.
5. Зараженные жуки и in vitro мутантная библиотека извлечения ДНК
ПРИМЕЧАНИЕ: Экстракция ДНК была выполнена с использованием набора для очистки ДНК и РНК в соответствии с протоколом производителя, кратко изложенным ниже.
- Гомогенизировать объединенные личинки (максимум 4 мг на микрофьюжную трубку), добавив 1-2 мл жидкого азота и измельчив пестиком.
- Разморозить in vitro выращенные мутантные культуры из запасов глицерина на льду. Гранулируют клетки центрифугированием при 9 600 × г в течение 10 мин перед лизисом клеток.
- Добавьте 300 мкл раствора лизиса тканей и клеток в образцы in vitro и in vivo. Добавить 5 мкл 10 мг/мл протеиназы К, инкубировать смесь при 60 °C в течение 15 мин, а затем поместить на лед на 3-5 мин.
- Добавьте 150 мкл реагента осаждения белка к лизатам и тщательно вихрь. Гранулируют белковый мусор центрифугированием при 9 600 × г в течение 10 мин.
- Перенесите супернатант в микрофьюжную трубку 1,5 мл. Добавьте 500 мкл изопропанола к надпосытной веществу и осторожно перевергируйте пробирки по меньшей мере 40 раз перед инкубацией при -20°С в течение 1 ч или на ночь.
- Гранулируют осажденную ДНК путем центрифугирования при 9 600 × г в течение 10 мин. Отбросьте супернатант и добавьте ледяной 70% этанол в гранулу ДНК.
- Центрифуга при ≥10 000 × г в течение 5 мин. Выбросьте супернатант и оставьте образцы сушиться на воздухе не менее 1 ч.
- Повторное суспендирование ДНК из образцов in vitro и in vivo в 100 мкл буфера Low-TE.
- Храните образцы при -20 °C.
6. Подготовка библиотеки секвенирования
ПРИМЕЧАНИЕ: Протокол и реагенты для подготовки библиотеки ДНК адаптированы и модифицированы из инструкций, предоставленных производителем комплекта для подготовки библиотеки ДНК.
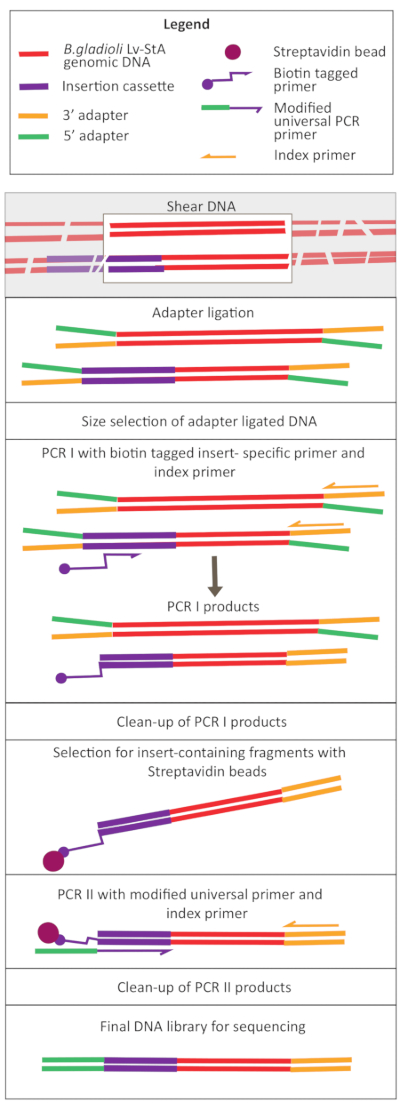
Рисунок 2:Схема этапов подготовки библиотеки ДНК. После сдвига и лигирования адаптера модифицированный протокол включает этап выбора шарика стрептавидина для обогащения фрагментов ДНК, содержащих вставную кассету. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
- Разбавьте образцы до концентрации 20 нг/мкл и объема 100 мкл и удерживайте их на льду.
- Сдвиг in vivo и in vitro образца ДНК с помощью ультразвукового аппарата. Установите ультразвуковой аппарат на мощность 70%. Вихрь образцов ненадолго и сдвиг в течение 1 мин 30 с.
ПРИМЕЧАНИЕ: Настройки ультразвукового прибора будут отличаться в зависимости от прибора. В этом случае размер фрагмента составлял 200-400 bp, что подходит для этого подхода секвенирования 150 bp, сопряженного конца (см. шаг 9.1). Параметры сдвига могут быть отрегулированы в соответствии с требованиями экспериментатора. - Проверьте, была ли ДНК ссжена до нужного диапазона размеров (в данном случае 200-400 bp). Загрузка 5 мкл нестрепаемой и среженной ДНК после смешивания с гелевым нагрузочным красителем в соотношении 1:1 на 1,6% агарозном геле при 250 В в течение 40 мин(рисунок 3А,В).
- Подготовка концов фрагментов, необходимых для перевязки адаптера
- К 50 мкл среженной ДНК добавляют к концу препарата реагенты, приведенные в библиотечном наборе для приготовления: 3 мкл ферментной смеси и 7 мкл реакционного буфера и хорошо перемешивают путем пипетирования. Установите термоциклер с нагретой крышкой при ≥ 75 °C и инкубирует образцы в течение 30 мин при 20 °C и 30 мин при 65 °C. Удерживать при 4 °C.
- Лигирование адаптера
- Для лигирования адаптером добавьте следующие реагенты к продуктам конечной стадии приготовления: 30 мкл Ligation Master Mix, 1 мкл Ligation Enhancer и 2,5 мкл разбавленный адаптер. Тщательно перемешайте путем пипетки и инкубирует образец в течение 15 мин при 20 °C в термоциклере с отсоехшей крышкой.
- Через 15 мин добавляют 3 мкл фермента (урацил ДНК гликозилаза + ДНК гликозилаза-лиаза Эндонуклеаза VIII) (см. Таблицу материалов). Хорошо перемешайте путем пипетки и инкубирует образец в течение 15 мин при 37 °C в термоциклере с крышкой, нагретой при ≥47 °C.
ПРИМЕЧАНИЕ: Протокол может быть приостановлен на этом этапе, а образцы могут быть сохранены при -20 °C.
- Подбор размера адаптерно-лигированных фрагментов ДНК, нацеленных на 250 bp
- Вихрь раствора магнитного шарика (см. Таблицу материалов)и поместите его при комнатной температуре в течение 30 мин перед использованием.
- Добавьте 0,3 раза шариков к 96,5 мкл лигированной смеси ДНК и тщательно перемешайте, пипетируя. Инкубировать бисероплетение в течение 5 мин.
ПРИМЕЧАНИЕ: Наличие солей и полиэтиленгликоля в бисерной смеси облегчает осаждение фрагментов ДНК на шариках. Низкое соотношение бусин к молекулам ДНК приводит к связыванию с бусинами только более крупных фрагментов ДНК. В этом случае фрагменты ДНК длиной выше 250 bp связаны с шариками. - Поместите трубки на магнитную подставку, чтобы стянуть бусины и удалить фрагменты ДНК нежелательного размера. Дайте шарикам отстояться в течение 5 минут, а затем перенесите прозрачный супернатант в новую микрофьюжную трубку (сохраните супернатант).
- Добавьте 0,15x свежего бисера к супернатанту и хорошо перемешайте, пипетировка. Инкубировать смесь шариков в течение 5 минут, а затем поместить трубки на магнитную подставку, чтобы стянуть шарики, связанные с целевой ДНК. Подождите 5 минут, а затем выбросьте супернатант (сохраните бусины).
ПРИМЕЧАНИЕ: Такое соотношение бусин к ДНК приводит к связыванию фрагментов желаемого размера 250 bp. - Выложите бусины на магнитную подставку, добавьте 200 мкл 80% этанола (свежеприготовленного) и подождите 30 с. Выбрасывайте и выбрасывайте этанол тщательно, не нарушая бусины на магнитной подставке. Повторите этот шаг.
- После последней стирки удалите следы этанола с бусин, а затем высушите бусины на воздухе в течение 2 минут, пока они не покажутся глянцевыми, но не полностью высохшими. Не пересушивайте бусины.
- Снимите трубки с магнитного стенда и добавьте 17 мкл 10 мМ Tris-HCl или 0,1x TE (Low-TE). Перемешать путем пипетки ~10 раз и инкубировать смесь при комнатной температуре в течение 2 мин.
- Поместите трубки обратно на магнитную подставку и подождите 5 минут. Как только шарики уляжется, перенесите супернатант ДНК в новую трубку.
- ПЦР I для добавления метки биотина к фрагментам ДНК, содержащим вставную кассету
- Добавьте метку биотинилированной грунтовки к фрагментам ДНК, содержащим Tn5-вставную кассету, используя транспозонно-специфическую биотинилированную грунтовку(таблица 5)и индексную грунтовку. Подготовьте мастер-микс ПЦР в соответствии с таблицей 6 и следуйте условиям ПЦР для теплового циклира, перечисленных в таблице 7.
- Очистка ПЦР I без подбора размера
- Вихрь 0,9x бусины и поместите их при комнатной температуре не менее чем на 30 минут перед очисткой.
- Добавьте 0,9 бусины в продукты ПЦР и тщательно перемешайте.
- Поместите бусины на магнитную подставку, чтобы стянуть бусины.
- Удалите прозрачный супернатант и дважды промыть связанную с шариками ДНК 200 мкл свежеприготовленного 80% этанола.
- Удалите этанол после этапов стирки и высушите шарики на воздухе, пока они не будут выглядеть глянцевыми, но не слишком сухими.
- Добавьте 32 мкл 10 мМ Tris-HCl или 0,1X TE (Low-TE) и инкубируют шарики в течение 5 мин. Поместите смесь обратно на магнитную подставку и перенесите супернатант в свежую микрофьюжную трубку.
- Связывание биотинилированных фрагментов ДНК с шариками стрептавидина
- Повторное суспендирование 32 мкл бусин стрептавидина в 1x буфере связывания и промывки. Трижды вымойте бусины с буфером, поместившись на магнитную подставку.
- Добавьте 32 мкл 2-кратного буфера для связывания и промывки и повторно суспендируете бусины. К этому добавляют 32 мкл очищенных продуктов PCR 1. Тщательно перемешать и инкубировать при комнатной температуре в течение 30 мин.
- Поместите смесь шарика и ДНК на магнитную подставку на 2 мин. Пипетка из супернатанта в виде биотин-помеченной ДНК, содержащей край вставки, связывается со стрептавидином на шариках.
- Вымойте бусины с 500 мкл 1x буфера для связывания и промывки, а затем вымойте бусины с 200 мкл Low-TE. Повторное суспендирование связанных с ДНК шариков в 17 мкл Low-TE.
- PCR II для добавления адаптеров к фрагментам, содержащим край вставной кассеты
- Готовят мастер-микс, как показано в таблице 8,используя индексные праймеры и модифицированные универсальные ПЦР-праймеры, перечисленные в таблице 5. Добавьте 15 мкл связанных с ДНК шариков стрептавидина с предыдущего этапа в смесь ПЦР. В таблице 7 приведены условия теплового циклира.
- Очистите продукты ПЦР без выбора размера, как указано на этапе 6.8 этого протокола. Элюют конечные продукты ДНК в 30 мкл молекулярной воды.
- Храните образцы при -20 °C и используйте их для секвенирования.
7. Секвенирование и анализ
- Виртуализация библиотеки с помощью технологии виртуализации с высокой пропускной способностью. Отрегулируйте глубину виртуализации в зависимости от размера библиотеки транспозонов, как указано ниже. Оцените качество чтения с помощью FastQC7. Выберите чтение, содержащее край вставки Tn5 на 5-м конце считывания, и удалите последовательность краев вставки с помощью Cutadapt8 и/или Trimmomatic9.
ПРИМЕЧАНИЕ: Здесь использовался парный подход к секвенированию для нацеливания на 150 bp на чтение и в общей сложности 8 Mio чтения. Чтобы получить репрезентативный набор данных, убедитесь, что общее число секвенированных считывания превышает максимально возможное число мутантов в библиотеке, то есть общее оценочное число колоний из шага 2.9. В качестве справки, этот протокол нацелен на 40-кратный максимально возможный размер библиотеки. Другие успешные исследования с использованием Tn-seq для аналогичной цели секвенировали общее число считывания, близкое к 25-кратному фактическому числу уникальных вставок в соответствующей библиотеке мутантов22,23. - Учитывая, что мутации на концах генов не являются функционально разрушительными, обрежьте на 5% оба конца аннотаций генов файла GFF эталонного генома. Сопоставьте обрезанные чтения с эталонным геномом с помощью Bowtie210.
- Рассчитайте количество вставок из числа уникальных позиций 5' в файле выравнивания BAM.
- Используя FeatureCounts11,получите количество генов попадания для каждого реплицированного образца.
- Используя пакет DESeq212 в RStudio, рассчитайте разницу в численности мутантов между различными условиями.
Результаты
Бактерии, связанные с хозяином, могут использовать несколько факторов для установления ассоциации, включая те, которые опосредовывают адгезию, подвижность, хемотаксис, стрессовые реакции или специфические транспортеры. В то время как факторы, важные для взаимодействия патогена ихозяина,были зарегистрированы для несколькихбактерий 13, 14,15, 16,17,18,включая членов рода Burkholderia19,20,меньше исследований изучали молекулярные механизмы, используемые полезными симбионтами для колонизации21,22,23 . Используя транспозонное секвенирование вставки, цель состояла в том, чтобы идентифицировать молекулярные факторы, которые позволяют B. gladioli колонизировать жуков L. villosa.
Транспозонно-опосредованный мутагенез выполняли с использованием плазмиды pRL27, которая несет транспозон Tn5 и кассету с резистентностью к канамицину, окруженную инвертными повторными сайтами. Плазмиду вводили в клетки-мишени B. gladioli Lv-StA путем конъюгации с плазмидным донором штаммом E. coli WM3064 (как показано на рисунке 1). После конъюгации конъюгационную смесь, содержащую реципиент B. гладиолусов и донорские клетки E. coli, накладывали на селективные агаровые пластины, содержащие канамицин. Отсутствие DAP на пластинах устраняло донорские клетки E. coli, а наличие канамицина отбирали для успешных B. гладиолусов Lv-StA трансконъюганты. Объединенная библиотека мутантов B. gladioli Lv-StA, полученная в результате сбора 100 000 трансконъюгантных колоний, была подготовлена для секвенирования с использованием модифицированного набора для подготовки библиотеки ДНК и пользовательских праймеров. На рисунке 2 выделены этапы подготовки библиотеки ДНК. Секвенирование дало 4 Mio парных считывания; 3 736 генов из 7 468 генов у B. гладиолусов Lv-StA были нарушены.
Чтобы идентифицировать мутантов, которые были дефектными колонизации в хозяине, библиотека мутантов B. gladioli Lv-StA была заражена яйцами жуков и выращена in vitro в среде KB в качестве контроля. Размер узкого места колонизации in vivo был рассчитан до эксперимента. Известное количество клеток B. gladioli Lv-StA было заражено на яйцах жуков, а количество колонизирующих клеток в только что вылупившихся первых личинках были получены путем обшивки суспензии от каждой личинки и подсчета колониеобразующих единиц на особь. Эти расчеты были сделаны для того, чтобы количество колонизирующих клеток было достаточным для оценки всех или высокого процента мутантов в библиотеке на способность колонизировать хозяина. Кроме того, время роста между условиями in vitro и in vivo было нормализовано на основе количества бактериальных поколений, чтобы сделать эти образцы сопоставимыми.
После того, как яйца вылупились, 1 296 личинок были собраны в 13 бассейнах. Соответствующие мутантные культуры in vitro выращивали и хранили в виде запасов глицерина. ДНК выращенных in vivo и in vitro библиотек мутантов была извлечена и фрагментирована в ультразвуковом аппарате. На рисунке 3 показано распределение размеров среженной ДНК, где большинство фрагментов охватывают от 100 до 400 bp, как и ожидалось. За этим шагом последовал модифицированный протокол подготовки библиотеки ДНК для секвенирования. На каждом этапе протокола проверялась концентрация оставшейся ДНК, чтобы убедиться, что шаги были выполнены правильно и отслеживать потери ДНК. Проверка качества (см. Таблицу материалов)перед секвенированием показала, что библиотеки ДНК содержали неожиданно большие (>800 bp) фрагменты ДНК, и это было более выражено в библиотеках in vivo. Учитывая сложность оптимизации кластеризации фрагментов в полосах секвенирования, для достижения желаемого количества считывания необходимо было увеличить глубину секвенирования до 10 млн парных считывания в библиотеках in vivo. Анализ результатов секвенирования показал, что в среднем 4 Mio чтения в библиотеках in vivo и 3,1 Mio чтения в библиотеках in vitro содержали край Transposon в 5' конце Read-1(таблица 9),что было удовлетворительно для этого эксперимента. Распределение 24 224 уникальных вставок по геному B. гладиолусов в исходной библиотеке показано на рисунке 4. Анализ, проведенный с использованием DESeq2, показал, что численность 271 мутанта значительно различалась между условиями in vivo и in vitro.
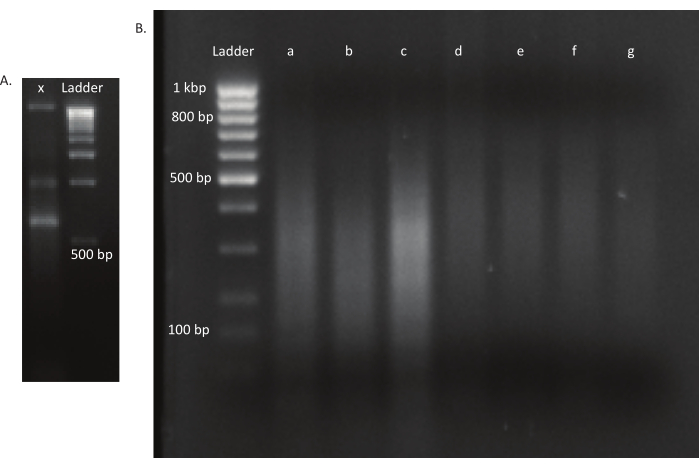
Рисунок 3:Агарозные гели мутанта и библиотеки ДНК. (А) Агарозный гель с неслышаной ДНК мутанта в полосе X и лестницей 1 kbp для масштаба. (B) Гель со среженной библиотекой ДНК. Размеры полос лестницы в первой полосе указаны с левой стороны. Первые три полосы a, b и c содержат среженные фрагменты ДНК библиотек in vivo. Полосы d, e, f и g содержат среженные фрагменты ДНК библиотек in vitro. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
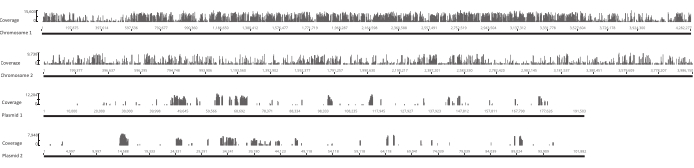
Рисунок 4:Расположение уникальных мест вставки в исходной библиотеке по четырем репликонам в геноме Гладиолусов Буркхолдерии Lv-StA. Каждый стержень вдоль оси X расположен в месте вставки. Высота полосы вдоль оси Y соответствует количеству считывания, связанных с этим сайтом. Обратите внимание, что две хромосомы и две плазмиды показаны в полной длине и, таким образом, имеют разные масштабы на оси X. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
| King's B средний/ агар | |
| Пептон (соя) | 20 г/л |
| К2ХПО4 | 1,5 г/л |
| MgSO4.7H2O | 1,5 г/л |
| Агар | 15 г/л |
| Растворенный в дистиллированной воде | |
| LB средний/агар | |
| Триптон | 10 г/л |
| Дрожжевой экстракт | 5 г/л |
| НаКл | 10 г/л |
| Растворенный в дистиллированной воде | |
Таблица 1: Компоненты носителей.
| Нет. | Грунтовки | Последовательность | Температура отжига ПЦР (°C) | |
| 1 | tpnRL17–1RC | 5'-CGTTACATCCCTGGCTTGTT-3' | 58.2 | |
| 2 | tpnRL13–2RC | 5'-ТКГТГАААГГТТТГЦТГ-3' | ||
Таблица 2: Буквари для подтверждения успешности спряжения.
| Компонент | Объем (мкл) |
| Вода, очищенная от ВЭЖХ | 4.92 |
| 10x Buffer S (высокая специфичность) | 1 |
| MgCl2 (25 мМ) | 0.2 |
| дНТП (2 мМ) | 1.2 |
| Грунтовка 1 (10 пмоль/мкл) | 0.8 |
| Грунтовка 2 (10 пмоль/мкл) | 0.8 |
| Taq (5 Едк/мкл) | 0.08 |
| Итого мастермикса | 9 |
| Шаблон | 1 |
Таблица 3: Мастер-микс ПЦР для подтверждения успешности конъюгации. Сокращения: ВЭЖХ = высокоэффективная жидкостная хроматография; dNTPs = дезоксинуклеозидтрифосфат.
| Стремянка | Температура °C | Время | Циклов |
| Начальная денатурация | 95 | 3 мин | 1 |
| Денатурация | 95 | 40 с | |
| Отжиг | 58.2 | 40 с | от 30 до 35 |
| Расширение | 72 | 1-2 мин | |
| Окончательное расширение | 72 | 4 мин | 1 |
| Держать | 4 | ∞ | |
Таблица 4: Условия ПЦР для подтверждения успешности конъюгации.
| Грунтовки | Последовательность | Тм °C | Использование | Источник | |
| Транспозонно-специфическая биотинилированная праймер | 5'-Биотин-ACAGGAACACTTAACGGCTGACATG -3' | 63.5 | 6.7.1. ПЦР I | Обычай | |
| Модифицированная универсальная ПЦР-праймер | 5'- ААТГАТАЦГГГГГАККАКГАГАГАЦК TACACTCTTTCCCTACACGACGCTC TTCCGATCTGAATTCATCGATGAT ГГТТГАГАТГТГТ – 3' | 62 | 6.10.1. ПЦР II | Обычай | |
| Индексная букварь | Обратитесь к руководству по эксплуатации | 6.7.1. ПЦР I и 6.10.1. ПЦР II | NEBNext Мультиплекс Олиго для Illumina (Индекс праймеров набор 1) | ||
| Адаптер | Обратитесь к руководству по эксплуатации | 6.5. Лигирование адаптера | Набор для подготовки библиотеки ДНК NEBNext Ultra II для Illumina | ||
Таблица 5: Праймеры и адаптер для ПЦР I и II при подготовке библиотеки ДНК.
| ПЦР-микс | (мкл) |
| Адаптерно-лигированные фрагменты ДНК | 15 |
| NEBNext Ultra II Q5 мастер-микс | 25 |
| Индексная грунтовка (10 пмоль/мкл) | 5 |
| Транспозонно-специфическая биотинилированная праймера (10 пмоль/мкл) | 5 |
| Общий объем | 50 |
Таблица 6: Подготовка библиотеки ДНК -мастер-микс ПЦР I.
| Стремянка | Температура | Время | Циклов |
| Начальная денатурация | 98 °С | 30 с | 1 |
| Денатурация | 98 °С | 10 с | от 6 до 12 |
| Отжиг | 65 °С | 30 с | |
| Расширение | 72 °С | 30 с | |
| Окончательное расширение | 72 °С | 2 мин | 1 |
| Держать | 16 °С | ∞ | |
Таблица 7: Подготовка библиотеки ДНК -условия ПЦР I и II.
| ПЦР-микс | (мкл) |
| ДНК, отобранная из бисера | 15 |
| NEBNext Ultra II Q5 мастер-микс | 25 |
| Индексная букварь | 5 |
| Модифицированный универсальный ПЦР-праймер | 5 |
| Общий объем | 50 |
Таблица 8: Подготовка библиотеки ДНК -мастер-микс PCR II.
| Библиотеки | Инвиво-1 | Инвиво-2 | Инвиво-3 | Инвитро-1 | Инвитро-2 | Инвитро-3 | Оригинальная библиотека | |
| Нет. количества считываний (PE) | 56,57,710 | 39,19,051 | 30,65,849 | 35,73,494 | 28,83,440 | 36,61,956 | 46,09,410 | |
| Нет. считывания, содержащего Tn – край на 5' конце Read-1 | 54,15,880 | 37,31,169 | 29,36,247 | 33,00,499 | 27,35,705 | 33,50,402 | 41,53,270 | |
| Общая скорость выравнивания Bowtie2 (%) (только чтение-1) | 95.53% | 83.71% | 89.87% | 80.79% | 78.00% | 73.06% | 74.92% | |
| Количество уникальных вставок | 8,539 | 4,134 | 7,183 | 18,930 | 18,421 | 20,438 | 24,224 | |
| Количество пораженных генов | 1575 | 993 | 1450 | 2793 | 2597 | 3037 | 3736 | |
Таблица 9: Сводка выходных данных секвенирования и частоты вставки транспозонов на библиотеку. Аббревиатура: PE = парный конец.
Обсуждение
Была создана библиотека транспозонов B. gladioli для выявления важных факторов колонизации хозяина в симбиотическом взаимодействии между жуками L. villosa и бактериями B. gladioli. Основными шагами в протоколе были конъюгация, инфекция хозяина, подготовка библиотеки ДНК и секвенирование.
Поскольку многие штаммы Burkholderia поддаются генетической модификации путем конъюгации24,25,плазмида, несущая транспозон и кассету вставки антибиотика, была успешно конъюгирована в штамм-мишень B. gladioli Lv-StA из E. coli. Предыдущие попытки трансформации электропорацией дали очень низкие до почти никакого уровня трансформанты гладиолусов B. Целесообразно оптимизировать технику трансформации для организма-мишени, чтобы эффективно выдавать большое количество трансформантов.
Один раунд конъюгации и 40 конъюгационных пятен нарушили 3 736 генов у B. gladioli Lv-StA. Оглядываясь назад, можно сказать, что для разрушения большинства из 7 468 генов и получения насыщенной библиотеки потребуется несколько раундов конъюгации. Примечательно, что время инкубации при сопряжении не позволяло превышать 12-18 ч, что является концом фазы экспоненциального роста B. гладиолусов. Разрешение конъюгации за пределами фазы экспоненциального роста бактериальных клеток снижает шансы на успех получения трансконъюгантов26. Поэтому период конъюгации следует корректировать в соответствии с ростом видов бактерий.
Для успешного проведения эксперимента, связанного с заражением мутантных библиотек у хозяина, важно оценить размер узкого места бактериальной популяции во время колонизации и разнообразие мутантов в библиотеке до заражения1,2,27. При подготовке к эксперименту мы оценили минимальное количество жуков, которые должны быть заражены, чтобы иметь высокую вероятность того, что каждый мутант в библиотеке будет отобран и допущен к колонизации. Также были рассчитаны приблизительное время генерации бактерий in vivo и количество поколений за время эксперимента. Затем культура in vitro была выращена до сопоставимого числа поколений путем корректировки времени инкубации. Для аналогичного эксперимента с инфекцией на других немоделовых хозяевах желательна способность поддерживать лабораторную культуру и постоянный источник организмов-хозяев.
Вслед за ростом мутантной библиотеки in vivo и in vitro и коллекции образцов был проведен модифицированный протокол подготовки библиотеки ДНК для секвенирования вставки транспозонов. Модификация протокола включала разработку пользовательских праймеров ПЦР и добавление шагов ПЦР для выбора фрагментов ДНК, содержащих вставную кассету. Поскольку протокол был настроен, дополнительные циклы ПЦР в протоколе увеличивали риск чрезмерной амплификации и получения гибридизированных фрагментов адаптер-адаптер в конечных библиотеках. Следовательно, рекомендуется заключительный этап очистки (без выбора размера) после двух PCR, поскольку он помогает в удалении этих фрагментов. Распределение по размерам библиотек ДНК все еще было шире, чем ожидалось. Однако увеличение глубины секвенирования обеспечило достаточные данные, которые были отфильтрованы в ходе биоинформатического анализа, получив удовлетворительные результаты.
Поскольку транспозонно-опосредованный мутагенез генерирует тысячи случайных вставок в одном эксперименте, можно создать насыщенную библиотеку мутантов, которая содержит все, кроме тех мутантов, где гены, необходимые для роста бактерий, были нарушены. Мы, скорее всего, не работали с насыщенной мутантной библиотекой, учитывая оценки основных генов в других исследованиях Burkholderia sp. 28,29. Тем не менее, ненасыщенная библиотека помогает в изучении различных генов-кандидатов для дальнейших исследований с использованием целевого мутагенеза. Перед экспериментами также важно помнить, что некоторые транспозоны имеют специфические сайты-мишени вставки, которые увеличивают обилие мутантов в определенных локусах в геноме30. Известно, что транспозоны Mariner нацелены на AT-сайты для вставки31,а транспозоны Tn5 имеют смещение GC32,33. Включение шагов во время биоинформатического анализа для распознавания горячих точек для транспозонных вставок поможет в оценке любой предвзятости распределения.
Несмотря на склонность к неудачам, хорошо продуманный эксперимент по секвенированию транспозонной вставки может стать мощным инструментом для идентификации многих условно важных генов у бактерий в рамках одного эксперимента. Например, дюжина генов Burkholderia seminalis, важных для подавления некроза листьев орхидеи, была идентифицирована путем объединения транспозонного мутагенеза и геномики34. Помимо Burkholderia,несколько генов адгезии и подвижности и транспортеров были идентифицированы как важные факторы колонизации у symbionts Snodgrassella alvi apis mellifera (Honeybee)22и у симпозиумов Vibrio fischerii Euprymna scolopes (гавайский кальмар бобтейла)23 с использованием транспозонно-инстиционного мутагенеза.
В качестве альтернативного подхода транспозонный мутагенез может сопровождаться скринингом отдельных мутантов с использованием селективных сред вместо секвенирования. Фенотипический скрининг или биоанализы для выявления недостатков, таких как подвижность, производство биологически активных вторичных метаболитов или специфические ауксотрофии, возможны. Например, скрининг библиотеки транспозонов мутантов Burkholderia insecticola (переназначенных в род Caballeronia35)был ключевым в выявлении того, что симбионты используют гены подвижности для колонизации Riptortus pedestris,их насекомого-хозяина36. Кроме того, с использованием транспозонного мутагенеза и фенотипического скрининга биосинтетический кластер генов для биоактивного вторичного метаболита кариоинненцина был идентифицирован в Burkholderia caryophylli37. Ауксотрофный мутант Burkholderia pseudomallei был идентифицирован после транспозонного мутагенеза и скрининга и является возможным ослабленным кандидатом на вакцину против мелиоидоза, опасного заболевания у людей и животных38. Таким образом, транспозонный мутагенез и секвенирование являются ценным подходом в изучении молекулярных признаков бактерий, которые важны для взаимодействия с их соответствующими хозяевами в патогенных или мутуалистических ассоциациях.
Раскрытие информации
Авторы заявляют, что у них нет конфликта интересов, относящегося к исследованию.
Благодарности
Мы благодарны Junbeom Lee за предоставление штамма E. coli WM3064 + pRL27 для сопряжения и руководства в процедуре, Катрин Хюффмайер за помощь в устранении неполадок во время генерации библиотеки мутантов и профессору Андре Родригесу за поддержку сбора насекомых и получения разрешений. Мы также благодарим Ребекку Янке и Дагмар Клебш за поддержку в сборе и выращивании насекомых. Мы выражаем признательность бразильским властям за предоставление следующих разрешений на доступ, сбор и экспорт образцов насекомых: разрешение SISBIO No 45742-1, 45742-7 и 45742-10, процесс CNPq No 01300.004320/2014-21 и 01300.0013848/2017-33, IBAMA Nr. 14BR016151DF и 20BR035212/DF). Это исследование было поддержано финансированием исследовательских грантов Немецкого научного фонда (DFG) FL1051/1-1 и KA2846/6-1.
Материалы
| Name | Company | Catalog Number | Comments |
| 2,6- Diaminopimelic Acid | Alfa Aesar | B22391 | For E.coli WM3064+ pRL27 |
| Agar - Agar | Roth | 5210 | |
| Agarose | Biozym | 840004 | |
| AMPure beads XP (magentic beads + polyethylene glycol + salts) | Beckman Coulter | A63880 | Size selection in step 6.6 |
| Bleach (NaOCl) 12% | Roth | 9062 | |
| Bowtie2 v.2.4.2 | Bioinfromatic tool for read mapping. Reference 10 in main manuscript. | ||
| Buffer-S | Peqlab | PEQL01-1020 | For PCRs |
| Cell scraper | Sarstedt | 83.1830 | |
| Cutadapt v.2.10 | Bioinformatic tool for removing specific adapter sequences from the reads. Reference 8 in main manuscript. | ||
| DESeq2 | RStudio package for assessing differential mutant abundance. Usually used for RNAseq analysis. Reference 12 in main manuscript. | ||
| DNA ladder 100 bp | Roth | T834.1 | |
| dNTPs | Life Technology | R0182 | PCR for confirming success of conjugation |
| EDTA, Di-Sodium salt | Roth | 8043 | |
| Epicentre MasterPure Complete DNA and RNA Purification Kit | Lucigen | MC85200 | |
| Ethidium bromide | Roth | 2218.1 | |
| FastQC v.0.11.8 | Bioinformatic tool for assessing the quality of sequencing data. Reference 7 in main manuscript. | ||
| FeatureCounts v.2.0.1 | Bioinformatic tool to obtain read counts per genomic feature. Reference 11 in main manuscript. | ||
| Glycerol | Roth | 7530 | |
| K2HPO4 | Roth | P749 | |
| Kanamycin sulfate | Serva | 26899 | |
| KCl | Merck | 4936 | |
| KH2PO4 | Roth | 3904 | |
| MgSO4.7H2O | Roth | PO27 | |
| Na2HPO4 | Roth | P030 | |
| NaCl | Merck | 6404 | |
| NEBNext Multiplex Oligos for Illumina (Index primers set 1) | New England Biolabs | E7335S | |
| NEBNext Ultra II DNA library prep kit for Illumina | New England Biolabs | E7645S | |
| Peptone (soybean) | Roth | 2365 | For Burkholderia gladioli Lv-StA KB-medium |
| peqGOLD 'Hot' Taq- DNA Polymerase | VWR | PEQL01-1020 | PCR for confirming success of conjugation |
| Petri plates - 145 x 20 mm | Roth | XH90.1 | For selecting transconjugants |
| Petri plates - 90 x 16 mm | Roth | N221.2 | |
| Qiaxcel (StarSEQ GmbH, Germany) | Quality check after DNA library preparation | ||
| Streptavidin beads | Roth | HP57.1 | |
| Taq DNA polymerase | VWR | 01-1020 | |
| Trimmomatic v.0.36 | Bioinformatic tool for trimming low quality reads and also adapter sequences. Reference 9 in main manuscript. | ||
| Tris -HCl | Roth | 9090.1 | |
| Tryptone | Roth | 2366 | For Escherichia coli WM3064+pRL27 LB medium |
| Ultrasonicator | Bandelin | GM 70 HD | For shearing |
| USER enzyme (uracil DNA glycosylase + DNA glycosylase- lyase Endonuclease VIII) | New England Biolabs | E7645S | Ligation step 6.5.2 |
| Yeast extract | Roth | 2363 |
Ссылки
- Cain, A. K., et al. A decade of advances in transposon-insertion sequencing. Nature Reviews Genetics. 21 (9), 526-540 (2020).
- Chao, M. C., Abel, S., Davis, B. M., Waldor, M. K. The design and analysis of transposon insertion sequencing experiments. Nature Reviews Microbiology. 14 (2), 119-128 (2016).
- Barquist, L., Boinett, C. J., Cain, A. K. Approaches to querying bacterial genomes with transposon-insertion sequencing. RNA Biology. 10 (7), 1161-1169 (2013).
- Flórez, L. V., et al. Antibiotic-producing symbionts dynamically transition between plant pathogenicity and insect-defensive mutualism. Nature Communications. 8 (1), 15172 (2017).
- Flórez, L. V., Kaltenpoth, M. Symbiont dynamics and strain diversity in the defensive mutualism between Lagria beetles and Burkholderia. Environmental Microbiology. 19 (9), 3674-3688 (2017).
- Flórez, L. V., et al. An antifungal polyketide associated with horizontally acquired genes supports symbiont-mediated defense in Lagria villosa beetles. Nature Communications. 9 (1), 2478 (2018).
- . FastQC A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2012)
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Liao, Y., Smyth, G. K., Shi, W. FeatureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2014).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Gaytán, M. O., Martínez-Santos, V. I., Soto, E., González-Pedrajo, B. Type three secretion system in attaching and effacing pathogens. Frontiers in Cellular and Infection Microbiology. 6, 129 (2016).
- Hachani, A., Wood, T. E., Filloux, A. Type VI secretion and anti-host effectors. Current Opinion in Microbiology. 29, 81-93 (2016).
- Deep, A., Chaudhary, U., Gupta, V. Quorum sensing and bacterial pathogenicity: From molecules to disease. Journal of Laboratory Physicians. 3 (1), 4-11 (2011).
- Silva, A. J., Benitez, J. A. Vibrio cholerae biofilms and cholera pathogenesis. PLOS Neglected Tropical Diseases. 10 (2), 0004330 (2016).
- Navarro-Garcia, F., Ruiz-Perez, F., Cataldi, &. #. 1. 9. 3. ;., Larzábal, M. Type VI secretion system in pathogenic Escherichia coli: structure, role in virulence, and acquisition. Frontiers in Microbiology. 10, 1965 (2019).
- Ribet, D., Cossart, P. How bacterial pathogens colonize their hosts and invade deeper tissues. Microbes and Infection. 17 (3), 173-183 (2015).
- Schwarz, S., et al. Burkholderia Type VI secretion systems have distinct roles in eukaryotic and bacterial cell interactions. PLoS Pathogens. 6 (8), 1001068 (2010).
- Jones, C., et al. Kill and cure: genomic phylogeny and bioactivity of Burkholderia gladioli bacteria capable of pathogenic and beneficial lifestyles. Microbial Genomics. 7 (1), 000515 (2021).
- Takeshita, K., Kikuchi, Y. Riptortuspedestris and Burkholderia symbiont: an ideal model system for insect-microbe symbiotic associations. Research in Microbiology. 168 (3), 175-187 (2017).
- Powell, J. E., et al. Genome-wide screen identifies host colonization determinants in a bacterial gut symbiont. Proceedings of the National Academy of Sciences of the United States of America. 113 (48), 13887-13892 (2016).
- Brooks, J. F., et al. Global discovery of colonization determinants in the squid symbiont Vibrio fischeri. Proceedings of the National Academy of Sciences of the United States of America. 111 (48), 17284-17289 (2014).
- Somprasong, N., McMillan, I., Karkhoff-Schweizer, R. R., Mongkolsuk, S., Schweizer, H. P. Methods for genetic manipulation of Burkholderia gladioli pathovar cocovenenans. BMC Research Notes. 3 (308), (2010).
- Garcia, E. C. Burkholderia thailandensis: Genetic manipulation. Current Protocols in Microbiology. 45, 1-15 (2017).
- Headd, B., Bradford, S. A. The conjugation window in an Escherichia coli K-12 strain with an IncFII plasmid. Applied and Environmental Microbiology. 86 (17), 00948 (2020).
- Van Opijnen, T., Camilli, A. Transposon insertion sequencing: A new tool for systems-level analysis of microorganisms. Nature Reviews Microbiology. 11 (7), 435-442 (2013).
- Gallagher, L. A., Ramage, E., Patrapuvich, R., Weiss, E., Brittnacher, M., Manoil, C. Sequence-defined transposon mutant library of Burkholderia thailandensis. mBio. 4 (6), 00604-00613 (2013).
- Wong, Y. -. C., et al. Candidate essential genes in Burkholderia cenocepacia J2315 identified by genome-wide TraDIS. Frontiers in Microbiology. 7, 1288 (2016).
- Moule, M. G., et al. Genome-wide saturation mutagenesis of Burkholderia pseudomallei K96243 predicts essential genes and novel targets for antimicrobial development. mBio. 5 (1), 00926 (2014).
- Ding, Q., Tan, K. S. Himar1 transposon for efficient random mutagenesis in Aggregatibacter actinomycetemcomitans. Frontiers in Microbiology. 8, 1842 (2017).
- Green, B., Bouchier, C., Fairhead, C., Craig, N. L., Cormack, B. P. Insertion site preference of Mu, Tn5, and Tn7 transposons. Mobile DNA. 3, 3 (2012).
- Lodge, J. K., Weston-Hafer, K., Berg, D. E. Transposon Tn5 target specificity: Preference for insertion at G/C pairs. Genetics. 120 (3), 645-650 (1988).
- Ará Ujo, W. L., et al. Genome sequencing and transposon mutagenesis of Burkholderia seminalis TC3.4.2R3 identify genes contributing to suppression of orchid necrosis caused by B. gladioli. Molecular Plant-microbe Interactions: MPMI. 29 (6), 435-446 (2016).
- Dobritsa, A. P., Samadpour, M. Reclassification of Burkholderiainsecticola as Caballeroniainsecticola comb. nov. and reliability of conserved signature indels as molecular synapomorphies. International Journal of Systematic and Evolutionary Microbiology. 69 (7), 2057-2063 (2019).
- Ohbayashi, T., et al. Insect's intestinal organ for symbiont sorting. Proceedings of the National Academy of Sciences of the United States of America. 112 (37), 5179-5188 (2015).
- Ross, C., Scherlach, K., Kloss, F., Hertweck, C. The molecular basis of conjugated polyyne biosynthesis in phytopathogenic bacteria. Angewandte Chemie International Edition. 53 (30), 7794-7798 (2014).
- Atkins, T., et al. A mutant of Burkholderia pseudomallei, auxotrophic in the branched chain amino acid biosynthetic pathway, is attenuated and protective in a murine model of melioidosis. Infection and Immunity. 70 (9), 5290-5294 (2002).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены