Method Article
Sequenciamento transposon-inserção como uma ferramenta para elucidar fatores de colonização bacteriana em um Birkholderia gladioli Simbionte de Besouros Lagria villosa
Neste Artigo
Resumo
Este é um método adaptado para identificar fatores de colonização de insetos candidatos em um simbionte benéfico da Burkholderia. O hospedeiro de besouro é infectado por uma biblioteca mutante aleatória gerada via mutagênese transposon, e a complexidade da biblioteca após a colonização é comparada a um controle cultivado in vitro.
Resumo
Inferir a função dos genes manipulando sua atividade é uma ferramenta essencial para entender os fundamentos genéticos da maioria dos processos biológicos. Os avanços na microbiologia molecular têm visto o surgimento de diversas técnicas de mutagênese para a manipulação de genes. Entre eles, o sequenciamento transposon-inserção (Tn-seq) é uma ferramenta valiosa para avaliar simultaneamente a funcionalidade de muitos genes candidatos de forma não alvo. A técnica tem sido fundamental para identificar mecanismos moleculares para a colonização de hospedeiros eucarióticos em vários micróbios patogênicos e alguns simbiontes benéficos.
Aqui, Tn-seq é estabelecido como um método para identificar fatores de colonização em um simbionte mutualista birmanês gladioli do besouro Lagria villosa. Por conjugação, a inserção mediada por Tn5 de um de resistência a antibióticos é realizada em locais genômicos aleatórios em B. gladioli. Para identificar o efeito das interrupções genéticas na capacidade das bactérias de colonizar o hospedeiro do besouro, a biblioteca gerada B. gladioli transposon-mutante é inoculada nos ovos de besouro, enquanto um controle é cultivado in vitro em um meio de cultura líquida. Depois de permitir tempo suficiente para colonização, o DNA é extraído das bibliotecas in vivo e in vitro cultivadas. Seguindo um protocolo de preparação da biblioteca de DNA, as amostras de DNA são preparadas para sequenciamento transposon-inserção. Fragmentos de DNA que contêm a borda de inserção transposon e o DNA bacteriano de flanqueamento são selecionados, e os locais de mutação são determinados por sequenciamento longe da borda de inserção transposon. Finalmente, analisando e comparando as frequências de cada mutante entre as bibliotecas in vivo e in vitro, pode-se prever a importância de genes simbiontes específicos durante a colonização do besouro.
Introdução
Burkholderia gladioli pode se envolver em uma associação simbiótica com besouros Lagria villosa, desempenhando um papel importante na defesa contra antagonistas microbianos do hospedeiro4,5,6. Besouros fêmeas abrigam várias cepas de B. gladioli em glândulas especializadas acessório ao sistema reprodutivo. Após a colocação de ovos, as fêmeas mancham células B. gladioli na superfície dos óvulos onde compostos antimicrobianos produzidos por B. gladioli inibem infecções por fungos entomopatômicos4,6. Durante o desenvolvimento embrionário tardio ou cedo após a escotilha das larvas, as bactérias colonizam invaginações cuticulares na superfície dorsal das larvas. Apesar dessa localização especializada e da rota de transmissão vertical dos symbionts, l. villosa pode presumivelmente também adquirir B. gladioli horizontalmente a partir do ambiente4. Além disso, pelo menos três cepas de B. gladioli foram encontradas em associação com L. villosa4,6. Entre eles, b. gladioli Lv-StA é o único que é favorável ao cultivo in vitro.
B. gladioli Lv-StA tem um tamanho genoma de 8,56 Mb6 e contém 7.468 genes. Qual desses genes são importantes para as bactérias B. gladioli colonizar o hospedeiro do besouro? Para responder a essa pergunta, utilizou-se o sequenciamento transposon-inserção (Tn-seq), método exploratório para identificar genes microbianos condicionalmente essenciais1,2,3. Uma biblioteca mutante de B. gladioli Lv-StA foi criada usando um transposon Tn5. Através da conjugação das células doadoras de Escherichia coli para B. gladioli Lv-StA, foi transferido um plasmid pRL27 que carrega o transposon Tn5 e um de resistência a antibióticos ladeado por repetições invertidas (Figura 1). Assim, foi gerado um conjunto de mutantes que carregam individualmente interrupções de 3.736 genes simbiontes(Figura 2).
A piscina mutante foi infectada em ovos de besouro para identificar os fatores de colonização e, como controle, também foi cultivada in vitro no meio B do Rei (KB). Após permitir tempo suficiente para colonização, larvas eclodidas foram coletadas e agrupadas para extração de DNA. Fragmentos de DNA contendo a inserção transposon e a região genômica flanqueada de B. gladioli Lv-StA foram selecionados usando um protocolo de preparação de biblioteca de DNA modificado para sequenciamento. A leitura do processamento de qualidade seguida de análise com o DESeq2 foi realizada para identificar genes específicos cruciais para b. gladioli Lv-StA colonizar larvas L. villosa quando transmitidas através da superfície do ovo.
Protocolo
1. Preparação de mídia e tampão
- Prepare as placas de mídia e ágar KB e LB, conforme indicado na Tabela 1, e autoclave a 121 °C, 15 psi, 20 min.
- Adicione 50 μg/mL de kanamycina esterilizada por filtro e 300 μM de ácido 2,6 diaminopimelic (DAP) ao meio LB autoclavado antes de culminar E.coli WM3064 + pRL27.
- Adicione 50 kanammicina esterilizada por filtro de μg/mL ao ágar KB autoclavado para despejar placas necessárias para selecionar transconjugantes B. gladioli Lv-StA bem sucedidos.
- Prepare 1x soro fisiológico tamponado de fosfato (PBS) misturando os seguintes componentes: NaCl 8 g/L, KCl 0.201 g/L, Na2HPO4 1,42 g/L e KH2PO4 0,272 g/L. Dissolva os sais em água destilada e autoclave a mistura a 121 °C, 15 psi, 20 min antes de usar. Armazene em temperatura ambiente.
- Prepare um tampão de ligação e lavagem de 2x dissolvendo os seguintes componentes: 10 mM Tris-HCl (pH 7.5), 1 mM de ácido tetraacético de etilenodiamina (EDTA) e 2 M NaCl em água destilada. Esterilize a mistura antes de usar. Armazene em temperatura ambiente.
- Prepare 1x Low-TE dissolvendo 10 mM Tris-HCl (pH 8.0) e 0,1 mM EDTA em água duplamente destilada. Esterilizar por autoclavagem a 121 °C, 15 psi, 20 min. Armazene em temperatura ambiente.
2. Conjugação para gerar a biblioteca mutante transposon
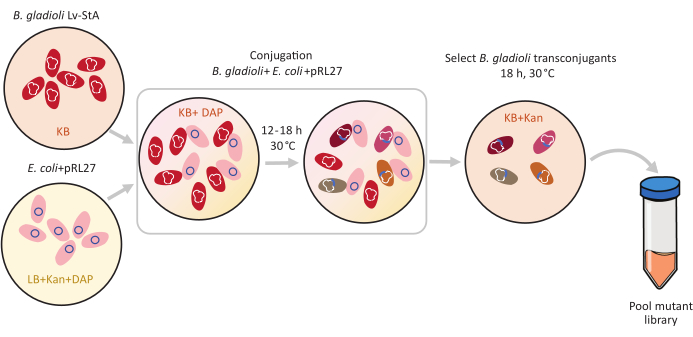
Figura 1: Etapas do protocolo de conjugação. O receptor de conjugação Burkholderia gladioli Lv-StA (vermelho) e o doador Escherichia coli contendo o plasmídeo pRL27 (rosa) são cultivados em ágar KB e LB, respectivamente, suplementados com kanamicina e DAP. Após a transferência conjugal do plasmídeo por 12-18 h a 30 °C, as células transconjugul. gladioli são selecionadas em KB contendo kanammicina e agrupadas. Abreviaturas: DAP = 2,6-ácido diaminopimelic; Kan = kanamicina. Clique aqui para ver uma versão maior desta figura.
- Sob um capô estéril, inocular uma cultura de doadores frescos de Escherichia coli WM3064 + pRL27 em 10 mL de lb médio suplementado com kanamicina e DAP. Células receptoras de Burkholderia gladioli Lv-StA em 5 mL de meio KB. Incubar as culturas a 30 °C durante a noite em um shaker a 250 rpm.
- Após o crescimento da noite para o dia, centrífuga 4 mL de cada uma das culturas a 9.600 × g por 6 min para pelotar as células. Descarte o supernatante.
- Sob um capô estéril, lave as culturas celulares pelleted em KB médio contendo DAP e finalmente resuspenque as culturas separadamente em 4 mL de meio KB + DAP.
- Em um tubo fresco de 15 mL, misture 250 μL das células doadoras E. coli lavadas com 1 mL das células receptoras B. gladioli Lv-StA lavadas.
- Local 10 μL desta mistura de célula de conjugação em placas de ágar KB contendo DAP. Deixe a placa descansar sem ser perturbada no capô estéril à temperatura ambiente por 1h. Em seguida, incubar as placas com as manchas de conjugação a 30 °C para 12-18 h.
NOTA: O período de conjugação pode ser ajustado de acordo com a espécie-alvo. No entanto, um longo período de conjugação aumenta o risco de dupla inserções ou integração plasmida no genoma. Para bactérias de crescimento lento, permita períodos de conjugação mais longos. - Após a incubação, adicione 2-4 mL de 1x PBS nas placas sob um capô estéril e use um raspador de células para liberar os pontos de conjugação bacteriana cultivados do ágar. Pipeta a mistura de células conjugadas em tubos de microfuça de 2 mL.
- Pelota as células por centrifugação a 9.600 × g por 2 min. Descarte o supernatante e lave a pelota duas vezes em 1 mL de 1x PBS, pipetando para cima e para baixo. Resuspense a última pelota em 1200 μL de 1x PBS. Faça diluições antes de emplacamento se o número de células na mistura estiver acima de 1 × 104.
- Misture bem e espalhe 200 μL da mistura celular em grandes placas de ágar KB (6 ou mais, se necessário) suplementada com kanamicina e incubada a 30 °C durante a noite.
NOTA: Colônias mutantes alvo aparecem dentro de 30 h nas placas de ágar seletivas. Devido ao marcador de resistência a antibióticos, apenas colônias mutantes aparecem na placa de ágar seletivo. Portanto, espera-se que todas as colônias sejam transconjulgantes bem sucedidas. - Conte o número total de colônias transconjugut em três placas e extrapolule para calcular o número aproximado de mutantes obtidos em todas as placas. Para aumentar as chances de obter uma biblioteca representativa, certifique-se de que o número total de colônias seja várias vezes maior do que o número total de genes no genoma. Para confirmar o sucesso da conjugação, realize um PCR visando o de inserção usando 10-20 colônias de amostras, conforme descrito na seção 3.
NOTA: O objetivo é garantir que o número de colônias seja pelo menos 10 vezes maior do que o número de genes em todo o genoma, neste caso, > 75.000 mutantes. No entanto, é geralmente desafiador estimar com precisão o número de colônias que corresponderiam a uma biblioteca totalmente representativa. O número de genes únicos mutados não é evidente neste momento, dado que as interrupções em genes essenciais não são capturadas, muitas vezes existem vários locais de mutação diferentes para o mesmo gene, e mutações geradas com transposons Tn5 não são totalmente aleatórias. - Sob um capô estéril, raspe colônias das placas adicionando 1-2 mL de 1x PBS no ágar. Misture a mistura celular raspada das placas em tubos de 50 mL. Vortex a biblioteca para misturar completamente e, em seguida, dividir 4 mL da biblioteca mutante agrupada em vários criotubos. Adicione 1 mL de 70% de glicerol aos tubos e armazene a -80 °C.
3. Eletroforese de PCR e gel para confirmar inserções bem sucedidas em B. gladioli Lv-StA
- Para confirmar a presença da inserção, escolha colônias mutantes individuais das placas de seleção na etapa 2.9 e realize um PCR visando o de inserção usando os primers listados na Tabela 2. Prepare o mix mestre pcr de acordo com a Tabela 3 e coloque as condições no cicloviário térmico conforme descrito na Tabela 4.
- Execute os produtos PCR em um gel de 1,6% de agarose por eletroforese (250 V, 40 min) para verificar se os fragmentos de DNA amplificados são do comprimento esperado de 1580 bp.
4. Infecção na piscina mutante em ovos de besouro
- Etapas de lavagem da biblioteca
- Descongele uma alíquota da biblioteca mutante preparada no gelo. Centrifugar a 2.683 × g por 10 min e remover o supernaspe. Sob um capô estéril, lave as células com 4 mL de 1x PBS para remover qualquer meio restante das células. Resuspense as células em 4 mL de 1x PBS.
- Conte o número de células em uma alíquota da biblioteca usando uma câmara de contagem de células. Dilui uma parte da biblioteca para 2 × 106 células/μL em 1x PBS.
- Vortex a biblioteca aliquot completamente para misturar toda a biblioteca de forma homogênea antes de tomar o volume necessário.
- Esterilização de embreagem de ovos e infecção in vivo
- Selecione uma embreagem de ovo L. villosa. Conte o número de ovos e continue se a embreagem contiver mais de 100 ovos.
- Esterilize toda a embreagem de ovo.
- Adicione 200 μL de 70% de etanol e lave suavemente os ovos por 5 minutos. Retire o etanol e lave os ovos duas vezes com água autoclavada.
- Adicione 200 μL de alvejante de 12% (NaOCl) e lave suavemente os ovos por 30 s. Retire o alvejante imediatamente e lave os ovos novamente três vezes com 200 μL de água autoclavada.
- Infecte 2 × 106 células/μL da biblioteca mutante lavada na embreagem de ovo esterilizada (2,5 μL por ovo).
- Dois dias após a escotilha de larvas de besouro infectado, colete 100 2ª larvas instar por tubo de microfuge de 1,5 mL e armazene a -80 °C.
-
Controle in vitro da biblioteca mutante
- Sob um capô estéril, inoculado 250 μL de 2 × 106 células/μL da biblioteca mutante lavada em 10 mL de KB médio contendo kanamicina.
- Incubar a cultura mutante in vitro a 30 °C por 20 h.
NOTA: Calcule a duração da incubação para corresponder ao número aproximado de gerações de WT B. gladioli Lv-StA in vivo durante a colonização. - Após a incubação de 20 h, adicione um volume igual de 70% de glicerol à cultura mutante in vitro e armazene-o a -80 °C.
5. Besouros infectados e extração de DNA de biblioteca mutante in vitro
NOTA: As extrações de DNA foram realizadas utilizando um kit de purificação de DNA e RNA de acordo com o protocolo do fabricante brevemente descrito abaixo.
- Homogeneize as larvas agrupadas (máximo de 4 mg por tubo microfuge) adicionando 1-2 mL de nitrogênio líquido e esmagando com um pilão.
- Descongele as culturas mutantes in vitro cultivadas de estoques de glicerol no gelo. Pelota as células por centrifugação a 9.600 × g por 10 minutos antes da lise celular.
- Adicione 300 μL de solução de tecido e lise celular às amostras in vitro e in vivo. Adicione 5 μL de 10 mg/mL Proteinase K, incubar a mistura a 60 °C por 15 min e, em seguida, coloque no gelo por 3-5 min.
- Adicione 150 μL de reagente de precipitação proteica aos lises e vórtices completamente. Pelota os detritos proteicos por centrifugação a 9.600 × g por 10 min.
- Transfira o supernatante para um tubo de microfuça de 1,5 mL. Adicione 500 μL de isopropanol ao supernasciente e inverta suavemente os tubos pelo menos 40 vezes antes de incubar a -20 °C por 1h ou durante a noite.
- Pelota o DNA precipitado por centrifugação a 9.600 × g por 10 min. Descarte o supernasce e adicione 70% de etanol gelado à pelota de DNA.
- Centrifugar a ≥ 10.000 × g por 5 min. Descarte o supernatante e deixe as amostras secas ao ar por pelo menos 1h.
- Resuspend o DNA das amostras in vitro e in vivo em 100 μL de buffer Low-TE.
- Armazene as amostras a -20 °C.
6. Preparação da biblioteca de sequenciamento
NOTA: O protocolo e os reagentes para a preparação da biblioteca de DNA são adaptados e modificados a partir das instruções fornecidas pelo fabricante do kit de preparação da biblioteca de DNA.
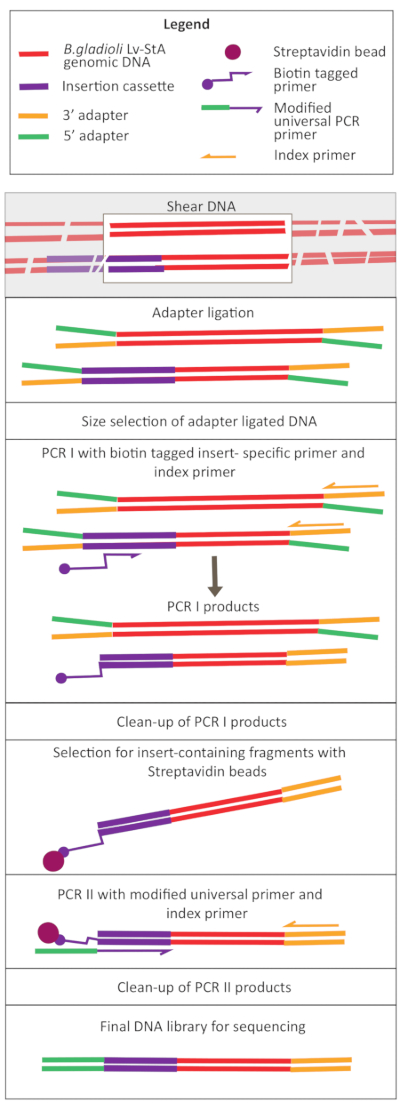
Figura 2: Esquema das etapas de preparação da biblioteca de DNA. Após a rolagem e a ligadura do adaptador, o protocolo modificado inclui uma etapa de seleção de contas streptavidin para enriquecer fragmentos de DNA contendo o de inserção. Clique aqui para ver uma versão maior desta figura.
- Diluir as amostras para concentração de 20 ng/μL e volume de 100 μL e mantê-las no gelo.
- Shear in vivo e in vitro amostra DNA usando um ultrassônico. Coloque o ultrassônico em 70% de potência. Vórtice as amostras brevemente e tesoura para 1 min 30 s.
NOTA: As configurações do ultrassonicador diferem entre os instrumentos. Neste caso, o tamanho do fragmento foi de 200-400 bp, o que é apropriado para esta abordagem de sequenciamento de 150 bp, emparelhado-end (ver passo. 9.1). Os parâmetros de shearing podem ser ajustados de acordo com as exigências do experimentador. - Verifique se o DNA foi coletado na faixa de tamanho desejada (neste caso, 200-400 bp). Carregue 5 μL do DNA descompanhado e rosqueado depois de misturar com corante de carregamento de gel em uma proporção de 1:1 em um gel de 1,6% de agarose executado a 250 V por 40 min(Figura 3A, B).
- Preparação de extremidades de fragmento necessárias para ligadura adaptador
- A 50 μL do DNA desarquiado, adicione os reagentes de preparação final dados no kit de preparação da biblioteca: 3 μL da mistura de enzimas e 7 μL de tampão de reação e misture bem por pipetação. Coloque um cicloviário térmico com uma tampa aquecida a ≥ 75 °C e incubar as amostras por 30 min a 20 °C e 30 min a 65 °C. Mantenha a 4 °C.
- Ligadura adaptador
- Para a ligadura do adaptador, adicione os seguintes reagentes aos produtos da etapa final de preparação: 30 μL Ligation Master Mix, 1 μL Ligation Enhancer e adaptador diluído de 2,5 μL. Misture bem por pipetar e incubar a amostra por 15 min a 20 °C no cicloviário térmico com a tampa aquecida desligada.
- Após 15 min, adicione 3 μL da enzima (uracil DNA glicosylase + DNA glicosylase-lyase Endonuclease VIII) (ver a Tabela de Materiais). Misture bem por pipetar e incubar a amostra por 15 min a 37 °C em um cicloviário térmico com a tampa aquecida a ≥47 °C.
NOTA: O protocolo pode ser pausado nesta etapa, e as amostras podem ser armazenadas a -20 °C.
- Seleção de tamanho de fragmentos de DNA ligados por adaptador de 250 bp
- Vórtice a solução de contas magnéticas (ver a Tabela dos Materiais) e coloque-a em temperatura ambiente por 30 minutos antes do uso.
- Adicione 0,3x de contas a 96,5 μL da mistura de DNA ligada e misture bem a tubulação. Incubar a mistura de contas por 5 minutos.
NOTA: A presença de sais e polietilenoglicol na mistura de contas facilita a precipitação de fragmentos de DNA nas contas. Uma baixa proporção de contas para moléculas de DNA leva à ligação de apenas fragmentos maiores de DNA às contas. Neste caso, fragmentos de DNA acima de 250 bp de comprimento estão ligados às contas. - Coloque os tubos em um suporte magnético para puxar as contas e remover fragmentos de DNA de tamanho indesejado. Que as contas se contentem com 5 minutos e depois transfiram o supernatante claro para um novo tubo de microfuge (mantenha o supernatante).
- Adicione 0,15x de contas frescas ao supernasciente e misture bem a pipetar bem. Incubar a mistura de contas por 5 minutos e, em seguida, colocar os tubos em um suporte magnético para puxar para baixo as contas ligadas ao DNA alvo. Aguarde 5 minutos e depois descarte o supernaspeso (fique com as contas).
NOTA: Esta proporção de contas ao DNA leva à ligação de fragmentos do tamanho desejado de 250 bp. - Com as contas no suporte magnético, adicione 200 μL de 80% de etanol (recém-preparado) e espere por 30 s. Pipeta para fora e descartar a lavagem do etanol cuidadosamente sem perturbar as contas no suporte magnético. Repita este passo.
- Após a última lavagem, remova os traços de etanol das contas e, em seguida, seque as contas por 2 minutos até que pareçam brilhantes, mas não completamente secas. Não seque demais as contas.
- Remova os tubos do suporte magnético e adicione 17 μL de 10 mM Tris-HCl ou 0,1x TE (Low-TE). Misture por pipetar ~10 vezes e incubar a mistura à temperatura ambiente por 2 minutos.
- Coloque os tubos de volta no suporte magnético e espere por 5 minutos. Uma vez que as contas se estabeleceram, transfira o DNA supernatante para um novo tubo.
- PCR I para adicionar biotina tag a fragmentos de DNA contendo o de inserção
- Adicione uma etiqueta de primer biotinína aos fragmentos de DNA contendo o de inserção Tn5 usando o primer biotinína(Tabela 5)e uma cartilha de índice. Prepare o mix mestre pcr de acordo com a Tabela 6 e siga as condições de PCR para o cicloviário térmico listado na Tabela 7.
- Limpeza do PCR I sem seleção de tamanho
- Contas de vórtice 0,9x e coloque-as em temperatura ambiente por pelo menos 30 minutos antes da limpeza.
- Adicione 0,9x contas aos produtos PCR e misture bem.
- Coloque as contas em um suporte magnético para puxar as contas.
- Remova o supernanato claro e lave o DNA ligado a contas com 200 μL de etanol recém-preparado duas vezes.
- Retire o etanol após as etapas de lavagem e seque as contas até que pareçam brilhantes, mas não muito secas.
- Adicione 32 μL de 10 mM Tris-HCl ou 0,1X TE (Low-TE) e incuba as contas por 5 min. Coloque a mistura de volta no suporte magnético e transfira o supernatante para um tubo de microfuça fresco.
- Vinculando fragmentos de DNA biotinilados a contas de streptavidina
- Resuspend 32 μL de contas streptavidinas em 1x Tampão de ligação e lavagem. Lave as contas com o tampão três vezes enquanto estiver em um suporte magnético.
- Adicione 32 μL de 2x tampão de ligação e lavagem e resuspenja as contas. Para isso, adicione 32 μL dos produtos PCR 1 limpos. Misture bem e incubar à temperatura ambiente por 30 minutos.
- Coloque a mistura de DNA em um suporte magnético por 2 minutos. Pipeta fora do supernante como DNA marcado por biotina contendo a borda de inserção liga-se ao streptavidin nas contas.
- Lave as contas com 500 μL de 1x Tampão de ligação e lavagem e, em seguida, lave as contas com 200 μL de Low-TE. Resuspend as contas ligadas ao DNA em 17 μL de Low-TE.
- PCR II para adicionar adaptadores aos fragmentos que contêm a borda do de inserção
- Prepare um mix mestre, como mostrado na Tabela 8, utilizando os primers de índice e primers pcr universais modificados listados na Tabela 5. Adicione 15 μL das contas de streptavidina vinculadas ao DNA da etapa anterior à mistura PCR. Consulte a Tabela 7 para obter as condições do cicloviário térmico.
- Limpe os produtos PCR sem seleção de tamanho, conforme dado na etapa 6.8 deste protocolo. Elute os produtos finais de DNA em 30 μL de água de grau molecular.
- Armazene as amostras a -20 °C e use-as para sequenciar.
7. Sequenciamento e análise
- Sequenciar a biblioteca usando tecnologia de sequenciamento de alto rendimento. Ajuste a profundidade de sequenciamento dependendo do tamanho da biblioteca transposon, conforme observado abaixo. Avalie a qualidade da leitura com FastQC7. Selecione leituras contendo a borda de inserção Tn5 na extremidade de 5' da leitura e remova a sequência de borda de inserção usando Cutadapt8 e/ou Trimmomatic9.
NOTA: Aqui, uma abordagem de sequenciamento de extremidade emparelhada foi usada para atingir 150 bp por leitura e um total de 8 leituras de Mio. Para obter um conjunto de dados representativo, certifique-se de que o número total de leituras sequenciadas exceda o número máximo possível de mutantes na biblioteca, ou seja, o número total estimado de colônias a partir da etapa 2.9. Como referência, este protocolo visava 40 vezes o tamanho máximo possível da biblioteca. Outros estudos bem-sucedidos usando Tn-seq para um propósito semelhante sequenciaram um número total de leituras próximas de 25 vezes do número real de inserções únicas na biblioteca mutante correspondente22,23. - Considerando que as mutações nas extremidades dos genes não são funcionalmente disruptivas, corte 5% das duas extremidades das anotações genéticas do arquivo GFF genoma de referência. Mapear as leituras aparadas para o genoma de referência usando Bowtie210.
- Calcule o número de inserções a partir do número de posições únicas de 5' no arquivo BAM de alinhamento.
- Usando FeatureCounts11, obtenha o número de genes de sucesso para cada amostra de réplica.
- Usando o pacote DESeq212 no RStudio, calcule a diferença nas abundâncias mutantes entre diferentes condições.
Resultados
As bactérias associadas ao hospedeiro podem empregar vários fatores para estabelecer uma associação, incluindo aquelas que mediam a adesão, motilidade, quimiotaxis, respostas ao estresse ou transportadores específicos. Embora fatores importantes para as interações patógenos-hospedeiros tenham sido relatados para várias bactérias13,14,15,16,17,18, incluindo membros do gênero Burkholderia19,20, menos estudos exploraram os mecanismos moleculares usados por simbiontos benéficos para colonização21,22,23 . Utilizando o sequenciamento da inserção transposon, o objetivo foi identificar fatores moleculares que permitem que B. gladioli colonize besouros L. villosa.
A mutagênese mediada pela Transposon foi realizada usando o plasmídeo pRL27, que carrega um transposon Tn5 e um de resistência à kanamicina ladeado por locais repetidos. O plasmídeo foi introduzido nas células B. gladioli Lv-StA por conjugação com o doador plasmídeo E. coli WM3064 (como mostrado na Figura 1). Após a conjugação, a mistura de conjugação contendo b. gladioli receptor e células doadoras E. coli foram banhadas em placas de ágar seletiva contendo kanamicina. A ausência de DAP nas placas eliminou as células E. coli doador, e a presença de kanamicina selecionada para os transconjugantes B. gladioli Lv-StA bem sucedidos. A biblioteca mutante B. gladioli Lv-StA obtida na colheita das 100.000 colônias transconjugantes foi preparada para sequenciamento usando um kit de preparação de biblioteca de DNA modificado e primers personalizados. A Figura 2 destaca as etapas de preparação da biblioteca de DNA. Sequenciamento rendeu 4 leituras emparelhadas mio; 3.736 genes de 7.468 genes em B. gladioli Lv-StA foram interrompidos.
Para identificar mutantes defeituosos na colonização, a biblioteca mutante B. gladioli Lv-StA foi infectada nos ovos de besouro e cultivada in vitro no meio KB como controle. O tamanho do gargalo de colonização in vivo foi calculado antes do experimento. Um número conhecido de células B. gladioli Lv-StA foi infectado em ovos de besouro, e o número de células colonizadoras em larvas recém-eclodidas foi obtido por chapeamento de uma suspensão de cada larva e contando unidades formadoras de colônias por indivíduo. Esses cálculos foram feitos para garantir que o número de células colonizadoras seja suficiente para avaliar todos ou uma alta porcentagem dos mutantes na biblioteca por sua capacidade de colonizar o hospedeiro. Além disso, o tempo de crescimento entre condições in vitro e in vivo foi normalizado com base no número de gerações bacterianas para tornar essas amostras comparáveis.
Após a eclosão dos ovos, 1.296 larvas foram coletadas em 13 piscinas. As culturas mutantes in vitro correspondentes foram cultivadas e armazenadas como estoques de glicerol. O DNA das bibliotecas mutantes in vivo e in vitro cultivadas foi extraído e fragmentado em um ultrassônico. A Figura 3 mostra a distribuição de tamanho do DNA desarquiado, onde a maioria dos fragmentos se estende entre 100 e 400 bp, como esperado. Esta etapa foi seguida pelo protocolo modificado de preparação da biblioteca de DNA para sequenciamento. Em cada etapa do protocolo, a concentração do DNA restante foi verificada para garantir que as etapas fossem executadas corretamente e para rastrear perdas de DNA. Uma verificação de qualidade (veja a Tabela de Materiais) antes do sequenciamento revelou que as bibliotecas de DNA continham fragmentos de DNA inesperadamente grandes (>800 bp), e isso foi mais pronunciado nas bibliotecas in vivo. Dada a dificuldade em otimizar o agrupamento de fragmentos nas pistas de sequenciamento, foi necessário aumentar a profundidade de sequenciamento para 10 leituras emparelhadas de Mio nas bibliotecas in vivo para atingir o número desejado de leituras. A análise dos resultados do sequenciamento revelou que uma média de 4 Mio lê nas bibliotecas in vivo e 3,1 Mio lê nas bibliotecas in vitro continha a borda Transposon no final de 5' da Tabela9, o que foi satisfatório para este experimento. A distribuição das 24.224 inserções únicas em todo o genoma B. gladioli na biblioteca original é mostrada na Figura 4. Uma análise realizada por meio do DESeq2 revelou que as abundâncias de 271 mutantes eram significativamente diferentes entre as condições in vivo e in vitro.
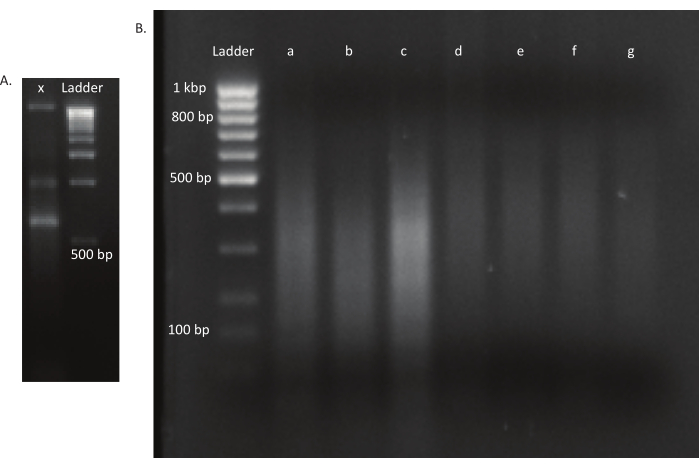
Figura 3: Géis agarose de uma biblioteca de mutante e DNA. (A) Gel de agarose com DNA descomproxido de um mutante na pista x e uma escada de 1 kbp para escala. (B) Gel com biblioteca de DNA desarquiada. Os tamanhos da banda da escada na primeira pista são indicados no lado esquerdo. As três primeiras pistas a, b e c contêm fragmentos de DNA desarmados das bibliotecas in vivo. As pistas d, e, f e g contêm fragmentos de DNA das bibliotecas in vitro. Clique aqui para ver uma versão maior desta figura.
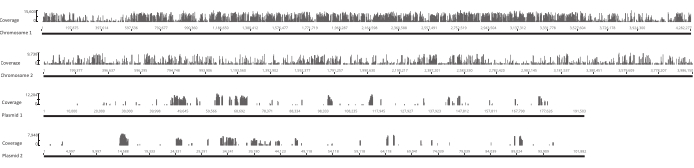
Figura 4: Localização de locais de inserção únicos na biblioteca original através dos quatro replicons no genoma Burkholderia gladioli Lv-StA. Cada barra ao longo do eixo x está localizada em um local de inserção. A altura de uma barra ao longo do eixo y corresponde ao número de leituras associadas a esse local. Observe que os dois cromossomos e dois plasmídeos são mostrados em comprimento total e, portanto, têm escalas diferentes no eixo x. Clique aqui para ver uma versão maior desta figura.
| M de B do rei/ ágar | |
| Peptone (soja) | 20 g/L |
| K2HPO4 | 1,5 g/L |
| MgSO4.7H2O | 1,5 g/L |
| Ágar | 15 g/L |
| Dissolvido em água destilada | |
| Meio/ágar LB | |
| Triptona | 10 g/L |
| Extrato de levedura | 5 g/L |
| NaCl | 10 g/L |
| Dissolvido em água destilada | |
Tabela 1: Componentes de mídia.
| Não. | Primers | Seqüenciar | PcR annealing temp. (°C) | |
| 1 | tpnRL17-1RC | 5'-CGTTACATCTGGCTTGTT-3' | 58.2 | |
| 2 | tpnRL13-2RC | 5'-TCGTGAAGAAGGTGTTGCTG-3' | ||
Tabela 2: Primers para confirmar o sucesso da conjugação.
| Componente | Volume (μL) |
| Água purificada pelo HPLC | 4.92 |
| 10x Buffer S (alta especificidade) | 1 |
| MgCl2 (25 mM) | 0.2 |
| dNTPs (2 mM) | 1.2 |
| Primer 1 (10 pmol/μL) | 0.8 |
| Primer 2 (10 pmol/μL) | 0.8 |
| Taq (5 U/μL) | 0.08 |
| Mastermix total | 9 |
| Modelo | 1 |
Tabela 3: Mistura mestre pcr para confirmar o sucesso da conjugação. Abreviaturas: HPLC = cromatografia líquida de alto desempenho; dNTPs = triphosfato desoxiconucleosídeo.
| Passos | Temperatura °C | Hora | Ciclos |
| Denaturação Inicial | 95 | 3 min. | 1 |
| Desnaturação | 95 | 40 s | |
| Recozimento | 58.2 | 40 s | 30 a 35 |
| Extensão | 72 | 1-2 min | |
| Extensão Final | 72 | 4 min. | 1 |
| Segurar | 4 | ∞ | |
Tabela 4: Condições de PCR para confirmar o sucesso da conjugação.
| Primers | Seqüenciar | Tm °C | Usar | Fonte | |
| Primer biotinilado específico para transposon | 5'-Biotin-ACAGGAACACTTAACGGCTGACATG -3' | 63.5 | 6.7.1. PCR I | Costume | |
| Primer Universal PCR modificado | 5'- AATGATACGGCGACCACCGAGATC TACACTCTCTCTACACGACGCTC TTCCGATCTGAATTCATCGATGAT GGTTGAGATGTGT – 3' | 62 | 6.10.1. PCR II | Costume | |
| Primer de índice | Consulte o manual do fabricante | 6.7.1. PCR I & 6.10.1. PCR II | NEBNext Multiplex Oligos para Illumina (Primers de índice definidos 1) | ||
| Adaptador | Consulte o manual do fabricante | 6.5. Ligadura do adaptador | Kit de preparação da biblioteca de DNA NEBNext Ultra II para Illumina | ||
Tabela 5: Primers e adaptador para PCR I e II durante a preparação da biblioteca de DNA.
| Mistura PCR | (μL) |
| Fragmentos de DNA ligados por adaptador | 15 |
| NEBNext Ultra II Q5 master mix | 25 |
| Primer de índice (10 pmol/ μL) | 5 |
| Primer biotinilado específico transposon (10 pmol/ μL) | 5 |
| Volume total | 50 |
Tabela 6: Preparação da biblioteca de DNA-PCR I master mix.
| Passos | Temperatura | Hora | Ciclos |
| Denaturação Inicial | 98 °C | 30 s | 1 |
| Desnaturação | 98 °C | 10 s | 6 a 12 |
| Recozimento | 65 °C | 30 s | |
| Extensão | 72 °C | 30 s | |
| Extensão Final | 72 °C | 2 min. | 1 |
| Segurar | 16 °C | ∞ | |
Tabela 7: Preparação da biblioteca de DNA-Condições PCR I e II.
| Mistura PCR | (μL) |
| DNA selecionado por contas | 15 |
| NEBNext Ultra II Q5 master mix | 25 |
| Primer de índice | 5 |
| Primer universal pcr modificado | 5 |
| Volume total | 50 |
Tabela 8: Preparação da biblioteca de DNA-MIX MESTRE PCR II.
| Bibliotecas | Invivo-1 | Invivo-2 | Invivo-3 | Invitro-1 | Invitro-2 | Invitro-3 | Biblioteca original | |
| Não. de leituras (PE) | 56,57,710 | 39,19,051 | 30,65,849 | 35,73,494 | 28,83,440 | 36,61,956 | 46,09,410 | |
| Não. de leituras contendo Tn – borda no final de 5' de Read-1 | 54,15,880 | 37,31,169 | 29,36,247 | 33,00,499 | 27,35,705 | 33,50,402 | 41,53,270 | |
| Taxa de alinhamento global bowtie2 (%) (somente leitura-1) | 95.53% | 83.71% | 89.87% | 80.79% | 78.00% | 73.06% | 74.92% | |
| Número de inserções únicas | 8,539 | 4,134 | 7,183 | 18,930 | 18,421 | 20,438 | 24,224 | |
| Número de genes atingidos | 1575 | 993 | 1450 | 2793 | 2597 | 3037 | 3736 | |
Tabela 9: Resumo da saída de sequenciamento e frequência de inserção transposon por biblioteca. Abreviação: PE = ponta emparelhada.
Discussão
Uma biblioteca mutante B. gladioli transposon foi gerada para identificar importantes fatores de colonização hospedeira na interação simbiótica entre besouros L. villosa e bactérias B. gladioli. Os principais passos do protocolo foram conjugação, infecção por hospedeiro, preparação da biblioteca de DNA e sequenciamento.
Como muitas cepas de Burkholderia são favoráveis à modificação genética por conjugação24,25, o plasmídeo que carrega o transposon e de inserção de antibióticos foi conjugado com sucesso na cepa B. gladioli Lv-StA da E. coli. Tentativas anteriores de transformação por eletroporação renderam-se muito baixas a quase nenhum transformador B. gladioli. É aconselhável otimizar a técnica de transformação do organismo alvo para produzir eficientemente um grande número de transformadores.
Uma rodada de conjugação e 40 pontos de conjugação interromperam 3.736 genes em B. gladioli Lv-StA. Em retrospectiva, múltiplas rodadas de conjugação seriam necessárias para interromper a maioria dos 7.468 genes e obter uma biblioteca saturada. Notavelmente, o tempo de incubação durante a conjugação não foi permitido exceder 12-18 h, que é o fim da fase de crescimento exponencial de B. gladioli. Permitir a conjugação além da fase exponencial de crescimento das células bacterianas reduz as chances de sucesso da obtenção de transconjugantes26. Portanto, o período de conjugação deve ser ajustado de acordo com o crescimento das espécies bacterianas.
Para realizar com sucesso um experimento envolvendo a infecção de bibliotecas mutantes em um hospedeiro, é importante avaliar o tamanho do gargalo populacional bacteriano durante a colonização e a diversidade de mutantes na biblioteca antes da infecção1,2,27. Em preparação para o experimento, estimamos o número mínimo de besouros que devem ser infectados para ter uma grande chance de que cada mutante na biblioteca seja amostrado e autorizado a colonizar. Também foram calculados o tempo aproximado de geração bacteriana in vivo e o número de gerações durante a duração do experimento. A cultura in vitro foi então cultivada a um número comparável de gerações, ajustando o tempo de incubação. Para um experimento de infecção semelhante em outros hospedeiros não-modelos, a capacidade de manter uma cultura de laboratório e uma fonte constante dos organismos hospedeiros é desejável.
Após o crescimento da biblioteca mutante in vivo e in vitro e coleta de amostras, foi realizado um protocolo modificado de preparação da biblioteca de DNA para sequenciamento de inserção transposon. A modificação no protocolo envolveu a concepção de primers PCR personalizados e a adição de etapas pcr para selecionar para fragmentos de DNA contendo o de inserção. Como o protocolo foi personalizado, ciclos adicionais de PCR no protocolo aumentaram o risco de superamplificação e obtenção de fragmentos de adaptador-adaptador hibridizado nas bibliotecas finais. Assim, recomenda-se uma etapa final de limpeza (sem seleção de tamanho) após os dois PCRs, pois ajuda na remoção desses fragmentos. A distribuição de tamanho das bibliotecas de DNA ainda era mais ampla do que o esperado. No entanto, o aumento da profundidade de sequenciamento forneceu dados suficientes que foram filtrados durante a análise bioinformática, obtendo resultados satisfatórios.
Como a mutagênese mediada pela transposon gera milhares de inserções aleatórias em um único experimento, é possível gerar uma biblioteca saturada de mutantes que contém todos, exceto aqueles mutantes onde genes essenciais para o crescimento bacteriano foram interrompidos. Provavelmente não trabalhamos com uma biblioteca mutante saturada, dadas as estimativas de genes essenciais em outros estudos sobre Burkholderia sp. 28,29. Uma biblioteca não saturada, no entanto, ajuda a explorar vários genes candidatos para estudos adicionais usando mutagênese direcionada. Antes dos experimentos, também é importante lembrar que alguns transposons têm locais-alvo de inserção específicos que aumentam a abundância de mutantes em certos loci no genoma30. Os transposons mariner são conhecidos por atingir os locais AT para inserção31, e os transposons Tn5 têm um viés GC32,33. Incluir etapas durante a análise de bioinformática para reconhecer hotspots para inserções transposon ajudará na avaliação de qualquer viés de distribuição.
Embora propenso a contratempos, um experimento de sequenciamento de inserção transposon bem projetado pode ser uma ferramenta poderosa para identificar muitos genes condicionalmente importantes em bactérias dentro de um único experimento. Por exemplo, uma dúzia de genes em Burkholderia seinalis importantes para a supressão da necrose da folha de orquídeas foram identificados pela combinação de mutagênese transposon e genômica34. Além da Burkholderia,vários genes e transportadores de adesão e motilidade foram identificados como importantes fatores de colonização em Snodgrassella alvi symbionts de Apis mellifera (Honeybee)22, e nos symbionts vibrio fischerii de euprymna scolopes (lula de bobtail havaiano)23 usando a abordagem mutagense transposon-inserção.
Como uma abordagem alternativa, a mutagênese transposon pode ser seguida pela triagem de mutantes individuais usando mídia seletiva em vez de sequenciamento. A triagem fenotípica ou os bioensatórios para identificar deficiências, como motilidade, produção de metabólitos secundários bioativos ou auxotropicais específicos, são viáveis. Por exemplo, a triagem de um inseto burkholderia (redesignado para o gênero Caballeronia35) a biblioteca mutante transposon tem sido fundamental para identificar que os simmbiontos empregam genes de motilidade para colonizar pedestris Riptortus, seu hospedeiro de insetos36. Além disso, utilizando mutagênese transposon e triagem fenotípica, o aglomerado genético biossintético para a cariomicenária bioativa secundária metabolite foi identificado em Burkholderia caryophylli37. Um mutante auxotrófico de Burkholderia pseudomallei foi identificado após mutagênese transposon e triagem e é um possível candidato à vacina atenuada contra a melioidose, uma doença perigosa em humanos e animais38. Assim, a mutagênese e sequenciamento transposon é uma abordagem valiosa no estudo dos traços moleculares das bactérias que são importantes para as interações com seus respectivos hospedeiros em associações patogênicas ou mutualistas.
Divulgações
Os autores declaram que não têm conflito de interesses relativos ao estudo.
Agradecimentos
Agradecemos a Junbeom Lee por fornecer a cepa E. coli WM3064+pRL27 para conjugação e orientação no procedimento, Kathrin Hüffmeier por ajudar na solução de problemas durante a geração de bibliotecas mutantes, e prof. André Rodrigues para apoiar a coleta de insetos e a aquisição de licenças. Agradecemos também a Rebekka Janke e Dagmar Klebsch pelo apoio na coleta e criação dos insetos. Reconhecemos as autoridades brasileiras para a concessão das seguintes autorizações de acesso, coleta e exportação de espécimes de insetos: autorização do SISBIO Nº 45742-1, 45742-7 e 45742-10, processo CNPq nº 01300.004320/2014-21 e 01300.0013848/2017-33, IBAMA Nº 14BR016151DF e 20BR035212/DF). Esta pesquisa foi apoiada por financiamento da German Science Foundation (DFG) Research Grants FL1051/1-1 e KA2846/6-1.
Materiais
| Name | Company | Catalog Number | Comments |
| 2,6- Diaminopimelic Acid | Alfa Aesar | B22391 | For E.coli WM3064+ pRL27 |
| Agar - Agar | Roth | 5210 | |
| Agarose | Biozym | 840004 | |
| AMPure beads XP (magentic beads + polyethylene glycol + salts) | Beckman Coulter | A63880 | Size selection in step 6.6 |
| Bleach (NaOCl) 12% | Roth | 9062 | |
| Bowtie2 v.2.4.2 | Bioinfromatic tool for read mapping. Reference 10 in main manuscript. | ||
| Buffer-S | Peqlab | PEQL01-1020 | For PCRs |
| Cell scraper | Sarstedt | 83.1830 | |
| Cutadapt v.2.10 | Bioinformatic tool for removing specific adapter sequences from the reads. Reference 8 in main manuscript. | ||
| DESeq2 | RStudio package for assessing differential mutant abundance. Usually used for RNAseq analysis. Reference 12 in main manuscript. | ||
| DNA ladder 100 bp | Roth | T834.1 | |
| dNTPs | Life Technology | R0182 | PCR for confirming success of conjugation |
| EDTA, Di-Sodium salt | Roth | 8043 | |
| Epicentre MasterPure Complete DNA and RNA Purification Kit | Lucigen | MC85200 | |
| Ethidium bromide | Roth | 2218.1 | |
| FastQC v.0.11.8 | Bioinformatic tool for assessing the quality of sequencing data. Reference 7 in main manuscript. | ||
| FeatureCounts v.2.0.1 | Bioinformatic tool to obtain read counts per genomic feature. Reference 11 in main manuscript. | ||
| Glycerol | Roth | 7530 | |
| K2HPO4 | Roth | P749 | |
| Kanamycin sulfate | Serva | 26899 | |
| KCl | Merck | 4936 | |
| KH2PO4 | Roth | 3904 | |
| MgSO4.7H2O | Roth | PO27 | |
| Na2HPO4 | Roth | P030 | |
| NaCl | Merck | 6404 | |
| NEBNext Multiplex Oligos for Illumina (Index primers set 1) | New England Biolabs | E7335S | |
| NEBNext Ultra II DNA library prep kit for Illumina | New England Biolabs | E7645S | |
| Peptone (soybean) | Roth | 2365 | For Burkholderia gladioli Lv-StA KB-medium |
| peqGOLD 'Hot' Taq- DNA Polymerase | VWR | PEQL01-1020 | PCR for confirming success of conjugation |
| Petri plates - 145 x 20 mm | Roth | XH90.1 | For selecting transconjugants |
| Petri plates - 90 x 16 mm | Roth | N221.2 | |
| Qiaxcel (StarSEQ GmbH, Germany) | Quality check after DNA library preparation | ||
| Streptavidin beads | Roth | HP57.1 | |
| Taq DNA polymerase | VWR | 01-1020 | |
| Trimmomatic v.0.36 | Bioinformatic tool for trimming low quality reads and also adapter sequences. Reference 9 in main manuscript. | ||
| Tris -HCl | Roth | 9090.1 | |
| Tryptone | Roth | 2366 | For Escherichia coli WM3064+pRL27 LB medium |
| Ultrasonicator | Bandelin | GM 70 HD | For shearing |
| USER enzyme (uracil DNA glycosylase + DNA glycosylase- lyase Endonuclease VIII) | New England Biolabs | E7645S | Ligation step 6.5.2 |
| Yeast extract | Roth | 2363 |
Referências
- Cain, A. K., et al. A decade of advances in transposon-insertion sequencing. Nature Reviews Genetics. 21 (9), 526-540 (2020).
- Chao, M. C., Abel, S., Davis, B. M., Waldor, M. K. The design and analysis of transposon insertion sequencing experiments. Nature Reviews Microbiology. 14 (2), 119-128 (2016).
- Barquist, L., Boinett, C. J., Cain, A. K. Approaches to querying bacterial genomes with transposon-insertion sequencing. RNA Biology. 10 (7), 1161-1169 (2013).
- Flórez, L. V., et al. Antibiotic-producing symbionts dynamically transition between plant pathogenicity and insect-defensive mutualism. Nature Communications. 8 (1), 15172 (2017).
- Flórez, L. V., Kaltenpoth, M. Symbiont dynamics and strain diversity in the defensive mutualism between Lagria beetles and Burkholderia. Environmental Microbiology. 19 (9), 3674-3688 (2017).
- Flórez, L. V., et al. An antifungal polyketide associated with horizontally acquired genes supports symbiont-mediated defense in Lagria villosa beetles. Nature Communications. 9 (1), 2478 (2018).
- . FastQC A quality control tool for high throughput sequence data Available from: https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ (2012)
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet.journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: A flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Liao, Y., Smyth, G. K., Shi, W. FeatureCounts: An efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30 (7), 923-930 (2014).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Gaytán, M. O., Martínez-Santos, V. I., Soto, E., González-Pedrajo, B. Type three secretion system in attaching and effacing pathogens. Frontiers in Cellular and Infection Microbiology. 6, 129 (2016).
- Hachani, A., Wood, T. E., Filloux, A. Type VI secretion and anti-host effectors. Current Opinion in Microbiology. 29, 81-93 (2016).
- Deep, A., Chaudhary, U., Gupta, V. Quorum sensing and bacterial pathogenicity: From molecules to disease. Journal of Laboratory Physicians. 3 (1), 4-11 (2011).
- Silva, A. J., Benitez, J. A. Vibrio cholerae biofilms and cholera pathogenesis. PLOS Neglected Tropical Diseases. 10 (2), 0004330 (2016).
- Navarro-Garcia, F., Ruiz-Perez, F., Cataldi, &. #. 1. 9. 3. ;., Larzábal, M. Type VI secretion system in pathogenic Escherichia coli: structure, role in virulence, and acquisition. Frontiers in Microbiology. 10, 1965 (2019).
- Ribet, D., Cossart, P. How bacterial pathogens colonize their hosts and invade deeper tissues. Microbes and Infection. 17 (3), 173-183 (2015).
- Schwarz, S., et al. Burkholderia Type VI secretion systems have distinct roles in eukaryotic and bacterial cell interactions. PLoS Pathogens. 6 (8), 1001068 (2010).
- Jones, C., et al. Kill and cure: genomic phylogeny and bioactivity of Burkholderia gladioli bacteria capable of pathogenic and beneficial lifestyles. Microbial Genomics. 7 (1), 000515 (2021).
- Takeshita, K., Kikuchi, Y. Riptortuspedestris and Burkholderia symbiont: an ideal model system for insect-microbe symbiotic associations. Research in Microbiology. 168 (3), 175-187 (2017).
- Powell, J. E., et al. Genome-wide screen identifies host colonization determinants in a bacterial gut symbiont. Proceedings of the National Academy of Sciences of the United States of America. 113 (48), 13887-13892 (2016).
- Brooks, J. F., et al. Global discovery of colonization determinants in the squid symbiont Vibrio fischeri. Proceedings of the National Academy of Sciences of the United States of America. 111 (48), 17284-17289 (2014).
- Somprasong, N., McMillan, I., Karkhoff-Schweizer, R. R., Mongkolsuk, S., Schweizer, H. P. Methods for genetic manipulation of Burkholderia gladioli pathovar cocovenenans. BMC Research Notes. 3 (308), (2010).
- Garcia, E. C. Burkholderia thailandensis: Genetic manipulation. Current Protocols in Microbiology. 45, 1-15 (2017).
- Headd, B., Bradford, S. A. The conjugation window in an Escherichia coli K-12 strain with an IncFII plasmid. Applied and Environmental Microbiology. 86 (17), 00948 (2020).
- Van Opijnen, T., Camilli, A. Transposon insertion sequencing: A new tool for systems-level analysis of microorganisms. Nature Reviews Microbiology. 11 (7), 435-442 (2013).
- Gallagher, L. A., Ramage, E., Patrapuvich, R., Weiss, E., Brittnacher, M., Manoil, C. Sequence-defined transposon mutant library of Burkholderia thailandensis. mBio. 4 (6), 00604-00613 (2013).
- Wong, Y. -. C., et al. Candidate essential genes in Burkholderia cenocepacia J2315 identified by genome-wide TraDIS. Frontiers in Microbiology. 7, 1288 (2016).
- Moule, M. G., et al. Genome-wide saturation mutagenesis of Burkholderia pseudomallei K96243 predicts essential genes and novel targets for antimicrobial development. mBio. 5 (1), 00926 (2014).
- Ding, Q., Tan, K. S. Himar1 transposon for efficient random mutagenesis in Aggregatibacter actinomycetemcomitans. Frontiers in Microbiology. 8, 1842 (2017).
- Green, B., Bouchier, C., Fairhead, C., Craig, N. L., Cormack, B. P. Insertion site preference of Mu, Tn5, and Tn7 transposons. Mobile DNA. 3, 3 (2012).
- Lodge, J. K., Weston-Hafer, K., Berg, D. E. Transposon Tn5 target specificity: Preference for insertion at G/C pairs. Genetics. 120 (3), 645-650 (1988).
- Ará Ujo, W. L., et al. Genome sequencing and transposon mutagenesis of Burkholderia seminalis TC3.4.2R3 identify genes contributing to suppression of orchid necrosis caused by B. gladioli. Molecular Plant-microbe Interactions: MPMI. 29 (6), 435-446 (2016).
- Dobritsa, A. P., Samadpour, M. Reclassification of Burkholderiainsecticola as Caballeroniainsecticola comb. nov. and reliability of conserved signature indels as molecular synapomorphies. International Journal of Systematic and Evolutionary Microbiology. 69 (7), 2057-2063 (2019).
- Ohbayashi, T., et al. Insect's intestinal organ for symbiont sorting. Proceedings of the National Academy of Sciences of the United States of America. 112 (37), 5179-5188 (2015).
- Ross, C., Scherlach, K., Kloss, F., Hertweck, C. The molecular basis of conjugated polyyne biosynthesis in phytopathogenic bacteria. Angewandte Chemie International Edition. 53 (30), 7794-7798 (2014).
- Atkins, T., et al. A mutant of Burkholderia pseudomallei, auxotrophic in the branched chain amino acid biosynthetic pathway, is attenuated and protective in a murine model of melioidosis. Infection and Immunity. 70 (9), 5290-5294 (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados