Method Article
Administration efficace d’interférence ARN orale (ARNi) aux moustiques anophèles gambiens adultes
Dans cet article
Résumé
L’administration orale d’ARNd produit par des bactéries, une méthode d’administration de l’interférence ARN (ARNi) couramment utilisée chez Caenorhabditis elegans, a été appliquée avec succès ici aux moustiques adultes. Notre méthode permet des études robustes de génétique inverse et des études de vecteurs bloquant la transmission sans l’utilisation de l’injection.
Résumé
L’interférence ARN est un outil très utilisé pour l’analyse génétique inverse depuis deux décennies. Chez les moustiques adultes, l’administration d’ARN double brin (ARNds) a été réalisée principalement par injection, ce qui nécessite beaucoup de temps et ne convient pas aux applications sur le terrain. Pour surmonter ces limitations, nous présentons ici une méthode plus efficace pour l’activation robuste de l’ARNi par administration orale d’ARNds à Anopheles gambiae adulte. De longs ARNd ont été produits dans la souche HT115 (DE3) d’Escherichia coli , et une suspension concentrée de bactéries contenant de l’ARNd tuées thermiquement dans du saccharose à 10% a été offerte sur des boules de coton ad-libitum aux moustiques adultes. Les boules de coton ont été remplacées tous les 2 jours pendant toute la durée du traitement. L’utilisation de cette méthode pour cibler le doubleex (un gène impliqué dans la différenciation sexuelle) ou la tête de fourche (qui code pour un facteur de transcription des glandes salivaires) a entraîné une réduction de l’expression du gène cible et / ou du signal d’immunofluorescence des protéines, mesurée par PCR quantitative en temps réel (qRT-PCR) ou microscopie confocale à fluorescence, respectivement. Des défauts dans la morphologie des glandes salivaires ont également été observés. Cette méthode très flexible, conviviale, peu coûteuse et rapide d’administration de l’ARNds pourrait être largement applicable aux gènes cibles importants pour la physiologie des insectes vecteurs et au-delà.
Introduction
De nombreuses maladies sont transmises par les moustiques, ce qui fait de l’étude de la physiologie et de la génétique des moustiques une entreprise importante. L’utilisation de l’ARNi dans ces organismes a été importante au cours des 20 dernières années et a permis la caractérisation fonctionnelle de nombreux gènes de moustiques 1,2,3,4,5. La technique la plus couramment utilisée pour l’administration d’ARNd a été la microinjection, qui présente l’inconvénient de blesser les moustiques et nécessite beaucoup de temps et d’efforts. Des méthodes d’administration orale d’ARNi ont été testées, mais principalement au stade larvaire des moustiques 6,7,8,9. L’administration orale d’ARNds chez les moustiques adultes n’a pas été pleinement explorée et pourrait être un outil utile pour l’étude de la biologie des vecteurs et de la lutte antivectorielle.
Le paludisme est transmis par les moustiques anophèles lorsqu’un moustique femelle infecté prend un repas de sang d’un hôte non infecté et injecte de la salive contenant des parasites du paludisme10. Pour finalement être transmis dans la salive d’un moustique, le parasite doit surmonter de nombreux obstacles, notamment échapper au système immunitaire des moustiques, traverser la barrière de l’intestin moyen et envahir les glandes salivaires11. L’architecture des glandes salivaires (SG) des moustiques est la clé de l’invasion parasitaire et cette architecture est contrôlée à la fois par des facteurs de transcription clés exprimés par les glandes salivaires ainsi que par des déterminants du dimorphisme sexuel. Plusieurs facteurs de transcription hautement conservés sont nécessaires pour la spécification cellulaire et le maintien homéostatique des glandes salivaires et pour la production et la sécrétion de protéines salivaires qui fonctionnent dans l’alimentation sanguine 12,13,14. La tête de fourche (Fkh) est un facteur de transcription de l’hélice ailée qui fonctionne comme un régulateur majeur de la structure et de la fonction SG de l’insecte (basé sur des études chez les mouches des fruits et la teigne du ver à soie)15,16,17,18,19,20. Dans les SG de drosophiles, Fkh fonctionne avec Sage, un facteur de transcription hélicoïque-boucle-hélice (bHLH) spécifique à SG, pour favoriser la survie sg et la production de salive19. Un co-régulateur important et positif de la production de salive chez la drosophile est CrebA, un facteur de transcription par fermeture éclair de leucine bien étudié qui régule à la hausse l’expression des gènes de la voie sécrétoire 21,22,23. Il existe également un fort degré de différenciation morphologique dans les glandes salivaires féminines qui joue probablement un rôle clé, non seulement dans l’alimentation sanguine, mais aussi dans la capacité des parasites à envahir ce tissu24.
De nombreux gènes impliqués dans la détermination de la survie, de la structure, de la physiologie et du dimorphisme sexuel des glandes salivaires ont des profils d’expression spatio-temporelle complexes 25,26,27, et les méthodes traditionnelles d’administration de l’ARNd pour induire l’ARNi ne sont pas toujours efficaces pour cibler ces types de gènes dans ce tissu ou dans d’autres. Cependant, l’administration orale d’ARNds au stade larvaire Aedes aegypti et de moustiques An. gambiae a été utilisée avec succès pour faire taire la forme spécifique à la femelle du gène dsx 9,28. Des études antérieures utilisant l’ARNds dans les glandes salivaires des moustiques ont révélé que, bien que de grandes quantités d’ARNds aient été nécessaires, l’effet de silencieux était relativement durable (au moins 13 jours)29. Ici, la capacité de la souche HT115 (DE3) d’E. coli tuée par la chaleur exprimant un ARNd spécifique à la séquence pour dsx, fkh ou CrebA à induire le silence de ces gènes par ARNi chez les moustiques femelles adultes a été testée. L’administration orale d’ARNd a induit l’élimination des gènes chez An. gambiae, avec des réductions claires des niveaux d’ARNm et avec des phénotypes compatibles avec la perte de fonction de ces gènes. Ainsi, cette approche fonctionnera probablement pour détruire la fonction d’une variété de gènes des glandes salivaires.
Protocole
1. Clonage de l’ARNds en vecteur d’expression d’E. coli
- Sélectionnez la séquence de gènes cible à insérer dans un vecteur approprié pour l’expression de l’ARNds. Récupérez les valeurs d’expression de Vectorbase.org à l’aide de la méthode suivante.
- Recherchez un gène d’intérêt (p. ex., tableau 1) dans le champ de recherche de la page d’accueil.
- Dans la page de gène résultante, accédez au 8. Section de transcriptomique .
- Recherchez les expériences pertinentes d’expression génique de séquençage d’ARN et de microréseaux.
- Transcrivez les valeurs d’intérêt dans le tableur et créez un tableau de données.
- Choisissez un plasmide disponible dans le commerce avec au moins un promoteur T7 à utiliser. Si le plasmide sélectionné n’a qu’un seul promoteur T7 (comme le font la plupart des plasmides commerciaux), incluez un deuxième promoteur T7 dans l’amorce inverse à utiliser pour l’amplification de l’ADNds pour le gène d’intérêt.
REMARQUE: La séquence d’ARNd pour les gènes cibles peut être sélectionnée à l’aide de l’application Web E-ARNi pour la conception de réactifsARNi 30. L’ARNds long (environ 400 pb) ou l’ARNds en épingle à cheveux court (ARNh) peuvent être conçus en fonction de séquences de gènes spécifiques. Ces séquences doivent être amplifiées et séquencées pour confirmation d’identité avant le clonage. Les régions génétiques, les plasmides et les promoteurs sélectionnés utilisés dans cette étude sont énumérés dans le dossier supplémentaire 1. - Effectuez le clonage selon une procédure simple en une étape décrite précédemment 9,31. À cette fin, purifier le produit PCR et ligaturer l’ADN plasmidique linéarisé. Utilisez le produit de la ligature pour la transformation par choc thermique des cellules E. coli compétentes32. Sélectionnez les cellules transformées par le biais d’un filtrage bleu/blanc. Confirmez l’orientation de l’insert à l’aide d’une PCR d’amorce T7 et confirmez la séquence à l’aide d’amorces M13.
REMARQUE: Les dépistages blancs / bleus peuvent être utilisés lorsque le plasmide sélectionné pour la transformation porte le gène lacZ qui code pour la β-galactosidase. Les colonies blanches doivent contenir l’insert souhaité dans la lacZ et peuvent être sélectionnées pour confirmer davantage la présence et l’orientation de la séquence cible33. - Purifier le plasmide de la première transformation et l’utiliser pour transformer E. coli HT115 (DE3) compétent comme décrit précédemment34. Après confirmation que le plasmide avec l’insert est présent dans le E. coli HT115 (DE3) compétent, fabriquer des stocks de glycérol de bactéries à usage unique.
REMARQUE: Un ARNds témoin non apparenté approprié doit être acquis ou préparé à être utilisé dans chaque expérience. Dans ce cas, la séquence du gène non apparenté aintegumenta (fourmi) d’Arabidopsis thaliana est utilisée.
2. Préparation de bactéries tuées par la chaleur exprimant l’ARNds
- Cultiver une culture à partir d’une seule colonie bactérienne de la souche HT115 (DE3) d’E. coli contenant le plasmide exprimant l’ARNd dans 50 mL de bouillon Luria (LB) contenant 100 μg/mL d’ampicilline et 12,5 μg/mL de tétracycline, sur un agitateur de plate-forme (180 tr/min) à 37 °C pendant 12 h.
- Diluer la culture bactérienne (1:1000) dans 2x milieux de tryptone de levure (2x YT) contenant 100 μg/mL d’ampicilline et 12,5 μg/mL de tétracycline.
- Induire la production d’ARNd en ajoutant 40 μM (concentration finale) d’isopropyle β-D-1-thiogalactopyranoside (IPTG).
- Lorsque les cellules atteignent un O.D.600 = 0,4, environ après 2 h d’induction à 37 °C avec agitation à 180 tr/min, préparer une suspension concentrée de bactéries tuées par la chaleur comme décrit par Taracena et al 9. Abreuver les cellules par centrifugation (4000 x g, 4 °C, 10 min) et laver les cellules dans un volume de tampon phosphate de sodium (PBS).
- Tourner à nouveau dans les mêmes conditions, suspendre à nouveau dans PBS à 1/100 du volume initial et placer à 70 °C pendant 1 h.
- Faire 400 μL aliquotes des bactéries tuées par la chaleur et stocker ces aliquotes à -20 °C jusqu’à une utilisation ultérieure (ne pas stocker plus d’une semaine). Cette suspension de bactéries tuées par la chaleur contient l’ARNd spécifique pour les expériences d’ARNi. Effectuez cette procédure à la fois pour les bactéries dsRNA du gène cible et pour le contrôle dsRNA non apparenté à utiliser dans chaque expérience.
3. Nourrir les moustiques avec des bactéries tuées par la chaleur exprimant l’ARNds
- Décongeler une aliquote de suspension de bactéries dsRNA (HT115 (DE3)) et mélanger avec 1,6 mL de solution de sucre à 12 % contenant 0,2 % de méthylparabène.
- Faites tremper une petite boule de coton dans cette solution et placez la boule de coton trempée dans une cage contenant des moustiques de 5 jours. Assurez-vous que les moustiques se nourrissent de cette solution, en capturant simultanément le sucre et les bactéries contenant de l’ARNd.
- Changez la boule de coton trempée dans une solution d’ARNd-sucre tous les deux jours pendant 8 jours consécutifs.
- Gardez les cages à moustiques dans des conditions constantes, c’est-à-dire 27 °C et 80% d’humidité relative avec une photopériode de 12 h:12 h de lumière: photocycle sombre, séparé par une période de 30 min à l’aube et de 30 min au crépuscule.
4. Les niveaux d’expression des gènes cibles du test
- Anesthésiez à froid les moustiques en plaçant le récipient sur la glace pendant une minute ou jusqu’à ce que les moustiques cessent de bouger. Une fois les moustiques anesthésiés, placez-les sur une surface froide pour isoler les femelles en vue de leur dissection.
- Vaporisez 70% d’éthanol sur les moustiques et placez-les sur une surface en verre avec du PBS. Avec une paire de pinces, fixez la tête du moustique et tirez le thorax très lentement, ce qui permet aux glandes salivaires d’être libérées dans le PBS.
- Gardez les glandes salivaires dans le PBS glacé jusqu’à ce que 10 personnes aient été disséquées. Pool Ten SG pour l’extraction de l’ARN en utilisant la méthode guanidinium thiocyanate-phénol-chloroforme. Suspendre la pastille d’ARN dans 30 μL d’eau sans RNase.
- Utilisez 1 μL aliquote de l’ARN extrait du SG à l’étape précédente, pour lire l’absorbance à 260 et 280 nm et calculer la concentration d’ARN de chaque échantillon en multipliant avec le facteur de dilution. Un rapport 260/280 de ~2,0 indique un ARN de bonne qualité.
- Utilisez 1 μg de l’ARN purifié pour synthétiser de l’ADN complémentaire (ADNc) à l’aide d’un kit commercial de transcription inverse.
- Effectuer une dilution 1:10 de l’ADNc pour préparer une réaction RT-PCR selon les recommandations du fabricant. Pour chaque échantillon, préparez une réaction pour le gène cible et, en parallèle, mettez en place une réaction avec le gène d’entretien ménager (HK). Définissez chaque réaction génique dans un triplicate technique pour éliminer l’impact de la variation aléatoire de la méthode.
REMARQUE: Ici, le gène S7 ribosomique An. gambiae (GeneBank: L20837.1) et l’actine (VectorBase: AGAP000651) ont été utilisés comme gènes HK. - Utilisez tous les apprêts à une concentration finale de 300 nM, en suivant les indications du fabricant SYBR-green. Amplifier avec des conditions PCR standard : 95 °C pendant 10 min, suivi de 40 cycles de 15 s à 95 °C et de 60 s à 60 °C.
REMARQUE: Pour quantifier l’expression des gènes, la méthode delta-delta-Ct (ΔΔCt) est utilisée. Delta Ct (ΔCt) est la différence entre le Ct du gène cible et le Ct du gène d’entretien ménager. ΔΔCt est la différence entre le ΔCt du groupe expérimental et le ΔCt du groupe témoin35.
5. Évaluation phénotypique : alimentation sanguine réussie
- Pour évaluer la capacité à se nourrir dans le sang, placez des groupes de 15 moustiques femelles traitées avec de l’ARNd cible et contrôlez sur de petites cages (12 cm de diamètre) et affamez-les pendant 4 h.
- À l’aide d’un bain-marie circulant réglé à 37 °C, de mangeoires à moustiques en verre (24 mm de diamètre) et d’une membrane de parafilm offrent du sang de mouton défibriné aux moustiques.
REMARQUE: Le sang peut être acquis auprès d’un vendeur commercial qui le prélève de manière aseptique sur des animaux sains, donneurs d’origine américaine et défibrome manuellement sans anticoagulants ni additifs. - Par observation directe, comptez et enregistrez le nombre de tentatives d’enquête pour obtenir avec succès un repas de sang des cinq premières femelles à devenir complètement engorgées dans chaque groupe.
REMARQUE: Pour éviter des changements métaboliques importants chez les moustiques, qui pourraient interférer avec les ressources énergétiques ayant un impact sur le comportement de recherche de sang, la famine a été réduite au minimum (4 h). En conséquence, tous les moustiques ne chercheraient pas avidement la farine de sang et nous avons limité le nombre de femelles engorgées à cinq (un tiers du total de chaque groupe), afin de réduire l’effet des variables temporelles telles que l’exposition à l’odeur humaine, le changement de température entre les chambres et les surfaces d’alimentation, etc.
6. Évaluation phénotypique : morphologie des glandes salivaires et régulation négative des protéines pertinentes
- Isolez les tissus frais dans 1x solution saline tamponnée au phosphate (PBS) comme décrit à l’étape 4.2 et fixez-les dans de l’acétone glacée pendant 90 s. Rincer plusieurs fois dans 1x PBS après avoir retiré l’acétone. Incuber avec des anticorps primaires pendant la nuit à 4 °C avec de l’antisérum (voir tableau des matériaux) dilué dans 1x PBS.
REMARQUE : Voir le tableau des matériaux pour l’identification des anticorps primaires utilisés pour les protéines salivaires (protéine antiplaquettaire d’anophèle , AAPP; Mucin 2, MUC2), facteurs de transcription SG (Fork Head, fkh; Sauge, sauge; Protéine A de liaison aux éléments de réponse cyclique-AMP, CrebA), et un marqueur des vésicules sécrétoires (Rab11). Ces anticorps sont utilisés comme lectures pour la forme et la fonction SG. Cependant, tout anticorps adapté à l’immunofluorescence devrait convenir à ce protocole. - Laver dans 1x PBS plusieurs fois. Ajouter les anticorps secondaires (fluorescents) dilués dans 1x PBS, et incuber dans l’obscurité à température ambiante pendant 2 h. Ajouter n’importe quelle contre-tache [telle que le 4′,6-diamidino-2-phénylindole (DAPI; ADN), l’agglutinine de germe de blé (WGA; pour la chitine), la phalloïdine (pour la F-actine) et/ou le Rouge du Nil (pour les lipides)] 30 min avant la fin de l’incubation de 2 h.
- Laver trois fois dans 1x PBS. Ensuite, montez les tissus dans du glycérol à 100% sur une lame de microscope standard avec un couvercle de 1 mm d’épaisseur et stockez-les à -20 ° C jusqu’à l’imagerie à l’aide d’un microscope confocal à fluorescence.
REMARQUE : Pour obtenir des données quantitatives, les paramètres d’imagerie doivent être maintenus constants. Ici, seules les images de projection d’intensité maximale à travers tout le volume 3D du tissu ont été incluses, et toute la quantification de l’image a été normalisée entre les traitements (dans le cadre d’une expérience) en fonction du signal DAPI dans les restes tissulaires non SG (corps gras, cuticule ou tête) également présents sur la diapositive.
Résultats
Pour commencer, les données d’expression des microréseaux de VectorBase ont été utilisées pour analyser des cibles potentielles à travers les stades de développement 36,37 afin de déterminer le statut d’expression de tous les gènes pertinents pour la présente étude (tableau 1). Comme prévu, tous les gènes cibles que nous avons choisis ont montré une expression chez les SG adultes. Les niveaux d’aapp et de sauge étaient particulièrement élevés (tableau 1). Il convient également de noter les niveaux élevés d’expression de f-Agdsx chez les SG9 de sexe féminin adulte.
Des segments spécifiques de chaque gène ont été évalués pour être utilisés comme ARNds à l’aide de l’application Web E-ARNi pour la conception de réactifs ARNi30. Les régions d’environ 400 pb contenant des séquences uniques à chaque gène cible ont ensuite été clonées (Figure 1A), transformées en souches bactériennes appropriées et utilisées pour préparer des suspensions de bactéries tuées par la chaleur, qui ont été induites pour produire de l’ARNd. Les moustiques adultes ont été nourris pendant 8 jours avec les boules de coton imbibées de saccharose contenant les suspensions bactériennes d’ARNds pour f-Agdsx, fkh ou fourmi (le contrôle négatif non lié).
Pour l’analyse de l’alimentation à l’ARNi des moustiques femelles, il a d’abord été déterminé si l’alimentation en aRNds f-Agdsx ou fkh induisait un silençage génique. Une réduction de 98,8 % (±2,1) des niveaux de transcription fkh a été observée dans le groupe nourri avec de l’ARNm fkh (Figure 1B), ce qui indique que l’ARNds a très efficacement réduit l’abondance des transcriptions fkh dans les SG. Étonnamment, les niveaux d’ARNm fkh ont été réduits de 82,0 % (±18,9) chez les moustiques traités avec de l’ARNds pour f-Agdsx, qui présentaient une réduction de 89,86 % (±4,48 ) de f-Agdsx , suggérant que fkh pourrait être une cible de F-Dsx dans la glande salivaire. Parallèlement à la réduction significative des niveaux d’expression de fkh, les moustiques fkh-knockdown ont montré une augmentation significative du nombre de tentatives de sondage nécessaires pour nourrir le sang. Ces moustiques ont présenté, en moyenne, cinq fois plus de tentatives d’alimentation que le groupe témoin ou les moustiques nourris à l’ARNd f-Agdsx pour être complètement engorgés de sang (Figure 1C). Cela a conduit à se demander si les traitements de l’ARNi fkh knockdown ont provoqué des changements dans la localisation et / ou la distribution des principaux régulateurs transcriptionnels (SG TF Sage et CrebA) (Figure 2), des protéines sécrétées (AAPP et mucine) (Figure 3) et de la machinerie sécrétoire [Nile Red (lipides) et Rab11 (vésicules sécrétoires)] (Figure 4). Il est important de noter que des différences substantielles dans l’intensité de la coloration ont été observées entre différentes régions du lobe, lobes et SG individuels.
Comme prévu, les niveaux de coloration à la sauge et à la CrebA ont été nettement réduits dans tous les lobes SG suivant l’ARNi fkh (figure 2B) par rapport à l’ARNi témoin des fourmis (figure 2A). Les réductions des valeurs d’intensité maximale les plus élevées (lignes pointillées rouges et étiquettes numériques) et des valeurs d’intensité maximale les plus basses (lignes pointillées bleues et étiquettes numériques) dans les profils de balayage linéaire ont suggéré des réductions dans les zones de signal élevé et faible dans le tissu (figures 2A, B). Ces données suggèrent que An. gambiae fkh RNAi est efficace et que fkh régule la production et/ou la stabilité des SG TF Sage et CrebA chez An. gambiae, analogue à leur relation génétique dans Drosophila SGs 19,38,39.
Lors de l’examen des protéines composant la salive très abondantes, les niveaux de protéine antiplaquettaire anophèle (AAPP)40,41 ont été réduits dans les trois lobes SG après l’ARNi fkh, par rapport au traitement ARNi témoin (Figure 3A, B; vert). D’autre part, aucun changement dans les niveaux de mucine n’a été observé (Figure 3A, B; violet). Ces données suggèrent que Fkh contribue différemment à l’expression de différents gènes de protéines salivaires.
Enfin, deux marqueurs de sécrétion ont été observés (figures 4A,B) : Rab11 (vésicules associées aux endosomes de recyclage apical)42 et Nile Red (lipides). Une réduction de la fluorescence Rab11 a été observée dans les lobes latéraux distaux (DL) après un traitement par ARNi fkh (Figure 4A v vs 4B v; vert). Cependant, une augmentation du signal Rab11 dans les lobes médial (M) et latéral proximal (PL) (Figure 4A vii, ix vs 4B vii, ix; vert) s’est également produite. Aucune différence perceptible n’a été observée dans le signal rouge du Nil (figures 4A, B; violet) après l’ARNi fkh par rapport au traitement ARNi témoin. Ces données suggèrent que la réduction du fkh peut modifier certaines actions sécrétoires de la machinerie d’une manière complexe qui diffère entre les lobes SG.
| Jeu de données: | Goltsev | Neira Oviedo | Neira Oviedo | Boulanger | Boulanger | Boulanger | Boulanger | ||
| symbole de gène | fonction | AGAP ID | embryon (25 heures) | Larves L3 | L3 SG | femelle adulte corps (3 jours) | mâle adulte corps (3 jours) | femelle adulte SG (3 jours) | mâle adulte SG (3 jours) |
| L’AAPP | protéine de salive | AGAP009974 | 3.92 | 4.38 | 4.33 | 3.81 | 2.46 | 11.92 | 2.69 |
| CrebA | Facteur txn | AGAP001464 | 6.28 | 5.22 | 5.92 | 2.99 | 2.96 | 3.27 | 3.13 |
| " | Facteur txn | AGAP011038 | 4.50 | 4.46 | 5.23 | 2.96 | 2.86 | 3.05 | 2.88 |
| Dsx | Facteur txn | AGAP004050 | 4.91 | 5.39 | 5.55 | 3.72 | 4.00 | 4.57 | 4.01 |
| fkh | Facteur txn | AGAP001671 | 5.18 | 4.67 | 5.25 | 2.99 | 3.09 | 3.21 | 3.05 |
| MUC2 | protéine de salive | AGAP012020 | 4.59 | 5.53 | 5.63 | 2.96 | 3.07 | 3.08 | 3.26 |
| Rab11 | trafic vésiculeux | AGAP004559 | 10.21 | 7.47 | 8.60 | 4.90 | 3.79 | 3.38 | 2.96 |
| sauge | Facteur txn | AGAP013335 | 5.32 | 5.96 | 8.89 | 3.40 | 3.33 | 7.37 | 7.23 |
Tableau 1 : Profils moyens d’expression de microréseaux log2 pour An. gambie gènes d’intérêt. Les noms de gènes, la catégorie fonctionnelle, les identificateurs Vectorbase (AGAP) et les données moyennes d’expression de microréseau log2 recueillies à partir de Vectorbase sont présentées. Ces données indiquent que nos gènes d’intérêt (impliqués dans la biologie et la sécrétion des cellules des glandes salivaires (SG)) sont exprimés et enrichis au stade larvaire 3 (L3) et chez les SG adultes, par rapport aux individus entiers.
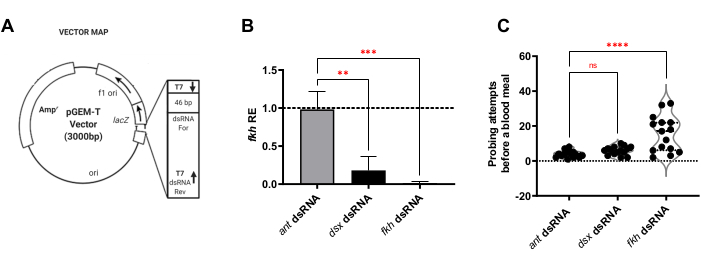
Figure 1 : f-Agdsx et fkh knockdown chez An. gambiae adulte réduit les niveaux d’ARNm fkh dans les SG et affecte la capacité féminine à se nourrir dans le sang. (A) Image représentative de la conception du plasmide utilisée pour la production d’ARNds dans cette méthodologie. La deuxième séquence promotrice T7 est ajoutée au plasmide en l’incluant dans l’amorce 3' utilisée pour amplifier l’insert à cloner dans le plasmide pGEMT. Le plasmide est ensuite transformé en bactéries E. coli HT115 (DE3) et une solution d’alimentation est constituée d’une suspension de bactéries tuées par la chaleur induite dans de l’eau sucrée à 10%. (B) Les animaux nourris avec une solution d’alimentation à ARNds pour f-Agdsx ou fkh ont montré des niveaux significativement plus faibles de transcriptions de fkh (ANOVA unidirectionnelle avec comparaisons multiples; n = 15). Cependant, seul le groupe nourri avec de l’ARNd (C) fkh a montré une différence significative dans le nombre de tentatives de morsure nécessaires pour acquérir un repas de sang. Les moustiques de ce groupe avaient besoin, en moyenne, de cinq fois plus de tentatives d’enquête pour obtenir un repas sanguin réussi que ce dont avaient besoin le groupe témoin ou les groupes nourris au dsx-dsRNA (ANOVA unidirectionnelle avec plusieurs comparaisons; n = 15). Les barres d’erreur indiquent l’erreur type de la moyenne (MEB). Chaque expérience a été menée dans trois répliques biologiques distinctes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
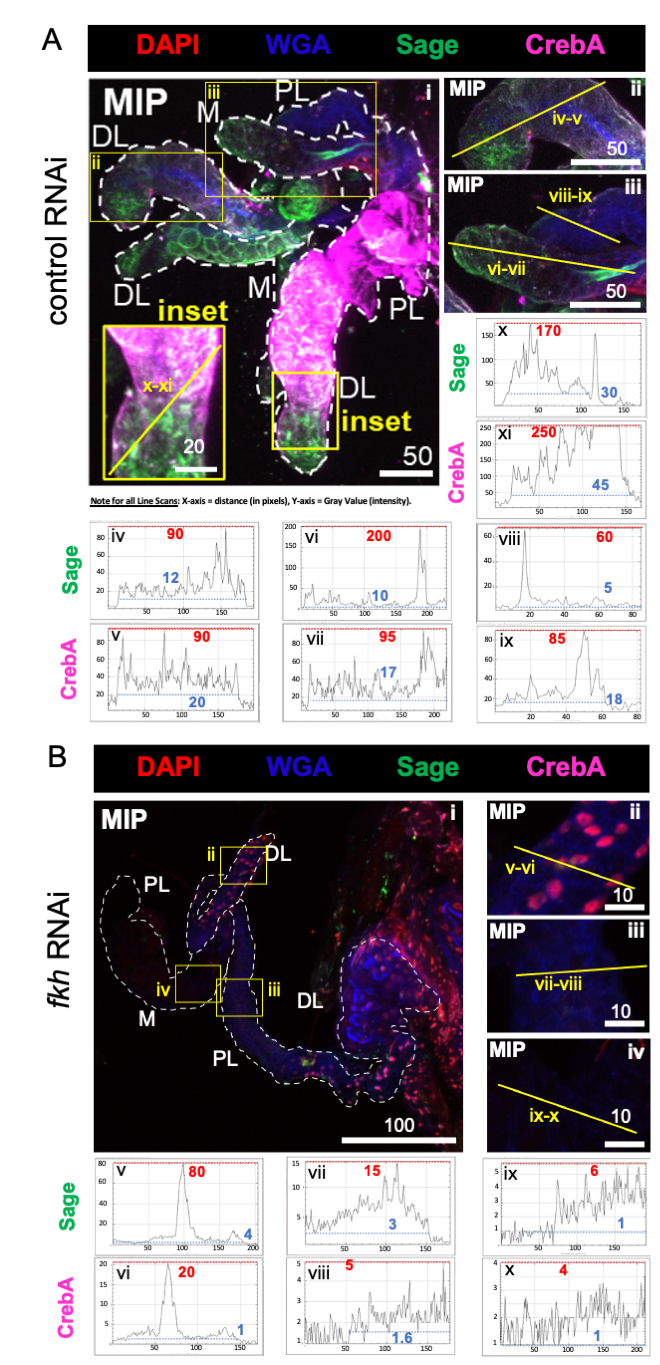
Figure 2 : L’abattage du fkh dans les glandes salivaires adultes d’An. gambiae réduit les niveaux de facteur de transcription SG. On voit des images représentatives du jour 13 des AG de la femelle adulte An. gambiae après 8 jours (jours 5-13) d’exposition orale à (A) un contrôle dsRNA non apparenté (fourmi) ou (B) dsRNA ciblant la tête de fourche SG TF (fkh, AGAP001671) dans 10% de saccharose coloré avec les colorants DAPI (ADN; rouge), agglutinine de germe de blé marquée (WGA, chitine / O-GlcNAcylation; bleu), antisérum contre les SG TF Sage (vert) et CrebA (violet). Les longueurs de barres d’échelle indiquées sont des microns. Les SG (i) sont soulignés par des tirets blancs. Les lignes jaunes dans les images de lobes zoomés (des régions entourées de cases jaunes et étiquetées « encart ») indiquent où les balayages linéaires de l’intensité du signal ont été effectués. Les intensités des canaux verts et violets correspondant aux balayages de ligne pour chaque lobe zoomé sont tracées (toujours de gauche à droite dans le SG) dans les graphiques sous les images; Axe X = distance (en pixels) et axe Y = unité grise (intensité en pixels). La plage dynamique de l’intensité des pixels est délimitée par des lignes pointillées rouges (maximum) et bleues (minimum) et les valeurs correspondantes sont affichées sur chaque graphique. MIP = projection 3D d’intensité maximale sur toute la profondeur SG. DL: lobe latéral distal; M : lobe médial ; PL: lobe latéral proximal; SD: canal salivaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
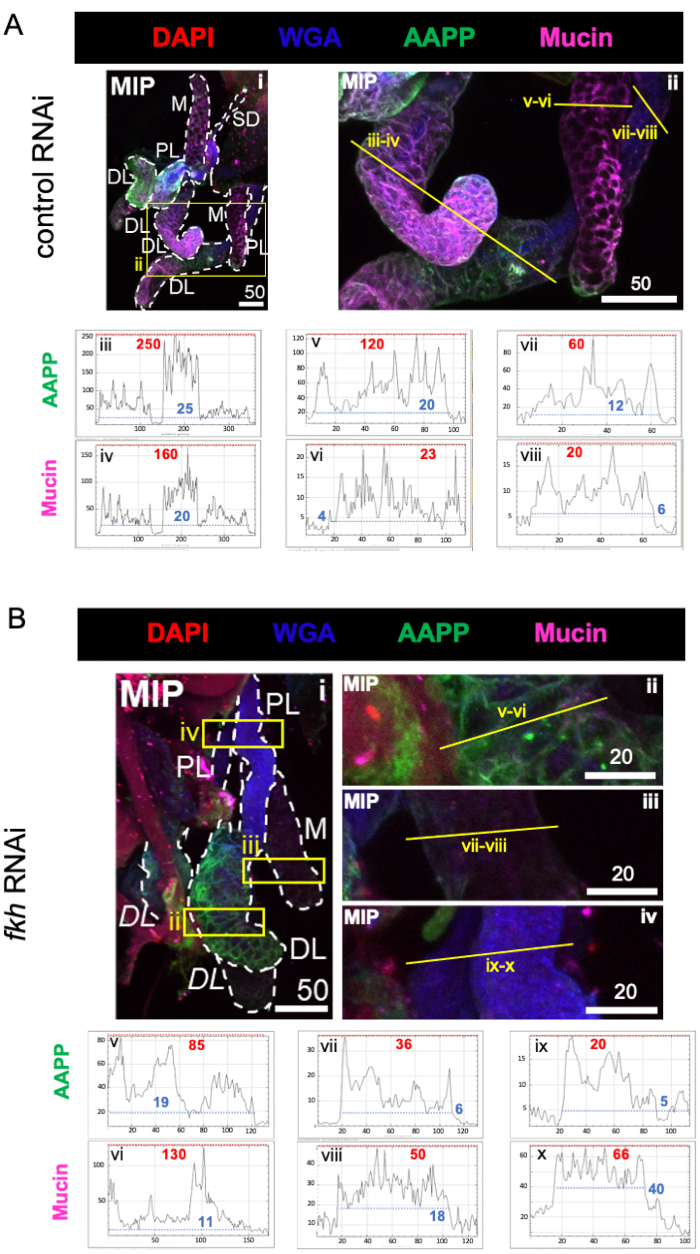
Figure 3 : L’élimination du fkh dans les glandes salivaires adultes d’An. gambiae réduit les niveaux de protéines sécrétées par SG. Sont montrées des images représentatives du jour 13 des AG de la femelle adulte An. gambiae après 8 jours (jours 5-13) d’exposition orale à (A) un contrôle dsRNA non apparenté (fourmi), ou (B) dsRNA ciblant la tête de fourche SG TF (fkh, AGAP001671) dans 10% de saccharose coloré avec les colorants DAPI (ADN; rouge), agglutinine de germe de blé marquée (WGA, chitine / O-GlcNAcylation; bleu), et les protéines salivaires AAPP (vert) et Mucine (MUC2, violet). Les longueurs de barres d’échelle indiquées sont des microns. Les SG (i) sont soulignés par des tirets blancs. Les lignes jaunes dans les images de lobes zoomés (des régions entourées de boîtes jaunes) indiquent où les balayages linéaires de l’intensité du signal ont été effectués. Les intensités des canaux verts et violets correspondant aux balayages de ligne pour chaque lobe sont tracées (toujours de gauche à droite dans le SG) dans les graphiques sous les images; Axe X = distance (en pixels) et axe Y = unité grise (intensité en pixels). La plage dynamique de l’intensité des pixels est délimitée par des lignes pointillées rouges (maximum) et bleues (minimum) et les valeurs correspondantes sont affichées sur chaque graphique. MIP = projection 3D d’intensité maximale sur toute la profondeur SG. DL: lobe latéral distal; M : lobe médial ; PL: lobe latéral proximal; SD: canal salivaire. Les étiquettes italiques « DL » (Bi) indiquent deux régions visibles du même lobe DL. Veuillez cliquer ici pour voir une version agrandie de cette figure.
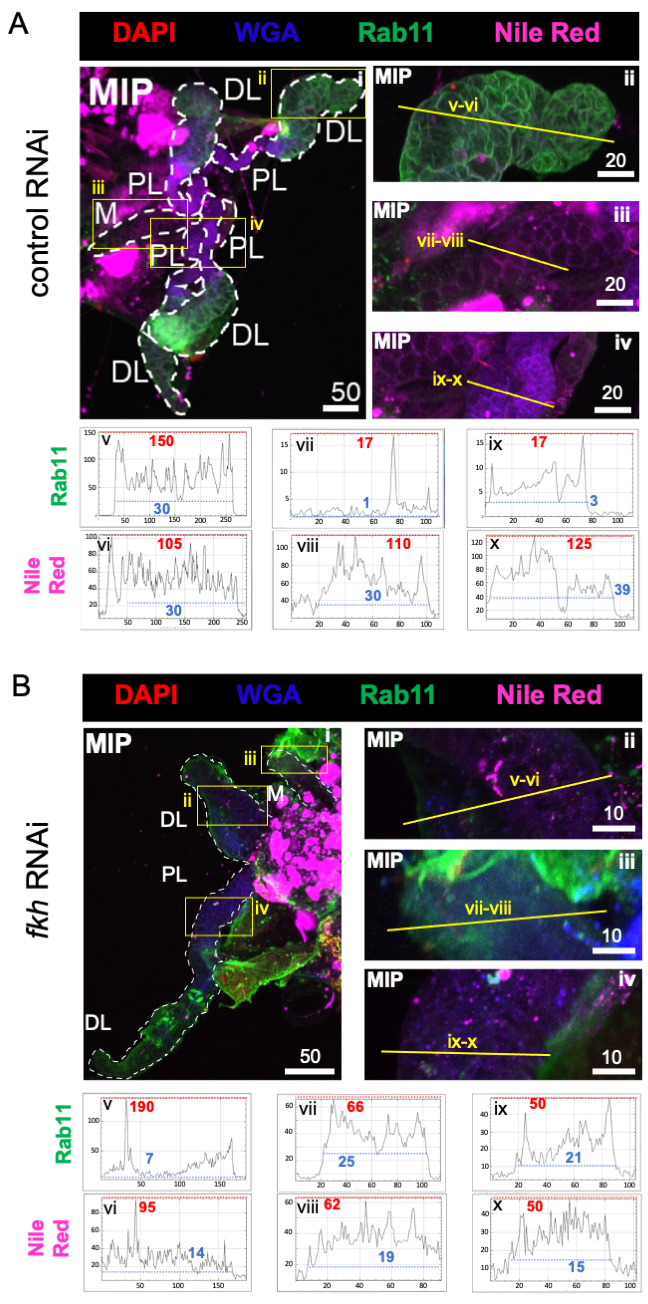
Figure 4 : L’abattage du fkh dans les glandes salivaires adultes d’An. gambiae réduit les marqueurs de sécrétion de SG. On voit des images représentatives du jour 13 des SG an. gambiae femelles adultes après 8 jours (jours 5-13) d’exposition orale à (A) un contrôle dsRNA non apparenté (fourmi), ou (B) dsRNA ciblant la tête de fourche SG TF (fkh, AGAP001671) dans 10% de saccharose coloré avec les colorants DAPI (ADN; rouge), agglutinine de germe de blé marqué (WGA, chitine / O-GlcNAcylation; bleu), Rouge du Nil (lipides; violet) et antisérums contre le marqueur de vésicule endosome de recyclage Rab11 (vert). Les longueurs de barres d’échelle indiquées sont des microns. Les SG (i) sont soulignés par des tirets blancs. Les lignes jaunes dans les images de lobes zoomés (des régions entourées de boîtes jaunes) indiquent où les balayages linéaires de l’intensité du signal ont été effectués. Les intensités des canaux verts et violets correspondant aux balayages linéaires pour chaque lobe sont tracées (toujours de gauche à droite dans le SG) dans les graphiques sous les images; Axe X = distance (en pixels) et axe Y = unité grise (intensité en pixels). La plage dynamique de l’intensité des pixels est délimitée par des lignes pointillées rouges (maximum) et bleues (minimum) et les valeurs correspondantes sont affichées sur chaque graphique. MIP = projection 3D d’intensité maximale sur toute la profondeur SG. DL: lobe latéral distal; M : lobe médial ; PL: lobe latéral proximal; SD: canal salivaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Dossier supplémentaire 1. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
La capacité de délivrer efficacement l’ARNd aux moustiques An. gambiae par voie orale a de vastes implications pour les études de biologie vectorielle en laboratoire et sur le terrain. La microinjection a longtemps été acceptée comme le mode d’administration préféré des produits chimiques, des anticorps, de l’ARNi et des stratégies de modification génétique chez les moustiques43,44. Les conséquences d’une manipulation physique importante, de dommages cellulaires et de stress peuvent être évitées par l’utilisation de l’administration orale, qui pourrait également convenir à des applications à grande échelle ou sur le terrain. Des travaux antérieurs ont suggéré que l’ARNi agit de manière omniprésente chez un moustique adulteindividuel 29, permettant des effets dans tous les tissus, y compris les glandes salivaires. En nourrissant les moustiques avec un grand nombre d’E. coli exprimant l’ARNd qui sont digérés de manière asynchrone sur une longue période, on peut potentiellement obtenir une exposition cohérente et uniforme à l’ARNi chez tous les individus dans une cage. Cette méthode permet de nourrir un grand nombre de moustiques et d’analyser la variabilité potentielle des phénotypes résultants en fonction du gène cible. Cependant, une considération importante est la possibilité d’une distribution hétérogène des bactéries, et donc de l’ARNds, dans la fibre de coton. Les 400 μL de bactéries utilisés quotidiennement pour l’alimentation en sucre des moustiques contiendraient environ ≤4,6 μg d’ARNds, comme décrit et calculé précédemment9, mais la quantité d’ARNds ingérée par chaque moustique n’a pas été déterminée individuellement. Si la construction d’ARNd devient routinière, ce protocole de traitement simple permet une assimilation rapide de cette technique par tout chercheur sur les moustiques. A priori, le temps passé pendant le traitement (30 min par jour) est insignifiant par rapport au temps nécessaire pour apprendre et appliquer la microinjection à des échantillons de taille similaire.
L’alimentation en ARNds est couramment utilisée pour des études de génétique inverse dans l’organisme modèle Caenorhabditis elegans45. Ce niveau élevé d’utilisation souligne la valeur de l’approche d’administration orale. La construction d’une bibliothèque à l’échelle du génome d’An. gambiae dans E. coli transformé, similaire à celle qui existe dans C. elegans46,47, permettrait un dépistage génétique inversé rapide chez les moustiques à une échelle accrue. Cependant, il est important de noter que l’efficacité de la méthode dépend dans une large mesure des niveaux endogènes de transcription et si l’expression n’est pas limitée au tissu cible mais exprimée plus largement 4,8,44. En outre, il existe des preuves que certains insecticides pourraient induire un évitement comportemental des moustiques48, et se nourrir avec des bactéries qui induisent potentiellement des effets indésirables en eux pourrait déclencher des modèles similaires d’évitement. Dans le cadre contrôlé du laboratoire, où les moustiques n’avaient pas de source de nourriture alternative, ils n’avaient pas le choix d’éviter l’eau sucrée avec E. coli et le besoin d’une source nutritive l’emporterait probablement sur l’instinct d’éviter les bactéries. Toutefois, cela devrait être envisagé si la stratégie devait être utilisée dans des contextes moins contrôlés.
Il peut être possible de cibler plusieurs gènes simultanément (en utilisant une construction, plusieurs constructions ou un mélange d’isolats bactériens transformés), mais d’autres études sont nécessaires pour évaluer l’efficacité. Une autre considération importante à ce stade est l’évaluation des effets hors cible ou synergiques possibles lors de l’utilisation de cibles uniques ou multiples. L’établissement de gènes et de groupes de contrôle appropriés est une partie importante de la conception expérimentale. De plus, il est tentant de spéculer que cette approche pourrait être utilisée pour cibler d’autres agents pathogènes ou virus49. Des travaux antérieurs sur l’induction de l’ARNi chez les moustiques ont été effectués dans des conditions où le réactif était directement injecté, de sorte que E. coli n’était pas présent. E. coli peut fournir un compartiment protecteur permettant la libération plus lente de l’ARNds au fil du temps, garantissant que l’exposition est plus ou moins continue sur une période beaucoup plus longue29.
Enfin, ces résultats montrent que les effets de cette technique sont accordables en ajustant le délai (durée et jour de début) de l’exposition et la quantité d’E. coli utilisée. Cette fonctionnalité nous a permis d’étudier les fonctions des gènes essentiels (dsx et fkh) en identifiant les conditions optimales de knockdown par essais et erreurs. Cela augmente considérablement la probabilité que les gènes cibles d’intérêt puissent être étudiés à l’aide de cette technique.
En résumé, il a été constaté que l’administration orale d’ARNi aux moustiques adultes peut être simple, polyvalente et une approche puissante pour étudier la fonction des gènes des moustiques et pour la création d’outils nouveaux et malléables pour la lutte antivectorielle des maladies transmises par les moustiques.
Déclarations de divulgation
Les auteurs signalent qu’ils n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Les auteurs tiennent à remercier le personnel et les scientifiques de la Direction de l’entomologie et de la Division des maladies parasitaires et du paludisme du CDC, ainsi que Brian Trigg et Michelle Chiu pour leur aide à la préparation des bactéries à JHU et / ou des discussions utiles sur ce travail. Nous remercions l’insectaire JHMRI et le directeur Chris Kizito pour l’accès et l’élevage des moustiques An. gambiae . Nous remercions Wei Huang (JHSPH) pour son aide dans l’obtention des plasmides PJet GFP et pPB47 GFP pour une utilisation dans cette étude. Le financement de ce travail a été fourni par: NIH R21AI153588 (à DJA), une bourse postdoctorale du Johns Hopkins Malaria Research Institute (à MW); et par une subvention de la Good Ventures Foundation et de l’Open Philanthropy Project à la CDC Foundation intitulée Support cryopreservation and suppression of female development in mosquitoes to help research for malaria, Open Philanthropy Project, 2017. Nous apprécions profondément l’aide du personnel du JHU Microscope Facility et le soutien de subvention applicable des NIH pour le microscope utilisé (NIH Grant #: S10OD016374). Les résultats et conclusions de ce manuscrit sont ceux des auteurs et ne représentent pas nécessairement les points de vue du CDC. L’utilisation de noms commerciaux est uniquement à des fins d’identification et n’implique pas l’approbation des Centers for Disease Control and Prevention, du Public Health Service ou du département américain de la Santé et des Services sociaux.
matériels
| Name | Company | Catalog Number | Comments |
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | |
| 2x Yeast Extract Tryptone (2xYT) Medium | BD Difco | DF0440-17 | |
| AAPP | n/a | n/a | Antisera. 1:50 dilution (rabbit); gift from Fabrizio Lombardo |
| AccuStart II PCR Supermix | Quantabio | 95137-100 | |
| Agarose | Millipore Sigma | A9539 | |
| Ampicillin | Millipore Sigma | A5354 | |
| Anopheles gambiae G3 | BioDefense and Emerging Infections (BEI) Malaria Research and Reference Reagent Resource Center (MR4) | MRA-112 | |
| BugDorm | BioQuip | 1452 | |
| Centrifuge 5810R | Eppendorf | P022628181 | |
| CrebA | DSHB | CrebA Rbt-PC | Antisera. 1:50 dilution (rabbit); generated by the Andrew Lab |
| Damiens diet | BioServ | ||
| DAPI | Life Technologies | n/a | 4′,6-diamidino-2-phenylindole; 1:200 dilution. |
| Defibrinated sheep blood | HemoStat | DSB050 | |
| Escherichia coli HT115 (DE3) | |||
| Ethidium bromide | Millipore Sigma | E7637 | |
| High-Capacity cDNA Reverse Transcription Kit | Thermo Fisher Scientific | 4368814 | |
| Isopropyl β-D-1-thiogalactopyranoside | Millipore Sigma | I5502 | |
| JM109 Competent cells | Promega | L2005 | |
| Luria Broth Media | Thermo Fisher Scientific | 10855001 | |
| Mucin 2 | Proteintech | Muc2; 27 675-1-AP | Antisera. 1:100 dilution (mouse). |
| Nanodrop 2000 | Thermo Fisher Scientific | ||
| Nile Red | Sigma | n/a | Lipid dye; 1:50 dilution. |
| Owl EasyCast B2 Mini Gel Horizontal Electrophoresis | Thermo Fisher Scientific | Model B2 | |
| pGEMT easy | Promega | A3600 | |
| Power SYBR-green PCR master MIX | Applied Biosystems | 4367659 | |
| PureLink PCR purification kit | Thermo Fisher Scientific | K31001 | |
| QuantaStudio 6 | Applied Biosystems | ||
| QuantStudio6 Real Time PCR System | Applied Biosystems | ||
| Rab11 | n/a | n/a | Antisera. 1:100 dilution (rabbit); generated by the Andrew Lab |
| Rh-WGA | Vector Labs | n/a | Rhodamine-conjugated wheat germ agglutinin (chitin, O-GlcNAcylation dye); 1:40 dilution |
| Sage | n/a | n/a | Antisera. 1:50 dilution (rat); generated by the Andrew Lab |
| T4 DNA ligase | Promega | M1801 | |
| Tetracycline | Millipore Sigma | 87128 | |
| Trizol | Thermo Fisher Scientific | 15596018 | |
| Zeiss LSM700 fluorescence confocal microscope | Zeiss | ||
| ANTIBODIES | |||
| Chicken anti-Rat IgG (H+L), Alexa Fluor 647 | Thermo Fisher Scientific | A21472 | |
| Goat anti-Mouse IgG (H+L), Alexa Fluor 647 | Thermo Fisher Scientific | A28181 | |
| IgG (H+L) Goat anti-Rabbit, Alexa Fluor 488 | Thermo Fisher Scientific | A27034 | |
| Rabbit anti-Goat IgG (H+L), Alexa Fluor 488 | Thermo Fisher Scientific | A27012 | |
| PRIMERS | |||
| ACT-2f: TACAACTCGATCATGAAGTGCGA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| ACT-3r: CCCGGGTACATGGTGGTACCGC CGGA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| FKH_RNAi_F: GCCGACTTATGCTTAGCCCA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| FKH_RNAi_R: TAGCCGTCAATTCCTCCTGC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| newDSX-f: AGAGGGCGGGGAAATTCTAGT | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| newDSX-r: GGGCTTGTGGCAGTACGAATA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| S7qf1: AGAACCAGCAGACCACCATC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| S7qr1: GCTGCAAACTTCGGCTATTC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
Références
- Hoa, N. T., Keene, K. M., Olson, K. E., Zheng, L. Characterization of RNA interference in an Anopheles gambiae cell line. Insect Biochemistry and Molecular Biology. 33, 949-957 (2003).
- Caplen, N., Zheng, Z., Falgout, B., Morgan, R. Inhibition of viral gene expression and replication in mosquito cells by dsRNA-triggered RNA interference | Elsevier enhanced reader. Molecular Therapy. 6, 243-251 (2002).
- Brown, A. E., Catteruccia, F. Toward silencing the burden of malaria: progress and prospects for RNAi-based approaches. BioTechniques. , 38-44 (2006).
- Airs, P. M., Bartholomay, L. C. RNA interference for mosquito and mosquito-borne disease control. Insects. 8, (2017).
- Blandin, S., et al. Reverse genetics in the mosquito Anopheles gambiae: targeted disruption of the Defensin gene. EMBO Reports. 3 (9), 852-856 (2002).
- Garver, L., Dimopoulos, G. Protocol for RNAi assays in adult mosquitoes (A. gambiae). Journal of Visualized Experiments: JoVE. (5), e230 (2007).
- Whyard, S., et al. Silencing the buzz: a new approach to population suppression of mosquitoes by feeding larvae double-stranded RNAs. Parasites & Vectors. 8, 96 (2015).
- Wiltshire, R. M., Duman-Scheel, M. Advances in oral RNAi for disease vector mosquito research and control. Current Opinion in Insect Science. 40, 18-23 (2020).
- Taracena, M. L., Hunt, C. M., Benedict, M. Q., Pennington, P. M., Dotson, E. M. Downregulation of female doublesex expression by oral-mediated RNA interference reduces number and fitness of Anopheles gambiae adult females. Parasites & Vectors. 12, 170 (2019).
- Grassi, B. Studi di uno zoologo sulla malaria. Real Accademia dei Lincei. 3, 229 (1901).
- Smith, R. C., Jacobs-lorena, M. Plasmodium - Mosquito interactions: A tale of roadblocks and detours. Advances in Insect Physiology. 39, (2010).
- Das, S., et al. Transcriptomic and functional analysis of the Anopheles gambiae salivary gland in relation to blood feeding. BMC Genomics. 11, 1-14 (2010).
- Francischetti, I. M. B., Valenzuela, J. G., Pham, V. M., Garfield, M. K., Ribeiro, J. M. C. Toward a catalog for the transcripts and proteins (sialome) from the salivary gland of the malaria vector Anopheles gambiae. Journal of Experimental Biology. 205, 2429-2451 (2002).
- Henderson, K. D., Isaac, D. D., Andrew, D. J. Cell fate specification in thedrosophila salivary gland: The integration of homeotic gene function with the DPP signaling cascade. Developmental Biology. 205, 10-21 (1999).
- Mach, V., Ohno, K., Kokubo, H., Suzuki, Y. The Drosophila fork head factor directly controls larval salivary gland-specific expression of the glue protein gene Sgs3. Nucleic Acids Research. 24 (12), 2387-2394 (1996).
- Weiserova, M., et al. Mini-Mu transposition of bacterial genes on the transmissible plasmid. Folia Microbiologica. 32 (5), 368-375 (1987).
- Abrams, E. W., Mihoulides, W. K., Andrew, D. J. Fork head and Sage maintain a uniform and patent salivary gland lumen through regulation of two downstream target genes, PH4αSG1 and PH4αSG2. Development. 133, 3517-3527 (2006).
- Myat, M. M., Isaac, P. P., Andrew, D. J. Early genes required for salivary gland fate determination and morphogenesis in Drosophila melanogaster. Advances in Dental Research. 14, 89-98 (2000).
- Fox, R. M., Vaishnavi, A., Maruyama, R., Andrew, D. J. Organ-specific gene expression: the bHLH protein Sage provides tissue specificity to Drosophila FoxA. Development of Cell Biology. 140, 2160-2171 (2013).
- Maruyama, R., Grevengoed, E., Stempniewicz, P., Andrew, D. J. Genome-wide analysis reveals a major role in cell fate maintenance and an unexpected role in endoreduplication for the Drosophila FoxA gene fork head. PLOS ONE. 6, 20901 (2011).
- Johnson, D. M., et al. CrebA increases secretory capacity through direct transcriptional regulation of the secretory machinery, a subset of secretory cargo, and other key regulators. Traffic. 21, 560-577 (2020).
- Fox, R. M., Hanlon, C. D., Andrew, D. J. The CrebA/Creb3-like transcription factors are major and direct regulators of secretory capacity. Journal of Cell Biology. 191, 479-492 (2010).
- Abrams, E. W., Andrew, D. J. CrebA regulates secretory activity in the Drosophila salivary gland and epidermis. Development. 132, 2743-2758 (2005).
- Wells, M. B., Andrew, D. J. Anopheles salivary gland architecture shapes plasmodium sporozoite availability for transmission. mBio. 10 (4), 01238 (2019).
- Pei-Wen, L., Xiao-Cong, L., Jin-Bao, G., Yan, L., Xiao-Guang, C. Molecular cloning, characterization and expression analysis of sex determiantion gene doublesex from Anopheles gambiae (Diptera: Culicidae). Acta Entomologica Sinica. 58 (2), 122-131 (2015).
- Scali, C., Catteruccia, F., Li, Q., Crisanti, A. Identification of sex-specific transcripts of the Anopheles gambiae doublesex gene. The Journal of Experimental Biology. 208, 3701-3709 (2005).
- Price, D. C., Egizi, A., Fonseca, D. M. Characterization of the doublesex gene within the Culex pipiens complex suggests regulatory plasticity at the base of the mosquito sex determination cascade. BMC Evolutionary Biology. 15, 1-13 (2015).
- Mysore, K., et al. siRNA-mediated silencing of doublesex during female development of the dengue vector mosquito Aedes aegypti. PLoS Neglected Tropical Diseases. 9, 1-21 (2015).
- Boisson, B., et al. Gene silencing in mosquito salivary glands by RNAi. FEBS Letters. 580, 1988-1992 (2006).
- Horn, T., Boutros, M. E-RNAi: a web application for the multi-species design of RNAi reagents-2010 update. Nucleic Acids Research. 38, 332-339 (2010).
- Taracena, M. L., et al. Genetically modifying the insect gut microbiota to control chagas disease vectors through systemic RNAi. PLoS Neglected Tropical Diseases. 9, (2015).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular Cloning: A Laboratory Manual. , (1989).
- Ullmann, A., Jacob, F., Monod, J. Characterization by in vitro complementation of a peptide corresponding to an operator-proximal segment of the β-galactosidase structural gene of Escherichia coli. Journal of Molecular Biology. 24, 339-343 (1967).
- Timmons, L. Bacteria-mediated RNAi-General outline. Carnegie Institution of Washington. , (2000).
- Pfaffl, M. W. Relative quantification. Real-time PCR. , 63-82 (2004).
- Neira-Oviedo, M., et al. The RNA-Seq approach to studying the expression of mosquito mitochondrial genes. Insect Molecular Biology. 20, 141-152 (2011).
- Baker, D. A., et al. A comprehensive gene expression atlas of sex- and tissue-specificity in the malaria vector, Anopheles gambiae. BMC Genomics. 12, (2011).
- Loganathan, R., Hoon, J., Wells, M. B., Andrew, D. J. Secrets of secretion - How studies of the Drosophila salivary gland have informed our understanding of the cellular networks underlying secretory organ form and function. Cellular Networks in Development. , 143 (2021).
- Chung, S., Hanlon, C. D., Andrew, D. J. Building and specializing epithelial tubular organs: The Drosophila salivary gland as a model system for revealing how epithelial organs are specified, form and specialize. Wiley Interdisciplinary Reviews: Developmental Biology. 3, 281-300 (2014).
- Yoshida, S., et al. Inhibition of collagen-induced platelet aggregation by anopheline antiplatelet protein, a saliva protein from a malaria vector mosquito. Blood. 111, 2007-2014 (2008).
- Wells, M. B., Villamor, J., Andrew, D. J. Salivary gland maturation and duct formation in the African malaria mosquito Anopheles gambiae. Scientific Reports. 7 (1), 601 (2017).
- Takahashi, S., et al. Rab11 regulates exocytosis of recycling vesicles at the plasma membrane. Journal of Cell Science. 125, 4049-4057 (2012).
- Catteruccia, F., Levashina, E. A. RNAi in the malaria vector, Anopheles gambiae. Methods in Molecular Biology. 555, 63-75 (2009).
- Balakrishna Pillai, A., et al. RNA interference in mosquito: understanding immune responses, double-stranded RNA delivery systems and potential applications in vector control. Insect Molecular Biology. 26, 127-139 (2017).
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30, 313-321 (2003).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421, 231-237 (2003).
- Carrasco, D., et al. Behavioural adaptations of mosquito vectors to insecticide control. Current Opinion in Insect Science. 34, 48-54 (2019).
- Magalhaes, T., et al. Induction of RNA interference to block Zika virus replication and transmission in the mosquito Aedes aegypti. Insect Biochemistry and Molecular Biology. , 111 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon