Method Article
Administração eficaz de interferência oral de RNA (RNAi) para mosquitos anofeeles gâmbias adultos
Neste Artigo
Resumo
A administração oral do dsRNA produzido por bactérias, um método de entrega para interferência de RNA (RNAi) que é rotineiramente utilizado em caenorhabditis elegans, foi aplicada com sucesso aqui em mosquitos adultos. Nosso método permite estudos robustos de genética reversa e estudos de vetores de bloqueio de transmissão sem o uso de injeção.
Resumo
A interferência do RNA tem sido uma ferramenta fortemente utilizada para análise genética reversa por duas décadas. Em mosquitos adultos, a administração de RNA (dsRNA) de dupla encalhada tem sido realizada principalmente por injeção, o que requer tempo significativo e não é adequado para aplicações de campo. Para superar essas limitações, aqui apresentamos um método mais eficiente para ativação robusta do RNAi por meio da entrega oral do dsRNA para anofeeles gâmbiae adulto. DsRNAs longas foram produzidas na cepa Escherichia coli HT115 (DE3), e uma suspensão concentrada de bactérias contendo dsRNA em 10% de sacarose foi oferecida em bolas de algodão ad-libitum para mosquitos adultos. Bolas de algodão foram substituídas a cada 2 dias durante o tratamento. O uso deste método para atingir o doublesex (gene envolvido na diferenciação de sexo) ou a cabeça de garfo (que codifica um fator de transcrição da glândula salivar) resultou em uma expressão genética alvo reduzida e/ou sinal de imunofluorescência proteica, medida por PCR quantitativo em Tempo Real (qRT-PCR) ou microscopia confocal de fluorescência, respectivamente. Também foram observados defeitos na morfologia das glândulas salivares. Este método altamente flexível, fácil de usar, de baixo custo e eficiente de entrega de dsRNA pode ser amplamente aplicável a genes-alvo importantes para a fisiologia vetorial de insetos e além.
Introdução
Muitas doenças são transmitidas por mosquitos, tornando o estudo da fisiologia do mosquito e da genética um importante empreendimento. O uso de RNAi nesses organismos tem sido proeminente nos últimos 20 anos e permitiu a caracterização funcional de muitos genes de mosquitos 1,2,3,4,5. A técnica mais utilizada para a entrega de dsRNA tem sido a microinjeção, que tem as desvantagens de que pode ferir os mosquitos e requer tempo e esforço significativos. Foram testados métodos de entrega oral para RNAi, mas principalmente na fase larval dos mosquitos 6,7,8,9. A entrega oral de dsRNA em mosquitos adultos não foi totalmente explorada e pode ser uma ferramenta útil para o estudo da biologia vetorial e do controle vetorial.
A malária é transmitida por mosquitos Anopheles quando um mosquito fêmea infectado toma uma refeição sanguínea de um hospedeiro não infectado e injeta saliva contendo parasitas maláricos10. Para, em última análise, ser transmitido na saliva de um mosquito, o parasita deve superar muitos obstáculos, incluindo a fuga do sistema imunológico do mosquito, a travessia da barreira do meio do bico e a invasão das glândulas salivares11. A arquitetura da glândula salivar de mosquitos (SG) é a chave para a invasão de parasitas e que a arquitetura é controlada tanto por fatores de transcrição expressos na glândula salivar, como por determinantes do dimorfismo sexual. Vários fatores de transcrição altamente conservados são necessários para especificação celular e manutenção homeostática das glândulas salivares e para a produção e secreção de proteínas salivares que funcionam na alimentação sanguínea 12,13,14. Fork head (Fkh) é um fator de transcrição de hélice alada que funciona como um dos principais reguladores da estrutura e função de SG de insetos (baseado em estudos em moscas de frutas e na mariposa de bicho-da-seda)15,16,17,18,19,20. Nos SGs de Drosophila, Fkh funciona com Sage, um fator de transcrição de helix-helix -helix (bHLH) específico da SG, para promover a sobrevivência de SG e a produção de saliva19. Um importante e positivo co-regulador da produção de saliva em Drosophila é o CrebA, um fator de transcrição de zíper de leucina bem estudado que regula a expressão dos genes de viasecretary 21,22,23. Há também um forte grau de diferenciação morfológica nas glândulas salivares femininas que provavelmente desempenha um papel fundamental, não apenas na alimentação sanguínea, mas também na capacidade dos parasitas de invadir esse tecido24.
Muitos dos genes envolvidos na determinação da sobrevivência da glândula salivar, estrutura, fisioterápria e dimorfismo sexual têm perfis complexos de expressão espostetorial 25,26,27, e os métodos tradicionais de entrega do dsRNA para induzir o RNAi nem sempre são eficientes em direcionar esses tipos de genes neste ou em outros tecidos. No entanto, a entrega oral de dsRNA no estágio larval dos mosquitos Aedes aegypti e An. gambiae tem sido usada com sucesso para silenciar a forma específica feminina do gene dsx 9,28. Estudos anteriores utilizando dsRNA em glândulas salivares de mosquitos descobriram que, embora grandes quantidades de dsRNA fossem necessárias, o efeito de silenciamento era relativamente duradouro (pelo menos 13 dias)29. Aqui, foi testada a capacidade da cepa E. coli HT115 (DE3) expressando dsRNA específico de sequência para dsx, fkh ou CrebA para induzir o silenciamento RNAi desses genes em mosquitos fêmeas adultas. Administração oral de dsRNA induzida knockdown genético em An. gambiae, com reduções claras nos níveis de mRNA e com fenótipos consistentes com a perda de função desses genes. Assim, essa abordagem provavelmente trabalhará para derrubar a função de uma variedade de genes da glândula salivar.
Protocolo
1. Clonagem dsRNA em vetor de expressão E. coli
- Selecione a sequência de genes de destino para inserir em um vetor apropriado para a expressão de dsRNA. Recupere os valores de expressão de Vectorbase.org usando o seguinte método.
- Procure por um gene de interesse (por exemplo, Tabela 1) na caixa de pesquisa da página inicial.
- Na página genética resultante, navegue até o 8. Seção de transcrição .
- Procure os experimentos relevantes de expressão genética RNA-seq e microarray listados.
- Transcreva valores de interesse no software de planilha e crie uma tabela de dados.
- Selecione um plasmid comercialmente disponível com pelo menos um promotor T7 a ser usado. Se o plasmídeo selecionado tiver apenas um promotor T7 (como a maioria dos plasmídeos comerciais fazem), inclua um segundo promotor T7 na cartilha reversa a ser usada para a amplificação do dsDNA para o gene de interesse.
NOTA: A sequência dsRNA para os genes alvo pode ser selecionada usando o aplicativo web E-RNAi para o design dos reagentes RNAi30. Ou dsRNA longo (aproximadamente 400 bp) ou dsRNA de pino curto (shRNA) podem ser projetados com base em sequências genéticas específicas. Essas sequências devem ser amplificadas e sequenciadas para confirmação de identidade antes da clonagem. As regiões genéticas selecionadas, plasmídeos e promotores utilizados neste estudo estão listados no Arquivo Suplementar 1. - Realizar clonagem de acordo com um procedimento simples de uma etapa descrito anteriormente 9,31. Para isso, purifique o produto PCR e ligate ao DNA plasmídeo linearizado. Use o produto da ligadura para a transformação de choque térmico das células E. coli competentes32. Selecione as células transformadas através da triagem azul/branco. Confirme a orientação da inserção usando um PCR T7 primer e confirme a sequência usando primers M13.
NOTA: As triagens brancas/azuis podem ser usadas quando o plasmídeo selecionado para transformação carrega o gene lacZ que codifica para β-galactosidase. As colônias brancas devem conter a inserção desejada dentro do lacZ e podem ser selecionadas para confirmar ainda mais a presença e orientação da sequênciade destino 33. - Purifique o plasmídeo da primeira transformação e use-o para transformar o competente E. coli HT115 (DE3) como descrito anteriormente34. Após a confirmação de que o plasmídeo com a inserção está presente no competente E. coli HT115 (DE3), fazer estoques de glicerol de bactérias para uso único.
NOTA: Um dsRNA de controle não relacionado apropriado deve ser adquirido ou preparado para uso em todos os experimentos. Neste caso, a sequência para o gene aintegumenta não relacionado (formiga) de Arabidopsis thaliana é usada.
2. Preparação de bactérias mortas pelo calor expressando dsRNA
- Cresça uma cultura de uma única colônia bacteriana de E. coli strain HT115 (DE3) contendo o dsRNA expressando plasmídeo em 50 mL de Caldo de Luria (LB) contendo 100 μg/mL de ampicillina e 12,5 μg/mL de tetraciclina, em um agitador de plataforma (180 rpm) a 37 °C para 12 h.
- Diluir a cultura bacteriana (1:1000) em 2x levedura triptona (2x YT) mídia contendo 100 μg/mL de ampicillina e 12,5 μg/mL de tetraciclina.
- Induzir a produção de dsRNA adicionando 40 μM (concentração final) isopropílico β-D-1-thiogalactopyranoside (IPTG).
- Quando as células atingirem um O.D.600 = 0,4, aproximadamente após 2h de indução a 37 °C com agitação a 180 rpm, prepare uma suspensão concentrada de bactérias mortas pelo calor, como descrito por Taracena et al 9. Pelotar as células por centrifugação (4000 x g, 4 °C, 10 min) e lavar células em um volume de tampão fosfato de sódio (PBS).
- Gire novamente sob as mesmas condições, suspenda novamente na PBS para 1/100 do volume inicial e coloque a 70 °C por 1h.
- Faça 400 alíquotas de μL das bactérias mortas pelo calor e armazene essas alíquotas a -20 °C até que usem mais (não guarde por mais de uma semana). Esta suspensão de bactérias mortas pelo calor contém o dsRNA específico para os experimentos RNAi. Realize este procedimento tanto para o gene-alvo dsRNA-bacteria quanto para que o controle dsRNA não relacionado seja usado em cada experimento.
3. Alimentar mosquitos com bactérias mortas pelo calor expressando dsRNA
- Descongele uma alíquota de suspensão de bactérias dsRNA (HT115 (DE3) e misture com 1,6 mL de solução de açúcar de 12% contendo 0,2% de metilparaben.
- Mergulhe uma pequena bola de algodão nesta solução e coloque a bola de algodão encharcada dentro de uma gaiola contendo mosquitos de 5 dias de idade. Certifique-se de que os mosquitos se alimentam dessa solução, pegando simultaneamente o açúcar e as bactérias contendo dsRNA.
- Troque a bola de algodão encharcada em solução de açúcar dsRNA a cada dois dias por 8 dias consecutivos.
- Mantenha as gaiolas do mosquito em condições constantes, ou seja, 27 °C e 80% de umidade relativa com um fotoperodo de 12 h:12 h luz: fotociclo escuro, separado por um amanhecer de 30 min e 30 min de anoitecer.
4. Ensaio de níveis de expressão genética de alvo
- Anestesize os mosquitos colocando o recipiente no gelo por um minuto ou até que os mosquitos parem de se mover. Uma vez que os mosquitos são anestesiados, coloque-os em uma superfície fria para isolar as fêmeas para dissecção.
- Pulverize 70% de etanol para os mosquitos e coloque-os em uma superfície de vidro com PBS. Com um par de fórceps, fixar a cabeça do mosquito firme e puxar o tórax muito lentamente, permitindo que as glândulas salivares sejam liberadas na PBS.
- Mantenha as glândulas salivares em PBS gelada até que 10 indivíduos tenham sido dissecados. Pool Dez OGs para extração de RNA usando o método guanidinium tiocianato-fenol-clorofórmio. Suspenda a pelota de RNA em 30 μL de água sem RNase.
- Use 1 μL aliquot do RNA extraído do SG na etapa anterior, para ler a absorvência em 260 e 280 nm e calcular a concentração de RNA de cada amostra multiplicando-se com o fator de diluição. Uma razão de 260/280 de ~2.0 indica RNA de boa qualidade.
- Use 1 μg do RNA purificado para sintetizar DNA complementar (cDNA) usando um kit de transcrição reversa comercial.
- Faça uma diluição de 1:10 do cDNA para preparar uma reação RT-PCR de acordo com as recomendações do fabricante. Para cada amostra, prepare uma reação para o gene alvo e, em paralelo, configure uma reação com o gene de limpeza (HK). Defina cada reação genética em um triplicado técnico para eliminar o impacto da variação aleatória do método.
NOTA: Aqui, o gene An. gambiae ribosomal S7 (GeneBank: L20837.1) e actin (VectorBase: AGAP000651) têm sido usados como genes HK. - Use todos os primers em uma concentração final de 300 nM, seguindo as indicações do fabricante verde-SYBR. Amplie com condições padrão de PCR: 95 °C para 10 min, seguido por 40 ciclos de 15 s a 95 °C e 60 s a 60 °C.
NOTA: Para quantificar a expressão genética, o método delta-delta-ct (ΔΔCt) é usado. Delta Ct (ΔCt) é a diferença entre o TC do gene alvo e o TC do gene de limpeza. ΔΔCt é a diferença entre o ΔCt do grupo experimental e o ΔCt do grupo de controle35.
5. Avaliação fenotípica: alimentação sanguínea bem sucedida
- Para avaliar a capacidade de alimentação sanguínea, configure grupos de 15 mosquitos fêmeas tratados com alvo e controle dsRNA em gaiolas pequenas (12 cm de diâmetro) e esflam fome por 4h.
- Utilizando um banho de água circulante de 37 °C, alimentadores de mosquitos de vidro (24 mm de diâmetro) e membrana parafilm, oferecem sangue desfibriado para os mosquitos.
NOTA: O sangue pode ser adquirido de um fornecedor comercial que o atrai de animais saudáveis e doadores de origem americana e desfibrila manualmente sem anticoagulantes ou aditivos. - Por observação direta, conte e regise o número de tentativas de sondagem para adquirir com sucesso uma refeição sanguínea das cinco primeiras fêmeas para se tornar totalmente engorgado em cada grupo.
NOTA: Para evitar mudanças metabólicas significativas nos mosquitos, que poderiam interferir com recursos energéticos que afetam o comportamento de busca de sangue, a fome foi mantida ao mínimo (4h). Como resultado, nem todo mosquito procuraria avidamente a refeição sanguínea e limitamos a contagem das fêmeas engorgadas a cinco (um terço do total de cada grupo), para reduzir o efeito de variáveis de tempo como exposição ao odor humano, mudança de temperatura entre as câmaras e as superfícies de alimentação, etc.
6. Avaliação fenotípica: Morfologia das glândulas salivares e baixa regulação de proteínas relevantes
- Isole o tecido fresco em 1x salina tamponada com fosfato (PBS) conforme descrito na etapa 4.2 e fixe em acetona gelada para 90 s. Enxágüe várias vezes em 1x PBS depois de remover a acetona. Incubar com anticorpos primários durante a noite a 4 °C com antiserum (ver Tabela de Materiais) diluído em 1x PBS.
NOTA: Ver Tabela de Materiais para identificação dos anticorpos primários utilizados para proteínas salivares (proteína anti plaqueta Anopheles , AAPP; Mucin 2, MUC2), fatores de transcrição de SG (Fork Head, fkh; Sábio, sábio; Proteína de ligação de elementos de resposta cíclica-AMP A, CrebA), e um marcador de vesículas secretas (Rab11). Estes anticorpos são usados como leituras para forma e função SG. No entanto, qualquer anticorpo adequado para imunofluorescência deve ser adequado para este protocolo. - Lave em 1x PBS várias vezes. Adicione anticorpos secundários (fluorescentes) diluídos em 1x PBS, e incubar no escuro à temperatura ambiente por 2 h. Adicione qualquer contra-retenção [como 4′,6-diamidino-2-fenilôdole (DAPI; DNA), agglutinina de germe de trigo (WGA; para quitina), fhalloidina (para F-actin) e/ou Vermelho Nilo (para lipídios)] 30 min antes do fim da incubação de 2 h.
- Lave três vezes em 1x PBS. Em seguida, monte os tecidos em 100% glicerol em um slide de microscópio padrão com uma tampa de 1 mm de espessura e armazene a -20 °C até a imagem usando um microscópio confocal de fluorescência.
NOTA: Para obter dados quantitativos, as configurações de imagem devem ser mantidas constantes. Aqui, apenas imagens de projeção de intensidade máxima através de todo o volume 3D do tecido foram incluídas, e toda a quantificação de imagem foi normalizada entre os tratamentos (dentro de um experimento) com base no sinal DAPI em restos de tecido não-SG (corpo de gordura, cutícula ou cabeça) também presentes no slide.
Resultados
Para começar, os dados de expressão de microarray do VectorBase foram usados para digitalizar alvos potenciais em estágiosde desenvolvimento 36,37 para determinar o status de expressão de todos os genes relevantes para o estudo atual (Tabela 1). Como esperado, todos os nossos genes-alvo escolhidos mostraram expressão em SGs adultos. Os níveis de aapp e sálvia foram particularmente altos (Tabela 1). Também foram observados os altos níveis de expressão de f-Agdsx em SGs mulheres adultas9.
Segmentos específicos de cada gene foram avaliados para uso como dsRNA utilizando o aplicativo web E-RNAi para o projeto de reagentes RNAi30. As regiões de ~400 bp contendo sequências exclusivas de cada gene alvo foram então clonadas (Figura 1A), transformadas nas cepas bacterianas apropriadas, e usadas para preparar suspensões de bactérias mortas pelo calor, que foram induzidas a produzir dsRNA. Mosquitos adultos foram alimentados por 8 dias nas bolas de algodão encharcadas de sacarose contendo as suspensões bacterianas de dsRNA para f-Agdsx,fkh ou formiga (o controle negativo não relacionado).
Para a análise da alimentação RNAi de mosquitos fêmeas, foi primeiro determinado se f-Agdsx ou fkh dsRNA-feedings induziram silenciamento genético. Uma redução de 98,8% (±2,1) nos níveis de transcrição fkh foi observada no grupo alimentado com fkh-dsRNA (Figura 1B), indicando que o dsRNA reduziu muito efetivamente a abundância de transcrições fkh em SGs. Surpreendentemente, os níveis de fkh mRNA foram reduzidos em 82,0% (±18,9) nos mosquitos tratados com dsRNA para f-Agdsx, que tiveram 89,86% (±4,48) de redução f-Agdsx , sugerindo que fkh poderia ser um alvo de F-Dsx na glândula salivar. Concomitante com a redução significativa dos níveis de expressão fkh , os mosquitos fkh-knockdown apresentaram um aumento significativo no número de tentativas de sondagem necessárias para alimentar o sangue. Esses mosquitos apresentaram, em média, cinco vezes mais tentativas de alimentação do que o grupo controle ou f-Agdsx dsRNA alimentado por mosquitos para serem completamente engorgados com sangue (Figura 1C). Isso levou a perguntar se os tratamentos rnai fkh knockdown causaram alterações na localização e/ou distribuição dos principais reguladores transcricionais (SG TFs Sage e CrebA) (Figura 2), proteínas secretadas (AAPP e mucina) (Figura 3) e máquinas secretary [Nilo Vermelho (lipídios) e Rab11 (vesículas secretas)] (Figura 4). É importante ressaltar que diferenças substanciais na intensidade das manchas foram observadas em diferentes regiões do lobo, lóbulos e SGs individuais.
Como previsto, os níveis de sálvia e mancha de CrebA foram significativamente reduzidos em todos os lóbulos de SG após fkh RNAi (Figura 2B) em comparação com o controle de formigas RNAi (Figura 2A). Reduções tanto nos maiores valores de intensidade máxima (linhas vermelhas e rótulos numéricos) quanto nos menores valores de intensidade máxima (linhas de traço azul e etiquetas numéricas) em perfis de varredura de linha sugeriram reduções em áreas de sinal alto e baixo dentro do tecido (Figuras 2A,B). Esses dados sugerem que An. gambiae fkh RNAi é eficaz e que fkh regula a produção e/ou estabilidade dos SG TFs Sage e CrebA em An. gambiae, análogo à sua relação genética em Drosophila SGs 19,38,39.
Quando considerados proteínas altamente abundantes do componente saliva, os níveis de proteína anti-plaqueta Anopheles (AAPP)40,41 foram reduzidos em todos os três lóbulos de SG após fkh RNAi, em comparação com o tratamento RNAi de controle (Figura 3A,B; verde). Por outro lado, não foram observadas alterações nos níveis de Mucina (Figura 3A,B; roxo). Esses dados sugerem que Fkh contribui de forma diferente para a expressão de diferentes genes de proteína saliva.
Por fim, foram observados dois marcadores de secreção (Figuras 4A,B): Rab11 (vesículas associadas a endosários de reciclagem apical)42 e Nilo Vermelho (lipídios). A fluorescência rab11 reduzida foi observada em lóbulos laterais distais (DL) após o tratamento fkh RNAi (Figura 4A v vs. 4B v; verde). No entanto, também ocorreu o aumento do sinal Rab11 nos lobos medial (M) e proximal lateral (PL) (Figura 4A vii, ix vs. 4B vii, ix; verde). Não foi observada diferença perceptível no sinal vermelho do Nilo (Figuras 4A,B; roxo) após fkh RNAi em comparação com o tratamento RNAi de controle. Esses dados sugerem que a redução de fkh pode alterar alguma ação de máquinas secretas de forma complexa que difere entre os lóbulos SG.
| Dataset: | Goltsev | Neira Oviedo | Neira Oviedo | Padeiro | Padeiro | Padeiro | Padeiro | ||
| símbolo genético | função | AGAP ID | embrião (25 horas.) | Larvas L3 | L3 SG | mulher adulta corpo (3 dias) | homem adulto corpo (3 dias) | mulher adulta SG (3 dias) | homem adulto SG (3 dias) |
| AAPP | proteína saliva | AGAP009974 | 3.92 | 4.38 | 4.33 | 3.81 | 2.46 | 11.92 | 2.69 |
| CrebA | fator txn | AGAP001464 | 6.28 | 5.22 | 5.92 | 2.99 | 2.96 | 3.27 | 3.13 |
| " | fator txn | AGAP011038 | 4.50 | 4.46 | 5.23 | 2.96 | 2.86 | 3.05 | 2.88 |
| dsx | fator txn | AGAP004050 | 4.91 | 5.39 | 5.55 | 3.72 | 4.00 | 4.57 | 4.01 |
| fkh | fator txn | AGAP001671 | 5.18 | 4.67 | 5.25 | 2.99 | 3.09 | 3.21 | 3.05 |
| MUC2 | proteína saliva | AGAP012020 | 4.59 | 5.53 | 5.63 | 2.96 | 3.07 | 3.08 | 3.26 |
| Rab11 | tráfico vesicular | AGAP004559 | 10.21 | 7.47 | 8.60 | 4.90 | 3.79 | 3.38 | 2.96 |
| sálvia | fator txn | AGAP013335 | 5.32 | 5.96 | 8.89 | 3.40 | 3.33 | 7.37 | 7.23 |
Tabela 1: Perfis de expressão de microarray log2 médios para An. gambiae genes de interesse. São mostrados nomes genéticos, categoria funcional, identificadores de Vectorbase (AGAP) e dados de expressão de microarray de log2 coletados do Vectorbase. Esses dados indicam que nossos genes de interesse (envolvidos na biologia celular e secreção da glândula salivar (SG) são expressos e enriquecidos no estágio larval 3 (L3) e nos GGs adultos, em comparação com indivíduos inteiros.
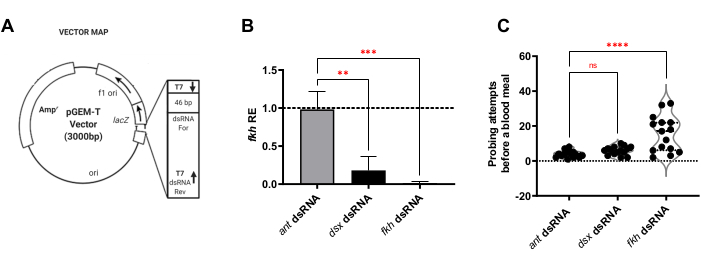
Figura 1: f-Agdsx e fkh knockdown em adulto An. gambiae reduz os níveis de fkh mRNA nos OGs e afeta a capacidade feminina de se alimentar no sangue. (A) Imagem representativa do design plasmídeo utilizado para a produção de dsRNA nesta metodologia. A segunda sequência de promotores T7 é adicionada ao plasmídeo, incluindo-o no primer de 3' usado para amplificar a inserção a ser clonada no plasmídeo pGEMT. O plasmídeo é então transformado em bactérias E. coli HT115 (DE3) e uma solução alimentar é feita de uma suspensão de bactérias induzidas pelo calor em 10% de água açucarada. (B) Os animais alimentados com uma solução de alimentação dsRNA para f-Agdsx ou fkh, apresentaram níveis significativamente mais baixos de transcrições de fkh (ANOVA unidirecional com múltiplas comparações; n=15). No entanto, apenas o grupo alimentado com fkh dsRNA (C) mostrou uma diferença significativa no número de tentativas de mordida necessárias para adquirir uma refeição de sangue. Os mosquitos deste grupo precisavam, em média, cinco vezes mais do que o número de tentativas de sondagem para obter uma refeição sanguínea bem sucedida do que o necessário pelo controle ou pelos grupos alimentados por dsx-dsRNA (ANOVA unidirecional com múltiplas comparações; n=15). As barras de erro indicam o Erro Padrão da Média (SEM). Cada experimento foi conduzido em três réplicas biológicas separadas. Clique aqui para ver uma versão maior desta figura.
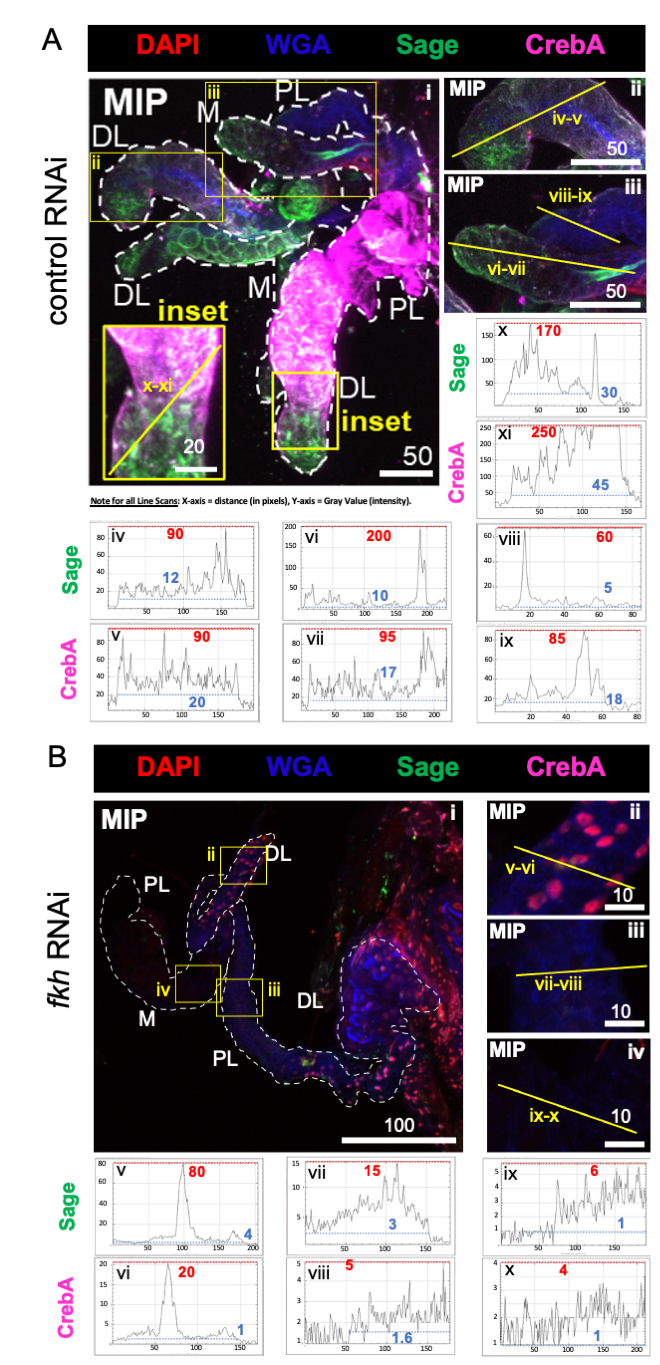
Figura 2: fkh knockdown em glândulas salivares an . gambiae adulto reduz os níveis de fator de transcrição SG. São mostradas imagens representativas do dia 13 fêmeas an . gambiae após 8 dias (dias 5-13) de exposição oral a (A) controle dsRNA não relacionado (formiga) ou (B) dsRNA visando a cabeça de garfo SG TF (fkh, AGAP001671) em 10% de sacarose manchada com os corantes DAPI (DNA; vermelho), aglutinina de germe de trigo rotulada (WGA, chitin/ O-GlcNAcylation; azul), antisera contra os SG TFs Sage (verde) e CrebA (roxo). Os comprimentos da barra de escala mostrados são mícrons. Os SGs (i) são delineados com traços brancos. Linhas amarelas em imagens de lobos ampliados (das regiões cercadas por caixas amarelas e rotuladas de "inset") indicam onde foram realizadas as varreduras de linha de intensidade do sinal. As intensidades do canal verde e roxo correspondentes às varreduras de linha para cada lobo zoom são traçadas (sempre da esquerda para a direita no SG) nos gráficos abaixo das imagens; Eixo X = distância (em pixels) e eixo Y = unidade cinza (intensidade de pixel). O alcance dinâmico da intensidade do pixel é delimitado por linhas pontilhadas vermelhas (máximas) e azuis (mínimas) e os valores correspondentes são mostrados em cada gráfico. MIP = projeção 3D de intensidade máxima através de toda a profundidade SG. DL: lobo lateral distal; M: rímbo medial; PL: lobo lateral proximal; SD: ducto salivar. Clique aqui para ver uma versão maior desta figura.
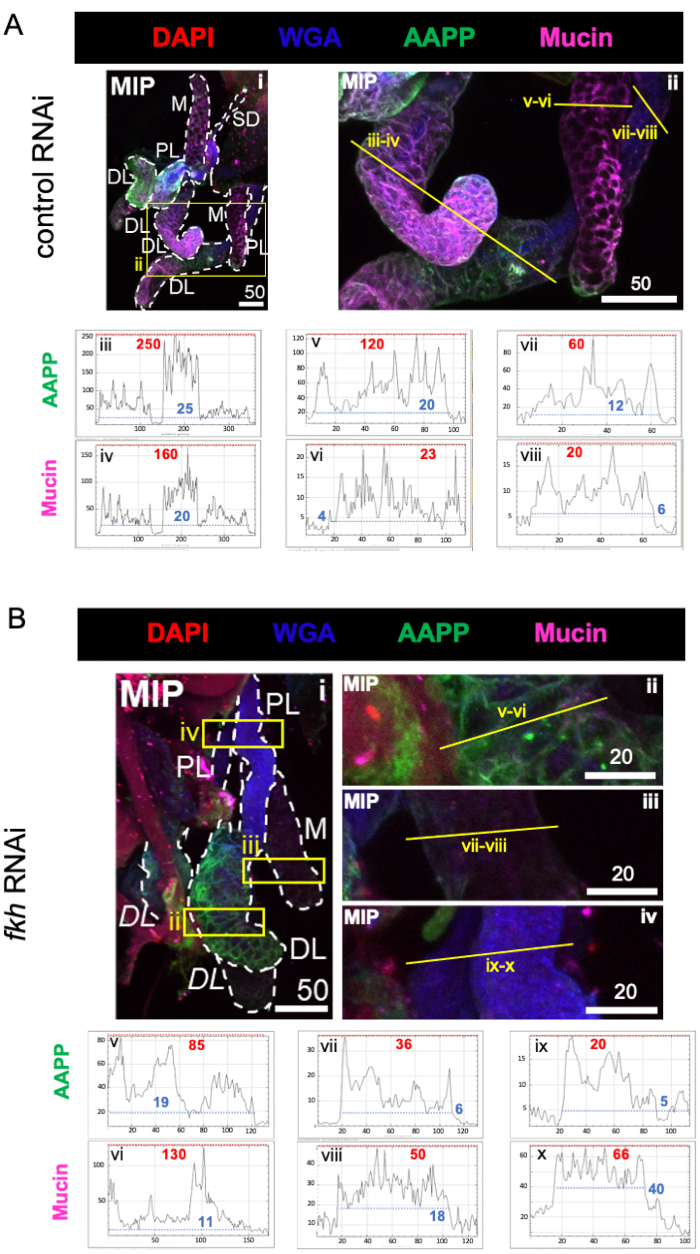
Figura 3: fkh knockdown em glândulas salivares an. gambiae adulta reduz os níveis de proteína secretado SG. São mostradas imagens representativas do dia 13 fêmeas an. gambiae após 8 dias (dias 5-13) de exposição oral a (A) controle dsRNA não relacionado (formiga), ou (B) dsRNA visando a cabeça de garfo SG TF (fkh,AGAP001671) em 10% de sacarose manchada com os corantes DAPI (DNA; vermelho), aglutina de germe de trigo rotulado (WGA, chitin/ O-GlcNAcylation; azul), e as proteínas saliva AAPP (verde) e Mucin (MUC2, roxo). Os comprimentos da barra de escala mostrados são mícrons. Os SGs (i) são delineados com traços brancos. Linhas amarelas em imagens de lobos ampliados (das regiões cercadas por caixas amarelas) indicam onde foram realizadas as varreduras de linha de intensidade do sinal. As intensidades do canal verde e roxo correspondentes às varreduras de linha para cada lobo são traçadas (sempre da esquerda para a direita no SG) nos gráficos abaixo das imagens; Eixo X = distância (em pixels) e eixo Y = unidade cinza (intensidade de pixel). O alcance dinâmico da intensidade do pixel é delimitado por linhas tracejadas vermelhas (máximas) e azuis (mínimas) e os valores correspondentes são mostrados em cada gráfico. MIP = projeção 3D de intensidade máxima através de toda a profundidade SG. DL: lobo lateral distal; M: rímbo medial; PL: lobo lateral proximal; SD: ducto salivar. Os rótulos itálicos "DL" (Bi) indicam duas regiões visíveis do mesmo lobo DL. Clique aqui para ver uma versão maior desta figura.
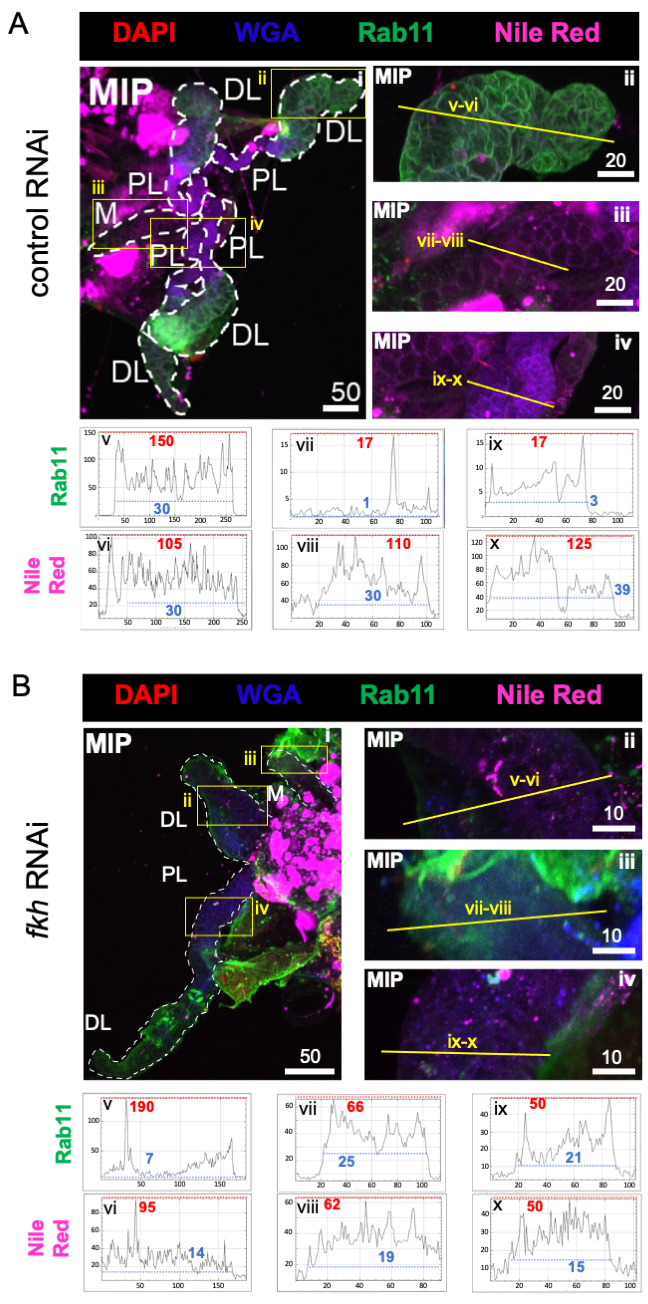
Figura 4: fkh knockdown em glândulas salivares an . gambiae adulto reduz marcadores de secreção SG. São mostradas imagens representativas do 13º dia feminino an. gambiae SGs após 8 dias (dias 5-13) de exposição oral a (A) controle dsRNA não relacionado (formiga), ou (B) dsRNA visando a cabeça de garfo SG TF (fkh, AGAP001671) em 10% de sacarose manchada com os corantes DAPI (DNA; vermelho), aglutinina de germe de trigo rotulada (WGA, chitin/ O-GlcNAcylation; azul), Vermelho-nilo (lipídios; roxo) e antisera contra o marcador de vesícula de reciclagem Rab11 (verde). Os comprimentos da barra de escala mostrados são mícrons. Os SGs (i) são delineados com traços brancos. Linhas amarelas em imagens de lobos ampliados (das regiões cercadas por caixas amarelas) indicam onde foram realizadas as varreduras de linha de intensidade do sinal. As intensidades do canal verde e roxo correspondentes às varreduras de linha para cada lobo são traçadas (sempre da esquerda para a direita no SG) nos gráficos abaixo das imagens; Eixo X = distância (em pixels) e eixo Y = unidade cinza (intensidade de pixel). O alcance dinâmico da intensidade do pixel é delimitado por linhas tracejadas vermelhas (máximas) e azuis (mínimas) e os valores correspondentes são mostrados em cada gráfico. MIP = projeção 3D de intensidade máxima através de toda a profundidade SG. DL: lobo lateral distal; M: rímbo medial; PL: lobo lateral proximal; SD: ducto salivar. Clique aqui para ver uma versão maior desta figura.
Arquivo complementar 1. Clique aqui para baixar este Arquivo.
Discussão
A capacidade de efetivamente entregar dsRNA para mosquitos An. gambiae por alimentação oral tem amplas implicações para estudos de biologia vetorial tanto no laboratório quanto no campo. A microinjeção tem sido aceita há muito tempo como o modo preferido de entrega de produtos químicos, anticorpos, RNAi e estratégias de modificação genética em mosquitos43,44. A consequência de manipulação física substancial, danos celulares e estresse pode ser evitada pelo uso de parto oral, que também pode ser potencialmente adequado para aplicações em larga escala ou de campo. Trabalhos anteriores sugerem que a RNAi age de forma onipresente dentro de um mosquito adulto individual29, permitindo efeitos em todos os tecidos, incluindo glândulas salivares. Alimentando mosquitos com um grande número de E. coli expressando dsRNA que são digeridos assíncronamente durante um longo período de tempo, pode-se potencialmente alcançar exposição consistente e uniforme ao RNAi em todos os indivíduos em uma gaiola. Este método permite alimentar um grande número de mosquitos e analisar a variabilidade potencial dos fenótipos resultantes, dependendo do gene alvo. No entanto, uma consideração importante é a possibilidade de distribuição heterogênea das bactérias e, consequentemente, dsRNA, na fibra de algodão. Os 400 μL de bactérias utilizados diariamente para a alimentação de açúcar do mosquito conteriam aproximadamente ≤ 4,6 μg de dsRNA, como descrito e calculado anteriormente9, mas a quantidade de dsRNA ingerida por cada mosquito não foi determinada individualmente. Se a construção de construções de dsRNA se tornar rotina, este simples protocolo de tratamento permite a rápida assimilação dessa técnica por qualquer pesquisador de mosquitos. A priori, o gasto de tempo durante o tratamento (30 minutos por dia) é trivial em comparação com o tempo gasto para aprender e aplicar microinjeção a tamanhos amostrais semelhantes.
A alimentação de dsRNA é rotineiramente usada para estudos de genética reversa no organismo modelo Caenorhabditis elegans45. Esse alto nível de uso ressalta o valor da abordagem de entrega oral. A construção de uma biblioteca de genoma an. gambiae em E. coli transformada, semelhante à que existe em C. elegans46,47, permitiria uma rápida triagem genética reversa em mosquitos em maior escala. No entanto, é importante notar que a eficiência do método depende em grande medida dos níveis endógenos da transcrição e se a expressão não se limita ao tecido alvo, mas expressa de forma mais ampla 4,8,44. Além disso, há evidências de que alguns inseticidas poderiam induzir a evasão comportamental dos mosquitos48, e alimentar-se com bactérias que potencialmente induzem efeitos adversos neles poderia desencadear padrões semelhantes de evasão. No ambiente controlado do laboratório, onde os mosquitos não tinham uma fonte de alimento alternativa, eles não tinham escolha de evitar a água de açúcar com E. coli e a necessidade de uma fonte nutritiva provavelmente substituiria o instinto de evitar as bactérias. No entanto, isso deve ser considerado se a estratégia deveria ser usada em ambientes menos controlados.
Pode ser possível atingir múltiplos genes simultaneamente (usando um construto, construções múltiplas ou uma mistura de isolados bacterianos transformados), mas estudos adicionais são necessários para avaliar a eficácia. Outra consideração importante para este ponto é a avaliação de possíveis efeitos fora do alvo ou sinérgicos ao usar alvos únicos ou múltiplos. O estabelecimento de genes e grupos de controle adequados é uma parte importante do projeto experimental. Além disso, é tentador especular que essa abordagem poderia ser usada para atingir outros patógenos ou vírus49. Trabalho prévio para indução rnai em mosquitos foi realizado em condições em que o reagente foi injetado diretamente, por isso E. coli não estava presente. O E. coli pode fornecer um compartimento de proteção permitindo a liberação mais lenta do dsRNA ao longo do tempo, garantindo que a exposição seja mais ou menos contínua durante um período muito mais longo29.
Finalmente, esses resultados mostram que os efeitos desta técnica são incapazes de ajustar o período de tempo (comprimento e dia de início) de exposição e a quantidade de E. coli utilizada. Esse recurso nos permitiu estudar as funções de genes essenciais (dsx e fkh) identificando condições ideais de knockdown por tentativa e erro. Isso aumenta muito a probabilidade de que genes de interesse alvo possam ser investigados usando essa técnica.
Em resumo, verificou-se que a entrega oral de RNAi para mosquitos adultos pode ser simples, versátil e uma abordagem poderosa para estudar a função genética do mosquito e para a criação de novas e maleáveis ferramentas para o controle vetorial de doenças transmitidas por mosquitos.
Divulgações
Os autores relatam que não têm conflitos de interesse para divulgar.
Agradecimentos
Os autores desejam agradecer à equipe e aos cientistas do Ramo de Entomologia e da Divisão de Doenças Parasitárias e Malária do CDC, e Brian Trigg e Michelle Chiu pela assistência com a preparação de bactérias na JHU e/ou discussões úteis deste trabalho. Agradecemos ao JHMRI Insectary e ao gerente Chris Kizito pelo acesso e criação de mosquitos An. gambiae . Agradecemos a Wei Huang (JHSPH) pela ajuda na obtenção de plasmídeos PJet GFP e pPB47 GFP para uso neste estudo. O financiamento para este trabalho foi fornecido por: NIH R21AI153588 (para DJA), uma Bolsa de Pós-doutorado do Johns Hopkins Malaria Research Institute (para MW); e por uma bolsa da Good Ventures Foundation e do Open Philanthropy Project para a Fundação CDC intitulada Support criopreservation and suppression of female development in mosquitos to assist research for malaria, Open Philanthropy Project, 2017. Agradecemos profundamente a assistência da equipe do JHU Microscópio Facility e o suporte de subvenção do NIH aplicável para o microscópio usado (NIH Grant #: S10OD016374). Os achados e conclusões deste manuscrito são dos autores e não representam necessariamente as opiniões do CDC. O uso de nomes comerciais é apenas para identificação e não implica endosso dos Centros de Controle e Prevenção de Doenças, do Serviço Público de Saúde ou do Departamento de Saúde e Serviços Humanos dos EUA.
Materiais
| Name | Company | Catalog Number | Comments |
| 1 Kb Plus DNA Ladder | Thermo Fisher Scientific | 10787018 | |
| 2x Yeast Extract Tryptone (2xYT) Medium | BD Difco | DF0440-17 | |
| AAPP | n/a | n/a | Antisera. 1:50 dilution (rabbit); gift from Fabrizio Lombardo |
| AccuStart II PCR Supermix | Quantabio | 95137-100 | |
| Agarose | Millipore Sigma | A9539 | |
| Ampicillin | Millipore Sigma | A5354 | |
| Anopheles gambiae G3 | BioDefense and Emerging Infections (BEI) Malaria Research and Reference Reagent Resource Center (MR4) | MRA-112 | |
| BugDorm | BioQuip | 1452 | |
| Centrifuge 5810R | Eppendorf | P022628181 | |
| CrebA | DSHB | CrebA Rbt-PC | Antisera. 1:50 dilution (rabbit); generated by the Andrew Lab |
| Damiens diet | BioServ | ||
| DAPI | Life Technologies | n/a | 4′,6-diamidino-2-phenylindole; 1:200 dilution. |
| Defibrinated sheep blood | HemoStat | DSB050 | |
| Escherichia coli HT115 (DE3) | |||
| Ethidium bromide | Millipore Sigma | E7637 | |
| High-Capacity cDNA Reverse Transcription Kit | Thermo Fisher Scientific | 4368814 | |
| Isopropyl β-D-1-thiogalactopyranoside | Millipore Sigma | I5502 | |
| JM109 Competent cells | Promega | L2005 | |
| Luria Broth Media | Thermo Fisher Scientific | 10855001 | |
| Mucin 2 | Proteintech | Muc2; 27 675-1-AP | Antisera. 1:100 dilution (mouse). |
| Nanodrop 2000 | Thermo Fisher Scientific | ||
| Nile Red | Sigma | n/a | Lipid dye; 1:50 dilution. |
| Owl EasyCast B2 Mini Gel Horizontal Electrophoresis | Thermo Fisher Scientific | Model B2 | |
| pGEMT easy | Promega | A3600 | |
| Power SYBR-green PCR master MIX | Applied Biosystems | 4367659 | |
| PureLink PCR purification kit | Thermo Fisher Scientific | K31001 | |
| QuantaStudio 6 | Applied Biosystems | ||
| QuantStudio6 Real Time PCR System | Applied Biosystems | ||
| Rab11 | n/a | n/a | Antisera. 1:100 dilution (rabbit); generated by the Andrew Lab |
| Rh-WGA | Vector Labs | n/a | Rhodamine-conjugated wheat germ agglutinin (chitin, O-GlcNAcylation dye); 1:40 dilution |
| Sage | n/a | n/a | Antisera. 1:50 dilution (rat); generated by the Andrew Lab |
| T4 DNA ligase | Promega | M1801 | |
| Tetracycline | Millipore Sigma | 87128 | |
| Trizol | Thermo Fisher Scientific | 15596018 | |
| Zeiss LSM700 fluorescence confocal microscope | Zeiss | ||
| ANTIBODIES | |||
| Chicken anti-Rat IgG (H+L), Alexa Fluor 647 | Thermo Fisher Scientific | A21472 | |
| Goat anti-Mouse IgG (H+L), Alexa Fluor 647 | Thermo Fisher Scientific | A28181 | |
| IgG (H+L) Goat anti-Rabbit, Alexa Fluor 488 | Thermo Fisher Scientific | A27034 | |
| Rabbit anti-Goat IgG (H+L), Alexa Fluor 488 | Thermo Fisher Scientific | A27012 | |
| PRIMERS | |||
| ACT-2f: TACAACTCGATCATGAAGTGCGA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| ACT-3r: CCCGGGTACATGGTGGTACCGC CGGA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| FKH_RNAi_F: GCCGACTTATGCTTAGCCCA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| FKH_RNAi_R: TAGCCGTCAATTCCTCCTGC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| newDSX-f: AGAGGGCGGGGAAATTCTAGT | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| newDSX-r: GGGCTTGTGGCAGTACGAATA | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| S7qf1: AGAACCAGCAGACCACCATC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
| S7qr1: GCTGCAAACTTCGGCTATTC | CDC Biotechnology Core Facility Branch | n/a | qRT-PCR primer |
Referências
- Hoa, N. T., Keene, K. M., Olson, K. E., Zheng, L. Characterization of RNA interference in an Anopheles gambiae cell line. Insect Biochemistry and Molecular Biology. 33, 949-957 (2003).
- Caplen, N., Zheng, Z., Falgout, B., Morgan, R. Inhibition of viral gene expression and replication in mosquito cells by dsRNA-triggered RNA interference | Elsevier enhanced reader. Molecular Therapy. 6, 243-251 (2002).
- Brown, A. E., Catteruccia, F. Toward silencing the burden of malaria: progress and prospects for RNAi-based approaches. BioTechniques. , 38-44 (2006).
- Airs, P. M., Bartholomay, L. C. RNA interference for mosquito and mosquito-borne disease control. Insects. 8, (2017).
- Blandin, S., et al. Reverse genetics in the mosquito Anopheles gambiae: targeted disruption of the Defensin gene. EMBO Reports. 3 (9), 852-856 (2002).
- Garver, L., Dimopoulos, G. Protocol for RNAi assays in adult mosquitoes (A. gambiae). Journal of Visualized Experiments: JoVE. (5), e230 (2007).
- Whyard, S., et al. Silencing the buzz: a new approach to population suppression of mosquitoes by feeding larvae double-stranded RNAs. Parasites & Vectors. 8, 96 (2015).
- Wiltshire, R. M., Duman-Scheel, M. Advances in oral RNAi for disease vector mosquito research and control. Current Opinion in Insect Science. 40, 18-23 (2020).
- Taracena, M. L., Hunt, C. M., Benedict, M. Q., Pennington, P. M., Dotson, E. M. Downregulation of female doublesex expression by oral-mediated RNA interference reduces number and fitness of Anopheles gambiae adult females. Parasites & Vectors. 12, 170 (2019).
- Grassi, B. Studi di uno zoologo sulla malaria. Real Accademia dei Lincei. 3, 229 (1901).
- Smith, R. C., Jacobs-lorena, M. Plasmodium - Mosquito interactions: A tale of roadblocks and detours. Advances in Insect Physiology. 39, (2010).
- Das, S., et al. Transcriptomic and functional analysis of the Anopheles gambiae salivary gland in relation to blood feeding. BMC Genomics. 11, 1-14 (2010).
- Francischetti, I. M. B., Valenzuela, J. G., Pham, V. M., Garfield, M. K., Ribeiro, J. M. C. Toward a catalog for the transcripts and proteins (sialome) from the salivary gland of the malaria vector Anopheles gambiae. Journal of Experimental Biology. 205, 2429-2451 (2002).
- Henderson, K. D., Isaac, D. D., Andrew, D. J. Cell fate specification in thedrosophila salivary gland: The integration of homeotic gene function with the DPP signaling cascade. Developmental Biology. 205, 10-21 (1999).
- Mach, V., Ohno, K., Kokubo, H., Suzuki, Y. The Drosophila fork head factor directly controls larval salivary gland-specific expression of the glue protein gene Sgs3. Nucleic Acids Research. 24 (12), 2387-2394 (1996).
- Weiserova, M., et al. Mini-Mu transposition of bacterial genes on the transmissible plasmid. Folia Microbiologica. 32 (5), 368-375 (1987).
- Abrams, E. W., Mihoulides, W. K., Andrew, D. J. Fork head and Sage maintain a uniform and patent salivary gland lumen through regulation of two downstream target genes, PH4αSG1 and PH4αSG2. Development. 133, 3517-3527 (2006).
- Myat, M. M., Isaac, P. P., Andrew, D. J. Early genes required for salivary gland fate determination and morphogenesis in Drosophila melanogaster. Advances in Dental Research. 14, 89-98 (2000).
- Fox, R. M., Vaishnavi, A., Maruyama, R., Andrew, D. J. Organ-specific gene expression: the bHLH protein Sage provides tissue specificity to Drosophila FoxA. Development of Cell Biology. 140, 2160-2171 (2013).
- Maruyama, R., Grevengoed, E., Stempniewicz, P., Andrew, D. J. Genome-wide analysis reveals a major role in cell fate maintenance and an unexpected role in endoreduplication for the Drosophila FoxA gene fork head. PLOS ONE. 6, 20901 (2011).
- Johnson, D. M., et al. CrebA increases secretory capacity through direct transcriptional regulation of the secretory machinery, a subset of secretory cargo, and other key regulators. Traffic. 21, 560-577 (2020).
- Fox, R. M., Hanlon, C. D., Andrew, D. J. The CrebA/Creb3-like transcription factors are major and direct regulators of secretory capacity. Journal of Cell Biology. 191, 479-492 (2010).
- Abrams, E. W., Andrew, D. J. CrebA regulates secretory activity in the Drosophila salivary gland and epidermis. Development. 132, 2743-2758 (2005).
- Wells, M. B., Andrew, D. J. Anopheles salivary gland architecture shapes plasmodium sporozoite availability for transmission. mBio. 10 (4), 01238 (2019).
- Pei-Wen, L., Xiao-Cong, L., Jin-Bao, G., Yan, L., Xiao-Guang, C. Molecular cloning, characterization and expression analysis of sex determiantion gene doublesex from Anopheles gambiae (Diptera: Culicidae). Acta Entomologica Sinica. 58 (2), 122-131 (2015).
- Scali, C., Catteruccia, F., Li, Q., Crisanti, A. Identification of sex-specific transcripts of the Anopheles gambiae doublesex gene. The Journal of Experimental Biology. 208, 3701-3709 (2005).
- Price, D. C., Egizi, A., Fonseca, D. M. Characterization of the doublesex gene within the Culex pipiens complex suggests regulatory plasticity at the base of the mosquito sex determination cascade. BMC Evolutionary Biology. 15, 1-13 (2015).
- Mysore, K., et al. siRNA-mediated silencing of doublesex during female development of the dengue vector mosquito Aedes aegypti. PLoS Neglected Tropical Diseases. 9, 1-21 (2015).
- Boisson, B., et al. Gene silencing in mosquito salivary glands by RNAi. FEBS Letters. 580, 1988-1992 (2006).
- Horn, T., Boutros, M. E-RNAi: a web application for the multi-species design of RNAi reagents-2010 update. Nucleic Acids Research. 38, 332-339 (2010).
- Taracena, M. L., et al. Genetically modifying the insect gut microbiota to control chagas disease vectors through systemic RNAi. PLoS Neglected Tropical Diseases. 9, (2015).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular Cloning: A Laboratory Manual. , (1989).
- Ullmann, A., Jacob, F., Monod, J. Characterization by in vitro complementation of a peptide corresponding to an operator-proximal segment of the β-galactosidase structural gene of Escherichia coli. Journal of Molecular Biology. 24, 339-343 (1967).
- Timmons, L. Bacteria-mediated RNAi-General outline. Carnegie Institution of Washington. , (2000).
- Pfaffl, M. W. Relative quantification. Real-time PCR. , 63-82 (2004).
- Neira-Oviedo, M., et al. The RNA-Seq approach to studying the expression of mosquito mitochondrial genes. Insect Molecular Biology. 20, 141-152 (2011).
- Baker, D. A., et al. A comprehensive gene expression atlas of sex- and tissue-specificity in the malaria vector, Anopheles gambiae. BMC Genomics. 12, (2011).
- Loganathan, R., Hoon, J., Wells, M. B., Andrew, D. J. Secrets of secretion - How studies of the Drosophila salivary gland have informed our understanding of the cellular networks underlying secretory organ form and function. Cellular Networks in Development. , 143 (2021).
- Chung, S., Hanlon, C. D., Andrew, D. J. Building and specializing epithelial tubular organs: The Drosophila salivary gland as a model system for revealing how epithelial organs are specified, form and specialize. Wiley Interdisciplinary Reviews: Developmental Biology. 3, 281-300 (2014).
- Yoshida, S., et al. Inhibition of collagen-induced platelet aggregation by anopheline antiplatelet protein, a saliva protein from a malaria vector mosquito. Blood. 111, 2007-2014 (2008).
- Wells, M. B., Villamor, J., Andrew, D. J. Salivary gland maturation and duct formation in the African malaria mosquito Anopheles gambiae. Scientific Reports. 7 (1), 601 (2017).
- Takahashi, S., et al. Rab11 regulates exocytosis of recycling vesicles at the plasma membrane. Journal of Cell Science. 125, 4049-4057 (2012).
- Catteruccia, F., Levashina, E. A. RNAi in the malaria vector, Anopheles gambiae. Methods in Molecular Biology. 555, 63-75 (2009).
- Balakrishna Pillai, A., et al. RNA interference in mosquito: understanding immune responses, double-stranded RNA delivery systems and potential applications in vector control. Insect Molecular Biology. 26, 127-139 (2017).
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in caenorhabditis elegans. Nature. 391, 806-811 (1998).
- Kamath, R. S., Ahringer, J. Genome-wide RNAi screening in Caenorhabditis elegans. Methods. 30, 313-321 (2003).
- Kamath, R. S., et al. Systematic functional analysis of the Caenorhabditis elegans genome using RNAi. Nature. 421, 231-237 (2003).
- Carrasco, D., et al. Behavioural adaptations of mosquito vectors to insecticide control. Current Opinion in Insect Science. 34, 48-54 (2019).
- Magalhaes, T., et al. Induction of RNA interference to block Zika virus replication and transmission in the mosquito Aedes aegypti. Insect Biochemistry and Molecular Biology. , 111 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados