Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Cellule unique réutilisable pour des analyses épigénomiques itératives
Dans cet article
Résumé
Le présent protocole décrit une méthode unicellulaire pour les analyses épigénomiques itératives utilisant une cellule unique réutilisable. La cellule unique réutilisable permet d’analyser plusieurs marques épigénétiques dans la même cellule unique et de valider statistiquement les résultats.
Résumé
Les analyses actuelles de l’épigénome unicellulaire sont conçues pour un usage unique. La cellule est éliminée après une seule utilisation, ce qui empêche l’analyse de plusieurs marques épigénétiques dans une seule cellule et nécessite des données provenant d’autres cellules pour distinguer le signal du bruit de fond expérimental dans une seule cellule. Cet article décrit une méthode permettant de réutiliser la même cellule unique pour des analyses épigénomiques itératives.
Dans cette méthode expérimentale, les protéines cellulaires sont d’abord ancrées à un polymère polyacrylamide au lieu de les réticuler avec la protéine et l’ADN, atténuant ainsi les biais structurels. Cette étape critique permet des expériences répétées avec la même cellule unique. Ensuite, une amorce aléatoire avec une séquence d’échafaudage pour la ligature de proximité est recuite à l’ADN génomique, et la séquence génomique est ajoutée à l’amorce par extension en utilisant une ADN polymérase. Par la suite, un anticorps contre un marqueur épigénétique et des IgG témoins, chacun marqué avec des sondes ADN différentes, sont liés aux cibles respectives dans la même cellule unique.
La ligature de proximité est induite entre l’amorce aléatoire et l’anticorps en ajoutant un ADN connecteur avec des séquences complémentaires à la séquence d’échafaudage de l’amorce aléatoire et à la sonde anticorps-ADN. Cette approche intègre l’information sur les anticorps et les séquences génomiques proches dans un seul produit d’ADN de ligature de proximité. En permettant des expériences répétées avec la même cellule unique, cette méthode permet une augmentation de la densité de données d’une cellule rare et une analyse statistique en utilisant uniquement des données d’IgG et d’anticorps provenant de la même cellule. Les cellules individuelles réutilisables préparées par cette méthode peuvent être stockées pendant au moins quelques mois et réutilisées ultérieurement pour élargir la caractérisation épigénétique et augmenter la densité des données. Cette méthode offre de la flexibilité aux chercheurs et à leurs projets.
Introduction
La technologie unicellulaire entre dans l’ère de la multiomique unicellulaire, qui intègre des technologies omiques unicellulairesindividuelles 1. Récemment, la transcriptomique unicellulaire a été combinée avec des méthodes de détection de l’accessibilité de la chromatine (scNMT-seq2 et SHARE-seq3) ou des modifications des histones (Paired-seq4 et Paired-Tag5). Plus récemment, la transcriptomique et la protéomique unicellulaires ont été intégrées à l’accessibilité de la chromatine (DOGMA-seq6). Ces méthodes utilisent un marquage basé sur la transposase pour détecter l’accessibilité de la chromatine ou les modifications des histones.
Les approches basées sur la transposase clivent l’ADN génomique et ajoutent un code-barres ADN à la fin du fragment d’ADN génomique. Chaque fragment génomique clivé ne peut accepter que jusqu’à deux codes-barres ADN (= une marque épigénétique par site de clivage), et l’ADN génomique au site de clivage est perdu. Par conséquent, les approches basées sur le clivage ont un compromis entre le nombre de marques épigénétiques testées et la densité du signal. Cela entrave l’analyse de plusieurs marques épigénétiques dans la même cellule. Une méthode épigénomique unicellulaire qui ne clive pas l’ADN génomique a été développée pour surmonter ce problème 7,8.
En plus de la question dérivée du clivage mentionnée ci-dessus, les approches basées sur la transposase ont d’autres limites. Dans l’analyse de l’épigénome unicellulaire, il est essentiel de connaître l’emplacement des histones et des protéines associées à l’ADN sur le génome. Dans les approches actuelles, cela est accompli en utilisant des cellules uniques non fixées et en conservant les interactions protéine-ADN et protéine-protéine. Cependant, cela génère un fort biais vers les régions chromatiniques accessibles, même dans l’analyse des modifications des histones 9. L’emplacement des histones et des protéines associées au génome sur le génome peut être préservé sans réticulation protéine-ADN et protéine-protéine, à l’aide d’un échafaudage en polyacrylamide 7,8. Cette approche réduit le biais structurel observé dans les approches actuelles qui dépendent des interactions protéine-ADN et protéine-protéine.
Les approches basées sur la transposase ne peuvent acquérir des signaux qu’une seule fois à partir d’une seule cellule. Par conséquent, il est difficile de délimiter l’épigénome complet d’une seule cellule en raison de la chute des signaux. Des cellules uniques réutilisables ont été développées pour surmonter les limitations actuelles en permettant une analyse épigénomique itérative dans la même cellule unique.
Protocole
Remarque : Une représentation schématique de la méthode est illustrée à la figure 1.
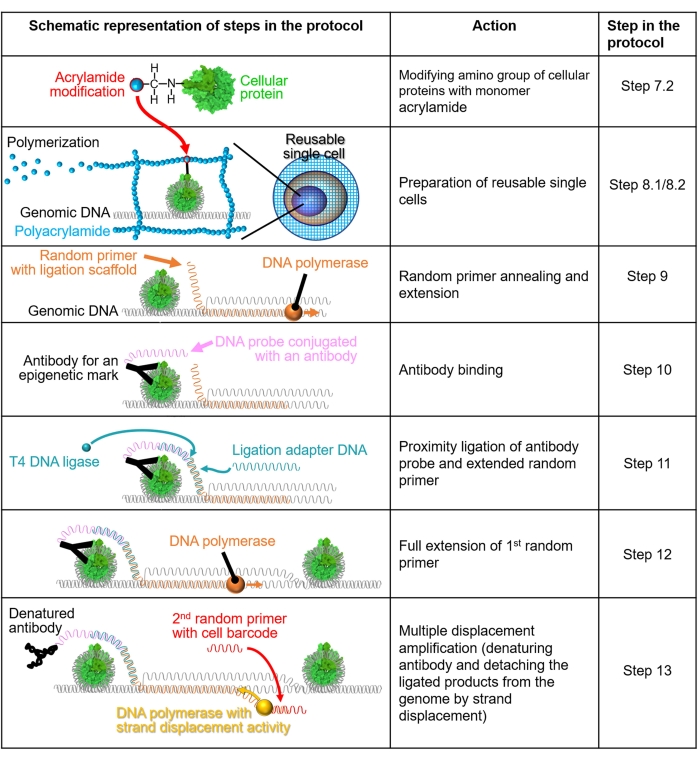
Figure 1 : Représentation schématique du flux de travail du protocole. Les étapes 7.2 à 13 sont expliquées au moyen de représentations schématiques. Chaque ligne indique une étape du protocole. Une protéine cellulaire colorée en vert est un nucléosome humain généré à partir d’une structure cristalline (PDB : 6M4G). Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Équilibrage des colonnes de dessalage
Remarque : Les colonnes de dessalement sont équilibrées comme décrit dans les étapes suivantes. Les colonnes de dessalage équilibrées sont utilisées aux étapes 2.1, 3.4 et 4.6.
- Retirez la fermeture inférieure d’une colonne de dessalage (coupure de 7 kDa, volume de billes de résine de 0,5 mL, voir le tableau des matériaux) et desserrez le bouchon en haut de la colonne de dessalage.
- Placer la colonne dans un tube à faible liaison de protéines de 1,5 mL (un tube de collecte, voir le tableau des matériaux) et centrifuger à 1 500 × g pendant 1 min à température ambiante pour éliminer la solution de stockage dans la colonne.
REMARQUE: Utilisez une centrifugeuse à rotor oscillant pour aplatir le haut du lit de billes. - Retirer le flux dans le tube de 1,5 mL et ajouter 300 μL de tampon phosphate NaCl 150 mM/100 mM, pH 8,0 (voir le tableau 1), sur le lit de résine.
- Centrifuger à 1 500 × g pendant 1 min à température ambiante et retirer le débit dans le tube de collecte.
- Répétez les étapes 1.3-1.4 trois fois de plus.
- Supprimez le tampon du tube de collecte et placez la colonne dans un nouveau tube de collecte.
- Utilisez la colonne de dessalage équilibrée aux étapes 2.1, 3.4 et 4.4.
2. Échange tampon d’anticorps
REMARQUE: Retirez le glycérol, l’arginine et l’azoture de sodium des anti-H3K27ac 10, anti-H3K27me3 10, anti-Med111 et anti-Pol II10 (voir la composition tampon indiquée dans le tableau 1). Toutes les procédures suivantes sont effectuées sous une hotte propre pour éviter la contamination par la DNase. Durée : 1 h
- Appliquer la solution d’anticorps (100 μL, voir tableau 2) sur une colonne de dessalage équilibrée préparée après l’étape 1.
- Centrifuger à 1 500 × g pendant 2 min à 4 °C et transférer le débit du tube collecteur dans un tube à faible liaison de protéines de 1,5 mL.
- Mesurer la concentration d’IgG en utilisant l’absorbance à 280 nm12 (utiliser un spectrophotomètre de microvolume).
- Transférer la solution d’anticorps dans une cassette d’ultrafiltration (seuil de poids moléculaire 100 kDa, 0,5 mL, voir le tableau des matériaux) et centrifuger à 12 000 × g pendant 5 min à 4 °C.
- Mesurer la concentration d’IgG en utilisant l’absorbance à 280 nm.
- Répétez les étapes 2.4-2.5 jusqu’à ce que la concentration d’IgG atteigne 1 mg/mL.
3. Activation des anticorps
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 2,5 h
- Dissoudre 1 mg de succinimidyl 6-hydrazinonicotinate acétone hydrazone (S-HyNic, voir le tableau des matières) avec 100 μL de N,N-diméthylformamide anhydre (DMF, voir le tableau des matières).
REMARQUE: Le DMF est un solvant organique inflammable et une toxine hépatique puissante absorbée par la peau. Portez des gants, des lunettes de sécurité et une blouse de laboratoire. Suivez les consignes de sécurité de l’établissement. Jetez les articles de laboratoire usagés conformément aux directives de l’établissement. - Ajouter 0,6 μL de S-HyNic/DMF à 100 μL de la solution d’anticorps (1 mg/mL dans 150 mM de NaCl/100 mM de phosphate de sodium, pH8,0. Voir le tableau 2 pour les anticorps utilisés et les IgG témoins.
- Incuber à température ambiante pendant 2 h (à l’abri de la lumière).
- Appliquer 100 μL d’anticorps ayant réagi au S-HyNic sur le dessus de la colonne de dessalage équilibrée (voir étape 1) et centrifuger à 1 500 × g pendant 2 min à 4 °C pour prélever l’échantillon.
- Jeter la colonne de dessalage après utilisation.
REMARQUE: La stabilité des groupes HyNic sur les protéines et autres biomolécules varie. Il est recommandé de conjuguer immédiatement les biomolécules modifiées par HyNic.
4. Activation de la sonde ADN
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 2,5 h
- Dissoudre une pastille d’une sonde d’ADN modifiée aux amines pour détecter la présence d’anticorps ou d’IgG témoins (sonde anticorps, tableau 3) avec 20 μL de tampon de phosphate de sodium de 150 mM/100 mM, pH 8,0.
REMARQUE: Recherchez une pastille / film mince et transparent au fond du tube. Appliquez le tampon directement sur la pastille. Si le granulé n’est pas visible, il peut s’être détaché et adhérer au mur ou au couvercle. Dans ce cas, centrifugez le tube et recherchez une pastille au fond du tube. - Dissoudre 1 mg de 4-formylbenzoate de succinimidyle (S-4FB, voir le tableau des matières) avec 50 μL de DMF anhydre et ajouter 10 μL de DMF à la sonde d’anticorps dissoute.
- Ajouter 4 μL de S-4FB/DMF préparé à l’étape 4.2, mélanger et incuber à température ambiante pendant 2 h (à l’abri de la lumière).
- Appliquez 34 μL de sonde d’anticorps à réaction S-4FB sur le dessus de la colonne de dessalage équilibrée (voir étape 1).
- Appliquer 15 μL de tampon phosphate de sodium 150 mM NaCl/100 mM, pH 8,0, sur le dessus du lit de gel après avoir complètement absorbé l’échantillon, et centrifuger à 1 500 × g pendant 2 min à 4 °C pour recueillir la sonde d’anticorps modifiée par S-4FB.
- Utilisez le flux pour la conjugaison suivante; mesurer l’absorbance à 260 nm; et calculer le taux de récupération de la sonde anticorps.
5. Conjugaison de l’anticorps modifié par S-HyNic et de la sonde d’anticorps modifiée par S-4FB
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 2 h
- Mélangez l’anticorps modifié par S-HyNic et la sonde d’anticorps modifiée par S-4FB, puis pipeter la solution de haut en bas pour mélanger.
- Incuber pendant 2 h à température ambiante (à l’abri de la lumière).
- Pour éteindre la réaction, ajoutez 478,8 μL de solution de trempe et de stockage (voir Tableau 1).
- Transférer la solution d’anticorps conjuguée à la sonde d’anticorps dans une cassette d’ultrafiltration (seuil de poids moléculaire 100 kDa).
- Centrifuger pendant 5 min à 12 000 × g, 4 °C.
- Vérifiez le volume de la solution à l’intérieur de la cassette par pipetage.
- Répétez les étapes 5.5-5.6 jusqu’à ce que le volume atteigne 100 μL et conservez à -20 °C.
NOTE: La concentration d’IgG est mesurée par ELISAsandwich 13,14,15 à l’aide d’un étalon IgG.
6. Préparation des billes magnétiques du noyau
REMARQUE : Dans cette méthode, une seule cellule est incorporée dans une bille d’acrylamide bicouche (voir la figure 2). Le noyau est une perle de polyacrylamide magnétique. La couche externe est constituée de polyacrylamide seul. Les perles magnétiques de base sont générées dans cette section. Cette section n’est pas essentielle pour l’expérience. Durée : 3 h
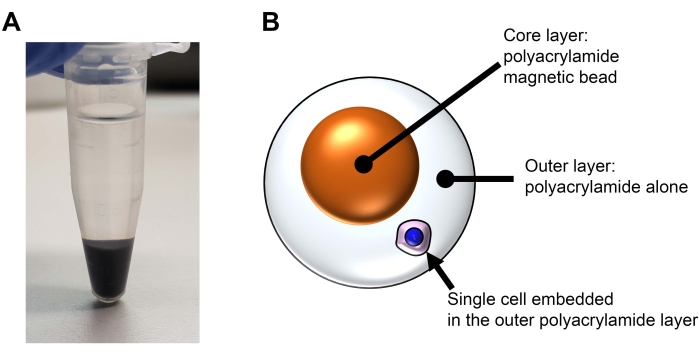
Figure 2 : Structure d’une bille de polyacrylamide bicouche pour la visibilité et une manipulation facile dans les expériences REpi-seq. (A) Nanoparticules magnétiques de l’étape 6.6 après centrifugation. Les nanoparticules magnétiques sont modifiées avec de l’acrylamide monomère et intégrées dans la bille magnétique de polyacrylamide représentée en B. (B) Représentation schématique d’une cellule unique réutilisable avec une bille magnétique en polyacrylamide. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Mélanger 50 μL de tampon de bicarbonate de sodium 1 M, pH 8,5 et 450 μL de solution d’acrylamide à 40 %.
NOTE: L’acrylamide est une neurotoxine. Portez des gants, un protecteur oculaire et une blouse de laboratoire. Suivez les consignes de sécurité de l’établissement. Jetez les articles de laboratoire usagés conformément aux directives de l’établissement. - Suspendre 1 g de poudre d’oxyde de fer fonctionnalisée par un ester NHS (voir le tableau des matériaux) dans le tampon acrylamide/bicarbonate de sodium et incuber à 4 °C pendant une nuit.
REMARQUE: Parce que les billes d’acrylamide sont transparentes, il peut être difficile de voir et de manipuler la position des perles. L’inclusion d’un cordon de noyau en oxyde de fer améliore la visibilité et facilite la manipulation car la position des billes peut être contrôlée à l’aide d’un aimant. Cependant, si les utilisateurs sont familiers avec les expériences REpi-seq, l’utilisation des billes de polyacrylamide magnétiques à noyau peut être ignorée. - Transférer la suspension de nanobilles dans 2 tubes (1,5 mL), centrifuger à 21 300 × g pendant 1 h (utiliser un rotor incliné) à 4 °C et retirer le surnageant.
- Suspendre la suspension inférieure avec 1 mL d’acrylamide/bis-acrylamide à 40 % (19:1, voir le tableau des matières).
NOTE: Le bis-acrylamide est une neurotoxine. Portez des gants et une blouse de laboratoire. Suivez les consignes de sécurité de l’établissement. Jetez les articles de laboratoire usagés conformément aux directives de l’établissement. - Centrifuger à 21 300 × g pendant 1 h (utiliser un rotor incliné) à 4 °C.
- Centrifuger à 5 000 × g pendant 30 min (utiliser un rotor oscillant sans frein) à 4 °C.
- Retirez le surnageant à l’aide d’une pipette P1000 à aspiration lente.
- Régler le volume à 400 μL d’acrylamide/bis-acrylamide à 40 % (19:1, voir le tableau des matières).
- Ajouter 25 μL de solution de persulfate d’ammonium à 10 % (voir tableau 1).
REMARQUE: Le persulfate d’ammonium est un agent oxydant puissant. La poussière en suspension dans l’air contenant du persulfate d’ammonium peut irriter les yeux, le nez, la gorge, les poumons et la peau au contact. Portez des gants, des lunettes de sécurité et une blouse de laboratoire. Suivez les consignes de sécurité de l’établissement. Jetez les articles de laboratoire usagés conformément aux directives de l’établissement. - Pour générer des billes de polyacrylamide magnétiques à noyau, transférer 0,5 μL de la suspension d’oxyde de fer modifié à l’acrylamide dans un tube PCR.
- Ajouter 50 μL d’huile minérale N,N,N,N',N’tétraméthyléthylènediamine/huile minérale à 4 % (TEMED , voir le tableau 1) et incuber pendant une nuit à température ambiante.
REMARQUE : TEMED est un solvant inflammable. Travaillez sous un capot. Ne pas inhaler. Tenir à l’écart des flammes nues, des surfaces chaudes et des sources d’inflammation. Portez des gants, des lunettes de sécurité et une blouse de laboratoire. Suivez les consignes de sécurité de l’établissement. Jetez les articles de laboratoire usagés conformément aux directives de l’établissement.
7. Modifier le groupe aminé des protéines cellulaires avec l’acrylamide monomère
REMARQUE: REpi-seq a été conçu pour analyser l’épigénome de cellules de souris et humaines au niveau de la cellule unique. Chaque étape doit être optimisée lors de l’utilisation de cette méthode sur des cellules d’espèces autres que la souris ou l’homme.
- Récolte des cellules
NOTE: Durée: 30 min- Mesurer la concentration et la viabilité des cellules à l’aide d’un compteur de cellules (voir le tableau des matériaux).
Remarque : La viabilité des cellules à cette étape affecte le nombre de cellules sont des cellules vivantes dans l’analyse des données. - Ajuster la concentration cellulaire à 1 × 105 cellules/ml avec un milieu de culture (*) contenant 10 % de sérum fœtal bovin.
NOTE: *Le milieu de culture est un milieu de culture optimal pour les cellules d’intérêt. - Transférer 1 mL de la suspension cellulaire dans un tube de 1,5 mL.
- Centrifuger la suspension cellulaire à 240 × g pendant 5 min à 4 °C et retirer le surnageant.
- Ajouter 1 mL de solution saline tamponnée au phosphate (PBS), mélanger les cellules par pipetage doux et centrifuger la suspension cellulaire à 240 × g pendant 5 min à 4 °C.
- Retirez le surnageant.
REMARQUE: Toutes les étapes ci-dessus doivent être effectuées dans une hotte de nettoyage à flux laminaire pour éviter toute contamination.
- Mesurer la concentration et la viabilité des cellules à l’aide d’un compteur de cellules (voir le tableau des matériaux).
- Modifier le groupe amino des protéines cellulaires avec de l’acrylamide monomère
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 1,5 h- Ajouter 1 mL de solution de modification du groupe amino (voir le tableau 1) à la pastille de cellule et suspendre la pastille de cellule par pipetage doux.
- Incuber le tube sur de la glace pendant 1 h et centrifuger la suspension cellulaire à 240 × g pendant 5 min à 4 °C.
- Retirer le surnageant et remettre les cellules en suspension avec 100 mL d’acrylamide à 4 % / 1 mM D’EDTA/PBS contenant un colorant intercalateur pour l’ADN (voir le tableau 1, 1 cellule/μL).
8. Préparation de cellules individuelles réutilisables
- Préparation de cellules individuelles réutilisables (version manuelle)
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 9 h/96 cellules- Mélanger 1 mL de suspension cellulaire (1 cellule/μL) et 199 mL de 1 mM D’EDTA/PBS contenant un colorant intercalateur pour l’ADN (voir le tableau 1).
- Transférer 200 μL de la suspension cellulaire dans chaque puits de plaques à fond plat de 96 puits (total de 10 plaques, voir le tableau des matériaux).
- Placez le couvercle sur la plaque de 96 puits et scannez les 10 plaques à l’aide d’un microscope à balayage (voir le tableau des matériaux) pour identifier les puits contenant une seule cellule.
- Transférer le contenu du puits contenant une seule cellule dans un tube PCR.
- Inclinez la plaque (vers l’opérateur) et attendez quelques minutes jusqu’à ce que la cellule unique soit descendue jusqu’au bord inférieur du puits.
- Placez l’embout de la pipette dans le coin inférieur et transférez la cellule unique et le tampon (aspirer 210 μL/puits = 200 μL de tampon + 10 μL d’air) dans un tube de PCR.
REMARQUE: Assurez-vous d’utiliser un embout P200 à faible rétention. - Vérifiez le puits à l’aide d’un microscope à fluorescence pour confirmer le transfert.
- Si une seule cellule est encore dans le puits, ajouter 200 μL/puits de 1 mM D’EDTA/PBS contenant un colorant intercalateur pour l’ADN.
- Ensuite, répétez le transfert.
- Centrifuger le tube PCR à 240 × g pendant 5 min à 4 °C à l’aide d’un rotor pivotant sans frein.
REMARQUE: Le freinage provoque un écoulement tourbillonnant d’eau dans le tube, ce qui perturbe la précipitation de la cellule unique. Lors de la centrifugation avec un rotor oscillant sans frein, la cellule unique coulera toujours au fond du tube. Cependant, si un rotor incliné est utilisé, la cellule unique se fixe à la paroi latérale du tube PCR et pourrait être perdue. - Retirer 195 μL du surnageant à une vitesse de pipetage très lente.
- Ajouter 195 μL/tube de solution Acrylamide/Bis-acrylamide/APS (voir tableau 1).
- Centrifuger le tube PCR à 240 × g pendant 5 min à 4 °C à l’aide d’un rotor pivotant sans frein.
- Enlever 195 μL du surnageant à vitesse de pipetage très lente.
- Transférer les 3 μL inférieurs dans le tube PCR contenant 4 % d’huile minérale TEMED/et une bille de polyacrylamide magnétique (générée à l’étape 6).
REMARQUE: Assurez-vous d’utiliser un conseil P10 à faible rétention. Distribuer la cellule près de la bille de polyacrylamide magnétique. Dans l’huile minérale, le liquide contenant la cellule unique adhère à la surface du cordon de polyacrylamide magnétique par tension superficielle. - Incuber pendant la nuit à température ambiante.
REMARQUE: Ce processus génère une bille de gel d’acrylamide bicouche. Le noyau est une perle de gel magnétique. La couche externe est constituée de gel de polyacrylamide contenant une seule cellule. La cellule unique est intégrée dans la couche externe du gel. Point d’arrêt sûr : Après avoir lavé les cellules individuelles réutilisables avec 50 % de glycérol/1 mM d’EDTA/0,05 % de tampon Tween 20/0,5 % BSA/TBS, les cellules individuelles réutilisables peuvent être conservées à -20 °C jusqu’à 6 mois.
- Préparation de cellules individuelles réutilisables (version semi-automatisée)
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 3 h/96 cellules- Mélanger 1 mL de suspension cellulaire (1 cellule/μL) et 199 mL de 1 mM D’EDTA/PBS contenant un colorant intercalateur pour l’ADN (voir le tableau 1).
- Transférer 200 μL de la suspension cellulaire dans chaque puits d’une plaque de nanopuits de 4 nL (voir la figure 3 et le tableau des matériaux) et placer la plaque de nanopuits de 4 nL sur un robot automatisé de prélèvement de cellules uniques (voir le tableau des matériaux).
- Placez une plaque PCR à 96 puits comme plaque de destination sur le robot automatisé de prélèvement de cellules uniques. S’assurer que chaque puits contient 200 μL/solution d’acrylamide/bis-acrylamide/APS (voir le tableau 1).
- Transférer une seule cellule d’un nanopuits de 4 nL vers un puits de la plaque PCR à 96 puits (voir la vidéo supplémentaire S1).
- Placer un couvercle sur la plaque PCR à 96 puits et centrifuger la plaque de 96 puits à 240 × g pendant 5 min à 4 °C à l’aide d’un rotor oscillant sans frein.
- Placez la plaque PCR à 96 puits sur le pont d’un robot de manutention automatique des liquides (voir le tableau des matériaux, figure 4, et le code supplémentaire 1).
- Faites glisser et déposez le code supplémentaire 1 dans la fenêtre du logiciel (voir le tableau des matériaux) du robot de manutention des liquides.
- Exécutez le programme sur le robot de manipulation automatique des liquides (voir Vidéo supplémentaire S2 et Vidéo supplémentaire S3).
- Retirer 195 μL du surnageant à une vitesse de pipetage très lente.
- Ajouter 50 μL d’huile minérale TEMED à 4 %.
REMARQUE : Les étapes 8.2.8.1 à 8.2.8.2 peuvent être effectuées par pipetage manuel.
- Incuber pendant la nuit à température ambiante (figure 5). Point d’arrêt sûr : Après avoir lavé les cellules individuelles réutilisables avec 50 % de glycérol/1 mM d’EDTA/0,05 % de tampon Tween 20/0,5 % BSA/TBS, conservez les cellules individuelles réutilisables à -20 °C jusqu’à 6 mois.
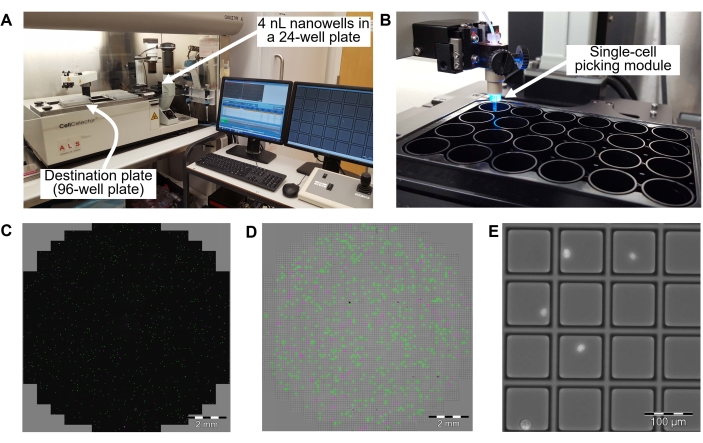
Figure 3: Prélèvement et transfert automatiques d’une seule cellule dans une plaque PCR à 96 puits à l’étape 8.2 . (A) Vue d’ensemble d’un système de prélèvement de cellules uniques. Un robot de prélèvement de cellules unique est dans une hotte de nettoyage à flux laminaire pour éviter la contamination. (B) Une plaque de 24 puits avec 4 nanopuits nL à l’intérieur du puits. (C) Distribution des cellules dans un puits à partir de la plaque de 24 puits. Les points verts sont des cellules identifiées comme une seule cellule dans chaque nanopuits de 4 nL. Les points magenta sont des cellules identifiées comme des doublets ou des multiplets de cellules. (D) Image en fond clair du puits dans la plaque de 24 puits. Un carré vert est un nanopuits de 4 nL contenant une seule cellule. Un carré magenta est un nanopuits de 4 nL contenant plusieurs cellules. (E) Champ amplifié de quelque 4 nanopuits. Les points lumineux sont des cellules uniques dans 4 nanopuits de nL. Le système de prélèvement de cellules uniques identifie les nanopuits contenant une seule cellule en acquérant des images en fond clair et en fluorescence de cellules avec coloration DAPI. Les cellules individuelles identifiées sont transférées du nanopuits de 4 nL à un puits d’une plaque PCR de 96 puits. Barres d’échelle = 2 mm (C, D), 100 μm (E). Abréviation = DAPI = 4',6-diamidino-2-phénylindole. Veuillez cliquer ici pour voir une version agrandie de cette figure.
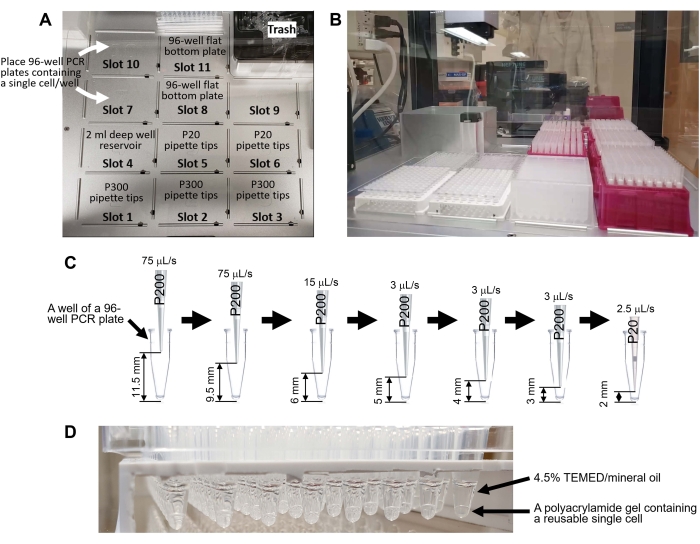
Figure 4 : Génération de cellules individuelles réutilisables à l’aide d’un robot de manipulation de liquides. (A) Un pont du robot de manutention des liquides. Le tablier comporte 11 emplacements pour les racks de pointe de pipettes (pointe P300 : emplacements 1 à 3, pointe P20 : fentes 5-6), plaque de 96 puits de 2 ml de puits profonds (fente 4), deux plaques PCR de 96 puits contenant une seule cellule par puits (fentes 7 et 10) et deux plaques à fond plat de 96 puits pour les déchets liquides (fentes 8 et 11). (B) Le pont après la mise en place de l’ustensile de laboratoire. (C) Représentation schématique du pipetage robotisé à l’étape 8.8.1. Le programme élimine le surnageant sans aspirer une seule cellule du fond de la plaque PCR à 96 puits. (D) Cellules individuelles réutilisables générées à l’aide du code supplémentaire 1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
9. Recuit et extension aléatoire de l’apprêt
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Les astérisques (*) aux étapes suivantes indiquent qu’un séparateur magnétique peut être utilisé pour contrôler la position des billes de polyacrylamide contenant une cellule unique réutilisable dans le tube. Cependant, l’utilisation du séparateur magnétique n’est pas indispensable. En descendant lentement la pointe de la pipette le long de la paroi du tube, les billes sont poussées vers le haut pour le lavage ou l’échange de tampon. Durée : 9 h
- Retirer l’huile minérale par pipetage(*) et laver (*) 5 fois avec 200 μL de 1x tampon TP Mg(-) (diluer 10x tampon TP Mg(-) à 1x tampon avec de l’eau ultrapure, voir tableau 1).
- Retirer le surnageant (*) et ajouter 15 μL de tampon de recuit (voir tableau 1).
- Incuber pendant 1 h sur glace.
NOTE: Le but de cette incubation est de perméabiliser le plasma et les membranes nucléaires et de livrer l’amorce aléatoire au noyau cellulaire. - Placer les tubes PCR sur un thermocycleur et chauffer à 94 °C pendant 3 min.
REMARQUE: La taille du tube est de 0,2 mL. Le volume de la solution est d’environ 20 μL, y compris le volume de la bille de polyacrylamide. La température du couvercle du thermocycleur est de 105 °C. - Transférer les tubes dans un bloc métallique refroidi à la glace et incuber pendant 2 min.
- Ajouter 4 μL de mélange MgSO4/NaCl/dNTP (voir le tableau 1) et mélanger avec un mélangeur vortex à vitesse moyenne.
- Ajouter 1 μL de Bst polymérase, gros fragment (voir le tableau des matériaux) et mélanger à l’aide d’un mélangeur vortex à vitesse moyenne.
- Incuber pendant 4 h sur agitateur (600 tr/min à 4 °C).
REMARQUE: Le but de cette incubation est de délivrer la Bst polymérase au noyau cellulaire. - Placez le tube PCR sur un thermocycleur et exécutez l’un des programmes suivants.
- Exécutez un programme de 4 h : 10 °C pendant 30 min, 20 °C pendant 30 min et 25 °C pendant 180 min.
- Vous pouvez également exécuter un programme de nuit : 4 °C pendant 4 h, 10 °C pendant 2 h, 20 °C pendant 2 h, 25 °C pendant 4 h et maintenir à 4 °C.
REMARQUE: La taille du tube est de 0,2 mL. Le volume de la solution est d’environ 25 μL, y compris le volume de la bille de polyacrylamide. La température du couvercle du thermocycleur est de 105 °C. Point d’arrêt sûr: Arrêtez l’expérience ici jusqu’à 1 jour en stockant les cellules individuelles réutilisables à 4 ° C.
10. Liaison aux anticorps
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 1,5 h
- Ajouter 1,625 μL de solution NaCl/EDTA/BSA (voir tableau 1) et mélanger en tourbillonnant à basse vitesse.
NOTE: Cette étape vise à 1) faciliter la chélation de Mg2+ par EDTA, 2) stabiliser la liaison de l’amorce aléatoire étendue de 300 mM NaCl, et 3) bloquer la liaison non spécifique des anticorps à l’aide d’albumine sérique bovine (BSA) dans la réaction ultérieure. - Incuber pendant 1 h sur de la glace et ajouter 0,1 μg/mL chacun d’anticorps et contrôler les IgG conjuguées avec Antibody Probe (préparé à l’étape 5).
- Incuber toute la nuit sur la glace en secouant doucement sur un shaker.
11. Ligature de proximité de la sonde anticorps et amorce aléatoire étendue de manière proximale
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Les astérisques (*) dans les étapes suivantes indiquent où un séparateur magnétique peut être utilisé pour contrôler la position des billes de polyacrylamide contenant une cellule unique réutilisable dans le tube. Cependant, l’utilisation du séparateur magnétique n’est pas indispensable. En descendant lentement la pointe de la pipette le long de la paroi du tube, les billes peuvent être poussées vers le haut pour le lavage ou l’échange tampon. Durée : 6 h
- Lavez (*) deux fois avec 200 μL de 1x tampon TPM-T (20 min d’incubation à chaque lavage) sur de la glace. Diluer 10x tampon TPM-T (Tris-HCl / Chlorure de potassium / Sulfate de magnésium / Triton X-100) à 1x avec de l’eau ultrapure).
- Retirer (*) le surnageant et laver (*) une fois avec 1x tampon ADN ligase T4 (voir le tableau des matières).
- Retirer (*) le surnageant et ajouter 19 μL de solution d’adaptateur de ligature (voir tableau 1).
- Incuber pendant 1 h à 25 °C.
- Ajouter 1 μL d’ADN ligase T4 (voir le tableau des matériaux) et mélanger le tube sur un mélangeur vortex à vitesse moyenne.
- Placez le tube sur un thermocycleur et exécutez le programme suivant : programme de ligature de proximité, 4 h à 16 °C et 30 min à 25 °C.
REMARQUE: La taille du tube est de 0,2 mL. Le volume de la solution est d’environ 25 μL, y compris le volume de la bille de polyacrylamide. La température du couvercle du thermocycleur est à température ambiante. - Laver (*) deux fois brièvement avec 200 μL de 1x tampon Bst Mg(-) EDTA(+) (voir tableau 1; préparer 1x tampon à partir de 10x tampon stock) et conserver la cellule à 4 °C, pendant une nuit. Point d’arrêt sûr: Arrêtez l’expérience ici jusqu’à 1 jour en stockant les cellules individuelles réutilisables à 4 ° C.
12. Extension complète de la 1ère amorce aléatoire
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 4,5 h
- Laver deux fois avec 200 μL 1x tampon Bst Mg(-) EDTA(-) (voir le tableau 1, préparer 1x tampon à partir de 10x tampon stock), retirer le surnageant et ajouter 20 μL de mélange Bst/dNTP/MgSO4 (voir Tableau 1).
- Mélanger avec un mélangeur vortex à vitesse moyenne et incuber pendant 4 h sur un agitateur orbital (à 6 °C et 500 tr/min).
- Placez le tube sur un thermocycleur et exécutez le programme suivant: programme d’extension complète: 10 °C pendant 1 h, 20 °C pendant 1 h, 30 °C pendant 1 h, 40 °C pendant 1 h, 50 °C pendant 1 h, 65 °C pendant 1 h, 94 °C pendant 10 min, et maintenez à 4 °C.
REMARQUE: Point d’arrêt sûr: L’expérience peut être arrêtée ici jusqu’à 1 jour en stockant les cellules individuelles réutilisables à 4 ° C.
13. Amplification à déplacement multiple
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 2,5 h (étapes 13.1-13.2) + 15 min (étapes 13.3-13.4) + 1 jour (étapes 13.5-13.10)
- Ajouter 0,4 μL de 100 μM 2nd amorce aléatoire (voir tableau 3) et mélanger avec un mélangeur vortex à vitesse moyenne.
- Incuber pendant 2 h à 6 °C et 500 tr/min sur un agitateur orbital, placer le tube sur un thermocycleur et chauffer à 94 °C pendant 3 min.
REMARQUE: La taille du tube est de 0,2 mL. Le volume de la solution est d’environ 25,4 μL, y compris le volume de la bille de polyacrylamide. La température du couvercle du thermocycleur est de 105 °C. - Placez les tubes dans un bloc métallique refroidi à la glace et ajoutez 1 μL/tube d’ADN polymérase Bst.
- Vortex à vitesse lente, placez les tubes sur un cycleur thermique et exécutez le programme suivant:
4 °C pendant 4 h, 10 °C pendant 30 min, 20 °C pendant 30 min, 30 °C pendant 30 min, 40 °C pendant 30 min, 50 °C pendant 30 min, 65 °C pendant 60 min, 94 °C pendant 3 min, et maintenir à 4 °C.
REMARQUE: La taille du tube est de 0,2 mL. Le volume de la solution est d’environ 26,4 μL, y compris le volume de la bille de polyacrylamide. La température du couvercle du thermocycleur est de 105 °C. Point d’arrêt sûr: Arrêtez l’expérience ici jusqu’à 1 jour en stockant les cellules individuelles réutilisables à 4 ° C. - Recueillir le surnageant (environ 20 μL) et le transférer dans un tube PCR.
- Conserver le surnageant collecté à -80 °C.
- Ajouter 20 μL/tube de tampon Tween 20/0,1x TE à 0,05 % à un tube de PCR contenant la cellule unique réutilisable (voir le tableau 1) et incuber la cellule unique réutilisable à 4 °C, pendant une nuit. Point d’arrêt sûr: Arrêtez l’expérience ici pendant quelques jours en prolongeant le temps d’incubation.
- Prélever le surnageant et combiner le surnageant avec l’échantillon prélevé à l’étape 13.5.
- Répétez les étapes 13.7-13.8 une fois de plus. Faire tremper la cellule unique réutilisable dans un tampon 50 % glycérol/5 mM EDTA/0,5 % BSA/0,05 % Tween20/TBS et l’incuber pendant 30 min sur un agitateur orbital (4 °C, 600 tr/min). Conserver la cellule unique réutilisable à -20 °C jusqu’à la prochaine expérience.
- Ajouter 40 μL d’Exo-master mix (voir tableau 1) et placer le tube sur un thermocycleur et exécuter le programme suivant: i) 95 °C pendant 5 min, ii) 95 °C pendant 30 s, iii) 60 °C 30 s, iv) 72 °C pendant 30 s, v) répéter les étapes ii-iv 19 fois, vi) 72 °C pendant 5 min, vii) maintenir à 4 °C.
REMARQUE: La taille du tube est de 0,2 mL. Le volume de la solution est d’environ 45 μL, y compris le volume de la bille de polyacrylamide. La température du couvercle du thermocycleur est de 105 °C. Point d’arrêt sûr: L’expérience peut être arrêtée ici pendant au moins quelques jours en stockant l’échantillon à -80 °C.
14. Purification du phénol-chloroforme et précipitation du polyéthylène glycol
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 1,5 h
- Transférer le produit dans un tube à faible liaison d’ADN de 0,5 mL et ajouter 100 μL de phénol:chloroforme:alcool isoamylique.
NOTE: Phénol: Chloroforme: Alcool isoamylique provoque une irritation et éventuellement des brûlures par contact. Portez des gants, des lunettes de sécurité et une blouse de laboratoire. Utiliser uniquement avec une ventilation adéquate ou porter un respirateur approprié. Suivez les consignes de sécurité de l’établissement. Jetez les articles de laboratoire usagés conformément aux directives de l’établissement. - Agiter pendant 30 s à la main et centrifuger à 12 000 × g pendant 10 min à 4 °C.
- Recueillir la phase aqueuse (80 μL) dans un tube à faible liaison d’ADN de 0,5 mL et ajouter 40,84 μL/tube de mélange acrylamide/MgCl2 linéaire (voir Tableau 1).
- Ajouter 47,06 μL/tube de PEG8000 à 50 % (p/v) (sans RNase) et mélanger par pipetage.
- Incuber pendant 20 min à température ambiante et centrifuger à 240 × g pendant 10 min à température ambiante.
- Retirer le surnageant et ajouter 400 μL/tube d’éthanol à 80 % (EtOH).
- Laver avec 80% d’EtOH, retirer le surnageant à l’aide d’un aspirateur et sécher le granulé à l’air.
- Suspendre la pastille avec 20 μL d’EDTA/10 mM de Tris-HCl, tampon pH 7,4, et conserver la solution à -80 °C.
REMARQUE : Mesurer la concentration d’ADN à l’aide d’un colorant intercalateur double brin spécifique de l’ADN (voir le tableau des matériaux). Point d’arrêt sûr: L’expérience peut être arrêtée ici en toute sécurité pendant au moins une semaine.
15. Transcription in vitro
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase et la RNase. Durée : 5 h
- Décongeler l’ADN des produits dérivés d’une seule cellule (à partir de l’étape 14) et préparer une bibliothèque mixte de produits dérivés d’une seule cellule en mélangeant 2 μL/cellule des produits d’ADN (volume total 20 μL).
- Ajouter 26 μL de mélange maître pour transcription in vitro (voir le tableau des matériaux et le protocole du fabricant) et mélanger par pipetage.
- Placer le tube PCR sur un thermocycleur et incuber pendant 4 h à 37 °C.
REMARQUE: La taille du tube est de 0,2 mL. Le volume de la solution est de 46 μL. La température du couvercle du thermocycleur est de 105 °C. - Ajouter 5 μL de tampon DNase I 10x (voir le tableau des matériaux) et ajouter 4 μL de DNase I (sans RNase, 4 U, voir le tableau des matériaux).
- Mélanger et incuber pendant 15 min à 37 °C et transférer l’échantillon dans un tube de 0,5 mL.
- Ajouter 300 μL/tube de thiocyanate-phénol-chloroforme de guanidinium (voir le tableau des matières) et mélanger par tourbillon doux.
REMARQUE : Le thiocyanate-phénol-chloroforme de guanidinium peut entraîner de graves brûlures chimiques par contact. Portez des gants, des lunettes de sécurité et une blouse de laboratoire. Utiliser uniquement avec une ventilation adéquate ou porter un respirateur approprié. Suivez les directives de sécurité de l’établissement. Jetez les articles de laboratoire usagés conformément aux directives de l’établissement. - Incuber pendant 5 min à R.T. sur un agitateur, puis conserver les échantillons à -80 °C jusqu’à 3 jours.
REMARQUE: Point d’arrêt sûr: L’expérience peut être arrêtée ici en toute sécurité jusqu’à 3 jours.
16. Purification de l’ARN
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase et la RNase. Durée : 2 h
- Ajouter 80 μL/tube de chloroforme à l’échantillon à partir de l’étape 15.7 et agiter vigoureusement le tube à la main pendant 15 s.
- Incuber pendant 2 à 15 min à température ambiante et centrifuger l’échantillon à 12 000 × g pendant 15 min à 4 °C.
- Prélever et transférer la phase aqueuse de l’échantillon (~200 μL) dans un nouveau tube de 1,5 mL.
- Ajouter 600 μL/tube de thiocyanate-phénol-chloroforme de guanidinium et transférer l’échantillon dans un tube de 1,5 mL.
- Ajouter 180 μL/tube de chloroforme et agiter vigoureusement le tube à la main pendant 15 s.
- Centrifuger l’échantillon à 12 000 × g pendant 10 min à 4 °C.
- Prélever et transférer la phase aqueuse de l’échantillon (~450 μL) dans un tube de 1,5 mL.
- Ajouter 60 μL/tube d’acrylamide linéaire (5 μg/μL) et ajouter 400 μL/tube d’isopropanol à 100 % à la phase aqueuse.
- Incuber le tube à température ambiante pendant 10 min et le centrifuger à 12 000 × g pendant 10 min à 4 °C.
- Retirez le surnageant avec précaution.
- Lavez la pastille d’ARN 3 fois avec 200 μL d’éthanol à 75% (eau sans EtOH/RNase), retirez le surnageant et séchez la pastille d’ARN à l’air.
REMARQUE : Ne laissez pas l’ARN sécher complètement car la pastille peut perdre de sa solubilité. - Resuspendre la pastille d’ARN dans 20 μL d’eau exempte de RNase contenant un inhibiteur de l’ARNase (1 μL/20 μL) et dissoudre la pastille par pipetage.
- Mesurer l’absorbance à 260 nm.
17. Transcription inverse
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 1 h
- Ajouter 7 μL/tube d’amorce de transcription inverse + mélange dNTP (voir les tableaux 1 et 3) et placer les tubes sur un cycleur thermique.
REMARQUE: La taille du tube est de 0,2 mL. Le volume de la solution est de 27 μL. La température du couvercle du thermocycleur est de 105 °C. - Chauffer à 65 °C pendant 5 min et placer les tubes sur de la glace pendant au moins 1 min.
- Ajouter 14 μL/tube de mélange maître à transcriptase inverse (voir tableau 1) et mélanger par pipetage.
- Placez les tubes sur un cycleur thermique, et incuberez-les à 55 °C pendant 10 min, puis à 80 °C pendant 10 min.
REMARQUE: La taille du tube est de 0,2 mL. Le volume de la solution est de 60 μL. La température du couvercle du thermocycleur est de 105 °C.
18. Deuxième synthèse du brin
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 2,5 h
- Ajouter 40 μL/tube d’eau ultrapure et diviser la solution de 100 μL en deux tubes de PCR de 0,2 mL (50 μL/tube).
- Ajouter 60 μL/tube de mélange de synthèse second brin (voir tableau 1) et placer les tubes sur un thermocycleur et exécuter le programme suivant: i) 95 °C pendant 5 min, ii) 95 °C pendant 30 s, iii) 60 °C pendant 30 s, iv) 72 °C pendant 30 s, v) répéter les étapes ii-iv 20 fois, et vi) maintenir à 4 °C.
REMARQUE: La taille du tube est de 0,2 mL. Le volume de la solution est de 110 μL. La température du couvercle du thermocycleur est de 105 °C. - Ajouter 4,4 μL/tube de 0,5 M d’EDTA (20 mM final) et conserver à -80 °C, pendant une nuit.
REMARQUE: Point d’arrêt sûr: L’expérience peut être arrêtée ici en toute sécurité jusqu’à quelques jours. - Purifier l’ADN par purification phénol-chloroforme et précipitation du polyéthylèneglycol (décrite à l’étape 14).
REMARQUE: Point d’arrêt sûr: L’expérience peut être arrêtée ici en toute sécurité pendant au moins une semaine.
19. Restriction de la digestion enzymatique et sélection de la taille
REMARQUE: La procédure suivante est effectuée sous un capot propre pour éviter la contamination par la DNase. Durée : 3 h (étapes 19.1-19.7)
- Mesurer la concentration d’ADN de l’ADN purifié (à partir de l’étape 18.4) en mesurant l’absorbance à 260 nm.
- Transférer 6 μg d’ADN dans un tube PCR, ajouter 30 μL de tampon de digestion 10x et ajuster le volume à 294 μL avec de l’eau ultrapure.
- Ajouter 6 μL d’enzyme de restriction BciVI et incuber à 37 °C pendant 1 h.
- Effectuer la précipitation de l’EtOH avec du polyacrylamide linéaire.
- Ajouter 60 μL/tube d’acétate de sodium 3 M (pH 5,2), puis ajouter 40 μL/tube d’acrylamide linéaire (5 mg/mL, voir le tableau des matières).
- Ajouter 400 μL/tube d’EtOH et incuber pendant une nuit à -20 °C.
REMARQUE: Point d’arrêt sûr: L’expérience peut être arrêtée ici en toute sécurité pendant une journée. - Centrifuger 12 000 × g pendant 10 min à 4 °C et retirer le surnageant.
- Lavez le granulé deux fois avec 80% d’EtOH et séchez le granulé.
- Dissoudre la pastille avec 20 μL de tampon 1xTE et ajouter 4 μL/tube de tampon frais 6x Gel Loading.
- Charger l’échantillon dans un gel d’agarose à 5 % / tampon TAE 0,5x et effectuer une électrophorèse (50 V pendant 40 min).
- Coupez et collectez le gel à plus de 50 pb (voir Figure 6) et extrayez l’ADN à l’aide d’un kit d’extraction de gel.
REMARQUE: Point d’arrêt sûr: L’expérience peut être arrêtée ici en toute sécurité pendant au moins une semaine. - Construire la bibliothèque de séquençage à l’aide d’une trousse de préparation de la bibliothèque d’ADN (voir le tableau des matériaux)16,17.
Résultats
Les cellules simples K562 ont été générées à l’aide du protocole décrit à l’étape 8 (voir la figure 5). Des cellules individuelles ont été intégrées dans la couche externe de la bille de polyacrylamide. L’ADN cellulaire a été coloré et visualisé à l’aide d’un colorant intercalateur pour la coloration de l’ADN.

Discussion
Cet article décrit le protocole étape par étape pour l’analyse multiépigénomique unicellulaire récemment rapportée utilisant des cellules uniques réutilisables7. Dans les paragraphes suivants, nous discutons des points critiques, en soulignant les limites potentielles du protocole.
L’un des points critiques tout au long du protocole (des étapes 7.2 à 13) est d’éviter la contamination par la DNase. Une seule cellule n’a que deux copies d’ADN génomi...
Déclarations de divulgation
Les Drs Ohnuki et Tosato sont les co-inventeurs d’un brevet intitulé « Methods for preparing a reusable single cell and methods for analysis the epigenome, transcriptome and genome of a single cell » (EP3619307 et US20200102604). La demande de brevet a été déposée en partie sur la base des résultats préliminaires liés à la technologie décrite dans le manuscrit actuel. L’invention ou les inventions décrites et revendiquées dans cette demande de brevet ont été faites alors que les inventeurs étaient des employés à temps plein du gouvernement des États-Unis. Par conséquent, en vertu de la partie 7 du Code of Federal Regulations, 45, tous les droits, titres et intérêts relatifs à cette demande de brevet ont été ou devraient être cédés par la loi au gouvernement des États-Unis. Le gouvernement des États-Unis transfère une partie des redevances qu’il reçoit à ses inventeurs employés en vertu de l’article 3710c du Code des États-Unis.
Remerciements
Nous remercions les Drs David Sanchez-Martin et Christopher B. Buck pour leurs commentaires au cours de la phase de conceptualisation du projet. Nous remercions également le Genomics Core, le Center for Cancer Research, le National Cancer Institute, les National Institutes of Health pour leur aide dans les expériences préliminaires, et le Collaborative Bioinformatics Resource, CCR, NCI, NIH pour leurs conseils en matière d’analyse informatique. Nous remercions Mme Anna Word pour son aide à l’optimisation des ADN polymérases utilisées dans la méthode. Ce travail a utilisé les ressources informatiques du cluster Biowulf (http://hpc.nih.gov) du NIHHPC. Ce projet est soutenu par le programme intra-muros du Center for Cancer Research, du National Cancer Institute, des National Institutes of Health, du NCI Director’s Innovation Award (#397172) et des fonds fédéraux du National Cancer Institute sous le contrat n ° HHSN261200800001E. Nous remercions les Drs Tom Misteli, Carol Thiele, Douglas R. Lowy et tous les membres du Laboratoire d’oncologie cellulaire pour leurs commentaires productifs.
matériels
| Name | Company | Catalog Number | Comments |
| 10x CutSmart buffer | New England BioLabs | B6004 | 10x Digestion buffer |
| 200 proof ethanol | Warner-Graham Company | 200 proof | Ethanol |
| 5-Hydroxymethylcytosine (5-hmC) Monoclonal Antibody [HMC/4D9] | Epigentek | A-1018-100 | Anti-5hmC |
| Acridine Orange/Propidium Iodide Stain | Logos Biosystems | F23001 | Cell counter |
| Acrylamide solution, 40% in H2O, for molecular biology | MilliporeSigma | 01697-500ML | 40% acrylamide solution |
| All-in-One Fluorescence Microscope BZ-X710 | Keyence | BZ-X710 | Scanning microscope |
| Amicon Ultra-0.5 Centrifugal Filter Unit | MilliporeSigma | UFC510024 | Ultrafiltration cassette |
| Ammonium persulfate for molecular biology | MilliporeSigma | A3678-100G | Ammonium persulfate powder |
| Anhydrous DMF | Vector laboratories | S-4001-005 | Anhydrous N,N-dimethylformamide (DMF) |
| Anti-RNA polymerase II CTD repeat YSPTSPS (phospho S5) antibody [4H8] | Abcam | ab5408 | Anti-Pol II |
| Anti-TRAP220/MED1 (phospho T1457) antibody | Abcam | ab60950 | Anti-Med1 |
| BciVI | New England BioLabs | R0596L | BciVI |
| Bovine Serum Albumin solution, 20 mg/mL in H2O, low bioburden, protease-free, for molecular biology | MilliporeSigma | B8667-5ML | 20% BSA (Table 7) |
| Bst DNA Polymerase, Large Fragment | New England BioLabs | M0275L | Bst DNA polymerase |
| BT10 Series 10 µl Barrier Tip | NEPTUNE | BT10 | P10 low-retention tip |
| CellCelector | Automated Lab Solutions | N/A | Automated single cell picking robot |
| CellCelector 4 nl nanowell plates for single cell cloning, Plate S200-100 100K, 24 well,ULA | Automated Lab Solutions | CC0079 | 4 nL nanowell plate |
| Chloroform | MilliporeSigma | Chloroform | |
| Corning Costar 96-Well, Cell Culture-Treated, Flat-Bottom Microplate | Corning | 3596 | Flat-bottom 96-well plates |
| Deep Vent (exo-) DNA Polymerase | New England BioLabs | M0259L | Exo- DNA polymerase |
| DNA LoBind Tubes, 0.5 mL | Eppendorf | 30108035 | 0.5 mL DNA low-binding tube |
| DNA Oligo, 1st random primer | Integrated DNA Technologies | N/A, see Table 3 | 1st random primer |
| DNA Oligo, 2nd random primer Cell#01 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#02 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#03 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#04 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#05 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#06 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#07 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#08 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#09 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#10 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#11 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd random primer Cell#12 | Integrated DNA Technologies | N/A, see Table 3 | 2nd random primer |
| DNA Oligo, 2nd synthesis primer | Integrated DNA Technologies | N/A, see Table 3 | 2nd synthesis primer |
| DNA Oligo, Ligation Adaptor | Integrated DNA Technologies | N/A, see Table 3 | Ligation Adaptor |
| DNA Oligo, Reverse Transcription primer | Integrated DNA Technologies | N/A, see Table 3 | Reverse Transcription primer |
| DNase I (RNase-free) | New England BioLabs | M0303L | DNase I (RNase-free, 4 U). |
| DNase I Reaction Buffer | New England BioLabs | B0303S | 10x DNase I buffer (NEB) |
| dNTP Mix (10 mM each) | Thermo Fisher | R0192 | 10 mM dNTPs |
| Fetal Bovine Serum, USA origin, Heat-inactivated | MilliporeSigma | F4135-500ML | Fetal bovine serum |
| HiScribe T7 High Yield RNA Synthesis Kit | New England BioLabs | E2040S | In-vitro-transcription master mix |
| Histone H3K27ac antibody | Active motif | 39133 | Anti-H3K27ac |
| Histone H3K27me3 antibody | Active motif | 39155 | Anti-H3K27me3 |
| IgG from rabbit serum | Millipore Sigma | I5006-10MG | Control IgG |
| Iron oxide(II,III) magnetic nanopowder, 30 nm avg. part. size (TEM), NHS ester functionalized | MilliporeSigma | 747467-1G | NHS ester functionalized 30 nm iron oxide powder |
| K-562 | American Type Culture Collection (ATCC) | CCL-243 | cells |
| Linear Acrylamide (5 mg/mL) | Thermo Fisher | AM9520 | Linear Acrylamide |
| LUNA-FL Dual Fluorescence Cell Counter | Logos Biosystems | L20001 | Cell counter |
| LUNA Cell Counting Slides, 50 Slides | Logos Biosystems | L12001 | Cell counter |
| Mineral oil, BioReagent, for molecular biology, light oil | MilliporeSigma | M5904-500ML | Mineral oil |
| N,N,N′,N′-Tetramethylethylenediamine for molecular biology | MilliporeSigma | T7024-100ML | N,N,N′,N′-Tetramethylethylenediamine |
| NaCl (5 M), RNase-free | Thermo Fisher | AM9760G | 5M NaCl |
| NanoDrop Lite | Thermo Fisher | 2516 | Microvolume spectrophotometer |
| NEST 2 mL 96-Well Deep Well Plate, V Bottom | Opentrons | N/A | 2 mL deep well 96-well plate |
| Non-skirted 96-well PCR plate | Genesee Scientific | 27-405 | 96-well PCR plate |
| NuSive GTG Agarose | Lonza | 50081 | Agarose |
| OmniPur Acrylamide: Bis-acrylamide 19:1, 40% Solution | MilliporeSigma | 1300-500ML | 40%Acrylamide/Bis-acrylamide |
| OT-2 lab robot | Opentrons | OT2 | Automated liquid handling robot |
| Paraformaldehyde, EM Grade, Purified, 20% Aqueous Solution | Electron Microscopy Sciences | 15713 | 20% Pararmaldehyde |
| PBS (10x), pH 7.4 | Thermo Fisher | 70011044 | 10x PBS |
| PIPETMAN Classic P1000 | GILSON | F123602 | A P1000 pipette |
| Protein LoBind Tubes, 1.5 mL | Eppendorf | 925000090 | 1.5 mL Protein low-binding tube |
| QIAgen Gel Extraction kit | Qiagen | 28706 | A P1000 pipette |
| Quant-iT PicoGreen dsDNA Assay | Thermo Fisher | P11495 | dsDNA specific intercalator dye |
| Quick Ligation kit | New England BioLabs | M2200L | T4 DNA ligase (NEB) |
| RNaseOUT Recombinant Ribonuclease Inhibitor | Thermo Fisher | 10777019 | RNAse inhibitor |
| S-4FB Crosslinker (DMF-soluble) | Vector laboratories | S-1004-105 | Succinimidyl 4-formylbenzoate (S-4FB) |
| S-HyNic | Vector laboratories | S-1002-105 | Succinimidyl 6-hydrazinonicotinate acetone hydrazone (S-HyNic) |
| Sodium Acetate, 3 M, pH 5.2, Molecular Biology Grade | MilliporeSigma | 567422-100ML | 3M Sodium acetate (pH 5.2) |
| Sodium bicarbonate, 1M buffer soln., pH 8.5 | Alfa Aesar | J60408 | 1M sodium bicarbonate buffer, pH 8.5 |
| Sodium phosphate dibasic for molecular biology | MilliporeSigma | S3264-250G | Na2HPO4 |
| Sodium phosphate monobasic for molecular biology | MilliporeSigma | S3139-250G | NaH2PO4 |
| SuperScript IV reverse transcriptase | Thermo Fisher | 18090050 | Reverse transcriptase |
| SYBR Gold Nucleic Acid Gel Stain (10,000x Concentrate in DMSO) | Thermo Fisher | S11494 | An intercalator dye for DNA |
| T4 DNA Ligase Reaction Buffer | New England BioLabs | B0202S | 10x T4 DNA ligase reaction buffer |
| ThermoPol Reaction Buffer Pack | New England BioLabs | B9004S | 10x TPM-T buffer (Tris-HCl/Pottasium chloride/Magnesium sulfate/Triton X-100) |
| TRIzol LS reagent | Thermo Fisher | 10296-028 | Guanidinium thiocyanate-phenol-chloroform extraction |
| TruSeq Nano DNA library prep kit | Illumina | 20015965 | A DNA library preparation kit (see also the manufacturer's instruction) |
| Ultramer DNA Oligo, Anti-5hmC_Ab#005 | Integrated DNA Technologies | N/A, see Table 3 | An amine-modified DNA probe for antibody |
| Ultramer DNA Oligo, Anti-H3K27ac_Ab#002 | Integrated DNA Technologies | N/A, see Table 3 | An amine-modified DNA probe for antibody |
| Ultramer DNA Oligo, Anti-H3K27me3_Ab#003 | Integrated DNA Technologies | N/A, see Table 3 | An amine-modified DNA probe for antibody |
| Ultramer DNA Oligo, Anti-Med1_Ab#004 | Integrated DNA Technologies | N/A, see Table 3 | An amine-modified DNA probe for antibody |
| Ultramer DNA Oligo, Anti-Pol II_Ab#006 | Integrated DNA Technologies | N/A, see Table 3 | An amine-modified DNA probe for antibody |
| Ultramer DNA Oligo, Control IgG_Ab#001 | Integrated DNA Technologies | N/A, see Table 3 | An amine-modified DNA probe for control IgG |
| UltraPure 0.5 M EDTA, pH 8.0 | Thermo Fisher | 15575020 | 0.5M EDTA, pH 8.0 |
| UltraPure DNase/RNase-Free Distilled Water | Thermo Fisher | 10977023 | Ultrapure water |
| Zeba Splin Desalting Columns, 7K MWCO, 0.5 mL | Thermo Fisher | 89882 | Desalting column |
Références
- Perkel, J. M. Single-cell analysis enters the multiomics age. Nature. 595 (7868), 614-616 (2021).
- Clark, S. J., et al. scNMT-seq enables joint profiling of chromatin accessibility DNA methylation and transcription in single cells. Nature Communications. 9 (1), 781 (2018).
- Ma, S., et al. Chromatin potential identified by shared single-cell profiling of RNA and Chromatin. Cell. 183 (4), 1103-1116 (2020).
- Zhu, C., et al. An ultra high-throughput method for single-cell joint analysis of open chromatin and transcriptome. Nature Structural & Molecular Biology. 26 (11), 1063-1070 (2019).
- Zhu, C., et al. Joint profiling of histone modifications and transcriptome in single cells from mouse brain. Nature Methods. 18 (3), 283-292 (2021).
- Mimitou, E. P., et al. Scalable, multimodal profiling of chromatin accessibility, gene expression and protein levels in single cells. Nature Biotechnology. 39 (10), 1246-1258 (2021).
- Ohnuki, H., Venzon, D. J., Lobanov, A., Tosato, G. Iterative epigenomic analyses in the same single cell. Genome Research. 31 (10), 1819-1830 (2021).
- Tosato, G., Ohnuki, H. . Methods of preparing a re-usable single cell and methods fro analyzing the epigenome, transcriptome, and genome of a single cell. , (2021).
- Harada, A., et al. A chromatin integration labelling method enables epigenomic profiling with lower input. Nature Cell Biology. 21 (2), 287-296 (2019).
- Egelhofer, T. A., et al. An assessment of histone-modification antibody quality. Nature Structural & Molecular Biology. 18 (1), 91-93 (2011).
- Marina, R. J., et al. TET-catalyzed oxidation of intragenic 5-methylcytosine regulates CTCF-dependent alternative splicing. EMBO Journal. 35 (3), 335-355 (2016).
- Weast, R. C., Weast, R. C., Astle, M. J., Beyer, W. H., et al. . Handbook of chemistry and physics: a ready-reference book of chemical and physical data. 56th edn. , (1975).
- Aydin, S. A short history, principles, and types of ELISA, and our laboratory experience with peptide/protein analyses using ELISA. Peptides. 72, 4-15 (2015).
- Gan, S. D., Patel, K. R. Enzyme immunoassay and enzyme-linked immunosorbent assay. Journal of Investigative Dermatology. 133 (9), 12 (2013).
- Porstmann, T., Kiessig, S. T. Enzyme immunoassay techniques. An overview. Journal of Immunological Methods. 150 (1-2), 5-21 (1992).
- Chao, H. P., et al. Systematic evaluation of RNA-Seq preparation protocol performance. BMC Genomics. 20 (1), 571 (2019).
- Song, Y., et al. A comparative analysis of library prep approaches for sequencing low input translatome samples. BMC Genomics. 19 (1), 696 (2018).
- Rotem, A., et al. Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state. Nature Biotechnology. 33 (11), 1165-1172 (2015).
- Ku, W. L., et al. Single-cell chromatin immunocleavage sequencing (scChIC-seq) to profile histone modification. Nature Methods. 16 (4), 323-325 (2019).
- Carter, B., et al. Mapping histone modifications in low cell number and single cells using antibody-guided chromatin tagmentation (ACT-seq). Nature Communications. 10 (1), 3747 (2019).
- Maskell, D. P., et al. Structural basis for retroviral integration into nucleosomes. Nature. 523 (7560), 366-369 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon