Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Combinaison d’organoïdes humains et de la technologie Organ-on-a-Chip pour modéliser des fonctionnalités spécifiques à la région intestinale
Dans cet article
Résumé
Les organoïdes intestinaux dérivés de la biopsie et les technologies d’organes sur puce sont combinés dans une plate-forme microphysiologique pour récapituler la fonctionnalité intestinale spécifique à la région.
Résumé
La muqueuse intestinale est une barrière physique et biochimique complexe qui remplit une myriade de fonctions importantes. Il permet le transport, l’absorption et le métabolisme des nutriments et des xénobiotiques tout en facilitant une relation symbiotique avec le microbiote et en limitant l’invasion des micro-organismes. L’interaction fonctionnelle entre les différents types de cellules et leur environnement physique et biochimique est vitale pour établir et maintenir l’homéostasie des tissus intestinaux. La modélisation de ces interactions complexes et de la physiologie intestinale intégrée in vitro est un objectif formidable avec le potentiel de transformer la façon dont de nouvelles cibles thérapeutiques et de nouveaux candidats médicaments sont découverts et développés.
Les technologies Organoïdes et Organ-on-a-Chip ont récemment été combinées pour générer des puces intestinales pertinentes pour l’homme adaptées à l’étude des aspects fonctionnels de la physiologie intestinale et de la physiopathologie in vitro. Les organoïdes dérivés des biopsies du petit (duodénum) et du gros intestin sont ensemencés dans le compartiment supérieur d’une puce d’organe, puis se développent avec succès en monocouches tout en préservant les caractéristiques cellulaires, moléculaires et fonctionnelles distinctes de chaque région intestinale. Les cellules endothéliales microvasculaires spécifiques du tissu intestinal humain sont incorporées dans le compartiment inférieur de la puce de l’organe pour recréer l’interface épithéliale-endothéliale. Cette nouvelle plate-forme facilite l’exposition luminale aux nutriments, aux médicaments et aux micro-organismes, permettant des études sur le transport intestinal, la perméabilité et les interactions hôte-microbe.
Ici, un protocole détaillé est fourni pour l’établissement de puces intestinales représentant le duodénum humain (puce du duodénum) et le côlon (puce du côlon), et leur culture ultérieure sous des déformations continues et de type péristaltisme. Nous démontrons des méthodes d’évaluation du métabolisme des médicaments et de l’induction du CYP3A4 dans la puce du duodénum à l’aide d’inducteurs et de substrats prototypiques. Enfin, nous fournissons une procédure étape par étape pour la modélisation in vitro de la perturbation de la barrière médiée par l’interféron gamma (IFNγ) (syndrome de l’intestin perméable) dans une puce du côlon, y compris des méthodes d’évaluation de l’altération de la perméabilité paracellulaire, des changements dans la sécrétion de cytokines et du profilage transcriptomique des cellules de la puce.
Introduction
L’intestin humain est un organe complexe et multitâche capable de s’auto-régénérer. Il est divisé en petit et gros intestin. La fonction principale de l’intestin grêle est de digérer davantage les aliments provenant de l’estomac, d’absorber tous les nutriments et de transmettre les résidus au gros intestin, qui récupère l’eau et les électrolytes. L’intestin grêle est en outre divisé en plusieurs régions anatomiquement distinctes: le duodénum, le jéjunum et l’iléon, chacune étant adaptée pour remplir des fonctions spécifiques. Par exemple, le duodénum aide à décomposer le chyme (contenu de l’estomac) pour permettre la bonne absorption des nutriments impliquant des protéines, des glucides, des vitamines et des minéraux dans le jéjunum. Cette partie proximale de l’intestin grêle est également le principal site d’absorption et de métabolisme des médicaments intestinaux, et elle se caractérise par l’expression plus élevée des enzymes métabolisant les médicaments (par exemple, le CYP3A4) par rapport à leur expression dans l’iléon et le côlon1. En plus de son rôle principal dans la digestion et l’absorption des nutriments, l’intestin est également une barrière efficace contre les contenus luminaux potentiellement nocifs, tels que les micro-organismes pathogènes, les métabolites microbiens, les antigènes alimentaires et les toxines 2,3. Il est à noter que le côlon humain est habité par un grand nombre de micro-organismes, dépassant de loin celui des cellules totales du corps humain, qui offrent de nombreux avantages pour la nutrition, le métabolisme et l’immunité. Par conséquent, le maintien de l’intégrité de la barrière muqueuse formée par les cellules épithéliales intestinales est essentiel pour permettre la relation symbiotique entre le microbiote intestinal et les cellules hôtes en les séparant physiquement afin d’éviter une activation inutile des cellules immunitaires2. En outre, la mort cellulaire intestinale programmée joue un rôle essentiel en tant que mécanisme d’autoprotection empêchant les cellules infectées de persister ou de proliférer – disséminant ainsi des agents pathogènes potentiels3 – tandis que l’auto-renouvellement continu de l’épithélium intestinal tous les quatre à sept jours compense la perte cellulaire assurant l’intégrité de la barrière et l’homéostasie tissulaire. Les déficiences des fonctions intestinales décrites, y compris l’absorption des nutriments, l’intégrité de la barrière ou le déséquilibre dans la mort et l’auto-renouvellement des cellules intestinales, peuvent entraîner le développement d’une gamme de troubles gastro-intestinaux, y compris la malnutrition et les maladies inflammatoires de l’intestin (MII)2,3.
Auparavant, des modèles animaux et des lignées cellulaires intestinales transformées dérivées du cancer ont été utilisés pour étudier les fonctions physiologiques et physiopathologiques du tissu intestinal humain. Cependant, les préoccupations de plus en plus importantes concernant la transférabilité de la recherche animale à l’homme, causées par la présence de disparités importantes entre les deux espèces, ont mis en évidence la nécessité de méthodes alternatives pertinentes pour l’homme4. Les lignées cellulaires intestinales in vitro couramment utilisées comprennent les cellules T84, Caco-2 et HT29. Bien qu’ils imitent certains aspects de la fonction de barrière intestinale et du transport membranaire, ils se caractérisent par une expression altérée des enzymes métabolisant les médicaments5, des récepteurs de surface et des transporteurs4. De plus, ils manquent de spécificité du segment intestinal et ne parviennent pas à récapituler la complexité de l’épithélium intestinal, chaque modèle ne contenant qu’un seul des cinq types de cellules épithéliales présentes dans l’intestin6.
Récemment, des cultures organoïdes intestinales humaines établies à partir de biopsies fraîches de l’intestin grêle et du côlon 7,8 ou de cellules souches pluripotentes induites (CSPi)9 ont été introduites comme modèles expérimentaux alternatifs ayant le potentiel de compléter, de réduire et peut-être de remplacer l’expérimentation animale à l’avenir. Bien que les CSPi puissent être obtenues de manière non invasive, l’établissement d’organoïdes à partir de CSPi nécessite l’utilisation de protocoles complexes et longs (avec plusieurs étapes expérimentales) et génère des cultures ressemblant à du tissu fœtal humain. En revanche, les organoïdes dérivés de la biopsie sont hautement évolutifs, car ils peuvent tirer parti de la capacité de renouvellement inhérente du tissu intestinal et peuvent être indéfiniment passés et propagés in vitro. Il est important de noter que les organoïdes dérivés de la biopsie maintiennent les caractéristiques spécifiques de la maladie et de la région intestinale du tissu primaire à partir duquel ils ont été développés et émulent la diversité cellulaire de l’épithélium intestinal. Les organoïdes peuvent être utilisés comme avatars spécifiques au patient in vitro pour démêler la biologie et la pathogenèse de divers troubles gastro-intestinaux et améliorer leur gestion thérapeutique. Bien que les organoïdes intestinaux aient atteint un degré impressionnant de fonctionnalité physiologique, ils ne parviennent toujours pas à reproduire la complexité des organes natifs en raison de leur manque de composants stromaux critiques – y compris les vaisseaux sanguins, le tissu conjonctif, les nerfs périphériques et les cellules immunitaires – ainsi que de stimulation mécanique. Les paramètres mécaniques, tels que l’écoulement, la contrainte de cisaillement, l’étirement et la pression, sont connus pour influencer la morphogenèse tissulaire et l’homéostasie in vivo et il a déjà été démontré qu’ils amélioraient la maturation des cellules in vitro10,11,12,13. Un autre inconvénient important des systèmes organoïdes est l’inaccessibilité de la lumière et, par conséquent, du côté apical de l’épithélium. Cela représente un défi pour l’étude de divers mécanismes associés à l’expression polarisée des transporteurs d’ions et de médicaments, aux interactions hôte-microbiome et aux tests de toxicité pharmaceutique. Enfin, les cultures organoïdes souffrent d’une variabilité considérable de taille, de morphologie et de fonction, en raison de la nature stochastique du processus d’auto-organisation in vitro et des choix de destin cellulaire. Par conséquent, pour réaliser le plein potentiel des organoïdes intestinaux dans la modélisation des maladies, le dépistage des médicaments et la médecine régénérative, il est nécessaire d’explorer de nouvelles stratégies qui réduisent la variabilité du développement organoïde, améliorent l’accès au compartiment luminal et intègrent les interactions cellule-cellule manquantes.
La technologie Organ-on-a-Chip a introduit de nombreuses techniques pour l’incorporation de forces mécaniques et d’écoulement de fluide dans les cultures de cellules intestinales in vitro. Cependant, étant donné que la plupart des études initiales de preuve de concept ont utilisé des lignées cellulaires dérivées du cancer qui ne présentaient pas une diversité cellulaire suffisante, la pertinence de ces systèmes a été remise en question. Récemment, nous avons combiné de manière synergique des organoïdes intestinaux et la technologie organ-on-a-chip pour incorporer les meilleures caractéristiques de chaque approche dans un système in vitro 14,15,16. La puce intestinale résultante récapitule l’architecture multicellulaire de l’épithélium intestinal, la présence d’une interface tissulaire épithéliale-endothéliale et les forces mécaniques de l’écoulement et de l’étirement des fluides, permettant l’émulation des fonctions au niveau des organes in vitro. De plus, l’utilisation d’organoïdes dérivés de tissus primaires (qui peuvent être échantillonnés dans différentes régions de l’intestin humain) comme matériau de départ augmente la polyvalence de ce modèle, car des puces représentant le duodénum, le jéjunum, l’iléon et le côlon humains peuvent être établies à la suite de procédures d’ensemencement et de culture similaires. Il est important de noter que les puces intestinales permettent d’évaluer en temps réel: l’intégrité de la barrière intestinale; activité de la bordure de la brosse et des enzymes du métabolisme des médicaments; production de mucines; sécrétion de cytokines; et l’interaction des cellules intestinales avec des micro-organismes pathogènes et commensaux, comme le démontrent les rapports publiés précédemment. Notamment, lorsque les puces intestinales ont été établies à l’aide d’organoïdes générés à partir de tissus de différents individus, ces modèles ont capturé la variabilité attendue entre donneurs dans les réponses fonctionnelles à divers médicaments et traitements. Dans l’ensemble, la fusion des organoïdes avec la technologie Organ-on-a-Chip ouvre la porte à des modèles plus avancés, personnalisés et pertinents in vivo qui pourraient améliorer la pertinence physiologique et la précision des résultats in vitro ainsi que leur extrapolation aux humains. Ici, un protocole détaillé est présenté pour établir la puce intestinale et son application dans les études des fonctions physiologiques des deux segments intestinaux: duodénum et côlon. Tout d’abord, les méthodes d’évaluation de l’activité de l’enzyme métabolisant le médicament CYP3A4 dans la puce du duodénum, ainsi que son induction par des composés prototypiques tels que la rifampicine et la vitamine D3, sont décrites. Deuxièmement, les étapes nécessaires pour modéliser « l’intestin qui fuit » dans la puce du côlon sont décrites dans le protocole, la perturbation de la barrière épithéliale étant effectuée à l’aide de cytokines caractéristiques impliquées dans la pathogenèse de la MII. En bref, les organoïdes dérivés de biopsies humaines sont propagés in vitro, soumis à une digestion enzymatique et introduits dans le canal supérieur de la puce. En présence d’une perfusion continue avec des milieux enrichis en facteur de croissance, ils se développent en une monocouche épithéliale confluente avec une architecture 3D et une surface cellulaire apicale facilement accessible. Le compartiment inférieur de la puce « vasculaire » est ensemencé de cellules endothéliales microvasculaires isolées du petit ou du gros intestin. L’épithélium et l’endothélium sont séparés par une membrane extensible poreuse, qui facilite les interactions paracrines entre les deux tissus et, lorsqu’ils sont soumis à des déformations cycliques, émule les mouvements de l’intestin humain semblables à ceux du péristaltisme. La co-culture est maintenue dans les conditions d’écoulement dynamique générées par la perfusion luminale et vasculaire avec des milieux de culture cellulaire appropriés. Enfin, nous décrivons de nombreux types d’essais et d’analyses des paramètres qui peuvent être effectués directement sur puce ou à partir d’effluents de culture cellulaire échantillonnés.
Access restricted. Please log in or start a trial to view this content.
Protocole
REMARQUE: Toutes les cultures cellulaires doivent être manipulées à l’aide d’une technique aseptique appropriée.
Les organoïdes intestinaux humains utilisés dans cette étude ont été obtenus de l’Université Johns Hopkins et toutes les méthodes ont été effectuées conformément aux directives et réglementations approuvées. Tous les protocoles expérimentaux ont été approuvés par le Conseil d’examen institutionnel de l’Université Johns Hopkins (IRB #NA 00038329).
1. Préparation des réactifs de culture cellulaire
- Préparer le milieu de croissance organoïde humain en suivant les instructions du fabricant (Table des matériaux) et le compléter avec 100 μg/mL de primocine.
- Préparer le milieu de croissance des cellules endothéliales microvasculaires humaines en suivant les instructions du fabricant (table des matériaux) et compléter avec 1:20 vol/vol FBS et 50 μg/ml au lieu de 100 μg/ml de primocine.
- Remettre en suspension Y-27632 (inhibiteur de rho-kinase) dans du BSA stérile à 0,1 % dans du DPBS pour préparer une solution mère de 10 mM. Aliquote dans des microtubes stériles et conserver à -20 °C jusqu’à 6 mois.
- Remettre en suspension CHIR99021 (inhibiteur de GSK-3) dans du diméthylsulfoxyde stérile (DMSO) pour préparer une solution mère de 5 mM. Aliquote dans des microtubes stériles et conserver à -20 °C jusqu’à 6 mois.
- Préparer la solution de digestion organoïde humaine en mélangeant une solution de dissociation organoïde 1:1 vol/vol (Table des matériaux) avec une solution de DPBS (Table des matériaux) et en la complétant avec une solution mère de 10 μM de Y-27632. Ce réactif doit être rendu frais pour chaque utilisation.
2. Culture de cellules endothéliales microvasculaires intestinales humaines (HIMEC)
- Initier la culture HIMEC (Table of Materials) 7 jours avant de l’ensemencer sur la puce. Seuls les HIMECs aux passages 1 à 6 peuvent être utilisés pour l’ensemencement des copeaux.
- Ajouter 5 mL de la solution du facteur de fixation (table des matériaux) dans une fiole T150; incuber à 37 °C pendant 1 min et jeter la solution.
- Ajouter 19 mL de milieu de croissance des cellules endothéliales préchauffé à température ambiante (voir étape 1.2) à la fiole.
- Décongeler un flacon congelé d’HIMEC (2 millions de cellules/flacon) dans un bain-marie à 37°C.
- Transférer le contenu du flacon dans la fiole et bercer doucement la fiole pour répartir uniformément les cellules. Placez la fiole dans un incubateur à 37 °C pendant la nuit pour permettre aux cellules endothéliales d’adhérer.
- Remplacer le milieu par 20 mL de milieu de croissance des cellules endothéliales préchauffé à température ambiante le lendemain et tous les 3 jours par la suite. Les cultures devraient atteindre 90% de confluence le jour de la récolte cellulaire pour les expériences sur puce.
3. Microfabrication et préparation de la puce
- Obtenir des copeaux d’un fournisseur commercial (Tableau des matériaux). Déballez les copeaux et placez-les dans le berceau à copeaux placé dans une boîte de Pétri carrée (Table des matériaux). Étiquetez la face avant de chaque support de puce avec les conditions expérimentales (Figure 1A).
REMARQUE: Bien que le protocole présenté repose sur l’utilisation d’une puce et d’un instrumentation spécifiques disponibles dans le commerce, il existe plusieurs dispositifs microfluidiques proposés par différents fournisseurs, qui peuvent offrir des avantages alternatifs, mais n’ont pas la capacité d’intégrer stretch. En outre, la microfabrication des organes sur puces peut être effectuée « en interne » selon les procédures décrites dans Huh et al.21.
4. Activation et revêtement ECM de la membrane
- Préparation de la solution d’activation
- Amener les réactifs ER-1 et ER-2 (table des matériaux) à température ambiante pour les équilibrer avant utilisation. L’ER-1 est sensible à la lumière et doit être manipulé dans l’obscurité.
- Reconstituer l’ER-1 dans 10 mL de réactif ER-2 pour obtenir une concentration finale de 0,5 mg/mL et confirmer que l’ER-1 est complètement dissous.
- Activation de surface
- Poussez doucement 50 μL de la solution ER-1 à travers les deux canaux de la puce (Figure 1A).
- Retirez tout excès de solution ER-1 de la surface de la puce à l’aide d’un aspirateur.
- Inspectez les puces pour vous assurer que toutes les puces ont reçu la solution ER-1. En cas de bulles d’air, introduisez plus de solution ER-1 jusqu’à ce que les bulles soient complètement éliminées.
- Incuber les copeaux à l’intérieur de la chambre de la lampe UV (Table des matériaux) pendant 15 min. Vérifiez que la lampe UV est réglée sur le réglage Cohérent.
- Ramenez les puces activées par ER-1 dans l’armoire de biosécurité (BSC). Aspirer la solution ER-1 des deux canaux. Laver tout résidu de la solution ER-1 avec 200 μL d’ER-2.
- Poussez 200 μL de solution saline tamponnée au phosphate (DBPS) stérile de Dulbecco à travers les canaux (Table des matériaux) et laissez le DBPS dans les canaux jusqu’à l’étape suivante.
- Préparation de matrice extracellulaire (ECM) et revêtement membranaire
- Préparation de solutions mères de composants ECM
- Reconstituer le collagène IV (Table des matériaux) et la fibronectine (Table des matériaux) à l’aide d’eau stérile de qualité de culture cellulaire (Table des matériaux) jusqu’à une concentration finale de 1 mg/mL. Préparer les aliquotes et les conserver à -20°C jusqu’à l’utilisation.
- Préparation de la solution de travail ECM pour le revêtement membranaire
- Préparez 1,5 mL de chaque solution ECM pour le canal supérieur et inférieur pour chaque 12 puces à enduire. La solution ECM doit toujours être préparée juste avant utilisation.
- Pour le canal supérieur, mélanger le collagène IV et la matrice solubilisée de la membrane basale (BMM) (Table des matériaux) dans du DBPS stérile froid à 200 μg/mL et 100 μg/mL, respectivement. Pour le canal inférieur, mélanger le collagène IV et la fibronectine dans du DPBS stérile froid à 200 μg/mL et 30 μg/mL, respectivement.
- Revêtement ECM des puces
- Aspirez le DBPS des deux canaux des puces et remplacez-le par la solution de travail ECM appropriée pour chaque canal (Figure 1A).
- Inspectez chaque puce pour vous assurer qu’elle a reçu la solution de revêtement. En cas de bulles d’air, poussez à travers plus de solution de revêtement jusqu’à ce que toutes les bulles soient complètement éliminées.
- Ajouter le DPBS stérile au berceau de la puce et placer la boîte de Petri contenant les copeaux dans un incubateur à 37 °C. Incuber pendant la nuit pour permettre aux protéines ECM de former des liaisons ioniques avec la membrane PDMS activée. Si vous le souhaitez, les cellules peuvent être ensemencées à tout moment entre 2 h et 1 jour après l’enrobage des copeaux. Alternativement, les copeaux enrobés peuvent être stockés à 4 ° C pendant la nuit, suivis d’une incubation nocturne à 37 ° C et d’un ensemencement de copeaux.
- Préparation de solutions mères de composants ECM
5. Ensemencement de cellules endothéliales microvasculaires intestinales (HIMEC) dans le canal inférieur de la puce
REMARQUE: Les HIMEC de l’intestin grêle et du côlon sont ensemencés dans le canal inférieur du duodénum et de la puce du côlon, respectivement.
- Préparer des chips
- Transférez les puces revêtues d’ECM au BSC. Aspirer doucement le revêtement ECM des deux canaux des puces, puis laver les deux canaux avec 200 μL de milieu de croissance organoïde et de milieu de croissance cellulaire endothéliale, respectivement.
- Conservez les copeaux lavés dans un incubateur à 37 °C jusqu’à ce que les cellules endothéliales soient ensemencées.
- Récolter les cellules endothéliales
- Apportez la fiole de culture HIMEC au BSC et lavez-la à l’aide de DPBS stérile.
- Ajouter 3 mL de solution de dissociation à la fiole et la placer dans l’incubateur à 37°C pendant 2 min pour permettre un détachement complet de la cellule.
- Recueillir les cellules dans un tube conique de 15 mL et ajouter un milieu de croissance cellulaire endothélial jusqu’à ce qu’il atteigne 10 mL. Échantillon de 15 à 20 μL de la suspension cellulaire pour le comptage cellulaire. Centrifuger à 150 x g pendant 5 min.
- Aspirer soigneusement le surnageant et suspendre à nouveau les HIMECs à une densité de 8-10 x 106 cellules /mL dans les milieux de croissance des cellules endothéliales.
- Ensemencez des cellules endothéliales dans le canal inférieur de la puce
- Apportez la boîte de Petri contenant les copeaux à l’ESB et retirez délicatement tous les milieux du canal inférieur à l’aide d’une pipette de 1 000 μL.
- Introduisez 10 à 15 μL de suspension HIMEC dans le canal inférieur de la puce. Inspectez la puce juste après l’ensemencement pour assurer une densité d’ensemencement optimale (80 % à 90 % de la couverture) et une distribution homogène des cellules dans le canal (Figure 1D). Si la densité d’ensemencement est supérieure ou inférieure à celle prévue ou inégale, lavez le canal 2x avec 200 μL de milieu de croissance cellulaire endothéliale et répétez la procédure d’ensemencement.
- Inverser la boîte de Petri immédiatement après l’ensemencement de chaque lot de six copeaux pour permettre la fixation des cellules endothéliales sur le côté inférieur de la membrane PDMS. Placer les boîtes de Petri dans l’incubateur à 37 °C pendant 30 min à 1 h, ou jusqu’à ce que les HIMEC dans le canal inférieur se soient fixés à la membrane (Figure 1D).
- Laver les copeaux
- Poussez doucement 200 μL de milieux de croissance des cellules endothéliales à travers l’entrée du canal inférieur pour éliminer toutes les cellules non attachées et reconstituer les nutriments dans le milieu.
- Laver les cellules qui ne se sont pas fixées dans le canal supérieur avec 200 μL de milieu de croissance organoïde complété par 5 μM CHIR99021 et 10 μM Y-27632.
- Placez les copeaux dans l’incubateur à 37 °C jusqu’à ce que vous procédiez à l’ensemencement des cellules épithéliales.
6. Ensemencement de fragments organoïdes dans le canal supérieur de la puce
REMARQUE: Les organoïdes isolés à partir de biopsies de diverses régions intestinales peuvent être cultivés dans la puce intestinale7. Suivez les procédures décrites dans Fujii et al. pour l’isolement des cryptes intestinales humaines et l’établissement de cultures organoïdes22. Ici, les organoïdes duodénaux et coliques sont utilisés pour générer respectivement les puces du duodénum et du côlon. Compte tenu de la grande variabilité d’un lot à l’autre et d’un donneur à l’autre dans la formation et la croissance des organoïdes, il est suggéré d’effectuer une évaluation pilote de la densité cellulaire dans la culture en suspension organoïde (format de plaque à 24 puits) pour atteindre la densité d’ensemencement optimale de 8 millions de cellules / mL.
- Évaluation pilote du nombre de cellules/puits de culture organoïde statique.
REMARQUE: La procédure suivante consiste uniquement à déterminer le nombre de puits de culture de suspension organoïde requis pour l’ensemencement des copeaux. Les cellules individuelles résultantes ne doivent pas être utilisées pour l’ensemencement des copeaux.- Aspirer soigneusement le surnageant de trois puits de la plaque de culture organoïde. Ajouter 500 μL de solution de dissociation BMM glacée (Table des matériaux) à chaque puits et utiliser un grattoir en plastique pour détacher le BMM solubilisé du plastique.
- Recueillir la suspension organoïde dans un tube conique stérile à faible teneur en protéines à l’aide d’une pipette glacée de 1 000 μL. Incuber sur de la glace pendant 60 min, en mélangeant toutes les 15 minutes en secouant doucement le tube. Centrifuger à 300 x g pendant 5 min à 4°C.
- Aspirer le surnageant et ajouter 2 mL de trypsine. Incuber à 37°C pendant 30 min pour digérer les organoïdes au niveau de la cellule unique et ajouter 10 mL de milieu de croissance organoïde complet pour arrêter la réaction enzymatique. Centrifuger à 300 x g pendant 5 min à 4°C.
- Remettre en suspension la pastille cellulaire dans 1 mL de milieu de croissance organoïde complet et compter le nombre total de cellules à l’aide d’un hématocytomètre selon les méthodes standard.
- Préparation de fragments organoïdes et leur ensemencement dans la puce
REMARQUE: Les procédures suivantes peuvent être utilisées pour ensemencer des organoïdes duodénaux ou coliques dans le canal supérieur de la puce. La mise en place d’une monocouche épithéliale avec une cytoarchitecture 3D pertinente in vivo dépend de la présence d’un flux15,23 et d’un ensemencement réussi de fragments organoïdes.- Aspirer soigneusement le milieu à partir du nombre de puits de la culture organoïde statique, ce qui est suffisant, comme déterminé à l’étape 6.1, pour atteindre une densité d’ensemencement finale de 8 millions de cellules / mL. Ajouter 500 μL de solution de dissociation BMM glacée à chaque puits.
- Détachez le BMM solubilisé de la surface des puits à l’aide d’un grattoir en plastique ou d’une pipette de 1 000 μL. Recueillir la suspension dans un tube conique à faible teneur en protéines de 15 mL.
- Conservez la suspension sur de la glace pendant 60 min, en mélangeant toutes les 15 minutes en secouant doucement le tube. Procéder à la centrifugation à 300 x g pendant 5 min à 4°C. Une pastille organoïde bien définie doit être visible après la centrifugation. Si une couche de gel transparente est observée au-dessus de la pastille cellulaire (résidus de BMM solubilisé) (Figure 1B), procédez comme suit.
- Mélanger le surnageant et la pastille cellulaire et ajouter un volume égal de solution de dissociation BMM dans le tube. Mélanger doucement et conserver sur de la glace pendant 10 minutes supplémentaires, et procéder à la centrifugation à 300 x g pendant 5 min à 4 °C.
- Si nécessaire, répétez l’étape 6.2.3.1 jusqu’à ce qu’aucun résidu de BMM solubilisé ne soit présent.
- Jeter le surnageant et remettre en suspension la pastille organoïde avec une solution de digestion organoïde (voir étape 1.5). Utilisez un volume suffisant de solution de digestion organoïde pour assurer une immersion complète de la pastille. 2 mL de solution conviennent à environ 2 400 à 12 000 organoïdes de taille moyenne (Figure 1D, Post-ensemencement). Incuber à 37°C pendant 1-3 min.
- Ajouter Advanced DMEM/F-12 au tube pour arrêter la réaction enzymatique. Utilisez quatre fois le volume de la solution de digestion utilisée. Centrifuger à 300 x g pendant 5 min à 4°C.
- Aspirer le surnageant et remettre en suspension la pastille de fragments organoïdes dans un milieu de croissance organoïde complet complété par 5 μM CHIR99021 et 10 μM Y-27632 pour atteindre 8 millions de cellules/mL. Préparer des aliquotes de 360 μL de la suspension dans des tubes stériles de 1,5 mL à faible teneur en protéines pour éviter la réduction de la densité cellulaire due à la fixation sur les parois.
- Retirez le milieu du canal supérieur des copeaux enduits. Ajouter 30 μL de suspension cellulaire dans le canal supérieur de chaque puce. Incuber les copeaux pendant la nuit dans un incubateur à 37 °C pour permettre aux fragments organoïdes d’adhérer à la membrane revêtue d’ECM (Figure 1D).
7. Culture dynamique de la puce intestinale - initiation et maintien du flux et des mouvements semblables à ceux du péristaltisme
- Préparation et dégazage des milieux
- Pour maintenir un flux laminaire constant à travers la puce, laissez la température du média s’équilibrer à la température ambiante, puis soumettez-le à une filtration sous vide pendant 10 minutes à l’aide d’une pompe à vide et de tubes coniques filtrants en PVDF (dégazage du média).
- Amorçage de modules portables
REMARQUE: Les modules portables sont des réservoirs qui agissent comme une interface entre les puces et le module de culture permettant de répéter l’échantillonnage et le dosage des milieux.- Ouvrez les modules portables, dans le BSC, et placez-les sur les plateaux des modules de culture, qui sont des conteneurs spécialisés pour l’alignement des modules portables.
- Ajouter 3 mL de milieu de croissance organoïde complet prééquilibré complété par 5 μM CHIR99021 et 10 μM Y-27632 au réservoir d’entrée supérieur et 3 mL de milieu de croissance cellulaire endothéliale prééquilibré dans le réservoir d’entrée inférieur (Figure 1C). Ajouter 300 μL du même milieu dans les réservoirs de sortie respectifs.
- Amenez les plateaux dans l’incubateur à 37 °C et glissez-les dans le module de culture (Figure 1C). Utilisez les commandes sur l’écran du module de culture pour exécuter le « Prime Cycle » (1 min de long). Lorsque la barre d’état indique « Prêt », le cycle principal est terminé. Répétez le cycle Prime pour vous assurer que suffisamment de gouttelettes se sont formées pour connecter avec succès les puces.
- Assurez-vous que tous les modules portables sont amorcés et ont des gouttelettes de média visibles.
- Introduction du flux
REMARQUE: L’écoulement est généralement initié 24 heures après l’ensemencement des fragments organoïdes pour permettre aux cellules épithéliales intestinales de se fixer fermement à la membrane.- Lavez les deux canaux des copeaux ensemencés avec 200 μL du milieu respectif pour éliminer les bulles ou les débris cellulaires. Laissez de petites gouttelettes de média sur les orifices après le lavage. Faites glisser le porte-puce dans le module portable (Figure 1C).
- Placez les modules portables sur les plateaux, puis dans le module de culture. Utilisez les commandes sur l’écran du module de culture pour programmer les conditions de culture de puce d’organe appropriées (débit et étirement).
REMARQUE: Pour les conditions standard de culture de puces du duodénum et du côlon, réglez le débit à 30 μL / h15 et 60 μL / h16 respectivement, pour les canaux supérieur et inférieur. Cependant, les débits de chaque canal sont contrôlés indépendamment et peuvent être réglés entre 0 et 1 000 μL / h. Des seringues ou des pompes péristaltiques peuvent être utilisées, au lieu des modules de culture présentés ici, pour introduire un flux laminaire dans les puces microfluidiques. Cependant, l’établissement de connexions fluidiques fiables entre les puces et les pompes à l’aide de tubes et de connecteurs peut être techniquement difficile lorsque la perfusion simultanée de plusieurs puces est nécessaire. - Démarrez le « Cycle de régulation » (2 h de long) qui met sous pression le milieu de culture dans un module portable et une puce pour empêcher la nucléation des bulles d’air. Les conditions programmées reprendront après l’achèvement du cycle de régulation.
- Changement de média
- Préparer les milieux frais pour les deux canaux et les reconstituer toutes les 48 h en ajoutant 2 mL de milieu frais aux réservoirs d’entrée (figure 1C).
- Mettez les modules de culture en pause et apportez les plateaux à BSC. Aspirer les milieux dans tous les réservoirs et les reconstituer avec des milieux frais. Ramenez les plateaux et redémarrez le flux.
- Introduction de l’étirement
REMARQUE: Laissez les cellules croître jusqu’à 100% de confluence avant l’application de la souche cyclique. L’étirement est généralement introduit 3 jours après l’ensemencement ou 48 heures après le début de la culture fluidique. Une déformation cyclique à 2 % à la fréquence de 0,2 ou 0,15 Hz, respectivement pour la puce intestinale du duodénum11 et la puce intestinale du côlon24 , est appliquée pendant les 24 premières heures. Il est ensuite augmenté à 10 % pour la durée restante de la culture de puces intestinales afin de ressembler étroitement à la souche cyclique subie par les cellules épithéliales intestinales in vivo (Figure 1D)25. Le module de culture peut prendre en charge l’application d’une déformation cyclique de 2% à 12% et d’une fréquence allant jusqu’à 0,4 Hz.- Pour introduire l’étirement de la culture fluidique en cours, mettez le module de culture en pause. À l’aide des commandes à l’écran, modifiez les paramètres d’étirement sur 2 % d’étirement, fréquence de 0,2 ou 0,15 Hz et redémarrez le module de culture.
- Après 24 h, répétez l’étape 7.5.1 pour appliquer une fréquence d’étirement de 10 %, 0,2 ou 0,15 Hz.
8. Induction du CYP450 à l’aide d’inducteurs CYP prototypiques dans la puce du duodénum
REMARQUE: Le test d’induction du cytochrome P450 (CYP450) permet d’évaluer si le composé d’essai augmente les niveaux d’ARNm et / ou l’activité catalytique d’enzymes spécifiques du CYP450. Ici, nous décrivons le protocole pour l’évaluation de l’induction du CYP3A4 par la norme et le régulateur recommandés par l’industrie in vitro in vitro inducteurs , la rifampicine (RIF) et la 1,2-dihydroxy vitamine D3 (VD3). La méthode présentée peut être utilisée pour identifier le potentiel de divers composés d’essai à induire différentes isoformes du CYP450 dans le tissu intestinal humain. Des ensembles spécifiques d’amorces et de substrats de sonde devront être sélectionnés pour chaque isoforme enzymatique à évaluer.
- Exposition aux inducteurs du CYP
- Préparer des solutions mères de 20 mM RIF, 100 mM VD3 et 200 mM de testostérone (table des matériaux) en utilisant du DMSO stérile.
ATTENTION : La testostérone est une substance contrôlée de l’annexe III. Suivez les procédures réglementaires lors de la manipulation. - Préparer le milieu doseur avec des inducteurs de CYP en diluant les solutions mères dans un milieu de croissance organoïde complet et un milieu de croissance des cellules endothéliales pour atteindre un RIF de 20 μM, soit 100 nmol/L de VD3. Préparer le contrôle du véhicule en diluant le DMSO dans les milieux respectifs pour obtenir 0,1% de DMSO.
- Mettez le module de culture en pause et apportez les plateaux à BSC. Remplacer le média dans tous les réservoirs d’entrée par 2 mL de milieu de dosage avec inducteurs ou commande du véhicule. Retournez les modules portables dans le module de culture et redémarrez le débit à 30 μL/h.
- Après 24 h, remplacez le support par une solution inductrice et répétez les étapes 8.1.2 à 8.1.3. Les solutions inductrices doivent être préparées fraîches tous les jours et réapprovisionnées toutes les 24 heures au cours de l’expérience, qui dure généralement de 48 à 72 heures.
- Préparer des solutions mères de 20 mM RIF, 100 mM VD3 et 200 mM de testostérone (table des matériaux) en utilisant du DMSO stérile.
- Incubation avec un substrat prototypique (testostérone)
- Le jour de la récolte, préparez la solution de substrat de sonde de testostérone avec les inducteurs correspondants dans Advanced DMEM / F12 pour obtenir une concentration finale de 200 μM. Omettez l’ajout du sérum au milieu car cela pourrait interférer avec l’analyse LCMS.
- Apportez les plateaux à BSC et aspirez le milieu de dosage de tous les réservoirs. Lavez et remplacez les réservoirs d’entrée supérieur et inférieur par un milieu DMEM/F12 avancé chaud et un milieu de croissance des cellules endothéliales, respectivement.
- Retirez le milieu de lavage des réservoirs et remplacez-le par 1 mL de solution de substrat de sonde, préparé à l’étape 8.2.1. Perfuser les copeaux à un débit élevé de 1 000 μL/h pendant 5 min et aspirer les réservoirs de sortie supérieur et inférieur. Renvoyer les copeaux dans le module de culture et incuber pendant 1 h sous le débit constant de 300 μL/h.
- Analyse des données
- Prélèvement d’échantillons pour l’analyse LCMS
- Aliquote 200 μL de solution d’arrêt contenant de l’acétonitrile avec 0,1% d’acide formique dans des tubes pré-marqués de 1,5 mL et les placer sur de la glace. La composition de la solution d’arrêt LCMS peut varier en fonction du substrat qui doit être analysé.
- Une fois le traitement terminé, arrêtez le flux et ramenez les plateaux à BSC. Prélever 100 μL d’effluent dans le réservoir de sortie supérieur et l’ajouter au tube correspondant contenant la solution d’arrêt (figure 2A). Placez les tubes immédiatement sur de la glace carbonique. Conservez les échantillons à -80 °C avant de procéder à l’analyse.
- Extraire et analyser les échantillons en utilisant des techniques HPLC ou LC-MS/MS, en surveillant la formation de métabolite-6β-hydroxytestostérone (6β-OH-T). L’activité de l’enzyme CYP peut être exprimée sous forme de protéine pmol/min/mg où pmol fait référence à la quantité de métabolite (6β-OH-T) formée au cours de la réaction. La teneur totale en protéines par puce (protéine; mg) est déterminée en effectuant un essai de Bradford décrit à l’étape 8.3.2.

L’activité d’induction du pli est calculée par : activité CYP (induite) / activité CYP (véhicule). - Si vous collectez des échantillons pour l’analyse de l’ARNm, reportez-vous à l’étape 8.3.3.
- Lyse des cellules pour l’analyse de l’expression des protéines
REMARQUE: L’extraction des protéines des cellules sur puce est effectuée à l’aide d’un tampon de lyse des protéines, complété par des inhibiteurs de la protéase et de la phosphatase (Table des matériaux). Les protéines extraites sont ensuite quantifiées à l’aide du test de Bradford et utilisées dans l’analyse en aval.- Détachez les puces des modules portables et placez-les dans une boîte de Pétri. Lavez les deux canaux avec 200 μL de DPBS stérile.
- Bloquez la sortie du canal inférieur avec une pointe de pipette filtrante de 200 μL. Perfuser 50 μL de la solution de dissociation à travers le canal inférieur et incuber pendant 2 min à température ambiante.
- Inspecter pour confirmer le détachement complet des cellules endothéliales. Pipette de haut en bas 1-2 fois et retirer la solution de dissociation du canal. Répétez le lavage.
- Bloquez la sortie du canal supérieur avec une pointe de pipette filtrante de 200 μL. Perfuser 75 μL de tampon de lyse des protéines à travers le canal supérieur. Laissez l’embout de la pipette inséré, pour bloquer l’entrée du canal supérieur et incuber pendant 5 min à température ambiante. Pipettez de haut en bas 5 à 10 fois et recueillez les lysats cellulaires dans un tube pré-étiqueté de 1,5 mL.
- Répétez l’étape 8.3.2.4. jusqu’à ce qu’un détachement complet des cellules soit observé. Recueillir les lysats cellulaires dans le même tube de 1,5 mL et les stocker à -80 °C jusqu’à l’analyse.
- Extraire la fraction protéique selon des méthodes standard et quantifier la protéine totale à l’aide du test de Bradford (Table des matériaux).
- Lyse des cellules pour l’analyse de l’expression génique
REMARQUE: La lyse cellulaire sur puce pour l’extraction de l’ARN peut être réalisée à l’aide d’un tampon de lyse de l’ARN, complété par du 2-mercaptoéthanol à 0,1% (Table des matériaux). Alternativement, un tampon de lyse d’ARN à base de phénol peut être utilisé si des rendements élevés d’ARN de haute qualité adaptés à l’analyse NGS et microréseaux sont nécessaires.- Suivez les étapes 8.3.2.1 à 8.3.2.2 pour préparer la puce intestinale à la lyse.
- Bloquez la sortie du canal supérieur à l’aide d’une pointe de pipette filtrante de 200 μL. Perfuser 150 μL de tampon de lyse de l’ARN à travers le canal supérieur. Laissez l’embout de la pipette inséré pour bloquer l’entrée du canal supérieur. Incuber pendant 2 min à température ambiante. Pour la lyse à l’aide d’un tampon de lyse d’ARN à base de phénol, utilisez 350 μL du réactif.
- Pipettez de haut en bas 5 à 10 fois et recueillez les lysats cellulaires dans un tube pré-étiqueté de 1,5 mL. Répétez l’étape avec un autre tampon de lyse d’ARN de 150 μL et collectez. Conserver les lysats cellulaires à -80 °C jusqu’à l’analyse. En cas d’analyse ultérieure du séquençage de l’ARN, procéder à l’analyse dans les 1 mois suivant la lyse.
- Extraire l’ARN total des lysats cellulaires à l’aide d’un kit de purification de l’ARN.
- Transcrivez en ADNc à l’aide d’un kit de transcriptase inverse et effectuez une PCR en temps réel à l’aide d’un cycleur PCR en temps réel et des amorces et tampons appropriés. Quantifier les résultats à l’aide de la méthode 2-ΔΔCt .
- Prélèvement d’échantillons pour l’analyse LCMS
9. Perturbation de la barrière épithéliale à l’aide de cytokines pro-inflammatoires dans la puce du côlon
NOTE: Ce protocole décrit la perturbation de la barrière épithéliale intestinale par l’interféron gamma cytokine (IFNγ)26,27,28,29. La cytokine est dosée dans le canal inférieur de la puce du côlon compte tenu de l’expression basolatérale du récepteur IFNγ sur les cellules épithéliales intestinales. Le stimulus pro-inflammatoire est introduit dans la puce le jour 5 de la culture, dès quel’application P a été stabilisée en dessous de 0,5 x 10-6 cm / s. Un schéma posologique similaire peut être utilisé pour d’autres cytokines pro-inflammatoires et agents perturbateurs de barrière.
- Stimulation avec IFNγ
- Préparer une solution mère de 100 μg/mL d’IFNγ (Table des matériaux) dans de l’eau de culture cellulaire stérile. Conserver la solution mère à -80°C au cours de l’expérience. Pour chaque expérience, utilisez toujours un stock frais d’IFNγ et évitez plus de trois cycles de congélation et de dégel.
- Préparer la solution posologique d’IFNγ en diluant la solution mère dans un milieu de croissance des cellules endothéliales dégazées pour atteindre une concentration finale de 10-100 ng/mL.
- Apportez les plateaux à BSC et retirez le milieu des réservoirs d’entrée du canal inférieur et remplacez-le par 3 mL du milieu contenant de l’IFNγ. Actualisez quotidiennement le milieu posologique DE l’IFNγ.
- Placez les plateaux dans le module de culture et perfuser les copeaux à un débit élevé de 1 000 μL/h pendant 5 min pour initier le traitement IFNγ. Rétablissez le débit à 60 μL/h et poursuivez la culture fluidique.
- Analyse des données
- Évaluation de la fonction barrière épithéliale. La perméabilité apparente épithéliale (application P), ou fonction barrière, peut être mesurée à différents moments du post-traitement de la culture avec IFNγ, dans les milieux effluents des réservoirs d’entrée et de sortie des deux canaux. Le traceur fluorescent doit être ajouté au milieu de croissance organoïde du canal supérieur 4 h avant l’évaluation del’application P. En règle générale, 3 kDa Dextran Cascade Blue est utilisé comme traceur fluorescent et est ajouté dans le milieu 24 h avant.
- Mettez le module de culture en pause et apportez les plateaux au BSC.
- Étiquetez et préparez une plaque à paroi noire de 96 puits avec 100 μL de DPBS par puits. À l’aide d’une pipette multicanal de 200 μL, recueillir et ajouter 50 μL d’effluents de tous les réservoirs aux puits respectifs (figure 2B).
- Pour préparer la courbe standard, diluer le milieu contenant 100 μg/mL de 3 kDa dextran Cascade Blue 1:3 dans du DPBS. Effectuez ensuite des dilutions en série en utilisant une dilution trois fois supérieure du milieu de croissance des cellules endothéliales dans le DPBS.
- Lisez la fluorescence à une excitation de 375 nm et à une émission de 420 nm à l’aide d’un lecteur de plaques. Utilisez les valeurs OD mesurées pour calculer la perméabilité apparente (application P) comme suit :
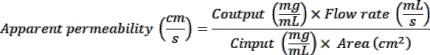
- Coloration par immunofluorescence des cellules sur puce
- Laver à l’aide de 200 μL de DPBS pour chaque canal, trois fois.
- Perfuser 200 μL de la solution fixatrice (4% PFA dans DPBS) dans chaque canal. Incuber pendant 15 min à température ambiante.
- Répétez l’étape de lavage 9.2.2.1. À ce stade, les copeaux lavés peuvent être conservés jusqu’à 7 jours à 4 °C.
- Perméabiliser les cellules à l’aide de 200 μL de solution de perméabilisation (0,1 % de Triton-X 100 dans du sérum d’âne normal (NDS) à 10 % dans le DPBS) pour chaque canal. Incuber pendant 30 min à température ambiante. Répétez l’étape de lavage 9.2.2.1.
- Bloquez les cellules de la puce en utilisant 200 μL de solution bloquante (10% de NDS dans le DPBS) pour chaque canal. Incuber pendant 1 h à température ambiante.
- Diluer les anticorps primaires dans une solution d’anticorps (5% NDS dans DPBS) comme suit: anti-Zonula Occludens 1 (ZO-1) (1:100, marqueur de jonction serrée épithéliale), anti-Occludine (1:100, marqueur de jonction serrée épithéliale), anti-Claudin 4 (1:100, marqueur de jonction serrée épithéliale), anti-E-cadhérine (1:100, marqueur de jonctions d’adhérence épithéliale (Tableau des matériaux). Perfuser 200 μL de la solution d’anticorps primaire à travers chaque canal et incuber pendant la nuit à 4 °C. Répétez l’étape de lavage 9.2.2.1.
- Diluer les anticorps secondaires dans une solution d’anticorps (1:300, 5% NDS dans DPBS). Perfuser 200 μL de cette solution à travers chaque canal et incuber pendant 2 h à température ambiante. Répétez l’étape de lavage 9.2.2.1. Si vous le souhaitez, la phalloïdine, un marqueur cytosquelettique, peut être ajoutée à la solution d’anticorps secondaire.
- Préparer une solution de 50 μg/mL de 4′,6-diamidino-2-phénylindole (DAPI) dans du DPBS et l’utiliser pour perfuser 200 μL à travers chaque canal. Incuber à température ambiante pendant 15 min. Répétez l’étape de lavage. À ce stade, les copeaux colorés peuvent être conservés jusqu’à 14 jours à 4 °C.
- Évaluation du clivage de la caspase 3
- Isoler et quantifier la quantité totale de protéines des cellules épithéliales comme décrit à l’étape 8.3.2. Omettez l’étape de lavage 8.3.2.1. Diluer les échantillons avec du DPBS à une concentration finale de 400 μg/mL.
- Quantifier les niveaux de caspase 3 totale et clivée à l’aide du kit de détection de caspase 3 en suivant le protocole du fabricant.
- Évaluation de la sécrétion de cytokines pro-inflammatoires
- Utiliser les effluents recueillis à la sortie des deux canaux de la puce du côlon pour quantifier les cytokines pro-inflammatoires en phase aiguë sécrétées, en suivant les protocoles fournis par le fabricant. Effectuer une dilution 5 fois pour le kit humain V-PLEX Vascular Injury Panel 2 et une dilution double pour le V-PLEX Human Proinflammatory Panel II.
- Évaluation de la fonction barrière épithéliale. La perméabilité apparente épithéliale (application P), ou fonction barrière, peut être mesurée à différents moments du post-traitement de la culture avec IFNγ, dans les milieux effluents des réservoirs d’entrée et de sortie des deux canaux. Le traceur fluorescent doit être ajouté au milieu de croissance organoïde du canal supérieur 4 h avant l’évaluation del’application P. En règle générale, 3 kDa Dextran Cascade Blue est utilisé comme traceur fluorescent et est ajouté dans le milieu 24 h avant.
Access restricted. Please log in or start a trial to view this content.
Résultats
La figure 1D résume la chronologie de la culture de la puce intestinale et illustre les cellules endothéliales intestinales et les organoïdes avant et après l’ensemencement sur la puce. De plus, il démontre les différences morphologiques distinctes entre le duodénum et les puces du côlon, mises en évidence par la présence de formations ressemblant à des villosités dans la puce du duodénum et représentatives de l’architecture de l’intestin grêle.
Access restricted. Please log in or start a trial to view this content.
Discussion
La combinaison de la technologie des organes sur puce et des organoïdes intestinaux est prometteuse pour une modélisation précise de la physiologie intestinale humaine et de la physiopathologie. Ici, nous fournissons un protocole étape par étape simple et robuste (décrit à la figure 1) pour l’établissement de la puce intestinale contenant l’épithélium de l’intestin grêle ou du côlon dérivé de la biopsie et des cellules endothéliales microvasculaires intestinales co-culti...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Gauri Kulkarni, Athanasia Apostolou, Lorna Ewart, Carolina Lucchesi et Magdalena Kasendra sont des employés actuels ou anciens d’Emulate Inc. et peuvent détenir des actions. Emulate Inc. est la société qui fabrique les dispositifs de puce d’organe et a publié des brevets pertinents pour le travail énoncé dans cet article.
Remerciements
Nous remercions le professeur Mark Donowitz pour avoir fourni les organoïdes dérivés de la biopsie intestinale et Brett Clair pour la conception des illustrations scientifiques de la puce, du module portable et du module de culture. Toutes les autres illustrations scientifiques ont été générées à l’aide du BioRender.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| small intestine Human Intestinal Microvascular Endothelial Cells | AlphaBioRegen | ALHE15 | 0.5 cells M/ml ; cryopreserved |
| colon Human Intestinal Microvascular Endothelial Cells | AlphaBioRegen | ALHE16 | 0.5 cells M/ml ; cryopreserved |
| Biopsy-derived Human Duodenal Organoids | John Hopkin's University | - | The organoids were provided by Professor Mark Donowitz (Institutional Review Board Number: NA_00038329). |
| Biopsy-derived Human Colonic Organoids | John Hopkin's University | - | The organoids were provided by Professor Mark Donowitz (Institutional Review Board Number: NA_00038329). |
| Zoë CM-1™ Culture Module | Emulate Inc. | - | Culture module |
| Orb-HM1™ Hub Module | Emulate Inc. | - | 5% CO2, vacuum stretch, and power supply |
| Chip-S1™ Stretchable Chip | Emulate Inc. | - | Organ-Chip |
| Pod™ Portable Modules | Emulate Inc. | - | Portable module |
| UV Light Box | Emulate Inc. | - | - |
| Chip Cradle | Emulate Inc. | - | 1 per square culture dish |
| Steriflip®-HV Filters | EMD Millipore | SE1M003M00 | 0.45 μm PVDF filter |
| Square Cell Culture Dish (120 x 120 mm) | VWR | 82051-068 | - |
| Handheld vacuum aspirator | Corning | 4930 | - |
| Aspirating pipettes | Corning / Falcon | 357558 | 2 mL, polystyrene, individually wrapped |
| Aspirating tips | - | Sterile (autoclaved) | |
| Serological pipettes | - | 2 mL, 5 mL, 10 mL, and 25 mL low endotoxin, sterile | |
| Pipette | P20, P200, P1000 and standard multichannel | ||
| Pipette tips | P20, P200, and P1000. | ||
| Conical tubes (Protein LoBind® Tubes) | Eppendorf | 0030122216; 0030122240 | 15 mL, 50 mL tubes |
| Eppendorf Tubes® lo-bind | Eppendorf | 022431081 | 1.5 mL tubes |
| 96 wells black walled plate | - | - | for epithelial permeability analysis |
| Microscope (with camera) | - | - | For bright-field imaging |
| Water bath (or beads) | - | - | Set to 37°C |
| Vacuum set-up | - | - | Minimum pressure: -70 kPa |
| Cell scrapers | Biotium | 220033 | |
| T75 flasks | BD Falcon | 353136 | Cell culture flask |
| Emulate Reagent-1 (ER-1) | Emulate Inc. | - | Chip coating solution |
| Emulate Reagent-2 (ER-2) | Emulate Inc. | - | Chip coating solution |
| Dulbecco’s PBS (DPBS) | Corning | 21-031-CV | 1X |
| Cell Culture Grade Water | Corning | MT25055CV | |
| Trypan blue | Sigma | 93595 | For cell counting |
| TryplE Express | ThermoFisher Scientific | 12604013 | Organoids dissociation and endothelium cells detachment solution |
| Advanced DMEM/F12 | ThermoFisher Scientific | 12634028 | Medium |
| IntestiCult™ Organoid Growth Medium (Human) | Stem Cell technologies | 06010 | Organoid Growth Medium |
| Endothelial Cell Growth Medium MV 2 | Promocell | C-22121 | Endothelial medium |
| Fetal bovine serum (FBS) | Sigma | F4135 | Serum |
| Primocin™ | InvivoGen | ANT-PM-1 | antimicrobial agent |
| Attachment Factor™ | Cell Systems | 4Z0-210 | coating solution for flask |
| Matrigel - Growth Factor Reduced | Corning | 356231 | Solubilized basement membrane matrix |
| Collagen IV | Sigma | C5533 | ECM component |
| Fibronectin | Corning | 356008 | ECM component |
| Y-27632 | Stem Cell technologies | 72304 | organoid media supplement |
| CHIR99021 | Reprocell | 04-0004-10 | organoid media supplement |
| Cell Recovery Solution | Corning | 354253 | Basement mebrane matrix dissociationsolution |
| Bovine Serum Albumin (BSA) | Sigma | A9576 | 30%, Sterile |
| Cell Culture Grade Water | Corning | MT25055CV | Sterile, Water |
| DMSO | Sigma | D2650 | solvent |
| 3KDa Dextran Cascade Blue | Invitrogen | D7132 | 10 mg powder |
| Rifampicin (RIF) | Sigma | Cat# R3501 | CYP inducer |
| Testosterone hydrate | Sigma | T1500 | CYP substrate |
| 1,25-dihyroxy Vitamin D3 (VD3) | Sigma | Cat# D1530 | CYP inducer |
| Acetonitrile with 0.1% (v/v) Formic acid | Sigma | 159002 | LCMS stop solution |
| IFNγ | Peprotech | 300-02 | |
| 4% Paraformaldehyde (PFA) | EMS | 157-4 | Fixative |
| Triton-X 100 | Sigma | T8787 | |
| Normal Donkey Serum (NDS) | Sigma | 566460 | |
| anti-Occludin | ThermoFisher Scientific | 33-1500 | tight junctions marker |
| anti-Claudin 4 | ThermoFisher Scientific | 36-4800 | tight junctions marker |
| anti-E-cadherin | Abcam | ab1416 | epithelial adherens junctions marker |
| anti-VE-cadherin | Abcam | ab33168 | endothelial adherent junctions marker |
| anti- Zonula Occludens 1 (ZO-1) | Thermo Fischer | 339194 | tight junctions marker |
| DAPI | ThermoFisher Scientific | 62248 | nuclear stain |
| 2-mercaptoethanol | Sigma | M6250 | |
| PureLink RNA Mini Kit | Invitrogen | 12183020 | RNA lysis, isolation and purification kit |
| SuperScript™ IV VILO™ Master Mix | Invitrogen | 11756050 | reverse transcriptase kit |
| TaqMan™ Fast Advanced Master Mix | Applied Biosystems | 4444557 | qPCR reagent |
| QuantStudio™ 5 Real-Time PCR System | Applied Biosystems | A28573 | Real-time PCR cycler |
| 18S primer | ThermoFisher Scientific | Hs99999901_s1 | Eukaryotic 18S rRNA |
| CYP3A4 primer | ThermoFisher Scientific | Hs00604506_m1 | Cytochrome family 3 subfamily A member 4 |
| Pierce™ Coomassie Plus (Bradford) Assay Kit | ThermoFisher Scientific | 23236 | Protein quantification kit |
| MSD Tris lysis buffer | Meso Scale Diagnostics | R60TX-3 | Protein lysis buffer |
| Cleaved/Total Caspase-3 Whole Cell Lysate Kit | Meso Scale Diagnostics | K15140D | Caspase 3 detection kit |
| V-PLEX Vascular Injury Panel 2 Human Kit | Meso Scale Diagnostics | K15198D | |
| V-PLEX Human Proinflammatory Panel II (4-Plex) | Meso Scale Diagnostics | K15053D | |
| Zeiss LSM 880 | Zeiss | - | Confocal microscope |
| Zeiss LD plan-Neofluar 20x/0.40 Korr M27 | Zeiss | - | 20X long-distance objective lenses |
| Zeiss AXIOvert.A1 | Zeiss | - | Brightfield microscope |
| Zeiss LD A-Plan 10X/0.25 Ph1 | Zeiss | - | 10X objective lenses |
Références
- Fritz, A., et al. Expression of clinically relevant drug-metabolizing enzymes along the human intestine and their correlation to drug transporters and nuclear receptors: An intra-subject analysis. Basic and Clinical Pharmacology and Toxicology. 124 (3), 245-255 (2019).
- Okumura, R., Takeda, K. Maintenance of intestinal homeostasis by mucosal barriers. Inflammation and Regeneration. 38 (1), 1-8 (2018).
- Delgado, M. E., Grabinger, T., Brunner, T. Cell death at the intestinal epithelial front line. FEBS Journal. 283 (14), 2701-2719 (2016).
- Mestas, J., Hughes, C. C. W. Of mice and not men: Differences between mouse and human immunology. The Journal of Immunology. 172 (5), 2731-2738 (2004).
- Sun, H., Chow, E. C. Y., Liu, S., Du, Y., Pang, K. S. The Caco-2 cell monolayer: Usefulness and limitations. Expert Opinion on Drug Metabolism and Toxicology. 4 (4), 395-411 (2008).
- Yu, H., et al. The contributions of human mini-intestines to the study of intestinal physiology and pathophysiology. Annual Review of Physiology. 79, 291-312 (2017).
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459 (7244), 262-265 (2009).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett’s epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Spence, J. R., et al. Directed differentiation of human pluripotent stem cells into intestinal tissue in vitro. Nature. 470 (7332), 105-110 (2011).
- Durel, J. F., Nerurkar, N. L. Mechanobiology of vertebrate gut morphogenesis. Current Opinion in Genetics and Development. 63, 45-52 (2020).
- Gayer, C. P., Basson, M. D. The effects of mechanical forces on intestinal physiology and pathology. Cellular Signalling. 21 (8), 1237-1244 (2009).
- Xu, Y., et al. Mechanical stimulation activates Piezo1 to promote mucin2 expression in goblet cells. Journal of Gastroenterology and Hepatology (Australia). 36 (11), 3127-3139 (2021).
- Navabi, N., McGuckin, M. A., Lindén, S. K. Gastrointestinal cell lines form polarized epithelia with an adherent mucus layer when cultured in semi-wet interfaces with mechanical stimulation. PLoS One. 8 (7), 68761(2013).
- Kasendra, M., et al. Development of a primary human Small Intestine-on-a-Chip using biopsy-derived organoids. Scientific Reports. 8 (1), 1-14 (2018).
- Kasendra, M., et al. Duodenum intestine-chip for preclinical drug assessment in a human relevant model. eLife. 9, 50135(2020).
- Apostolou, A., et al. A novel microphysiological colon platform to decipher mechanisms driving human intestinal permeability. Cellular and Molecular Gastroenterology and Hepatology. 12 (5), 1719-1741 (2021).
- Jalili-Firoozinezhad, S., et al. Author correction: A complex human gut microbiome cultured in an anaerobic intestine-on-a-chip. Nature Biomedical Engineering. 3 (7), 583(2019).
- Yin, J., et al. Fluid shear stress enhances differentiation of jejunal human enteroids in Intestine-Chip. American Journal of Physiology-Gastrointestinal and Liver Physiology. 320 (3), 258-271 (2021).
- Sontheimer-Phelps, A., et al. Human colon-on-a-chip enables continuous in vitro analysis of colon mucus layer accumulation and physiology. Cellular and Molecular Gastroenterology and Hepatology. 9 (3), 507-526 (2020).
- Tovaglieri, A., et al. Species-specific enhancement of enterohemorrhagic E. coli pathogenesis mediated by microbiome metabolites. Microbiome. 7 (1), 43(2019).
- Huh, D., et al. Microfabrication of human organs-on-chips. Nature Protocols. 8 (11), 2135-2157 (2013).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nature Protocols. 10 (10), 1474-1485 (2015).
- Kim, H. J., Huh, D., Hamilton, G., Ingber, D. E. Human gut-on-a-chip inhabited by microbial flora that experiences intestinal peristalsis-like motions and flow. Lab on a Chip. 12 (12), 2165-2174 (2012).
- Sarna, S. K. Colonic motility: From bench side to bedside. Morgan & Claypool Life Sciences. , San Rafael (CA). (2010).
- Basson, M. D. Paradigms for mechanical signal transduction in the intestinal epithelium - category: Molecular, cell, and developmental biology. Digestion. 68 (4), 217-225 (2003).
- Wang, F., et al. Interferon-gamma and tumor necrosis factor-alpha synergize to induce intestinal epithelial barrier dysfunction by up-regulating myosin light chain kinase expression. The American Journal of Pathology. 166 (2), 409-419 (2005).
- Nava, P., et al. Interferon-γ regulates intestinal epithelial homeostasis through converging β-Catenin signaling pathways. Immunity. 32 (3), 392-402 (2010).
- Madara, J. L., Stafford, J. Interferon-γ directly affects barrier function of cultured intestinal epithelial monolayers. Journal of Clinical Investigation. 83 (2), 724-727 (1989).
- Bruewer, M., et al. Proinflammatory cytokines disrupt epithelial barrier function by apoptosis-independent mechanisms. The Journal of Immunology. 171 (11), 6164-6172 (2003).
- Jones, S. C., et al. Adhesion molecules in inflammatory bowel disease. Gut. 36 (5), 724-730 (1995).
- Uhlar, C. M., Whitehead, A. S. Serum amyloid A, the major vertebrate acute-phase reactant. European Journal of Biochemistry. 265 (2), 501-523 (1999).
- Pérez-González, C., Ceada, G., Matejčić, M., Trepat, X. Digesting the mechanobiology of the intestinal epithelium. Current Opinion in Genetics & Development. 72, 82-90 (2022).
- In, J., et al. Enterohemorrhagic escherichia coli reduces mucus and intermicrovillar bridges in human stem cell-derived colonoids. Cellular and Molecular Gastroenterology and Hepatology. 2 (1), 48-62 (2016).
- Grassart, A., et al. Bioengineered human organ-on-chip reveals intestinal microenvironment and mechanical forces impacting shigella infection. Cell Host and Microbe. 26 (3), 435-444 (2019).
- Kerns, S. J., et al. Human immunocompetent organ-on-chip platforms allow safety profiling of tumor-targeted t-cell bispecific antibodies. eLife. 10, 67106(2021).
- Bhatia, S. N., Ingber, D. E. Microfluidic organs-on-chips. Nature Biotechnology. 32 (8), 760-772 (2014).
- Ingber, D. E. Reverse engineering human pathophysiology with organs-on-chips. Cell. 164 (6), 1105-1109 (2016).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon