Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Sonde β-galactosidase associée à la sénescence fluorescente rouge lointain pour l’identification et l’enrichissement de cellules tumorales sénescentes par cytométrie en flux
Dans cet article
Résumé
Un protocole de quantification fluorescente par cytométrie en flux des cellules cancéreuses sénescentes induites par des médicaments de chimiothérapie en culture cellulaire ou dans des modèles tumoraux murins est présenté. Les procédures facultatives comprennent le co-immunomarquage, la fixation d’échantillons pour faciliter l’analyse de grands lots ou de points temporels, et l’enrichissement de cellules sénescentes viables par tri cytométrique en flux.
Résumé
La sénescence cellulaire est un état d’arrêt prolifératif induit par des dommages biologiques qui s’accumulent normalement au fil des ans dans les cellules vieillissantes, mais qui peut également émerger rapidement dans les cellules tumorales en réponse aux dommages induits par divers traitements contre le cancer. La sénescence des cellules tumorales est généralement considérée comme indésirable, car les cellules sénescentes deviennent résistantes à la mort et bloquent la rémission tumorale tout en exacerbant la tumeur maligne et la résistance au traitement. Par conséquent, l’identification des cellules tumorales sénescentes est d’un intérêt continu pour la communauté de recherche sur le cancer. Il existe divers tests de sénescence, dont beaucoup sont basés sur l’activité du marqueur de sénescence bien connu, la bêta-galactosidase associée à la sénescence (SA-β-Gal).
Typiquement, le test SA-β-Gal est réalisé à l’aide d’un substrat chromogène (X-Gal) sur des cellules fixes, avec le dénombrement lent et subjectif des cellules sénescentes « bleues » par microscopie optique. Des tests améliorés utilisant des substrats fluorescents SA-β-Gal perméables aux cellules, y compris C12-FDG (vert) et DDAO-Galactoside (DDAOG; rouge lointain), ont permis l’analyse de cellules vivantes et l’utilisation de plates-formes d’analyse fluorescentes à haut débit, y compris des cytomètres en flux. C12-FDG est une sonde bien documentée pour SA-β-Gal, mais son émission fluorescente verte chevauche l’autofluorescence cellulaire intrinsèque (AF) qui survient pendant la sénescence en raison de l’accumulation d’agrégats de lipofuscine. En utilisant la sonde rouge lointain SA-β-Gal DDAOG, l’autofluorescence cellulaire verte peut être utilisée comme paramètre secondaire pour confirmer la sénescence, ajoutant ainsi de la fiabilité au test. Les canaux de fluorescence restants peuvent être utilisés pour la coloration de la viabilité cellulaire ou l’immunomarquage fluorescent facultatif.
En utilisant la cytométrie en flux, nous démontrons l’utilisation de DDAOG et de l’autofluorescence de la lipofuscine comme test à double paramètre pour l’identification des cellules tumorales sénescentes. La quantification du pourcentage de cellules sénescentes viables est effectuée. Si vous le souhaitez, une étape facultative d’immunomarquage peut être incluse pour évaluer les antigènes de surface cellulaire d’intérêt. Les cellules sénescentes identifiées peuvent également être triées cytométriquement en flux et collectées pour analyse en aval. Les cellules sénescentes collectées peuvent être immédiatement lysées (par exemple, pour des tests immunologiques ou des analyses omiques) ou cultivées.
Introduction
Les cellules sénescentes s’accumulent normalement dans les organismes au fil des ans au cours du vieillissement biologique normal, mais peuvent également se développer rapidement dans les cellules tumorales en réponse aux dommages induits par divers traitements contre le cancer, y compris la radiothérapie et la chimiothérapie. Bien qu’elles ne prolifèrent plus, les cellules tumorales sénescentes induites par le traitement (TIS) peuvent contribuer à la résistance au traitement et entraîner une récidive 1,2,3. Les facteurs sécrétés par les cellules TIS peuvent exacerber la tumeur maligne en favorisant l’évasion immunitaire ou les métastases 4,5. Les cellules TIS développent des phénotypes complexes et spécifiques au contexte, des profils métaboliques modifiés et des réponses immunitaires uniques 6,7,8. Par conséquent, l’identification et la caractérisation des cellules tumorales TIS induites par diverses approches de traitement du cancer est un sujet d’intérêt continu pour la communauté de recherche sur le cancer.
Pour détecter les cellules tumorales TIS, les tests de sénescence conventionnels sont largement utilisés, principalement basés sur la détection d’une activité accrue de l’enzyme marqueur de sénescence, la bêta-galactosidase lysosomale GLB19. La détection à un pH lysosomal presque neutre (plutôt qu’acide) permet une détection spécifique de la bêta-galactosidase associée à la sénescence (SA-β-Gal)10. Un test SA-β-Gal standard utilisé depuis plusieurs décennies utilise X-Gal (5-bromo-4-chloro-3-indolyl-β-D-galactopyranoside), un substrat chromogène bleu de la bêta-galactosidase, pour détecter la SA-β-Gal dans les cellules fixes par microscopieoptique 11. Le test X-Gal permet la confirmation visuelle qualitative du TIS à l’aide de réactifs et d’équipements de laboratoire couramment disponibles. Un microscope optique transmis de base est le seul instrument nécessaire pour évaluer la présence du chromogène bleu. Cependant, la procédure de coloration X-Gal peut manquer de sensibilité, nécessitant parfois plus de 24 heures pour que la couleur se développe. La coloration est suivie d’un score subjectif à faible débit des cellules sénescentes individuelles basé sur le comptage des cellules présentant un certain niveau d’intensité du chromogène bleu au microscope optique. Comme X-Gal est imperméable aux cellules, ce test nécessite des cellules fixées au solvant, qui ne peuvent pas être récupérées pour une analyse en aval. Lorsque vous travaillez avec des échantillons limités d’animaux ou de patients, cela peut être un inconvénient majeur.
Des dosages SA-β-Gal améliorés utilisant des substrats enzymatiques fluorescents perméants aux cellules, y compris le C 12-FDG (5-dodécanoylaminofluorescéine Di-β-D-Galactopyranoside, vert) et le DDAOG (9H-(1,3-dichloro-9,9-diméthylacridin-2-one-7-yl) β-D-galactopyranoside, rouge lointain) ont déjà paru dans la littérature12,13,14,15. La structure chimique de la sonde et les caractéristiques optiques du DDAOG sont illustrées à la figure supplémentaire S1. Ces sondes perméables permettent l’analyse de cellules vivantes (plutôt que fixes), et les sondes fluorescentes plutôt que chromogènes facilitent l’utilisation de plates-formes d’analyse fluorescentes rapides à haut débit, y compris des instruments de dépistage à haute teneur et des cytomètres en flux. Les cytomètres de flux de tri permettent la récupération de populations enrichies de cellules sénescentes vivantes à partir de cultures cellulaires ou de tumeurs pour une analyse en aval (par exemple, Western blotting, ELISA ou « omiques »). L’analyse de fluorescence fournit également un signal quantitatif, permettant une détermination plus précise du pourcentage de cellules sénescentes dans un échantillon donné. Des sondes fluorescentes supplémentaires, y compris des sondes de viabilité et des anticorps marqués au fluorophore, peuvent facilement être ajoutées pour l’analyse multiplexée de cibles au-delà de SA-β-Gal.
Semblable au DDAOG, le C12-FDG est une sonde fluorescente pour SA-β-Gal, mais son émission fluorescente verte chevauche la FA cellulaire intrinsèque, qui survient lors de la sénescence en raison de l’accumulation d’agrégats de lipofuscine dans les cellules16. En utilisant la sonde DDAOG rouge lointain, la FA cellulaire verte peut être utilisée comme paramètre secondaire pour confirmer la sénescence17. Cela améliore la fiabilité du test en utilisant un deuxième marqueur en plus du SA-β-Gal, qui peut souvent être peu fiable en tant que marqueur unique de la sénescence18. Comme la détection de la FA endogène dans les cellules sénescentes est une approche sans marquage, il s’agit d’un moyen rapide et simple d’étendre la spécificité de notre test basé sur DDAOG.
Dans ce protocole, nous démontrons l’utilisation de DDAOG et de la FA comme test rapide de cytométrie en flux à double paramètre pour l’identification de cellules tumorales TIS viables à partir de cultures in vitro ou isolées de tumeurs traitées par des médicaments établies chez la souris (Figure 1). Le protocole utilise des fluorophores compatibles avec une large gamme d’analyseurs et de trieurs de cytométrie en flux commerciaux standard (tableau 1). La quantification du pourcentage de cellules sénescentes viables à l’aide de l’analyse par cytométrie en flux standard est activée. Si vous le souhaitez, une étape facultative d’immunomarquage peut être effectuée pour évaluer les antigènes de surface cellulaire d’intérêt en même temps que la sénescence. Les cellules sénescentes identifiées peuvent également être enrichies à l’aide de la méthodologie standard de tri cellulaire activé par fluorescence (FACS).
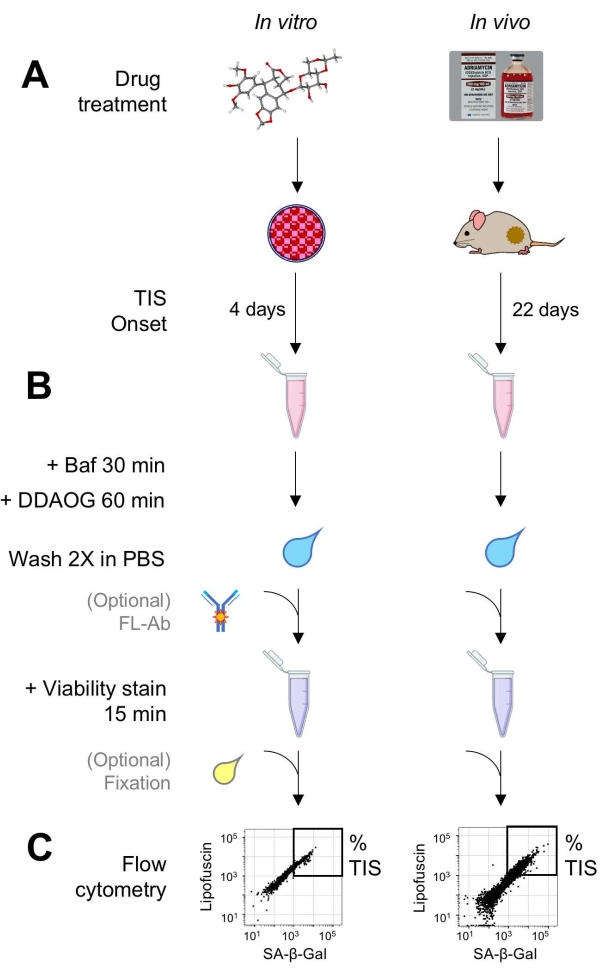
Figure 1 : Flux de travail expérimental. Un schéma résumant les points clés du test DDAOG. (A) Un médicament inducteur de TIS est ajouté à des cellules cultivées de mammifères ou administré à des souris porteuses de tumeurs. Un délai est alors prévu pour l’apparition du TIS: pour les cellules, 4 jours après le traitement; pour les souris, 22 jours au total, avec trois traitements tous les 5 jours plus 7 jours de récupération. Les cellules sont récoltées ou les tumeurs sont dissociées en suspension. (B) Les échantillons sont traités avec Baf pour ajuster le pH lysosomal pour la détection de SA-β-Gal pendant 30 min; ensuite, la sonde DDAOG est ajoutée pendant 60 minutes pour détecter SA-β-Gal. Les échantillons sont lavés 2x dans du PBS, et une tache de viabilité est brièvement ajoutée (15 min). En option, les échantillons peuvent être colorés avec des anticorps fluorescents dans des canaux de fluorescence ouverts et / ou fixés pour une analyse ultérieure. (C) Les échantillons sont analysés à l’aide d’un cytomètre en flux standard. Les cellules viables sont visualisées dans des diagrammes de points montrant le DDAOG rouge (indiquant SA-β-Gal) par rapport à l’autofluorescence verte (lipofuscine). Une porte pour déterminer le pourcentage de cellules TIS est établie en fonction des échantillons témoins non traités (non représentés). Si un cytomètre de tri (FACS) est utilisé, les cellules TIS peuvent être collectées et remises en culture pour d’autres tests in vitro ou lysées et traitées pour des essais de biologie moléculaire. Abréviations : DDAO = 9H-(1,3-dichloro-9,9-diméthylacridin-2-one); DDAOG = DDAO-Galactoside; TIS = sénescence induite par la thérapie; FL-Ab = anticorps conjugué au fluorophore; Baf = Bafilomycine A1; SA-β-Gal = bêta-galactosidase associée à la sénescence; PBS = solution saline tamponnée au phosphate; FACS = tri cellulaire activé par fluorescence. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Fluorophore | Détecte | Ex/Em (nm) | Cytomètre laser (nm) | Détecteur cytomètre / filtre passe-bande (nm) |
| Le | SA-β-Gal | 645/6601 | 640 | 670 / 30 |
| AF | Lipofuscine | < 600 | 488 | 525 / 50 |
| CV450 | Viabilité | 408/450 | 405 | 450 / 50 |
| PE | Anticorps/marqueur de surface | 565/578 | 561 | 582 / 15 |
Tableau 1 : Fluorophores et spécifications optiques du cytomètre. Les spécifications du cytomètre utilisées dans ce protocole sont répertoriées pour un instrument avec un total de 4 lasers et 15 détecteurs d’émission. DDAOG détecté à 645/660 nm est la forme de la sonde clivée par SA-β-Gal1. Le DDAOG onculé peut présenter une fluorescence de faible niveau à 460/610 nm, mais il est éliminé par les étapes de lavage prévues dans le protocole. Abréviations : DDAO = 9H-(1,3-dichloro-9,9-diméthylacridin-2-one); DDAOG = DDAO-Galactoside; AF = autofluorescence; PE = phycoérythrine; SA-β-Gal = bêta-galactosidase associée à la sénescence.
Protocole
Tous les travaux sur les animaux décrits ont été approuvés par le comité institutionnel de soin et d’utilisation des animaux de l’Université de Chicago.
1. Préparation et stockage des solutions mères
REMARQUE: Si les cellules doivent être triées en flux, toutes les solutions doivent être préparées à l’aide de techniques stériles et filtrées à travers un dispositif filtrant de 0,22 μm.
- Préparer une solution mère de DDAO-Galactoside à 5 mg/mL dans du DMSO. Aliquote à 50 μL par tube (ou volume souhaité). Conserver à −20 °C dans l’obscurité jusqu’à 1 an.
- Préparer une solution mère de Bafilomycine A1 à 1 mM dans du DMSO. Aliquote à 50 μL par tube (ou volume souhaité). Conserver à −20 °C jusqu’à 6 mois.
- Préparer une solution mère de Calcein Violet 450 AM à 1 mM dans du DMSO. Aliquote à 50 μL par tube (ou volume souhaité). Conserver à −20 °C dans l’obscurité jusqu’à 1 an.
- Pour le traitement in vitro de cellules cultivées, préparer des solutions mères concentrées de 10 mM d’agent(s) induisant la sénescence dans le solvant approprié et stériliser à l’aide de filtres à seringue de 0,2 μm. Conserver à −20 °C ou selon les directives du fabricant.
REMARQUE: Pour l’administration d’agents de chimiothérapie induisant la sénescence in vivo (à des souris présentant des tumeurs établies), les agents doivent être de qualité USP et dilués dans une solution saline à partir d’un stock concentré juste avant l’injection. - Préparer un milieu de culture complet pour la ou les lignées cellulaires utilisées.
REMARQUE: Par exemple, préparez un milieu pour les cellules B16-F10 ou A549 avec DMEM 1x + 10% FBS + 1x substitut de glutamine + 1x pénicilline / streptomycine. Les milieux doivent être maintenus stériles. D’autres formulations de milieux spécifiques à la lignée cellulaire peuvent être utilisées. Certains composants tels que le glutathion peuvent, dans certains cas, interférer avec l’apparition de la sénescence. Des essais empiriques de diverses formulations de milieux doivent être effectués si le début de la sénescence est plus faible que prévu ou n’a pas été observé avec les agents de chimiothérapie témoins. - Préparez les tampons de coloration et de lavage.
- Préparer 1% d’albumine sérique bovine (BSA) dans 1x PBS pour une utilisation dans les procédures de coloration. Dissoudre 2 g de BSA dans 200 mL de PBS et agiter pendant 10 minutes, ou jusqu’à dissolution complète, à température ambiante.
- Préparez 0,5% de BSA dans 1x PBS comme tampon de lavage. Diluer 100 mL du BSA à 1 % préparé à l’étape 1.6.1 dans 100 mL de 1x PBS pour 0,5 % de BSA.
- Conserver les tampons à 4 °C jusqu’à 1 mois.
- Préparer 4% de paraformaldéhyde dans 1x PBS. Utiliser des ampoules de paraformaldéhyde scellées disponibles dans le commerce (p. ex. 16 % v/v) pour plus de commodité et de stabilité : 2,5 mL de PFA à 16 % + 7,5 mL de 1x PBS (= 10 mL de PFA à 4 %). Ajustez le volume préparé en fonction du volume total nécessaire par expérience.
REMARQUE: Préparez-vous au besoin pour la fixation de la cellule seulement; Préparez frais à chaque fois. - Préparer le tampon de tri FACS: 1x PBS, 1 mM EDTA, 25 mM HEPES, 1% BSA (pH 7,2). Filtrer stérile à travers un dispositif de filtration de 0,22 μm et conserver à 4 °C jusqu’à 1 mois.
REMARQUE: Préparez-vous au besoin pour le tri FACS uniquement. Les formulations tampons de tri en flux peuvent varier d’un instrument FACS à l’autre. La formulation ci-dessus est compatible avec l’instrument utilisé dans cette étude (voir le tableau des matériaux). Consultez les directives du fabricant. - Préparer la solution de dissociation tumorale : 20 μg/mL de Liberase TL + 100 μg/mL de DNAse I dans un milieu RPMI-1640 (sans FBS ni autres suppléments). Il est utile de conserver sous la main des solutions mères de Liberase TL (préparée et conservée selon les directives du fabricant) et de DNAse I (100 mg/mL dans de l’eau bidistillée [ddH2O], stockée à −20°C).
REMARQUE: Préparez-vous au besoin si vous utilisez uniquement des tumeurs; Préparez frais à chaque fois.
2. Induction de la sénescence par des agents chimiothérapeutiques dans des cellules cancéreuses en culture
REMARQUE : Toutes les étapes de manipulation cellulaire décrites dans cette section doivent être effectuées dans une enceinte de biosécurité en utilisant des pratiques stériles. Cette section est écrite pour les types de cellules adhérentes. Les cellules de suspension peuvent être utilisées avec les modifications appropriées, comme indiqué.
- Cultiver des lignées cellulaires cancéreuses selon le protocole standard du fournisseur ou du laboratoire qui a fourni la ou les lignées cellulaires spécifiques utilisées.
NOTE: Les cellules à passage bas (p < 10) sont généralement préférées car il y aura des niveaux plus faibles de sénescence réplicative, c’est-à-dire un fond inférieur, dans les échantillons de cellules non traitées. - Un jour avant l’induction de la sénescence par des médicaments, récolter des cellules avec de la trypsine-EDTA 0,25% (ou comme recommandé). Neutraliser la trypsine en ajoutant un volume égal de milieu de culture complet et transférer la suspension cellulaire dans un tube conique stérile.
Remarque : Cette étape n’est pas nécessaire pour les cellules de suspension. - Compter les cellules à l’aide de la méthode standard de l’hémocytomètre et enregistrer les cellules/mL. Cellules à plaques à 1 × 10 3-10× 103 cellules/cm2 dans des boîtes de culture en plastique standard à 6 puits.
REMARQUE: La densité optimale de placage dépend du taux de prolifération des cellules et doit être déterminée par l’utilisateur. Les cellules devraient être en phase logarithmique de croissance à une confluence d’environ 10 % à 20 % au moment du traitement (c.-à-d. après une incubation de 18 à 24 heures après le placage). La densité de départ (cellules/mL) des cellules en suspension doit être déterminée par l’utilisateur. Les plaques à six puits produisent généralement suffisamment de cellules sénescentes par puits pour un échantillon standard d’analyse de cytométrie en flux. En cas de tri en flux, une surface beaucoup plus grande (p. ex. plusieurs plaques P150) devrait être utilisée pour permettre la récupération d’un nombre suffisant de cellules sénescentes pour les essais en aval (≥1 × 106). - Incuber les cellules plaquées pendant la nuit (18-24 h) dans un incubateur à 37 °C avec 5% de CO2 et un bac d’humidité.
- Traiter les cellules plaquées avec un ou plusieurs agents chimiothérapeutiques induisant la sénescence. Inclure au moins un témoin positif, p. ex. l’étoposide (ETO) ou la bléomycine (BLM). Préparez des puits en double par médicament. Inclure un ensemble traité avec véhicule uniquement comme contrôle.
REMARQUE : Une courbe de dose pour chaque agent expérimental doit être testée par l’utilisateur afin de déterminer la concentration optimale pour l’induction de la sénescence dans la ou les lignées cellulaires utilisées. - Incuber les cellules pendant 4 jours dans un incubateur à 37 °C avec 5% de CO2 et un bac d’humidité pour permettre l’apparition de la sénescence. Examiner quotidiennement les changements morphologiques attendus à l’aide d’un microscope optique.
NOTE: Des temps d’incubation de 3 à 5 jours peuvent être acceptables en fonction du taux d’apparition de la sénescence. Les milieux peuvent être changés et l’agent réappliqué (ou non), comme souhaité, pour favoriser des conditions de croissance saines tout en atteignant un pourcentage acceptable de cellules sénescentes. - Après le début de la sénescence, récolter les cellules en ajoutant 0,25% de trypsine-EDTA pendant 5 min à 37 °C. Lorsque les cellules sont dissociées en suspension, neutraliser la trypsine avec un volume égal de milieu complet.
REMARQUE: Cette étape n’est pas nécessaire pour les cellules qui se développent en suspension. Si la coloration des marqueurs de surface doit être effectuée, éviter l’utilisation de trypsine-EDTA car elle peut détruire temporairement les antigènes de surface sur les cellules. Au lieu de cela, dissociez délicatement la monocouche à l’aide d’un grattoir cellulaire en plastique stérile (ou d’un réactif de dissociation alternatif conçu pour préserver les antigènes de surface). - Compter les cellules de chaque échantillon à l’aide d’un hémocytomètre. Calculer les cellules/mL pour chaque échantillon.
REMARQUE: Le bleu de trypan peut être ajouté pour évaluer le pourcentage de cellules mortes à ce stade (c’est-à-dire en raison d’un traitement médicamenteux), mais la mort cellulaire sera également déterminée avec un colorant de viabilité fluorescent pendant le flux de travail de coloration DDAOG. - Aliquote ≥0,5 × 106 cellules par échantillon dans des tubes microcentrifugés de 1,7 mL.
REMARQUE : Le nombre de cellules par échantillon doit être normalisé pour tous les échantillons. - Centrifuger les tubes pendant 5 min à 1 000 × g dans une microcentrifugeuse à 4 °C. Retirez le surnageant.
REMARQUE : Si une microcentrifugeuse réfrigérée n’est pas disponible, il peut être acceptable d’effectuer des centrifugations à température ambiante pour certains types de cellules résilientes. - Passez à la coloration DDAOG à la section 4.
3. Induction de la sénescence par des médicaments de chimiothérapie dans les tumeurs établies chez la souris
REMARQUE: Si les cellules tumorales doivent être triées par FACS, assurez-vous de la stérilité à chaque étape en travaillant dans une armoire de biosécurité et en travaillant avec des instruments, des procédures et des réactifs stériles.
- Créer des modèles tumoraux de souris en injectant des cellules cancéreuses par voie sous-cutanée, selon des méthodes standard (par exemple, Appelbe et al.19).
REMARQUE : Le nombre de cellules cancéreuses à injecter, le site d’injection et la souche de souris appropriée doivent être optimisés pour chaque protocole. Ici, les cellules B16-F10 ont été injectées par voie sous-cutanée à 1 × 106 cellules dans 0,1 mL de solution saline (1 × 107 cellules/mL).- Vérifiez que la viabilité des cellules utilisant le bleu de trypan est de >90% avant d’effectuer des injections. Anesthésiez les souris avec de l’isoflurane.
- Anesthésier les souris femelles C57/BL6 âgées de 6 à 7 semaines avec un mélange d’isoflurane à 3 % et d’air et les maintenir dans ces conditions dans une chambre d’induction placée dans une enceinte de biosécurité stérile. Confirmez l’anesthésie en pinçant doucement le pied de la souris. Appliquez une pommade vétérinaire stérile sur les deux yeux pour empêcher le séchage de la cornée pendant la procédure. Pendant la procédure, maintenez la température corporelle de la souris à l’aide d’une lampe chauffante.
- Travaillant à l’intérieur d’une enceinte de biosécurité stérile, retirez la souris de la chambre d’induction et placez-la en contact avec un cône nasal fournissant 3% d’isoflurane. Rasez la zone de flanc au site d’injection à l’aide d’un rasoir électrique propre. Mélanger brièvement la suspension cellulaire préparée en retournant manuellement le tube juste avant l’injection et injecter la suspension cellulaire par voie sous-cutanée dans le(s) flanc(s) rasé(s) à l’aide d’une seringue stérile de 0,5 mL munie d’une aiguille stérile de 27 G. Retirez la souris du capot et transférez-la dans la cage de récupération.
- Dans la cage de récupération, surveiller les signes vitaux des souris en continu jusqu’à ce qu’elles aient repris suffisamment conscience pour maintenir la position couchée sternale, démontrer le réflexe de redressement et être capables de se déplacer en toute sécurité dans la cage. Ne laissez pas les souris sans surveillance et ne retournez pas les animaux qui ont subi une inoculation de cellules tumorales en compagnie d’autres animaux jusqu’à ce qu’ils soient complètement rétablis. Surveiller quotidiennement toutes les souris inoculées pour détecter la perte de poids, la réduction de l’activité / mobilité et les symptômes neurologiques; Euthanasier les souris présentant des symptômes graves dans n’importe quelle catégorie. Pour les souris présentant des symptômes de douleur après l’inoculation, administrer la buprénorphine (0,1-0,2 mg / kg) une fois par voie sous-cutanée.
REMARQUE : Les souris présentant une douleur persistante après la buprénorphine doivent être euthanasiées.
- En commençant 5 à 7 jours après l’inoculation des cellules cancéreuses, mesurez la croissance tumorale avec des étriers tous les 2-3 jours. Initier un traitement prosénescent lorsque la tumeur a atteint 50 mm 3 ± 10 mm3 en volume.
NOTE: Dans ce travail, des doses de chlorhydrate de doxorubicine de qualité USP (DOX) ou de doxorubicine liposomale pégylée (PLD) à 10 mg / kg ont été administrées en injection de chlorure de sodium à 0,9% (USP). Les médicaments ont été injectés par voie intrapéritonéale 3x, une fois tous les 5 jours, à partir du moment où les tumeurs ont atteint 50 mm 3 ± 10 mm3. Les souris se sont rétablies pendant 7 jours après le traitement final pour permettre l’apparition du TIS dans les tumeurs. Les dosages d’induction de la sénescence et les conditions pour d’autres traitements et / ou modèles tumoraux doivent être optimisés. - 7 jours après le traitement médicamenteux final, sacrifier les souris par surdosagede CO 2 et luxation cervicale ou d’autres méthodes approuvées conformément aux directives de travail des animaux de laboratoire. Extrayez les tumeurs et recueillez-les dans des tubes stériles ou des plaques à 6 puits remplies d’un milieu de croissance RPMI stérile (pour préserver la viabilité pendant le traitement).
REMARQUE: Si vous effectuez un examen histologique (par exemple, X-Gal ou immunohistochimie), les tumeurs peuvent être divisées ici, avec une moitié congelée dans un milieu d’incorporation O.C.T. et cryosectionnée en utilisant des procédures standard pour l’histologie des tissus congelés. La moitié tumorale restante devrait fournir un matériau abondant pour la dissociation et la coloration DDAOG. - Transférer une tumeur dans une boîte en plastique P100 contenant 5 ml de milieu RPMI. Hacher la tumeur en morceaux à l’aide d’un scalpel.
- Transférer 5 mL de la suspension de morceaux tumoraux contenant des cellules en suspension et des débris dans un tube conique de 15 mL. Utilisez l’embout le plus large d’une pipette sérologique de 25 ml pour transférer cette suspension en présence de gros débris. Rincer le plat avec un volume supplémentaire de RPMI stérile pour recueillir les matériaux. Boucher et placer le tube conique sur de la glace.
- Répétez les étapes 3.45-3.5 pour les tumeurs restantes. Utilisez une plaque et un scalpel séparés pour chaque tumeur pour éviter la contamination croisée, ou rincez bien avec du PBS entre les tumeurs. Utilisez 5 mL de milieu frais pour hacher chaque tumeur.
- Préparer la solution de dissociation tumorale : 20 μg/mL de Liberase TL + 100 μg/mL de DNAse I dans un milieu RPMI-1640 (sans FBS).
REMARQUE: De nombreuses formulations efficaces pour les solutions de dissociation tumorale existent et peuvent inclure une variété d’enzymes et d’autres composants de différents fabricants. Les concentrations optimales de composants peuvent varier considérablement selon les types de tumeurs. Si les globules rouges sont fortement présents dans la tumeur, la lyse des globules rouges peut également être effectuée; Si les cellules mortes sont un problème, une trousse d’élimination des cellules mortes peut être utilisée. Il est fortement recommandé à l’utilisateur de déterminer indépendamment les conditions optimales de dissociation tumorale qui assurent une viabilité élevée et une faible présence de cellules contaminantes, de matériaux conjonctifs et de débris. - Centrifuger tous les échantillons tumoraux dans des tubes coniques pendant 5 min à 1 000 × g (4 °C). Retirez le surnageant.
- Ajouter 1-5 mL de solution de dissociation tumorale à chaque échantillon tumoral, en fonction du volume de matériel tumoral. Assurez-vous qu’il y a 1-2 mL en excès de la pastille de matériel tumoral dans le tube. Vortex à une vitesse modérée pour mélanger.
- Placer les échantillons dans un incubateur à 37 °C avec rotation rapide pendant 45 min. Vortex brièvement toutes les 15 minutes.
- Filtrer chaque échantillon à travers une crépine cellulaire de 100 μm dans un tube conique de 50 mL. Si les échantillons sont trop visqueux pour passer à travers le filtre, ajouter 10 mL de milieu RPMI-1640 à diluer. Rincer les filtres avec un milieu RPMI pour recueillir les cellules résiduelles.
- Utilisez un hémocytomètre pour compter les cellules/ml pour chaque échantillon.
- Aliquote deux réplicas ou plus de 5 × 106 cellules par échantillon tumoral.
- Centrifuger pendant 5 min à 1 000 × g (4 °C). Retirez le surnageant.
- (Facultatif) Cryoconserver les échantillons tumoraux pour une coloration DDAOG ultérieure si vous le souhaitez.
- Resuspendre la pastille de cellules tumorales dissociées dans un milieu de cryoconservation : 50% FBS, 40% RPMI-1640, 10% DMSO, préparé dans des conditions stériles à 5 × 106 cellules/mL.
- Aliquote 1 mL de suspension cellulaire dans chaque cryovial.
- Congeler les cryovials pendant 24 h dans un récipient de congélation de cellules isopropanol à −80 °C; Ensuite, transférer dans un cryostockage d’azote liquide pour un stockage à plus long terme (>1 semaine).
- Lorsque la coloration est souhaitée, décongeler les cryovials sur la glace et procéder à la coloration DDAOG dans la section 4.
REMARQUE: Certaines tumeurs peuvent ne pas rester viables par cryoconservation, et la résilience à ce processus doit être évaluée par l’utilisateur pour le modèle tumoral d’intérêt.
- Passez à la section 4 pour la coloration DDAOG.
4. Coloration DDAOG de SA-β-Gal dans des échantillons cellulaires ou tumoraux
- Diluer 1 mM de Bafilomycine A1 à 1:1 000x dans un milieu DMEM (sans FBS) pour une concentration finale de 1 μM.
- Ajouter la solution préparée de Baf-DMEM aux échantillons de granulés cellulaires (à partir de l’étape 2.11 ou 3.16) pour une concentration de 1 × 106 cellules/mL.
REMARQUE : Par exemple, si vous utilisez 0,5 × 106 cellules par échantillon, ajoutez 0,5 mL de Baf-DMEM. Pour les tumeurs, 5 × 106 cellules peuvent être colorées dans 5 mL de Baf-DMEM. - Incuber pendant 30 min à 37 °C (sans CO2) sur un rotateur/agitateur réglé à vitesse lente.
REMARQUE: Évitez les incubateurs de CO2 pour le processus de coloration, qui peuvent acidifier les solutions et ainsi interférer avec la coloration Baf et DDAOG. - Sans laver, ajouter la solution mère DDAOG (5 mg/mL) à 1:500x (10 μg/mL final) à chaque échantillon. Pipette à mélanger. Remplacer sur un rotateur/agitateur à 37 °C (sans CO2) pendant 60 min. Protéger de la lumière directe.
- Centrifuger les tubes pendant 5 min à 1 000 x g à 4 °C. Retirez le surnageant.
- Laver avec 1 ml de BSA glacé à 0,5 % par tube et à la pipette pour mélanger. Centrifuger les tubes pendant 5 min à 1 000 x g à 4 °C et retirer le surnageant. Répétez cette étape 2x pour bien laver les cellules. Retirez le surnageant et continuez.
REMARQUE: Il est important d’effectuer les étapes de lavage de l’étape 4.6 pour éliminer le DDAOG onculé, qui peut présenter une émission de fluorescence indésirable (460/610 nm).
REMARQUE : En cas d’immunomarquage des marqueurs de surface cellulaire, passez à la section 5 ci-dessous. - (Facultatif) Fixation et stockage de cellules colorées DDAOG pour analyse ultérieure
- Ajouter goutte à goutte 0,5 mL de paraformaldéhyde glacé à 4 % à chaque échantillon lavé. Pipette à mélanger.
- Incuber pendant 10 min à température ambiante.
- Lavez les cellules 2x avec 1 mL de PBS.
- Conservez les échantillons jusqu’à 1 semaine à 4 °C avant l’analyse par cytométrie en flux.
Remarque : Pour les échantillons fixes, ignorez l’étape 4.8.
- Diluer le stock Calcein Violet 450 AM (1 mM) à 1:1 000x dans 1% BSA-PBS (1 μM final). Ajouter 300 μL (pour les échantillons de cellules en culture) ou 1 000 μL (pour les échantillons tumoraux) aux pastilles de cellules lavées à partir de l’étape 4.6. Incuber pendant 15 min sur de la glace dans l’obscurité.
- Passez à la configuration de la cytométrie en flux (section 6).
5. (Facultatif) Immunomarquage pour les marqueurs de surface cellulaire en combinaison avec DDAOG
REMARQUE : Comme pour toute expérience de cytométrie en flux, des échantillons témoins à coloration unique contenant uniquement du DDAOG et des anticorps fluorescents seulement doivent être préparés pour déterminer la diaphonie (le cas échéant) entre les canaux de fluorescence. Si la diaphonie est observée, une compensation de cytométrie de flux standard doit être effectuée20.
- Resuspendre les pastilles de cellule obtenues à l’étape 4.6 dans 100 μL de tampon de coloration (1% BSA dans 1x PBS).
- Ajouter le réactif bloquant le récepteur Fc approprié pour l’espèce cellulaire (souris ou humain) au titrage recommandé par le fabricant. Incuber pendant 10 min à 24 °C.
- Ajouter des anticorps conjugués au fluorophore au titrage recommandé par le fabricant (ou déterminé par l’utilisateur). Incuber pendant 20 min sur glace, à l’abri de la lumière.
- Centrifuger les tubes pendant 5 min à 1 000 × g à 4 °C. Retirez le surnageant.
- Laver avec 1 ml de tampon de lavage glacé (0,5 % BSA-PBS) par tube et pipette pour mélanger. Centrifuger les tubes pendant 5 min à 1 000 × g à 4 °C et retirer le surnageant. Répétez cette étape 2x pour bien laver les cellules.
- Diluer 1 mM Calcein Violet 450 AM à 1:1 000x dans 1% BSA-PBS. Ajouter 300 μL aux pastilles de cellules lavées à partir de l’étape 5.5. Incuber pendant 15 min sur de la glace dans l’obscurité.
- Procéder à l’analyse par cytométrie en flux (sections 6 et 7).
6. Configuration du cytomètre en flux et acquisition de données
- Transférer les échantillons cellulaires dans des tubes compatibles avec l’instrument de cytométrie en flux. Placez les tubes sur de la glace et gardez-les à l’abri de la lumière.
NOTE: Si des agrégats sont observés dans les suspensions cellulaires, faire passer la suspension dans des crépines cellulaires de 70 à 100 μm avant l’analyse. N’utilisez pas de crépines de 40 μm, car elles peuvent exclure certaines des plus grandes cellules sénescentes. - Dans le logiciel référencé (voir le tableau des matériaux), ouvrez les graphiques suivants : 1) diagramme de points FSC-A vs SSC-A, 2) histogramme de canal violet, 3) diagramme de points de canal rouge lointain (par exemple, APC-A) ou de canal vert (par exemple, FITC-A).
REMARQUE : Des diagrammes d’exclusion de doublets et des histogrammes monocanaux peuvent également être utilisés, mais ne sont pas strictement obligatoires. - Initier l’acquisition des données du cytomètre.
- Placez l’échantillon témoin du véhicule uniquement taché avec DDAOG sur l’orifice d’admission. À une faible vitesse d’admission, commencez à acquérir des données d’échantillonnage.
- Ajustez les tensions FSC et SSC de sorte que > 90 % des événements soient contenus dans le graphique. Si les cellules ne s’adaptent pas bien au tracé, abaissez le paramètre de mise à l’échelle de la zone à 0,33-0,5 unité.
- Retirez l’échantillon du véhicule uniquement sans enregistrer de données.
- (Facultatif) Ajouter une gouttelette de microsphères d’étalonnage arc-en-ciel dans un tube cytométrique avec 1 mL de PBS. Placez le tube sur l’orifice d’admission du cytomètre. Commencez à acquérir des exemples de données.
- Ajustez les tensions des canaux violet, vert et rouge lointain de sorte que le pic supérieur de la microsphère arc-en-ciel soit compris entre 10 4 et 105 unités de fluorescence relative dans chaque canalet que tous les pics soient bien séparés dans chaque canal. Record de 10 000 événements. Retirez le tube.
- Placer l’échantillon témoin positif (p. ex. BLM, ETO) coloré au DDAOG sur l’orifice d’admission. À faible vitesse, acquérir des échantillons de données. Observez les événements dans les canaux FSC, SSC, violet, vert et rouge lointain pour vous assurer que plus de 90% des événements sont contenus dans toutes les placettes. Recherchez une augmentation du signal AF et DDAOG par rapport au contrôle du véhicule uniquement.
- Si vous utilisez un cytomètre de tri, lancez le tri à cette étape.
- Aux fins de la tenue des dossiers, consigner 10 000 cellules pour l’échantillon témoin et chaque échantillon trié.
- Trier la quantité souhaitée de cellules (≥1 × 106 convient généralement) dans un tube de collecte approprié à l’instrument avec 3 à 5 ml de milieu de culture.
- Après le tri, passez à la culture ou à l’analyse en aval.
- Passez à la section 7 pour l’analyse de routine des échantillons triés.
- Si vous utilisez des anticorps fluorescents, optimisez les tensions des canaux ici en utilisant les échantillons non colorés, monocolorés et à double coloration préparés dans la section 5.
REMARQUE: Pour le cytomètre en flux étalonné utilisé ici, les tensions optimales des canaux se situent généralement entre 250 et 600 (milieu de gamme), mais les tensions optimales et les plages de tension de canal varient d’un instrument à l’autre. Évitez d’utiliser des tensions à des plages très basses ou élevées, qui peuvent supprimer le signal ou amplifier le bruit. - Après avoir terminé les étapes 6.1 à 6.5 et apporté les ajustements nécessaires aux réglages du cytomètre, consignez les données de tous les échantillons. Assurez-vous que les paramètres restent uniformes pour tous les enregistrements d’échantillons. Enregistrez ≥10 000 événements par échantillon de cellules cultivées ou 100 000 ≥ événements par échantillon de cellules tumorales.
REMARQUE : Bien que l’établissement de points de contrôle et l’analyse puissent être effectués à l’aide d’un logiciel d’acquisition de données (p. ex., FACSDiva), un flux de travail complet d’établissement de points de contrôle et d’analyse à effectuer après l’acquisition à l’aide d’un logiciel d’analyse distinct (FlowJo) est décrit à la section 7 ci-dessous. L’analyse post-acquisition est privilégiée pour réduire le temps passé au poste de travail du cytomètre et profiter des outils supplémentaires inclus dans le logiciel d’analyse dédié. - Enregistrez des exemples de données au format de fichier .fcs. Exportez les fichiers vers un poste de travail équipé d’un logiciel d’analyse de cytométrie en flux (par exemple, FlowJo). Passez à la section 7.
7. Analyse des données de cytométrie en flux
REMARQUE: Le flux de travail présenté utilise le logiciel FlowJo. Un logiciel alternatif d’analyse des données de cytométrie en flux peut être utilisé si les étapes clés décrites dans cette section sont suivies de la même manière.
- À l’aide du logiciel FlowJo, ouvrez les fichiers de données .fcs de l’étape 6.7.
- Ouvrez la fenêtre de mise en page.
- Faites glisser et déposez tous les échantillons dans la fenêtre de mise en page.
- Portes des cellules viables.
- Double-cliquez d’abord sur les exemples de données pour la commande du véhicule uniquement pour ouvrir sa fenêtre de données.
- Visualisez les données sous forme d’histogramme à canal violet. Identifier les cellules viables colorées par CV450 en fonction de leur fluorescence plus brillante que les cellules mortes.
- Dessinez une porte à l’aide de l’outil d’histogramme à porte unique pour inclure uniquement les cellules viables. Nommez la porte viable.
- Ensuite, à partir de la fenêtre de disposition d’exemple, faites glisser la porte viable sur les autres échantillons de cellules pour appliquer la porte uniformément.
- Dans la fenêtre de mise en page, visualisez tous les échantillons sous forme d’histogrammes de canal violet (viabilité). Vérifier que le contrôle cellulaire viable est approprié pour tous les échantillons avant de procéder; Si ce n’est pas le cas, apportez les ajustements nécessaires.
REMARQUE: La coloration de viabilité peut présenter des variations selon les traitements ou les tumeurs.
- Portes des cellules sénescentes.
- Double-cliquez sur les données de cellule viable fermées pour le contrôle du véhicule uniquement pour ouvrir sa fenêtre de données.
- Visualisez les données sous forme de diagramme à points pour le canal rouge lointain (DDAOG) par rapport au canal vert (AF).
- Dessinez une porte à l’aide de l’outil de contrôle rectangulaire pour inclure <5 % des cellules DDAOG+ et AF+ (quadrant supérieur droit). Nommez la porte sénescente.
- Ensuite, à partir de la fenêtre de disposition des exemples, faites glisser la porte sénescente sur les sous-ensembles viables des autres échantillons de cellules pour appliquer la porte uniformément.
- Dans la fenêtre de mise en page, faites glisser et déposez tous les sous-ensembles de cellules viables répertoriés dans la section 7.4. Visualisez tous les échantillons viables sous forme de diagrammes à points rouge lointain (p. ex., APC-A) ou en chenaux verts (p. ex., FITC-A).
- S’assurer que la barrière sénescente dessinée à l’étape 7.5.3 est visible sur toutes les placettes et que la barrière de contrôle du véhicule seulement présente ≤5 % à 10 % de cellules sénescentes.
- Une fois que le pourcentage de cellules sénescentes a été déterminé à l’aide des étapes ci-dessus, présenter les données résultantes à l’aide des diagrammes FlowJo, résumés dans un tableau de données et/ou analysés statistiquement à l’aide d’un logiciel standard.
Résultats
Plusieurs expériences ont été réalisées pour démontrer la comparabilité du DDAOG au X-Gal et au C12-FDG pour la détection de la sénescence par SA-β-Gal. Tout d’abord, X-Gal a été utilisé pour colorer les cellules sénescentes du mélanome B16-F10 induites par ETO (Figure 2A). Une couleur bleue intense s’est développée dans un sous-ensemble de cellules traitées par ETO, tandis que d’autres cellules présentaient une coloration bleue moins intense. La morpholog...
Discussion
Au cours de la dernière décennie, la cytométrie en flux est devenue une plate-forme de dosage plus courante dans la recherche sur le cancer en raison de la popularité émergente de l’immunologie tumorale, du développement de cytomètres en flux à moindre coût et de l’amélioration des installations d’instrumentation partagées dans les établissements universitaires. Les tests multicolores sont maintenant standard, car la plupart des nouveaux instruments sont équipés de réseaux optiques violet, bleu-vert ...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer pour cette étude.
Remerciements
Nous remercions le Cytometry and Antibody Core Facility de l’Université de Chicago pour son soutien sur l’instrumentation de cytométrie en flux. Le Centre de recherche animale de l’Université de Chicago a fourni un logement pour animaux.
matériels
| Name | Company | Catalog Number | Comments |
| Bafilomycin A1 | Research Products International | B40500 | |
| Bleomycin sulfate | Cayman | 13877 | |
| Bovine serum albumin (BSA) | US Biological | A1380 | |
| Calcein Violet 450 AM viability dye | ThermoFisher Scientific | 65-0854-39 | eBioscience |
| DPP4 antibody, PE conjugate | Biolegend | 137803 | Clone H194-112 |
| Cell line: A549 human lung adenocarcinoma | American Type Culture Collection | CCL-185 | |
| Cell line: B16-F10 mouse melanoma | American Type Culture Collection | CRL-6475 | |
| Cell scraper | Corning | 3008 | |
| Cell strainers, 100 µm | Falcon | 352360 | |
| DDAO-Galactoside | Life Technologies | D6488 | |
| DMEM medium 1x | Life Technologies | 11960-069 | |
| DMSO | Sigma | D2438 | |
| DNAse I | Sigma | DN25 | |
| Doxorubicin, hydrochloride injection (USP) | Pfizer | NDC 0069-3032-20 | |
| Doxorubicin, PEGylated liposomal (USP) | Sun Pharmaceutical | NDC 47335-049-40 | |
| EDTA 0.5 M | Life Technologies | 15575-038 | |
| Etoposide | Cayman | 12092 | |
| FBS | Omega | FB-11 | |
| Fc receptor blocking reagent | Biolegend | 101320 | Anti-mouse CD16/32 |
| Flow cytometer (cell analyzer) | Becton Dickinson (BD) | Various | LSRFortessa |
| Flow cytometer (cell sorter) | Becton Dickinson (BD) | Various | FACSAria |
| GlutaMax 100x | Life Technologies | 35050061 | |
| HEPES 1 M | Lonza | BW17737 | |
| Liberase TL | Sigma | 5401020001 | Roche |
| Paraformaldehyde 16% | Electron Microscopy Sciences | 15710 | |
| Penicillin/Streptomycin 100x | Life Technologies | 15140122 | |
| Phosphate buffered saline (PBS) 1x | Corning | MT21031CV | Dulbecco's PBS (without calcium and magnesium) |
| Rainbow calibration particles, ultra kit | SpheroTech | UCRP-38-2K | 3.5-3.9 µm, 2E6/mL |
| RPMI-1640 medium 1x | Life Technologies | 11875-119 | |
| Sodium chloride 0.9% (USP) | Baxter Healthcare Corporation | 2B1324 | |
| Software for cytometer data acquisition, "FACSDiva" | Becton Dickinson (BD) | n/a | Contact BD for license |
| Software for cytometer data analysis, "FlowJo" | TreeStar | n/a | Contact TreeStar for license |
| Trypsin-EDTA 0.25% | Life Technologies | 25200-114 |
Références
- Saleh, T., Tyutyunyk-Massey, L., Gewirtz, D. A. Tumor cell escape from therapy-induced senescence as a model of disease recurrence after dormancy. Cancer Research. 79 (6), 1044-1046 (2019).
- Wang, B., Kohli, J., Demaria, M. Senescent cells in cancer therapy: friends or foes. Trends in Cancer. 6 (10), 838-857 (2020).
- Prasanna, P. G., et al. Therapy-induced senescence: Opportunities to improve anticancer therapy. Journal of the National Cancer Institute. 113 (10), 1285-1298 (2021).
- Velarde, M. C., Demaria, M., Campisi, J. Senescent cells and their secretory phenotype as targets for cancer therapy. Interdisciplinary Topics in Gerontology and Geriatrics. 38, 17-27 (2013).
- Ou, H. L., et al. Cellular senescence in cancer: from mechanisms to detection. Molecular Oncology. 15 (10), 2634-2671 (2021).
- Hernandez-Segura, A., Nehme, J., Demaria, M. Hallmarks of cellular senescence. Trends in Cell Biology. 28 (6), 436-453 (2018).
- Bojko, A., Czarnecka-Herok, J., Charzynska, A., Dabrowski, M., Sikora, E. Diversity of the senescence phenotype of cancer cells treated with chemotherapeutic agents. Cells. 8 (12), 1501 (2019).
- Mikuła-Pietrasik, J., Niklas, A., Uruski, P., Tykarski, A., Książek, K. Mechanisms and significance of therapy-induced and spontaneous senescence of cancer cells. Cellular and Molecular Life Sciences. 77 (2), 213-229 (2020).
- Lee, B. Y., et al. Senescence-associated beta-galactosidase is lysosomal beta-galactosidase. Aging cell. 5 (2), 187-195 (2006).
- Itahana, K., Itahana, Y., Dimri, G. P. Colorimetric detection of senescence-associated β galactosidase. Methods in Molecular Biology. 965, 143-156 (2013).
- Dimri, G. P., et al. A biomarker that identifies senescent human cells in culture and in aging skin in vivo. Proceedings of the National Academy of Sciences of the United States of America. 92 (20), 9363-9367 (1995).
- Debacq-Chainiaux, F., Erusalimsky, J. D., Campisi, J., Toussaint, O. Protocols to detect senescence-associated beta-galactosidase (SA-betagal) activity, a biomarker of senescent cells in culture and in vivo. Nature Protocols. 4 (12), 1798-1806 (2009).
- Noppe, G., et al. Rapid flow cytometric method for measuring senescence associated beta-galactosidase activity in human fibroblasts. Cytometry A. 75 (11), 910-916 (2009).
- Tung, C. -. H., et al. In vivo imaging of β-galactosidase activity using far red fluorescent switch. Cancer Research. 64 (5), 1579-1583 (2004).
- Gong, H., et al. beta-Galactosidase activity assay using far-red-shifted fluorescent substrate DDAOG. Analytical Biochemistry. 386 (1), 59-64 (2009).
- Terman, A., Brunk, U. T. Lipofuscin: Mechanisms of formation and increase with age. APMIS. 106 (2), 265-276 (1998).
- Georgakopoulou, E. A., et al. Specific lipofuscin staining as a novel biomarker to detect replicative and stress-induced senescence. A method applicable in cryo-preserved and archival tissues. Aging. 5 (1), 37-50 (2013).
- Wang, B., Demaria, M. The quest to define and target cellular senescence in cancer. Cancer Research. 81 (24), 6087-6089 (2021).
- Appelbe, O. K., Zhang, Q., Pelizzari, C. A., Weichselbaum, R. R., Kron, S. J. Image-guided radiotherapy targets macromolecules through altering the tumor microenvironment. Molecular Pharmaceutics. 13 (10), 3457-3467 (2016).
- Maciorowski, Z., Chattopadhyay, P. K., Jain, P. Basic multicolor flow cytometry. Current Protocols in Immunology. 117, 1-38 (2017).
- Fan, Y., Cheng, J., Zeng, H., Shao, L. Senescent cell depletion through targeting BCL-family proteins and mitochondria. Frontiers in Physiology. 11, 593630 (2020).
- Kim, K. M., et al. Identification of senescent cell surface targetable protein DPP4. Genes and Development. 31 (15), 1529-1534 (2017).
- Flor, A. C., Kron, S. J. Lipid-derived reactive aldehydes link oxidative stress to cell senescence. Cell Death Discovery. 7 (9), 2366 (2016).
- Jochems, F., et al. The Cancer SENESCopedia: A delineation of cancer cell senescence. Cell reports. 36 (4), 109441 (2021).
- Fallah, M., et al. Doxorubicin and liposomal doxorubicin induce senescence by enhancing nuclear factor kappa B and mitochondrial membrane potential. Life Sciences. 232, 116677 (2019).
- Kasper, M., Barth, K. Bleomycin and its role in inducing apoptosis and senescence in lung cells - modulating effects of caveolin-1. Current Cancer Drug Targets. 9 (3), 341-353 (2009).
- Muthuramalingam, K., Cho, M., Kim, Y. Cellular senescence and EMT crosstalk in bleomycin-induced pathogenesis of pulmonary fibrosis-an in vitro analysis. Cell Biology International. 44 (2), 477-487 (2020).
- Flor, A. C., Wolfgeher, D., Wu, D., Kron, S. J. A signature of enhanced lipid metabolism, lipid peroxidation and aldehyde stress in therapy-induced senescence. Cell Death Discovery. 3, 17075 (2017).
- Burd, C. E., et al. Monitoring tumorigenesis and senescence in vivo with a p16(INK4a)-luciferase model. Cell. 152 (1-2), 340-351 (2013).
- Liu, J. Y., et al. Cells exhibiting strong p16 (INK4a) promoter activation in vivo display features of senescence. Proceedings of the National Academy of Sciences of the United States of America. 116 (7), 2603-2611 (2019).
- Wang, L., Lankhorst, L., Bernards, R. Exploiting senescence for the treatment of cancer. Nature Reviews Cancer. 22 (6), 340-355 (2022).
- Baek, K. -. H., Ryeom, S. Detection of oncogene-induced senescence in vivo. Methods in Molecular Biology. 1534, 185-198 (2017).
- González-Gualda, E., Baker, A. G., Fruk, L., Muñoz-Espín, D. A guide to assessing cellular senescence in vitro and in vivo. The FEBS Journal. 288 (1), 56-80 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon