Method Article
Un modèle murin pour la transition de Streptococcus pneumoniae de colonisateur à pathogène lors d’une co-infection virale récapitule la maladie exacerbée par l’âge
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Cet article décrit un nouveau modèle murin pour la transition du pneumocoque d’un colonisateur asymptomatique à un agent pathogène pathogène au cours d’une infection virale. Ce modèle peut être facilement adapté pour étudier les interactions polymicrobiennes et hôte-pathogène au cours des différentes phases de la progression de la maladie et entre divers hôtes.
Résumé
Streptococcus pneumoniae (pneumocoque ) est un colonisateur asymptomatique du nasopharynx chez la plupart des individus, mais peut évoluer vers un agent pathogène pulmonaire et systémique lors de l’infection par le virus de la grippe A (IAV). L’âge avancé augmente la susceptibilité de l’hôte à la pneumonie pneumococcique secondaire et est associé à une aggravation de l’issue de la maladie. Les facteurs de l’hôte à l’origine de ces processus ne sont pas bien définis, en partie à cause d’un manque de modèles animaux qui reproduisent la transition de la colonisation asymptomatique à la maladie clinique grave.
Cet article décrit un nouveau modèle murin qui recrée la transition des pneumocoques du portage asymptomatique à la maladie lors d’une infection virale. Dans ce modèle, les souris sont d’abord inoculées par voie intranasale avec des pneumocoques cultivés par biofilm pour établir un portage asymptomatique, suivi d’une infection par le VAI du nasopharynx et des poumons. Il en résulte une dissémination bactérienne dans les poumons, une inflammation pulmonaire et des signes évidents de maladie pouvant évoluer vers la létalité. Le degré de maladie dépend de la souche bactérienne et des facteurs de l’hôte.
Fait important, ce modèle reproduit la susceptibilité au vieillissement, car par rapport aux jeunes souris, les souris âgées présentent une maladie clinique plus grave et succombent à la maladie plus fréquemment. En séparant le portage et la maladie en étapes distinctes et en offrant la possibilité d’analyser les variantes génétiques de l’agent pathogène et de l’hôte, ce modèle de co-infection S. pneumoniae/IAV permet l’examen détaillé des interactions d’un pathobioonte important avec l’hôte à différentes phases de la progression de la maladie. Ce modèle peut également servir d’outil important pour identifier des cibles thérapeutiques potentielles contre la pneumonie pneumococcique secondaire chez les hôtes sensibles.
Introduction
Streptococcus pneumoniae (pneumocoque) sont des bactéries à Gram positif qui résident asymptomatiquement dans le nasopharynx de la plupart des individus en bonne santé 1,2. Favorisés par des facteurs qui ne sont pas complètement définis, les pneumocoques peuvent passer de colonisateurs bénins du nasopharynx à des agents pathogènes qui se propagent à d’autres organes entraînant des infections graves, notamment l’otite moyenne, la pneumonie et la bactériémie3. La présentation de la pneumococcie dépend, en partie, des différences spécifiques à la souche, y compris le sérotype, qui est basé sur la composition des polysaccharides capsulaires. Plus de 100 sérotypes ont été caractérisés jusqu’à présent, et certains sont associés à des infections plus invasives 4,5. Plusieurs autres facteurs augmentent le risque de pneumococcie. L’un de ces facteurs est l’infection virale, où le risque de pneumonie pneumococcique est multiplié par 100 par IAV 6,7. Historiquement, S. pneumoniae est l’une des causes les plus fréquentes de pneumonie bactérienne secondaire après la grippe et est associée à de pires résultats8. Un autre facteur de risque majeur est l’âge avancé. En fait, S. pneumoniae est la principale cause de pneumonie bactérienne d’origine communautaire chez les personnes âgées de plus de 65 ans 9,10. Les personnes âgées représentent la majorité (>75 %) des décès dus à la pneumonie et à la grippe, ce qui indique que les deux facteurs de risque - le vieillissement et l’infection par le VAI - aggravent de façon synergique la susceptibilité à la maladie11,12,13,14. Cependant, les mécanismes par lesquels l’infection virale provoque la transition des pneumocoques du colonisateur asymptomatique à l’agent pathogène invasif et la façon dont cela est façonné par les facteurs de l’hôte restent mal définis. Cela est dû en grande partie à l’absence d’un modèle de petit animal qui récapitule la transition de la colonisation pneumococcique asymptomatique à la maladie clinique critique.
Les études de co-infection ont classiquement été modélisées chez des souris inoculées avec des pneumocoques directement dans les poumons 7 jours après l’infection grippale15,16. Cela reproduit la susceptibilité à la pneumonie bactérienne secondaire et est idéal pour étudier comment les réponses immunitaires antivirales altèrent les défenses antibactériennes17. Cependant, des études longitudinales chez l’homme ont démontré que le portage pneumococcique dans le nasopharynx, où les bactéries peuvent former des biofilms asymptomatiques18, est uniformément associé à des maladies invasives 19,20. Les isolats bactériens provenant d’infections de l’oreille moyenne, des poumons et du sang sont génétiquement identiques à ceux trouvés dans le nasopharynx20. Ainsi, pour étudier la transition du portage asymptomatique à la maladie invasive à la suite d’une infection par le VAI, un modèle a été établi dans lequel des souris ont reçu par voie intranasale des pneumocoques cultivés par biofilm suivis d’une infection par le VAI du nasopharynx21,22. L’infection virale des voies respiratoires supérieures a entraîné des changements dans l’environnement de l’hôte qui ont entraîné la dispersion des pneumocoques à partir de biofilms et leur propagation aux voies respiratoires inférieures21. Ces bactéries dispersées avaient une expression régulée à la hausse de facteurs de virulence importants pour l’infection, les convertissant de colonisateurs en agents pathogènes21. Ces observations mettent en évidence l’interaction complexe entre le virus, l’hôte et la bactérie et démontrent que les changements de l’hôte déclenchés par l’infection virale ont un impact direct sur le comportement pneumococcique, qui, à son tour, modifie le cours de l’infection bactérienne. Cependant, ce modèle ne récapitule pas les signes graves de maladie observés chez les humains, probablement parce que le virus est limité aux fosses nasales et que les effets systémiques de l’infection virale sur l’immunité de l’hôte et les lésions pulmonaires ne sont pas récapitulés.
Nous avons récemment établi un modèle qui intègre l’interaction complexe entre l’hôte et les agents pathogènes, mais qui imite aussi plus étroitement la gravité de la maladie observée chez l’homme23. Dans ce modèle, les souris sont d’abord infectées par voie intranasale par des pneumocoques cultivés par biofilm pour établir un portage asymptomatique, suivi d’une infection par le VAI du nasopharynx et des poumons. Cela a entraîné une dissémination bactérienne dans les poumons, une inflammation pulmonaire et une maladie qui a évolué vers la létalité chez une fraction des jeunes souris23. Cette étude antérieure a démontré que l’infection virale et bactérienne modifiait la défense de l’hôte : l’infection virale favorisait la dissémination bactérienne et la colonisation bactérienne antérieure altérait la capacité de l’hôte à contrôler les niveaux d’IAV pulmonaires23. L’examen de la réponse immunitaire a révélé que l’infection par le VAI diminuait l’activité antibactérienne des neutrophiles, tandis que la colonisation bactérienne atténuait la réponse à l’interféron de type I essentielle à la défense antivirale23. Fait important, ce modèle reproduisait la susceptibilité au vieillissement. Par rapport aux jeunes souris, les souris âgées présentaient des signes de maladie plus tôt, présentaient une maladie clinique plus grave et succombaient à une infection plus fréquemment23. Les travaux présentés dans ce manuscrit montrent que le degré de maladie dépend également de la souche bactérienne, car les souches invasives de pneumocoque affichent une dissémination plus efficace lors de l’infection par le VAI, montrent des signes plus manifestes d’inflammation pulmonaire et entraînent des taux accélérés de maladie par rapport aux souches non invasives. Ainsi, ce modèle de co-infection S. pneumoniae/IAV permet l’examen détaillé des facteurs pathogènes et des facteurs de l’hôte et est bien adapté à l’étude des réponses immunitaires aux infections polymicrobiennes aux différentes phases de la progression de la maladie.
Protocole
Toutes les études sur les animaux ont été réalisées conformément aux recommandations du Guide pour le soin et l’utilisation des animaux de laboratoire. Toutes les procédures ont été approuvées par le comité de soin et d’utilisation des animaux de l’établissement de l’Université de Buffalo.
1. Préparation de milieux chimiquement définis (MDP)
- Préparez les stocks comme suit:
- Dissoudre les composés du mélange I énumérés au tableau 1 dans 100 mL d’eau ultrapure en agitant les mélanges. Conserver dans des aliquotes de 200 μL à −20 °C.
- Dissoudre les composés du mélange II énumérés au tableau 1 dans 20 mL de NaOH 0,1 M en agitant. Conserver dans des aliquotes de 100 μL à −20 °C.
- Dissoudre les composés du mélange III énumérés au tableau 1 dans 1 mL d’eau ultrapure pendant l’agitation. Conserver dans des aliquotes de 10 μL à 4 °C.
- Dissoudre le composé IV du mélange indiqué au tableau 1 dans 1 mL d’eau ultrapure en agitant le mélange. Conserver dans des aliquotes de 10 μL à −20 °C.
- Dissoudre d’abord les composés énumérés dans le tableau 2 dans 15 mL d’eau ultrapure en agitant. Ajuster le pH à 7,0 avec quelques gouttes de 0,1 M de NaOH et ajuster le volume final à 20 mL en utilisant de l’eau ultrapure. Conserver dans 1 mL d’aliquotes à −20 °C.
- Dissoudre les composés énumérés au tableau 3 dans 90 mL d’eau ultrapure sur une plaque chauffante à 50 °C en agitant. Ajustez le pH à 7,0 avec 0,1 M de NaOH, puis ajustez le volume final à 100 mL avec de l’eau ultrapure. Conserver dans des aliquotes de 5 mL à −20 °C.
- Préparer le bouillon de démarrage frais à chaque fois en dissolvant les composés du tableau 4 dans 70 ml d’eau ultrapure en agitant.
- Au stock de démarrage frais, ajoutez les mélanges suivants dans l’ordre : 200 μL de stock de Mix I (tableau 1), 80 μL de Mix II (Tableau 1), 10 μL de Mix III (Tableau 1), 10 μL de Mix IV (Tableau 2), 1 mL de Vitamin Stock (Tableau 3) et 5 mL de Amino Acid Stock (Tableau 4).
- Une fois les bouillons ajoutés, ajustez le volume final à 100 mL en ajoutant 30 mL d’eau ultrapure dans le bécher.
- Compléter le MDP avec des composés du tableau 4. Une fois bien mélangé, stériliser par filtre et conserver à 4 °C pendant un maximum de 2 semaines.
2. Culture du biofilm de S. pneumoniae
- Préparer le milieu RP-10 en mélangeant 445 mL de RPMI 1640 avec 50 mL de sérum fœtal bovin inactivé par la chaleur et 5 mL de pénicilline/streptomycine à 10 000 U/mL et 10 000 μg/mL, respectivement.
- Cultivez la lignée cellulaire de carcinome mucoépidermoïde NCI-H292 (H292). Ajouter les cellules d’un flacon acheté à 5 mL de milieu RP-10 dans une fiole traitée par culture tissulaire T-25. Incuber à 37 °C/5% CO2 pendant 3-5 jours pour atteindre 100% de confluence.
- Vérifiez les cellules au microscope optique en utilisant un grossissement 10x pour évaluer la confluence.
REMARQUE: Lorsque toutes les cellules sont en contact avec d’autres cellules et qu’il n’y a pas d’espaces entre les deux, la confluence souhaitée à 100% est atteinte. - Lavez les cellules 2x dans 5 mL de PBS à température ambiante. Assurez-vous que le tampon est exempt de calcium pour éviter de chélater l’EDTA à l’étape suivante.
- Ajouter 1 mL de trypsine-EDTA dans la fiole et incuber à 37 °C/5 % de CO2 pendant 5 à 10 minutes jusqu’à ce que les cellules se détachent. Neutraliser avec 4 mL de milieu RP-10. Mélanger doucement en pipetant de haut en bas et transférer dans un tube conique de 50 mL.
- Ajouter 500 μL de suspension cellulaire par puits à une plaque de 24 puits traitée par culture tissulaire. À partir d’une fiole confluente de T-25, attendez-vous à 2 × 10 6-4× 106 cellules/mL.
- Le lendemain, vérifiez les cellules au microscope optique pour vous assurer qu’elles sont confluentes, comme à l’étape 2.3. Si ce n’est pas le cas, incuber plus longtemps.
- Une fois que les cellules H292 sont confluentes à 100 % dans la plaque de 24 puits, lavez doucement les cellules 3x avec 1 mL de PBS à température ambiante pour vous assurer qu’il ne reste aucun milieu contenant des antibiotiques ou des débris.
- Après avoir lavé les cellules, ajouter 250 μL/puits de paraformaldéhyde à 4 % pour fixer les cellules. Incuber pendant 1 h sur glace ou toute la nuit à 4 °C.
- La nuit précédant la fixation cellulaire, étaler la souche d’intérêt de S. pneumoniae sur des plaques de gélose au sang et incuber pendant une nuit à 37 °C/5 % de CO2.
REMARQUE : Les données présentées ici portent sur les souches de S. pneumoniae suivantes obtenues par échange collaboratif : isolat d’otite moyenne EF3030 24 du sérotype 19F, souche D3925 d’Avery de sérotype 2 classique et isolat de bactériémie de sérotype 4TIGR4 26. Les souches sont également disponibles dans les collections publiques référencées dans le Tableau des matériaux. - Préparer le MDP plus oxyrase (0,15 U/mL) en ajoutant 100 μL d’oxyrase (30 U/mL) à 20 mL de MDP.
NOTE: L’oxyrase est utilisée pour éliminer l’oxygène afin de permettre la croissance efficace de S. pneumoniae en culture liquide27. - Inoculer les bactéries de la plaque dans du CDM + oxyrase frais en lavant les bactéries de la plaque en ajoutant 1 mL de CDM + oxyrase et en soulevant doucement les colonies bactériennes à l’aide du côté d’un embout de pipette de 1 mL, en prenant soin de ne pas gratter l’agar. Alternativement, utilisez une boucle d’inoculation pour soulever les bactéries et les inoculer dans un tube contenant 1 mL du CDM + oxyrase.
- Diluer les bactéries dans CDM + oxyrase à une DO initialede 600 de 0,05.
- Cultiver les bactéries dans un tube conique de 50 mL à coiffage lâche à 37 °C/5 % de CO 2 jusqu’à ce qu’une DO600 de 0,2 soit atteinte (cela prendra entre2 et 5 h). Vérifiez la DO600 toutes les heures pour vous assurer qu’elle ne dépasse pas 0,2.
- Une fois que la DO a atteint 0,2, faire tourbillonner le tube de culture bactérienne. Ensemencer 0,5 mL de bactéries sur les cellules H292 fixées et ajouter 0,5 mL de MDP + milieu oxyrase par puits. Ajouter 1 mL de CDM + oxyrase aux puits de contrôle sans bactéries. Incuber la plaque pendant 48 h à 34 °C/5% CO2.
NOTE: La croissance à 34 ° C est utilisée pour imiter plus étroitement la température plus basse dans le nasopharynx21. - Toutes les 12 heures suivant l’ensemencement initial, retirer délicatement 0,5 mL du milieu et reconstituer avec 0,5 mL de CDM + oxyrase fraîche. Veillez à ne pas perturber la formation du biofilm. Vérifiez le fond de la plaque pour le biofilm et recherchez une nébulosité croissante au fil du temps en raison de la croissance du biofilm. Pour contrôler la contamination, vérifiez les puits sans bactéries pour vous assurer que les puits de contrôle restent clairs.
- À 48 h après l’inoculation, retirer le surnageant et laver 2x très doucement avec 1 mL de PBS. Remettez en suspension dans 1 mL de MDP frais et pipette vigoureusement de haut en bas pour soulever le biofilm. Pour chaque souche bactérienne, regrouper les bactéries de tous les puits dans un tube conique de 50 mL. Bien mélanger en inclinant doucement le tube bien bouché de haut en bas plusieurs fois.
- Dans le tube conique de 50 mL, ajouter 40 % de glycérol dans le MDP à des volumes égaux pour obtenir une suspension bactérienne avec une concentration finale de 20 % de glycérol. Aliquote 1 mL dans des tubes microcentrifugeurs, surgeler sur de la glace sèche et économiser à −80 °C.
- Avant utilisation, énumérer les bactéries en décongelant une partie aliquote sur de la glace, en faisant tourner le tube à 1 700 × g pendant 5 minutes, en retirant le surnageant, en remettant la pastille en suspension dans 1 mL de PBS et en plaquant les dilutions en série sur des plaques de gélose au sang28.
- Faire pousser les plaques de gélose pendant une nuit à 37 °C/5 % de CO2 et compter les colonies aux dilutions pertinentes pour obtenir la concentration bactérienne en unités formant colonies (UFC)/mL.
REMARQUE: Il est recommandé de dénombrer les bactéries dans les stocks au moins un jour après la congélation ou plus tard, car il y a une baisse de la viabilité bactérienne dans les premières 24 heures. Les aliquotes congelées stockées peuvent être utilisées pour l’infection ultérieure de souris pendant un maximum de 2 mois.
3. Inoculation intranasale de souris avec S. pneumoniae cultivé en biofilm
- Achetez des souris et utilisez-les à l’âge souhaité.
REMARQUE: Les souris âgées de 3-4 mois sont préférées aux jeunes hôtes modèles, et les souris âgées de 21 à 24 mois peuvent être utilisées pour modéliser des personnes âgées de >65 ans29 ans. Les données présentées ici concernent des souris mâles C57BL/6. - Décongeler les aliquotes bactériennes cultivées sur le biofilm sur la glace et faire tourner à 1 700 × g pendant 5 minutes. Retirer et jeter soigneusement le surnageant sans perturber la pastille, laver les bactéries en remettant la pastille en suspension dans 1 mL de PBS et essorer à nouveau à 1 700 × g pendant 5 min. Retirer le surnageant et remettre en suspension la pastille dans le volume nécessaire pour atteindre la concentration désirée (viser 5 × 106 UFC/10 μL pour l’inoculation intranasale). Confirmer les quantités de bactéries administrées en plaquant l’inoculum préparé sur des plaques de gélose au sang comme à l’étape 2.19.
- Inoculer les souris par voie intranasale avec 5 × 106 UFC en pipetant 5 μL de l’inoculum dilué dans chaque naris. Assurez-vous de tenir fermement les souris, en stabilisant la tête, jusqu’à ce que le volume soit inhalé (généralement dans les secondes qui suivent le pipetage du volume dans les narines). Effectuez cette étape en l’absence d’anesthésie pour prévenir l’aspiration pulmonaire de l’inoculum.
4. Infection virale par le virus de la grippe A (IAV)
- À 48 heures après l’inoculation intranasale de S. pneumoniae, décongeler la souche d’IAV d’intérêt sur la glace.
REMARQUE : Les données présentées ici concernent une souche adaptée à la souris du virus de la grippe A A/PR/8/34 H1N1 qui a été obtenue par échange collaboratif30. - Une fois que le virus a dégelé, diluer le virus dans du PBS à la concentration désirée; viser 20 unités de formation de plaques (UFP)/50 μL pour l’infection intratrachéale et 200 UFP/10 μL pour l’infection intranasale. Pour les groupes simulés infectés et uniquement bactériens, utilisez PBS pour inoculer les souris.
- Placez un lubrifiant ophtalmique sur les yeux des souris avant l’anesthésie. Anesthésier les souris à l’aide d’isoflurane à 5% et confirmer l’anesthésie par un pincement ferme des orteils.
- Une fois que l’animal est anesthésié, retirez-le de la chambre isoflurane et infectez immédiatement les souris anesthésiées avec 50 μL (20 UFP) d’IAV par voie intratrachéale à l’aide d’une pince à épiler émoussée pour retirer la langue de la bouche et pipeter le volume de liquide dans la trachée.
- Placez les souris dans une cage séparée et surveillez jusqu’à leur récupération complète (elles sont capables de maintenir une position couchée sternale [capable de se tenir debout sur la poitrine]).
- Après la récupération, inoculer immédiatement par voie intranasale aux souris 10 μL (200 UFP) de IAV en utilisant la méthode d’inoculation de l’étape 3.3.
- Souris domestiques qui ont subi une infection bactérienne et virale simple ou double avec le même groupe d’infection et les séparer des autres groupes.
5. Surveillance des souris pour détecter les symptômes de la maladie
- Surveillez les souris quotidiennement pendant au moins 10 jours et notez aveuglément les signes de maladie comme suit:
- Score comme suit pour la perte de poids: 0 = 5% ou moins; 1 = 5 %-10 %; 2 = 10 %-15 %; 3 = 20 % ou plus. Euthanasier les souris en utilisant l’inhalation de CO2 lorsque le score de perte de poids est de 3.
- Score suivant pour l’activité : 0 = normal/actif; 1 = en mouvement mais légèrement diminué; 2 = diminué; 3 = gravement diminué/léthargique (ne bouge qu’au toucher), 4 = coma/immobile. Euthanasier les souris lorsque le score d’activité est de 3.
- Score suivant pour la posture : 0 = pas d’intuition (normale); 1 = posture légèrement voûtée; 2 = intuition sévère. Euthanasier les souris lorsque le score de posture est de 2.
- Le score suivant pour les yeux : 0 = normal ; 1 = saillie; 1 = coulé; 1 = fermé; 1 = décharge. Il peut s’agir d’une combinaison. Additionnez les totaux pour le score final de l’œil.
- Score suivant pour la respiration: 0 = respiration normale; 1 = irrégulier ou altéré (taux supérieur/inférieur); 2 = laborieux (effort exagéré ou halètement). Euthanasier les souris lorsque le score respiratoire est de 2.
- Sur la base des critères ci-dessus, additionnez les scores individuels pour un score clinique total de sain (0) à extrêmement malade (15). Considérez que toute souris affichant un score total supérieur à 2 est malade. Euthanasier sans cruauté toute souris affichant un score total supérieur à 9 ou les scores indiqués pour chaque critère et les marquer sur la courbe de survie.
6. Traitement des tissus infectés pour le dénombrement bactérien
- À 48 heures suivant l’infection par le VIA, euthanasier les souris.
- Placez la souris en décubitus dorsal. En utilisant de l’éthanol à 70%, vaporisez la poitrine et l’abdomen de la souris pour nettoyer la fourrure. À l’aide d’une pince, pincez la fourrure et la peau au milieu de la souris et coupez la fourrure avec des ciseaux à dissection de 4,5 pouces pour exposer la zone du foie jusqu’à la poitrine.
- Collecte de sang
- À l’aide de ciseaux à dissection, couper doucement dans la cavité péritonéale pour exposer le foie. À l’aide d’une pince, exposez la veine porte hépatique située au sommet du foie, près du diaphragme. Couper la veine porte hépatique à l’aide des ciseaux de dissection. Une fois que le sang commence à s’accumuler dans la cavité péritonéale, prélever 10 μL de sang à l’aide d’une micropipette et placer dans 90 μL de solution anticoagulante (solution d’EDTA à 50 mM dans du PBS) dans un tube microcentrifugé pour le placage de la charge bactérienne.
- Utilisez une micropipette P-1000 pour recueillir le reste du sang, placez-le dans un tube de prélèvement sanguin et centrifugez à 7 600 × g pendant 2 minutes pour recueillir le sérum. Conservez les sérums dans des tubes de microcentrifugation à -80 °C pour une analyse ultérieure de toute cytokine ou métabolite souhaité.
- Prélèvement pulmonaire
- À l’aide de ciseaux à dissection, faites une découpe sur les côtés de la cage thoracique exposée et tirez doucement les côtes vers la tête de la souris pour exposer le cœur. Insérez une aiguille de 25 G fixée à une seringue de 10 ml préremplie de PBS dans le ventricule droit et commencez à perfuser lentement. Recherchez le blanchiment des poumons comme indicateur de perfusion réussie. Rincer lentement pour éviter de casser le tissu pulmonaire.
- Soulevez le cœur avec la pince et faites une coupe pour séparer les poumons et le cœur. Une fois séparé, ramassez tous les lobes du poumon avec la pince et rincez dans un plat avec du PBS stérile pour éliminer tout sang résiduel. Dans une boîte de Pétri, hacher les poumons en petits morceaux et bien mélanger. Retirer la moitié du mélange pulmonaire pour déterminer l’UFC bactérienne ou l’UFP virale et le placer dans un tube à fond rond de 15 mL prérempli de 0,5 mL de PBS pour homogénéisation.
REMARQUE : Il est important de ne pas prélever différents lobes du même poumon pour les diverses évaluations. Au lieu de cela, tous les lobes doivent être hachés, bien mélangés et analysés de manière égale pour les différentes évaluations. - Retirer l’autre moitié du poumon pour la cytométrie en flux (section 7 ci-dessous) et la placer dans une plaque de 24 puits non traitée par culture tissulaire avec chaque puits prérempli de 0,5 mL de RP-10. Laisser à température ambiante jusqu’au traitement.
- Collection de nasopharynx
- Au niveau du cou, utilisez les ciseaux de dissection pour couper la fourrure, puis coupez le muscle et exposez la trachée.
NOTE: La trachée est une structure en forme de tube située sous le muscle. - Placez de petites pinces sous la trachée à une distance de 1 cm de la mâchoire de la souris pour la stabiliser. À l’aide de ciseaux à dissection, faire doucement une fente de 0,1 cm sur la partie antérieure de la trachée, en évitant de couper complètement la trachée.
- Préparez une seringue de 1 mL remplie de 0,5 mL de PBS avec un tube de 0,58 mm attaché à une aiguille de 25 G. Recueillir le lavage nasal en insérant le tube dans la trachée en remontant vers le nasopharynx. Une fois que la résistance est ressentie dans la cavité nasale, placez un tube microcentrifuge au nez et rincez lentement le PBS à travers la trachée pour recueillir le lavage nasal.
- Placez la souris en position couchée. Vaporisez la tête de la souris avec de l’éthanol. Utilisez des ciseaux à dissection pour couper la fourrure et un coussinet mystatique pour exposer l’os de la tête de la souris.
- À l’aide des ciseaux à dissection, faites une coupe de 1 cm sur les côtés de la mandibule et entre les yeux. À l’aide de forceps, retirez lentement les os du visage du corps pour exposer la cavité nasale.
- Utilisez une pince pour retirer délicatement le tissu nasal et placez-le dans un tube inférieur rond prérempli de 0,5 ml de PBS pour l’homogénéisation.
- Au niveau du cou, utilisez les ciseaux de dissection pour couper la fourrure, puis coupez le muscle et exposez la trachée.
- Pour homogénéiser le tissu collecté, nettoyez d’abord la sonde homogénéisante en la mettant dans de l’éthanol à 70% et en allumant l’homogénéisateur à 60% de puissance pendant 30 s. Répétez l’étape dans de l’eau stérile pendant 10 s. Homogénéiser chaque tissu pendant 1 min. Nettoyez la sonde homogénéisante dans de l’eau stérile entre chaque échantillon et dans un tube frais d’éthanol à 70 % entre chaque organe et groupe d’échantillons.
- Dénombrement des nombres bactériens
- Une fois que tous les organes ont été prélevés et homogénéisés, diluer en série sur des plaques de gélose au sang. Pour calculer l’UFC totale, utilisez 10 μL dans la plaque et notez le volume final en mL pour chaque échantillon. Plaquer les échantillons du nasopharynx sur des plaques de gélose au sang additionnées de gentamicine de 3 μg/mL pour sélectionner la croissance de S. pneumoniae tout en inhibant la croissance d’autres microorganismes qui colonisent ce tissu. Incuber pendant une nuit à 37 °C/5% CO2.
- Pour énumérer l’UFC bactérienne pour le poumon et le nasopharynx, comptez d’abord les colonies sur les plaques de gélose au sang. Ensuite, utilisez l’équation (1) et l’équation (2) pour calculer la quantité par mL et le nombre total.
Quantité par mL = nombre de colonies × facteur de dilution × 100 (1)
Nombre total = quantité par mL × volume total par échantillon (2)
NOTE: Dans l’équation (1), 100 est utilisé pour multiplier puisque 10 μL est plaqué, ce qui est une dilution 100 fois de 1 mL. Le volume total par échantillon dans l’équation (2) provient de l’étape 6.7.1, ce qui donne la limite de détection de 100 par organe. - Pour énumérer l’UFC bactérienne Pour la bactériémie, comptez d’abord les colonies sur les plaques de gélose au sang. Ensuite, utilisez l’équation (3) pour déterminer la quantité par mL de sang.
Quantité par mL de sang = nombre de colonies × facteur de dilution × 100 × 10 (3)
NOTE: Dans l’équation (3), 100 est utilisé lorsque 10 μL est plaqué, ce qui correspond à une dilution de 100 fois de 1 mL, et 10 indique une dilution de 1:10 du sang dans l’anticoagulant. Il en résulte une limite de détection de 1 000/mL.
7. Traitement des échantillons pulmonaires pour la cytométrie en flux
- Préparez le support requis comme suit :
- Préparer le PR-10 tel que décrit à l’étape 2.1.
- Préparer le tampon de digestion en mélangeant le RP-10 avec 2 mg/mL de collagénase et 30 μL/mL de DNase I.
- Préparer le tampon de lyse en dissolvant 8,29 g deNH4Cl, 1 g deNaHCO3 et 0,038 g d’EDTA dans 1 L deH2O.
- Préparer 10x tampon FACS en mélangeant 450 mL de HBSS avec 50 mL de FBS inactivé par la chaleur et 5 g d’azoture de sodium.
- Préparer 1x tampon FACS en diluant 50 mL de 10x tampon FACS dans 450 mL de HBSS.
- Prélever les échantillons pulmonaires de l’étape 6.4.3 et les placer dans une plaque à 24 puits. Ajouter 500 μL de tampon de digestion à chaque puits. Incuber pendant 45 min jusqu’à 1 h à 37 °C/5% CO2.
- Préremplir 50 ml de tubes coniques pour chaque échantillon avec 5 mL de RP-10. Lorsque l’incubation est terminée, placez un filtre de 100 μm au sommet du tube conique de 50 ml et mouillez-le avec 1 mL de RP-10.
- À l’aide d’une micropipette P-1000, déplacez les poumons digérés et placez-les sur le filtre. Utilisez le piston d’une seringue de 3 ml pour écraser l’organe. Rincer 2x avec 1 mL de RP-10 à chaque fois.
- Faire tourner les échantillons à 4 °C et 327 × g pendant 5 min. Aspirer le surnageant et remettre en suspension la pastille dans 1 mL de tampon de lyse. Laisser reposer 3 min pour permettre la lyse des globules rouges. Neutraliser avec 5 mL de RP-10.
- Faire tourner les échantillons à 4 °C et 327 × g pendant 5 min. Aspirer le surnageant, remettre en suspension la pastille dans 1 mL de RP-10 et prélever 10 μL pour compter les échantillons.
- Faire tourner les échantillons à 4 °C et 327 × g pendant 5 min. Aspirer le surnageant et remettre en suspension la pastille dans le RP-10 à 2 × 10 6-4 × 106 cellules/mL. Ajouter 60 μL de chaque échantillon dans une plaque de 96 puits pour colorer les types de cellulesdésirés 23 énumérés à l’étape 7.9, tableau 5 et tableau 6.
- Faire tourner la plaque à 4 °C et 327 × g pendant 5 min.
- Pendant ce temps, préparez les mélanges maîtres d’anticorps, fluorescents moins un (FMO), et les témoins à coloration unique avec les anticorps souhaités. Pour colorer les leucocytes polymorphonucléaires (PMN), les macrophages, les monocytes, les cellules dendritiques et les lymphocytes T, utilisez les anticorps et les dilutions finales énumérés dans les tableaux 5 et 6. Utiliser un volume total de 100 μL/puits du mélange d’anticorps. Suivez les dilutions indiquées dans les tableaux pour déterminer le volume approprié du mélange maître et les anticorps individuels requis.
- Lorsque l’essorage est terminé (étape 7.8), décanter le surnageant, remettre les pastilles en suspension dans 100 μL des mélanges d’anticorps, des FMO ou des témoins à coloration unique, et incuber sur de la glace pendant 30 minutes dans l’obscurité.
- Laver les cellules 2x en ajoutant 150 μL de tampon FACS dans les puits et en faisant tourner la plaque à 4 °C et 327 × g pendant 5 min.
- Lorsque l’essorage est terminé, décanter le surnageant, remettre les pastilles en suspension dans 100 μL de tampon de fixation et incuber sur de la glace pendant 20 min.
- Laver les cellules 2x en ajoutant 150 μL de tampon FACS dans les puits et en faisant tourner la plaque à 4 °C et 327 × g pendant 5 min.
- Préparer des tubes FACS marqués avec 200 μL de tampon FACS. Remettez les pastilles en suspension dans 150 μL de tampon FACS. Filtrer individuellement chaque échantillon dans leur tube FACS correspondant à l’aide d’un filtre de 100 μm. Conserver sur la glace ou à 4 °C et à l’abri de la lumière jusqu’à ce que vous soyez prêt à analyser.
- Analysez les cellules à l’aide d’un cytomètre en flux.
8. Dosage de plaque pour le dénombrement de l’IAV
- Préparez le support requis comme suit :
- Préparer le milieu infectieux en dissolvant 2,5 g d’albumine sérique bovine (BSA) dans 40 mL de DMEM en agitant à 37 °C pendant 10-20 minutes jusqu’à dissolution. Filtrer-stériliser en 460 ml de DMEM.
- Préparer la cellulose microcristalline à 2,4 % en dissolvant 1,2 mg de cellulose microcristalline dans 50 mL deH2O. Autoclave sur le liquide et conserver à température ambiante.
- Préparer 5 % de BSA DMEM en dissolvant 2,5 g de BSA dans 40 mL de DMEM en agitant à 37°C pendant 10-20 min. Ajouter les 10 mL restants de DMEM pour obtenir un volume final de 50 mL. Filtrer-stériliser et conserver à 4 °C.
- Préparer 2x MEM/0,5% BSA en mélangeant 1 mL de DMEM BSA à 5% avec 9 mL de 2x MEM.
- Préparer un milieu de recouvrement à faible viscosité en mélangeant un rapport de 1:1 de cellulose microcristalline à 2,4 % et 2 MEM/0,5 % de BSA avec 1 mg/mL de trypsine TPCK (inhibiteur de la chymotrypsine).
- Préparer EMEM/FBS à 10 % en mélangeant 450 mL de milieu essentiel minimal (EMEM) d’Eagle avec 50 mL de FBS inactivé par la chaleur.
- Cultivez la lignée cellulaire de rein canin Madin-Darby (MDCK). Ajouter les cellules d’un flacon acheté à 5 mL d’EMEM/FBS à 10 % dans une fiole traitée par culture tissulaire T-25. Incuber pendant 3 à 5 jours à 37 °C/5 % de CO2 jusqu’à ce que les cellules atteignent 100 % de confluence. Vérifiez la confluence comme à l’étape 2.3.
- Retirer et jeter le milieu de culture, et rincer 2x avec 5 ml de PBS à température ambiante. Ajouter 1 mL de trypsine-EDTA dans la fiole et incuber à 37 °C/5 % de CO2 pendant 10-15 minutes jusqu’à ce que les cellules se détachent. Une fois soulevé, neutraliser avec 4 ml d’EMEM/10 % de FBS pour obtenir une suspension cellulaire à 2 × 105 cellules/mL.
- Ensemencer les cellules MDCK dans une plaque traitée par culture tissulaire à 12 puits en ajoutant 1 mL de cellules en suspension par puits (à 2 × 105 cellules/puits) 1 jour avant de commencer le dosage de la plaque.
REMARQUE: Assurez-vous que les cellules atteignent 100% de confluence avant utilisation et incuber plus longtemps si nécessaire pour atteindre la confluence. - Pour être utilisées comme étalons, effectuer des dilutions en série 10 fois (106-10 1) du stock IAV (d’un titre connu) dans le milieu infectieux indiqué à l’étape 8.1.1. Faire 1,2 mL de chaque dilution à tester en trois exemplaires.
- Décongeler l’organe homogéné sur la glace. Faites tourner sur une centrifugeuse de table à 2 000 × g et recueillez le surnageant clair.
- Répétez l’étape 8.5 mais avec le surnageant des échantillons de l’étape 8.6.
- Aspirez le milieu des cellules et lavez 2x avec 1 mL de PBS pour enlever tout le FBS.
- Ajouter doucement 300 μL de chaque échantillon de dilution étalon ou dilué en série le long du côté de chaque puits, en commençant par la dilution la plus élevée jusqu’à la plus basse, et en trois exemplaires.
- Placer les plaques dans l’incubateur à 37 °C/5% CO2, en agitant la plaque toutes les 10 minutes pendant un total de 50 min. Assurez-vous de les placer à plat dans l’incubateur et ne les empilez pas.
- Après les 50 min, laver les cellules 2x avec 1 mL de PBS.
- Ajouter 2 mL du milieu superposé à faible viscosité dans chaque puits, à l’exception des puits à faible dilution et sans virus; À ceux-ci, ajoutez le milieu d’infection et la trypsine.
- Replacez la plaque dans l’incubateur à 37 °C/5% de CO 2 pendant2 à 4 jours pour obtenir des plaques visualisables à l’œil nu.
- Lavez rapidement les plaques en ajoutant 2 ml de PBS dans chaque puits sur le côté et agitez doucement pour suspendre le milieu de recouvrement à faible viscosité déposé.
- Jetez tout le volume de liquide dans le puits en pipetant doucement le milieu.
- Répétez le lavage une fois de plus avec 2 mL de PBS dans chaque puits, puis jetez tout le volume de liquide par pipetage doux.
- Pour fixer les plaques, ajoutez 500 μL de paraformaldéhyde à 4% dans chaque puits, agitez et laissez reposer pendant 30 minutes.
- Laver lentement sur le côté avec 1 mL de PBS; Ensuite, jetez doucement le liquide.
- Ajouter 500 μL de violet cristallin à 1 % (dilué dans de l’eau) à chaque puits pour couvrir la monocouche cellulaire. Incuber pendant 5 min.
- Laver avec 1 ml d’eau du robinet. Assurez-vous d’éliminer tout le liquide dans le puits par pipetage doux. Placez l’assiette à l’envers sur un mate-couches pour sécher pendant la nuit.
- Comptez les plaques visuellement et enregistrez les images sur n’importe quel imageur disponible.
Résultats
Des S. pneumoniae cultivés sur biofilm (figure 1A) ont été utilisés pour infecter des souris (figure 1B) à l’aide d’un petit inoculum de 10 μL administré par voie intranasale à des souris non anesthésiées. Cet inoculum de petit volume entraîne un portage pneumococcique constant limité au nasopharynx (figures 2A, groupes +sp) tout en évitant la propagation systémique (groupes figure 2B, C +sp). Deux jours après l’inoculation intranasale, les souris ont été infectées par un virus de la grippe A H1N1 adapté aux murins A/PR/8/34 (IAV)22,30 administré par voie intranasale et intratrachéale pour obtenir une administration constante de quantités spécifiques au nasopharynx et aux poumons 23.
Ici, le modèle a été utilisé pour comparer l’évolution de la maladie à la suite d’une infection virale chez des souris déficientes par voie intranasale avec différentes souches de S. pneumoniae, y compris TIGR4 et D39, qui sont des souches invasives entraînant une pneumonie qui évolue vers une bactériémie, et EF3030, qui est une souche d’otite moyenne 21,24,25,26,31. La présentation de la maladie chez les souris co-infectées par S. pneumoniae/IAV dépendait de la souche bactérienne (figure 2). Bien qu’il n’y ait pas eu de différence significative dans le nombre de bactéries du nasopharynx (figure 2A) entre les souches, S. pneumoniae TIGR4 et D39, mais pas EF3030, disséminées dans les poumons 48 heures après l’infection par le VAI (figure 2B). Quarante pour cent des souris infectées par voie intranasale par S. pneumoniae TIGR4 ont présenté une dissémination bactérienne dans les poumons, et parmi celles-ci, la moitié d’entre elles sont devenues bactériémiques (Figure 2C), ce qui correspond aux résultats antérieurs23.
Les souris infectées par voie intranasale par S. pneumoniae D39 ont montré une dissémination plus efficace, car une propagation aux poumons a été observée chez 100 % des souris co-infectées (figure 2B). Comme pour S. pneumoniae TIGR4, la moitié des personnes ont présenté une bactériémie (figure 2C). Dans le suivi de la survie globale, quelle que soit la souche bactérienne, le taux de survie des souris co-infectées était significativement inférieur à celui des souris individuellement confrontées à S. pneumoniae seule pour toutes les souches testées (Figure 2D). Par rapport aux souris témoins exposées avec le IAV seul, les souris infectées par voie intranasale par S. pneumoniae TIGR4 et D39, mais pas EF3030, ont affiché des taux accélérés de maladie. Au jour 2 après l’infection par le VAI, 30 % (D39) et 20 % (TIGR4) des souris avaient succombé, tandis que les groupes témoins composés uniquement d’IAV n’ont commencé à succomber qu’au jour 5 après la provocation (Figure 2D). Les souris co-infectées par S. pneumoniae EF3030 et IAV présentaient des symptômes retardés, plus semblables à ceux des témoins IAV seuls (Figure 2D). Ces résultats démontrent que le modèle de co-infection entraîne chez les jeunes souris en bonne santé une maladie dépendante de la souche bactérienne, ce qui le rend idéal pour explorer les facteurs bactériens requis à chaque étape de la progression de la maladie.
Ce modèle a été utilisé pour évaluer la présence de diverses cellules immunitaires dans les poumons (types de cellules et stratégie de déclenchement à la figure 3) à la suite d’une infection par le VAI chez des souris inoculées par voie intranasale avec différentes souches de S. pneumoniae. Les souches bactériennes D39 et TIGR4, qui se sont dispersées dans les poumons à la suite d’une infection par le VAI, ont provoqué une augmentation significative par rapport à la ligne de base (non infectées) de l’afflux de cellules immunitaires inflammatoires de la circulation, telles que les neutrophiles (PMN) et les monocytes, alors que EF3030 ne l’a pas fait (Figure 4A-C). L’infection par le VAI à elle seule a entraîné une augmentation significative par rapport à la ligne de base de l’afflux de cellules immunitaires importantes pour la défense de l’hôte contre l’infection virale, telles que les cellules NK et les cellules T gamma-delta (Figure 4A-C). Ces réponses antivirales ont été émoussées de façon significative chez les souris infectées par S. pneumoniae par voie intranasale avant la provocation virale (figure 4A-C). Ceci est cohérent avec des études antérieures évaluant les réponses aux cytokines qui ont révélé que le transport de S. pneumoniae atténuait la production d’interférons de type I et nuisait à la capacité de l’hôte à contrôler les charges d’IAV dans les poumons23. Ces résultats démontrent que le modèle de co-infection peut être utilisé pour étudier comment les réponses immunitaires changent dans les infections mono par rapport aux infections polymicrobiennes.
Ce modèle a également été utilisé pour évaluer l’effet du vieillissement sur l’évolution de la maladie à la suite d’une infection par le VAI chez des souris infectées par voie intranasale par S. pneumoniae TIGR4. Chez les souris infectées isolément, les titres viraux ne variaient pas entre les cohortes jeunes et âgées (figure 5A)23. Comme dans les études précédentes23, les souris âgées ont présenté des signes de maladie plus précoces et significativement plus graves que leurs homologues jeunes, comme le démontrent les scores cliniques plus élevés (Figure 5B). Conformément aux symptômes de la maladie, les souris âgées inoculées avec S. pneumoniae ont commencé à mourir plus rapidement dans les 24 heures suivant l’infection par le VAI et toutes ont succombé à la maladie, tandis que les jeunes témoins ont survécu à l’infection à un taux significativement plus élevé (33%) (Figure 5C). Ces résultats démontrent que le modèle de co-infection peut être utilisé pour détecter une maladie plus grave chez les hôtes vulnérables, ce qui le rend idéal pour explorer les facteurs de l’hôte qui confèrent une résistance ou une susceptibilité à la co-infection.
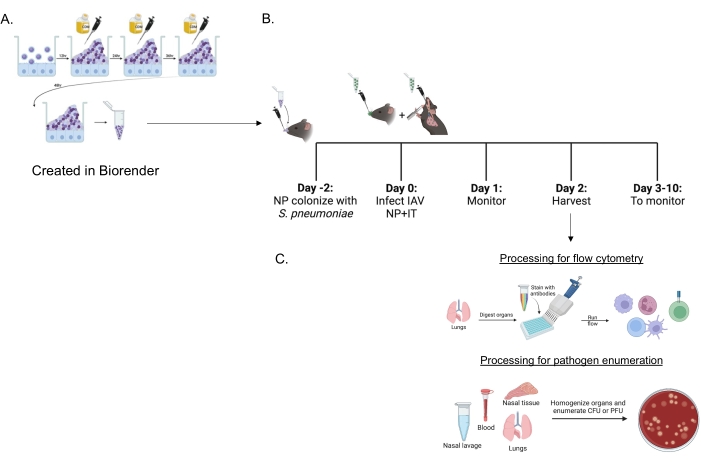
Figure 1 : Chronologie de la co-infection et du traitement des organes pour l’évaluation de l’afflux de cellules immunitaires et de la charge pathogène. (A) Streptococcus pneumoniae est cultivé dans des biofilms. (B) Les souris sont inoculées par voie intranasale avec 5 × 106 UFC de la souche S. pneumoniae cultivée sur biofilm indiquée pour établir un portage nasopharyngé ou ne sont pas traitées. Quarante-huit heures plus tard, les souris sont soit simulées traitées avec PBS, soit reçoivent 200 PFU du virus de la grippe A PR8 par voie intranasale et 20 PFU par voie intratrachéale. Les souris sont surveillées au fil du temps pour les scores cliniques de la maladie et la survie. (C) 48 h après l’infection par le VIA, on évalue l’UFC bactérienne ou l’UFP virale dans les différents organes ou l’afflux de cellules immunitaires dans les poumons. Abréviations : UFC = unités formant colonies; PFU = unités formant des plaques; IAV = virus de la grippe A PR8; IT = intratrachéale; NP = nasopharyngé. Veuillez cliquer ici pour voir une version agrandie de cette figure.
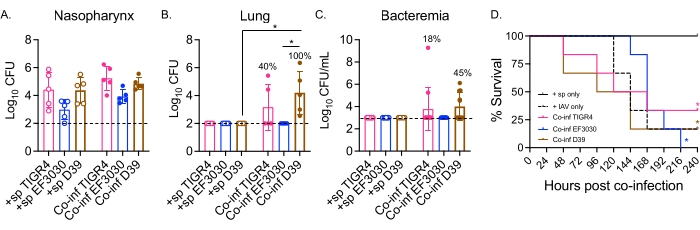
Figure 2 : La double infection intranasale/intratrachéale par le VAI des souris inoculées par S. pneumoniae entraîne une propagation bactérienne et une maladie qui dépend de la souche bactérienne. De jeunes souris C57BL/6 (B6) mâles (âgées de 10 à 12 semaines) ont été infectées comme le montre la figure 1. Le nombre de bactéries dans (A) le nasopharynx, (B) les poumons et (C) le sang a été déterminé 48 heures après l’infection par le VAI. (B,C) Les pourcentages indiquent la fraction de souris qui ont présenté une propagation. (D) La survie a été surveillée pendant 10 jours après l’infection par le virus de l’IAV. Les données regroupées de (A, B) n = 5, (C) n = 11 et (D) n = 6 souris par groupe sont présentées. Chaque cercle correspond à une souris, et les lignes pointillées indiquent la limite de détection. (A-C)*, indique une différence significative (p < 0,05) entre les groupes indiqués, tels que déterminés par le test de Kruskal-Wallis. (D) *, indique une différence significative (p < 0,05) entre les souris +sp et Co-inf par souche bactérienne, déterminée par le test log-rank (Mantel-Cox). Abréviations : +sp = souris infectées par voie intranasale par des bactéries utilisant uniquement la souche indiquée; Co-inf = souris infectées par des bactéries infectées par le virus de l’IAV; IAV = souris ayant reçu le virus de la grippe A; UFC = unités formant colonies. Veuillez cliquer ici pour voir une version agrandie de cette figure.
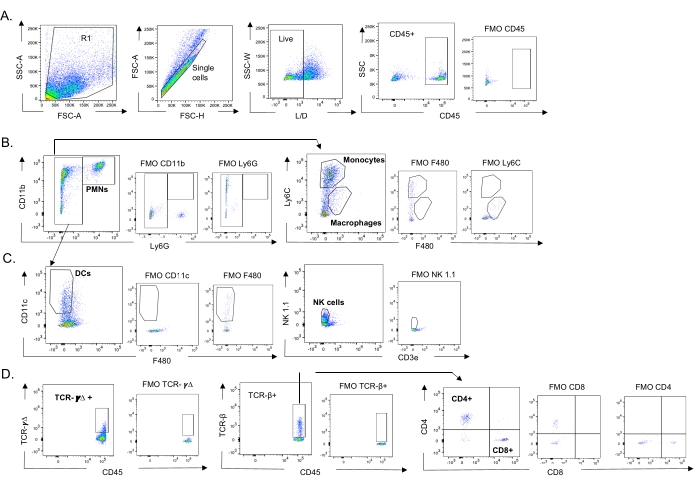
Figure 3 : Stratégie de contrôle des cellules immunitaires. Les poumons ont été récoltés et l’afflux de cellules immunitaires a été déterminé par cytométrie en flux. La stratégie de contrôle représentative des différents types de cellules est présentée. (A) CD45+, cellules individuelles vivantes ont été fermées et les pourcentages de (B) PMN (Ly6G +, CD11b +), macrophages (Ly6G-, Ly6C-, F480+) et monocytes (Ly6G-, Ly6C +), (C) DCs (Ly6G-, CD11c+) et NK (NK1.1+, CD3-), (D) TCR- γΔ et CD8 (CD8+, TCRβ+) et CD4 (CD4+, TCRβ+ ) ont été déterminées. Abréviations : SSC-A = aire de pic de diffusion latérale; FSC-A = aire du pic de diffusion vers l’avant; FSC-H = hauteur du pic de diffusion vers l’avant; SSC-W = largeur latérale du pic de dispersion; L/D = vivant/mort; FMO = fluorescent moins un; NK = tueur naturel; PMN = leucocyte polymorphonucléaire; DC = cellule dendritique; TCR = récepteur des lymphocytes T. Veuillez cliquer ici pour voir une version agrandie de cette figure.
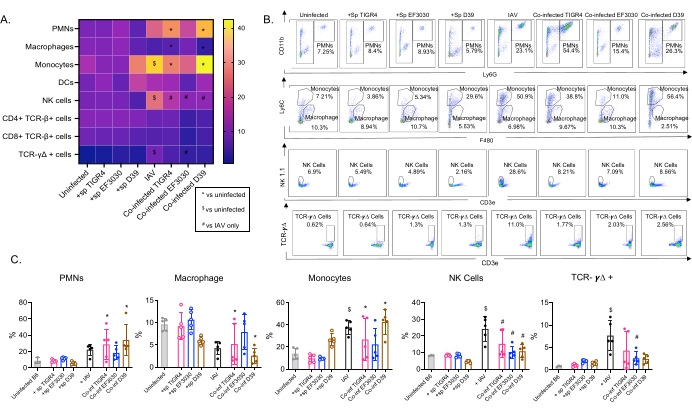
Figure 4 : Les réponses immunitaires pulmonaires dépendent de la souche bactérienne. Les jeunes souris mâles C57BL/6 (âgées de 10 à 12 semaines) n’étaient pas infectées, inoculées seules avec la souche indiquée de Streptococcus pneumoniae (+sp), individuellement infectées par le IAV (IAV) ou co-infectées par S. pneumoniae et IAV (Co-inf). Quarante-huit heures après l’infection par le VAI (voir le plan expérimental à la figure 1), les poumons ont été prélevés et l’afflux de cellules immunitaires a été déterminé par cytométrie en flux suivant la stratégie de déclenchement de la figure 3. (A) Les pourcentages moyens de chaque type de cellule indiqué à l’intérieur de la grille CD45 sont affichés pour tous les groupes de traitement sur la carte thermique. (B) Des diagrammes de points représentatifs des types de cellules qui présentaient des différences significatives entre les traitements sont présentés pour chaque groupe de souris. (C) Les pourcentages des types de cellules immunitaires indiqués sont indiqués. Chaque cercle correspond à une souris. (A,C) Les données regroupées de n = 5 souris par groupe sont présentées. *, indique une différence significative (p < 0,05) entre Co-inf et non infecté; $, indique un écart significatif entre IAV et non infecté; #, indique une différence significative entre Co-inf et IAV seul. Les différences significatives entre les groupes de provocation pour chaque type de cellule ont été déterminées par ANOVA suivie du test de Tukey. Abréviations : NK = tueur naturel; PMN = leucocyte polymorphonucléaire; DC = cellule dendritique; TCR = récepteur des lymphocytes T; IAV = virus de la grippe A. Veuillez cliquer ici pour voir une version agrandie de cette figure.
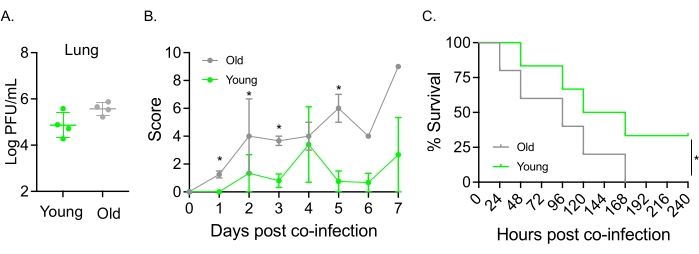
Figure 5 : Vieillissement et sensibilité accrue de l’hôte à la co-infection IAV/Streptococcus pneumoniae. Des souris mâles C57BL/6 jeunes (10-12 semaines) et âgées (21-22 mois) ont été co-infectées par S. pneumoniae TIGR4 i.n. et IAV i.n. et i.t. (comme dans la figure 1) ou individuellement confrontées à l’IAV seul. (A) Les titres viraux ont été déterminés 48 heures plus tard. Les astérisques indiquent une signification statistique (p < 0,05) telle que déterminée par le test t de Student. Les données sont regroupées à partir de n = 4 souris par groupe. (B) le score clinique et (C) la survie ont été surveillés au fil du temps. (B) La moyenne ± MEB combinée à partir de n = 6 souris par groupe est indiquée. Les astérisques indiquent une signification statistique (p < 0,05) entre les souris jeunes et les souris âgées au point temporel indiqué, tel que déterminé par le test de Mann-Whitney. (C) Les données sont regroupées à partir de n = 6 souris par groupe. Les astérisques indiquent une signification statistique (p < 0,05) entre les souris jeunes et les souris âgées, déterminée par le test de log-rank (Mantel-Cox). Abréviations : IAV = virus de la grippe A; i.n. = intranasale; i.t. = intratrachéale; SEM = erreur type de la moyenne. La figure 5A est reproduite avec la permission de Joma et coll.23. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Mix I stock pour CDM | |
| Adénine | 0,1 g |
| D-Alanine | 0,25 g |
| CaCl2 Anhydre | 0,025 g |
| Sulfate de manganèse | 0,03 g |
| Cyanocobalamine | 100 μL de 10 mg/mL de stock |
| Acide para-aminobenzoïque | 400 μL de 5 mg/mL de stock |
| Pyridoxamine 2HCl | 100 μL de 10 mg/mL de stock |
| Stock Mix II pour MDP | |
| Guanine | 0,05 g |
| Uracile | 0,05 g |
| Stock Mix III pour MDP | |
| Nitrate ferrique 9H2O | 50 mg/mL |
| Sulfate ferrique 7H2O | 10 mg/mL |
| Stock Mix IV pour MDP | |
| Bêta-nicotinamide adénine dinucléotide | 25 mg/mL |
Tableau 1: Stocks des combinaisons I, II, III et IV pour le MDP. Abréviation : MDP = milieu chimiquement défini.
| Mélange de vitamines pour MDP | |
| Chlorhydrate de pyridoxal | 0,8 g |
| Thiamine Cl2 | 0,4 g |
| Riboflavine | 0,4 g |
| Ca-pantothénate | 0,4 g |
| Biotine | 0,04 g |
| Acide folique | 0,4 g |
| Niacinamide | 0,4 g |
Tableau 2 : Mélange de vitamines pour le MDP. Abréviation : MDP = milieu chimiquement défini.
| Stock d’acides aminés pour MDP | |
| L-Alanine | 0,480 g |
| L-Arginine | 0,250 g |
| L-Asparagine | 0,700 g |
| Acide L-aspartique | 0,600 g |
| L-Cystéine | 1.000 g |
| L-Cystine | 0,100 g |
| Acide L-glutamique | 0,200 g |
| L-Glutamine | 0,780 g |
| L-Glycine | 0,350 g |
| L-Histidine | 0,300 g |
| L-Isoleucine | 0,430 g |
| L-Leucine | 0,950 g |
| L-Lysine | 0,880 g |
| L-méthionine | 0,250 g |
| L-phénylalanine | 0,550 g |
| L-Proline | 1,350 g |
| L-Sérine | 0,680 g |
| L-Thréonine | 0,450 g |
| L-tryptophane | 0,100 g |
| L-Valine | 0,650 g |
Tableau 3 : Stock d’acides aminés pour le MDP. Abréviation : MDP = milieu chimiquement défini.
| Stock de démarrage pour MDP | |
| Dextrose | 1,0 g |
| Sulfate de magnésium-7-hydrate | 0,070 g |
| Phosphate de potassium dibasique | 0,02 g |
| Phosphate de potassium monobasique | 0,1 g |
| Acétate de sodium anhydre | 0,45 g |
| Sodium Bicarbonate | 0,25 g |
| Phosphate de sodium dibasique | 0,735 g |
| Phosphate de sodium monobasique | 0,32 g |
| Suppléments finaux pour MDP | |
| Chlorure de choline | 0,1 g |
| L-cystéine HCl | 0,075 g |
| Sodium Bicarbonate | 0,25 g |
Tableau 4: Stock de démarrage et suppléments finals pour le MDP. Abréviation : MDP = milieu chimiquement défini.
| Anticorps/fluorophore | Clone | Facteur de dilution |
| L/D pour l’excitation UV | N/A | 0.38888889 |
| Ly6G AF 488 | 1A8 | 0.25 |
| CD11b APC | M1/70 | 0.25 |
| CD11c PE | N418 | 0.18055556 |
| Bloc Souris Fc | 2.4G2 | 0.11111111 |
| F4/80 PE Cy7 | BM8 | 0.18055556 |
| Ly6C BV605 | AL-21 | 0.25 |
| CD103 BV 421 | M290 | 0.18055556 |
| CD45 APC-eF-780 | 30-F11 | 0.18055556 |
Tableau 5 : Panel d’anticorps 1.
| Anticorps/fluorophore | Clone | Facteur de dilution |
| L/D pour l’excitation UV | N/A | 0.388888889 |
| TCR-β APC Cy7 | N° H57-597 | 0.180555556 |
| CD4 V450 (Bleu Pacifique) | RM4-5 | 0.25 |
| CD8 BV650 | 53-6.7 | 0.180555556 |
| Bloc Souris Fc | 2.4G2 | 0.111111111 |
| CD45 PE | 30-F11 | 0.180555556 |
| CD3 AF488 | 145-2C11 | 0.180555556 |
| TCR- γΔ APC | GL-3 | 0.180555556 |
| NK1.1 AF 700 | PK136 | 0.180555556 |
Tableau 6 : Panel d’anticorps 2.
Discussion
La plupart des études expérimentales existantes sur la co-infection à S. pneumoniae/IAV reposent sur l’administration de bactéries dans les poumons de souris pré-infectées par le virus de l’IAV. Ces modèles ont permis d’identifier les changements dans l’environnement pulmonaire et la réponse immunitaire systémique qui rendent l’hôte vulnérable à une infection bactérienne secondaire 15,16,17,32,33,34,35,36,37. Cependant, ces modèles n’ont pas réussi à imiter la transition de S. pneumoniae d’un colonisateur asymptomatique à un agent pathogène capable de causer des infections pulmonaires et systémiques graves. De plus, ces modèles ne conviennent pas à l’étude des facteurs de l’hôte et des interactions hôte-pathogène dans les voies respiratoires supérieures qui contribuent à la susceptibilité à l’infection. Un modèle antérieur pour le mouvement des pneumocoques du nasopharynx vers le poumon après une infection par le VAI reposait sur une infection bactérienne du nasopharynx suivie d’une infection virale. Cependant, il n’a pas réussi à reproduire les signes graves de maladie observés chez les patients humains21. Le modèle modifié d’infection murine décrit ici récapitule la transition de S. pneumoniae d’un portage asymptomatique à un agent pathogène qui cause une maladie clinique grave.
Une étape critique de ce modèle consiste à établir une infection à S. pneumoniae dans le nasopharynx. Streptococcus pneumoniae forme des biofilms et colonise le nasopharynx à différentes efficacités 21,38. Pour établir une infection constante, au moins 5 × 106 UFC des souches bactériennes cultivées sur biofilm testées jusqu’à présent sont nécessaires23. Il est recommandé que toute nouvelle souche bactérienne soit testée pour une infection stable du nasopharynx avant l’infection virale. Pour la co-infection virale, des études antérieures ont montré qu’une infection intranasale par IAV est nécessaire pour la dispersion de la bactérie du nasopharynx21,22,23. Dans ces études antérieures, 500 UFP de l’IAV pour l’administration intranasale ont été utilisées, tandis que dans cette étude, 200 UFP étaient suffisantes pour augmenter le nombre de bactéries dans le nasopharynx. L’infection par le VAI ne se limite pas aux voies respiratoires supérieures et peut se propager aux poumons39,40, ce qui est essentiel pour rendre l’environnement pulmonaire plus permissif pour les infections bactériennes15,16,41. L’administration de l’IAV aux poumons peut être réalisée par administration intranasale ou par installation intratrachéale de souris anesthésiées. Des travaux antérieurs avec des souris BALB / cByJ ont révélé que l’administration intranasale entraîne une pneumonie virale21; cependant, l’accès de l’inoculum aux poumons après l’inoculation intranasale est plus restreint chez les souris C57BL/6. Chez les souris C57BL/6, une installation intratrachéale est nécessaire pour une administration régulière du virus23. Dans ce modèle, la colonisation bactérienne préalable accélère la présentation des symptômes de la maladie après une infection virale23. Comme l’infection virale peut elle-même causer des symptômes de la maladie avec une variation potentielle de la cinétique, il est recommandé de tester d’abord une gamme de doses pour toute nouvelle souche virale testée et de choisir une dose qui révèle une cinétique accélérée chez les hôtes co-infectés.
Les poumons fournissent une autre lecture critique pour l’évaluation de la maladie dans ce modèle. Pour l’évaluation de la charge pathogène et de l’afflux de cellules immunitaires, un poumon de la même souris peut être utilisé. Cependant, comme la gravité de l’infection et de l’inflammation peut différer d’un lobe à l’autre, il est recommandé de ne pas prendre différents lobes du même poumon pour les différentes évaluations. Au contraire, tous les lobes peuvent être hachés en petits morceaux, bien mélangés ensemble, puis analysés également pour les différentes évaluations. De même, le nasopharynx peut être utilisé pour le dénombrement de l’UFC bactérienne ou PFU virale et la réponse immunitaire. Cependant, le nombre de cellules obtenues à partir des lavages et des tissus est trop faible pour effectuer une cytométrie en flux sans regrouper les échantillons de souris du même groupe. Alternativement, l’inflammation dans le nasopharynx peut être évaluée histologiquement23.
Une caractéristique essentielle de ce modèle est qu’il récapitule la maladie clinique observée chez les patients. Chez les humains, la pneumonie pneumococcique secondaire à la suite d’une infection par le VAI entraîne souvent des signes évidents de maladie, notamment de la toux, de la dyspnée, de la fièvre et des douleurs musculaires pouvant entraîner des hospitalisations, une insuffisance respiratoire etmême la mort8,15,42,43. Ce modèle récapitule les signes graves de maladie clinique observés chez l’homme en termes de difficulté à respirer (reflétée dans le score respiratoire) et de malaise global (reflété dans les scores de posture et de mouvement) affichés par les souris, ainsi que de décès chez certains des jeunes témoins en bonne santé. Les symptômes exacerbés de la maladie chez les souris co-infectées sont probablement le résultat à la fois de la dissémination bactérienne dans les poumons et de l’altération de la clairance virale chez les souris atteintes de portage pneumococcique23. Une limite du modèle est que l’incidence de la maladie clinique et la dissémination bactérienne à partir du nasopharynx varient d’une souris à l’autre et sont influencées par la souche bactérienne, l’âge de l’hôte et le génotype21,22,23. En conséquence, pour les souches invasives, la progression de l’infection localisée (sans bactériémie détectable) à la mort peut se produire dans les 24 heures. Par conséquent, pour une véritable évaluation de la propagation systémique, la bactériémie doit être suivie sur des intervalles plus courts (toutes les 6 à 12 heures). De même, le score de la maladie peut changer rapidement, en particulier dans les 72 premières heures suivant la co-infection. Par conséquent, pour suivre de près les symptômes de la maladie, il est conseillé de surveiller les souris trois fois par jour pendant les jours 1 à 3 après l’infection par le VAI.
En résumé, ce modèle reproduit le déplacement de S. pneumoniae d’un colonisateur asymptomatique du nasopharynx à un agent pathogène capable de causer une maladie pulmonaire et systémique lors de l’infection par le VAI. Dans ce modèle, l’IAV déclenche la transition de S. pneumoniae en modifiant le comportement bactérien dans le nasopharynx, en augmentant la propagation bactérienne au poumon et en modifiant l’immunité antibactérienne23. De même, le portage bactérien atténue les réponses immunitaires antivirales et altère l’élimination de l’IAV des poumons23. Cela rend ce modèle idéal pour analyser les changements dans les réponses immunitaires dans les infections simples par rapport aux infections polymicrobiennes. De plus, l’évolution de la maladie après une co-infection dépend, en partie, de la souche de pneumocoques présente dans le nasopharynx. Par conséquent, le modèle est adapté pour disséquer les facteurs bactériens nécessaires à la colonisation asymptomatique par rapport à la transition pathogène de S. pneumoniae. Enfin, ce modèle reproduit la susceptibilité du vieillissement aux co-infections et, bien que cela n’ait pas été testé ici, il peut être facilement utilisé pour évaluer l’impact des antécédents de l’hôte sur l’évolution de la maladie. En conclusion, la séparation du portage et de la maladie en étapes distinctes offre la possibilité d’analyser les variantes génétiques des agents pathogènes et de l’hôte, ce qui permet d’examiner en détail les interactions d’un pathobionte important avec l’hôte à différentes phases de la progression de la maladie. À l’avenir, ce modèle pourra être utilisé pour adapter les options de traitement aux hôtes vulnérables.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous tenons à remercier Nick Lenhard pour la lecture critique et l’édition de ce manuscrit. Nous tenons également à remercier Andrew Camilli et Anthony Campagnari pour les souches bactériennes et Bruce Davidson pour les souches virales. Ce travail a été soutenu par la subvention du National Institute of Health (R21AG071268-01) à J.L. et le National Institute of Health Grants (R21AI145370-01A1), (R01AG068568-01A1), (R21AG071268-01) à E.N.B.G.
matériels
| Name | Company | Catalog Number | Comments |
| 4-Aminobenzoic acid | Fisher | AAA1267318 | Mix I stock |
| 96-well round bottom plates | Greiner Bio-One | 650101 | |
| 100 µm Filters | Fisher | 07-201-432 | |
| Adenine | Fisher | AC147440250 | Mix I stock |
| Avicel | Fisher | 501785325 | Microcyrstalline cellulose |
| BD Cytofix Fixation Buffer | Fisher | BDB554655 | Fixation Buffer |
| BD Fortessa | Flow cytometer | ||
| BD Intramedic Polyethylene Tubing | Fisher | 427410 | Tubing for nasal lavage |
| BD Disposable Syringes with Luer-Lok Tips (1 mL) | Fisher | 14-823-30 | |
| BD Microtainer Capillary Blood Collector and BD Microgard Closure | Fisher | 02-675-185 | Blood collection tubes |
| Beta-Nicotinamide adenine dinucleotide | Fisher | AAJ6233703 | Mix IV stock |
| Biotin | Fisher | AC230090010 | Vitamin stock |
| C57BL/6J mice | The Jackson Laboratory | #000644 | Mice used in this study |
| Calcium Chloride Anhydrous | Fisher Chemical | C77-500 | Mix I stock |
| CD103 BV 421 | BD Bioscience | BDB562771 | Clone: M290 DF 1:200 |
| CD11b APC | Invitrogen | 50-112-9622 | Clone: M1/70, DF 1:300 |
| CD11c PE | BD Bioscience | BDB565592 | Clone: N418 DF 1:200 |
| CD3 AF 488 | BD Bioscience | OB153030 | Clone: 145-2C11 DF 1:200 |
| CD4 V450 | BD Horizon | BDB560470 | Clone: RM4.5 DF 1:300 |
| CD45 APC eF-780 | BD Bioscience | 50-112-9642 | Clone: 30-F11 DF 1:200 |
| CD45 PE | Invitrogen | 50-103-70 | Clone: 30-F11 DF 1:200 |
| CD8α BV 650 | BD Horizon | BDB563234 | Clone: 53-6.7 DF 1:200 |
| Choline chloride | Fisher | AC110290500 | Final supplement to CDM |
| Corning Disposable Vacuum Filter/Storage Systems | Fisher | 09-761-107 | Filter sterilzation apparatus |
| Corning Tissue Culture Treated T-25 Flasks | Fisher | 10-126-9 | |
| Corning Costar Clear Multiple Well Plates | Fisher | 07-201-590 | |
| Corning DMEM With L-Glutamine and 4.5 g/L Glucose; Without Sodium Pyruvate | Fisher | MT10017CM | |
| Cyanocobalamin | Fisher | AC405925000 | Mix I stock |
| D39 | National Collection of Type Culture (NCTC) | NCTC 7466 | Streptococcus pneumoniae strain |
| D-Alanine | Fisher | AAA1023114 | Mix I stock |
| D-Calcium pantothenate | Fisher | AC243301000 | Vitamin stock |
| Dextrose | Fisher Chemical | D16-500 | Starter stock |
| Dnase | Worthington Biochemical | LS002147 | |
| Eagles Minimum Essential Medium | ATCC | 30-2003 | |
| EDTA | VWR | BDH4616-500G | |
| EF3030 | Center for Disease Control and Prevention | Available via the isolate bank request | Streptococcus pneumoniae strain, request using strain name |
| F480 PE Cy7 | BD Bioscience | 50-112-9713 | Clone: BMB DF 1:200 |
| Falcon 50 mL Conical Centrifuge Tubes | Fisher | 14-432-22 | 50 mL round bottom tube |
| Falcon Round-Bottom Polypropylene Test Tubes With Cap | Fisher | 14-959-11B | 15 mL round bottom tube |
| Falcon Round-Bottom Polystyrene Test Tubes (5 mL) | Fisher | 14-959-5 | FACS tubes |
| FBS | Thermofisher | 10437-028 | |
| Ferric Nitrate Nonahydrate | Fisher | I110-100 | Mix III stock |
| Fisherbrand Delicate Dissecting Scissors | Fisher | 08-951-5 | Instruments used for harvest |
| Fisherbrand Disposable Inoculating Loops | Fisher | 22-363-602 | Inoculating loops |
| Fisherbrand Dissecting Tissue Forceps | Fisher | 13-812-38 | Forceps for harvest |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5 mL | Fisher | 05-408-137 | Micocentrifuge tubes |
| Fisherbrand Sterile Syringes for Single Use (10 mL) | Fisher | 14-955-459 | |
| Folic Acid | Fisher | AC216630500 | Vitamin stock |
| Gibco RPMI 1640 (ATCC) | Fisher | A1049101 | |
| Gibco DPBS, no calcium, no magnesium | Fisher | 14190250 | |
| Gibco HBSS, calcium, magnesium, no phenol red | Fisher | 14025134 | |
| Gibco MEM (Temin's modification) (2x), no phenol red | Fisher | 11-935-046 | |
| Gibco Penicillin-Streptomycin (10,000 U/mL) | Fisher | 15-140-122 | |
| Gibco Trypan Blue Solution, 0.4% | Fisher | 15-250-061 | |
| Gibco Trypsin-EDTA (0.25%), phenol red | Fisher | 25-200-056 | |
| Glycerol (Certified ACS) | Fisher | G33-4 | |
| Glycine | Fisher | AA3643530 | Amino acid stock |
| Guanine | Fisher | AAA1202414 | Mix II stock |
| Invitrogen UltraComp eBeads Compensation Beads | Fisher | 50-112-9040 | |
| Iron (II) sulfate heptahydrate | Fisher | AAA1517836 | Mix III stock |
| L-Alanine | Fisher | AAJ6027918 | Amino acid stock |
| L-Arginine | Fisher | AAA1573814 | Amino acid stock |
| L-Asparagine | Fisher | AAB2147322 | Amino acid stock |
| L-Aspartic acid | Fisher | AAA1352022 | Amino acid stock |
| L-Cysteine | Fisher | AAA1043518 | Amino acid stock |
| L-Cysteine hydrochloride monohydrate | Fisher | AAA1038914 | Final supplement to CDM |
| L-Cystine | Fisher | AAA1376218 | Amino acid stock |
| L-Glutamic acid | Fisher | AC156211000 | Amino acid stock |
| L-Glutamine | Fisher | O2956-100 | Amino acid stock |
| L-Histidine | Fisher | AC166150250 | Amino acid stock |
| LIFE TECHNOLOGIES LIVE/DEAD Fixable Blue Dead Cell Stain Kit, for UV excitation | Invitrogen | 50-112-1524 | Clone: N/A DF 1:500 |
| L-Isoleucine | Fisher | AC166170250 | Amino acid stock |
| L-Leucine | Fisher | BP385-100 | Amino acid stock |
| L-Lysine | Fisher | AAJ6222514 | Amino acid stock |
| L-Methionine | Fisher | AAA1031822 | Amino acid stock |
| Low endotoxin BSA | Sigma Aldrich | A1470-10G | |
| L-Phenylalanine | Fisher | AAA1323814 | Amino acid stock |
| L-Proline | Fisher | AAA1019922 | Amino acid stock |
| L-Serine | Fisher | AC132660250 | Amino acid stock |
| L-Threonine | Fisher | AC138930250 | Amino acid stock |
| L-Tryptophan | Fisher | AAA1023014 | Amino acid stock |
| L-Valine | Fisher | AAA1272014 | Amino acid stock |
| Ly6C BV 605 | BD Bioscience | BDB563011 | Clone: AL-21 DF 1:300 |
| Ly6G AF 488 | Biolegend | NC1102120 | Clone: IA8, DF 1:300 |
| Madin-Darby Canine Kidney (MDCK) cells | American Type Culture Collection (ATCC) | CCL-34 | MDCK cell line for PFU analuysis |
| Magnesium Sulfate 7-Hydrate | Fisher | 60-019-68 | CDM starter stock |
| Manganese Sulfate | Fisher | M113-500 | Mix I stock |
| MilQ water | Ultra-pure water | ||
| Mouse Fc Block | BD Bioscience | BDB553142 | Clone: 2.4G2 DF 1:100 |
| MWI VETERINARY PURALUBE VET OINTMENT | Fisher | NC1886507 | Eye lubricant for infection |
| NCI-H292 mucoepidermoid carcinoma cell line | ATCC | CRL-1848 | H292 lung epithelial cell line for biofilm growth |
| Niacinamide | Fisher | 18-604-792 | Vitamin stock |
| NK 1.1 AF 700 | BD Bioscience | 50-112-4692 | Clone: PK136 DF 1:200 |
| Oxyrase For Broth 50Ml Bottle 1/Pk | Fisher | 50-200-5299 | To remove oxygen from liquid cultures |
| Paraformaldehyde 4% in PBS | Thermoscientific | J19932-K2 | |
| Pivetal Isoflurane | Patterson Veterinary | 07-893-8440 | Isoflurane for anesthesia during infection |
| Potassium Phosphate Dibasic | Fisher Chemical | P288-500 | Starter stock |
| Potassium Phosphate Monobasic | Fisher Chemical | P285-500 | Starter stock |
| Pyridoxal hydrochloride | Fisher | AC352710250 | Vitamin stock |
| Pyridoxamine dihydrochloride | Fisher | AAJ6267906 | Mix I stock |
| Riboflavin | Fisher | AC132350250 | Vitamin stock |
| Sodium Acetate | VWR | 0530-500G | Starter stock |
| Sodium Azide | Fisher Bioreagents | BP922I-500 | For FACS buffer |
| Sodium Bicarbonate | Fisher Chemical | S233-500 | Starter stock and final supplement to CDM |
| Sodium Phosphate Dibasic | Fisher Chemical | S374-500 | Starter stock |
| Sodium Phosphate Monobasic | Fisher Chemical | S369-500 | Starter stock |
| TCR APC | BD Bioscience | 50-112-8889 | Clone: GL-3 DF 1:200 |
| TCRβ APC-Cy7 | BD Pharmigen | BDB560656 | Clone: H57-597 DF 1:200 |
| Thermo Scientific Blood Agar with Gentamicin | Fisher | R01227 | Blood agar plates with the antibiotic gentamicin |
| Thermo Scientific Trypsin, TPCK Treated | Fisher | PI20233 | |
| Thiamine hydrochloride | Fisher | AC148991000 | Vitamin stock |
| TIGR4 | ATCC | BAA-334 | Streptococcus pneumoniae strain |
| Uracil | Fisher | AC157300250 | Mix II stock |
| Worthington Biochemical Corporation Collagenase, Type 2, 1 g | Fisher | NC9693955 |
Références
- Kadioglu, A., Weiser, J. N., Paton, J. C., Andrew, P. W. The role of Streptococcus pneumoniae virulence factors in host respiratory colonization and disease. Nature Reviews Microbiology. 6 (4), 288-301 (2008).
- Obaro, S., Adegbola, R. The pneumococcus: Carriage, disease and conjugate vaccines. Journal of Medical Microbiology. 51 (2), 98-104 (2002).
- Chong, C. P., Street, P. R. Pneumonia in the elderly: A review of the epidemiology, pathogenesis, microbiology, and clinical features. Southern Medical Journal. 101 (11), 1141-1145 (2008).
- Kadioglu, A., Andrew, P. W. Susceptibility and resistance to pneumococcal disease in mice. Briefings in Functional Genomics and Proteomics. 4 (3), 241-247 (2005).
- Ganie, F., et al. Structural, genetic, and serological elucidation of Streptococcus pneumoniae serogroup 24 serotypes: Discovery of a new serotype, 24C, with a variable capsule structure. Journal of Clinical Microbiology. 59 (7), 0054021 (2021).
- Centers for Disease Control and Prevention. Estimates of deaths associated with seasonal influenza --- United States. MMWR. Morbidity and Mortality Weekly Report. 59 (33), 1057-1062 (2010).
- Shrestha, S., et al. Identifying the interaction between influenza and pneumococcal pneumonia using incidence data. Science Translational Medicine. 5 (191), (2013).
- McCullers, J. A. Insights into the interaction between influenza virus and pneumococcus. Clinical Microbiology Reviews. 19 (3), 571-582 (2006).
- Pneumococcal Disease Global Pneumococcal Disease and Vaccine. Centers for Disease Control and Prevention Available from: https://www.cdc.gov/pneumococcal/global.html (2018)
- Grudzinska, F. S., et al. Neutrophils in community-acquired pneumonia: Parallels in dysfunction at the extremes of age. Thorax. 75 (2), 164-171 (2020).
- Boe, D. M., Boule, L. A., Kovacs, E. J. Innate immune responses in the ageing lung. Clinical and Experimental Immunology. 187 (1), 16-25 (2017).
- Krone, C. L., van de Groep, K., Trzcinski, K., Sanders, E. A., Bogaert, D. Immunosenescence and pneumococcal disease: An imbalance in host-pathogen interactions. The Lancet Respiratory Medicine. 2 (2), 141-153 (2014).
- Cho, S. J., et al. Decreased NLRP3 inflammasome expression in aged lung may contribute to increased susceptibility to secondary Streptococcus pneumoniae infection. Experimental Gerontology. 105, 40-46 (2018).
- Disease Burden of Influenza. Centers for Disease Control and Prevention Available from: https://www.cdc.gov/flu/about/burden/index.html (2018)
- McCullers, J. A. The co-pathogenesis of influenza viruses with bacteria in the lung. Nature Reviews Microbiology. 12 (4), 252-262 (2014).
- McCullers, J. A., Rehg, J. E. Lethal synergism between influenza virus and Streptococcus pneumoniae: Characterization of a mouse model and the role of platelet-activating factor receptor. The Journal of Infectious Diseases. 186 (3), 341-350 (2002).
- Metzger, D. W., Sun, K. Immune dysfunction and bacterial coinfections following influenza. Journal of Immunology. 191 (5), 2047-2052 (2013).
- Chao, Y., Marks, L. R., Pettigrew, M. M., Hakansson, A. P. Streptococcus pneumoniae biofilm formation and dispersion during colonization and disease. Frontiers in Cellular and Infection Microbiology. 4, 194 (2014).
- Bogaert, D., De Groot, R., Hermans, P. W. Streptococcus pneumoniae colonisation: The key to pneumococcal disease. The Lancet Infectious Diseases. 4 (3), 144-154 (2004).
- Simell, B., et al. The fundamental link between pneumococcal carriage and disease. Expert Review of Vaccines. 11 (7), 841-855 (2012).
- Marks, L. R., Davidson, B. A., Knight, P. R., Hakansson, A. P. Interkingdom signaling induces Streptococcus pneumoniae biofilm dispersion and transition from asymptomatic colonization to disease. mBio. 4 (4), 00438 (2013).
- Reddinger, R. M., Luke-Marshall, N. R., Sauberan, S. L., Hakansson, A. P., Campagnari, A. A. Streptococcus pneumoniae modulates Staphylococcus aureus biofilm dispersion and the transition from colonization to invasive disease. mBio. 9 (1), 02089 (2018).
- Joma, B. H., et al. A murine model for enhancement of Streptococcus pneumoniae pathogenicity upon viral infection and advanced age. Infection and Immunity. 89 (8), 0047120 (2021).
- Andersson, B., et al. Identification of an active disaccharide unit of a glycoconjugate receptor for pneumococci attaching to human pharyngeal epithelial cells. Journal of Experimental Medicine. 158 (2), 559-570 (1983).
- Avery, O. T., Macleod, C. M., McCarty, M. Studies on the chemical nature of the substance inducing transformation of pneumococcal types: Induction of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III. The Journal of Experimental Medicine. 79 (2), 137-158 (1944).
- Tettelin, H., et al. Complete genome sequence of a virulent isolate of Streptococcus pneumoniae. Science. 293 (5529), 498-506 (2001).
- Tothpal, A., Desobry, K., Joshi, S. S., Wyllie, A. L., Weinberger, D. M. Variation of growth characteristics of pneumococcus with environmental conditions. BMC Microbiology. 19 (1), 304 (2019).
- Bou Ghanem, E. N., et al. Extracellular adenosine protects against Streptococcus pneumoniae lung infection by regulating pulmonary neutrophil recruitment. PLoS Pathogens. 11 (8), 1005126 (2015).
- Bou Ghanem, E. N., et al. The alpha-tocopherol form of vitamin E boosts elastase activity of human PMNs and their ability to kill Streptococcus pneumoniae. Frontiers in Cellular and Infection Microbiology. 7, 161 (2017).
- Tait, A. R., Davidson, B. A., Johnson, K. J., Remick, D. G., Knight, P. R. Halothane inhibits the intraalveolar recruitment of neutrophils, lymphocytes, and macrophages in response to influenza virus infection in mice. Anesthesia & Analgesia. 76 (5), 1106-1113 (1993).
- Aaberge, I. S., Eng, J., Lermark, G., Lovik, M. Virulence of Streptococcus pneumoniae in mice: A standardized method for preparation and frozen storage of the experimental bacterial inoculum. Microbial Pathogenesis. 18 (2), 141-152 (1995).
- McCullers, J. A., Bartmess, K. C. Role of neuraminidase in lethal synergism between influenza virus and Streptococcus pneumoniae. The Journal of Infectious Diseases. 187 (6), 1000-1009 (2003).
- Smith, A. M., McCullers, J. A. Secondary bacterial infections in influenza virus infection pathogenesis. Current Topics in Microbiology and Immunology. 385, 327-356 (2014).
- Cundell, D. R., Gerard, N. P., Gerard, C., Idanpaan-Heikkila, I., Tuomanen, E. I. Streptococcus pneumoniae anchor to activated human cells by the receptor for platelet-activating factor. Nature. 377 (6548), 435-438 (1995).
- Ballinger, M. N., Standiford, T. J. Postinfluenza bacterial pneumonia: Host defenses gone awry. Journal of Interferon & Cytokine Research. 30 (9), 643-652 (2010).
- Sun, K., Metzger, D. W. Inhibition of pulmonary antibacterial defense by interferon-gamma during recovery from influenza infection. Nature Medicine. 14 (5), 558-564 (2008).
- Nakamura, S., Davis, K. M., Weiser, J. N. Synergistic stimulation of type I interferons during influenza virus coinfection promotes Streptococcus pneumoniae colonization in mice. Journal of Clinical Investigation. 121 (9), 3657-3665 (2011).
- Blanchette-Cain, K., et al. Streptococcus pneumoniae biofilm formation is strain dependent, multifactorial, and associated with reduced invasiveness and immunoreactivity during colonization. mBio. 4 (5), 00745 (2013).
- Rello, J., Pop-Vicas, A. Clinical review: Primary influenza viral pneumonia. Critical Care. 13 (6), 235 (2009).
- Torres, A., Loeches, I. M., Sligl, W., Lee, N. Severe flu management: A point of view. Intensive Care Medicine. 46 (2), 153-162 (2020).
- Bakaletz, L. O. Viral-bacterial co-infections in the respiratory tract. Current Opinion in Microbiology. 35, 30-35 (2017).
- Palacios, G., et al. Streptococcus pneumoniae coinfection is correlated with the severity of H1N1 pandemic influenza. PLoS One. 4 (12), 8540 (2009).
- Dhanoa, A., Fang, N. C., Hassan, S. S., Kaniappan, P., Rajasekaram, G. Epidemiology and clinical characteristics of hospitalized patients with pandemic influenza A (H1N1) 2009 infections: The effects of bacterial coinfection. Virology Journal. 8, 501 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon