Method Article
Um modelo de camundongo para a transição de Streptococcus pneumoniae de colonizador para patógeno após coinfecção viral recapitula a doença exacerbada pela idade
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este artigo descreve um novo modelo de camundongo para a transição de pneumococo de um colonizador assintomático para um patógeno causador de doença durante a infecção viral. Este modelo pode ser facilmente adaptado para estudar interações polimicrobianas e patógeno-hospedeiro durante as diferentes fases de progressão da doença e em vários hospedeiros.
Resumo
O Streptococcus pneumoniae (pneumococo) é um colonizador assintomático da nasofaringe na maioria dos indivíduos, mas pode evoluir para patógeno pulmonar e sistêmico após a infecção pelo vírus influenza A (IAV). A idade avançada aumenta a suscetibilidade do hospedeiro à pneumonia pneumocócica secundária e está associada a desfechos agravados da doença. Os fatores do hospedeiro que conduzem esses processos não estão bem definidos, em parte devido à falta de modelos animais que reproduzam a transição da colonização assintomática para a doença clínica grave.
Este artigo descreve um novo modelo de camundongo que recria a transição de pneumococos de carreador assintomático para doença após infecção viral. Nesse modelo, camundongos são primeiro inoculados intranasalmente com pneumococos cultivados com biofilme para estabelecer carreamento assintomático, seguido por infecção por IAV tanto na nasofaringe quanto nos pulmões. Isso resulta em disseminação bacteriana para os pulmões, inflamação pulmonar e sinais óbvios de doença que podem evoluir para letalidade. O grau da doença é dependente da cepa bacteriana e dos fatores do hospedeiro.
É importante ressaltar que esse modelo reproduz a suscetibilidade do envelhecimento, pois em comparação com camundongos jovens, os camundongos velhos apresentam doença clínica mais grave e sucumbem à doença com mais frequência. Ao separar carreador e doença em etapas distintas e fornecer a oportunidade de analisar as variantes genéticas do patógeno e do hospedeiro, este modelo de co-infecção S. pneumoniae/IAV permite o exame detalhado das interações de um importante patobionte com o hospedeiro em diferentes fases da progressão da doença. Esse modelo também pode servir como uma importante ferramenta para identificar potenciais alvos terapêuticos contra pneumonia pneumocócica secundária em hospedeiros suscetíveis.
Introdução
Streptococcus pneumoniae (pneumococo) são bactérias Gram-positivas que residem assintomático na nasofaringe da maioria dos indivíduos saudáveis 1,2. Promovido por fatores não completamente definidos, o pneumococo pode transitar de colonizadores benignos da nasofaringe para patógenos que se disseminam para outros órgãos, resultando em infecções graves, incluindo otite média, pneumonia e bacteremia3. A apresentação da doença pneumocócica é, em parte, dependente de diferenças específicas da cepa, incluindo o sorotipo, que é baseado na composição dos polissacarídeos capsulares. Existem mais de 100 sorotipos caracterizados até o momento, e alguns estão associados a infecções mais invasivas 4,5. Vários outros fatores aumentam o risco de doença pneumocócica. Um desses fatores é a infecção viral, em que o risco de pneumonia pneumocócica é aumentado em 100 vezes pelo IAV 6,7. Historicamente, o S. pneumoniae é uma das causas mais comuns de pneumonia bacteriana secundária após influenza e está associado a piores desfechos8. Outro importante fator de risco é a idade avançada. De fato, o S. pneumoniae é a principal causa de pneumonia bacteriana adquirida na comunidade em idosos acima de 65 anos 9,10. Os idosos são responsáveis pela maioria (>75%) dos óbitos por pneumonia e influenza, indicando que os dois fatores de risco - envelhecimento e infecção por VAI - pioram sinergicamente a suscetibilidade à doença11,12,13,14. No entanto, os mecanismos pelos quais a infecção viral provoca a transição do pneumococo de colonizador assintomático para patógeno invasivo e como este é moldado por fatores do hospedeiro permanecem pouco definidos. Isso se deve em grande parte à ausência de um modelo animal de pequeno porte que recapitule a transição da colonização pneumocócica assintomática para doença clínica crítica.
Estudos de co-infecção têm sido classicamente modelados em camundongos inoculados com pneumococos diretamente nos pulmões 7 dias após a infecção por influenza15,16. Isso reproduz a suscetibilidade à pneumonia bacteriana secundária e é ideal para estudar como as respostas imunes antivirais prejudicam as defesas antibacterianas17. Entretanto, estudos longitudinais em humanos têm demonstrado que o carreamento pneumocócico na nasofaringe, onde a bactéria pode formar biofilmesassintomáticos18, está uniformemente associado a doenças invasivas19,20. Isolados bacterianos de infecções de orelha média, pulmão e sangue são geneticamente idênticos aos encontrados na nasofaringe20. Assim, para estudar a transição de carreamento assintomático para doença invasiva após infecção por VA, estabeleceu-se um modelo em que camundongos receberam pneumococo cultivado com biofilme por via intranasal seguido de infecção por IAV na nasofaringe21,22. A infecção viral das vias aéreas superiores levou a alterações no ambiente do hospedeiro que levaram à dispersão dos pneumococos dos biofilmes e sua disseminação para as vias aéreas inferiores21. Essas bactérias dispersas apresentaram expressão regulada de fatores de virulência importantes para a infecção, convertendo-as de colonizadoras em patógenos21. Essas observações destacam a complexa interação entre vírus, hospedeiro e bactéria e demonstram que as alterações no hospedeiro desencadeadas pela infecção viral têm impacto direto no comportamento pneumocócico, que, por sua vez, altera o curso da infecção bacteriana. No entanto, esse modelo falha em recapitular os sinais graves de doença observados em humanos, provavelmente porque o vírus é limitado à cavidade nasal, e os efeitos sistêmicos da infecção viral na imunidade do hospedeiro e danos pulmonares não são recapitulados.
Recentemente, estabelecemos um modelo que incorpora a complexa interação entre o hospedeiro e os patógenos, mas também mimetiza mais de perto a gravidade da doença observada em humanos23. Neste modelo, camundongos são primeiramente infectados intranasalmente com pneumococos cultivados com biofilme para estabelecer carreamento assintomático, seguido por infecção por IAV tanto na nasofaringe quanto nos pulmões. Isso resultou em disseminação bacteriana para os pulmões, inflamação pulmonar e doença que progrediu para letalidade em uma fração de camundongos jovens23. Este estudo prévio demonstrou que tanto a infecção viral quanto a bacteriana alteraram a defesa do hospedeiro: a infecção viral promoveu disseminação bacteriana e a colonização bacteriana prévia prejudicou a capacidade do hospedeiro de controlar os níveis pulmonares de VAI23. O exame da resposta imune revelou que a infecção por IAV diminuiu a atividade antibacteriana dos neutrófilos, enquanto a colonização bacteriana atenuou a resposta do interferon tipo I crítica para a defesa antiviral23. É importante ressaltar que esse modelo reproduzia a suscetibilidade do envelhecimento. Comparados a camundongos jovens, os camundongos velhos apresentaram sinais de doença mais precocemente, apresentaram doença clínica mais grave e sucumbiram à infecção com maior frequência23. O trabalho apresentado neste manuscrito mostra que o grau da doença também é dependente da cepa bacteriana, pois cepas invasivas de pneumococo apresentam disseminação mais eficiente na infecção por IAV, mostram sinais mais evidentes de inflamação pulmonar e resultam em taxas aceleradas de doença em comparação com cepas não invasivas. Assim, este modelo de co-infecção S. pneumoniae/IAV permite o exame detalhado de fatores patogênicos e do hospedeiro e é adequado para estudar respostas imunes a infecções polimicrobianas nas diferentes fases da progressão da doença.
Protocolo
Todos os estudos em animais foram realizados de acordo com as recomendações do Guide for the Care and Use of Laboratory Animals. Todos os procedimentos foram aprovados pelo Comitê Institucional de Cuidados e Uso de Animais da Universidade de Búfalo.
1. Preparação de meios quimicamente definidos (MDL)
- Prepare os estoques da seguinte forma:
- Dissolver os compostos da mistura I listados na Tabela 1 em 100 mL de água ultrapura agitando. Conservar em alíquotas de 200 μL a −20 °C.
- Dissolver os compostos da mistura II listados na Tabela 1 em 20 mL de NaOH 0,1 M agitando. Conservar em alíquotas de 100 μL a −20 °C.
- Dissolver os compostos da mistura III listados na Tabela 1 em 1 mL de água ultrapura agitando. Conservar em alíquotas de 10 μL a 4 °C.
- Dissolver o composto da mistura IV indicado na Tabela 1 em 1 mL de água ultrapura agitando. Conservar em alíquotas de 10 μL a −20 °C.
- Dissolver os compostos listados na Tabela 2 inicialmente em 15 mL de água ultrapura agitando. Ajustar o pH para 7,0 com algumas gotas de NaOH 0,1 M e ajustar o volume final para 20 mL usando água ultrapura. Conservar em alíquotas de 1 ml a −20 °C.
- Dissolver os compostos indicados no quadro 3 em 90 ml de água ultrapura numa placa quente a 50 °C enquanto agita. Ajustar o pH para 7,0 com NaOH 0,1 M e, em seguida, ajustar o volume final para 100 mL usando água ultrapura. Conservar em alíquotas de 5 ml a -20 °C.
- Faça o estoque de partida sempre dissolvendo os compostos da Tabela 4 em 70 mL de água ultrapura enquanto agita.
- À calção inicial fresca, adicionar por ordem as seguintes misturas: 200 μL de caldo Mix I (Quadro 1), 80 μL de stock Mix II (Quadro 1), 10 μL de stock Mix III (Quadro 1), 10 μL de stock Mix IV (Quadro 2), 1 ml de stock de vitaminas (Tabela 3) e 5 mL de stock de aminoácidos (Tabela 4).
- Uma vez adicionados os estoques, ajustar o volume final para 100 mL adicionando 30 mL de água ultrapura ao copo.
- Completar o MDL com compostos da Tabela 4. Uma vez misturado completamente, filtrar-esterilizar e armazenar a 4 °C por um máximo de 2 semanas.
2. Cultivo do biofilme de S. pneumoniae
- Preparar o meio RP-10 misturando 445 mL de RPMI 1640 com 50 mL de soro fetal bovino (SFB) inativado pelo calor e 5 mL de penicilina/estreptomicina a 10.000 U/mL e 10.000 μg/mL, respectivamente.
- Cultivar a linhagem celular do carcinoma mucoepidermóide NCI-H292 (H292). Adicionar as células de um frasco para injetáveis comprado a 5 ml de meio RP-10 num balão T-25 tratado com cultura de tecidos. Incubar a 37 °C/5% CO2 por 3-5 dias até atingir 100% de confluência.
- Verificar as células ao microscópio de luz com aumento de 10x para avaliar a confluência.
NOTA: Quando todas as células estão em contato com outras células e não há lacunas entre elas, então a confluência desejada de 100% é alcançada. - Lavar as células 2x em 5 mL de PBS à temperatura ambiente. Certifique-se de que o tampão está isento de cálcio para evitar a quelação do EDTA na etapa seguinte.
- Adicionar 1 ml de tripsina-EDTA ao balão e incubar a 37 °C/5% CO2 durante 5-10 min até que as células se desprendam. Neutralizar com 4 mL de meio RP-10. Misture suavemente pipetando para cima e para baixo e transfira para um tubo cônico de 50 mL.
- Adicionar 500 μL da suspensão celular por poço a uma placa de 24 poços tratada com cultura de tecidos. De um frasco T-25 confluente, esperar 2 × 10 6-4 × 106 células/mL.
- No dia seguinte, verifique as células sob um microscópio de luz para certificar-se de que estão confluentes, como no passo 2.3. Se não estiverem, incube por mais tempo.
- Uma vez que as células H292 estejam 100% confluentes na placa de 24 poços, lave suavemente as células 3x com 1 mL de PBS à temperatura ambiente para garantir que nenhum meio contendo antibióticos ou detritos permaneça.
- Após lavar as células, adicionar 250 μL/poço de paraformaldeído a 4% para fixar as células. Incubar durante 1 h no gelo ou durante a noite a 4 °C.
- Na noite anterior à fixação celular, estriar a cepa de interesse de S. pneumoniae em placas de ágar sangue e incubar durante a noite a 37 °C/5% CO2.
NOTA: Os dados aqui apresentados são com as seguintes cepas de S. pneumoniae obtidas via troca colaborativa: sorotipo 19F otite média isolado EF3030 24, sorotipo clássico 2 Avery cepa D3925 e sorotipo 4 bacteremia isolado TIGR426. As cepas também estão disponíveis em coleções públicas referenciadas na Tabela de Materiais. - Preparar MDL mais oxirase (0,15 U/mL) adicionando 100 μL de oxirase (30 U/mL) a 20 mL de MDL.
NOTA: A oxirase é utilizada para eliminar oxigênio e permitir o crescimento eficiente de S. pneumoniae em cultura líquida27. - Inocular as bactérias da placa em MDL + oxirase fresca, lavando as bactérias da placa adicionando 1 mL de MDL + oxirase e levantando suavemente as colônias bacterianas usando o lado de uma ponta de pipeta de 1 mL, tomando cuidado para não raspar o ágar. Alternativamente, use uma alça de inoculação para levantar as bactérias e inocular-as em um tubo contendo 1 mL de MDL + oxirase.
- Diluir as bactérias em MDL + oxirase para um ODinicial de 600 de 0,05.
- Cultivar as bactérias em um tubo cônico de 50 mL frouxamente tampado a 37 °C/5% CO 2 até que um OD600 de 0,2 seja atingido (isso levará entre2-5 h). Verifique o OD600 a cada hora para garantir que o OD não exceda 0,2.
- Uma vez que a DO tenha atingido 0,2, vórtex o tubo de cultura bacteriana. Semeando 0,5 mL da bactéria nas células H292 fixas e adicionando mais 0,5 mL de meio MDL + oxirase por poço. Adicionar 1 mL de MDL + oxirase aos poços controle sem bactérias. Incubar a placa durante 48 h a 34 °C/5% CO2.
NOTA: O crescimento a 34 °C é usado para mimetizar mais de perto a temperatura mais baixa na nasofaringe21. - A cada 12 h após a semeadura inicial, remova suavemente 0,5 mL do meio e reabasteça com 0,5 mL de MDL fresco + oxirase. Tenha cuidado para não interromper o biofilme em formação. Verifique o fundo da placa para biofilme e procure aumentar a nebulosidade com o passar do tempo devido ao crescimento do biofilme. Para controlar a contaminação, verifique os poços sem bactérias para garantir que os poços de controle permaneçam limpos.
- Às 48 h após a inoculação, retire o sobrenadante e lave 2x muito suavemente com 1 mL de PBS. Ressuspender em 1 mL de MDL fresco e pipetar para cima e para baixo vigorosamente para levantar o biofilme. Para cada cepa bacteriana, agrupe as bactérias de todos os poços em um tubo cônico de 50 mL. Misture bem inclinando suavemente o tubo bem tampado para cima e para baixo várias vezes.
- Ao tubo cônico de 50 mL, adicionar 40% de glicerol em CDM em volumes iguais para obter uma suspensão bacteriana com concentração final de 20% de glicerol. Aliquota 1 mL em tubos de microcentrífuga, congele rapidamente em gelo seco e economize a -80 °C.
- Antes do uso, enumerar as bactérias descongelando uma alíquota no gelo, girando o tubo a 1.700 × g por 5 min, removendo o sobrenadante, ressuspendendo o pellet em 1 mL de PBS e plaqueando diluições seriadas em placas de ágar sangue28.
- Cultivar as placas de ágar durante a noite a 37 °C/5% CO2 e contar as colónias em diluições relevantes para obter a concentração bacteriana em unidades formadoras de colónias (UFC)/ml.
NOTA: Recomenda-se enumerar as bactérias nos estoques pelo menos um dia após o congelamento ou mais tarde, pois há uma queda na viabilidade bacteriana dentro das primeiras 24 h. As alíquotas congeladas armazenadas podem ser usadas para infecção subsequente de camundongos por no máximo 2 meses.
3. Inoculação intranasal de camundongos com S. pneumoniae cultivada em biofilme
- Compre ratos e use na idade desejada.
NOTA: Camundongos com idade entre 3-4 meses de idade são preferidos para modelar hospedeiros jovens, e camundongos com idade entre 21-24 meses podem ser usados para modelar indivíduos idosos >65 anos de idade29. Os dados aqui apresentados são com camundongos machos C57BL/6. - Descongele as alíquotas bacterianas cultivadas em biofilme no gelo e gire a 1.700 × g por 5 min. Retire e descarte cuidadosamente o sobrenadante sem interromper o pellet, lave as bactérias ressuspendendo o pellet em 1 mL de PBS e gire novamente a 1.700 × g por 5 min. Retirar o sobrenadante e ressuspender o pellet no volume necessário para atingir a concentração desejada (almejar 5 ×10 6 UFC/10 μL para inoculação intranasal). Confirmar as quantidades de bactérias administradas plaqueando o inóculo preparado em placas de ágar sangue como na etapa 2.19.
- Inocular os camundongos por via intranasal com 5 × 106 UFC pipetando 5 μL do inóculo diluído em cada naris. Certifique-se de segurar os ratos firmemente, estabilizando a cabeça, até que o volume seja inalado (normalmente dentro de segundos após pipetar o volume nas narinas). Realizar esta etapa na ausência de anestesia para evitar aspiração pulmonar do inóculo.
4. Infecção viral pelo vírus influenza A (IAV)
- Às 48 h após a inoculação intranasal com S. pneumoniae, descongelar a cepa IAV de interesse no gelo.
NOTA: Os dados aqui apresentados são com uma cepa adaptada de camundongo do vírus influenza A A/PR/8/34 H1N1 que foi obtida via troca colaborativa30. - Uma vez descongelado o vírus, diluir o vírus em PBS até a concentração desejada; visam 20 unidades formadoras de placa (UFP)/50 μL para infecção intratraqueal e 200 UFP/10 μL para infecção intranasal. Para grupos infectados e somente com bactérias, use PBS para inocular os camundongos.
- Coloque lubrificante oftálmico nos olhos dos ratos antes da anestesia. Anestesiar os camundongos com isoflurano a 5% e confirmar a anestesia com pinça firme dos dedos.
- Uma vez anestesiado o animal, retire-o da câmara de isoflurano e infecte imediatamente os camundongos anestesiados com 50 μL (20 UFP) de VAI intratraquealmente usando pinça romba para puxar a língua para fora da boca e pipetando o volume de líquido pela traqueia.
- Coloque os ratos em uma gaiola separada e monitore até a recuperação completa (eles são capazes de manter a decúbito esternal [capaz de deitar ereto no peito]).
- Após a recuperação, inocular imediatamente os camundongos intranasalmente com 10 μL (200 UFP) de IAV usando o método de inoculação na etapa 3.3.
- Camundongos domésticos que sofreram infecção bacteriana e viral única ou dupla com o mesmo grupo de infecção e os separam dos outros grupos.
5. Monitorando os camundongos quanto aos sintomas da doença
- Monitore os camundongos diariamente por pelo menos 10 dias e pontue cegamente para sinais de doença da seguinte maneira:
- Escore igual ou inferior para perda de peso: 0 = 5% ou menos; 1 = 5%-10%; 2 = 10%-15%; 3 = 20% ou mais. Eutanasiar os camundongos usando inalação de CO2 quando o escore de perda de peso estiver em 3.
- Escore da seguinte forma para atividade: 0 = normal/ativo; 1 = movimento, mas ligeiramente diminuído; 2 = diminuído; 3 = diminuído gravemente/letárgico (só se move se tocado), 4 = coma/imóvel. Eutanasiar os ratos quando a pontuação de atividade estiver em 3.
- Escore da seguinte forma para postura: 0 = sem palpite (normal); 1 = postura levemente curvada; 2 = palpite severo. Eutanasiar os camundongos quando o escore postural estiver em 2.
- Escore igual para os olhos: 0 = normal; 1 = protrusão; 1 = afundado; 1 = fechado; 1 = alta. Pode ser uma combinação. Adicione os totais para a pontuação final do olho.
- Escore da seguinte forma para respiração: 0 = Respiração normal; 1 = irregular ou alterado (maior/menor índice); 2 = laborioso (esforço exagerado ou ofegante). Eutanasiar os ratos quando o escore respiratório estiver em 2.
- Com base nos critérios acima, soma-se os escores individuais para um escore clínico total de saudável (0) a extremamente doente (15). Considere que qualquer mouse que exibe uma pontuação total acima de 2 está doente. Eutanasiar humanamente todos os camundongos que apresentarem um escore total acima de 9 ou os escores indicados para cada critério e marcá-los na curva de sobrevivência.
6. Processamento de tecidos infectados para enumeração bacteriana
- Às 48 h após a infecção por IAV, eutanasiar os camundongos.
- Coloque o rato em decúbito dorsal. Usando etanol 70%, borrife o peito e o abdômen do camundongo para limpar o pelo. Usando pinças, aperte o pelo e a pele no meio do rato e corte o pelo com 4,5 em tesoura de dissecção para expor a área do fígado até o peito.
- Coleta de sangue
- Usando tesoura de dissecção, corte suavemente na cavidade peritoneal para expor o fígado. Usando pinças, expor a veia porta hepática na parte superior do fígado, perto do diafragma. Cortar a veia porta hepática com a tesoura de dissecção. Uma vez que o sangue começa a se acumular na cavidade peritoneal, colete 10 μL de sangue usando uma micropipeta e coloque em 90 μL de solução anticoagulante (solução de EDTA 50 mM em PBS) em um tubo de microcentrífuga para plaqueamento para carga bacteriana.
- Use uma micropipeta P-1000 para coletar o restante do sangue, coloque-o em um tubo de coleta de sangue e centrifugue a 7.600 × g por 2 min para coletar o soro. Guarde os soros em tubos de microcentrífuga a -80 °C para análise subsequente de qualquer citocina ou metabolito desejado.
- Coleta pulmonar
- Usando tesoura de dissecção, faça um corte nas laterais da caixa torácica exposta e puxe suavemente as costelas para cima em direção à cabeça do mouse para expor o coração. Insira uma agulha de 25 G acoplada a uma seringa de 10 mL pré-cheia com PBS no ventrículo direito e comece a perfundir lentamente. Procure clareamento dos pulmões como indicador de perfusão bem-sucedida. Lave lentamente para evitar a quebra do tecido pulmonar.
- Levante o coração com a pinça e faça um corte para separar os pulmões e o coração. Uma vez separado, pegue todos os lóbulos do pulmão com a pinça e enxágue em um prato com PBS estéril para remover qualquer sangue residual. Em uma placa de Petri, pique os pulmões em pequenos pedaços e misture bem. Remova metade da mistura pulmonar para determinação da UFC bacteriana ou da UFP viral e coloque-a em um tubo de fundo redondo de 15 mL pré-preenchido com 0,5 mL de PBS para homogeneização.
NOTA: É importante não tomar lobos diferentes do mesmo pulmão para as várias avaliações. Em vez disso, todos os lóbulos devem ser picados, bem misturados e analisados igualmente para as diferentes avaliações. - Remova a outra metade do pulmão para citometria de fluxo (seção 7 abaixo) e coloque-a em uma placa de 24 poços não tratada com cultura de tecidos, com cada poço pré-preenchido com 0,5 mL de RP-10. Deixar à temperatura ambiente até ao processamento.
- Coleção Nasofaringe
- No pescoço, use a tesoura de dissecção para cortar o pelo e, em seguida, cortar o músculo e expor a traqueia.
NOTA: A traqueia é uma estrutura semelhante a um tubo localizada sob o músculo. - Coloque pequenas pinças sob a traqueia a uma distância de 1 cm da mandíbula do rato para estabilizá-la. Com tesoura de dissecção, faça suavemente uma fenda de 0,1 cm na porção anterior da traqueia, evitando cortar completamente a traqueia.
- Prepare uma seringa de 1 mL preenchida com 0,5 mL de PBS com tubo de 0,58 mm acoplado a uma agulha de 25 G. Coletar a lavagem nasal inserindo a tubulação na traqueia indo para cima em direção à nasofaringe. Uma vez que a resistência é sentida entrando na cavidade nasal, coloque um tubo de microcentrífuga no nariz e lave lentamente o PBS através da traqueia para coletar a lavagem nasal.
- Coloque o mouse em decúbito ventral. Borrife a cabeça do rato com etanol. Use tesoura de dissecção para cortar o pelo e almofada mistáquica para expor o osso da cabeça do rato.
- Usando a tesoura de dissecção, faça um corte de 1 cm nas laterais da mandíbula e entre os olhos. Usando pinças, puxe lentamente os ossos faciais para longe do corpo para expor a cavidade nasal.
- Use pinça para remover suavemente o tecido nasal e coloque-o em um tubo de fundo redondo pré-preenchido com 0,5 mL de PBS para homogeneização.
- No pescoço, use a tesoura de dissecção para cortar o pelo e, em seguida, cortar o músculo e expor a traqueia.
- Para homogeneizar o tecido coletado, primeiro limpe a sonda homogeneizadora, colocando-a em etanol 70% e ligando o homogeneizador a 60% de potência por 30 s. Repita o passo em água estéril por 10 s. Homogeneizar cada tecido por 1 min. Limpar a sonda homogeneizadora em água estéril entre cada amostra e em um tubo fresco de etanol 70% entre cada órgão e grupo amostral.
- Enumeração dos números bacterianos
- Uma vez que todos os órgãos tenham sido colhidos e homogeneizados, diluições seriadas em placas de ágar sangue. Para calcular a UFC total, use 10 μL para placa e anote o volume final em mL para cada amostra. Plaquear as amostras de nasofaringe em placas de ágar sangue suplementadas com 3 μg/mL de gentamicina para selecionar o crescimento de S. pneumoniae enquanto inibe o crescimento de outros microrganismos que colonizam esse tecido. Incubar durante a noite a 37 °C/5% CO2.
- Para enumerar as UFC bacterianas para o pulmão e nasofaringe, primeiro conte as colônias nas placas de ágar sangue. Em seguida, use a equação (1) e a equação (2) para calcular a quantidade por mL e o número total.
Quantidade por ml = número de colónias × factor de diluição × 100 (1)
Número total = quantidade por mL × volume total por amostra (2)
NOTA: Na equação (1), 100 é usado para multiplicar, uma vez que 10 μL é plaqueado, o que é uma diluição de 100 vezes de 1 mL. O volume total por amostra na equação (2) é da etapa 6.7.1, que resulta no limite de detecção de 100 por órgão. - Para enumerar a UFC bacteriana para bacteremia, primeiro conte as colônias nas placas de ágar sangue. Em seguida, use a equação (3) para determinar a quantidade por mL de sangue.
Quantidade por mL de sangue = número de colônias × fator de diluição × 100 × 10 (3)
NOTA: Na equação (3), 100 é usado como 10 μL é plaqueado, que é uma diluição de 100 vezes de 1 mL, e 10 indica uma diluição de 1:10 do sangue em anticoagulante. Isso resulta no limite de detecção de 1.000/mL.
7. Processamento das amostras de pulmão para citometria de fluxo
- Prepare a mídia necessária da seguinte maneira:
- Prepare o RP-10 conforme descrito na etapa 2.1.
- Preparar o tampão de digestão misturando RP-10 com 2 mg/mL de colagenase e 30 μL/mL de DNase I.
- Preparar tampão de lise dissolvendo 8,29 g de NH4Cl, 1 g de NaHCO3 e 0,038 g de EDTA em 1 L de H2O.
- Preparar 10x tampão FACS misturando 450 mL de HBSS com 50 mL de FBS inativado pelo calor e 5 g de azida sódica.
- Preparar 1x tampão FACS diluindo 50 mL de tampão 10x FACS em 450 mL de HBSS.
- Colher as amostras de pulmão do passo 6.4.3 e colocar numa placa de 24 poços. Adicionar 500 μL de tampão de digestão a cada poço. Incubar durante 45 min até 1 h a 37 °C/5% CO2.
- Pré-encher tubos cônicos de 50 mL para cada amostra com 5 mL de RP-10. Quando a incubação terminar, colocar um filtro de 100 μm na parte superior do tubo cônico de 50 mL e molhá-lo com 1 mL de RP-10.
- Usando uma micropipeta P-1000, mova os pulmões digeridos e coloque-os no filtro. Use o êmbolo de uma seringa de 3 mL para amassar o órgão. Enxaguar 2x com 1 mL de RP-10 de cada vez.
- Girar as amostras a 4 °C e 327 × g durante 5 min. Aspirar o sobrenadante e ressuspender o pellet em 1 mL de tampão de lise. Deixe por 3 minutos para permitir a lise dos glóbulos vermelhos. Neutralizar com 5 mL de RP-10.
- Girar as amostras a 4 °C e 327 × g durante 5 min. Aspirar o sobrenadante, ressuspender o pellet em 1 mL de RP-10 e colher 10 μL para contagem das amostras.
- Girar as amostras a 4 °C e 327 × g durante 5 min. Aspirar o sobrenadante e ressuspender o pellet em RP-10 a 2 × 10 6-4 × 106 células/mL. Adicionar 60 μL de cada amostra em uma placa de 96 poços para corar para os tipos celulares desejados23 listados na etapa 7.9, Tabela 5 e Tabela 6.
- Gire a placa a 4 °C e 327 × g por 5 min.
- Enquanto isso, prepare as misturas mestras de anticorpos, florescente menos um (FMOs) e controles de coloração única com os anticorpos desejados. Para coloração para leucócitos polimorfonucleares (PMNs), macrófagos, monócitos, células dendríticas e células T, utilizar os anticorpos e diluições finais listados na Tabela 5 e Tabela 6. Use um volume total de 100 μL/poço da mistura de anticorpos. Siga as diluições indicadas nos quadros para determinar o volume adequado da mistura principal e os anticorpos individuais necessários.
- Quando o spin estiver feito (passo 7.8), decantar o sobrenadante, ressuspender os pellets em 100 μL das misturas de anticorpos, FMOs ou controles de coloração única e incubar no gelo por 30 min no escuro.
- Lavar as células 2x adicionando 150 μL de tampão FACS aos poços e girando a placa a 4 °C e 327 × g durante 5 min.
- Quando o spin estiver feito, decantar o sobrenadante, ressuspender os pellets em 100 μL de tampão de fixação e incubar no gelo por 20 min.
- Lavar as células 2x adicionando 150 μL de tampão FACS aos poços e girando a placa a 4 °C e 327 × g durante 5 min.
- Prepare tubos FACS marcados com 200 μL de tampão FACS. Ressuspender os pellets em 150 μL de tampão FACS. Filtrar individualmente cada amostra no tubo FACS correspondente utilizando um filtro de 100 μm. Manter no gelo ou a 4 °C e protegido da luz até estar pronto para analisar.
- Analise as células usando um citômetro de fluxo.
8. Ensaio de placa para enumeração do IAV
- Prepare a mídia necessária da seguinte maneira:
- Preparar o meio de infecção dissolvendo 2,5 g de albumina de soro bovino (BSA) em 40 mL de DMEM agitando a 37 °C por 10-20 min até dissolver. Filtrar-esterilizar em 460 mL de DMEM.
- Preparar 2,4% de celulose microcristalina dissolvendo 1,2 mg de celulose microcristalina em 50 mL de H2O. Autoclave no ajuste líquido e armazenar à temperatura ambiente.
- Preparar 5% de BSA DMEM dissolvendo 2,5 g de BSA em 40 mL de DMEM enquanto agita a 37°C por 10-20 min. Adicione os 10 mL restantes de DMEM para um volume final de 50 mL. Filtrar-esterilizar e armazenar a 4 °C.
- Preparar 2x MEM/0,5% BSA misturando 1 mL de 5% BSA DMEM com 9 mL de 2x MEM.
- Preparar meio de cobertura de baixa viscosidade misturando uma proporção de 1:1 de 2,4% de celulose microcristalina e 2x MEM/0,5% BSA com 1 mg/mL de TPCK (inibidor da quimotripsina) tripsina.
- Preparar EMEM/FBS a 10% misturando 450 mL de Eagle's Minimum Essential Medium (EMEM) com 50 mL de FBS inativado pelo calor.
- Cultivar a linhagem celular de rim canino Madin-Darby (MDCK). Adicionar as células de um frasco para injetáveis comprado a 5 ml de EMEM/FBS a 10% num balão T-25 tratado com cultura de tecidos. Incubar por 3-5 dias a 37 °C/5% CO2 até que as células atinjam 100% de confluência. Verifique se há confluência como na etapa 2.3.
- Retire e descarte o meio de cultura e enxágue 2x com 5 mL de PBS à temperatura ambiente. Adicionar 1 ml de tripsina-EDTA ao balão e incubar a 37 °C/ 5% CO2 durante 10-15 minutos até que as células se desprendam. Uma vez levantado, neutralizar com 4mL de EMEM/FBS a 10% para obter uma suspensão celular a 2 × 105 células/mL.
- Semeando as células MDCK em uma placa de cultura de tecido de 12 poços adicionando 1 mL de células ressuspensas por poço (a 2 × 10a 5 células/poço) 1 dia antes de iniciar o ensaio de placa.
NOTA: Certifique-se de que as células atinjam 100% de confluência antes do uso e incubem por mais tempo, se necessário, para atingir a confluência. - Para utilização como normas, efectuar diluições em série de 10 vezes (106-10 1) de existências IAV (de um título conhecido) no meio de infecção indicado no passo 8.1.1. Completar 1,2 ml de cada diluição para testar em triplicata.
- Descongelar o órgão homogeneiza no gelo. Gire para baixo em uma centrífuga de mesa a 2.000 × g e colete o sobrenadante claro.
- Repetir o passo 8.5, mas com o sobrenadante das amostras do passo 8.6.
- Aspirar o meio das células e lavar 2x com 1 mL de PBS para remover todo o SFB.
- Adicionar 300 μL de cada diluição padrão ou amostra diluída em série suavemente ao longo do lado de cada poço, começando pela diluição mais elevada até à mais baixa, e fazê-lo em triplicata.
- Colocar as placas na incubadora a 37 °C/5% CO2, agitando a placa a cada 10 min por um total de 50 min. Certifique-se de colocá-los na incubadora e não empilha-los.
- Após os 50 min, lavar as células 2x com 1 mL de PBS.
- Adicionar 2 mL do meio de cobertura de baixa viscosidade em cada poço, exceto nos poços de menor diluição e sem vírus; a estes, adicionar meio de infecção e tripsina.
- Coloque a placa de volta na incubadora a 37 °C/5% CO 2 por2-4 dias para obter placas que possam ser visualizadas a olho nu.
- Lave as placas adicionando 2 mL de PBS em cada poço rapidamente do lado e agite suavemente para suspender o meio de sobreposição de baixa viscosidade assentado.
- Descarte todo o volume de líquido no poço pipetando suavemente o meio.
- Repita a lavagem mais uma vez com 2 mL de PBS em cada poço e, em seguida, descarte todo o volume de líquido por pipetagem suave.
- Para fixar as placas, adicione 500 μL de paraformaldeído a 4% em cada poço, agite e deixe descansar por 30 min.
- Lave lentamente o lado com 1 mL de PBS; Em seguida, descarte suavemente o líquido.
- Adicionar 500 μL de violeta de cristal a 1% (diluído em água) a cada poço para cobrir a monocamada celular. Incubar por 5 min.
- Lavar com 1 mL de água da torneira. Certifique-se de descartar todo o líquido no poço por pipetagem suave. Coloque o prato de cabeça para baixo em uma almofada de fraldas para secar durante a noite.
- Conte as placas visualmente e salve as imagens em qualquer imageador disponível.
Resultados
S. pneumoniae cultivada com biofilme (Figura 1A) foi usada para infectar camundongos (Figura 1B) usando um pequeno inóculo de 10 μL administrado intranasalmente a camundongos não anestesiados. Esse inóculo de pequeno volume resulta em carreamento pneumocócico consistente restrito à nasofaringe (Figura 2A, grupos +sp), evitando disseminação sistêmica (grupos Figura 2B,C, +sp). Dois dias após a inoculação intranasal, os camundongos foram infectados com um vírus influenza A A/PR/8/34 (IAV) adaptado à múrbia22,30, administrado tanto por via intranasal quanto intratraqueal para obter uma entrega consistente de quantidades específicas para a nasofaringe e os pulmões 23.
Aqui, o modelo foi usado para comparar o curso da doença após infecção viral em camundongos desafiados intranasalmente com diferentes cepas de S. pneumoniae, incluindo TIGR4 e D39, que são cepas invasivas que resultam em pneumonia que evolui para bacteremia, e EF3030, que é uma cepa de otite média 21,24,25,26,31. A apresentação da doença em camundongos co-infectados com S. pneumoniae/IAV foi dependente da cepa bacteriana (Figura 2). Embora não tenha havido diferença significativa no número bacteriano da nasofaringe (Figura 2A) entre nenhuma das cepas, S. pneumoniae TIGR4 e D39, mas não EF3030, disseminaram-se para os pulmões por 48 h após a infecção pelo IAV (Figura 2B). Quarenta por cento dos camundongos infectados intranasalmente com S. pneumoniae TIGR4 apresentaram disseminação bacteriana para os pulmões, e destes, metade deles tornou-se bacterêmica (Figura 2C), consistente com achados anteriores23.
Camundongos infectados intranasalmente com S. pneumoniae D39 mostraram disseminação mais eficiente, pois a disseminação para os pulmões foi observada em 100% dos camundongos co-infectados (Figura 2B). Semelhante ao S. pneumoniae TIGR4, metade deles apresentou bacteremia (Figura 2C). No rastreamento da sobrevida global, independentemente da cepa bacteriana, a taxa de sobrevivência dos camundongos co-infectados foi significativamente menor do que os camundongos desafiados isoladamente com S. pneumoniae isoladamente para todas as cepas testadas (Figura 2D). Em comparação com os camundongos controle desafiados apenas com IAV, os camundongos intranasalmente infectados com S. pneumoniae TIGR4 e D39, mas não EF3030, exibiram taxas aceleradas de doença. No dia 2 pós-infecção com IAV, 30% (D39) e 20% (TIGR4) dos camundongos haviam sucumbido, enquanto os grupos controle somente com IAV não começaram a sucumbir até o dia 5 pós-desafio (Figura 2D). Os camundongos co-infectados com S. pneumoniae EF3030 e IAV apresentaram sintomas tardios, mais semelhantes aos controles somente com IAV (Figura 2D). Esses achados demonstram que o modelo de co-infecção resulta em doença em camundongos jovens saudáveis que é dependente de cepa bacteriana, o que o torna ideal para explorar os fatores bacterianos necessários em cada etapa da progressão da doença.
Esse modelo foi usado para avaliar a presença de várias células imunes nos pulmões (tipos celulares e estratégia de gating na Figura 3) após infecção por IAV em camundongos inoculados intranasalmente com diferentes cepas de S. pneumoniae. As cepas bacterianas D39 e TIGR4, que se dispersaram nos pulmões após a infecção pelo IAV, provocaram um aumento significativo acima do valor basal (não infectado) no influxo de células imunes inflamatórias da circulação, como neutrófilos (PMNs) e monócitos, enquanto EF3030 não o fez (Figura 4A-C). A infecção isolada por IAV provocou um aumento significativo acima da linha de base no influxo de células imunes importantes para a defesa do hospedeiro contra a infecção viral, como as células NK e as células T gama-delta (Figura 4A-C). Essas respostas antivirais foram significativamente embotadas em camundongos infectados intranasalmente com S. pneumoniae antes do desafio viral (Figura 4A-C). Isso é consistente com estudos anteriores avaliando as respostas de citocinas que descobriram que o carreamento de S. pneumoniae diminuiu a produção de interferons tipo I e prejudicou a capacidade do hospedeiro de controlar as cargas de IAV nos pulmões23. Esses achados demonstram que o modelo de coinfecção pode ser usado para estudar como as respostas imunes mudam em infecções mono versus polimicrobianas.
Esse modelo também foi usado para avaliar o efeito do envelhecimento no curso da doença após a infecção por IAV em camundongos infectados intranasalmente com S. pneumoniae TIGR4. Em camundongos isolados, os títulos virais não variaram entre as coortes jovens e idosas (Figura 5A)23. Como em estudos anteriores23, camundongos velhos apresentaram sinais de doença mais precoces e significativamente mais graves em comparação com seus colegas jovens, como demonstrado pelos escores clínicos mais altos (Figura 5B). Consistente com os sintomas da doença, camundongos velhos inoculados com S. pneumoniae começaram a morrer mais rapidamente dentro de 24 h após a infecção por IAV, e todos sucumbiram à doença, enquanto os controles jovens sobreviveram à infecção em uma taxa significativamente maior (33%) (Figura 5C). Esses achados demonstram que o modelo de coinfecção pode ser usado para detectar doença mais grave em hospedeiros vulneráveis, tornando-o ideal para explorar fatores do hospedeiro que conferem resistência ou suscetibilidade à coinfecção.
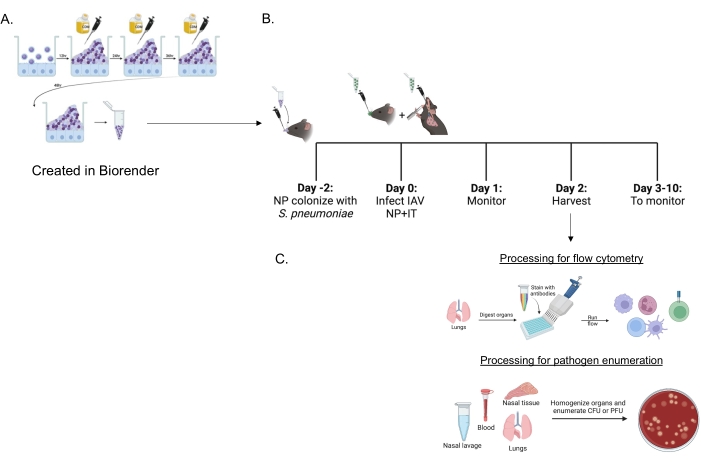
Figura 1: Linha do tempo da coinfecção e processamento de órgãos para a avaliação do influxo de células imunes e carga de patógenos . (A) Streptococcus pneumoniae são cultivados em biofilmes. (B) Camundongos são inoculados intranasalmente com 5 × 106 UFC da cepa indicada de S. pneumoniae cultivada com biofilme para estabelecer carreamento nasofaríngeo ou não tratada. Quarenta e oito horas depois, os camundongos são tratados com PBS ou recebem 200 UFP do vírus influenza A PR8 por via intranasal e 20 por via intratraqueal. Os camundongos são monitorados ao longo do tempo quanto aos escores clínicos da doença e à sobrevida. (C) Às 48 h após a infecção por IAV, são avaliadas UFC bacterianas ou UFP virais nos diferentes órgãos ou influxo de células imunes nos pulmões. Abreviaturas: UFC = unidades formadoras de colônias; UFP = unidades formadoras de placa; IAV = vírus influenza A PR8; TI = intratraqueal; NP = nasofaríngeo. Clique aqui para ver uma versão maior desta figura.
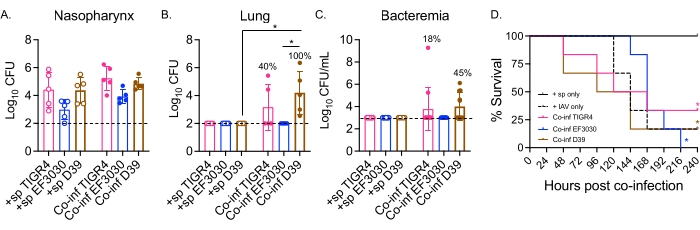
Figura 2: A dupla infecção intranasal/intratraqueal por IAV de camundongos inoculados com S. pneumoniae leva à disseminação bacteriana e doença dependente da cepa bacteriana. Camundongos C57BL/6 (B6) machos jovens (10-12 semanas de idade) foram infectados como na Figura 1. O número de bactérias na nasofaringe (A), (B) pulmões e (C) sangue foi determinado 48 h após a infecção por IAV. (B,C) As porcentagens denotam a fração de camundongos que exibiu disseminação. (D) A sobrevida foi monitorada por 10 dias após a infecção por IAV. Dados agrupados de (A,B) n = 5, (C) n = 11 e (D) n = 6 camundongos por grupo são mostrados. Cada círculo corresponde a um mouse, e as linhas tracejadas indicam o limite de detecção. (A-C)*, indica diferença significativa (p < 0,05) entre os grupos indicados, conforme determinado pelo teste de Kruskal-Wallis. (D) *, indica uma diferença significativa (p < 0,05) entre camundongos +sp e Co-inf por cepa bacteriana, conforme determinado pelo teste log-rank (Mantel-Cox). Abreviações: +sp = camundongos infectados intranasalmente com bactérias utilizando apenas a cepa indicada; Co-inf = camundongos infectados por bactérias que estavam infectados com IAV; IAV = camundongos que receberam o vírus influenza A; UFC = unidades formadoras de colônias. Clique aqui para ver uma versão maior desta figura.
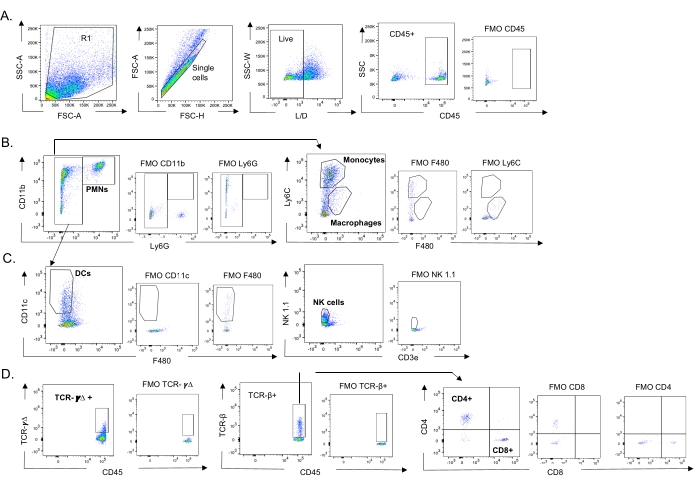
Figura 3: Estratégia de confinamento de células imunes. Os pulmões foram colhidos e o influxo de células imunes foi determinado por citometria de fluxo. A estratégia de gating representativa dos diferentes tipos celulares é mostrada. (A) CD45+, células únicas vivas foram acopladas e as porcentagens de (B) PMNs (Ly6G+, CD11b+), macrófagos (Ly6G-, Ly6C-, F480+) e monócitos (Ly6G-, Ly6C+), (C) DCs (Ly6G-, CD11c+) e células NK (NK1.1+, CD3-), (D) TCR-γΔ e CD8 (CD8+, TCRβ+) e CD4 (CD4+, TCRβ+ ) Células T foram determinadas. Abreviações: SSC-A = área do pico de dispersão lateral; FSC-A = área do pico de dispersão anterior; FSC-H = altura do pico de dispersão anterior; SSC-W = largura do pico de dispersão lateral; L/D = vivo/morto; FMO = fluorescente menos um; NK = assassino natural; PMN = leucócitos polimorfonucleares; DC = célula dendrítica; TCR = receptor de células T. Clique aqui para ver uma versão maior desta figura.
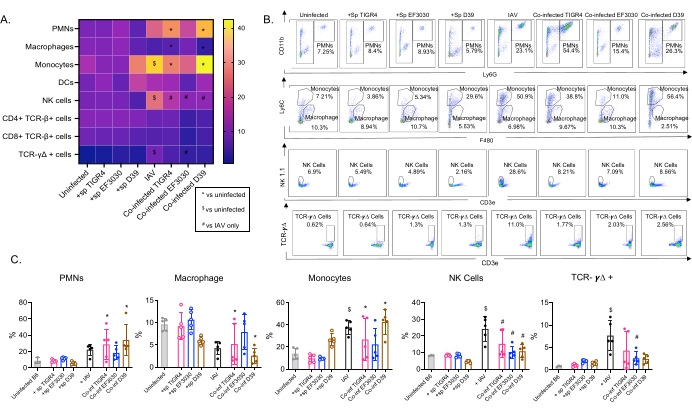
Figura 4: As respostas imunes pulmonares são bacterianas-dependentes. Camundongos jovens (10-12 semanas de idade) machos C57BL/6 não foram infectados, inoculados isoladamente com a cepa indicada de Streptococcus pneumoniae (+sp), desafiados isoladamente com IAV (IAV) ou coinfectados com S. pneumoniae e IAV (Co-inf). Quarenta e oito horas após a infecção pelo IAV (ver o desenho experimental na Figura 1), os pulmões foram colhidos e o influxo de células imunes foi determinado por citometria de fluxo seguindo a estratégia de gating na Figura 3. (A) As porcentagens médias de cada tipo de célula indicado dentro da porta CD45 são exibidas para todos os grupos de tratamento no mapa de calor. (B) Gráficos de pontos representativos dos tipos celulares que apresentaram diferenças significativas entre os tratamentos são mostrados para cada grupo de camundongos. (C) As porcentagens dos tipos de células imunes indicadas são mostradas. Cada círculo corresponde a um rato. (A,C) Dados agrupados de n = 5 camundongos por grupo são mostrados. *, indica diferença significativa (p < 0,05) entre Co-inf e não infectados; $, indica um significativo entre IAV e não infectados; #, indica uma diferença significativa entre Co-inf e IAV isoladamente. Diferenças significativas entre os grupos desafio para cada tipo celular foram determinadas por ANOVA seguida pelo teste de Tukey. Abreviações: NK = natural killer; PMN = leucócitos polimorfonucleares; DC = célula dendrítica; TCR = receptor de células T; IAV = vírus influenza A. Clique aqui para ver uma versão maior desta figura.
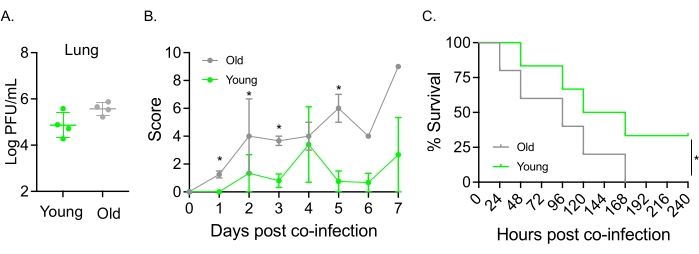
Figura 5: Envelhecimento e aumento da suscetibilidade do hospedeiro à coinfecção IAV/Streptococcus pneumoniae . Camundongos machos C57BL/6 jovens (10-12 semanas) e com idade (21-22 meses) foram coinfectados com S. pneumoniae TIGR4 i.n. e IAV i.n. e i.t. (como na Figura 1) ou desafiados isoladamente com IAV isoladamente. (A) Os títulos virais foram determinados 48 h depois. Os asteriscos indicam significância estatística (p < 0,05) determinada pelo teste t de Student. Os dados são agrupados a partir de n = 4 camundongos por grupo. (B) O escore clínico e (C) a sobrevida foram monitorados ao longo do tempo. (B) A média ± MEV agrupada de n = 6 camundongos por grupo é mostrada. Os asteriscos indicam significância estatística (p < 0,05) entre os camundongos jovens versus velhos no momento indicado, conforme determinado pelo teste de Mann-Whitney. (C) Os dados são agrupados a partir de n = 6 camundongos por grupo. Os asteriscos indicam significância estatística (p < 0,05) entre camundongos jovens versus velhos, conforme determinado pelo teste log-rank (Mantel-Cox). Abreviações: IAV = vírus influenza A; i.n. = intranasal; i.t. = intratraqueal; EPM = erro padrão da média. A Figura 5A é reimpressa com permissão de Joma et al.23. Clique aqui para ver uma versão maior desta figura.
| Mix I estoque para MDL | |
| Adenina | 0,1 gr |
| D-alanina | 0,25 gr |
| CaCl2 Anidro | 0,025 gr |
| Sulfato de Manganês | 0,03 gr |
| Cyanocobalamin | 100 μL de estoque de 10 mg/mL |
| Ácido Para-Aminobenzóico | 400 μL de 5 mg/mL de estoque |
| Piridoxamina 2HCl | 100 μL de estoque de 10 mg/mL |
| Mix II estoque para MDL | |
| Guanina | 0,05 gr |
| Uracil | 0,05 gr |
| Mix III estoque para MDL | |
| Nitrato férrico 9H2O | 50 mg/mL |
| Sulfato férrico 7H2O | 10 mg/mL |
| Mix IV estoque para MDL | |
| Dinucleotídeo de beta-nicotinamida adenina | 25 mg/mL |
Quadro 1: Misturar existências I, II, III e IV para MDL. Abreviação: MDL = mídia quimicamente definida.
| Estoque de mistura de vitaminas para MDL | |
| Cloridrato de piridoxal | 0,8 gr |
| Tiamina Cl2 | 0,4 gr |
| Riboflavina | 0,4 gr |
| Ca-pantotenato | 0,4 gr |
| Biotina | 0,04 gr |
| Ácido fólico | 0,4 gr |
| Niacinamida | 0,4 gr |
Tabela 2: Estoque de mix vitamínico para MDL. Abreviação: MDL = mídia quimicamente definida.
| Estoque de aminoácidos para MDL | |
| L-alanina | 0,480 gr |
| L-arginina | 0,250 gr |
| L-Asparagina | 0.700 g |
| Ácido L-aspártico | 0.600 g |
| L-cisteína | 1.000 gr |
| L-cistina | 0.100 gr |
| Ácido L-glutâmico | 0.200 g |
| L-Glutamina | 0,780 gr |
| L-Glicina | 0,350 gr |
| L-histidina | 0.300 gr |
| L-Isoleucina | 0,430 gr |
| L-Leucina | 0,950 gr |
| L-lisina | 0,880 gr |
| L-metionina | 0,250 gr |
| L-fenilalanina | 0,550 gr |
| L-Prolina | 1.350 gr |
| L-Serina | 0,680 gr |
| L-treonina | 0,450 gr |
| L-triptofano | 0.100 gr |
| L-Valina | 0,650 gr |
Tabela 3: Estoque de aminoácidos para MDL. Abreviação: MDL = mídia quimicamente definida.
| Estoque inicial para MDL | |
| Dextrose | 1,0 gr |
| Sulfato de magnésio-7-hidratado | 0,070 gr |
| Fosfato de potássio dibásico | 0,02 gr |
| Fosfato de potássio monobásico | 0,1 gr |
| Acetato de Sódio Anidro | 0,45 gr |
| Bicarbonato de sódio | 0,25 gr |
| Fosfato de Sódio Dibásico | 0,735 gr |
| Fosfato de Sódio Monobásico | 0.32 gr |
| Suplementos finais para MDL | |
| Cloreto de colina | 0,1 gr |
| L-cisteína HCl | 0,075 gr |
| Bicarbonato de sódio | 0,25 gr |
Quadro 4: Existências iniciais e suplementos finais para o MDL. Abreviação: MDL = mídia quimicamente definida.
| Anticorpo/Fluoróforo | Clone | Fator de diluição |
| L/D para excitação UV | N/A | 0.38888889 |
| Ly6G AF 488 | 1A8 | 0.25 |
| CD11b APC | M1/70 | 0.25 |
| CD11c PE | N418 | 0.18055556 |
| Bloco Fc do mouse | 2,4G2 | 0.11111111 |
| F4/80 PE Cy7 | BM8 | 0.18055556 |
| Ly6C BV605 | AL-21 | 0.25 |
| Placa CD103 BV 421 | M290 | 0.18055556 |
| CD45 APC-eF-780 | 30-F11 | 0.18055556 |
Tabela 5: Painel de anticorpos 1.
| Anticorpo/Fluoróforo | Clone | Fator de diluição |
| L/D para excitação UV | N/A | 0.388888889 |
| TCR-β APC Cy7 | H 57-597 | 0.180555556 |
| CD4 V450 (azul do Pacífico) | RM4-5 | 0.25 |
| CD8 BV650 | 53-6.7 | 0.180555556 |
| Bloco Fc do mouse | 2,4G2 | 0.111111111 |
| CD45 PE | 30-F11 | 0.180555556 |
| CD3 AF488 | 145-2C11 | 0.180555556 |
| TCR- γΔ APC | GL-3 | 0.180555556 |
| NK1.1 AF 700 | PK136 | 0.180555556 |
Tabela 6: Painel de anticorpos 2.
Discussão
A maioria dos estudos experimentais existentes de co-infecção por S. pneumoniae/IAV baseia-se na entrega bacteriana nos pulmões de camundongos pré-infectados com IAV. Esses modelos têm auxiliado na identificação de alterações no ambiente pulmonar e na resposta imune sistêmica que tornam o hospedeiro suscetível à infecção bacteriana secundária15,16,17,32,33,34,35,36,37. No entanto, esses modelos falharam em mimetizar a transição de S. pneumoniae de um colonizador assintomático para um patógeno capaz de causar infecções pulmonares e sistêmicas graves. Além disso, esses modelos não são adequados para estudar os fatores do hospedeiro e as interações patógeno-hospedeiro no trato respiratório superior que contribuem para a suscetibilidade à infecção. Um modelo prévio para o movimento de pneumococos da nasofaringe para o pulmão após a infecção por IAV baseava-se em infecção bacteriana da nasofaringe seguida de infecção viral. No entanto, não conseguiu reproduzir os sinais graves da doença observados em pacientes humanos21. O modelo de infecção murina modificado descrito aqui recapitula a transição de S. pneumoniae de carreador assintomático para patógeno causador de doença clínica grave.
Uma etapa crítica desse modelo é o estabelecimento da infecção por S. pneumoniae na nasofaringe. Streptococcus pneumoniae forma biofilmes e coloniza a nasofaringe em diferenteseficiências 21,38. Para estabelecer infecção consistente, são necessárias pelo menos 5 × 106 UFC das cepas bacterianas cultivadas em biofilme testadas até o momento23. Recomenda-se que qualquer nova cepa bacteriana seja testada para infecção estável da nasofaringe antes da infecção viral. Para a coinfecção viral, estudos prévios constataram que a infecção intranasal com IAV é necessária para a dispersão da bactéria da nasofaringe21,22,23. Nesses estudos anteriores, 500 UFP de IAV para parto intranasal foram utilizadas, enquanto neste estudo, 200 UFP foram suficientes para aumentar o número de bactérias na nasofaringe. A infecção pelo VAI não se limita às vias aéreas superiores e pode se disseminar para os pulmões39,40, o que é fundamental para tornar o ambiente pulmonar mais permissivo à infecção bacteriana15,16,41. A entrega de IAV aos pulmões pode ser alcançada por entrega intranasal ou instalação intratraqueal de camundongos anestesiados. Trabalhos anteriores com camundongos BALB/cByJ descobriram que o parto intranasal resulta em pneumonia viral21; no entanto, o acesso do inóculo aos pulmões após a inoculação intranasal é mais restrito em camundongos C57BL/6. Em camundongos C57BL/6, a instalação intratraqueal é necessária para a liberação consistente do vírus23. Nesse modelo, a colonização bacteriana prévia acelera a apresentação dos sintomas da doença após a infecção viral23. Como a própria infecção viral pode causar sintomas da doença com potencial variação na cinética, recomenda-se primeiro testar um intervalo de doses para qualquer nova cepa viral testada e escolher uma dose que revele cinética acelerada em hospedeiros coinfectados.
Os pulmões fornecem outra leitura crítica para a avaliação da doença neste modelo. Para a avaliação da carga de patógenos e do influxo de células imunes, um pulmão do mesmo camundongo pode ser usado. No entanto, como a gravidade da infecção e da inflamação pode diferir entre os lobos, recomenda-se não tomar lobos diferentes do mesmo pulmão para as várias avaliações. Em vez disso, todos os lóbulos podem ser picados em pequenos pedaços, misturados bem juntos e, em seguida, analisados igualmente para as diferentes avaliações. Da mesma forma, a nasofaringe pode ser usada para a enumeração de UFC bacteriana ou PFU viral e resposta imune. No entanto, o número de células obtidas das lavagens e do tecido é muito baixo para realizar citometria de fluxo sem agrupar as amostras de camundongos dentro do mesmo grupo. Alternativamente, a inflamação na nasofaringe pode ser avaliadahistologicamente23.
Uma característica crítica desse modelo é que ele recapitula a doença clínica observada nos pacientes. Em humanos, a pneumonia pneumocócica secundária após infecção por VAI frequentemente resulta em sinais óbvios da doença, incluindo tosse, dispneia, febre e dores musculares que podem levar a hospitalizações, insuficiência respiratória e até morte 8,15,42,43. Este modelo recapitula os sinais graves de doença clínica observados em humanos em termos de dificuldade em respirar (refletida no escore respiratório) e mal-estar geral (refletido nos escores de postura e movimento) exibidos pelos camundongos, bem como morte em alguns dos controles jovens saudáveis. Os sintomas exacerbados da doença em camundongos co-infectados são provavelmente resultado tanto da disseminação bacteriana para os pulmões quanto da depuração viral prejudicada em camundongos com carreamento pneumocócico23. Uma limitação do modelo é que a incidência de doença clínica e disseminação bacteriana da nasofaringe varia entre camundongos e é influenciada pela cepa bacteriana, idade do hospedeiro e genótipo21,22,23. Refletindo isso, para cepas invasivas, a progressão de infecção localizada (sem bacteremia detectável) para morte pode ocorrer dentro de 24 h. Portanto, para uma verdadeira avaliação da disseminação sistêmica, a bacteremia deve ser acompanhada em intervalos mais curtos (a cada 6-12 h). Da mesma forma, o escore da doença pode mudar rapidamente, particularmente nas primeiras 72 h após a coinfecção. Portanto, para acompanhar de perto os sintomas da doença, é aconselhável monitorar camundongos três vezes por dia durante os dias 1-3 após a infecção por IAV.
Em resumo, esse modelo replica o movimento de S. pneumoniae de um colonizador assintomático da nasofaringe para um patógeno capaz de causar doença pulmonar e sistêmica na infecção por IAV. Nesse modelo, o IAV desencadeia a transição de S. pneumoniae modificando o comportamento bacteriano na nasofaringe, aumentando a disseminação bacteriana para o pulmão e alterando a imunidade antibacteriana23. Da mesma forma, o carreamento bacteriano atenua as respostas imunes antivirais e prejudica a depuração do IAV dos pulmões23. Isso torna esse modelo ideal para analisar mudanças nas respostas imunes em infecções únicas versus polimicrobianas. Além disso, o curso da doença após a coinfecção é, em parte, dependente da cepa de pneumococo presente na nasofaringe. Portanto, o modelo é adequado para dissecar os fatores bacterianos necessários para a colonização assintomática versus transição patogênica de S. pneumoniae. Por fim, esse modelo reproduz a suscetibilidade do envelhecimento a coinfecções e, embora não tenha sido testado aqui, pode ser facilmente usado para avaliar o impacto do background do hospedeiro no curso da doença. Em conclusão, separar carreador e doença em etapas distintas oferece a oportunidade de analisar as variantes genéticas tanto do patógeno quanto do hospedeiro, permitindo o exame detalhado das interações de um importante patobionte com o hospedeiro em diferentes fases da progressão da doença. No futuro, esse modelo pode ser usado para adaptar opções de tratamento para hospedeiros vulneráveis.
Divulgações
Os autores não têm conflitos de interesse a declarar.
Agradecimentos
Agradecemos a Nick Lenhard pela leitura crítica e edição deste manuscrito. Também gostaríamos de agradecer a Andrew Camilli e Anthony Campagnari pelas cepas bacterianas e Bruce Davidson pelas cepas virais. Este trabalho foi apoiado pelo National Institute of Health Grant (R21AG071268-01) para J.L. e pelo National Institute of Health Grants (R21AI145370-01A1), (R01AG068568-01A1), (R21AG071268-01) para E.N.B.G.
Materiais
| Name | Company | Catalog Number | Comments |
| 4-Aminobenzoic acid | Fisher | AAA1267318 | Mix I stock |
| 96-well round bottom plates | Greiner Bio-One | 650101 | |
| 100 µm Filters | Fisher | 07-201-432 | |
| Adenine | Fisher | AC147440250 | Mix I stock |
| Avicel | Fisher | 501785325 | Microcyrstalline cellulose |
| BD Cytofix Fixation Buffer | Fisher | BDB554655 | Fixation Buffer |
| BD Fortessa | Flow cytometer | ||
| BD Intramedic Polyethylene Tubing | Fisher | 427410 | Tubing for nasal lavage |
| BD Disposable Syringes with Luer-Lok Tips (1 mL) | Fisher | 14-823-30 | |
| BD Microtainer Capillary Blood Collector and BD Microgard Closure | Fisher | 02-675-185 | Blood collection tubes |
| Beta-Nicotinamide adenine dinucleotide | Fisher | AAJ6233703 | Mix IV stock |
| Biotin | Fisher | AC230090010 | Vitamin stock |
| C57BL/6J mice | The Jackson Laboratory | #000644 | Mice used in this study |
| Calcium Chloride Anhydrous | Fisher Chemical | C77-500 | Mix I stock |
| CD103 BV 421 | BD Bioscience | BDB562771 | Clone: M290 DF 1:200 |
| CD11b APC | Invitrogen | 50-112-9622 | Clone: M1/70, DF 1:300 |
| CD11c PE | BD Bioscience | BDB565592 | Clone: N418 DF 1:200 |
| CD3 AF 488 | BD Bioscience | OB153030 | Clone: 145-2C11 DF 1:200 |
| CD4 V450 | BD Horizon | BDB560470 | Clone: RM4.5 DF 1:300 |
| CD45 APC eF-780 | BD Bioscience | 50-112-9642 | Clone: 30-F11 DF 1:200 |
| CD45 PE | Invitrogen | 50-103-70 | Clone: 30-F11 DF 1:200 |
| CD8α BV 650 | BD Horizon | BDB563234 | Clone: 53-6.7 DF 1:200 |
| Choline chloride | Fisher | AC110290500 | Final supplement to CDM |
| Corning Disposable Vacuum Filter/Storage Systems | Fisher | 09-761-107 | Filter sterilzation apparatus |
| Corning Tissue Culture Treated T-25 Flasks | Fisher | 10-126-9 | |
| Corning Costar Clear Multiple Well Plates | Fisher | 07-201-590 | |
| Corning DMEM With L-Glutamine and 4.5 g/L Glucose; Without Sodium Pyruvate | Fisher | MT10017CM | |
| Cyanocobalamin | Fisher | AC405925000 | Mix I stock |
| D39 | National Collection of Type Culture (NCTC) | NCTC 7466 | Streptococcus pneumoniae strain |
| D-Alanine | Fisher | AAA1023114 | Mix I stock |
| D-Calcium pantothenate | Fisher | AC243301000 | Vitamin stock |
| Dextrose | Fisher Chemical | D16-500 | Starter stock |
| Dnase | Worthington Biochemical | LS002147 | |
| Eagles Minimum Essential Medium | ATCC | 30-2003 | |
| EDTA | VWR | BDH4616-500G | |
| EF3030 | Center for Disease Control and Prevention | Available via the isolate bank request | Streptococcus pneumoniae strain, request using strain name |
| F480 PE Cy7 | BD Bioscience | 50-112-9713 | Clone: BMB DF 1:200 |
| Falcon 50 mL Conical Centrifuge Tubes | Fisher | 14-432-22 | 50 mL round bottom tube |
| Falcon Round-Bottom Polypropylene Test Tubes With Cap | Fisher | 14-959-11B | 15 mL round bottom tube |
| Falcon Round-Bottom Polystyrene Test Tubes (5 mL) | Fisher | 14-959-5 | FACS tubes |
| FBS | Thermofisher | 10437-028 | |
| Ferric Nitrate Nonahydrate | Fisher | I110-100 | Mix III stock |
| Fisherbrand Delicate Dissecting Scissors | Fisher | 08-951-5 | Instruments used for harvest |
| Fisherbrand Disposable Inoculating Loops | Fisher | 22-363-602 | Inoculating loops |
| Fisherbrand Dissecting Tissue Forceps | Fisher | 13-812-38 | Forceps for harvest |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5 mL | Fisher | 05-408-137 | Micocentrifuge tubes |
| Fisherbrand Sterile Syringes for Single Use (10 mL) | Fisher | 14-955-459 | |
| Folic Acid | Fisher | AC216630500 | Vitamin stock |
| Gibco RPMI 1640 (ATCC) | Fisher | A1049101 | |
| Gibco DPBS, no calcium, no magnesium | Fisher | 14190250 | |
| Gibco HBSS, calcium, magnesium, no phenol red | Fisher | 14025134 | |
| Gibco MEM (Temin's modification) (2x), no phenol red | Fisher | 11-935-046 | |
| Gibco Penicillin-Streptomycin (10,000 U/mL) | Fisher | 15-140-122 | |
| Gibco Trypan Blue Solution, 0.4% | Fisher | 15-250-061 | |
| Gibco Trypsin-EDTA (0.25%), phenol red | Fisher | 25-200-056 | |
| Glycerol (Certified ACS) | Fisher | G33-4 | |
| Glycine | Fisher | AA3643530 | Amino acid stock |
| Guanine | Fisher | AAA1202414 | Mix II stock |
| Invitrogen UltraComp eBeads Compensation Beads | Fisher | 50-112-9040 | |
| Iron (II) sulfate heptahydrate | Fisher | AAA1517836 | Mix III stock |
| L-Alanine | Fisher | AAJ6027918 | Amino acid stock |
| L-Arginine | Fisher | AAA1573814 | Amino acid stock |
| L-Asparagine | Fisher | AAB2147322 | Amino acid stock |
| L-Aspartic acid | Fisher | AAA1352022 | Amino acid stock |
| L-Cysteine | Fisher | AAA1043518 | Amino acid stock |
| L-Cysteine hydrochloride monohydrate | Fisher | AAA1038914 | Final supplement to CDM |
| L-Cystine | Fisher | AAA1376218 | Amino acid stock |
| L-Glutamic acid | Fisher | AC156211000 | Amino acid stock |
| L-Glutamine | Fisher | O2956-100 | Amino acid stock |
| L-Histidine | Fisher | AC166150250 | Amino acid stock |
| LIFE TECHNOLOGIES LIVE/DEAD Fixable Blue Dead Cell Stain Kit, for UV excitation | Invitrogen | 50-112-1524 | Clone: N/A DF 1:500 |
| L-Isoleucine | Fisher | AC166170250 | Amino acid stock |
| L-Leucine | Fisher | BP385-100 | Amino acid stock |
| L-Lysine | Fisher | AAJ6222514 | Amino acid stock |
| L-Methionine | Fisher | AAA1031822 | Amino acid stock |
| Low endotoxin BSA | Sigma Aldrich | A1470-10G | |
| L-Phenylalanine | Fisher | AAA1323814 | Amino acid stock |
| L-Proline | Fisher | AAA1019922 | Amino acid stock |
| L-Serine | Fisher | AC132660250 | Amino acid stock |
| L-Threonine | Fisher | AC138930250 | Amino acid stock |
| L-Tryptophan | Fisher | AAA1023014 | Amino acid stock |
| L-Valine | Fisher | AAA1272014 | Amino acid stock |
| Ly6C BV 605 | BD Bioscience | BDB563011 | Clone: AL-21 DF 1:300 |
| Ly6G AF 488 | Biolegend | NC1102120 | Clone: IA8, DF 1:300 |
| Madin-Darby Canine Kidney (MDCK) cells | American Type Culture Collection (ATCC) | CCL-34 | MDCK cell line for PFU analuysis |
| Magnesium Sulfate 7-Hydrate | Fisher | 60-019-68 | CDM starter stock |
| Manganese Sulfate | Fisher | M113-500 | Mix I stock |
| MilQ water | Ultra-pure water | ||
| Mouse Fc Block | BD Bioscience | BDB553142 | Clone: 2.4G2 DF 1:100 |
| MWI VETERINARY PURALUBE VET OINTMENT | Fisher | NC1886507 | Eye lubricant for infection |
| NCI-H292 mucoepidermoid carcinoma cell line | ATCC | CRL-1848 | H292 lung epithelial cell line for biofilm growth |
| Niacinamide | Fisher | 18-604-792 | Vitamin stock |
| NK 1.1 AF 700 | BD Bioscience | 50-112-4692 | Clone: PK136 DF 1:200 |
| Oxyrase For Broth 50Ml Bottle 1/Pk | Fisher | 50-200-5299 | To remove oxygen from liquid cultures |
| Paraformaldehyde 4% in PBS | Thermoscientific | J19932-K2 | |
| Pivetal Isoflurane | Patterson Veterinary | 07-893-8440 | Isoflurane for anesthesia during infection |
| Potassium Phosphate Dibasic | Fisher Chemical | P288-500 | Starter stock |
| Potassium Phosphate Monobasic | Fisher Chemical | P285-500 | Starter stock |
| Pyridoxal hydrochloride | Fisher | AC352710250 | Vitamin stock |
| Pyridoxamine dihydrochloride | Fisher | AAJ6267906 | Mix I stock |
| Riboflavin | Fisher | AC132350250 | Vitamin stock |
| Sodium Acetate | VWR | 0530-500G | Starter stock |
| Sodium Azide | Fisher Bioreagents | BP922I-500 | For FACS buffer |
| Sodium Bicarbonate | Fisher Chemical | S233-500 | Starter stock and final supplement to CDM |
| Sodium Phosphate Dibasic | Fisher Chemical | S374-500 | Starter stock |
| Sodium Phosphate Monobasic | Fisher Chemical | S369-500 | Starter stock |
| TCR APC | BD Bioscience | 50-112-8889 | Clone: GL-3 DF 1:200 |
| TCRβ APC-Cy7 | BD Pharmigen | BDB560656 | Clone: H57-597 DF 1:200 |
| Thermo Scientific Blood Agar with Gentamicin | Fisher | R01227 | Blood agar plates with the antibiotic gentamicin |
| Thermo Scientific Trypsin, TPCK Treated | Fisher | PI20233 | |
| Thiamine hydrochloride | Fisher | AC148991000 | Vitamin stock |
| TIGR4 | ATCC | BAA-334 | Streptococcus pneumoniae strain |
| Uracil | Fisher | AC157300250 | Mix II stock |
| Worthington Biochemical Corporation Collagenase, Type 2, 1 g | Fisher | NC9693955 |
Referências
- Kadioglu, A., Weiser, J. N., Paton, J. C., Andrew, P. W. The role of Streptococcus pneumoniae virulence factors in host respiratory colonization and disease. Nature Reviews Microbiology. 6 (4), 288-301 (2008).
- Obaro, S., Adegbola, R. The pneumococcus: Carriage, disease and conjugate vaccines. Journal of Medical Microbiology. 51 (2), 98-104 (2002).
- Chong, C. P., Street, P. R. Pneumonia in the elderly: A review of the epidemiology, pathogenesis, microbiology, and clinical features. Southern Medical Journal. 101 (11), 1141-1145 (2008).
- Kadioglu, A., Andrew, P. W. Susceptibility and resistance to pneumococcal disease in mice. Briefings in Functional Genomics and Proteomics. 4 (3), 241-247 (2005).
- Ganie, F., et al. Structural, genetic, and serological elucidation of Streptococcus pneumoniae serogroup 24 serotypes: Discovery of a new serotype, 24C, with a variable capsule structure. Journal of Clinical Microbiology. 59 (7), 0054021 (2021).
- Centers for Disease Control and Prevention. Estimates of deaths associated with seasonal influenza --- United States. MMWR. Morbidity and Mortality Weekly Report. 59 (33), 1057-1062 (2010).
- Shrestha, S., et al. Identifying the interaction between influenza and pneumococcal pneumonia using incidence data. Science Translational Medicine. 5 (191), (2013).
- McCullers, J. A. Insights into the interaction between influenza virus and pneumococcus. Clinical Microbiology Reviews. 19 (3), 571-582 (2006).
- Pneumococcal Disease Global Pneumococcal Disease and Vaccine. Centers for Disease Control and Prevention Available from: https://www.cdc.gov/pneumococcal/global.html (2018)
- Grudzinska, F. S., et al. Neutrophils in community-acquired pneumonia: Parallels in dysfunction at the extremes of age. Thorax. 75 (2), 164-171 (2020).
- Boe, D. M., Boule, L. A., Kovacs, E. J. Innate immune responses in the ageing lung. Clinical and Experimental Immunology. 187 (1), 16-25 (2017).
- Krone, C. L., van de Groep, K., Trzcinski, K., Sanders, E. A., Bogaert, D. Immunosenescence and pneumococcal disease: An imbalance in host-pathogen interactions. The Lancet Respiratory Medicine. 2 (2), 141-153 (2014).
- Cho, S. J., et al. Decreased NLRP3 inflammasome expression in aged lung may contribute to increased susceptibility to secondary Streptococcus pneumoniae infection. Experimental Gerontology. 105, 40-46 (2018).
- Disease Burden of Influenza. Centers for Disease Control and Prevention Available from: https://www.cdc.gov/flu/about/burden/index.html (2018)
- McCullers, J. A. The co-pathogenesis of influenza viruses with bacteria in the lung. Nature Reviews Microbiology. 12 (4), 252-262 (2014).
- McCullers, J. A., Rehg, J. E. Lethal synergism between influenza virus and Streptococcus pneumoniae: Characterization of a mouse model and the role of platelet-activating factor receptor. The Journal of Infectious Diseases. 186 (3), 341-350 (2002).
- Metzger, D. W., Sun, K. Immune dysfunction and bacterial coinfections following influenza. Journal of Immunology. 191 (5), 2047-2052 (2013).
- Chao, Y., Marks, L. R., Pettigrew, M. M., Hakansson, A. P. Streptococcus pneumoniae biofilm formation and dispersion during colonization and disease. Frontiers in Cellular and Infection Microbiology. 4, 194 (2014).
- Bogaert, D., De Groot, R., Hermans, P. W. Streptococcus pneumoniae colonisation: The key to pneumococcal disease. The Lancet Infectious Diseases. 4 (3), 144-154 (2004).
- Simell, B., et al. The fundamental link between pneumococcal carriage and disease. Expert Review of Vaccines. 11 (7), 841-855 (2012).
- Marks, L. R., Davidson, B. A., Knight, P. R., Hakansson, A. P. Interkingdom signaling induces Streptococcus pneumoniae biofilm dispersion and transition from asymptomatic colonization to disease. mBio. 4 (4), 00438 (2013).
- Reddinger, R. M., Luke-Marshall, N. R., Sauberan, S. L., Hakansson, A. P., Campagnari, A. A. Streptococcus pneumoniae modulates Staphylococcus aureus biofilm dispersion and the transition from colonization to invasive disease. mBio. 9 (1), 02089 (2018).
- Joma, B. H., et al. A murine model for enhancement of Streptococcus pneumoniae pathogenicity upon viral infection and advanced age. Infection and Immunity. 89 (8), 0047120 (2021).
- Andersson, B., et al. Identification of an active disaccharide unit of a glycoconjugate receptor for pneumococci attaching to human pharyngeal epithelial cells. Journal of Experimental Medicine. 158 (2), 559-570 (1983).
- Avery, O. T., Macleod, C. M., McCarty, M. Studies on the chemical nature of the substance inducing transformation of pneumococcal types: Induction of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III. The Journal of Experimental Medicine. 79 (2), 137-158 (1944).
- Tettelin, H., et al. Complete genome sequence of a virulent isolate of Streptococcus pneumoniae. Science. 293 (5529), 498-506 (2001).
- Tothpal, A., Desobry, K., Joshi, S. S., Wyllie, A. L., Weinberger, D. M. Variation of growth characteristics of pneumococcus with environmental conditions. BMC Microbiology. 19 (1), 304 (2019).
- Bou Ghanem, E. N., et al. Extracellular adenosine protects against Streptococcus pneumoniae lung infection by regulating pulmonary neutrophil recruitment. PLoS Pathogens. 11 (8), 1005126 (2015).
- Bou Ghanem, E. N., et al. The alpha-tocopherol form of vitamin E boosts elastase activity of human PMNs and their ability to kill Streptococcus pneumoniae. Frontiers in Cellular and Infection Microbiology. 7, 161 (2017).
- Tait, A. R., Davidson, B. A., Johnson, K. J., Remick, D. G., Knight, P. R. Halothane inhibits the intraalveolar recruitment of neutrophils, lymphocytes, and macrophages in response to influenza virus infection in mice. Anesthesia & Analgesia. 76 (5), 1106-1113 (1993).
- Aaberge, I. S., Eng, J., Lermark, G., Lovik, M. Virulence of Streptococcus pneumoniae in mice: A standardized method for preparation and frozen storage of the experimental bacterial inoculum. Microbial Pathogenesis. 18 (2), 141-152 (1995).
- McCullers, J. A., Bartmess, K. C. Role of neuraminidase in lethal synergism between influenza virus and Streptococcus pneumoniae. The Journal of Infectious Diseases. 187 (6), 1000-1009 (2003).
- Smith, A. M., McCullers, J. A. Secondary bacterial infections in influenza virus infection pathogenesis. Current Topics in Microbiology and Immunology. 385, 327-356 (2014).
- Cundell, D. R., Gerard, N. P., Gerard, C., Idanpaan-Heikkila, I., Tuomanen, E. I. Streptococcus pneumoniae anchor to activated human cells by the receptor for platelet-activating factor. Nature. 377 (6548), 435-438 (1995).
- Ballinger, M. N., Standiford, T. J. Postinfluenza bacterial pneumonia: Host defenses gone awry. Journal of Interferon & Cytokine Research. 30 (9), 643-652 (2010).
- Sun, K., Metzger, D. W. Inhibition of pulmonary antibacterial defense by interferon-gamma during recovery from influenza infection. Nature Medicine. 14 (5), 558-564 (2008).
- Nakamura, S., Davis, K. M., Weiser, J. N. Synergistic stimulation of type I interferons during influenza virus coinfection promotes Streptococcus pneumoniae colonization in mice. Journal of Clinical Investigation. 121 (9), 3657-3665 (2011).
- Blanchette-Cain, K., et al. Streptococcus pneumoniae biofilm formation is strain dependent, multifactorial, and associated with reduced invasiveness and immunoreactivity during colonization. mBio. 4 (5), 00745 (2013).
- Rello, J., Pop-Vicas, A. Clinical review: Primary influenza viral pneumonia. Critical Care. 13 (6), 235 (2009).
- Torres, A., Loeches, I. M., Sligl, W., Lee, N. Severe flu management: A point of view. Intensive Care Medicine. 46 (2), 153-162 (2020).
- Bakaletz, L. O. Viral-bacterial co-infections in the respiratory tract. Current Opinion in Microbiology. 35, 30-35 (2017).
- Palacios, G., et al. Streptococcus pneumoniae coinfection is correlated with the severity of H1N1 pandemic influenza. PLoS One. 4 (12), 8540 (2009).
- Dhanoa, A., Fang, N. C., Hassan, S. S., Kaniappan, P., Rajasekaram, G. Epidemiology and clinical characteristics of hospitalized patients with pandemic influenza A (H1N1) 2009 infections: The effects of bacterial coinfection. Virology Journal. 8, 501 (2011).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados