Method Article
Production de neurones 2-inductibles de neurogénine humaine dans un bioréacteur en suspension tridimensionnel
Dans cet article
Résumé
Cet article décrit un protocole pour la génération de neurones induits par l’homme dérivés de cellules souches pluripotentes dans un bioréacteur de suspension 3D de paillasse.
Résumé
La dérivation de cellules de lignées neuronales à partir de cellules souches pluripotentes induites par l’homme (CSPhi) a marqué une étape importante dans la recherche sur le cerveau. Depuis leur premier avènement, les protocoles ont été continuellement optimisés et sont maintenant largement utilisés dans la recherche et le développement de médicaments. Cependant, la très longue durée de ces protocoles conventionnels de différenciation et de maturation et la demande croissante de CSPi de haute qualité et de leurs dérivés neuronaux soulèvent la nécessité d’adopter, d’optimiser et de normaliser ces protocoles pour une production à grande échelle. Ce travail présente un protocole rapide et efficace pour la différenciation des hiPSC génétiquement modifiés exprimant la neurogénine 2 inductible par la doxycycline (iNGN2) en neurones à l’aide d’un bioréacteur de suspension tridimensionnel (3D) de paillasse.
En bref, les suspensions unicellulaires de CSPhi iNGN2 ont été autorisées à former des agrégats en 24 heures, et l’engagement de la lignée neuronale a été induit par l’ajout de doxycycline. Les agrégats ont été dissociés après 2 jours d’induction et les cellules ont été cryoconservées ou replaquées pour la maturation terminale. Les neurones iNGN2 générés ont exprimé des marqueurs neuronaux classiques dès le début et ont formé des réseaux neuritiques complexes dans la semaine 1 après le replating, indiquant une maturité croissante des cultures neuronales. En résumé, un protocole détaillé étape par étape pour la génération rapide de neurones dérivés de l’hiPSC dans un environnement 3D est fourni qui présente un grand potentiel comme point de départ pour la modélisation de la maladie, les criblages phénotypiques de médicaments à haut débit et les tests de toxicité à grande échelle.
Introduction
Les troubles neurologiques sont la principale cause d’invalidité dans le monde1. Une personne sur six est touchée, et l’incidence continue d’augmenter. Le fardeau financier qui en découle pour les sociétés et leurs systèmes de soins de santé est énorme. Une évaluation de 30 pays européens en 2010 a estimé les coûts annuels liés aux troubles mentaux et neurologiques à 800 milliards d’euros2. Le fardeau socio-économique croissant exige des stratégies de traitement efficaces, et bien que notre compréhension de la physiopathologie de la maladie ait considérablement augmenté, la traduction dans les cliniques est souvent insuffisante. En général, seulement 12 % des produits pharmaceutiques font l’objet d’essais cliniques, dont plus de 80 % échouent aux stades ultérieurs, principalement en raison de leur inefficacité ou d’une toxicité imprévue 3,4. Les raisons sont variées, mais la transférabilité limitée de l’expérimentation animale aux essais sur l’homme est de plus en plus mise en évidence5. Les modèles de cellules et de tissus humains in vitro peuvent combler le fossé de la traduction interspécifique, et les progrès de la technologie des cellules souches pluripotentes induites par l’homme (hiPSC) possèdent un grand potentiel à cet égard. Les CSPh sont largement utilisées en recherche fondamentale et partagent des caractéristiques essentielles avec les cellules souches embryonnaires (CSEh), telles qu’une capacité d’auto-renouvellement presque illimitée et la capacité de se différencier en trois couches germinales6, tout en contournant les préoccupations éthiques associées à la destruction embryonnaire.
La dérivation de cellules neuronales à partir de CSEh, et plus tard de CSPh, a marqué une étape importante dans la recherche sur le cerveau. Les protocoles de différenciation initiaux étaient basés sur l’application de facteurs de croissance imitant des étapes critiques au cours de l’embryogenèse, la plupart d’entre eux impliquant une double inhibition de la SMAD en suspension ou en cultures adhérentes 7,8,9. Les neurones matures ont été générés avec succès, mais plusieurs inconvénients de ces protocoles entravent encore leur large utilisation dans le développement de médicaments, tels que le faible rendement et la grande variabilité d’un lot à l’autre des neurones générés, ainsi que la charge de travail importante liée aux longs temps de culture. Des améliorations ont été obtenues par l’expression forcée de facteurs de transcription impliqués de manière critique dans la neurogenèse, et les membres de la famille des neurogénines (NGN), en particulier NGN2, ont été identifiés comme des facteurs efficaces10. L’expression ectopique de NGN2 médiée par le lentiviral dans les CSPhi a considérablement accéléré les premiers stades de la différenciation neuronale et induit le destin des cellules neuronales en seulement 1 semaine11. La maturation terminale ultérieure dans des co-cultures avec des astrocytes a donné des neurones fonctionnels d’une grande pureté et en grande quantité avec des propriétés reproductibles. L’édition génique dirigée sur site du locus refuge du site d’intégration des virus adéno-associés 1 (AAVS1) a ensuite été appliquée pour créer des lignes hiPSC avec une cassette d’expression stable et inductible par la doxycycline pour NGN212,13, minimisant ainsi les effets secondaires indésirables de l’administration de lentiviraux.
La différenciation robuste et efficace des neurones de la neurogénine 2 inductible par la doxycycline (iNGN2) présente un grand potentiel pour les criblages phénotypiques de médicaments à haut débit et les essais de toxicité10,14,15; et des progrès substantiels dans le biotraitement ont été réalisés au cours de la dernière décennie en mettant en œuvre des bioréacteurs pour une expansion et une différenciation évolutivesdes cellules 16,17,18,19. Cependant, la majorité des protocoles de différenciation sont optimisés pour les cultures adhérentes, et la traduction dans un environnement tridimensionnel (3D) nécessite souvent des modifications essentielles. Récemment, l’utilisation réussie d’un bioréacteur 3D de paillasse avec des caractéristiques de contrainte de cisaillement réduites a été rapportée pour l’expansion des CSPhi et pour la différenciation reproductible en hépatocytes, cardiomyocytes et neurones20. Ici, un protocole détaillé pour la génération et la caractérisation des neurones iNGN2 est fourni en utilisant le bioréacteur 3D de paillasse identique.
Protocole
NOTE: Toutes les manipulations cellulaires, ainsi que les boîtes de culture et les préparations de milieu, doivent être effectuées dans des conditions stériles. La hotte à flux laminaire doit être nettoyée à fond avant utilisation et après le traitement en essuyant toutes les surfaces avec de l’éthanol à 70%. Le protocole décrit est optimisé pour les différenciations neuronales dans un incubateur et un bioréacteur CERO 3D (ci-après dénommé bioréacteur de paillasse). Ce bioréacteur de paillasse offre quatre emplacements pour les tubes de bioréacteur spécialisés, chacun d’une capacité maximale de 50 mL. La température et les niveaux de CO2 sont contrôlés en permanence, et les paramètres de culture (p. ex., vitesse et temps de rotation) sont réglés indépendamment pour chaque tube. Toutes les étapes d’aspiration ont été effectuées à l’aide d’une pipette d’aspiration et d’une pompe à vide, sauf indication contraire.
1. Culture et expansion des CSPh
REMARQUE: La lignée NGN2 hiPSC inductible par la doxycycline BIONi010-C-13 est utilisée dans ce protocole. Le protocole d’expansion fourni ici est optimisé pour les boîtes de Petri de 6 cm, mais des formats de culture alternatifs peuvent être utilisés si vous préférez.
- Enrober des boîtes de Petri de 6 cm avec une matrice membranaire basale diluée dans le milieu Eagle’s medium modifié (DMEM)/F12 (-/-) de Dulbecco à froid à une concentration finale de 0,083 mg/mL/10 cm2. Incuber les plats enrobés à 37 °C pendant au moins 30 min.
REMARQUE: Un protocole détaillé pour la préparation de la solution matricielle membranaire basale se trouve dans les instructions du fabricant. Les hiPSC peuvent être cultivées sur d’autres matrices extracellulaires pour l’expansion. - Décongeler les hiPSC cryoconservés conformément au protocole utilisateur21 de la lignée cellulaire EBiSC dans un milieu d’entretien iPSC sans alimentation + inhibiteur de ROCK de 10 μM (Y-27632). Une densité de semis de 1 × 106 cellules viables par boîte de 60 mm est recommandée.
- Changez le milieu le lendemain pour un milieu d’entretien iPSC sans alimentation sans inhibiteur de ROCK et effectuez des changements quotidiens de milieu.
- Commencez à passer lorsque les cultures hiPSC atteignent une confluence de 60% à 80%. Vérifiez les zones différenciées et nettoyez les colonies manuellement si la zone dépasse 5%.
REMARQUE: Les CSPhi indifférenciées apparaissent sous forme de cellules rondes avec un nucléole proéminent et moins de cytoplasme. Des colonies plates et serrées se forment tôt après la décongélation ou le passage. Des exemples d’images en clair de cultures hiPSC sont présentées à la figure 1. De plus amples informations sont fournies dans le protocole utilisateur EBiSC Cell lineUser Protocol 21. Pour le passage, préparez des plats recouverts d’une matrice membranaire basale et un milieu d’entretien iPSC sans alimentation. - Aspirer le milieu à partir de cultures hiPSC et rincer les cellules 2x avec 0,5 mM d’acide éthylènediaminetétraacétique (EDTA) dans 1x solution saline tamponnée au phosphate de Dulbecco (DPBS) sans Ca 2+ et Mg2+ (DPS [-/-], voir Tableau des matériaux).
- Retirer le surnageant et ajouter 2 mL d’EDTA 0,5 mM dans 1x DPBS (-/-) aux boîtes de Petri de 6 cm. Incuber les cellules à 37 °C pendant 3 min dans l’incubateur.
- Aspirer 1,5 mL de la solution d’EDTA et poursuivre l’incubation pendant 3 à 5 minutes.
- Tapotez doucement la vaisselle pour faciliter le détachement des cellules.
REMARQUE : Vérifier le détachement par évaluation visuelle. Les colonies de CSPhi devraient commencer à se détacher après 5 minutes, mais un temps d’incubation plus long peut être nécessaire avec des cultures de CSPhi confluentes plus élevées. Si les colonies ne se détachent pas, augmentez le temps d’incubation jusqu’à 10 min, mais ne dépassez pas ce délai. - Ajouter 5 ml de milieu d’entretien iPSC sans alimentation aux boîtes de Petri de 6 cm et remettre doucement les colonies en suspension 2x avec une pipette sérologique de 10 ml ou à l’aide d’embouts de pipettes de grand calibre. Transférer les cellules dans un tube de 15 mL.
NOTA : Les CSPh sont très sensibles aux contraintes mécaniques; Il convient donc d’éviter les resuspensions multiples. La suspension cellulaire finale doit être constituée de petits fragments de colonie (50-200 μm). - Aspirer le surnageant des boîtes de culture enrobées et préparer 4 ml de milieu d’entretien iPSC sans alimentation par boîte de 6 cm.
- Transférer les petits fragments de colonie dans les plats de culture fraîchement préparés dans des rapports fractionnés de 1:10 à 1:40, et cultiver à 37 °C et 5% de CO2 avec des changements quotidiens du milieu.
NOTE: Les petites colonies devraient se fixer dans les 1 ou 2 heures suivant le passage.
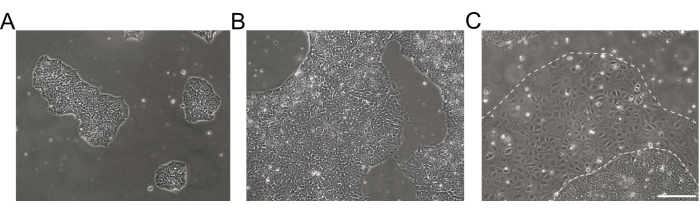
Figure 1 : Morphologie des cultures de cellules souches pluripotentes induites par l’homme. (A,B) Cultures de CSPhi de bonne qualité de différentes confluences montrant des colonies de CSPhi compactées avec une morphologie homogène et des bords définis. (C) culture hiPSC avec des grappes émergentes de cellules différenciées autour des bords de la colonie (ligne blanche pointillée). Barre d’échelle = 200 μm. Abréviation : hiPSC = cellule souche pluripotente induite par l’homme. Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Préculture des CSPh dans le système de bioréacteur de paillasse (jour-2)
REMARQUE: Commencez la préculture lorsque les cultures hiPSC atteignent une confluence entre 60% et 80%. Vérifiez les colonies hiPSC pour les zones différenciées. Pendant cette phase de préculture, les hiPSC sont maintenus dans un milieu de maintenance iPSC sans alimentation pendant 2 jours.
- Préparer le milieu de culture, composé d’un milieu d’entretien iPSC sans alimentation et d’un inhibiteur de ROCK de 10 μM (Y-27632).
- Aspirer complètement le milieu des CSPhi et rincer doucement les cellules avec 1x DPBS (-/-) deux fois.
- Ajouter 2,0 ml de solution trypsine-EDTA préchauffée aux boîtes de Petri de 6 cm et incuber les cellules pendant 3 min à 37 °C dans l’incubateur.
- Tapotez doucement les plats pour faciliter le détachement des cellules, ou incuber pendant 1-2 minutes de plus.
- Remettez les cellules en suspension dans 5 mL de milieu d’entretien iPSC sans alimentation + inhibiteur ROCK par boîte. Transférer la suspension cellulaire dans un tube de 15 mL ou 50 mL et mélanger doucement par pipetage pour assurer la singularisation de la cellule.
- Déterminer le nombre de cellules dans 100 μL de suspension cellulaire à l’aide d’un compteur de cellules automatisé comme décrit précédemment20. Transférer le volume correspondant pour 15 × 106 cellules par tube de bioréacteur dans un tube de 50 mL.
- Centrifuger les cellules à 300 × g pendant 3 min.
- Aspirer le surnageant et remettre les cellules en suspension dans 2 mL de milieu d’entretien iPSC sans alimentation + inhibiteur ROCK.
- Remplir chaque tube de 50 mL avec 18 mL de milieu (densité d’ensemencement cellulaire de 0,75 × 106 cellules/mL). Distribuer la suspension cellulaire dans les tubes du bioréacteur (20 mL par tube).
- Placez les tubes dans le système de bioréacteur. Réglez les paramètres de culture suivants : période de rotation de 2 s, vitesse de rotation de 60 tr/min, pas de pause d’agitation, 37 °C et 5 % de CO2 pour une durée illimitée20.
- Démarrez le programme de culture via l’écran du bioréacteur.
- Changez de média le lendemain. Laisser les agrégats se déposer dans les tubes du bioréacteur pendant ~5 min. Aspirez soigneusement le surnageant.
REMARQUE: Ne laissez pas les agrégats se déposer pendant plus de 10 minutes, car ils pourraient coller les uns aux autres et construire une suspension d’agrégats hétérogène. Il est recommandé de laisser ~5 mL de milieu de culture dans le tube du bioréacteur. - Ajouter 15 mL de milieu d’entretien iPSC frais sans alimentation sans inhibiteur de ROCK par tube, et poursuivre la culture dans le bioréacteur de paillasse pendant 24 h.
3. Différenciation des CSPhi en neurones précoces (jour 0)
- Préparer le milieu neurobasal (MNN) : 50 % DMEM/F-12 avec dipeptide de L-alanyl-L-glutamine stabilisé, 50 % de milieu neurobasal, 0,5x supplément sans sérum (50x), supplément 0,5x sans sérum basé sur la formulation N-1 de Bottenstein (100x), dipeptide de L-alanyl-L-glutamine stabilisé 0,5x, solution d’acides aminés non essentiels MEM 0,5x (100x), pyruvate de sodium 500 nM (100 mM), 50 nM 2-mercaptoéthanol (50 mM), solution d’insuline humaine 0,025% et 5 U/mL pénicilline-streptomycine.
REMARQUE: NBM doit être conservé à 4 ° C et peut être utilisé jusqu’à 2 semaines. - Commencez la différenciation neuronale en ajoutant de la doxycycline (DOX) aux cultures hiPSC. Pour cela, laissez les agrégats se déposer dans les tubes du bioréacteur. Aspirer soigneusement le surnageant des cellules, en laissant ~5 mL dans le tube, et ajouter 35 mL de milieu d’induction neurale (NIM) composé de NBM et de 2 μg/mL de DOX.
REMARQUE: Comme DOX est sensible à la lumière, il est recommandé d’éteindre la lumière pendant le travail. - Replacez les tubes dans le bioréacteur de paillasse et poursuivez la culture.
- Effectuez des changements de support tous les jours pendant 2 jours, comme décrit à l’étape 3.2.
NOTE: Après 4 jours en culture en suspension, les agrégats peuvent être dissociés et les neurones précoces cryoconservés ou directement replaqués pour la maturation terminale.
4. Cryoconservation des neurones précoces (jour 2)
REMARQUE: La cryoconservation n’est pas nécessaire et n’est pas critique pour le processus de différenciation, mais fortement recommandée car de grands stocks de neurones précoces peuvent être produits et stockés pour une maturation et des analyses ultérieures.
- Laissez les agrégats se déposer dans le tube du bioréacteur. Aspirer le surnageant comme décrit précédemment.
- Transférer les agrégats dans un tube stérile de 15 mL ou 50 mL et rincer doucement les granulats 2x avec 1x DPBS (-/-). Aspirez soigneusement le surnageant autant que possible sans déranger les agrégats.
- Ajouter 2 à 5 mL d’enzyme de dissociation cellulaire préchauffée, selon la taille de la pastille, et incuber les cellules pendant environ 10 minutes à 37 °C au bain-marie. Remettez doucement en suspension les agrégats sédimentés toutes les 2 minutes jusqu’à ce qu’ils se dissocient.
REMARQUE: Une suspension cellulaire presque homogène doit être obtenue après 7-10 min d’incubation. - Ajouter le triple volume de milieu NBM préchauffé et remettre les cellules en suspension avec précaution pour assurer la singularisation cellulaire.
- Déterminer le nombre de cellules et transférer le volume correspondant pour la cryoconservation dans un tube de 15 mL ou 50 mL.
REMARQUE : Une densité cellulaire de 5 à 10 × 10à 6 cellules/mL de milieu de congélation est recommandée. - Centrifuger les cellules à 300 × g pendant 3 min.
- Aspirer le surnageant et remettre doucement en suspension la pastille cellulaire dans le volume correspondant de milieu de congélation contenant 10% de diméthylsulfoxyde (DMSO).
- Aliquote la suspension cellulaire dans des flacons appropriés pour la cryoconservation (1 mL/flacon).
- Transférer immédiatement les flacons dans un récipient préréfrigéré à taux de congélation lent rempli de 2-propanol et placer le récipient à -80 °C pendant la nuit. Placez les flacons à -150 °C le lendemain pour un stockage à long terme.
REMARQUE: Le 2-Propanol liquide est très inflammable et peut causer des lésions oculaires au contact. Tenir à l’écart de la chaleur et porter des gants de protection ainsi que des lunettes.
5. Maturation des neurones dérivés de l’hiPSC dans des cultures monocouches
- Préparer des plats de culture enrobés de poly-L-ornithine / laminine pour la culture à long terme de neurones dérivés de l’hiPSC.
- Diluer la solution mère de poly-L-ornithine à 0,001 % dans 1x DPBS (-/-) et enrober la vaisselle pendant la nuit à 4 °C, ou pendant 4 h à 37 °C. Aspirer la solution de poly-L-ornithine et laver les plaques une fois avec 1x DPBS (-/-).
- Diluer la solution de laminine dans 1x DPBS (-/-) jusqu’à une concentration finale de 10 μg/mL et incuber les plats pendant une nuit à 4 °C, ou pendant 4 h à 37 °C.
REMARQUE: Un volume de 0,1 à 0,15 mL par cm 2 est recommandé pour les procédures de revêtement et de 0,2 mL par cm2 pour toutes les étapes de lavage respectives. D’autres substrats de revêtement peuvent être utilisés, mais les effets potentiels sur la fixation et la maturation des cellules doivent être évalués.
- Décongeler les cellules cryoconservées. Pour cela, placez le cryovial dans un bain-marie (réglé à 37 °C) et agitez-le pendant environ 1 min, jusqu’à ce qu’il reste un petit amas de suspension cellulaire gelée.
- Transférer délicatement la suspension cellulaire goutte à goutte dans un tube de 15 mL préparé avec 10 mL de MNB préchauffé. Rincer le cryovial avec 1 mL de NBM et transférer la suspension cellulaire dans le tube identique de 15 mL.
- Centrifuger les cellules à 300 × g pendant 3 min.
- Aspirer le surnageant et ajouter 1-2 mL de NIM complété par un inhibiteur de ROCK de 10 μM.
- Remettez soigneusement en suspension la pastille de cellule et déterminez les numéros de cellule.
- Aspirer la solution de laminine restante des boîtes de culture cellulaire enrobées et ensemencer les cellules à une densité d’ensemencement de 1 × 105 cellules/cm2 dans NIM complétée par un inhibiteur de ROCK de 10 μM.
- Basculez le milieu après 24 h en NIM sans inhibiteur de ROCK.
- Effectuer des changements moyens quotidiens pendant 4 jours.
- Après cette phase initiale d’induction NGN2, omettre le DOX et cultiver les cellules dans le MNN, avec des changements de demi-milieu 2x par semaine jusqu’à atteindre le stade de maturation souhaité.
REMARQUE: La co-culture de neurones iNGN2 avec des astrocytes est recommandée pour augmenter la survie, l’attachement, la maturité et l’activité électriquedes cellules 13,22.
6. Caractérisation des neurones dérivés de l’hiPSC
NOTE: La différenciation en dérivés neuronaux peut être évaluée par les techniques suivantes.
- Immunocytochimie et imagerie
- Aspirez le milieu des neurones dérivés de l’hiPSC et lavez les cellules une fois avec 1x DPBS avec Ca 2+ et Mg2+ (+/+).
REMARQUE: Pipeter soigneusement, de préférence sur les bords du puits, car les cellules peuvent se détacher très facilement de la surface. Un volume de 0,2 mL parcm2 est recommandé pour toutes les étapes de lavage afin d’assurer une couverture complète des cellules avec 1x DPBS (+/+). - Fixer les cellules neuronales à l’aide d’une solution de fixation contenant 4% de paraformaldéhyde dans du DPBS (+/+) pendant 15 min à température ambiante (RT). Un volume de 0,1 mL parcm2 est recommandé.
NOTE: Le paraformaldéhyde (4%) dans le DPBS est une solution dangereuse et irritante pour la peau avec une toxicité aiguë et une cancérogénicité potentielle. Tenir à l’écart de la chaleur, porter des gants et des lunettes de protection, éviter l’inhalation et utiliser la solution uniquement dans un environnement ventilé. - Rincer doucement les cellules 2x dans 1x DPBS (+/+) et procéder à la coloration.
REMARQUE: Les cellules fixes peuvent être stockées dans DPBS (+/+) à 4 °C jusqu’à 1 mois jusqu’à ce que le traitement soit ultérieur. - Aspirer la DPBS (+/+) à partir des échantillons. Perméabiliser les cellules et bloquer les sites de liaison non spécifiques avec 1x DPBS (+/+) contenant 1% BSA et 0,2% Triton-X-100 pendant 60 min à TA.
REMARQUE: Un volume de 0,2 mL par cm2 est recommandé. - Retirer le surnageant et incuber les cellules pendant une nuit à 4 °C, avec les anticorps primaires respectifs dilués dans un tampon de coloration (1x DPBS [+/+] contenant 1% de BSA) (voir le tableau des matériaux). Assurez-vous que les cellules sont complètement recouvertes par la solution.
REMARQUE: Un volume de 0,1-0,15 mL par cm2 est recommandé. - Aspirer le tampon de coloration et rincer les cellules 3x dans 1x DPBS (+/+).
- Diluer les anticorps secondaires correspondants (voir le tableau des matériaux) dans un tampon de coloration à une concentration finale de 1:1 000.
- Incuber les cellules dans une solution d’anticorps diluée pendant 1 h à TA dans l’obscurité. Assurez-vous que les cellules sont complètement recouvertes par la solution.
REMARQUE: Un volume de 0,1-0,15 mL par cm2 est recommandé. - Aspirer la solution d’anticorps secondaires et rincer les cellules 2x dans 1x DPBS (+/+).
- Contre-colorer les noyaux avec du 4',6-diamidino-2-phénylindole (DAPI), dilué dans du DPBS (+/+) pendant 5 min à TA.
REMARQUE: Un volume de travail de 0,1 à 0,15 mL par cm2 est recommandé. - Rincer les cellules 2x dans 1x DPBS (+/+).
- Conserver les cellules dans DPBS (+/+) à 4 °C jusqu’à l’imagerie.
REMARQUE: L’imagerie en fond clair convient pour suivre les changements morphologiques et la croissance des neurites. De plus, l’expression stéréotypée des marqueurs peut être évaluée par microscopie à fluorescence. Alors que les hiPSCs indifférenciés expriment OCT 3/4 et NANOG, la bêta-III-tubuline (TUBB3) et la protéine 2 associée aux microtubules (MAP2) peuvent servir de marqueurs neuronaux pour visualiser les neurites et les axones. Le réseau neuritique peut être évalué davantage en déterminant la longueur des neurites dans les images en fond clair ou fluorescentes.
- Aspirez le milieu des neurones dérivés de l’hiPSC et lavez les cellules une fois avec 1x DPBS avec Ca 2+ et Mg2+ (+/+).
- Analyses de l’expression génique par réaction en chaîne de la polymérase quantitative en temps réel (qPCR)
- Aspirer le milieu et rincer les cellules une fois dans 1x DPBS (-/-).
REMARQUE: Un volume de 0,2 mL par cm2 est recommandé. - Ajouter un tampon de lyse d’ARN froid et récolter les cellules en grattant.
- Isoler l’ARN à l’aide d’un kit à base de colonne approprié et déterminer les concentrations d’ARN par photospectrométrie UV.
- Générer de l’ADNc en utilisant 250 ng d’ARN total et un kit approprié pour la transcription inverse.
- Préparer des réactions qPCR de balise moléculaire contenant 2,5 ng d’ADNc en double. Utiliser un mélange principal approprié et les dosages d’amorcecorrespondants 20 (voir le tableau des matières).
- Exécutez la qPCR en appliquant 45 cycles avec un recuit d’apprêt à 60 °C pendant 20 s.
- Normaliser les niveaux relatifs d’expression génique à la moyenne des gènes d’entretien GAPDH, HPRT1 et GUSB. Appliquer la méthode ΔΔCt23, en utilisant des hiPSC indifférenciés comme étalonneur.
- Aspirer le milieu et rincer les cellules une fois dans 1x DPBS (-/-).
Résultats
Au cours des étapes initiales, les cultures adhérentes des CSPh sont détachées, singularisées et transférées en suspension (Figure 2). Les agrégats se forment en 24 heures et grossissent continuellement. Après 2 jours d’induction transgénique, les premiers neurones peuvent être cryoconservés pour des expériences ultérieures.
La prolifération persistante pendant les premiers jours en suspension entraîne une augmentation du nombre de cellules, atteignant son apogée après 2 jours d’induction (Figure 3A,B). Avec la prolifération, les agrégats commencent à croître. Par rapport au jour 0, le diamètre montre une augmentation de 50% au jour 2, et est presque doublé au jour 5 (Figure 3D). Bien qu’un diamètre croissant limite l’apport en nutriments à l’intérieur des agrégats, la viabilité des cellules n’est pas affectée au jour 2 ou au jour 5 de la différenciation (Figure 3C). Cependant, une culture prolongée de plus de 4 jours en suspension n’améliore pas davantage le rendement cellulaire, car les agrégats deviennent de plus en plus résistants à la singularisation enzymatique (Figure 3B).
Lors de la cryoconservation, les cellules du jour 2 sont décongelées et plaquées sur des boîtes enrobées de poly-L-ornithine (PLO)-laminine pour la maturation terminale. En général, les cellules se fixent très bien après la décongélation et commencent à étendre les neurites dès le début. Le profil d’expression génique temporel, ainsi que la coloration immunocytochimique des marqueurs neuronaux TUBB3 et MAP2, confirment l’identité des cellules neuronales (Figure 4A,B). En plus de l’augmentation des niveaux de TUBB3 et MAP2, les cultures neuronales sont enrichies en transcrits tau de protéines associées aux microtubules (MAPT), codant pour une protéine neuronale impliquée dans la stabilisation axonale, et montrent une baisse concomitante de l’expression du facteur de transcription régulateur de la pluripotence POU5F1. De plus, un réseau neuritique dense se forme dans la première semaine après la décongélation (Figure 4C). Ces changements morphologiques, en conjonction avec le profil transcriptionnel, suggèrent une maturation croissante des cultures neuronales.
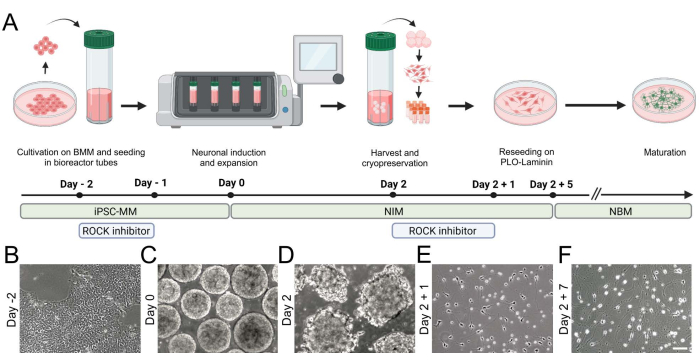
Figure 2 : Génération de neurones iNGN2 dérivés de l’hiPSC à l’aide d’un bioréacteur de paillasse. (A) Aperçu schématique du paradigme de différenciation, mettant en évidence les étapes clés de la culture cellulaire. (B-F) Les images en fond clair visualisent (B) les CSPhi et (C,D) la formation d’agrégats dans le bioréacteur de paillasse. (E,F) Les neurones iNGN2 étendent les neurites et subissent des changements morphologiques au cours de la différenciation. Barre d’échelle = 100 μm. Abréviations : BMM = matrice membranaire basale; iPSC-MM = support de maintenance iPSC; PLO = poly-L-ornithine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
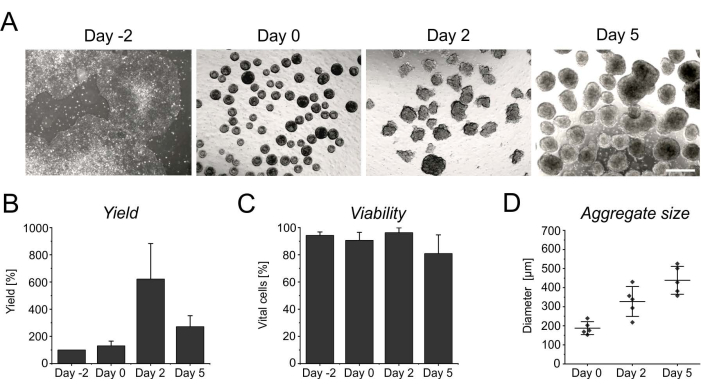
Figure 3 : Rendement cellulaire et viabilité pendant la formation et la croissance des agrégats. (A) Les images en fond clair montrent la formation d’agrégats dans les 2 jours suivant la culture dans le bioréacteur de paillasse, ainsi que l’augmentation de la taille des agrégats au fil du temps. Barre d’échelle = 500 μm. (B) Quantification du rendement cellulaire et (C) viabilité sur le cours de différenciation. Les diagrammes à barres représentent la moyenne + écart-type de quatre expériences indépendantes. (D) Le diamètre, indicatif de la taille des agrégats, a été déterminé de manière semi-automatique à l’aide du logiciel libre ImageJ (version 1.53). Tout d’abord, les images en fond clair ont été converties en images binaires, puis évaluées à l’aide de l’outil d’analyse des particules, en appliquant les paramètres suivants : taille > 2 500 μm 2, circularité 0,45-1, exclure les bords et inclure les trous. Les lignes horizontales indiquent la moyenne ± écart-type de cinq différenciations indépendantes. Les points simples représentent la moyenne des expériences de différenciation individuelles avec au moins 20 agrégats par point temporel. Veuillez cliquer ici pour voir une version agrandie de cette figure.
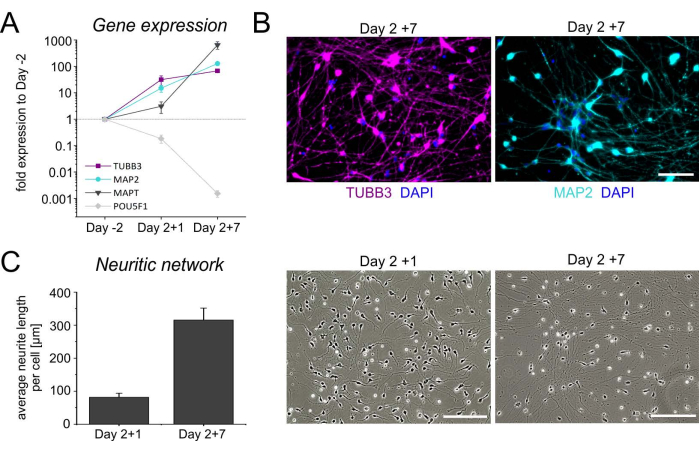
Figure 4 : Caractérisation des neurones iNGN2 dérivés de l’hiPSC. Profil d’expression génique temporel pour les gènes neuronaux TUBB3, MAP2 et MAPT, ainsi que le gène pluripotent associé aux cellules souches POU5F1. Les niveaux d’expression relative ont été normalisés aux gènes de référence GAPDH, HPRT1 et GUSB. Les CSPh indifférenciées (jour-2) ont été choisies comme étalonneur. Les symboles géométriques indiquent la moyenne ± écart-type de quatre différenciations indépendantes. (B) Images représentatives des neurones iNGN2 au jour 7 après la décongélation (jour 2 + 7), colorées pour la bêta-III-tubuline (TUBB3, magenta) et la protéine 2 associée aux microtubules (MAP2, cyan). Les noyaux cellulaires ont été contre-colorés avec du DAPI. Barres d’échelle = 100 μm. (C) Évaluation du réseau neuritique dans des images en fond clair de cultures neuronales après décongélation. La longueur totale des neurites a été déterminée dans une zone de 930,82 × 698,11μm2 à l’aide d’ImageJ (version 1.53). Après avoir ajusté la luminosité et le contraste, les images ont été converties en images 8 bits et les couleurs ont été inversées. Les neurites ont ensuite été squelettées, puis analysées à l’aide des plugins ImageJ « Skeletonize » et « Analyze Skeleton », respectivement. La longueur totale des neurites a été divisée par le nombre de somas dans la région d’intérêt pour obtenir la longueur moyenne des neurites / cellule. Les diagrammes à barres représentent la moyenne + écart-type de trois expériences indépendantes. Abréviations : DAPI = 4',6-diamidino-2-phénylindole. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Il a déjà été démontré que l’expression ectopique du facteur de transcription neuronal NGN2 accélère les premiers stades de la différenciation neuronale et induit l’engagement de la lignée neuronale dans les CSPhi dans la semaine suivant la culture12,13,20. Ce travail décrit un protocole détaillé pour la différenciation des hiPSCs BIONi010-C-13 génétiquement modifiés en neurones iNGN2 à l’aide d’un bioréacteur de paillasse.
Le bioréacteur CERO 3D est un bioréacteur à faible volume avec quatre emplacements pour tubes spécialisés, chacun d’une capacité maximale de 50 mL. Les niveaux de température et de CO2 sont contrôlés en permanence et les paramètres de culture (par exemple, la vitesse et le temps de rotation) sont régulés pour chaque tube, ce qui permet aux utilisateurs d’exécuter plusieurs approches de différenciation en parallèle. Les brise-ondes intégrés sur la paroi du tube intérieur permettent une perturbation du fluide avec une faible contrainte de cisaillement et maintiennent les agrégats 3D en suspension pendant la rotation contrôlée. L’accès limité aux nutriments, ainsi que les interactions 3D cellule-cellule et cellule-matrice extracellulaire (ECM) forcées, peuvent influencer de manière significative la différenciation cellulaire et le comportement24,25,26,27.
La culture de cellules sous forme d’agrégats 3D en suspension augmente ces interactions cellulaires, imitant ainsi plus étroitement l’environnement in vivo des tissus ou des organes28. La perturbation concomitante du milieu régule la taille des agrégats due au frottement mécanique, améliore les échanges gazeux et réduit le gradient des nutriments et des déchets dans le tube, tout en préservant les gradients physiologiques pertinents à l’intérieur des agrégats 29,30,31,32,33,34 . Bien que les changements de milieu soient effectués manuellement, le bioréacteur de paillasse réduit les coûts de main-d’œuvre et d’exploitation par rapport aux flacons de culture statique. Le bioréacteur offre également plusieurs avantages par rapport aux bioréacteurs à agitation entièrement automatisés, car la conception sans roue réduit la contrainte de cisaillement hydrodynamique à la surface de l’agrégat, améliorant ainsi non seulement la survie des cellules, mais limitant également les effets de la contrainte de cisaillement sur les CSPi sensibles, la différenciation cellulaire et la fonction35,36,37,38.
Les bioréacteurs à roues verticales, dans lesquels les cellules sont agitées par une grande roue verticale, ainsi que les bioréacteurs à bascule utilisant des sacs de culture gonflés attachés à une plate-forme motorisée, représentent des plates-formes de suspension 3D alternatives. Bien que excessivement testés pour la culture de CSPhi 17,18,19,39,40, on sait peu de choses sur leur applicabilité dans les approches de différenciation neuronale, et les rapports se limitent à l’expansion réussie des cellules précurseurs neurales de mammifères et d’humains dans les bioréacteurs à fustiger 41,42,43,44, avec un nombre rare d’études axées sur maturation44,45. En général, les plates-formes de suspension 3D automatisées offrent l’avantage de changements de support entièrement automatisés et contrôlés par ordinateur, réduisant ainsi les variations de manipulation et minimisant le risque de contamination. En outre, les paramètres nutritifs tels que le lactate et la concentration de glucose peuvent être surveillés en permanence. Cependant, la mise en place de ces systèmes nécessite souvent un investissement initial important, de l’espace de laboratoire et la formation de personnel qualifié. Le bioréacteur de paillasse représente une alternative compacte et facile à utiliser pour la culture de cellules en suspension, mais ne fournit pas de surveillance simultanée des nutriments.
Comme dans la majorité des protocoles utilisés pour la culture de cellules dans des plateformes de suspension 3D, la formation initiale d’agrégats représente une étape critique. Les hiPSCs sont cultivés sous forme de monocouches adhérentes et ensuite transférés sous forme de suspensions unicellulaires dans un environnement 3D. Pour minimiser la perte cellulaire à ce stade, plusieurs aspects doivent être pris en compte. La conception des tubes avec les brise-ondes intégrés signifie qu’un volume de culture minimal de 10 mL par tube est nécessaire pour assurer une perturbation optimale du milieu. Le volume critique ne doit donc pas être sous-coté à long terme. De plus, une densité de semis initiale de 7,5 millions de cellules par 10 mL de volume de culture et une vitesse de rotation de 60 tr/min sont fortement recommandées pour former des agrégats stables à court terme, bien que des adaptations puissent être nécessaires en fonction de la lignée cellulaire.
Après le transfert des CSPhi en suspension, des agrégats doivent être formés dans les 24 heures. Le premier changement moyen est critique, car les agrégats sont petits et peuvent ne pas se déposer correctement. Le temps de sédimentation des granulats peut être prolongé mais ne doit pas dépasser plus de 10 minutes, car les agrégats peuvent commencer à s’agglutiner et à croître de manière hétérogène. Pendant l’aspiration, un volume de culture minimal de 5 mL doit être laissé dans le tube et toute perturbation du milieu doit être évitée. Néanmoins, une perte de cellules et d’agrégats est à prévoir au cours des phases initiales. Le nombre de cellules indique un rendement cellulaire constant au cours des 2 premiers jours en culture, indiquant que la capacité proliférative élevée des CSPhi compense la perte cellulaire initiale. Une fois en suspension, la culture douce et la perturbation conduisent à des agrégats de taille homogène qui commencent à croître avec le temps.
La différenciation neuronale a été réalisée selon un protocole précédemment publié basé sur la surexpression de NGN2 induite par la doxycycline13, et les étapes individuelles ont été optimisées pour une culture 3D. Le facteur de transcription neuronale NGN2 est un moteur bien décrit dans la différenciation neuronale et accélère significativement l’induction neuronale11,13,46. Alors que la structuration conventionnelle avec des facteurs de croissance définis prend plusieurs semaines à 7,8,9, l’expression de NGN2 induit le destin des cellules neuronales en quelques jours. En plus du temps de culture raccourci, le protocole ne dépend ni de facteurs de croissance coûteux ni de matrices d’enrobage pour la culture initiale en suspension, ce qui réduit les coûts de culture supplémentaires.
De plus, une comparaison directe de la génération de neurones immatures induite par NGN2 dans des cultures adhérentes et en suspension a révélé une multiplication par 1,36 du nombre de cellules après 4 jours en culture à l’aide du bioréacteur de paillasse20, tandis que le volume de milieu requis pour la génération de 1 million de cellules devrait être réduit de moitié. L’utilisation du bioréacteur de paillasse pour la génération de neurones iNGN2 peut donc être bénéfique, car un plus grand nombre de cellules peut être produit avec un temps et un coût de manipulation réduits. Conformément aux rapports précédents, l’engagement de la lignée neuronale a été observé très tôt. Après 2 jours d’induction NGN2, une expression profonde de marqueurs neuronaux classiques tels que TUBB3, MAP2 et MAPT était évidente, soutenant l’applicabilité du bioréacteur de paillasse pour l’induction neuronale. En suivant ce protocole, environ 6,2 millions de neurones iNGN2 immatures peuvent être obtenus à partir de 1 million de CSPhi dans les 4 jours suivant la culture. Toutefois, la capacité de production peut varier d’un lot à l’autre et dépendre du volume de culture en suspension au moment de l’ensemencement.
Pour augmenter encore le rendement, le temps de culture a été prolongé de 3 jours supplémentaires; Cependant, bien que les agrégats soient devenus plus gros, ils sont également devenus très compacts et ne pouvaient pas être dissociés correctement pour la cryoconservation ou le replacage. Malgré les progrès réalisés dans les approches de culture 3D, les cultures neuronales 2D adhérentes sont toujours l’étalon-or pour les analyses fonctionnelles telles que les réseaux de microélectrodes ou l’imagerie calcique. La singularisation cellulaire est donc une condition préalable importante pour une monocouche neuronale et un réseau neuritique répartis de manière homogène. Un détachement mécanique plus fort des cellules ou des réactifs de dissociation agressifs peuvent être utilisés pour assurer la singularisation, mais avec des effets potentiels sur la viabilité cellulaire, la physiologie et la capacité de rattachement47,48. En ce qui concerne la nécessité de cellules individuelles pour des analyses et une maturation ultérieures, les agrégats ont été dissociés après 4 jours en suspension, et les neurones iNGN2 (jour 2) cryoconservés. La différenciation post-décongélation et la caractérisation des neurones iNGN2 ont confirmé une maturité croissante des cultures neuronales et la formation d’un réseau neuritique dense.
Le protocole fourni produit un nombre élevé de neurones iNGN2. Cependant, plusieurs limites doivent être prises en compte. Comme pour la plupart des plates-formes de culture en suspension, le transfert des CSPh d’une culture adhérente 2D à un environnement 3D est essentiel et souvent associé à une perte cellulaire profonde. D’autres pertes importantes de cellules et d’agrégats sont attendues lors des changements de milieux. Par rapport aux plates-formes automatisées, le temps de manipulation et le risque de contamination à l’aide du bioréacteur de paillasse sont augmentés, car les changements de milieu doivent être effectués manuellement. Outre le manque d’automatisation, le bioréacteur de paillasse n’offre pas la possibilité de surveiller les nutriments, et la capacité maximale de 50 ml par tube limite le potentiel de mise à l’échelle du protocole. Enfin, il est important de noter que, bien que NGN2 accélère l’engagement de la lignée neuronale, la durée de maturation terminale n’est pas raccourcie et nécessite tout de même une culture à long terme sur plusieurs semaines. Compte tenu de l’importance du soutien astrocytaire pour la fonction neuronale, l’attachement et la survie 49,50,51, les approches de co-culture devraient être envisagées et peuvent même être essentielles pour la culture à long terme.
En résumé, un protocole de différenciation 2D a été traduit avec succès dans un environnement 3D pour la génération rapide et reproductible de neurones iNGN2 en mettant en œuvre un bioréacteur de paillasse. Le protocole décrit produit de grandes quantités de neurones dérivés de l’iPSC de bonne qualité, qui peuvent servir de point de départ pour des tests de modèles complexes, des criblages de médicaments à haut débit et des tests de toxicité à grande échelle.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt concurrent.
Remerciements
Le projet EBiSC2 a reçu un financement de l’entreprise commune (JU) de l’initiative en matière de médicaments innovants 2 dans le cadre de la convention de subvention n° 821362. L’entreprise commune reçoit le soutien du programme de recherche et d’innovation Horizon 2020 de l’Union européenne et de l’EFPIA. Nous remercions Nadine J. Smandzich, Helene D. M. Hemmer, Johnn-Majd Balsters et Vanessa M. Nalewaja pour leur aide dans les analyses immunocytochimiques et d’expression génique, ainsi que Heike Arthen pour son excellent soutien technique. De plus, nous remercions Stephanie Bur pour la mise en place du programme de bioréacteurs. La figure 2A a été créée avec BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol 50 mM | Gibco | 11528926 | |
| 60 mm Nunclon Delta Surface | Nunc | 734-2040 | |
| Anti-beta III Tubulin antibody [TU-20] (dilution 1:1,000) | Abcam | ab7751 | |
| Applied Biosystems High-Capacity cDNA Reverse Transcription Kit | Thermo Fisher Scientific | 10400745 | |
| B-27 Supplement (50x) | Gibco | 11530536 | serum-free Supplement (50x) |
| BIONi010-C-13 hiPSC line | European Bank for induced pluripotent Stem Cell (EBiSC), BIONEER as Depositor | SAMEA103988285 | Bioneer is depositor in EBiSC and owner of the hiPSC line. |
| Biorender | Biorender.com | ||
| BSA Cell Culture grade | Thermo Fisher Scientific | 12330023 | |
| CERO 3D Incubator & Bioreactor | OLS OMNI Life Science | 2800000 | |
| CEROtubes Cell culture Tubes 50 mL | OLS OMNI Life Science | 2800005 | |
| Citavi 6 | Swiss Academic Software | ||
| CryoStor CS10 | Stemcell | 7930 | freezing medium containing 10% DMSO |
| Cytofix Fixation Solution | BD Biosciences | 554655 | fixation solution containing 4% paraformaldehyde |
| DAPI (NUCBLUE FIXED CELL STAIN) | Thermo Fisher Scientific | 12333553 | |
| Doxycycline hydrochloride (DOX) | Sigma-Aldrich | D3447 | |
| DPBS without Calcium and Magnesium (DPBS -/-) | Gibco | 14190250 | |
| DPBS with Calcium and Magnesium (DPBS +/+) | Gibco | 11580456 | |
| DMEM/F12 (-/-) | Gibco | 21331-020 | |
| DMEM/F-12, GlutaMAX Supplement | Gibco | 31331-028 | |
| EVOS XL Core Cell Imaging System | Thermo Fisher Scientific | ||
| Goat anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 555 | Thermo Fisher Scientific | A21424 | |
| GlutaMAX supplement | Gibco | 35050-038 | stabilized L-alanyl-L-glutamine dipeptid |
| ImageJ 1.51v | National Institute of Health | ||
| Insulin solution human | Sigma-Aldrich | I9278l | |
| Laminin | Merck | L2020 | |
| MACSQuant Analyzer | Miltenyi | ||
| MAP2 Monoclonal Antibody (dilution 1: 300) | Thermo Fisher Scientific | 13-1500 | |
| Matrigel Growth factor reduced, phenol-red free | Corning Life Science | 356231 | basement membrane matrix |
| MEM Non-Essential Amino Acids Solution (100x) | Gibco | 11140035 | |
| MicroAmp Optical Adhesive Film Kit | Thermo Fisher Scientific | 10095714 | |
| mTeSR1 | Stemcell | 85850 | feeder-free iPSC maintenance medium |
| N-2 Supplement (100x) | Gibco | 17502-048 | serum-free Supplement based on Bottenstein’s N-1 formulation |
| Neurobasal Medium | Gibco | 11570556 | |
| Nikon Eclipse TS2 | Nikon Instruments Europe B.V. | ||
| NucleoCounter-NC200 | ChemoMetec A/S | ||
| Origin 2021 | OriginLab | ||
| Penicillin-Streptomycin | Gibco | 11548876 | |
| Perm/Wash Buffer | BD Biosciences | 554723 | |
| Primer assay TUBB3(Hs00801390_s1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay GAPDH (Hs99999905_m1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay GUSB (Hs99999908_m1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay HPRT1 (Hs99999909_m1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay MAP2 (Hs00258900_m1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay MAPT (Hs00902194_m1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay POU5F1 (Hs04260367_gH) | Thermo Fisher Scientific | 11620099 | |
| Poly-L-ornithine 0.01% | Merck | P4957 | |
| RNeasy Plus Micro Kit (50)-Kit | Qiagen | 74034 | column-based RNA isolation kit |
| RLT Buffer | Qiagen | 79216 | cell lysis buffer |
| Sodium Pyruvat (100 mM) | Gibco | 12539059 | |
| StemPro Accutase | Gibco | 11599686 | cell dissociation enzyme |
| TAQMAN FAST ADVANCED MASTER MIX | Thermo Fisher Scientific | 11380912 | qPCR master mix |
| Triton-X-100 | Sigma-Aldrich | T8787 | |
| TrypLE Select Enzym | Gibco | 12563-011 | Trypsin-EDTA solution |
| UltraPure 0.5 M EDTA, pH 8.0 | Thermo Fisher Scientific | AM9260G | |
| Via1-Cassette | ChemoMetec A/S | 941-0012 | |
| Wide Bore Filtered Pipette Tips | Thermo Fisher Scientific | 10088880 | |
| X20 OPTICAL 96WELL FAST CLEAR REACTION PLATES | Thermo Fisher Scientific | 15206343 | |
| Y-27632 dihydrochloride, Rho kinase inhibitor (ROCK inhibitor) | Abcam | ab120129 |
Références
- Feigin, V. L., et al. The global burden of neurological disorders: translating evidence into policy. The Lancet. Neurology. 19 (3), 255-265 (2020).
- Olesen, J., Gustavsson, A., Svensson, M., Wittchen, H. -. U., Jönsson, B. The economic cost of brain disorders in Europe. European Journal of Neurology. 19 (1), 155-162 (2012).
- Preclinical Development: The Safety Hurdle Prior to Human Trials. American Pharmaceutical Review Available from: https://www.americanpharmaceuticalreview.com/Featured-Articles/187349-Preclinical-Development-The-Safety-Hurdle-Prior-to-Human-Trials (2016)
- van Norman, G. A. Phase II trials in drug development and adaptive trial design. JACC. Basic to Translational Science. 4 (3), 428-437 (2019).
- van Norman, G. A. Limitations of animal studies for predicting toxicity in clinical trials: part 2: potential alternatives to the use of animals in preclinical trials. JACC. Basic to Translational Science. 5 (4), 387-397 (2020).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Chambers, S. M., et al. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nature Biotechnology. 27 (3), 275-280 (2009).
- Shi, Y., Kirwan, P., Livesey, F. J. Directed differentiation of human pluripotent stem cells to cerebral cortex neurons and neural networks. Nature Protocols. 7 (10), 1836-1846 (2012).
- Reinhardt, P., et al. Derivation and expansion using only small molecules of human neural progenitors for neurodegenerative disease modeling. PloS One. 8 (3), 59252 (2013).
- Hulme, A. J., Maksour, S., St-Clair Glover, M., Miellet, S., Dottori, M. Making neurons, made easy: the use of Neurogenin-2 in neuronal differentiation. Stem Cell Reports. 17 (1), 14-34 (2022).
- Zhang, Y., et al. Rapid single-step induction of functional neurons from human pluripotent stem cells. Neuron. 78 (5), 785-798 (2013).
- Schmid, B., et al. Generation of two gene edited iPSC-lines carrying a DOX-inducible NGN2 expression cassette with and without GFP in the AAVS1 locus. Stem Cell Research. 52, 102240 (2021).
- Shih, P. -. Y., et al. Development of a fully human assay combining NGN2-inducible neurons co-cultured with iPSC-derived astrocytes amenable for electrophysiological studies. Stem Cell Research. 54, 102386 (2021).
- Chang, C. -. Y., et al. Induced pluripotent stem cell (iPSC)-based neurodegenerative disease models for phenotype recapitulation and drug screening. Molecules. 25 (8), 2000 (2000).
- Silva, M. C., Haggarty, S. J. Human pluripotent stem cell-derived models and drug screening in CNS precision medicine. Annals of the New York Academy of Sciences. 1471 (1), 18-56 (2020).
- Elanzew, A., Sommer, A., Pusch-Klein, A., Brüstle, O., Haupt, S. A reproducible and versatile system for the dynamic expansion of human pluripotent stem cells in suspension. Biotechnology Journal. 10 (10), 1589-1599 (2015).
- Davis, B. M., Loghin, E. R., Conway, K. R., Zhang, X. Automated closed-system expansion of pluripotent stem cell aggregates in a rocking-motion bioreactor. SLAS Technology. 23 (4), 364-373 (2018).
- Kropp, C., et al. Impact of feeding strategies on the scalable expansion of human pluripotent stem cells in single-use stirred tank bioreactors. Stem Cells Translational Medicine. 5 (10), 1289-1301 (2016).
- Borys, B. S., et al. Overcoming bioprocess bottlenecks in the large-scale expansion of high-quality hiPSC aggregates in vertical-wheel stirred suspension bioreactors. Stem Cell Research & Therapy. 12 (1), 55 (2021).
- Kwok, C. K., et al. Scalable expansion of iPSC and their derivatives across multiple lineages. Reproductive Toxicology. 112, 23-35 (2022).
- User Protocol for Human induced Pluripotent Stem Cells. EBiSC Available from: https://ebisc.org/docs/ebisc/EBiSC_User_Protocol_for_Human_induced_Pluripotent_Stem_Cells.pdf (2023)
- Neyrinck, K., et al. SOX9-induced generation of functional astrocytes supporting neuronal maturation in an all-human system. Stem Cell Reviews and Reports. 17 (5), 1855-1873 (2021).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Griffith, L. G., Swartz, M. A. Capturing complex 3D tissue physiology in vitro. Nature Reviews. Molecular Cell Biology. 7 (3), 211-224 (2006).
- Burdick, J. A., Vunjak-Novakovic, G. Engineered microenvironments for controlled stem cell differentiation. Tissue Engineering. Part A. 15 (2), 205-219 (2009).
- Baker, B. M., Chen, C. S. Deconstructing the third dimension: how 3D culture microenvironments alter cellular cues. Journal of Cell Science. 125, 3015-3024 (2012).
- Duval, K., et al. Modeling physiological events in 2D vs. 3D cell culture. Physiology. 32 (4), 266-277 (2017).
- Haycock, J. W. 3D cell culture: a review of current approaches and techniques. Methods in Molecular Biology. 695, 1-15 (2011).
- Sen, A., Kallos, M. S., Behie, L. A. Effects of hydrodynamics on cultures of mammalian neural stem cell aggregates in suspension bioreactors. Industrial and Engineering Chemical Research. 40 (23), 5350-5357 (2001).
- Gerecht-Nir, S., Cohen, S., Itskovitz-Eldor, J. Bioreactor cultivation enhances the efficiency of human embryoid body (hEB) formation and differentiation. Biotechnology and Bioengineering. 86 (5), 493-502 (2004).
- Zhao, F., et al. Effects of oxygen transport on 3-d human mesenchymal stem cell metabolic activity in perfusion and static cultures: experiments and mathematical model. Biotechnology Progress. 21 (4), 1269-1280 (2005).
- Cimetta, E., Figallo, E., Cannizzaro, C., Elvassore, N., Vunjak-Novakovic, G. Micro-bioreactor arrays for controlling cellular environments: design principles for human embryonic stem cell applications. Methods. 47 (2), 81-89 (2009).
- Kehoe, D. E., Jing, D., Lock, L. T., Tzanakakis, E. S. Scalable stirred-suspension bioreactor culture of human pluripotent stem cells. Tissue Engineering. Part A. 16 (2), 405-421 (2010).
- Sargent, C. Y., et al. Hydrodynamic modulation of embryonic stem cell differentiation by rotary orbital suspension culture. Biotechnology and Bioengineering. 105 (3), 611-626 (2010).
- Liu, N., Zang, R., Yang, S. -. T., Li, Y. Stem cell engineering in bioreactors for large-scale bioprocessing. Engineering in Life Sciences. 14 (1), 4-15 (2014).
- Wolfe, R. P., Leleux, J., Nerem, R. M., Ahsan, T. Effects of shear stress on germ lineage specification of embryonic stem cells. Integrative Biology: Quantitative Biosciences from Nano to Macro. 4 (10), 1263-1273 (2012).
- Wolfe, R. P., Ahsan, T. Shear stress during early embryonic stem cell differentiation promotes hematopoietic and endothelial phenotypes. Biotechnology and Bioengineering. 110 (4), 1231-1242 (2013).
- Shafa, M., et al. Impact of stirred suspension bioreactor culture on the differentiation of murine embryonic stem cells into cardiomyocytes. BMC Cell Biology. 12 (1), 53 (2011).
- Kwok, C. K., et al. Scalable stirred suspension culture for the generation of billions of human induced pluripotent stem cells using single-use bioreactors. Journal of Tissue Engineering and Regenerative Medicine. 12 (2), 1076-1087 (2018).
- Rodrigues, C. A. V., et al. Scalable culture of human induced pluripotent cells on microcarriers under xeno-free conditions using single-use vertical-wheel™ bioreactors. Journal of Chemical Technology & Biotechnology. 93 (12), 3597-3606 (2018).
- Gilbertson, J. A., Sen, A., Behie, L. A., Kallos, M. S. Scaled-up production of mammalian neural precursor cell aggregates in computer-controlled suspension bioreactors. Biotechnology and Bioengineering. 94 (4), 783-792 (2006).
- Baghbaderani, B. A., et al. Expansion of human neural precursor cells in large-scale bioreactors for the treatment of neurodegenerative disorders. Biotechnology Progress. 24 (4), 859-870 (2008).
- Baghbaderani, B. A., et al. Bioreactor expansion of human neural precursor cells in serum-free media retains neurogenic potential. Biotechnology and Bioengineering. 105 (4), 823-833 (2010).
- Serra, M., Brito, C., Costa, E. M., Sousa, M. F. Q., Alves, P. M. Integrating human stem cell expansion and neuronal differentiation in bioreactors. BMC Biotechnology. , 82 (2009).
- Zhao, S., et al. Generation of cortical neurons through large-scale expanding neuroepithelial stem cell from human pluripotent stem cells. Stem Cell Research & Therapy. 11 (1), 431 (2020).
- Pawlowski, M., et al. Inducible and deterministic forward programming of human pluripotent stem cells into neurons, skeletal myocytes, and oligodendrocytes. Stem Cell Reports. 8 (4), 803-812 (2017).
- Bajpai, R., Lesperance, J., Kim, M., Terskikh, A. V. Efficient propagation of single cells Accutase-dissociated human embryonic stem cells. Molecular Reproduction and Development. 75 (5), 818-827 (2008).
- Jager, L. D., et al. Effect of enzymatic and mechanical methods of dissociation on neural progenitor cells derived from induced pluripotent stem cells. Advances in Medical Sciences. 61 (1), 78-84 (2016).
- Allen, N. J. Astrocyte regulation of synaptic behavior. Annual Review of Cell and Developmental Biology. 30, 439-463 (2014).
- Verkhratsky, A., Nedergaard, M., Hertz, L. Why are astrocytes important. Neurochemical Research. 40 (2), 389-401 (2015).
- Jäkel, S., Dimou, L. Glial cells and their function in the adult brain: a journey through the history of their ablation. Frontiers in Cellular Neuroscience. 11, 24 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon