Method Article
Produzione di neuroni umani 2-inducibili in un bioreattore tridimensionale a sospensione
In questo articolo
Riepilogo
Questo articolo descrive un protocollo per la generazione di neuroni derivati da cellule staminali pluripotenti indotte umane in un bioreattore a sospensione 3D da banco.
Abstract
La derivazione di cellule del lignaggio neuronale da cellule staminali pluripotenti indotte umane (hiPSC) ha segnato una pietra miliare nella ricerca sul cervello. Fin dal loro primo avvento, i protocolli sono stati continuamente ottimizzati e sono ora ampiamente utilizzati nella ricerca e nello sviluppo di farmaci. Tuttavia, la lunghissima durata di questi protocolli convenzionali di differenziazione e maturazione e la crescente domanda di hiPSC di alta qualità e dei loro derivati neurali aumentano la necessità di adottare, ottimizzare e standardizzare questi protocolli per la produzione su larga scala. Questo lavoro presenta un protocollo rapido ed efficiente per la differenziazione di neurogenina 2 (iNGN2) geneticamente modificata, inducibile dalla doxiciclina che esprime hiPSC in neuroni utilizzando un bioreattore a sospensione tridimensionale (3D) da banco.
In breve, le sospensioni monocellulari di iNGN2-hiPSCs sono state autorizzate a formare aggregati entro 24 ore e l'impegno della linea neuronale è stato indotto dall'aggiunta di doxiciclina. Gli aggregati sono stati dissociati dopo 2 giorni di induzione e le cellule sono state crioconservate o riplaccate per la maturazione terminale. I neuroni iNGN2 generati hanno espresso marcatori neuronali classici nella fase iniziale e hanno formato reti neuritiche complesse entro 1 settimana dalla riplaccatura, indicando una crescente maturità delle colture neuronali. In sintesi, viene fornito un protocollo dettagliato passo-passo per la generazione rapida di neuroni derivati dall'hiPSC in un ambiente 3D che ha un grande potenziale come punto di partenza per la modellazione della malattia, screening fenotipici di farmaci ad alto rendimento e test di tossicità su larga scala.
Introduzione
I disturbi neurologici sono la principale causa di disabilità in tutto il mondo1. Una persona su sei ne è affetta e l'incidenza continua ad aumentare. L'onere finanziario associato alle società e ai loro sistemi sanitari è enorme. Una valutazione di 30 paesi europei nel 2010 ha stimato costi annuali di 800 miliardi di euro relativi ai disturbi mentali e neurologici2. Il crescente onere socioeconomico richiede strategie di trattamento efficaci e, sebbene la nostra comprensione della fisiopatologia della malattia sia aumentata notevolmente, la traduzione in clinica è spesso insufficiente. In generale, solo il 12% dei farmaci entra in studi clinici, di cui oltre l'80% fallisce durante le fasi successive, principalmente a causa di inefficacia o tossicità imprevista 3,4. Le ragioni sono varie, ma la limitata trasferibilità dalla sperimentazione animale nelle fasi precliniche alla sperimentazione umana è venuta sempre più alla ribalta5. I modelli cellulari e tissutali umani in vitro possono colmare il divario della traduzione interspecie e i progressi nella tecnologia delle cellule staminali pluripotenti indotte dall'uomo (hiPSC) possiedono un grande potenziale in questo senso. Le hiPSC sono ampiamente utilizzate nella ricerca di base e condividono caratteristiche essenziali con le cellule staminali embrionali (hESC), come una capacità di auto-rinnovamento quasi illimitata e la capacità di differenziarsi in tutti e tre gli strati germinali6, aggirando le preoccupazioni etiche associate alla distruzione degli embrioni.
La derivazione di cellule neuronali da hESC, e successivamente da hiPSC, ha segnato una pietra miliare nella ricerca sul cervello. I protocolli di differenziazione iniziale erano basati sull'applicazione di fattori di crescita che imitavano passaggi critici durante l'embriogenesi, la maggior parte dei quali coinvolgeva la doppia inibizione della SMAD in sospensione o in colture aderenti 7,8,9. I neuroni maturi sono stati generati con successo, ma diversi inconvenienti di questi protocolli ostacolano ancora il loro ampio uso nello sviluppo di farmaci, come la bassa resa e l'elevata variabilità da lotto a lotto dei neuroni generati, nonché l'ampio carico di lavoro legato ai lunghi tempi di coltura. I miglioramenti sono stati ottenuti grazie all'espressione forzata di fattori di trascrizione criticamente coinvolti nella neurogenesi e i membri della famiglia delle neurogenine (NGN), in particolare NGN2, sono stati identificati come driver efficaci10. L'espressione ectopica lentiviral-mediata di NGN2 nelle hiPSC ha accelerato significativamente le prime fasi della differenziazione neuronale e ha indotto il destino delle cellule neuronali entro solo 1 settimana11. La successiva maturazione terminale in co-colture con astrociti ha prodotto neuroni funzionali in elevata purezza e quantità con proprietà riproducibili. L'editing genetico diretto dal sito del locus safe-harbor del sito di integrazione del virus adeno-associato 1 (AAVS1) è stato quindi applicato per creare linee hiPSC con una cassetta di espressione stabile e inducibile dalla doxiciclina per NGN212,13, riducendo così al minimo gli effetti collaterali indesiderati della consegna lentivirale.
La differenziazione robusta ed efficiente dei neuroni della neurogenina 2 inducibile dalla doxiciclina 2 (iNGN2) ha un grande potenziale per screening fenotipici di farmaci ad alto rendimento e saggi di tossicità10,14,15; e progressi sostanziali nel bioprocessing sono stati fatti durante l'ultimo decennio implementando bioreattori per un'espansione e differenziazione cellulare scalabile16,17,18,19. Tuttavia, la maggior parte dei protocolli di differenziazione sono ottimizzati per le culture aderenti e la traduzione in un ambiente tridimensionale (3D) richiede spesso modifiche essenziali. Recentemente, l'uso di successo di un bioreattore 3D da banco con caratteristiche di stress di taglio ridotte è stato riportato per l'espansione di hiPSCs e per la differenziazione riproducibile in epatociti, cardiomiociti e neuroni20. Qui, viene fornito un protocollo dettagliato per la generazione e la caratterizzazione dei neuroni iNGN2 utilizzando l'identico bioreattore 3D da banco.
Protocollo
NOTA: Tutte le manipolazioni cellulari, così come i piatti di coltura e le preparazioni medie, devono essere eseguite in condizioni sterili. La cappa a flusso laminare deve essere pulita accuratamente prima dell'uso e dopo la lavorazione pulendo tutte le superfici con etanolo al 70%. Il protocollo descritto è ottimizzato per le differenziazioni neuronali in un incubatore e bioreattore 3D CERO (di seguito denominato bioreattore da banco). Questo bioreattore da banco offre quattro slot per tubi di bioreattori specializzati, ciascuno con una capacità massima di 50 ml. I livelli di temperatura e CO2 sono continuamente controllati e i parametri di coltivazione (ad esempio, velocità e tempo di rotazione) sono regolati per ogni tubo in modo indipendente. Tutte le fasi di aspirazione sono state eseguite utilizzando una pipetta di aspirazione e una pompa per vuoto, se non diversamente specificato.
1. Coltura ed espansione delle hiPSCs
NOTA: In questo protocollo viene utilizzata la linea NGN2 hiPSC inducibile dalla doxiciclina BIONi010-C-13. Il protocollo di espansione fornito qui è ottimizzato per piastre di Petri da 6 cm, ma è possibile utilizzare formati di coltura alternativi se si preferisce.
- Rivestire le piastre di Petri di 6 cm con matrice di membrana basale diluita nel mezzo di Eagle modificato (DMEM)/F12 (-/-) di Dulbecco a freddo ad una concentrazione finale di 0,083 mg/ml/10 cm2. Incubare i piatti rivestiti a 37 °C per almeno 30 minuti.
NOTA: Un protocollo dettagliato per la preparazione della soluzione di matrice della membrana basale può essere trovato nelle istruzioni del produttore. Le hiPSCs possono essere coltivate su matrici extracellulari alternative per l'espansione. - Disgelo di hiPSC crioconservate secondo il protocollo User Protocol21 della linea cellulare EBiSC in mezzo di mantenimento iPSC senza alimentatore + inibitore ROCK da 10 μM (Y-27632). Si raccomanda una densità di semina di 1 × 106 cellule vitali per piatto da 60 mm.
- Sostituire il terreno il giorno successivo con un mezzo di manutenzione iPSC privo di alimentatore senza inibitore ROCK ed eseguire modifiche giornaliere del fluido.
- Iniziare il passaggio quando le colture hiPSC raggiungono una confluenza del 60% -80%. Controllare le aree differenziate e pulire manualmente le colonie se l'area supera il 5%.
NOTA: Le hiPSC indifferenziate appaiono come cellule rotonde con un nucleolo prominente e meno citoplasma. Le colonie piatte e strettamente imballate si formano presto dopo lo scongelamento o il passaggio. Immagini esemplari in campo chiaro delle colture hiPSC sono mostrate nella Figura 1. Ulteriori informazioni sono fornite nel protocollo User Protocol21 della linea cellulare EBiSC. Per il passaggio, preparare piatti rivestiti con matrice di membrana basale e mezzo di manutenzione iPSC privo di alimentatore. - Aspirare il terreno da colture di hiPSC e sciacquare le cellule 2 volte con 0,5 mM di acido etilendiamminotetraacetico (EDTA) in 1x soluzione salina tamponata fosfato di Dulbecco (DPBS) senza Ca 2+ e Mg2+ (DPS [-/-], vedere Tabella dei materiali).
- Rimuovere il surnatante e aggiungere 2 mL di 0,5 mM EDTA in 1x DPBS (-/-) alle piastre di Petri da 6 cm. Incubare le cellule a 37 °C per 3 minuti nell'incubatore.
- Aspirare 1,5 mL della soluzione di EDTA e continuare l'incubazione per 3-5 minuti.
- Picchiettare delicatamente i piatti per facilitare il distacco delle cellule.
NOTA: Controllare il distacco mediante valutazione visiva. Le colonie di hiPSC dovrebbero iniziare a staccarsi dopo 5 minuti, ma potrebbe essere necessario un tempo di incubazione più lungo con colture hiPSC più confluenti. Se le colonie non si staccano, aumentare il tempo di incubazione fino a 10 minuti, ma non superare questo lasso di tempo. - Aggiungere 5 ml di terreno di mantenimento iPSC senza alimentatore alle piastre di Petri da 6 cm e risospendere delicatamente le colonie 2 volte con una pipetta sierologica da 10 mL o utilizzando punte per pipette a foro largo. Trasferire le cellule in una provetta da 15 ml.
NOTA: le hiPSC sono altamente sensibili alle sollecitazioni meccaniche; Pertanto, dovrebbero essere evitate risospensioni multiple. La sospensione cellulare finale dovrebbe consistere in piccoli frammenti di colonia (50-200 μm). - Aspirare il surnatante dalle piastre di coltura rivestite e preparare 4 ml di terreno di mantenimento iPSC senza alimentatore per piatto da 6 cm.
- Trasferire piccoli frammenti di colonia nei piatti di coltura appena preparati in rapporti di divisione da 1:10 a 1:40 e coltivare a 37 °C e 5% di CO2 con cambi giornalieri dei terreni.
NOTA: Le piccole colonie devono attaccarsi entro 1 o 2 ore dopo il passaggio.
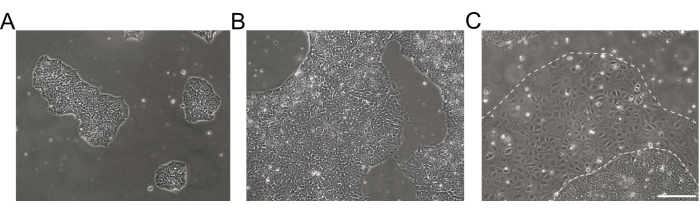
Figura 1: Morfologia di colture di cellule staminali pluripotenti indotte dall'uomo . (A,B) Colture hiPSC di buona qualità di diversa confluenza che mostrano colonie di hiPSC compattate con una morfologia omogenea e bordi definiti. (C) coltura hiPSC con gruppi emergenti di cellule differenziate attorno ai bordi della colonia (linea bianca tratteggiata). Barra di scala = 200 μm. Abbreviazione: hiPSC = cellula staminale pluripotente indotta dall'uomo. Fare clic qui per visualizzare una versione ingrandita di questa figura.
2. Precoltivazione di hiPSCs nel sistema di bioreattori da banco (giorno-2)
NOTA: Iniziare la precoltivazione quando le colture hiPSC raggiungono una confluenza tra il 60% e l'80%. Controllare le colonie hiPSC per aree differenziate. Durante questa fase di precoltivazione, le hiPSC vengono mantenute in un mezzo di mantenimento iPSC privo di alimentatore per 2 giorni.
- Preparare il terreno di coltura, costituito da terreno di mantenimento iPSC privo di alimentatore e inibitore ROCK da 10 μM (Y-27632).
- Aspirare completamente il mezzo dalle hiPSC e sciacquare delicatamente le cellule con 1x DPBS (-/-) due volte.
- Aggiungere 2,0 mL di soluzione di tripsina-EDTA preriscaldata alle piastre di Petri da 6 cm e incubare le cellule per 3 minuti a 37 °C nell'incubatore.
- Picchiettare delicatamente i piatti per facilitare il distacco delle cellule o incubare per 1-2 minuti in più.
- Risospendere le cellule in 5 ml di terreno di mantenimento iPSC senza alimentatore + inibitore ROCK per piatto. Trasferire la sospensione cellulare in un tubo da 15 mL o 50 mL e mescolare delicatamente mediante pipettaggio per garantire la singolarizzazione cellulare.
- Determinare il numero di celle in 100 μL di sospensione cellulare utilizzando un contatore automatico delle celle come descritto in precedenza20. Trasferire il volume corrispondente per 15 × 106 celle per tubo del bioreattore in un tubo da 50 ml.
- Centrifugare le cellule a 300 × g per 3 minuti.
- Aspirare il surnatante e risospendere le cellule in 2 mL di terreno di mantenimento iPSC privo di alimentatore + inibitore ROCK.
- Riempire ogni tubo da 50 mL con 18 mL di terreno (densità di semina cellulare di 0,75 × 106 celle/ml). Erogare la sospensione cellulare nei tubi del bioreattore (20 ml per provetta).
- Posizionare i tubi nel sistema del bioreattore. Impostare i seguenti parametri di coltivazione: 2 s di rotazione, velocità di rotazione di 60 giri/min, nessuna pausa di agitazione, 37 °C e 5% di CO2 per una durata illimitata20.
- Avviare il programma di coltivazione tramite il display del bioreattore.
- Cambia il supporto il giorno successivo. Lasciare che gli aggregati si depositino nei tubi del bioreattore per ~ 5 minuti. Aspirare con cura il surnatante.
NOTA: non lasciare che gli aggregati si depositino per più di 10 minuti, poiché potrebbero attaccarsi l'un l'altro e costruire una sospensione di aggregati eterogenei. Si consiglia di lasciare ~ 5 ml di terreno di coltura nel tubo del bioreattore. - Aggiungere 15 ml di terreno di mantenimento iPSC fresco senza alimentatore senza inibitore ROCK per provetta e continuare la coltivazione nel bioreattore da banco per 24 ore.
3. Differenziazione delle hiPSCs in neuroni precoci (giorno 0)
- Preparare il mezzo neurobasale (NBM): 50% DMEM/F-12 con L-alanil-L-glutammina dipeptide stabilizzato, 50% terreno neurobasale, 0,5x integratore senza siero (50x), 0,5x integratore senza siero basato sulla formulazione N-1 di Bottenstein (100x), 0,5x L-alanil-L-glutammina dipeptide stabilizzato, 0,5x MEM soluzione di aminoacidi non essenziali (100x), 500 nM piruvato di sodio (100 mM), 50 nM 2-mercaptoetanolo (50 mM), soluzione di insulina umana allo 0,025% e 5 U/mL di penicillina-streptomicina.
NOTA: NBM deve essere conservato a 4 °C e può essere utilizzato per un massimo di 2 settimane. - Iniziare la differenziazione neuronale aggiungendo doxiciclina (DOX) alle colture hiPSC. Per questo, lascia che gli aggregati si depositino nei tubi del bioreattore. Aspirare con attenzione il surnatante dalle cellule, lasciando ~ 5 ml nel tubo e aggiungere 35 ml di mezzo di induzione neurale (NIM) costituito da NBM e 2 μg / mL DOX.
NOTA: Poiché DOX è sensibile alla luce, si consiglia di spegnere la luce durante il lavoro. - Rimetti i tubi nel bioreattore da banco e continua la coltivazione.
- Eseguire le modifiche dei supporti ogni giorno per 2 giorni, come descritto nel passaggio 3.2.
NOTA: Dopo 4 giorni in coltura in sospensione, gli aggregati possono essere dissociati e i neuroni precoci crioconservati o direttamente riplaccati per la maturazione terminale.
4. Crioconservazione dei primi neuroni (giorno 2)
NOTA: La crioconservazione non è necessaria e non è critica per il processo di differenziazione, ma è altamente raccomandata in quanto grandi scorte di neuroni precoci possono essere prodotte e conservate per la successiva maturazione e analisi.
- Lasciare che gli aggregati si depositino nel tubo del bioreattore. Aspirare il surnatante come descritto in precedenza.
- Trasferire gli aggregati in un tubo sterile da 15 mL o 50 mL e sciacquare delicatamente gli aggregati 2x con 1x DPBS (-/-). Aspirare con attenzione il surnatante il più possibile senza disturbare gli aggregati.
- Aggiungere 2-5 ml di enzima di dissociazione cellulare preriscaldata, a seconda delle dimensioni del pellet, e incubare le cellule per circa 10 minuti a 37 °C a bagnomaria. Risospendere delicatamente gli aggregati sedimentati ogni 2 minuti fino a quando gli aggregati non si dissociano.
NOTA: Una sospensione cellulare quasi omogenea deve essere ottenuta dopo 7-10 minuti di incubazione. - Aggiungere il triplo volume del mezzo NBM preriscaldato e risospendere attentamente le celle per garantire la singolarizzazione cellulare.
- Determinare il numero di cellule e trasferire il volume corrispondente per la crioconservazione in una provetta da 15 ml o 50 ml.
NOTA: Si raccomanda una densità cellulare di 5-10 × 106 cellule/ml di mezzo di congelamento. - Centrifugare le cellule a 300 × g per 3 minuti.
- Aspirare il surnatante e risospendere delicatamente il pellet cellulare nel volume corrispondente di mezzo di congelamento contenente il 10% di dimetilsolfossido (DMSO).
- Aliquotare la sospensione cellulare in flaconcini idonei alla crioconservazione (1 mL/flaconcino).
- Trasferire immediatamente i flaconcini in un contenitore di congelamento prerefrigerato a bassa velocità riempito con 2-propanolo e porre il contenitore a -80 °C durante la notte. Introdurre i flaconcini a -150 °C il giorno successivo per la conservazione a lungo termine.
NOTA: Il liquido 2-propanolo è altamente infiammabile e può causare danni agli occhi al contatto. Tenere lontano da fonti di calore e indossare guanti protettivi e occhiali.
5. Maturazione di neuroni derivati da hiPSC in colture monostrato
- Preparare piatti di coltura rivestiti di poli-L-ornitina / laminina per la coltivazione a lungo termine di neuroni derivati da hiPSC.
- Diluire la soluzione madre di poli-L-ornitina allo 0,001% in 1x DPBS (-/-) e ricoprire le stoviglie per una notte a 4 °C o per 4 ore a 37 °C. Aspirare la soluzione di poli-L-ornitina e lavare le piastre una volta con 1x DPBS (-/-).
- Diluire la soluzione di laminina in 1x DPBS (-/-) fino ad una concentrazione finale di 10 μg/mL e incubare i piatti per una notte a 4 °C o per 4 ore a 37 °C.
NOTA: Si raccomanda un volume di 0,1-0,15 ml per cm 2 per le procedure di rivestimento e 0,2 ml per cm2 per tutte le rispettive fasi di lavaggio. È possibile utilizzare substrati di rivestimento alternativi, ma devono essere valutati i potenziali effetti sull'attaccamento e sulla maturazione delle cellule.
- Scongelare le cellule crioconservate. Per questo, posizionare il crioviale a bagnomaria (impostato a 37 ° C) e ruotarlo per circa 1 minuto, fino a quando non rimane un piccolo grumo di sospensione cellulare congelata.
- Trasferire con cautela la sospensione cellulare goccia a goccia su un tubo da 15 mL preparato con 10 mL di NBM preriscaldato. Risciacquare il crioviale con 1 mL di NBM e trasferire la sospensione cellulare nello stesso tubo da 15 mL.
- Centrifugare le cellule a 300 × g per 3 minuti.
- Aspirare il surnatante e aggiungere 1-2 ml di NIM integrato con inibitore ROCK da 10 μM.
- Risospendere attentamente il pellet cellulare e determinare il numero di celle.
- Aspirare la soluzione di laminina rimanente dalle piastre di coltura cellulare rivestite e seminare le cellule ad una densità di semina di 1 × 105 cellule / cm2 in NIM integrato con inibitore ROCK 10 μM.
- Commutare il mezzo dopo 24 ore su NIM senza inibitore ROCK.
- Eseguire cambi medi giornalieri per 4 giorni.
- Dopo questa fase iniziale di induzione di NGN2, omettere il DOX e coltivare le cellule in NBM, con cambi di semimedia 2 volte a settimana fino a raggiungere lo stadio di maturazione desiderato.
NOTA: Si raccomanda la co-coltura dei neuroni iNGN2 con gli astrociti per aumentare la sopravvivenza, l'attaccamento, la maturità e l'attività elettrica delle cellule13,22.
6. Caratterizzazione di neuroni derivati da hiPSC
NOTA: La differenziazione in derivati neuronali può essere valutata con le seguenti tecniche.
- Immunocitochimica e imaging
- Aspirare il mezzo dai neuroni derivati dall'hiPSC e lavare le cellule una volta con 1x DPBS con Ca 2+ e Mg2+ (+/+).
NOTA: Pipettare con attenzione, preferibilmente sui bordi del pozzetto, poiché le celle potrebbero staccarsi molto facilmente dalla superficie. Si consiglia un volume di 0,2 ml per cm2 per tutte le fasi di lavaggio per garantire una copertura completa delle celle con 1x DPBS (+/+). - Fissare le cellule neuronali utilizzando una soluzione di fissazione contenente il 4% di paraformaldeide in DPBS (+/+) per 15 minuti a temperatura ambiente (RT). Si raccomanda un volume di 0,1 ml per cm2 .
NOTA: La paraformaldeide (4%) nella DPBS è una soluzione pericolosa e irritante per la pelle con tossicità acuta e potenziale cancerogenicità. Tenere lontano da fonti di calore, indossare guanti e occhiali protettivi, evitare l'inalazione e utilizzare la soluzione solo in un ambiente ventilato. - Risciacquare delicatamente le celle 2x in 1x DPBS (+/+) e procedere con la colorazione.
NOTA: Le celle fisse possono essere conservate in DPBS (+/+) a 4 °C per un massimo di 1 mese fino a ulteriore elaborazione. - Aspirare il DPBS (+/+) dai campioni. Permeabilizzare le cellule e bloccare i siti di legame non specifici con 1x DPBS (+/+) contenente 1% BSA e 0,2% Triton-X-100 per 60 minuti a RT.
NOTA: Si raccomanda un volume di 0,2 ml per cm2 . - Rimuovere il surnatante e incubare le cellule per una notte a 4 °C, con i rispettivi anticorpi primari diluiti in tampone colorante (1x DPBS [+/+] contenente 1% BSA) (vedere Tabella dei materiali). Assicurarsi che le cellule siano completamente coperte dalla soluzione.
NOTA: Si raccomanda un volume di 0,1-0,15 ml per cm2 . - Aspirare il tampone colorante e risciacquare le celle 3x in 1x DPBS (+/+).
- Diluire i corrispondenti anticorpi secondari (vedere Tabella dei materiali) nel tampone colorante ad una concentrazione finale di 1:1.000.
- Incubare le cellule in soluzione anticorpale diluita per 1 ora a RT al buio. Assicurarsi che le cellule siano completamente coperte dalla soluzione.
NOTA: Si raccomanda un volume di 0,1-0,15 ml per cm2 . - Aspirare la soluzione anticorpale secondaria e sciacquare le cellule 2x in 1x DPBS (+/+).
- Controcolorare i nuclei con 4',6-diamidino-2-fenilindolo (DAPI), diluito in DPBS (+/+) per 5 minuti a RT.
NOTA: Si raccomanda un volume di lavoro di 0,1-0,15 ml per cm2 . - Risciacquare le celle 2x in 1x DPBS (+/+).
- Conservare le celle in DPBS (+/+) a 4 °C fino all'imaging.
NOTA: L'imaging in campo chiaro è adatto per tracciare i cambiamenti morfologici e la crescita dei neuriti. Inoltre, l'espressione stereotipata dei marcatori può essere valutata mediante microscopia a fluorescenza. Mentre le hiPSC indifferenziate esprimono OCT 3/4 e NANOG, la beta-III-tubulina (TUBB3) e la proteina 2 associata ai microtubuli (MAP2) possono servire come marcatori neuronali per la visualizzazione di neuriti e assoni. La rete neuritica può essere ulteriormente valutata determinando la lunghezza del neurite in immagini in campo chiaro o fluorescenti.
- Aspirare il mezzo dai neuroni derivati dall'hiPSC e lavare le cellule una volta con 1x DPBS con Ca 2+ e Mg2+ (+/+).
- Analisi dell'espressione genica mediante reazione a catena quantitativa della polimerasi in tempo reale (qPCR)
- Aspirare il mezzo e risciacquare le cellule una volta in 1x DPBS (-/-).
NOTA: Si raccomanda un volume di 0,2 ml per cm2 . - Aggiungere tampone di lisi RNA freddo e raccogliere le cellule grattando.
- Isolare l'RNA utilizzando un kit adatto basato su colonne e determinare le concentrazioni di RNA mediante fotospettrometria UV.
- Generare cDNA utilizzando 250 ng di RNA totale e un kit adatto per la trascrizione inversa.
- Preparare reazioni molecolari beacon qPCR contenenti 2,5 ng di cDNA in duplicato. Utilizzare una miscela master appropriata e i corrispondenti saggi di primer20 (vedi Tabella dei materiali).
- Eseguire la qPCR applicando 45 cicli con ricottura del primer a 60 °C per 20 s.
- Normalizzare i livelli di espressione genica relativa alla media dei geni housekeeping GAPDH, HPRT1 e GUSB. Applicare il metodo ΔΔCt23, utilizzando hiPSC indifferenziate come calibratore.
- Aspirare il mezzo e risciacquare le cellule una volta in 1x DPBS (-/-).
Risultati
Durante le fasi iniziali, le colture aderenti di hiPSC vengono staccate, singolarizzate, e trasferite in sospensione (Figura 2). Gli aggregati si formano entro 24 ore e crescono continuamente di dimensioni. Dopo 2 giorni di induzione del transgene, i primi neuroni possono essere crioconservati per esperimenti successivi.
La proliferazione persistente durante i primi giorni in sospensione produce un aumento del numero di cellule, raggiungendo il suo picco dopo 2 giorni di induzione (Figura 3A,B). Insieme alla proliferazione, gli aggregati iniziano a crescere. Rispetto al giorno 0, il diametro mostra un aumento del 50% al giorno 2 ed è quasi raddoppiato al giorno 5 (Figura 3D). Sebbene un diametro crescente limiti l'apporto di nutrienti all'interno degli aggregati, la vitalità delle cellule non è influenzata al giorno 2 o al giorno 5 della differenziazione (Figura 3C). Tuttavia, una coltivazione prolungata per più di 4 giorni in sospensione non migliora ulteriormente la resa cellulare, poiché gli aggregati diventano sempre più resistenti alla singolarizzazione enzimatica (Figura 3B).
Dopo la crioconservazione, le cellule del giorno 2 vengono scongelate e placcate su piatti rivestiti di poli-L-ornitina (PLO)-laminina per la maturazione terminale. In generale, le cellule si attaccano molto bene dopo lo scongelamento e iniziano presto ad estendere i neuriti. Il profilo di espressione genica temporale, così come la colorazione immunocitochimica per i marcatori neuronali TUBB3 e MAP2, conferma l'identità delle cellule neuronali (Figura 4A,B). Oltre all'aumento dei livelli di TUBB3 e MAP2, le colture neuronali sono arricchite in trascritti tau della proteina associati ai microtubuli (MAPT), che codificano per una proteina neuronale coinvolta nella stabilizzazione degli assoni, e mostrano un concomitante declino nell'espressione del fattore di trascrizione che regola la pluripotenza POU5F1. Inoltre, una fitta rete neuritica si forma entro la prima settimana dopo lo scongelamento (Figura 4C). Questi cambiamenti morfologici, in combinazione con il profilo trascrizionale, suggeriscono una crescente maturazione delle colture neuronali.
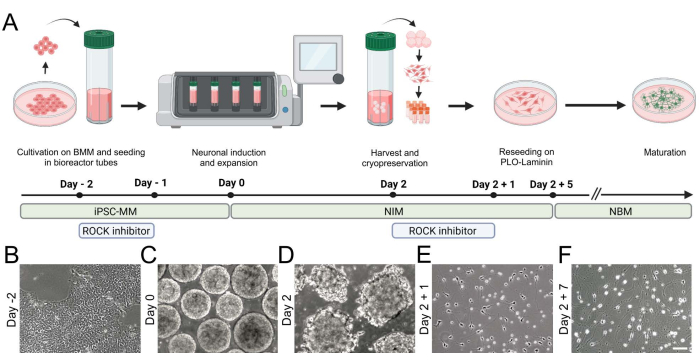
Figura 2: Generazione di neuroni iNGN2 derivati da hiPSC utilizzando un bioreattore da banco. (A) Panoramica schematica del paradigma di differenziazione, evidenziando le fasi chiave della coltura cellulare. (B-F) Le immagini in campo chiaro visualizzano (B) le hiPSC e (C,D) la formazione di aggregati nel bioreattore da banco. (E,F) I neuroni iNGN2 estendono i neuriti e subiscono cambiamenti morfologici durante la differenziazione. Barra di scala = 100 μm. Abbreviazioni: BMM = matrice di membrana basale; iPSC-MM = mezzo di manutenzione iPSC; PLO = poli-L-ornitina. Fare clic qui per visualizzare una versione ingrandita di questa figura.
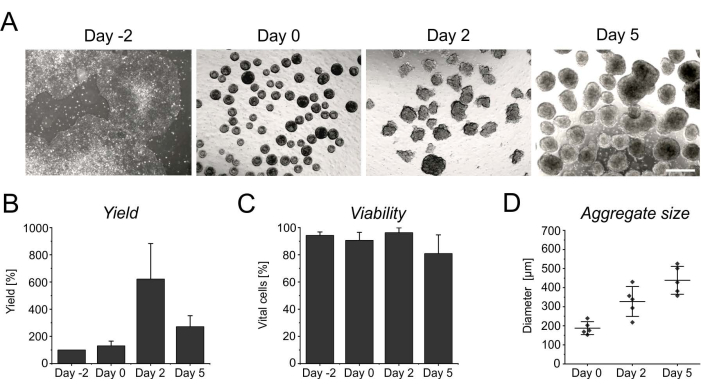
Figura 3: Resa cellulare e vitalità durante la formazione e la crescita degli aggregati. (A) Le immagini in campo chiaro mostrano la formazione di aggregati entro 2 giorni dalla coltura nel bioreattore da banco, nonché l'aumento delle dimensioni dell'aggregato nel tempo. Barra di scala = 500 μm. (B) Quantificazione della resa cellulare e (C) vitalità nel corso del differenziamento. I grafici a barre rappresentano la media + SD di quattro esperimenti indipendenti. (D) Il diametro, indicativo della dimensione degli aggregati, è stato determinato in modo semi-automatico utilizzando il software open source ImageJ (versione 1.53). In primo luogo, le immagini in campo chiaro sono state convertite in immagini binarie e quindi ulteriormente valutate utilizzando lo strumento di analisi delle particelle, applicando i seguenti parametri: dimensione > 2.500 μm 2, circolarità 0,45-1, escludere i bordi e includere i fori. Le linee orizzontali indicano la media ± SD di cinque differenziazioni indipendenti. I singoli punti rappresentano la media degli esperimenti di differenziazione individuale con almeno 20 aggregati per punto temporale. Fare clic qui per visualizzare una versione ingrandita di questa figura.
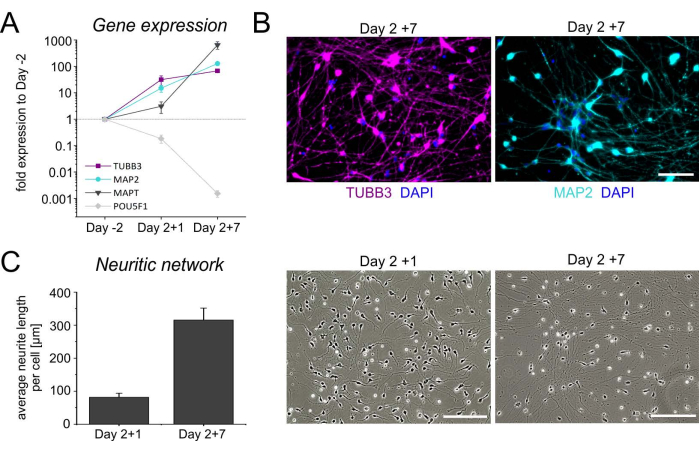
Figura 4: Caratterizzazione dei neuroni iNGN2 derivati da hiPSC. Profilo di espressione genica temporale per i geni neuronali TUBB3, MAP2 e MAPT, nonché per il gene associato alle cellule staminali pluripotenti POU5F1. I livelli di espressione relativa sono stati normalizzati ai geni di riferimento GAPDH, HPRT1 e GUSB. Le hiPSC indifferenziate (giorno-2) sono state scelte come calibratore. I simboli geometrici indicano la media ± SD di quattro differenziazioni indipendenti. (B) Immagini rappresentative dei neuroni iNGN2 al giorno 7 dopo lo scongelamento (giorno-2 + 7), colorate per beta-III-tubulina (TUBB3, magenta) e proteina 2 associata ai microtubuli (MAP2, ciano). I nuclei cellulari sono stati controcolorati con DAPI. Barre di scala = 100 μm. (C) Valutazione della rete neuritica in immagini in campo chiaro di colture neuronali dopo lo scongelamento. La lunghezza totale dei neuriti è stata determinata in un'area di 930,82 × 698,11 μm2 utilizzando ImageJ (versione 1.53). Dopo aver regolato la luminosità e il contrasto, le immagini sono state convertite in immagini a 8 bit e i colori sono stati invertiti. I neuriti sono stati successivamente scheletrati e quindi analizzati utilizzando rispettivamente i plugin ImageJ 'Skeletonize' e 'Analyze Skeleton'. La lunghezza totale dei neuriti è stata divisa per il numero di somi nella regione di interesse per ottenere la lunghezza media dei neuriti/cellula. I grafici a barre rappresentano la media + SD di tre esperimenti indipendenti. Abbreviazioni: DAPI = 4',6-diamidino-2-fenilindolo. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Discussione
L'espressione ectopica del fattore di trascrizione neuronale NGN2 ha precedentemente dimostrato di accelerare le prime fasi della differenziazione neuronale e indurre l'impegno della linea neuronale nelle hiPSC entro 1 settimana dalla coltivazione12,13,20. Questo lavoro descrive un protocollo dettagliato per la differenziazione di bioni010-C-13 hiPSCs geneticamente modificati in neuroni iNGN2 utilizzando un bioreattore da banco.
Il bioreattore 3D CERO è un bioreattore a basso volume con quattro slot per tubi specializzati, ciascuno con una capacità massima di 50 ml. I livelli di temperatura e CO2 sono continuamente controllati e i parametri di coltivazione (ad esempio, velocità e tempo di rotazione) sono regolati per ogni tubo, consentendo agli utenti di eseguire diversi approcci di differenziazione in parallelo. Gli interruttori d'onda integrati sulla parete del tubo interno consentono una perturbazione del fluido con un basso sforzo di taglio e mantengono gli aggregati 3D in sospensione durante la rotazione controllata. L'accesso limitato ai nutrienti, così come le interazioni forzate cellula-cellula 3D e cellula-matrice extracellulare (ECM), possono influenzare significativamente la differenziazione cellulare e il comportamento24,25,26,27.
La coltura di cellule come aggregati 3D in sospensione aumenta queste interazioni cellulari, imitando così più da vicino l'ambiente in vivo di tessuti o organi28. La perturbazione concomitante del mezzo regola la dimensione dell'aggregato a causa dell'attrito meccanico, migliora lo scambio di gas e riduce il gradiente di nutrienti e rifiuti nel tubo, preservando i gradienti fisiologici rilevanti all'interno degli aggregati 29,30,31,32,33,34. Sebbene le modifiche del mezzo vengano eseguite manualmente, il bioreattore da banco riduce i costi di manodopera e operativi rispetto ai palloni di coltura statici. Il bioreattore offre anche diversi vantaggi rispetto ai bioreattori completamente automatizzati con serbatoio di agitazione, poiché il design senza girante riduce lo sforzo di taglio idrodinamico sulla superficie aggregata, migliorando così non solo la sopravvivenza cellulare, ma anche limitando gli effetti dello stress di taglio sulle iPSC sensibili, sulla differenziazione cellulare e sulla funzione35,36,37,38.
I bioreattori a ruota verticale, in cui le cellule sono agitate da una grande girante verticale, così come i bioreattori a movimento oscillante che utilizzano sacchetti di coltura gonfiati attaccati a una piattaforma motorizzata, rappresentano piattaforme di sospensione 3D alternative. Sebbene eccessivamente testati per la coltivazione di hiPSCs 17,18,19,39,40, si sa poco sulla loro applicabilità negli approcci di differenziazione neuronale e i rapporti sono limitati all'espansione riuscita di cellule precursori neurali di mammiferi e umani nei bioreattori stir-tank 41,42,43,44, con un raro numero di studi incentrati su maturazione44,45. In generale, le piattaforme di sospensione 3D automatizzate offrono il vantaggio di cambi di mezzo completamente automatizzati e controllati da computer, riducendo così le variazioni nella manipolazione e minimizzando il rischio di contaminazione. Inoltre, i parametri nutrizionali come la concentrazione di lattato e glucosio possono essere monitorati continuamente. Tuttavia, la creazione di questi sistemi richiede spesso un investimento iniziale significativo, spazi di laboratorio e la formazione di personale qualificato. Il bioreattore da banco rappresenta un'alternativa compatta e facile da usare per la coltura di cellule in sospensione, ma non fornisce un monitoraggio simultaneo dei nutrienti.
Come nella maggior parte dei protocolli utilizzati per la coltivazione di cellule in piattaforme di sospensione 3D, la formazione iniziale di aggregati rappresenta un passaggio critico. Le hiPSC vengono coltivate come monostrati aderenti e successivamente trasferite come sospensioni a cella singola in un ambiente 3D. Per ridurre al minimo la perdita di cellule in quella fase, è necessario considerare diversi aspetti. Il design dei tubi con gli interruttori d'onda integrati significa che è necessario un volume di coltura minimo di 10 ml per tubo per garantire una perturbazione ottimale del fluido. Il volume critico non dovrebbe quindi essere sottovalutato a lungo termine. Inoltre, una densità di semina iniziale di 7,5 milioni di cellule per 10 ml di volume di coltura e una velocità di rotazione di 60 rpm sono altamente raccomandate per formare aggregati stabili a breve termine, anche se potrebbero essere necessari adattamenti a seconda della linea cellulare.
Dopo aver trasferito le hiPSC in sospensione, gli aggregati dovrebbero formarsi entro 24 ore. Il primo cambiamento del mezzo è critico, poiché gli aggregati sono piccoli e potrebbero non stabilizzarsi correttamente. Il tempo per la sedimentazione degli aggregati può essere prolungato ma non deve superare i 10 minuti, poiché gli aggregati possono iniziare ad aggregarsi e crescere in modo eterogeneo. Durante l'aspirazione, un volume di coltura minimo di 5 ml deve essere lasciato nel tubo e qualsiasi perturbazione del mezzo deve essere evitata. Tuttavia, è prevedibile una perdita di cellule e aggregati durante le fasi iniziali. La conta cellulare indica una resa cellulare costante nei primi 2 giorni di coltura, indicando che l'elevata capacità proliferativa delle hiPSC compensa la perdita cellulare iniziale. Una volta in sospensione, la coltivazione delicata e la perturbazione portano ad aggregati di dimensioni omogenee che iniziano a crescere nel tempo.
La differenziazione neuronale è stata eseguita secondo un protocollo precedentemente pubblicato basato sulla sovraespressione13 di NGN2 indotta dalla doxiciclina e le singole fasi sono state ottimizzate per una coltivazione 3D. Il fattore di trascrizione neuronale NGN2 è un driver ben descritto nella differenziazione neuronale e accelera significativamente l'induzione neuronale11,13,46. Mentre il pattern convenzionale con fattori di crescita definiti richiede da diverse settimane a mesi 7,8,9, l'espressione di NGN2 induce il destino delle cellule neuronali in pochi giorni. Oltre al tempo di coltivazione ridotto, il protocollo non dipende né da costosi fattori di crescita né da matrici di rivestimento per la coltura di sospensione iniziale, riducendo così ulteriormente i costi di coltivazione.
Inoltre, un confronto diretto della generazione guidata da NGN2 di neuroni immaturi in colture aderenti e in sospensione ha rivelato un aumento di 1,36 volte delle cellule dopo 4 giorni in coltura utilizzando il bioreattore da banco20, mentre il volume di mezzo richiesto per la generazione di 1 milione di cellule dovrebbe essere dimezzato. L'impiego del bioreattore da banco per la generazione di neuroni iNGN2 può quindi essere utile, poiché è possibile produrre un numero maggiore di cellule con tempi e costi di gestione inferiori. In linea con i rapporti precedenti, l'impegno della linea neuronale è stato osservato precocemente. Dopo 2 giorni di induzione di NGN2, era evidente una profonda espressione di marcatori neuronali classici come TUBB3, MAP2 e MAPT, a sostegno dell'applicabilità del bioreattore da banco per l'induzione neuronale. Seguendo questo protocollo, circa 6,2 milioni di neuroni iNGN2 immaturi possono essere ottenuti da 1 milione di hiPSC entro 4 giorni dalla coltura. Tuttavia, la capacità di produzione può variare tra i lotti e dipendere dal volume della coltura in sospensione durante la semina.
Per aumentare ulteriormente la resa, il tempo di coltivazione è stato esteso per ulteriori 3 giorni; Tuttavia, sebbene gli aggregati diventassero più grandi, diventavano anche altamente compatti e non potevano essere correttamente dissociati per la crioconservazione o la riplaccatura. Nonostante i progressi negli approcci di coltura 3D, le colture neuronali 2D aderenti sono ancora il gold standard per le analisi funzionali come array di microelettrodi o imaging del calcio. La singolarizzazione cellulare è quindi un prerequisito importante per un monostrato neuronale omogeneamente distribuito e una rete neuritica. Un distacco meccanico più forte delle cellule o reagenti di dissociazione duri possono essere utilizzati per garantire la singolarizzazione, ma con potenziali effetti sulla vitalità cellulare, sulla fisiologia e sulla capacità di riattaccamento47,48. Per quanto riguarda la necessità di singole cellule per ulteriori analisi e maturazione, gli aggregati sono stati dissociati dopo 4 giorni di sospensione e i neuroni iNGN2 (giorno 2) crioconservati. La differenziazione post-disgelo e la caratterizzazione dei neuroni iNGN2 hanno confermato una crescente maturità delle colture neuronali e la formazione di una fitta rete neuritica.
Il protocollo fornito produce un numero elevato di neuroni iNGN2. Tuttavia, è necessario considerare diverse limitazioni. Come per la maggior parte delle piattaforme di coltura in sospensione, il trasferimento di hiPSC da una coltura aderente 2D a un ambiente 3D è fondamentale e spesso associato a una profonda perdita cellulare. Ulteriori perdite significative di cellule e aggregati sono attese durante i cambiamenti dei media. Rispetto alle piattaforme automatizzate, il tempo di gestione e il rischio di contaminazione utilizzando il bioreattore da banco sono aumentati, poiché le modifiche del mezzo devono essere eseguite manualmente. A parte la mancanza di automazione, il bioreattore da banco non offre la possibilità di monitorare i nutrienti e la capacità massima di 50 ml per tubo limita il potenziale di upscaling del protocollo. Infine, è importante notare che, sebbene NGN2 acceleri l'impegno della linea neuronale, la durata della maturazione terminale non è ridotta e richiede ancora una coltura a lungo termine per diverse settimane. Data l'importanza del supporto astrocitico per la funzione neuronale, l'attaccamento e la sopravvivenza 49,50,51, gli approcci di co-coltura dovrebbero essere considerati, e potrebbero anche essere essenziali per la coltivazione a lungo termine.
In sintesi, un protocollo di differenziazione 2D è stato tradotto con successo in un ambiente 3D per la generazione rapida e riproducibile di neuroni iNGN2 implementando un bioreattore da banco. Il protocollo descritto produce elevate quantità di neuroni derivati da iPSC di buona qualità, che possono servire come punto di partenza per test di modelli complessi, screening di farmaci ad alto rendimento e saggi di tossicità su larga scala.
Divulgazioni
Gli autori non dichiarano interessi concorrenti.
Riconoscimenti
Il progetto EBiSC2 ha ricevuto finanziamenti dall'impresa comune (JU) Innovative Medicines Initiative 2 nell'ambito della convenzione di sovvenzione n. 821362. L'impresa comune riceve sostegno dal programma di ricerca e innovazione Horizon 2020 dell'Unione europea e dall'EFPIA. Ringraziamo Nadine J. Smandzich, Helene D. M. Hemmer, Johnn-Majd Balsters e Vanessa M. Nalewaja per la loro assistenza nelle analisi immunocitochimiche e di espressione genica, nonché Heike Arthen per il suo eccellente supporto tecnico. Inoltre, ringraziamo Stephanie Bur per l'istituzione del programma di bioreattori. La Figura 2A è stata creata con BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol 50 mM | Gibco | 11528926 | |
| 60 mm Nunclon Delta Surface | Nunc | 734-2040 | |
| Anti-beta III Tubulin antibody [TU-20] (dilution 1:1,000) | Abcam | ab7751 | |
| Applied Biosystems High-Capacity cDNA Reverse Transcription Kit | Thermo Fisher Scientific | 10400745 | |
| B-27 Supplement (50x) | Gibco | 11530536 | serum-free Supplement (50x) |
| BIONi010-C-13 hiPSC line | European Bank for induced pluripotent Stem Cell (EBiSC), BIONEER as Depositor | SAMEA103988285 | Bioneer is depositor in EBiSC and owner of the hiPSC line. |
| Biorender | Biorender.com | ||
| BSA Cell Culture grade | Thermo Fisher Scientific | 12330023 | |
| CERO 3D Incubator & Bioreactor | OLS OMNI Life Science | 2800000 | |
| CEROtubes Cell culture Tubes 50 mL | OLS OMNI Life Science | 2800005 | |
| Citavi 6 | Swiss Academic Software | ||
| CryoStor CS10 | Stemcell | 7930 | freezing medium containing 10% DMSO |
| Cytofix Fixation Solution | BD Biosciences | 554655 | fixation solution containing 4% paraformaldehyde |
| DAPI (NUCBLUE FIXED CELL STAIN) | Thermo Fisher Scientific | 12333553 | |
| Doxycycline hydrochloride (DOX) | Sigma-Aldrich | D3447 | |
| DPBS without Calcium and Magnesium (DPBS -/-) | Gibco | 14190250 | |
| DPBS with Calcium and Magnesium (DPBS +/+) | Gibco | 11580456 | |
| DMEM/F12 (-/-) | Gibco | 21331-020 | |
| DMEM/F-12, GlutaMAX Supplement | Gibco | 31331-028 | |
| EVOS XL Core Cell Imaging System | Thermo Fisher Scientific | ||
| Goat anti-Mouse IgG (H+L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor 555 | Thermo Fisher Scientific | A21424 | |
| GlutaMAX supplement | Gibco | 35050-038 | stabilized L-alanyl-L-glutamine dipeptid |
| ImageJ 1.51v | National Institute of Health | ||
| Insulin solution human | Sigma-Aldrich | I9278l | |
| Laminin | Merck | L2020 | |
| MACSQuant Analyzer | Miltenyi | ||
| MAP2 Monoclonal Antibody (dilution 1: 300) | Thermo Fisher Scientific | 13-1500 | |
| Matrigel Growth factor reduced, phenol-red free | Corning Life Science | 356231 | basement membrane matrix |
| MEM Non-Essential Amino Acids Solution (100x) | Gibco | 11140035 | |
| MicroAmp Optical Adhesive Film Kit | Thermo Fisher Scientific | 10095714 | |
| mTeSR1 | Stemcell | 85850 | feeder-free iPSC maintenance medium |
| N-2 Supplement (100x) | Gibco | 17502-048 | serum-free Supplement based on Bottenstein’s N-1 formulation |
| Neurobasal Medium | Gibco | 11570556 | |
| Nikon Eclipse TS2 | Nikon Instruments Europe B.V. | ||
| NucleoCounter-NC200 | ChemoMetec A/S | ||
| Origin 2021 | OriginLab | ||
| Penicillin-Streptomycin | Gibco | 11548876 | |
| Perm/Wash Buffer | BD Biosciences | 554723 | |
| Primer assay TUBB3(Hs00801390_s1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay GAPDH (Hs99999905_m1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay GUSB (Hs99999908_m1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay HPRT1 (Hs99999909_m1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay MAP2 (Hs00258900_m1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay MAPT (Hs00902194_m1) | Thermo Fisher Scientific | 11620099 | |
| Primer assay POU5F1 (Hs04260367_gH) | Thermo Fisher Scientific | 11620099 | |
| Poly-L-ornithine 0.01% | Merck | P4957 | |
| RNeasy Plus Micro Kit (50)-Kit | Qiagen | 74034 | column-based RNA isolation kit |
| RLT Buffer | Qiagen | 79216 | cell lysis buffer |
| Sodium Pyruvat (100 mM) | Gibco | 12539059 | |
| StemPro Accutase | Gibco | 11599686 | cell dissociation enzyme |
| TAQMAN FAST ADVANCED MASTER MIX | Thermo Fisher Scientific | 11380912 | qPCR master mix |
| Triton-X-100 | Sigma-Aldrich | T8787 | |
| TrypLE Select Enzym | Gibco | 12563-011 | Trypsin-EDTA solution |
| UltraPure 0.5 M EDTA, pH 8.0 | Thermo Fisher Scientific | AM9260G | |
| Via1-Cassette | ChemoMetec A/S | 941-0012 | |
| Wide Bore Filtered Pipette Tips | Thermo Fisher Scientific | 10088880 | |
| X20 OPTICAL 96WELL FAST CLEAR REACTION PLATES | Thermo Fisher Scientific | 15206343 | |
| Y-27632 dihydrochloride, Rho kinase inhibitor (ROCK inhibitor) | Abcam | ab120129 |
Riferimenti
- Feigin, V. L., et al. The global burden of neurological disorders: translating evidence into policy. The Lancet. Neurology. 19 (3), 255-265 (2020).
- Olesen, J., Gustavsson, A., Svensson, M., Wittchen, H. -. U., Jönsson, B. The economic cost of brain disorders in Europe. European Journal of Neurology. 19 (1), 155-162 (2012).
- Preclinical Development: The Safety Hurdle Prior to Human Trials. American Pharmaceutical Review Available from: https://www.americanpharmaceuticalreview.com/Featured-Articles/187349-Preclinical-Development-The-Safety-Hurdle-Prior-to-Human-Trials (2016)
- van Norman, G. A. Phase II trials in drug development and adaptive trial design. JACC. Basic to Translational Science. 4 (3), 428-437 (2019).
- van Norman, G. A. Limitations of animal studies for predicting toxicity in clinical trials: part 2: potential alternatives to the use of animals in preclinical trials. JACC. Basic to Translational Science. 5 (4), 387-397 (2020).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Chambers, S. M., et al. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nature Biotechnology. 27 (3), 275-280 (2009).
- Shi, Y., Kirwan, P., Livesey, F. J. Directed differentiation of human pluripotent stem cells to cerebral cortex neurons and neural networks. Nature Protocols. 7 (10), 1836-1846 (2012).
- Reinhardt, P., et al. Derivation and expansion using only small molecules of human neural progenitors for neurodegenerative disease modeling. PloS One. 8 (3), 59252 (2013).
- Hulme, A. J., Maksour, S., St-Clair Glover, M., Miellet, S., Dottori, M. Making neurons, made easy: the use of Neurogenin-2 in neuronal differentiation. Stem Cell Reports. 17 (1), 14-34 (2022).
- Zhang, Y., et al. Rapid single-step induction of functional neurons from human pluripotent stem cells. Neuron. 78 (5), 785-798 (2013).
- Schmid, B., et al. Generation of two gene edited iPSC-lines carrying a DOX-inducible NGN2 expression cassette with and without GFP in the AAVS1 locus. Stem Cell Research. 52, 102240 (2021).
- Shih, P. -. Y., et al. Development of a fully human assay combining NGN2-inducible neurons co-cultured with iPSC-derived astrocytes amenable for electrophysiological studies. Stem Cell Research. 54, 102386 (2021).
- Chang, C. -. Y., et al. Induced pluripotent stem cell (iPSC)-based neurodegenerative disease models for phenotype recapitulation and drug screening. Molecules. 25 (8), 2000 (2000).
- Silva, M. C., Haggarty, S. J. Human pluripotent stem cell-derived models and drug screening in CNS precision medicine. Annals of the New York Academy of Sciences. 1471 (1), 18-56 (2020).
- Elanzew, A., Sommer, A., Pusch-Klein, A., Brüstle, O., Haupt, S. A reproducible and versatile system for the dynamic expansion of human pluripotent stem cells in suspension. Biotechnology Journal. 10 (10), 1589-1599 (2015).
- Davis, B. M., Loghin, E. R., Conway, K. R., Zhang, X. Automated closed-system expansion of pluripotent stem cell aggregates in a rocking-motion bioreactor. SLAS Technology. 23 (4), 364-373 (2018).
- Kropp, C., et al. Impact of feeding strategies on the scalable expansion of human pluripotent stem cells in single-use stirred tank bioreactors. Stem Cells Translational Medicine. 5 (10), 1289-1301 (2016).
- Borys, B. S., et al. Overcoming bioprocess bottlenecks in the large-scale expansion of high-quality hiPSC aggregates in vertical-wheel stirred suspension bioreactors. Stem Cell Research & Therapy. 12 (1), 55 (2021).
- Kwok, C. K., et al. Scalable expansion of iPSC and their derivatives across multiple lineages. Reproductive Toxicology. 112, 23-35 (2022).
- User Protocol for Human induced Pluripotent Stem Cells. EBiSC Available from: https://ebisc.org/docs/ebisc/EBiSC_User_Protocol_for_Human_induced_Pluripotent_Stem_Cells.pdf (2023)
- Neyrinck, K., et al. SOX9-induced generation of functional astrocytes supporting neuronal maturation in an all-human system. Stem Cell Reviews and Reports. 17 (5), 1855-1873 (2021).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Griffith, L. G., Swartz, M. A. Capturing complex 3D tissue physiology in vitro. Nature Reviews. Molecular Cell Biology. 7 (3), 211-224 (2006).
- Burdick, J. A., Vunjak-Novakovic, G. Engineered microenvironments for controlled stem cell differentiation. Tissue Engineering. Part A. 15 (2), 205-219 (2009).
- Baker, B. M., Chen, C. S. Deconstructing the third dimension: how 3D culture microenvironments alter cellular cues. Journal of Cell Science. 125, 3015-3024 (2012).
- Duval, K., et al. Modeling physiological events in 2D vs. 3D cell culture. Physiology. 32 (4), 266-277 (2017).
- Haycock, J. W. 3D cell culture: a review of current approaches and techniques. Methods in Molecular Biology. 695, 1-15 (2011).
- Sen, A., Kallos, M. S., Behie, L. A. Effects of hydrodynamics on cultures of mammalian neural stem cell aggregates in suspension bioreactors. Industrial and Engineering Chemical Research. 40 (23), 5350-5357 (2001).
- Gerecht-Nir, S., Cohen, S., Itskovitz-Eldor, J. Bioreactor cultivation enhances the efficiency of human embryoid body (hEB) formation and differentiation. Biotechnology and Bioengineering. 86 (5), 493-502 (2004).
- Zhao, F., et al. Effects of oxygen transport on 3-d human mesenchymal stem cell metabolic activity in perfusion and static cultures: experiments and mathematical model. Biotechnology Progress. 21 (4), 1269-1280 (2005).
- Cimetta, E., Figallo, E., Cannizzaro, C., Elvassore, N., Vunjak-Novakovic, G. Micro-bioreactor arrays for controlling cellular environments: design principles for human embryonic stem cell applications. Methods. 47 (2), 81-89 (2009).
- Kehoe, D. E., Jing, D., Lock, L. T., Tzanakakis, E. S. Scalable stirred-suspension bioreactor culture of human pluripotent stem cells. Tissue Engineering. Part A. 16 (2), 405-421 (2010).
- Sargent, C. Y., et al. Hydrodynamic modulation of embryonic stem cell differentiation by rotary orbital suspension culture. Biotechnology and Bioengineering. 105 (3), 611-626 (2010).
- Liu, N., Zang, R., Yang, S. -. T., Li, Y. Stem cell engineering in bioreactors for large-scale bioprocessing. Engineering in Life Sciences. 14 (1), 4-15 (2014).
- Wolfe, R. P., Leleux, J., Nerem, R. M., Ahsan, T. Effects of shear stress on germ lineage specification of embryonic stem cells. Integrative Biology: Quantitative Biosciences from Nano to Macro. 4 (10), 1263-1273 (2012).
- Wolfe, R. P., Ahsan, T. Shear stress during early embryonic stem cell differentiation promotes hematopoietic and endothelial phenotypes. Biotechnology and Bioengineering. 110 (4), 1231-1242 (2013).
- Shafa, M., et al. Impact of stirred suspension bioreactor culture on the differentiation of murine embryonic stem cells into cardiomyocytes. BMC Cell Biology. 12 (1), 53 (2011).
- Kwok, C. K., et al. Scalable stirred suspension culture for the generation of billions of human induced pluripotent stem cells using single-use bioreactors. Journal of Tissue Engineering and Regenerative Medicine. 12 (2), 1076-1087 (2018).
- Rodrigues, C. A. V., et al. Scalable culture of human induced pluripotent cells on microcarriers under xeno-free conditions using single-use vertical-wheel™ bioreactors. Journal of Chemical Technology & Biotechnology. 93 (12), 3597-3606 (2018).
- Gilbertson, J. A., Sen, A., Behie, L. A., Kallos, M. S. Scaled-up production of mammalian neural precursor cell aggregates in computer-controlled suspension bioreactors. Biotechnology and Bioengineering. 94 (4), 783-792 (2006).
- Baghbaderani, B. A., et al. Expansion of human neural precursor cells in large-scale bioreactors for the treatment of neurodegenerative disorders. Biotechnology Progress. 24 (4), 859-870 (2008).
- Baghbaderani, B. A., et al. Bioreactor expansion of human neural precursor cells in serum-free media retains neurogenic potential. Biotechnology and Bioengineering. 105 (4), 823-833 (2010).
- Serra, M., Brito, C., Costa, E. M., Sousa, M. F. Q., Alves, P. M. Integrating human stem cell expansion and neuronal differentiation in bioreactors. BMC Biotechnology. , 82 (2009).
- Zhao, S., et al. Generation of cortical neurons through large-scale expanding neuroepithelial stem cell from human pluripotent stem cells. Stem Cell Research & Therapy. 11 (1), 431 (2020).
- Pawlowski, M., et al. Inducible and deterministic forward programming of human pluripotent stem cells into neurons, skeletal myocytes, and oligodendrocytes. Stem Cell Reports. 8 (4), 803-812 (2017).
- Bajpai, R., Lesperance, J., Kim, M., Terskikh, A. V. Efficient propagation of single cells Accutase-dissociated human embryonic stem cells. Molecular Reproduction and Development. 75 (5), 818-827 (2008).
- Jager, L. D., et al. Effect of enzymatic and mechanical methods of dissociation on neural progenitor cells derived from induced pluripotent stem cells. Advances in Medical Sciences. 61 (1), 78-84 (2016).
- Allen, N. J. Astrocyte regulation of synaptic behavior. Annual Review of Cell and Developmental Biology. 30, 439-463 (2014).
- Verkhratsky, A., Nedergaard, M., Hertz, L. Why are astrocytes important. Neurochemical Research. 40 (2), 389-401 (2015).
- Jäkel, S., Dimou, L. Glial cells and their function in the adult brain: a journey through the history of their ablation. Frontiers in Cellular Neuroscience. 11, 24 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon