Method Article
Morphogenèse tridimensionnelle dans l’intestin canin sur puce à l’aide d’organoïdes intestinaux dérivés de patients atteints de maladies inflammatoires de l’intestin
Dans cet article
Résumé
L’intégration d’organoïdes intestinaux canins et d’un système microfluidique Gut-on-a-Chip offre des modèles translationnels pertinents pour les maladies intestinales humaines. Les protocoles présentés permettent la morphogenèse 3D et la modélisation dynamique in vitro de l’intestin, ce qui contribue au développement de traitements efficaces pour les maladies intestinales chez les chiens et les humains avec One Health.
Résumé
Les intestins canins présentent des similitudes dans l’anatomie, la microbiologie et la physiologie avec ceux des humains, et les chiens développent naturellement des troubles intestinaux spontanés similaires à ceux des humains. Surmonter la limitation inhérente des organoïdes tridimensionnels (3D) dans l’accès à la surface apicale de l’épithélium intestinal a conduit à la génération de cultures monocouches bidimensionnelles (2D), qui exposent la surface luminale accessible à l’aide de cellules dérivées des organoïdes. L’intégration de ces organoïdes et des cultures monocouches dérivées d’organoïdes dans un système microfluidique Gut-on-a-Chip a permis de faire évoluer la technologie, permettant le développement de modèles intestinaux dynamiques in vitro plus pertinents sur le plan physiologique.
Dans cette étude, nous présentons un protocole pour générer une morphogenèse 3D de l’épithélium intestinal canin à partir d’échantillons de tissus intestinaux primaires obtenus chez des chiens atteints de maladies inflammatoires chroniques de l’intestin (MICI). Nous décrivons également un protocole pour générer et maintenir des cultures monocouches 2D et des systèmes intestins sur puce à l’aide de cellules dérivées des organoïdes intestinaux 3D. Les protocoles présentés dans cette étude servent de cadre de base pour la mise en place d’un système microfluidique Gut-on-a-Chip spécialement conçu pour les chiens. En jetant les bases de cette approche innovante, nous visons à étendre l’application de ces techniques à la recherche biomédicale et translationnelle, en nous alignant sur les principes de l’initiative One Health. En utilisant cette approche, nous pouvons développer des modèles in vitro dynamiques plus pertinents sur le plan physiologique pour l’étude de la physiologie intestinale chez les chiens et les humains. Cela a des implications importantes pour les applications biomédicales et pharmaceutiques, car cela peut aider à développer des traitements plus efficaces pour les maladies intestinales chez les deux espèces.
Introduction
La morphogenèse épithéliale intestinale a été largement étudiée à l’aide de modèles animaux de laboratoire, qui sont coûteux, prennent du temps et ne représentent pas avec précision les processus de développement humain1. De plus, les modèles de culture cellulaire 2D statique conventionnels n’ont pas la capacité d’imiter l’organisation spatiale complexe d’une architecture épithéliale 3D2. Par conséquent, il est nécessaire de mettre en place un protocole permettant d’induire une morphogenèse 3D in vitro à l’aide de cellules épithéliales intestinales provenant de modèles animaux pertinents pour l’homme afin de faire progresser notre compréhension de l’architecture épithéliale intestinale.
Les chiens de compagnie ont développé une anatomie intestinale et des compositions de microbiome remarquablement similaires à celles des humains en raison de leur environnement et de leur régime alimentaire communs pendant la domestication3. En plus de cette similitude, les humains et les chiens partagent diverses morbidités chroniques que l’on pense être attribuées à la santé intestinale. Les chiens, comme les humains, peuvent développer spontanément des maladies chroniques telles que l’obésité, le dysfonctionnement cognitif, le diabète sucré, les maladies inflammatoires de l’intestin (MII) et l’adénocarcinome colorectal 4,5,6,7,8,9,10. Malgré le développement et l’utilisation de cellules épithéliales humaines et murines dans des études antérieures sur l’intestinsur puce 2,11,12,13,14, l’épithélium intestinal canin n’a pas été utilisé jusqu’à présent. Notre nouvelle approche, qui utilise l’épithélium organoïde intestinal canin dans un système de culture dynamique avec une morphogenèse épithéliale 3D, a des implications importantes pour la médecine canine et humaine.
Les progrès récents dans la culture d’organoïdes intestinaux ont conduit à la mise en place de la culture d’organoïdes intestinaux canins15. Ce système de culture consiste à cultiver des cellules souches intestinales sous conditionnement morphogène défini, ce qui permet d’obtenir un modèle 3D aux propriétés d’auto-renouvellement dérivé de cellules souches adultes16. Cependant, la réalisation d’essais de transport ou de cocultures hôte-microbiome pose des difficultés avec ce modèle 3D en raison de la nature fermée de la lumière intestinale17. Pour remédier à ce problème, les chercheurs ont généré une monocouche 2D dérivée d’organoïdes intestinaux, permettant l’exposition de la surface luminale18,19. Cependant, les organoïdes 3D et les monocouches 2D sont maintenus dans des conditions statiques, qui ne reflètent pas avec précision la biomécanique in vivo du microenvironnement intestinal. La combinaison de la technologie des organoïdes canins dérivés des patients et de la morphogenèse 3D in vitro offre une opportunité pour la recherche translationnelle sur les maladies chroniques multifactorielles. Cette approche permet aux chercheurs de mettre au point des traitements plus efficaces qui profitent à la fois aux humains et aux chiens et de faire progresser la recherche translationnelle, en s’alignant sur l’initiative One Health, qui est une approche collaborative qui reconnaît l’interdépendance de la santé humaine, animale et environnementale. Il favorise la coopération interdisciplinaire afin de relever des défis complexes en matière de santé et d’obtenir des résultats optimaux en matière de santé pour tous. En comprenant les interdépendances entre les humains, les animaux et les écosystèmes, l’initiative vise à atténuer les risques liés aux maladies infectieuses émergentes, à la dégradation de l’environnement et à d’autres problèmes de santé communs20,21,22.
Ce protocole décrit des méthodes complètes pour la culture de cellules épithéliales intestinales canines obtenues à partir d’organoïdes de patients sur un microdispositif Gut-on-a-Chip avec une membrane poreuse à base de polydiméthylsiloxane (PDMS). L’établissement de la morphogenèse épithéliale 3D par l’intégration d’organoïdes intestinaux canins et cette technologie Gut-on-a-Chip nous permet d’étudier comment l’intestin se développe et maintient son organisation cellulaire et sa niche de cellules souches. Cette plateforme offre une occasion précieuse d’étudier l’impact des communautés de microbiome sur la santé intestinale et de comprendre comment ces communautés génèrent des métabolites microbiens qui contribuent à la physiopathologie intestinale14,23. Ces progrès peuvent maintenant être étendus aux échantillons intestinaux canins, offrant aux chercheurs la possibilité d’explorer la relation complexe entre le microbiome intestinal et la physiologie de l’hôte. Cela ouvre la voie à des informations précieuses sur les mécanismes sous-jacents de la physiopathologie intestinale et sur la compréhension du rôle potentiel des métabolites microbiens dans la santé canine et humaine, ainsi que dans diverses maladies. Le protocole utilisé pour l’intestin sur puce canin est reproductible, ce qui en fait un modèle expérimental approprié pour la médecine comparée, car cette approche permet d’étudier les interactions hôte-microbiome, les infections pathogènes et les effets thérapeutiques à base de probiotiques chez les chiens et les humains.
Protocole
L’étude a été approuvée et menée conformément au Comité institutionnel de protection et d’utilisation des animaux de l’Université de l’État de Washington (ASAF # 6993). Dans ce protocole, nous avons utilisé un dispositif microfluidique Gut-on-a-Chip bien établi en PDMS qui a été fabriqué en interne2 (Figure 1D). Des méthodes détaillées de fabrication du microdispositif Gut-on-a-Chip peuvent être trouvées dans les rapports précédents 2,24. Ce protocole démontre une intégration unique d’organoïdes intestinaux et d’un système microfluidique (Figure 2).
1. Activation de surface d’un Gut-on-a-Chip en PDMS
- Préparer une solution de polyéthylène (PEI) à 1 % en ajoutant 1 mL de solution PEI à 50 % dans 49 mL d’eau distillée (DI) dans un tube conique de 50 mL. Retournez le tube deux à trois fois pour bien mélanger la solution, puis filtrez la solution à l’aide d’un filtre seringue de 0,2 μm.
REMARQUE : Conservez-le à 4 °C. - Préparer une solution de glutaraldéhyde (GA) à 0,1 % en ajoutant 100 μL de solution à 50 % d’AG dans 49,9 mL d’AD dans un tube conique de 50 mL. Retournez le tube deux à trois fois pour bien mélanger la solution, puis filtrez la solution à l’aide d’un filtre seringue de 0,2 μm.
REMARQUE : Conservez-le à 4 °C et assurez-vous de le protéger de l’exposition à la lumière directe. - Placez l’appareil Gut-on-a-Chip dans un four sec réglé à 60 °C et incubez-le pendant au moins 30 min pour éliminer toute humidité restante.
- Exposez l’appareil Gut-on-a-Chip à un traitement UV et à l’ozone pendant 60 minutes à l’aide d’un générateur UV/Ozone.
REMARQUE : Pour assurer une activation optimale des surfaces PDMS pendant le traitement, maintenez une distance d’environ 3 cm ou moins entre la lampe UV et l’appareil. Évitez d’encombrer les appareils dans le générateur pour obtenir une activation efficace de la surface. - Fixez les tubes d’entrée, de dérivation et de sortie du microcanal supérieur à l’aide de pinces de liant pour les serrer. Fixez également le tube d’entrée pour le microcanal inférieur.
- Débranchez le tube de sortie du microcanal inférieur. Assurez-vous que le tube de dérivation du microcanal inférieur reste ouvert.
- À l’aide d’une micropipette P100, introduire 100 μL d’une solution de PEI à 1 % par l’orifice de sortie du microcanal inférieur.
REMARQUE : Il est recommandé d’effectuer ce processus pendant que l’appareil est encore chaud à cause de l’exposition aux UV/Ozone. Observez la solution de PEI s’écouler à travers le microcanal et la solution de PEI tombe pour sortir du tube de dérivation vers le microcanal inférieur. - Rebranchez le tube de sortie du microcanal inférieur.
- Fixez les tubes d’entrée, de dérivation et de sortie du microcanal inférieur en utilisant des pinces de reliure pour les serrer. Fixez également le tube d’entrée pour le microcanal supérieur.
- Débranchez le tube de sortie du microcanal supérieur. Assurez-vous que le tube de dérivation du microcanal supérieur reste ouvert.
- À l’aide d’une micropipette P100, introduire 100 μL d’une solution de PEI à 1 % par l’orifice de sortie du microcanal supérieur.
REMARQUE : Observez la solution de PEI s’écouler à travers le microcanal et la solution de PEI tombe pour sortir du tube de dérivation vers le microcanal supérieur. - Remettez en place le tube de sortie du microcanal supérieur.
- Une fois que les microcanaux supérieur et inférieur sont remplis d’une solution de PEI à 1 %, laissez l’appareil incuber à température ambiante (RT) pendant 10 minutes.
- Effectuez les étapes 1.5 à 1.12 avec une solution à 0,1 % d’AG.
- Une fois que les microcanaux supérieur et inférieur sont remplis d’une solution à 0,1 % d’AG, laissez l’appareil incuber à RT pendant 20 minutes.
- Effectuez les étapes 1.5 à 1.12 avec de l’eau DI pour éliminer tout excès de solution d’activation de surface.
- Placez les chips dans un four sec à 60 °C et laissez-les sécher toute la nuit.
REMARQUE : Assurez-vous de retirer les clips de reliure de tous les tubes pour éviter tout impact du tube. Ce processus de séchage est essentiel à l’activation uniforme de la surface du microcanal dans le Gut-on-a-Chip12.
2. Enrobage de matrice extracellulaire (MEC) et préparation du milieu de culture pour la culture Gut-on-a-Chip
- Préparer un mélange ECM avec un milieu basal organoïde de manière à ce que la concentration finale de collagène I et de Matrigel soit respectivement de 60 μg/mL et 2 % (vol/vol).
REMARQUE : Préparer 50 μL de mélange ECM par puce intestinale (c.-à-d. 20 μL de mélange ECM pour chaque microcanal supérieur et inférieur et 10 μL supplémentaires). Préparez-le le jour de l’utilisation et conservez cette solution à 4 °C ou placez-la sur de la glace jusqu’à ce qu’elle soit prête à l’emploi. - Sortez l’intestin sur puce qui a été traité avec des UV/Ozone, du PEI et du GA de l’étuve sèche.
REMARQUE : Inspecter sous un microscope à contraste de phase pour voir s’il y a de l’humidité résiduelle. - Laissez refroidir l’intestin sur puce dans une enceinte de sécurité biologique pendant 10 minutes.
- Fixez les tubes d’entrée, de dérivation et de sortie du microcanal supérieur à l’aide de pinces de liant pour les serrer. Fixez également le tube d’entrée pour le microcanal inférieur.
- Débranchez le tube de sortie du microcanal inférieur.
- À l’aide d’une micropipette P100, introduisez 20 μL de mélange ECM à travers l’orifice de sortie du microcanal inférieur.
REMARQUE : Observez le mélange ECM s’écouler à travers le microcanal sans piéger de bulles d’air. S’il y a une bulle d’air, introduisez un mélange ECM supplémentaire dans le microcanal inférieur jusqu’à ce que la bulle disparaisse. - Remettez en place le tube de sortie du microcanal inférieur.
- Fixez les tubes d’entrée, de dérivation et de sortie du microcanal inférieur en utilisant des pinces de reliure pour les serrer. Fixez également le tube d’entrée pour le microcanal supérieur.
- Débranchez le tube de sortie du microcanal supérieur.
- À l’aide d’une micropipette P100, introduire 20 μL de mélange ECM à travers l’orifice de sortie du microcanal supérieur.
- Remettez en place le tube de sortie du microcanal supérieur.
- Placer la puce dans un incubateur de CO 2 humidifié avec 5 % de CO2 à 37 °C pendant 1 h pour former la couche ECM sur la membrane PDMS traitée avec PEI et GA (Figure 3A).
REMARQUE : Assurez-vous que tous les tubes sont serrés pendant l’incubation. - Pendant l’incubation de l’enrobage ECM, préparer deux seringues de 1 mL contenant un milieu d’ensemencement à froid, qui est le milieu de culture organoïde dépourvu d’A8301 mais contenant 10 μM Y-27632 et 2,5 μM CHIR99021, comme indiqué précédemment dans Gut-on-a-Chip dérivé d’organoïdes humains2.
REMARQUE : L’A8301 a été retiré du milieu pour améliorer la fixation des cellules à l’ECM enrobé, comme indiqué précédemment2. Le jour 0 de la culture Gut-on-a-Chip ne nécessite qu’un écoulement de microcanaux supérieurs. Par conséquent, préparez une seringue pleine pour le canal supérieur et un minimum de 0,2 ml pour le canal inférieur. - Une fois le revêtement ECM terminé, retirez la puce de l’incubateur et relâchez la pince sur le tube de dérivation connecté au microcanal inférieur.
- Connectez une seringue de 1 mL remplie de milieu d’ensemencement à l’aiguille à extrémité émoussée qui est reliée au microcanal inférieur de l’intestin sur puce. Introduisez délicatement le milieu de semis (~50 μL) dans le tube de dérivation. Une fois que le tube est rempli avec le fluide introduit, fixez le tube de dérivation connecté au microcanal inférieur avec un clip de liant.
- Relâchez la pince du tube de sortie connecté au microcanal inférieur. Introduisez soigneusement le milieu de semis dans le microcanal inférieur pour lui permettre de s’écouler en douceur dans le système. Fixez le tube de sortie connecté au microcanal inférieur avec un clip de liant après la perfusion.
- Ensuite, ouvrez le tube de dérivation connecté au microcanal supérieur.
- Connectez une seringue de 1 ml remplie de milieu d’ensemencement à l’aiguille à extrémité émoussée qui est reliée au microcanal supérieur de l’intestin sur puce. Introduisez délicatement le milieu de semis (~50 μL) dans le tube de dérivation. Une fois que le tube est rempli avec le fluide introduit, fixez le tube de dérivation connecté au microcanal supérieur avec un clip de liant.
- Relâchez le tube de sortie connecté au microcanal inférieur. Introduisez soigneusement le milieu de semis dans le microcanal supérieur pour lui permettre de s’écouler en douceur dans le système. Fixez le tube de sortie connecté au microcanal supérieur avec un clip de liant après la perfusion.
3. Préparation de cellules organoïdes intestinales canines pour l’ensemencement
REMARQUE : Pour générer le modèle Gut-on-a-Chip, des organoïdes du côlon canin (appelés colonoïdes) dérivés de chiens atteints de MII ont été utilisés dans ce protocole. Ces colonoïdes ont été obtenus à partir de trois à cinq petits fragments de tissu colique biopsiés, selon une méthode précédemment rapportée15,18. Pour des résultats optimaux, il est crucial d’utiliser des colonoïdes canins qui ont subi un minimum de trois passages de culture afin d’établir des organoïdes stables adaptés aux applications in vitro. Il est recommandé de cultiver les colonoïdes canins pendant une durée d’au moins 3 à 4 jours pour faciliter la différenciation adéquate des cellules multi-lignées au sein des organoïdes, garantissant leur maturité fonctionnelle et leur adéquation aux expériences ultérieures dans le modèle Gut-on-a-Chip. La limite maximale du passage pour ce travail est inférieure à 20, comme l’indique une étude antérieure qui a démontré le phénotype et le caryotype inaltérés tout au long de 20 passages consécutifs25. La signalisation de ces donneurs est présentée dans le tableau supplémentaire S1.
- Cultivez les colonoïdes canins dans des plaques à 24 puits incorporées dans 30 μL de Matrigel 15,18 avec un milieu de culture organoïde répertorié dans le tableau des matériaux, qui est modifié à partir du milieu précédemment rapporté 15,26,27. Le milieu conditionné a été obtenu en cultivant des cellules Rspo1 et des cellules HEK293 modifiées pour sécréter Noggin28.
- Éliminez le milieu de culture organoïde par aspiration sous vide et introduisez 500 μL de solution de récupération cellulaire à une température glaciale dans chaque puits. Incuber pendant 30 minutes à 4 °C.
REMARQUE : En règle générale, trois puits sur une plaque de 24 puits d’organoïdes intestinaux canins matures fournissent une quantité suffisante de cellules dissociées pour ensemencer un seul dispositif Gut-on-a-Chip avec 40 à 50 organoïdes / un champ de vision à un grossissement x10. - Perturber mécaniquement les dômes Matrigel pendant 5 s à l’aide d’une micropipette P1000. Par la suite, rassemblez la suspension organoïde dans un tube conique de 15 mL.
- Centrifuger le tube conique à 200 × g et 4 °C pendant 5 min, puis retirer le surnageant.
- Introduire 1 mL de protéase de type trypsine à température ambiante, complétée par 10 μM Y-27632, et remettre en suspension la pastille cellulaire par pipetage à l’aide d’une micropipette P1000.
- Placez la suspension cellulaire dans un bain-marie réglé à 37 °C et incubez-la pendant 10 min en agitant périodiquement le mélange.
- Introduire 5 mL de milieu basal organoïde chaud et pipeter vigoureusement la suspension cellulaire à l’aide d’une micropipette P1000 jusqu’à ce qu’elle devienne trouble, sans amas cellulaires visibles.
- Passez la suspension cellulaire à travers un tamis cellulaire avec une coupure de 70 μm pour éliminer les débris de Matrigel et les grands amas de cellules.
- Centrifuger le tube conique à 200 × g et 4 °C pendant 5 min, puis remettre le granulé en suspension dans le milieu de semis. Pour l’ensemencement d’un dispositif Gut-on-a-Chip canin, utilisez 20 μL de milieu de semis pour remettre en suspension la pastille cellulaire (c.-à-d. utilisez 20 μL de milieu de semis lors de l’ensemencement d’un dispositif Gut-on-a-Chip).
- Modifier la concentration de cellules viables à 1 × 107 cellules/mL avec le milieu de semis comme indiqué précédemment2. Effectuez une évaluation de la viabilité à l’aide d’un hémocytomètre en combinant 10 μL de la suspension cellulaire avec 10 μL de bleu de trypan, puis observez les cellules au microscope.
REMARQUE : Il est crucial d’ajuster la concentration cellulaire de manière appropriée pour assurer une fixation cellulaire optimale et la formation d’une monocouche uniforme sur la membrane de la puce. Si le nombre initial de cellules est insuffisant, il peut en résulter un retard ou un échec de l’établissement d’une monocouche confluente. À l’inverse, si le nombre de cellules est excessif, les cellules non adhérentes peuvent s’agréger en amas à l’intérieur du canal, entraînant des effets de concentration indésirables.
4. Ensemencement et formation d’une monocouche cellulaire 2D
- Débranchez le tube de sortie du microcanal supérieur. Assurez-vous que le tube de dérivation du microcanal supérieur reste ouvert. Fixez à la fois l’entrée et la sortie du microcanal inférieur en utilisant des pinces de reliure pour les serrer.
- À l’aide d’une micropipette P100 ou P20, introduire 20 μL de la suspension cellulaire du protocole 3 dans l’orifice de sortie du microcanal supérieur (Figure 3B Ensemencement).
- Fixez le tube de dérivation et d’entrée du microcanal supérieur à l’aide de clips de liant pour le serrer. Ensuite, rattachez le tube de sortie au trou de sortie du microcanal supérieur, en veillant à ce que le tube reste ouvert tout au long du processus pour éviter toute pression appliquée sur le microcanal supérieur. Après cette étape, fixez lentement le tube de sortie du canal supérieur à l’aide d’un clip de liant pour le serrer.
- Vérifiez à l’aide d’un microscope que les cellules sont uniformément réparties dans le microcanal supérieur.
REMARQUE : Il est important de serrer le tube pour arrêter le mouvement du fluide dans le canal afin de permettre des conditions statiques stables jusqu’à ce que la fixation souhaitable de la cellule soit accomplie. - Placez la puce dans un incubateur de CO2 humidifié à 37 °C.
REMARQUE : Il faut environ 3 h pour que les cellules organoïdes intestinales canines se fixent au revêtement de l’ECM (Figure 3B , pièce jointe). - Connectez la seringue fixée au microcanal supérieur de l’intestin sur puce à un pousse-seringue positionné dans un incubateur de CO2 .
REMARQUE : Rincez soigneusement le fluide dans le microcanal à l’aide du bouton du pousse-seringue et retirez les cellules non liées. Examinez la puce au microscope pour vous assurer que toutes les cellules non attachées ont été efficacement éliminées. - Amorcer l’écoulement du milieu de culture à 30 μL/h avec le milieu de semis. Ce flux continu n’est destiné qu’au microcanal supérieur dans un premier temps jusqu’à l’établissement de la monocouche 2D sur un boyau sur puce. Pour le microcanal inférieur, laissez les microcanaux serrés et le fluide non rincé.
- Dès le lendemain de l’ensemencement des cellules, remplacez le milieu de culture par un milieu de culture organoïde, qui contient de l’A8301 et qui ne contient pas 10 μM Y-27632 et 2,5 μM CHIR99021, à partir du milieu de semis.
- Une fois la monocouche établie, initiez également l’écoulement continu du fluide vers le microcanal inférieur. Il faut généralement 2 à 3 jours pour que les cellules épithéliales organoïdes intestinales canines établissent des monocouches épithéliales (Figure 3C).
5. Mise en place de la morphogenèse 3D dans le Gut-on-a-Chip canin
REMARQUE : Après la formation des monocouches confluentes dans Gut-on-a-Chip, un écoulement moyen du canal supérieur et du canal inférieur et de la souche cellulaire a été introduit pour initier la morphogenèse 3D à la monocouche 2D, comme présenté à la figure 2.
- Introduire le milieu de culture organoïde dans les microcanaux supérieur et inférieur pour initier le développement de la morphogenèse 3D dans un système Gut-on-a-Chip. Pour obtenir une contrainte de cisaillement de 0,02 dyne/cm2 dans la conception Gut-on-a-Chip existante (c’est-à-dire un microcanal de 500 μm de hauteur), augmenter le débit à 50 μL/h29.
- Initier 10 % de la déformation cellulaire et 0,15 Hz de fréquence, comme recommandé avant2, à l’aide d’un bioréacteur régulé par ordinateur en appliquant une contrainte cyclique aux cellules cultivées in vitro. Ce processus appliquerait une aspiration sous vide à l’appareil Gut-on-a-Chip.
- Maintenez ces conditions de culture pendant au moins 2 à 3 jours. La morphogenèse 3D de la monocouche intestinale canine se produit généralement 2 à 3 jours après le début de l’écoulement du canal Dural et de l’aspiration sous vide (Figure 3C).
6. Caractérisation de l’intestin sur puce canin
- Imagerie de cellules vivantes
- Éliminez les bulles d’air dans le Gut-on-a-Chip en faisant couler doucement le fluide à l’aide du pousse-seringue.
- Débranchez le dispositif Gut-on-a-Chip du pousse-seringue.
REMARQUE : Évitez toute manœuvre qui peut appliquer une pression à l’intérieur de l’intestin sur puce. - Positionnez l’appareil sur un microscope pour capturer des images de l’épithélium 3D établi. Pour visualiser la structure des couches épithéliales 3D à l’aide d’un contraste de phase, utilisez des objectifs 10x et 20x (Figure 3C).
- Coloration par immunofluorescence
- Préparez la solution bloquante en dissolvant 1 g de BSA dans 50 mL de solution saline tampon phosphate (PBS) pour obtenir 2 % de BSA. Faire passer la solution à travers un filtre seringue de 0,2 μm pour la filtration. Conservez cette solution à une température de 4 °C.
- Préparer la solution perméabilisante en combinant 150 μL de Triton X-100 avec 50 mL de solution bloquante, ce qui donne une concentration finale de 0,3 % de Triton X-100. Faire passer la solution à travers un filtre seringue de 0,2 μm pour la filtration. Conservez cette solution à une température de 4 °C.
- Effectuer la fixation des cellules en injectant 100 μL d’un PFA à 4 % dans les microcanaux supérieur et inférieur, comme décrit aux étapes 2.4 à 2.11.
- Rincez les cellules en injectant 100 μL de PBS dans les microcanaux supérieur et inférieur, comme décrit aux étapes 2.4 à 2.11.
- Effectuer la perméabilisation des cellules en injectant 100 μL de Triton à 0,3 % dans les microcanaux supérieur et inférieur, comme décrit aux étapes 2.4 à 2.11. Placez l’appareil sur RT pendant 30 min.
- Rincez les cellules en injectant 100 μL de PBS dans les microcanaux supérieur et inférieur, comme décrit aux étapes 2.4 à 2.11.
- Effectuer un blocage des cellules pour empêcher la liaison non spécifique en injectant 100 μL de BSA à 2 % dans les microcanaux supérieur et inférieur, comme décrit aux étapes 2.4 à 2.11. Placez l’appareil sur RT pendant 1 h.
- Injecter 20 μL de la solution primaire d’anticorps diluée dans 2 % de BSA et la placer à RT pendant 3 h, suivie d’une nuit d’incubation à 4 °C.
REMARQUE : Assurez-vous que tous les tubes sont serrés pendant l’incubation à 4 °C pendant la nuit. La concentration d’un anticorps primaire doit être de 2 à 5 fois supérieure à la concentration recommandée pour la coloration monocouche ou organoïde 3D (figure supplémentaire S1). - Rincez les cellules en injectant 100 μL de PBS dans les microcanaux supérieur et inférieur, comme décrit aux étapes 2.4 à 2.11.
- Injecter 20 μL de la solution d’anticorps secondaire diluée dans 2 % de BSA et la placer à RT pendant 1 h.
REMARQUE : Assurez-vous que tous les tubes sont serrés pendant l’incubation. À partir de cette étape, il est nécessaire de protéger la configuration de l’appareil avec du papier d’aluminium pour éviter le photoblanchiment. - Rincez les cellules en injectant 100 μL de PBS dans les microcanaux supérieur et inférieur, comme décrit aux étapes 2.4 à 2.11.
- Préparer une solution combinée pour la contre-coloration de la F-actine et du DAPI (diamidino-2-phénylindole). Injecter 20 μL de la solution combinée dans les microcanaux supérieur et inférieur comme décrit aux étapes 2.4 à 2.11.
- Rincez les cellules en injectant 100 μL de PBS dans les microcanaux supérieur et inférieur, comme décrit aux étapes 2.4 à 2.11.
Réaliser une imagerie par fluorescence de l’architecture des cellules épithéliales 3D à l’aide d’un microscope à fluorescence ou d’un microscope confocal.
7. Fonction de barrière épithéliale
- Éliminez les bulles d’air dans le Gut-on-a-Chip en faisant couler doucement le fluide à l’aide du pousse-seringue. Assurez-vous que tous les tubes sont ouverts pendant la mesure.
- Retirez le Gut-on-a-Chip du pousse-seringue et placez-le à RT pendant au moins 10 minutes.
- Retirer les électrodes Ag/AgCl de la solution d’EtOH à 70 %.
- Placez deux électrodes Ag/AgCl dans l’entrée supérieure et la sortie inférieure, respectivement, pour mesurer la résistance de la couche épithéliale à l’aide d’un multimètre.
- Placez deux électrodes Ag/AgCl dans l’entrée inférieure et la sortie supérieure, respectivement. Indiquez la moyenne de ces deux valeurs en tant que valeur de résistance pour le Gut-on-a-Chip.
REMARQUE : Le TEER vierge doit être mesuré sur un Gut-on-a-Chip avec uniquement un revêtement ECM sans l’épithélium. - Calculer la valeur de la résistance électrique transépithéliale (TEER) (kΩ × cm2) peut être calculé à l’aide de l’équation (1).
TEER = (Ωt - Ωblanc) × A (1)
Où Ωt est une valeur de résistance mesurée à un moment précis depuis le début de l’expérience, Ωblanc est une valeur de résistance mesurée à l’instant sans l’épithélium, et A est la surface de la surface recouverte par une couche cellulaire (environ 0,11 cm2 pour ce modèle Gut-on-a-Chip29).- Calculer le TEER normalisé à l’aide de l’équation (2).
FEER =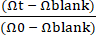 (2)
(2)
Où Ω0 est une valeur de résistance au point de temps de lecture initial de l’expérience, comme indiqué précédemment30 (Figure 4C).
- Calculer le TEER normalisé à l’aide de l’équation (2).
Résultats
Ce protocole facilite de manière fiable le développement spontané de la morphogenèse intestinale 3D dans un système Gut-on-a-Chip. Cette approche utilise des cellules épithéliales intestinales canines obtenues à partir d’organoïdes intestinaux dérivés de chiens atteints de maladies inflammatoires de l’intestin (MII) (Figure 1B). Des regroupements occasionnels de morphogenèse 3D de cellules épithéliales intestinales canines peuvent être observés dans l’ensemble du microcanal après 6 à 9 jours d’écoulement moyen (Figure 3C). Ces changements morphologiques peuvent être surveillés à l’aide de techniques de contraste de phase. Dans cette étude, nous avons utilisé des organoïdes dérivés de deux chiens diagnostiqués avec une MII. Notamment, une morphogenèse 3D réussie a été observée dans deux répétitions biologiques, chacune d’entre elles ayant été réalisée avec deux répétitions techniques. Les résultats de cette étude fournissent une base pour de futures recherches impliquant des organoïdes intestinaux dérivés d’autres donneurs canins. Ces résultats démontrent l’applicabilité et la répétabilité potentielles de notre approche expérimentale, qui a déjà été rapportée dans des échantillons humains. Ces résultats confirment une fois de plus que la technologie Gut-on-a-Chip est applicable aux cellules épithéliales intestinales canines, comme indiqué précédemment avec les études utilisant des cellules épithéliales intestinales humaines2.
Ce protocole a montré que la coloration par immunofluorescence peut être utilisée pour évaluer la structure 3D de monocouches dérivées d’organoïdes qui ont formé des structures ressemblant à des villosités dans des puces microfluidiques à l’aide de la microscopie à fluorescence conventionnelle (Figure 4 A,B). Ce protocole peut être adapté pour valider les phénotypes cellulaires différenciés et organisés spatialement par coloration par immunofluorescence. La visualisation d’une morphogenèse 3D au sein d’un Gut-on-a-Chip offre une excellente occasion d’étudier la réponse de l’hôte au cours de diverses interactions pathologiques 14,23,31. Lorsqu’elle est combinée avec des cellules épithéliales dérivées de patients donneurs, comme décrit précédemment chez l’homme, cette technologie peut être utilisée pour construire des modèles personnalisés de maladies intestinales13. Grâce à l’intégration de l’imagerie par immunofluorescence avec des techniques de visualisation de l’ARN ciblées comme l’hybridation in situ par fluorescence, il peut être possible d’analyser visuellement les transcriptomes et les protéomes de l’hôte dans un système Gut-on-a-Chip.
La préservation de l’intégrité de la membrane intestinale est vitale pour le maintien de l’homéostasie intestinale, et la plateforme Gut-on-a-Chip offre un avantage précieux en permettant une surveillance et une quantification précises de cette fonction cruciale. La mesure de l’ETER à l’aide de la technologie Gut-on-a-Chip offre plusieurs avantages. Par exemple, des études antérieures ont évalué avec succès le TEER lors de la co-culture de cellules intestinales avec des bactéries non pathogènes et probiotiques32, ainsi que dans des conditions d’intestin perméable23. Cela permet aux chercheurs d’étudier l’impact de différentes conditions sur la fonction de la barrière intestinale et d’identifier des interventions potentielles pour promouvoir la santé intestinale.
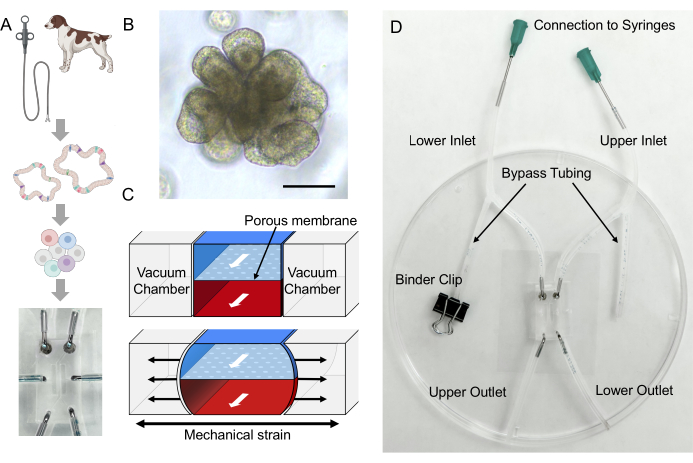
Figure 1 : Mise en place d’une MICI canine dérivée d’un patient Gut-on-a-Chip. (A) Intégration des organoïdes intestinaux dérivés du patient et de la plate-forme Gut-on-a-Chip. La biopsie endoscopique peut être effectuée pour isoler les cellules de la crypte intestinale afin de développer des organoïdes intestinaux spécifiques au donneur. Les cellules épithéliales peuvent être dissociées en cellules uniques à partir des organoïdes, puis ensemencées dans un intestin sur puce basé sur PDMS et cultivées dans un microenvironnement dynamique unique. (B) Images représentatives de colonoïdes provenant d’un chien atteint de MII. Barre d’échelle = 100 μm. (C) Ce schéma illustre un dispositif Gut-on-a-Chip constitué d’une membrane poreuse placée entre les microcanaux supérieur et inférieur. Le microcanal supérieur est indiqué par la zone bleue, tandis que le microcanal inférieur est indiqué par la zone rouge. Des chambres à vide sont présentes de chaque côté du microcanal, qui déforment la membrane poreuse pour imiter le mouvement péristaltique24. (D) Une installation de Gut-on-a-Chip canin comprend un Gut-on-a-Chip basé sur PDMS assemblé avec un tube qui est placé sur une lamelle de recouvrement 2,24. La tubulure de dérivation est essentielle pour éviter l’accumulation de pression dans le microcanal pendant la manipulation (c’est-à-dire la connexion aux seringues). Des pinces à liant sont utilisées pour serrer le tube. Les matériaux sensibles au volume peuvent être infusés via les trous ouverts de la sortie supérieure ou inférieure. Le milieu de culture organoïde peut être perfusé en connectant les seringues aux aiguilles à extrémité émoussée et en faisant circuler le flux à travers l’entrée supérieure et inférieure. Abréviations : MII = maladie inflammatoire de l’intestin ; PDMS = polydiméthylsiloxane. Veuillez cliquer ici pour voir une version agrandie de cette figure.
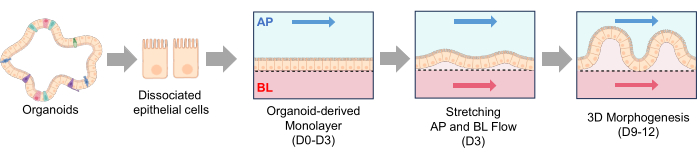
Figure 2 : Formation de structures ressemblant à des villosités dans les MICI canines Gut-on-a-Chip. Les cellules épithéliales dissociées ont été ensemencées dans un Gut-on-a-Chip enrobé d’ECM. Une fois que les cellules dissociées ont été attachées à la membrane PDMS, un flux apical a été initié pendant 3 jours (D0-D3). Lorsqu’une monocouche 2D confluente est formée (D3), un écoulement basolatéral avec étirement fréquent est initié (étirement, AP et BL Flow). Après 2-3 jours de double écoulement et d’étirement de la membrane, la monocouche 2D commence à développer une morphogenèse 3D, et des structures ressemblant à des villosités se forment après 9 jours de culture (morphogenèse 3D, D9-D12). Abréviations : ECM = matrice extracellulaire ; PDMS = polydiméthylsiloxane ; PA = apical ; BL = basolatéral. Veuillez cliquer ici pour voir une version agrandie de cette figure.
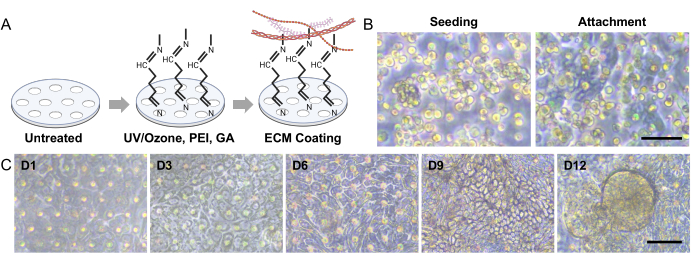
Figure 3 : Ensemencement d’organoïdes intestinaux canins et morphogenèse 3D dans un intestin sur puce. (A) Les étapes expérimentales pour l’activation de surface d’une membrane poreuse dans un Gut-on-a-Chip basé sur PDMS. L’utilisation du traitement UV/Ozone, du PEI et du traitement GA en conjonction facilite la réticulation des amines présentes dans les solutions ECM. Ce processus conduit à l’immobilisation stable des protéines ECM sur la membrane poreuse. (B) Les images de contraste de phase montrent la morphologie des cellules immédiatement après l’ensemencement (à gauche) et 3 à 5 h après l’ensemencement (à droite). La membrane poreuse après 3 h de semis présente des zones plus minces et plus sombres où des cellules individuelles se sont fixées, mettant en évidence le processus de fixation. (C) Les images de contraste de phase illustrent la morphogenèse 3D des monocouches intestinales au sein d’un système Gut-on-a-Chip. Ces monocouches ont été dérivées de chiens atteints de MICI et ces cellules organoïdes ont été cultivées pendant une période de 12 jours dans des conditions dynamiques, ce qui impliquait des mouvements d’écoulement de fluide et d’étirement. Barres d’échelle = 50 μm (B,C). Abréviations : MII = maladie inflammatoire de l’intestin ; ECM = matrice extracellulaire ; PDMS = polydiméthylsiloxane ; PEI = polyéthylénimine ; GA = glutaraldéhyde. Veuillez cliquer ici pour voir une version agrandie de cette figure.
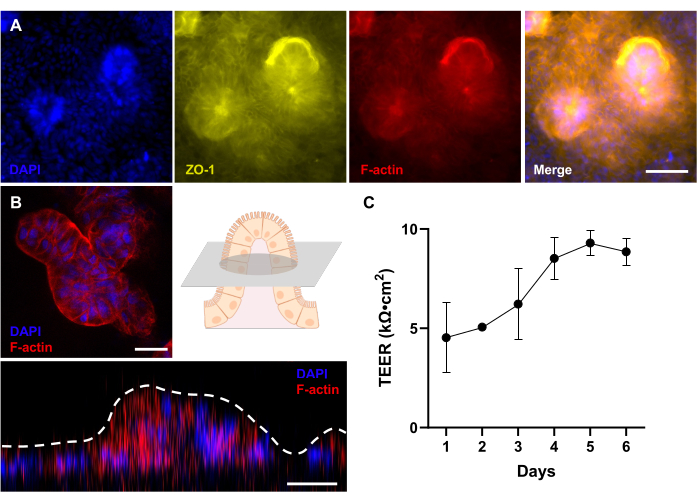
Figure 4 : Évaluation du développement morphologique 3D dans l’intestin sur puce canin dérivé du patient. (A) Imagerie par immunofluorescence d’un intestin sur puce (IBD) canin, montrant une vue descendante d’un épithélium 3D entièrement développé après 12 jours de culture, qui est évalué par un microscope à fluorescence. La protéine de jonction serrée (ZO-1) est visualisée en jaune ; la membrane à bordure en brosse (F-actine) apparaît en rouge ; et les noyaux sont colorés avec du DAPI et apparaissent bleus. (B) Imagerie par immunofluorescence d’une MII canine Gut-on-a-Chip à l’aide d’un microscope confocal avec une lentille à longue distance. Comme le montre le schéma, une image fluorescente d’une coupe transversale d’une structure en forme de villosité d’un épithélium 3D complètement développé après 12 jours de culture est montrée. De plus, l’empilement en Z montre une vue latérale de l’épithélium 3D, qui révèle la formation de structures ressemblant à des villosités. La membrane à bordure en brosse (F-actine) apparaît en rouge et les noyaux sont colorés avec du DAPI et apparaissent en bleu. (C) La fonction de barrière intestinale a été évaluée et mesurée par TEER dans des Gut-on-a-Chips canins dérivés de patients. Des valeurs TEER stables ont été atteintes au jour 5 de la culture sur Gut-on-a-Chip. Les barres d’erreur expriment le MEB des mesures. La valeur FEER a été mesurée sur deux répétitions biologiques avec une répétition technique. Barres d’échelle = 50 μm (A), 25 μm (B). Abréviations : MII = maladie inflammatoire de l’intestin ; DAPI = 4',6-diamidino-2-phénylindole ; TEER = résistance électrique transépithéliale. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire S1 : Caractérisation de l’anticorps polyclonal anti-ZO-1 dans des monocouches dérivées de colonoïdes canins et sur des dispositifs Gut-on-a-Chip. (A) Coloration par immunofluorescence de ZO-1 en jaune avec F-actine en rouge et leur image de superposition. Barre d’échelle = 25 μm. (B) La visualisation par immunofluorescence de ZO-1 en jaune dans des « puces intestinales perméables » a été réalisée dans diverses conditions, y compris la stimulation avec des bactéries probiotiques (LGG + Cytokines ou VSL # 3 + Cytokines) et des contrôles exempts de germes sans stimulation probiotique (Cytokines). Barre d’échelle = 50 μm. Cette figure est reproduite à partir de Min et al.23. Veuillez cliquer ici pour télécharger ce fichier.
Tableau supplémentaire S1 : Sommaire des renseignements sur les donneurs de tissus. Un tableau récapitulatif de l’âge, du sexe, de la race, de l’évaluation histopathologique et de l’indice d’activité des MII canines (CIBDAI) des donneurs. Le CIBDAI est un système de notation numérique utilisé pour déduire la gravité clinique de la MII33 canine. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Cette étude marque la démonstration pionnière de la compatibilité des organoïdes intestinaux canins avec le développement d’un modèle d’intestin sur puce MICI canin. L’intégration d’organoïdes intestinaux et de cultures monocouches dérivées d’organoïdes dans un système microfluidique (c’est-à-dire le système Gut-on-a-Chip) a fait progresser la technologie, permettant la création de modèles intestinaux in vitro qui imitent étroitement la dynamique physiologique et sont plus représentatifs des conditions biologiques. En particulier, étant donné qu’il existe très peu de rapports de culture d’intestins sur puce à l’aide d’organoïdes dérivés de MII chez l’homme, l’étude actuelle utilisant des intestins sur puce dérivés de MII canins peut fournir des informations de premier plan sur l’étude des MII chez l’homme.
Le développement réussi de la morphogenèse 3D de l’épithélium intestinal canin sur un intestin sur puce nécessite une attention particulière à plusieurs étapes critiques. Tout d’abord, la surface hydrophobe des canaux microfluidiques PDMS peut entraver l’adhésion de l’ECM et l’attachement cellulaire ultérieur, ce qui nécessite l’activation de la surface de la PDMS avant l’enrobage de l’ECM et l’ensemencement des cellules (voir la section 1 du protocole). Pour obtenir une culture monocouche stable, l’élimination des cellules non attachées en excès est cruciale après la fixation des cellules (étapes 4.6-4.7 du protocole). De plus, une stimulation dynamique, telle qu’un débit constant du milieu et un mouvement de vide de type péristaltique, est nécessaire pour la morphogenèse 3D de l’épithélium intestinal (étape 5.2 du protocole). Une manipulation prudente est essentielle pour éviter les bulles d’air dans le microcanal lors de toutes les étapes de la culture Gut-on-a-Chip.
Si vous rencontrez un mauvais ensemencement cellulaire dans l’intestin sur puce, cela peut être dû à un faible nombre de cellules ou à une mauvaise fixation des cellules. Pour résoudre le problème du faible nombre de cellules, il est important d’inspecter la santé des organoïdes intestinaux préparés en observant leur croissance dans Matrigel. La viabilité cellulaire peut être évaluée par coloration au bleu de trypan après dissociation cellulaire pour s’assurer que pas plus de 20 % des cellules sont mortes. Si le nombre de cellules viables est insuffisant, il est possible de tenter d’optimiser les conditions du milieu organoïde. Une autre possibilité est la dissociation incomplète des organoïdes, entraînant un excès d’amas cellulaires de plus de 70 μm qui sont piégés par le filtre. Pour résoudre ce problème, une option consiste à prolonger la durée du pipetage pendant la dissociation cellulaire. Alternativement, le tube conique de 15 ml peut être agité doucement toutes les minutes pendant le traitement avec une protéase de type trypsine. Une mauvaise fixation des cellules à l’intestin sur puce peut être due à un revêtement ECM inadéquat. Pendant le processus de revêtement, il est conseillé de vérifier soigneusement la présence de bulles d’air et d’empêcher leur formation en ajoutant doucement plus de solution de revêtement au besoin. L’encombrement des cellules et l’absence de lavage des cellules non attachées peuvent entraîner une monocouche initiale insuffisante. Dans un tel cas, une légère pulsation peut être appliquée en poussant le piston de la seringue. Ces étapes de dépannage peuvent aider à identifier et à résoudre les problèmes au cours du processus de culture Gut-on-a-Chip.
Bien que cette plate-forme Gut-on-a-Chip permette la création de couches épithéliales 3D ondulées, nous reconnaissons la nécessité d’une complexité biologique supplémentaire pour reproduire pleinement le microenvironnement intestinal. Il est crucial de prendre en compte les interactions entre les cellules épithéliales et mésenchymateuses, le dépôt d’ECM pour la régénération 3D et la présence de caractéristiques cryptes-villosités qui établissent une niche cellulaire souche appropriée. Les cellules stromales, telles que les fibroblastes, jouent un rôle essentiel dans la production de protéines ECM et la régulation de la morphogenèse intestinale34,35,36. L’inclusion de cellules mésenchymateuses dans ce modèle a le potentiel d’améliorer à la fois la morphogenèse et l’efficacité de l’attachement cellulaire. Les couches endothéliales, qui englobent le système vasculaire capillaire et les vaisseaux lymphatiques, jouent un rôle crucial dans la régulation du transport moléculaire et le recrutement des cellules immunitaires37,38. L’inclusion de cellules immunitaires dérivées de patients pourrait être essentielle dans la modélisation des maladies intestinales, car elle permet de démontrer l’interaction entre l’immunité innée et l’immunité adaptative, ainsi que l’établissement d’une immunité tissulaire spécifique39. Après l’achèvement de la morphogenèse 3D sur Gut-on-a-Chip, le milieu de culture organoïde peut être modifié en milieu de différenciation organoïde. Cela peut être une approche viable pour induire une différenciation cellulaire supplémentaire, en fonction des objectifs expérimentaux.
L’imagerie de la microarchitecture 3D in situ est difficile en raison de la longue distance de travail requise, qui peut être surmontée avec un objectif à longue distance. De plus, les méthodes de microfabrication et de collage couche par couche rendent difficile l’accès aux couches supérieures pour l’examen par MEB. Pour la conception actuelle de Gut-on-a-Chip, un pousse-seringue par microdispositif Gut-on-a-Chip est nécessaire, ce qui occupe l’espace de l’incubateur deCO2 et empêche les expériences à grande échelle. Des innovations sont nécessaires pour accroître l’évolutivité d’une plateforme conviviale et d’un criblage à haut débit.
Ces protocoles actuels permettent le développement spontané de couches épithéliales 3D in vitro, dépassant les limites des organoïdes 3D traditionnels, des monocouches 2D et des systèmes de culture de microdispositifs statiques. Ce microenvironnement intestinal dynamique in vitro peut être contrôlé par l’introduction de la co-culture de divers types de cellules. Des études antérieures ont exploré des méthodes de manipulation du microenvironnement de l’intestin sur puce, y compris la co-culture du microbiome intestinal14,23 et des cellules mononucléées périphériques30. Ce microenvironnement reconstitué a de nombreuses applications potentielles, notamment les tests de médicaments, les études mécanistiques fondamentales et la modélisation des maladies. Le microenvironnement reconstruit présente un potentiel important pour un large éventail d’applications, telles que les tests de médicaments 23,40,41 et la modélisation des maladies 12,13,14,30, ainsi que les études mécanistiques fondamentales de la morphogenèse intestinale 42. Divers tests peuvent être effectués soit en prélevant des surnageants pour l’évaluation des métabolites 43, soit en prélevant des cellules pour un examen génomique 2,32, soit en examinant visuellement les cellules à l’aide de colorants de cellules vivantes ou d’une fixation pour une imagerie d’immunofluorescence ultérieure 23,44.
Cette étude présente un protocole reproductible pour le développement de la morphogenèse 3D des couches épithéliales intestinales canines dans une plateforme Gut-on-a-Chip. La structure épithéliale 3D qui en résulte fournit une représentation plus réaliste du microenvironnement intestinal, ce qui présente un immense potentiel d’applications dans diverses études biomédicales. En utilisant cette architecture intestinale, nous pouvons mener davantage de recherches translationnelles et potentiellement obtenir des résultats prometteurs.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Nous tenons à remercier le service de médecine interne des petits animaux de la WSU (Dre Jillian Haines, Dre Sarah Guess, Shelley Ensign LVT) et la coordonnatrice des études cliniques de la WSU VTH, Valorie Wiss, pour leur soutien dans le recrutement des cas et la collecte d’échantillons auprès de citoyens scientifiques (patients donneurs). Ce travail a été soutenu en partie par le Bureau du directeur des National Institutes Of Health (K01OD030515 et R21OD031903 à Y.M.A.) et le programme de défi à l’étranger de la Société japonaise pour la promotion de la science à l’étranger (202280196 à l’I.N.). La figure 1A et la figure 3A ont été créées avec BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| Organoid basal medium | |||
| Advanced DMEM/F12 | Gibco | 12634-010 | |
| GlutaMAX | Gibco | 35050-061 | 2 mM, glutamine substitute |
| 1 M HEPES | VWR Life Science | J848-500ML | 10 mM |
| 100x penicillin–streptomycin | Corning | MT30009CI | 1x |
| Organoids and organoid medium | |||
| A-83-01 | PeproTech | 9094360 | 500 nM |
| B27 supplement | Gibco | 17504-044 | 1x |
| CHIR99021 | Reprocell | 04-0004-base | 2.5 µM |
| HEK293 cells engineered to secrete Noggin | Baylor College of Medicine | ||
| Murine EGF | PeproTech | 315-09-1MG | 50 ng/mL |
| Murine Wnt-3a | PeproTech | 315-20-10UG | 100 ng/mL |
| N-Acetyl-L-cysteine | Sigma | A9165-25G | 1 mM |
| N2 MAX Media supplement | Gibco | 17502-048 | 1x |
| Nicotinamide | Sigma | N0636-100G | 10 mM |
| Noggin Conditioned Medium | NA | NA | 10% vol/vol |
| Primocin | InvivoGen | ant-pm-1 | 100 µg/ml |
| R-spondin1 (Rspo1) cells | Trevigen | 3710-001-01 | Rspo1 cells |
| R-Spondin-1 Conditioned Medium | NA | NA | 20% vol/vol |
| SB202190 | Sigma-Aldrich | S7067-25MG | 10 µM |
| Y-27632 | StemCellTechnologies | 72308 | 10 µM |
| [Leu15 ]-Gastrin I human | Sigma-Aldrich | G9145-.5MG | 10 nM |
| Reagents | |||
| 4% Paraformaldehyde solution | Fisher Scientific | AAJ19943K2 | |
| Alexa Fluor 647 Phalloidin | Thermo Fisher Scientific | A22287 | x250 dilution |
| Anti-Rabbit IgG H&L labeled with Alexa Fluor 555 | Abcam | ab150078 | x1,000 dilution |
| Anti-ZO-1 polyclonal antibody | Thermo Fisher Scientific | 61-7300 | x50 dilution |
| Cell Recovery Solution | Corning | 354253 | |
| Collagen I, Rat Tail 3 mg/mL | Gibco | A10483-01 | |
| Diamidino-2-phenylindole (DAPI) | Thermo Fisher Scientific | 62248 | x1,000 dilution |
| EMS Glutaraldehyde Aqueous 50% | Electron Microscopy Sciences | 16320 | |
| Matrigel Matrix | Corning | 356255 | |
| Poly(ethyleneimine) solution | Sigma | 408700-250ML | |
| TrypLE Express | Gibco | 12604-021 | |
| Materials and Equipment | |||
| 24-well culture plates | Corning | 3524 | |
| 87V Industrial Multimeter | Fluke Corporation | ||
| Centrifuge | Eppendorf | 5910R | |
| CO2 incubator | Eppendorf | C170i | |
| DMi8 fluorescence microscope | Leica microsystems | DMi8 | |
| Dry oven | Fisher Scientific | 15-103-0519 | |
| FlexCell FX-5000 Tension system | Flexcell International Corporation | ||
| Inverted phase-contrast microscope | Leica microsystems | DMi1 | |
| SP8-X inverted confocal microscope | Leica microsystems | SP8-X | |
| Syringe pump | Braintree Scientific | model no. BS-8000 120V | |
| Syringe, 3 mL sterile | BD Biosciences | 14-823-435 | |
| Syringes, 1 mL sterile | BD Biosciences | 14-823-434 | |
| UV/ozone generator | Jelight Company | model no. 30 | |
| Software | |||
| LAS X imaging software | Leica microsystems |
Références
- Shanks, N., Greek, R., Greek, J. Are animal models predictive for humans. Philosophy, ethics, and humanities in medicine: PEHM. 4, 2 (2009).
- Shin, W., Kim, H. J. 3D in vitro morphogenesis of human intestinal epithelium in a gut-on-a-chip or a hybrid chip with a cell culture insert. Nature protocols. 17 (3), 910-939 (2022).
- Coelho, L. P., et al. Similarity of the dog and human gut microbiomes in gene content and response to diet. BMC Biome. 6 (72), 1-11 (2018).
- German, A. J. The growing problem of obesity in dogs and cats. The Journal of Nutrition. 136, 1940-1946 (1940).
- Patronek, G. J., Waters, D. J., Glickman, L. T. Comparative longevity of pet dogs and humans: Implications for gerontology research. Journals of Gerontology - Series A Biological Sciences and Medical Sciences. 52 (3), B171-B178 (1997).
- Lutz, T. A. Mammalian models of diabetes mellitus, with a focus on type 2 diabetes mellitus. Nature reviews. Endocrinology. 19 (6), 350-360 (2023).
- Allenspach, K., Culverwell, C., Chan, D. Long-term outcome in dogs with chronic enteropathies: 203 cases. Veterinary Record. 178 (15), 368 (2016).
- Patnaik, A. K., Hurvitz, A. I., Johnson, G. F. Canine gastrointestinal neoplasms. Veterinary Pathology. 14 (6), 547-555 (1977).
- Saito, T., et al. Immunohistochemical analysis of beta-catenin, e-cadherin and p53 in canine gastrointestinal epithelial tumors. Journal of Veterinary Medical Science. 82 (9), 1277-1286 (2020).
- Kopper, J. J., et al. Harnessing the biology of canine intestinal organoids to heighten understanding of inflammatory bowel disease pathogenesis and accelerate drug discovery: A One Health approach. Frontiers in Toxicology. 3, 1-13 (2021).
- Jalili-firoozinezhad, S., et al. A complex human gut microbiome cultured in an anaerobic intestine-on-a-chip. Nature Biomedical Engineering. 3 (7), 520-531 (2019).
- Shin, W., et al. Robust formation of an epithelial layer of human intestinal organoids in a polydimethylsiloxane-based Gut-on-a-Chip microdevice. Frontiers in Medical Technology. 2, (2020).
- Shin, Y. C., et al. Three-dimensional regeneration of patient-derived intestinal organoid epithelium in a physiodynamic mucosal Interface-on-a-Chip. Micromachines. 11 (7), 663 (2020).
- Tovaglieri, A., et al. Species-specific enhancement of enterohemorrhagic E. coli pathogenesis mediated by microbiome metabolites. Microbiome. 7 (1), 43 (2019).
- Chandra, L., et al. Derivation of adult canine intestinal organoids for translational research in gastroenterology. BMC Biology. 17 (1), 1-21 (2019).
- Sato, T., et al. Long-term expansion of epithelial organoids from human colon, adenoma, adenocarcinoma, and Barrett's epithelium. Gastroenterology. 141 (5), 1762-1772 (2011).
- Wilson, S. S., Tocchi, A., Holly, M. K., Parks, W. C., Smith, J. G. A small intestinal organoid model of non-invasive enteric pathogen-epithelial cell interactions. Mucosal Immunology. 8 (2), 352-361 (2015).
- Ambrosini, Y. M., et al. Recapitulation of the accessible interface of biopsy-derived canine intestinal organoids to study epithelial-luminal interactions. PLoS ONE. 15 (4), 1-17 (2020).
- Gabriel, V., et al. Canine intestinal organoids in a dual-chamber permeable support system. Journal of Visualized Experiments. 2 (181), 1-24 (2022).
- Yamada, A., et al. Confronting emerging zoonoses: the one health paradigm. Confronting Emerging Zoonoses: The One Health Paradigm. , 1 (2014).
- Lerner, H., Berg, C. The concept of health in One Health and some practical implications for research and education: what is One Health. Infection Ecology & Epidemiology. 5 (1), 25300 (2015).
- Garcia, S. N., Osburn, B. I., Jay-Russell, M. T. One Health for food safety, food security, and sustainable food production. Frontiers in Sustainable Food Systems. 4, 1-9 (2020).
- Min, S., et al. Live probiotic bacteria administered in a pathomimetic Leaky Gut Chip ameliorate impaired epithelial barrier and mucosal inflammation. Scientific Reports. 12 (1), 22641 (2022).
- Kim, H. J., Huh, D., Hamilton, G., Ingber, D. E. Human gut-on-a-chip inhabited by microbial flora that experiences intestinal peristalsis-like motions and flow. Lab on a Chip. 12 (12), 2165-2174 (2012).
- Qu, M., et al. Establishment of intestinal organoid cultures modeling injury-associated epithelial regeneration. Cell Research. 31 (3), 259-271 (2021).
- Sahoo, D. K., et al. Differential transcriptomic profiles following stimulation with lipopolysaccharide in intestinal organoids from dogs with inflammatory bowel disease and intestinal mast cell tumor. Cancers. 14 (14), 3525 (2022).
- Gabriel, V., et al. Standardization and maintenance of 3D canine hepatic and intestinal organoid cultures for use in biomedical research. Journal of Visualized Experiments. (179), 1-28 (2022).
- Heijmans, J., et al. ER stress causes rapid loss of intestinal epithelial stemness through activation of the unfolded protein response. Cell reports. 3 (4), 1128-1139 (2013).
- Shin, W., et al. A robust longitudinal co-culture of obligate anaerobic gut microbiome with human intestinal epithelium in an anoxic-oxic interface-on-a-chip. Frontiers in Bioengineering and Biotechnology. 7, 1-13 (2019).
- Shin, W., Kim, H. J. Intestinal barrier dysfunction orchestrates the onset of inflammatory host-microbiome cross-talk in a human gut inflammation-on-a-chip. Proceedings of the National Academy of Sciences of the United States of America. 115 (45), E10539-E10547 (2018).
- Park, G. S., et al. Emulating host-microbiome ecosystem of human gastrointestinal tract in vitro. Stem Cell Reviews and Reports. 13 (3), 321-334 (2017).
- Kim, H. J., Li, H., Collins, J. J., Ingber, D. E. Contributions of microbiome and mechanical deformation to intestinal bacterial overgrowth and inflammation in a human gut-on-a-chip. Proceedings of the National Academy of Sciences of the United States of America. 113 (1), E7-E15 (2016).
- Jergens, A. E., et al. A scoring index for disease activity in canine inflammatory bowel disease. Journal of Veterinary Internal Medicine. 17 (3), 291-297 (2003).
- Roulis, M., Flavell, R. A. Fibroblasts and myofibroblasts of the intestinal lamina propria in physiology and disease. Differentiation; research in biological diversity. 92 (3), 116-131 (2016).
- Göke, M., Kanai, M., Podolsky, D. K. Intestinal fibroblasts regulate intestinal epithelial cell proliferation via hepatocyte growth factor. The American Journal of Physiology. 274 (5), G809-G818 (1998).
- Powell, D. W., Pinchuk, I. V., Saada, J. I., Chen, X., Mifflin, R. C. Mesenchymal cells of the intestinal lamina propria. Annual Review of Physiology. 73, 213-237 (2011).
- Pappenheimer, J. R., Michel, C. C. Role of villus microcirculation in intestinal absorption of glucose: coupling of epithelial with endothelial transport. The Journal of Physiology. 553, 561-574 (2003).
- Agace, W. W. T-cell recruitment to the intestinal mucosa. Trends in Immunology. 29 (11), 514-522 (2008).
- Ambrosini, Y. M., Shin, W., Min, S., Kim, H. J. Microphysiological engineering of immune responses in intestinal inflammation. Immune Network. 20 (2), 13 (2020).
- Kasendra, M., et al. Development of a primary human Small Intestine-on-a-Chip using biopsy-derived organoids. Scientific Reports. 8 (1), 1-14 (2018).
- Sontheimer-Phelps, A., et al. Human Colon-on-a-Chip enables continuous in vitro analysis of colon mucus layer accumulation and physiology. Cellular and Molecular Gastroenterology and Hepatology. 9 (3), 507-526 (2020).
- Shin, W., et al. Human intestinal morphogenesis controlled by transepithelial morphogen gradient and flow- dependent physical cues in a microengineered Gut-on-a-Chip. iScience. 15, 391-406 (2019).
- Gijzen, L., et al. An Intestine-on-a-Chip model of plug-and-play modularity to study inflammatory processes. SLAS Technology. 25 (6), 585-597 (2020).
- Kim, H. J., Lee, J., Choi, J. H., Bahinski, A., Ingber, D. E. Co-culture of living microbiome with microengineered human intestinal villi in a gut-on-a-chip microfluidic device. Journal of Visualized Experiments. (114), 3-9 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon