Method Article
Analyse de la formation de complexes protéiques à des concentrations micromolaires par couplage de la microfluidique avec la photométrie de masse
Dans cet article
Résumé
Ce protocole combine la photométrie de masse avec un nouveau système microfluidique pour étudier les interactions protéine-protéine de faible affinité. Cette approche est basée sur la dilution rapide de complexes hautement concentrés en solution, ce qui permet des mesures de faible affinité et élargit l’applicabilité de la photométrie de masse.
Résumé
La photométrie de masse est une technologie polyvalente de mesure de masse qui permet d’étudier les interactions biomoléculaires et la formation complexe en solution sans étiquettes. La photométrie de masse est généralement adaptée à l’analyse d’échantillons dans la gamme de concentration 100 pM-100 nM. Cependant, dans de nombreux systèmes biologiques, il est nécessaire de mesurer des échantillons plus concentrés pour étudier les interactions de faible affinité ou transitoires. Ici, nous démontrons une méthode qui élargit efficacement la gamme de concentrations d’échantillons pouvant être analysées par photométrie de masse, de la nanomolaire à des dizaines de micromolaires.
Dans ce protocole, la photométrie de masse est combinée à un nouveau système microfluidique pour étudier la formation de complexes protéiques en solution dans la gamme de concentration micromolaire. Avec le système microfluidique, les utilisateurs peuvent maintenir un échantillon à une concentration plus élevée souhaitée, suivie d’une dilution à l’ordre nanomolaire - plusieurs millisecondes avant la mesure par photométrie de masse. En raison de la vitesse de dilution, les données sont obtenues avant que l’équilibre de l’échantillon ne se soit déplacé (c’est-à-dire la dissociation du complexe).
La technique est appliquée pour mesurer les interactions entre un anticorps d’immunoglobuline G (IgG) et le récepteur Fc néonatal, montrant la formation de complexes d’ordre élevé qui n’étaient pas quantifiables avec des mesures de photométrie de masse statique.
En conclusion, la combinaison de la photométrie de masse et de la microfluidique permet de caractériser des échantillons dans la gamme de concentration micromolaire et est efficace pour mesurer les interactions biomoléculaires avec des affinités plus faibles. Ces capacités peuvent être appliquées dans une gamme de contextes, y compris le développement et la conception de produits biothérapeutiques, permettant une caractérisation approfondie de diverses interactions protéine-protéine.
Introduction
Les interactions protéine-protéine sous-tendent la plupart des fonctions cellulaires, de la régulation immunitaire à la réplication et à la traduction de l’ADN. Par conséquent, il existe un besoin fondamental dans l’ensemble des sciences de la vie d’étudier un vaste éventail d’interactions à travers divers complexes hétérogènes qui se forment couramment. Cependant, leur détection, leur caractérisation et leur quantification sont souvent difficiles, en particulier pour les interactions de faible affinité1.
Les tests d’immunoprécipitation sont souvent utilisés pour détecter les interactions de haute affinité, mais pour les interactions de faible affinité et transitoires, la détection est largement irréalisable2. Des techniques de fluorescence peuvent également être utilisées, mais nécessitent l’ajout potentiellement perturbateur de marqueurs fluorescents2. La cryo-EM peut fournir un instantané structurel et une lecture d’ensemble des complexes protéiques formés avec une résolution spatiale élevée, mais nécessite également de travailler à des concentrations trop faibles pour l’imagerie d’interactions de faible affinité. La cryo-EM pose également des défis liés au coût, à l’accessibilité, à la préparation des échantillons et au temps d’analyse3.
De plus, la résonance plasmonique de surface (SPR) est devenue un moyen populaire de quantifier les interactions protéine-protéine, bien qu’elle nécessite une immobilisation des protéines, ce qui peut affecter l’équilibre de liaison et entraîner des taux d’on-on variables, réduisant ainsi la précision de la mesure 4,5. Elle implique également plusieurs étapes d’analyse avant la collecte et l’analyse des données6.
La photométrie de masse est une technique à molécule unique qui a été utilisée pour analyser les interactions protéine-protéine 5,6,7. Il fonctionne en mesurant la masse de molécules uniques ou de complexes en fonction de la lumière qu’elles diffusent lorsqu’elles atterrissent sur la surface d’une lamelle de verre8. Des mesures par photométrie de masse ont été utilisées pour quantifier les affinités de liaison à partir de l’abondance relative des partenaires de liaison et des complexes qu’ils forment5. Néanmoins, comme d’autres techniques à molécule unique, la concentration de l’échantillon à mesurer doit généralement être inférieure à 100 nM. Si la concentration est plus élevée, les molécules atterrissant sur la surface du verre se chevaucheront spatialement, ce qui entraînera une mauvaise qualité des données7. Par conséquent, les interactions plus faibles (KD ~ micromolaires), qui se dissocient à ces concentrations plus faibles, ne peuvent pas être mesurées de manière fiable car il n’est pas possible d’observer le mélange nécessaire d’espèces non liées et liées5.
Ici, nous décrivons une approche qui surmonte cette limitation basée sur un nouveau dispositif de photométrie de masse microfluidique couplé. Plus précisément, un système microfluidique est utilisé en combinaison avec le photomètre de masse pour élargir efficacement la gamme d’interactions pouvant être quantifiées par la photométrie de masse. Il a été démontré que la microfluidique offre un éventail de possibilités pour étudier les interactions protéine-protéine, y compris la dilution rapide pour détecter les interactions faibles 1,9. Le système décrit ici fonctionne en diluant rapidement l’échantillon jusqu’à 10 000 fois sur une puce microfluidique et en le faisant circuler immédiatement à travers la zone d’observation de la puce, ce qui permet à la mesure par photométrie de masse de commencer dans les 50 ms à partir du moment où les molécules ont commencé le processus de dilution10. La dilution se produit lorsque l’échantillon et le tampon sont combinés dans un mélangeur à valve Tesla inversée sur la puce, les débits relatifs des deux solutions déterminant la quantité de dilution qui se produit (voir l’étape 8 du protocole). Le débit est contrôlable à l’aide du logiciel de contrôle microfluidique. La modification du débit peut modifier la population relative de l’espèce, car elle peut avoir un impact sur le nombre d’atterrissages à la surface du verre, qui est mesuré par le photomètre de masse.
La vitesse du processus est suffisamment rapide pour que la mesure soit terminée avant que l’intégrité de l’interaction ne soit perturbée (pour plus de détails, voir également la discussion). Cela peut être compris à travers un bref regard sur la théorie des réactions du premier ordre, où 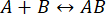 . La constante de vitesse directe (association) est kf, la constante de vitesse arrière (dissociation) est kb et la constante de dissociation d’équilibre (KD) est définie comme suit :
. La constante de vitesse directe (association) est kf, la constante de vitesse arrière (dissociation) est kb et la constante de dissociation d’équilibre (KD) est définie comme suit :
KD= kb/ kf
Pour la liaison aux protéines, kfest généralement limité par la diffusion des réactifs11 et est donc limité à la gamme de 106-10 7 M-1·s-1. Parce que la gamme de est limitée, une réaction de faible affinité (KD~micromolaires) aura kb≈ 1 s-1. C’est-à-dire, kb= kf · KD= (106 M-1·s-1) (10-6 M) = 1 s-1, avec une demi-vie du complexe d’environ 0,7 s11,12.
Notre exemple de système est la liaison de l’anticorps monoclonal IgG trastuzumab au domaine soluble du récepteur Fc néonatal IgG (FcRn), qui sont des partenaires d’interaction connus13. Des données précédemment publiées obtenues en utilisant uniquement la photométrie de masse conventionnelle (c’est-à-dire avec une dilution manuelle des échantillons) ont montré que les protéines forment plusieurs espèces. Les monomères FcRn, les dimères FcRn et les IgG non liées étaient clairement visibles, tandis que les complexes IgG-FcRn (à des rapports de 1:1 et 1:2) ont également été détectés (à un pH de 5,0), mais seulement avec une très faible abondance5. Cette observation soulève la question de savoir si la formation du complexe IgG-FcRn pourrait être plus clairement détectée si elle était mesurée à une concentration plus élevée. En effet, la combinaison de la photométrie de masse avec une approche de dilution rapide couplée décrite ici a fourni des preuves plus robustes de la formation complexe par une augmentation de leurs particules mesurées.
Le protocole de photométrie de masse et de microfluidique décrit ici permet de caractériser la formation de complexes avec un KD jusqu’à la gamme micromolaire. Une détermination empirique du KD nécessitera d’autres améliorations de la précision du capteur de débit, de la stabilité de la pompe, des variations d’une puce à l’autre et de l’emplacement de la mesure à l’intérieur de la fenêtre d’observation, car tous ces facteurs influenceraient le temps écoulé entre le moment où l’échantillon est dilué et celui où il est mesuré.
La même approche pourrait être appliquée pour étudier la liaison entre toutes les protéines solubles, à condition qu’elles aient des poids moléculaires distincts (séparés par au moins 25 kDa) qui se situent dans la plage appropriée pour l’analyse avec un photomètre de masse (30 kDa à 6 MDa). Les informations obtenues pourraient être utiles pour des études dans divers contextes, allant de l’acquisition d’une compréhension mécaniste des fonctions cellulaires à la conception de nouveaux médicaments biothérapeutiques.
Protocole
1. Préparation des instruments et lancement du logiciel
- Allumez le photomètre de masse et laissez-le allumé pendant au moins 1 h avant de commencer les mesures.
REMARQUE : En effet, l’instrument doit atteindre une température constante. Ne pas laisser le photomètre de masse allumé pendant 1 h peut entraîner un décalage de masse et, par conséquent, des résultats inexacts. - Allumez la table anti-vibration en la mettant sous tension et en appuyant sur le bouton d’isolement (sous le photomètre de masse). Les vibrations limitent les performances de l’instrument de photométrie de masse, c’est pourquoi la table anti-vibration est importante pour assurer une sensibilité maximale de l’instrument. Allumez le boîtier de microfluidique.
- Lancer le logiciel d’acquisition de données et le logiciel de contrôle microfluidique. Allumez le compresseur d’air.
2. Préparation d’échantillons de protéines, de tampons et de solutions de nettoyage
- Ajoutez 200 ml de tampon PBS (pH 7,4) dans une bouteille propre de 200 ml et 200 ml de tampon PBS (pH 5,0) dans une deuxième bouteille de 200 ml.
REMARQUE : Les bouteilles de 200 ml devront être connectées au capteur d’unité de débit « L » dans les étapes suivantes. Utilisez un tampon PBS pH 7,4 pour la mesure de l’étalonnage et un tampon PBS pH 5,0 pour la mesure de l’échantillon. - Dans un tube à centrifuger de 0,5 mL, mélanger les IgG (2 μM) et les FcRn (20 μM) dans un rapport de 1:10 pour un volume total de 60 μL.
- Les solutions mères pour FcRn et IgG sont respectivement de 91,9 μM et 13,5 μM. Préparez la réaction en mélangeant 9 μL d’IgG de base + 13 μL de FcRn de base + 38 μL de PBS (pH 5,0, température ambiante [RT]) dans un tube à centrifuger pour un volume total de 60 μL. Les concentrations finales pour les deux réactifs étaient de 2 μM pour les IgG et de 20 μM pour le FcRn. Gardez tous les stocks de protéines sur la glace tout au long de ce processus.
- Dans un autre tube à centrifuger de 0,5 mL, distribuer 20 μL de calibrant β-Amylase à une concentration de 20 μM.
- À titre d’exemple, pour préparer l’étalon β-Amylase utilisé ici, suivez la procédure décrite aux points 2.3.1.1 et 2.3.1.2.
- Mettre à nouveau en suspension 20 mg de poudre de β-amylase dans le flacon dans 3,57 mL de PBS (glycérol à 5 %), ce qui correspond à 5,6 mg/mL et à une concentration molaire de 100 μM (le poids moléculaire étant de 56 kDa).
- Pour diluer à 20 μM, combinez 200 μL de la pâte 100 μM avec 800 μL de PBS (pH 7,4, RT) dans un tube à centrifuger de 1 mL.
- À titre d’exemple, pour préparer l’étalon β-Amylase utilisé ici, suivez la procédure décrite aux points 2.3.1.1 et 2.3.1.2.
- Incuber les échantillons à RT pendant au moins 30 min.
REMARQUE : Commencez à préparer les dilutions de l’échantillon et de l’étalonnage après avoir allumé le photomètre de masse. Pour cette expérience, les échantillons ont été incubés pendant 120 min. - Dans trois tubes à centrifuger de 50 mL, aliquote : 50 mL de PBS (pH 7,4) - solution de nettoyage 1 (CS1), 50 mL de NaOH 0,5 M - solution de nettoyage 2 (CS2) et 50 mL de solution de nettoyage 100 % IPA - solution de nettoyage 3 (CS3).
3. Montage expérimental
- Pour charger l’échantillon et l’étalon, placez le tube à centrifuger de l’étalonnage à la position 4 et le tube à centrifuger de l’échantillon à la position 5 de la boîte de microfluidique (Figure 1).
- Pour charger les solutions de nettoyage, placez CS1, CS2 et CS3 aux positions 1, 2 et 3 de la boîte de microfluidique (Figure 1).
REMARQUE : L’échantillon, l’étalonnage et les solutions de nettoyage sont connectés au multi-interrupteur (commutateur m). Le m-switch est connecté au capteur de l’unité de débit « S ». - Vissez le bouchon qui se connecte à la ligne tampon sur la bouteille tampon de 200 ml (pH 7,4).
REMARQUE : La ligne tampon est connectée à une vanne d’arrêt, qui est connectée au capteur de l’unité de débit « L ». La vanne d’arrêt empêche tout effet de siphonnage qui peut se produire lorsque le flux tampon est arrêté. Le siphonnage peut entraîner la contamination du tampon par le fluide résiduel dilué qui retourne dans le réservoir tampon. - Placez la puce microfluidique sur une plaque de préparation.
Poussez l’autre extrémité du tube du capteur de l’unité de débit « S » dans l'« entrée de l’échantillon » du premier canal de la puce microfluidique (Figure 1). La ligne d’échantillonnage est terminée. - Poussez l’autre extrémité du tube du capteur de l’unité de débit « L » dans l'« entrée tampon » du premier canal de la puce microfluidique (Figure 1). La ligne tampon est terminée.
- Pour recueillir l’écoulement, poussez un tube jusqu’à la « sortie » du premier canal de la puce microfluidique ; positionnez l’autre extrémité de manière à ce qu’elle s’écoule dans une fiole ou un bécher (figure 1).
4. Amorçage des lignes d’échantillonnage et de tampon avec un tampon
- Ouvrez la vanne d’arrêt manuelle sur la conduite tampon.
- Dans le logiciel de contrôle microfluidique, réglez le débit de la ligne tampon à 1000 μL/min et assurez-vous que la pression de la ligne tampon ne dépasse pas 110 mbar (Figure 2). Le flux démarre automatiquement dans la ligne tampon (Figure 1).
REMARQUE : Si la pression de la ligne tampon dépasse 110 mbar, cela peut être dû à l’air passant à travers le capteur de débit. Si la pression ne revient pas à la normale en quelques secondes, il y a potentiellement un blocage (généralement dû à un tube pincé au niveau des connexions). Dans ce cas, arrêtez le débit et vérifiez les connexions dans la ligne tampon, en commençant par la vanne d’arrêt. - Dans le logiciel de contrôle microfluidique, sélectionnez la position 1 de l’interrupteur m (correspondant au tampon PBS), puis réglez le débit de la ligne d’échantillonnage à 8 μL/min et assurez-vous que la pression de la ligne d’échantillonnage ne dépasse pas 350 mbar. Le flux démarre automatiquement dans la ligne d’échantillonnage (Figure 1).
- À l’aide d’une pointe de pipette (ou d’un autre composant en plastique souple), appliquez une légère pression par le haut près de la ou des bulles piégées dans la puce afin de déloger les bulles d’air et de vous assurer qu’elles sont éliminées par la sortie.
REMARQUE : Assurez-vous d’éliminer toutes les bulles dans les sections « mélangeur » et « zone d’observation » (Figure 1) du canal utilisé. Veillez à ne pas pousser trop fort, car cela pourrait endommager la puce.
5. Placer la puce microfluidique sur le photomètre de masse et trouver la mise au point
- Appliquez une goutte d’huile d’immersion pour microscope sur l’objectif du photomètre de masse.
- Placez la puce microfluidique sur le support du photomètre de masse avec l'« entrée d’échantillon » vers le haut et maintenez-la fixée aux pinces de platine (Figure 1). Assurez-vous que tous les raccords de tube restent attachés.
- À l’aide du logiciel d’acquisition de données, déplacez la platine pour vous assurer que la « zone d’observation » du canal 1 est alignée avec l’objectif (Figure 1).
- Fermez le couvercle du photomètre de masse et appuyez sur l’option Dilution des gouttelettes Trouver la mise au point dans le logiciel d’acquisition de données.
- Vérifiez la bague de mise au point blanche dans le coin inférieur gauche du logiciel d’acquisition de données (Figure 3). Les trous dans l’anneau indiquent la présence d’une bulle d’air dans l’huile d’immersion ; Éliminez-le en augmentant la vitesse de la platine au maximum et en déplaçant doucement la platine latéralement.
- Une fois la mise au point terminée, attendez 2-3 minutes avant de prendre le premier enregistrement. Ensuite, appuyez sur Enregistrer pour enregistrer une mesure de 1 min et assurez-vous (en observant la mesure) qu’aucune impureté n’apparaît.
REMARQUE : Des impuretés peuvent se trouver sur la surface du verre ou dans le tampon. La valeur de netteté dans le logiciel d’acquisition de données doit être supérieure à 4,5 %.
6. Étalonnage par photométrie de masse
- Dans le logiciel de contrôle microfluidique, mettez l’interrupteur m en position 4 (correspondant à l’étalon) et assurez-vous que le débit de la ligne d’échantillonnage est réglé à 8 μL/min et que la pression de la ligne d’échantillonnage ne dépasse pas 350 mbar (Figure 2).
- L’étalon commencera à s’écouler à travers la puce. Attendez environ 1,5 à 2,5 minutes (ou jusqu’à ce que l’étalonnage soit visible de manière cohérente sur le logiciel d’acquisition de données). Le temps peut varier en fonction de la longueur du tube de la conduite d’échantillonnage.
- Une fois que l’étalonnage est observé de manière constante (c’est-à-dire que le nombre d’événements est suffisant pour une mesure précise de la photométrie de masse), dans le logiciel de contrôle de la microfluidique, réduisez le débit à 0,5 μL/min - le niveau de dilution cible pour cette expérience.
REMARQUE : Commencer avec un débit plus élevé réduit simplement le temps nécessaire au calibrateur pour atteindre la puce. - Assurez-vous qu’il n’y a pas « trop peu » ou « trop » de molécules qui atterrissent sur la surface de mesure (Figure 4).
- Si la densité de l’événement d’atterrissage n’est pas « idéale » (Figure 4), modifiez le débit dans le logiciel de contrôle de la microfluidique. Si la densité d’événements est trop faible, augmenter le débit de l’échantillon jusqu’à ce que des événements d’atterrissage bien séparés soient observés (mais ne dépassent pas 8 μL/min). S’il est trop élevé, réduisez le débit de l’échantillon (mais ne descendez pas en dessous de 0,1 μL/min).
- Si le volume d’étalonnage est faible dans le tube d’échantillon, changez la position de l’interrupteur m sur la ligne PBS pH 7.4 (position 1) pour éviter d’injecter de l’air.
- Appuyez sur Enregistrer et prenez une mesure de 60 s. Enregistrez le fichier dans le dossier de votre choix.
REMARQUE : Le logiciel d’acquisition de données produit des fichiers avec l’extension .mp.
7. Nettoyage de la ligne d’échantillonnage et arrêt de l’écoulement
- Dans le logiciel de contrôle microfluidique, passez à la position 2 de l’interrupteur m et modifiez la pression de l’échantillon à 800 mbar (Figure 2). Rincez le système avec du CS2 (NaOH) pendant 4 min.
- Passez à la position 3 de l’interrupteur m et rincez le système avec CS3 (IPA) pendant 4 min.
- Passez à la position 1 de l’interrupteur m et rincez le système avec CS1 (PBS) pendant 4 min.
- Dans le logiciel de contrôle microfluidique, arrêtez tout débit en réglant les pressions de la ligne d’échantillonnage et de la ligne tampon sur 0 et fermez la vanne tampon.
- Déconnectez la puce de la platine et replacez-la sur la plaque de préparation. Débranchez tous les tubes et placez l’extrémité du tube d’échantillon dans une bouteille de déchets. Nettoyez l’objectif avec de l’isopropanol et des lingettes.
8. Mesure de l’échantillon de photométrie de masse
- Dévissez le bouchon relié à la bouteille tampon de 200 ml (pH 7,4) et vissez-le à la bouteille tampon de 200 ml (pH 5,0).
- Répétez les étapes 3.4 à 5.6, mais utilisez le deuxième canal de la puce au lieu du premier.
- Dans le logiciel de contrôle microfluidique, mettez l’interrupteur m en position 5 (correspondant à l’échantillon), réglez le débit de la ligne d’échantillonnage à 8 μL/min et assurez-vous que la pression de la ligne d’échantillonnage ne dépasse pas 350 mbar (Figure 2).
- Répétez les étapes 6.2 à 6.4 pour mesurer l’échantillon.
- Pour calculer les débits à utiliser, assurez-vous que la différence de pli du débit correspond au facteur de dilution de l’échantillon souhaité. Par exemple, pour obtenir la dilution de 2000x ici, les débits diffèrent d’un facteur 2000 ; le débit tampon est de 1000 μL/min et le débit d’échantillon est de 0,5 μL/min. À la fin d’une expérience, laissez toujours les lignes propres en suivant le protocole de nettoyage.
9. Analyse des données
- Une fois l’acquisition des données terminée, lancez le logiciel d’analyse de données (Figure 5).
- Cliquez sur l’icône plus (+) en haut à gauche et sélectionnez le fichier d’étalonnage .mp. Le logiciel commencera à analyser le fichier chargé. Selon la taille et le nombre de mesures, cela peut prendre quelques minutes
- N’analysez pas les fichiers .mp dans le logiciel d’analyse de données lors de l’acquisition de données à l’aide d’un logiciel d’acquisition de données, car cela pourrait réduire la qualité des données acquises.
- Appuyez sur le bouton Créer un étalonnage de masse (en bas à droite). Une boîte de dialogue s’ouvrira et affichera un tableau rempli avec les valeurs de contraste des pics ajustés.
- Remplacez les valeurs par les valeurs de masse connues pour les pics ajustés (pour la β-amylase, ces valeurs sont 56 kDa, 112 kDa et 224 kDa) et appuyez sur Enregistrer. Le fichier d’étalonnage nouvellement créé (extension .mc) s’affiche sur le panneau d’étalonnage de masse (en bas à gauche du logiciel d’analyse de données) (Figure 6).
- Cliquez sur l’icône plus (+) en haut à gauche et sélectionnez le fichier d’exemple .mp.
- Pour créer un histogramme de masse, comme illustré à la figure 5, allez dans l’onglet Analyse , sélectionnez le mode Histogramme et l’option Tracé de masse . Ajustez la largeur du bac, les limites de masse et d’autres paramètres selon vos besoins.
- Personnalisez davantage le tracé dans l’onglet Figures si vous le souhaitez avant d’exporter des figures et/ou d’enregistrer l’ensemble de l’espace de travail en tant que fichier .dmp.
Résultats
La photométrie de masse a été utilisée pour mesurer l’interaction entre l’anticorps monoclonal IgG trastuzumab et le domaine soluble du récepteur Fc néonatal IgG (FcRn). Un mélange 1:10 des deux protéines (à 2 μM pour les IgG et 20 μM pour FcRn) a été dilué manuellement à 10 nM et 20 nM dans le PBS, respectivement.
L’étape de dilution est nécessaire car la photométrie de masse, une technique de mesure de masse à molécule unique, ne peut analyser que des échantillons dans la gamme 100 pM-100 nM. Tenter de mesurer des échantillons dont les concentrations se situent à l’extérieur de cette plage peut compromettre l’exactitude des résultats (figure 4). Cette expérience n’est pas incluse dans le présent Protocole, car elle a été décrite précédemment 5,6.
Dans les histogrammes de masse produits dans une expérience de photométrie de masse, l’intensité du signal de diffusion (ou « contraste ») pour chaque événement d’atterrissage est tracée sur l’axe des x, et elle est convertie en masse moléculaire via l’étape d’étalonnage masse-contraste. Pendant ce temps, l’axe des y indique le nombre de molécules comptées avec une masse donnée (ou contraste). Par conséquent, un pic indique la présence d’une population de molécules dans la plage des poids moléculaires indiquée sur l’axe des x. Le nombre d’événements (dénombrements) qui composent le pic reflète la taille de cette population.
La mesure par photométrie de masse avec dilution manuelle a permis d’obtenir un histogramme de masse où les deux pics les plus importants, basés sur les poids moléculaires attendus des protéines, correspondaient à des monomères FcRn non liés (~50 kDa) et à des monomères d’anticorps IgG (~150 kDa) (figure 7). À l’instar des données de photométrie de masse publiées précédemment5, les espèces non liées étaient proéminentes, tandis que les pics dans les gammes de masse qui correspondraient à des complexes étaient beaucoup moins apparents.
L’expérience a été répétée à l’aide d’un système microfluidique à dilution rapide pour diluer le mélange à la concentration nécessaire immédiatement avant la mesure par photométrie de masse. Cette approche a permis de détecter d’autres complexes de faible affinité, qui peuvent s’être dissociés lors de l’étape de dilution manuelle. Pour obtenir le même facteur de dilution de 2000x que celui utilisé lors de l’expérience de dilution manuelle, le mélange d’échantillons 1:10 (à une concentration de 2 μM pour les IgG et de 20 μM pour le FcRn) a été écoulé sur la puce microfluidique à un débit de 0,5 μL/min, avec un tampon PBS (pH 5,0) à un débit de 1 mL/min. Pour s’assurer que les protéines n’étaient pas dégradées au moment de la mesure, les IgG (2 μM) et FcRn (20 μM) ont été mesurés en flux avec le système microfluidique après 120 min d’incubation des échantillons. Les mesures individuelles des témoins n’ont montré aucune dégradation des protéines (figure supplémentaire 1)
Pour l’échantillon qui a subi une dilution rapide, les pics correspondant aux monomères FcRn (53 kDa), aux dimères FcRn (97 kDa) et aux monomères IgG (148 kDa) ont de nouveau pu être observés. De plus, deux pics supplémentaires ont été clairement observés à 196 kDa et 251 kDa, correspondant à des complexes IgG-FcRn avec des stœchiométries 1:1 et 1:2 (Figure 7). Les masses attendues pour ces deux complexes étaient respectivement de 200 kDa et 250 kDa. La variabilité de la mesure de la masse se situe à l’intérieur de l’erreur de mesure pour le photomètre de masse utilisé dans cette étude est de ±5 %14 (2 % pour le complexe 1:1 et 0,4 % pour le complexe 1:2).
La présence de ces complexes dans l’échantillon rapidement dilué uniquement est cohérente avec l’idée qu’ils ont tendance à se dissocier à des concentrations plus faibles, à une vitesse rapide par rapport à un processus de dilution manuelle, mais lente par rapport au processus de dilution rapide obtenu avec le système microfluidique15.
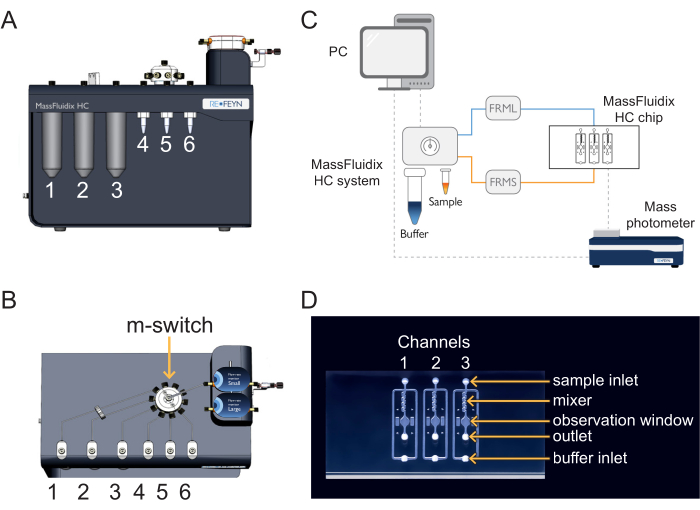
Figure 1 : Combinaison de la microfluidique et de la photométrie de masse. (A,B) La boîte microfluidique utilisée dans ce protocole, vue de l’avant (A) et (B) du haut. Les solutions de nettoyage sont placées dans les positions 1 à 3, et les échantillons et les calibrates dans les positions 4 à 6. Toutes les solutions sont connectées au m-switch, qui est connecté au capteur d’unité de débit « Small ». C) Vue d’ensemble de l’ensemble du système. L’ordinateur (en haut à gauche) est connecté au boîtier de microfluidique (illustré à côté du tampon et des tubes d’échantillon) et au photomètre de masse (en bas à droite), où se trouve la puce de microfluidique. Le petit débitmètre (FRMS) surveille le débit dans la conduite d’échantillonnage, tandis que le grand débitmètre (FRML) surveille le débit tampon. Le tube de l’échantillon et de la ligne tampon se connecte à un canal de la puce microfluidique, placé à l’intérieur du photomètre de masse. (D) Chaque puce a trois canaux. Le FRMS est connecté à l’entrée de l’échantillon et le FRML à l’entrée du tampon. La zone du mélangeur est l’endroit où la dilution rapide de l’échantillon a lieu, tandis que la zone d’observation est l’endroit où les mesures de photométrie de masse sont effectuées. La sortie est surveillée par un capteur de débit supplémentaire pour s’assurer qu’il n’y a pas de fuites dans la puce. Veuillez cliquer ici pour voir une version agrandie de cette figure.
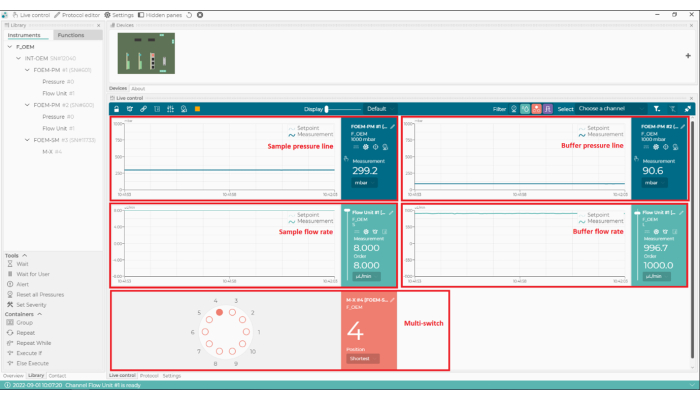
Figure 2 : Le logiciel de contrôle de la microfluidique. Avec ce logiciel, réglez et surveillez les débits, les conduites de pression du système microfluidique, et la position du multi-interrupteur (m-switch). Veuillez cliquer ici pour voir une version agrandie de cette figure.
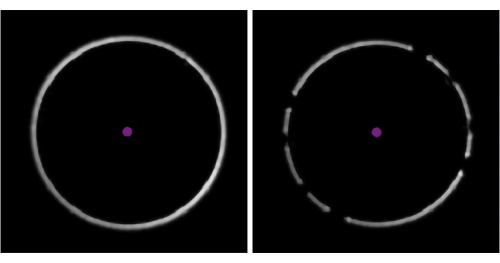
Figure 3 : Identification de la présence de bulles dans l’huile. Les exemples du logiciel d’acquisition de données montrent une bague de mise au point claire et ininterrompue (à gauche) et une bague « cassée », indiquant la présence de bulles d’air dans l’huile d’immersion (à droite). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Exemples représentatifs de concentrations d’échantillons où le nombre d’atterrissages de molécules est trop faible, idéal ou trop nombreux. Les molécules atterrissant à la surface de la zone d’observation dans la puce microfluidique apparaîtront comme des taches sombres dans la vue ratiométrique de l’image de photométrie de masse. De manière optimale, la concentration souhaitée doit permettre d’obtenir une densité idéale d’événements d’atterrissage lors de l’acquisition des données (au milieu). Si la densité de l’événement d’atterrissage est trop faible (en haut, « Trop peu »), il est impossible d’effectuer une analyse statistique précise des données de photométrie de masse. S’il est trop élevé (en bas, « Trop de molécules »), les molécules d’atterrissage se chevaucheront spatialement, ce qui entraînera une mauvaise qualité des données. Ces images ont été capturées avec le logiciel d’acquisition de données à l’aide d’un photomètre de masse. Veuillez cliquer ici pour voir une version agrandie de cette figure.
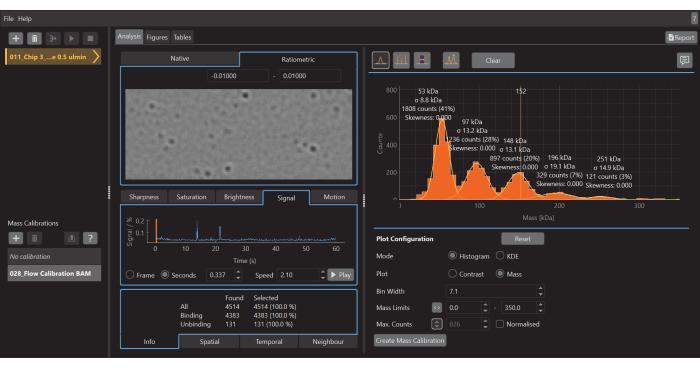
Figure 5 : Capture d’écran du logiciel d’analyse de données pour la photométrie de masse. Ici, les fichiers .mp exportés à partir du logiciel d’acquisition de données peuvent être chargés pour analyser les données. Des chiffres peuvent être générés à partir des données traitées. Veuillez cliquer ici pour voir une version agrandie de cette figure.
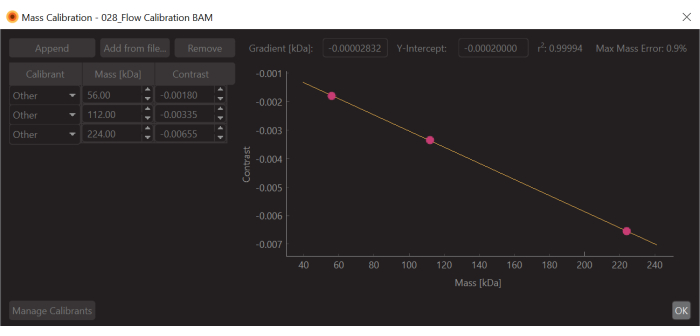
Figure 6 : Capture d’écran de l’étalonnage du contraste de masse pour cette expérience. L’étalon utilisé était la β-amylase, qui est connue pour former trois espèces : monomère (56 kDa), dimère (112 kDa) et tétramère (224 kDa). Veuillez cliquer ici pour voir une version agrandie de cette figure.
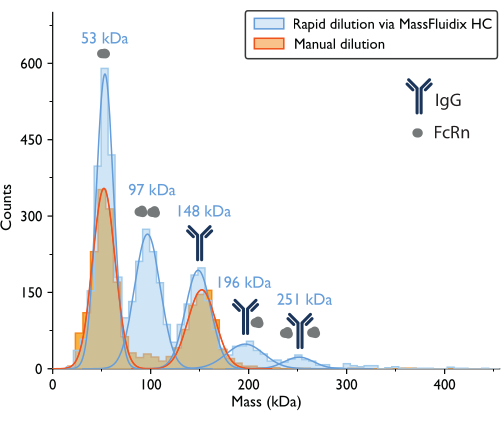
Figure 7 : Les histogrammes de masse ne révèlent une formation complexe qu’après une dilution rapide de l’échantillon. Les histogrammes de masse et les distributions gaussiennes les mieux ajustées correspondantes sont présentés pour les mesures d’échantillons contenant des IgG et FcRn après dilution manuelle (orange) ou dilution rapide via le système microfluidiqueHC (bleu). Les étiquettes de masse indiquent que les valeurs moyennes des courbes gaussiennes correspondent aux histogrammes de masse mesurés après dilution rapide. Après dilution manuelle, des pics correspondant aux monomères FcRn (52 kDa, mesurés avec la dilution manuelle) et aux monomères IgG (152 kDa) ont été observés. Après dilution rapide, en plus des monomères FcRn et IgG, des pics correspondant à des dimères FcRn et à des complexes IgG-FcRn avec stœchiométrie 1:1 et 1:2 ont également été clairement observés. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure 1 : Les mesures par photométrie de masse des échantillons de contrôle après dilution rapide ne montrent aucune dégradation des protéines. (A) Histogramme de masse et distribution gaussienne la mieux ajustée pour l’échantillon uniquement FcRn. La concentration initiale de l’échantillon avant la dilution rapide était de 20 μM, et le débit a été fixé à 0,5 μL/min. Des pics de masse correspondant aux monomères FcRn (53 kDa) et aux dimères FcRn (97 kDa) ont pu être observés. (B) Histogramme de masse et distribution gaussienne la mieux ajustée pour l’échantillon d’IgG (Herceptin) uniquement. La concentration initiale de l’échantillon avant la dilution rapide était de 2 μM et le débit a été fixé à 1 μL/min. Un seul pic de masse correspondant à un monomère IgG (155 kDa) a pu être observé. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Le protocole décrit ici fournit une méthode pour détecter et quantifier les interactions protéine-protéine de faible affinité. Il utilise un photomètre de masse couplé à un système microfluidique à dilution rapide. La photométrie de masse est un outil bioanalytique sans marquage qui permet de mesurer de manière fiable la masse moléculaire en solution pour les biomolécules16, pour celles comprises entre 30 kDa et 6 MDa. Comme la photométrie de masse est une technique à molécule unique qui analyse les échantillons un par un, elle est généralement limitée aux échantillons dans la gamme de concentration 100 pM-100 nM. Au-dessus de cette plage, les molécules atterrissant sur la surface du verre se chevaucheront spatialement, ce qui entraînera une mauvaise qualité des données ; En dessous de cette plage, on obtient trop peu de données pour effectuer une analyse robuste7. Une conséquence importante est qu’elle peut limiter l’étude des interactions protéiques à celles qui forment un mélange d’espèces liées et non liées dans cette plage.
Ici, nous avons détaillé un protocole étape par étape pour l’utilisation d’un système microfluidique à dilution rapide afin d’élargir efficacement la gamme de concentrations d’échantillons qui se prêtent à la photométrie de masse. En diluant l’échantillon sur la puce microfluidique, puis en le faisant circuler dans la fenêtre d’observation du détecteur dans un délai de 50 ms, le système capture les complexes présents dans l’échantillon non dilué avant que l’équilibre d’interaction ne se déplace. L’échantillon est livré en continu au détecteur lors des mesures individuelles. Dans ces conditions, 95% du complexe restera intact lorsque l’échantillon sera mesuré, même pour des interactions de faible affinité - avec un KD de l’ordre des micromolaires et des taux de dissociation aussi rapides que 1 s-1.
Cela peut être calculé comme suit : Pour une réaction 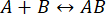 avec une vitesse directe kfet une vitesse arrière kb,
avec une vitesse directe kfet une vitesse arrière kb,
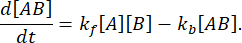
À l’équilibre, les concentrations des trois espèces (A, B et le complexe AB) restent constantes, donc 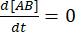 et
et 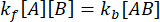 . Sous l’hypothèse conservatrice que la perturbation (dilution, dans ce cas) peut provoquer la dissociation du complexe, mais que la réaction directe (d’association) ne se produit pas, le terme kf [A] [B] peut être considéré comme négligeable, et la simplification suivante peut être faite :
. Sous l’hypothèse conservatrice que la perturbation (dilution, dans ce cas) peut provoquer la dissociation du complexe, mais que la réaction directe (d’association) ne se produit pas, le terme kf [A] [B] peut être considéré comme négligeable, et la simplification suivante peut être faite :
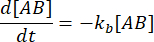
L’intégration donne l’expression suivante pour la concentration du complexe au moment après la perturbation de l’équilibre :
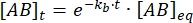
La fraction du complexe qui reste liée au temps t après la perturbation de l’équilibre est donc :
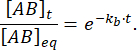
À = 50 ms, pour une réaction avec kb≈ 1 s-1, la fraction liée est de 0,95, soit 95%11,12.
La photométrie de masse a été utilisée ici et précédemment5 pour étudier la liaison de l’anticorps monoclonal IgG trastuzumab au domaine soluble du FcRn. Il a été rapporté que les deux partenaires de liaison se lient avec une affinité nanomolaire à un pH acidede 17. La photométrie de masse a été utilisée pour évaluer qualitativement l’abondance des complexes formés alors que les partenaires de liaison étaient à pH 5,0, et les échantillons ont été rapidement dilués par un système microfluidique supplémentaire. La procédure a été optimisée pour l’interaction protéine-protéine particulière sur la base des résultats précédemment rapportés5. La même procédure peut être utilisée pour étudier d’autres interactions, à condition que les utilisateurs aient des connaissances préalables ou optimisent les conditions expérimentales du système en question, telles que les tampons à utiliser, la concentration initiale en protéines, la stœchiométrie attendue et la quantité d’incubation nécessaire pour permettre à l’interaction d’atteindre un équilibre.
Lorsque le mélange IgG-FcRn a été dilué manuellement, il était difficile de détecter la présence de complexes IgG-FcRn, même si ces protéines sont connues pour interagir5. Cet article montre que l’approche de dilution rapide entraîne une augmentation notable de la quantité de ces complexes. Pour le même échantillon, lorsque la dilution rapide a été utilisée, les complexes FcRn-IgG 1:1 et les complexes FcRn-IgG 2:1 ont tous deux été clairement observés. Ces différences dans la formation des complexes démontrent l’importance d’étudier les systèmes d’interaction biomoléculaire à travers une large gamme de concentrations.
De plus, ces résultats démontrent également qu’il est simple d’utiliser la microfluidique avec l’analyse de molécules uniques pour capturer les interactions faibles, comblant ainsi une lacune importante de la méthode. La combinaison de la microfluidique à dilution rapide et de la photométrie de masse offre des avantages intéressants en raison des avantages de la photométrie de masse en tant que technique analytique. C’est-à-dire que la photométrie de masse ne nécessite pas d’étiquettes, implique une préparation minimale de l’échantillon et les mesures sont effectuées en solution. Pour ce protocole, un autre avantage clé de la photométrie de masse est sa capacité à distinguer et à quantifier toutes les espèces formées (à condition qu’elles aient une masse distincte de >30 kDa). Cela contraste avec la SPR, par exemple, qui peut mesurer les taux de liaison et de déliaison, mais ne peut pas facilement fournir des informations stœchiométriques8.
Pour ce protocole, ainsi que pour les expériences de photométrie de masse plus généralement, plusieurs considérations sont utiles. Tout d’abord, la concentration finale en protéines doit être dans la limite de ce que la photométrie de masse peut mesurer (100 pM-100 nM). La concentration initiale d’incubation doit également se situer dans la plage du système microfluidique (jusqu’à 90 μM) et être théoriquement supérieure au KD réel de l’interaction10. Le point de départ recommandé est un rapport de mélange de concentration de 1:1 entre les espèces en interaction à la concentration μM. Le rapport pourrait alors être varié à 1:2, 1:5 ou, comme dans le cas de cette interaction, 1:10. S’il n’y a pas d’information préalable sur les interactions protéiques, l’utilisateur devrait optimiser l’expérience, en commençant par une concentration élevée (recommandée 20 μM) pour chaque partenaire afin de déterminer si l’affinité des composants se situe dans la plage de concentration soutenue par la méthode présentée (c’est-à-dire que des complexes sont formés). L’optimisation peut également impliquer le choix d’autres conditions tampons pour favoriser les interactions ou le titrage de l’un des composants de l’interaction pour déterminer le bon rapport de mélange. Une fois ceux-ci déterminés, il est possible d’optimiser les concentrations et les débits pour permettre des conditions optimales pour l’étude et la méthode, par exemple, en diminuant les concentrations pour permettre une meilleure résolution des pics.
Deuxièmement, pour reproduire avec succès cette expérience, les impuretés doivent être minimisées. Les sources courantes d’impuretés connues pour affecter négativement les mesures de photométrie de masse comprennent d’autres protéines ou débris cellulaires qui restent après la purification, des tampons non filtrés, des détergents formant des micelles (s’ils sont présents à une concentration trop élevée) et des tampons contenant de fortes concentrations de sel, de glycérol ou d’autres composants. Comme indiqué dans le protocole ci-dessus, les bulles dans le système microfluidique doivent être éliminées. Des bulles peuvent se former dans le système de tuyauterie ou si les échantillons ont une tension superficielle élevée et sont sujets à la formation de mousse. Des bulles peuvent également se former dans l’huile d’immersion, ce qui peut être détecté à partir de la bague de mise au point (Figure 3). Si les bulles ne peuvent pas être éliminées en suivant les étapes décrites dans le protocole, une autre solution consiste à dégazer l’échantillon à l’aide d’un dessiccateur et d’une pompe à vide, en laissant l’échantillon sous pression réduite pendant quelques minutes. Il n’est pas recommandé de vortex ou d’agiter des solutions protéiques hautement concentrées, car ces actions peuvent favoriser la formation de bulles.
Bien que la mesure d’une interaction protéine-protéine spécifique soit démontrée ici, le même protocole peut être appliqué à d’autres systèmes d’interaction protéine-protéine sans modification significative. Une autre orientation future de ce protocole serait d’utiliser les mesures pour calculer les valeurs de KD pour les complexes identifiés, comme cela a été décrit ailleurs dans le contexte de la photométrie de masse 5,7. Alors que les études précédentes utilisaient des données provenant d’expériences impliquant une dilution manuelle et des interactions plus fortes, le principe d’analyse pourrait être facilement appliqué dans ce contexte, à condition que d’autres améliorations du dispositif microfluidique soient mises en œuvre (telles que l’augmentation de la précision du capteur de débit et de la stabilité de la pompe).
Au-delà des interactions protéine-protéine, il est probable que des applications plus larges soient utilisées pour l’approche combinée de la photométrie de masse et de la microfluidique à dilution rapide. La photométrie de masse peut être utilisée pour évaluer la pureté, l’agrégation et l’homogénéité de l’échantillon18,19 ; étudier l’oligomérisationdes protéines 20, l’assemblage macromoléculaire21 ou la polymérisation22 ; et dans d’autres domaines. L’analyse par photométrie de masse s’étend également au-delà des protéines ; Il a été utilisé pour étudier les interactions entre les acides nucléiques et les protéines23, les particules virales24 et les nanoparticules25. Ce protocole décrit donc une application importante d’un système microfluidique combiné de photométrie de masse - il permet la mesure directe des interactions protéine-protéine faibles au niveau des molécules et des complexes individuels. La valeur de l’application actuelle est élevée, car elle ouvre la possibilité de caractériser directement des interactions qui ont généralement été difficiles à étudier - avec une pertinence dans des domaines thérapeutiques critiques. Cette approche combinée pourrait également servir de base à un plus large éventail d’investigations pour des échantillons dont les concentrations peuvent atteindre des dizaines de micromolaires.
Déclarations de divulgation
Myndert Claasen et Zornitsa Kofinova sont des employés de Refeyn Ltd, qui produit le photomètre de masse et le système microfluidique utilisés dans cet article. Weston Struwe est actionnaire et consultant de Refeyn Ltd.
Remerciements
W.S. est soutenu par une bourse UKRI Future Leaders [MR/V02213X/1]. Le texte et les graphiques du manuscrit ont été préparés avec le soutien des membres de l’équipe de communication scientifique de Refeyn (Panagiota Paganopoulou, Neus Torres Tamarit et Catherine Lichten). Nous tenons également à remercier Camille Hetez, Sofia Ferreira et Matthias Langhorst pour leurs précieux commentaires.
matériels
| Name | Company | Catalog Number | Comments |
| 2-Propanol (Isopropanol) | VWR International LLC | 20880.320 | |
| Data acquisition software | Refeyn | AcquireMP (v2022 R1) | |
| Data analysis software | Refeyn | DiscoverMP (v2022 R1) | |
| FCRN, His-Tag | Sigma | SRP0624 | |
| Herceptin (IgG) | Cambridge Bioscience | HY-P9907-1mg | |
| Mass photometer | Refeyn | TwoMP | |
| Microfluidics box | Refeyn | MassFluidix HC system | |
| Microfluidics chip | Refeyn | MassFluidix HC chip | |
| Microfluidics control software | Fluigent | OxyGEN | |
| Phosphate Buffered Saline (PBS), 1x Ultra Pure | VWR International LLC | K812 | |
| Sodium Hydroxide (NaOH) | Sigma | S2770 | |
| β-Amylase, from sweet potato | Sigma | A8781 |
Références
- Arter, W. E., Levin, A., Krainer, G., Knowles, T. P. J. Microfluidic approaches for the analysis of protein-protein interactions in solution. Biophysical Reviews. 12 (2), 575-585 (2020).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry. B. 122 (49), 11554-11560 (2018).
- Li, Z. Editorial: Methods in structural biology: Cryo-electron microscopy. Frontiers in Molecular Biosciences. 9, 1041386 (2022).
- Herling, T. W., et al. A microfluidic platform for real-time detection and quantification of protein-ligand interactions. Biophysical Journal. 110 (9), 1957-1966 (2016).
- Soltermann, F., et al. Quantifying protein-protein interactions by molecular counting with mass photometry. Angewandte Chemie International Edition. 59 (27), 10774-10779 (2020).
- Wu, D., Piszczek, G. Rapid determination of antibody-antigen affinity by mass photometry. Journal of Visualized Experiments: JoVE. 168, 61784 (2021).
- Wu, D., Piszczek, G. Measuring the affinity of protein-protein interactions on a single-molecule level by mass photometry. Analytical Biochemistry. 592, 113575 (2020).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- MassFluidix® HC system for rapid dilution via microfluidics. Available from: https://www.refeyn.com/massfluidix-hc-system (2023)
- Pollard, T. D. A guide to simple and informative binding assays. Molecular Biology of the Cell. 21 (23), 4061-4067 (2010).
- Jarmoskaite, I., AlSadhan, I., Vaidyanathan, P. P., Herschlag, D. How to measure and evaluate binding affinities. eLife. 9, e57264 (2020).
- Monnet, C., et al. Selection of IgG variants with increased FcRn binding using random and directed mutagenesis: Impact on effector functions. Frontiers in Immunology. 6, 39 (2015).
- . Refeyn TwoMP: Transforming biomolecular characterisation Available from: https://www.refeyn.com/twomp-mass-photometer (2022)
- Lai, S. -. H., Tamara, S., Heck, A. J. R. Single-particle mass analysis of intact ribosomes by mass photometry and Orbitrap-based charge detection mass spectrometry. iScience. 24 (11), 103211 (2021).
- Wu, D., Piszczek, G. Standard protocol for mass photometry experiments. European Biophysics Journal. 50 (3-4), 403-409 (2021).
- Vaughn, D. E., Bjorkman, P. J. Structural basis of pH-dependent antibody binding by the neonatal Fc receptor. Structure. 6 (1), 63-73 (1998).
- Niebling, S., et al. Biophysical screening pipeline for Cryo-EM grid preparation of membrane proteins. Frontiers in Molecular Biosciences. 9, 882288 (2022).
- Paul, S. S., Lyons, A., Kirchner, R., Woodside, M. T. Quantifying oligomer populations in real time during protein aggregation using single-molecule mass photometry. ACS Nano. 16 (10), 16462-16470 (2022).
- Schulz, L., et al. Evolution of increased complexity and specificity at the dawn of form I Rubiscos. Science. 378 (6616), 155-160 (2022).
- Malay, A. D., et al. An ultra-stable gold-coordinated protein cage displaying reversible assembly. Nature. 569 (7756), 438-442 (2019).
- Hundt, N., Cole, D., Hantke, M. F., Miller, J. J., Struwe, W. B., Kukura, P. Direct observation of the molecular mechanism underlying protein polymerization. Science Advances. 8 (35), eabm7935 (2022).
- Acharya, A., et al. Distinct RPA domains promote recruitment and the helicase-nuclease activities of Dna2. Nature Communications. 12, 6521 (2021).
- Ebberink, E. H. T. M., Ruisinger, A., Nuebel, M., Thomann, M., Heck, A. J. R. Assessing production variability in empty and filled adeno-associated viruses by single molecule mass analyses. Molecular Therapy - Methods & Clinical Development. 27, 491-501 (2022).
- Melo, L., et al. Size distributions of gold nanoparticles in solution measured by single-particle mass photometry. The Journal of Physical Chemistry B. 125 (45), 12466-12475 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon