Method Article
Analisi della formazione di complessi proteici a concentrazioni micromolari mediante accoppiamento di microfluidica con fotometria di massa
In questo articolo
Riepilogo
Questo protocollo combina la fotometria di massa con un nuovo sistema microfluidico per studiare le interazioni proteina-proteina a bassa affinità. Questo approccio si basa sulla rapida diluizione di complessi altamente concentrati in soluzione, che consente misurazioni a bassa affinità e amplia l'applicabilità della fotometria di massa.
Abstract
La fotometria di massa è una tecnologia versatile di misurazione della massa che consente lo studio delle interazioni biomolecolari e della formazione di complessi in soluzione senza etichette. La fotometria di massa è generalmente adatta all'analisi di campioni nell'intervallo di concentrazione 100 pM-100 nM. Tuttavia, in molti sistemi biologici, è necessario misurare campioni più concentrati per studiare interazioni a bassa affinità o transitorie. Qui, dimostriamo un metodo che espande efficacemente la gamma di concentrazioni del campione che possono essere analizzate mediante fotometria di massa da nanomolari a decine di micromolari.
In questo protocollo, la fotometria di massa è combinata con un nuovo sistema microfluidico per studiare la formazione di complessi proteici in soluzione nell'intervallo di concentrazione micromolare. Con il sistema microfluidico, gli utenti possono mantenere un campione alla concentrazione più alta desiderata seguita da una diluizione nell'intervallo nanomolare, diversi millisecondi prima della misurazione della fotometria di massa. A causa della velocità della diluizione, i dati vengono ottenuti prima che l'equilibrio del campione si sia spostato (cioè la dissociazione del complesso).
La tecnica viene applicata per misurare le interazioni tra un anticorpo immunoglobulina G (IgG) e il recettore Fc neonatale, mostrando la formazione di complessi di ordine superiore che non erano quantificabili con misurazioni di fotometria di massa statica.
In conclusione, la combinazione di fotometria di massa e microfluidica consente di caratterizzare campioni nell'intervallo di concentrazione micromolare ed è abile nella misurazione delle interazioni biomolecolari con affinità più deboli. Queste capacità possono essere applicate in una vasta gamma di contesti, tra cui lo sviluppo e la progettazione di bioterapeutici, consentendo una caratterizzazione approfondita di diverse interazioni proteina-proteina.
Introduzione
Le interazioni proteina-proteina sottolineano la maggior parte delle funzioni cellulari, dalla regolazione immunitaria alla replicazione e traduzione del DNA. Di conseguenza, c'è un bisogno fondamentale in tutte le scienze della vita di studiare una vasta gamma di interazioni tra diversi complessi eterogenei che si formano comunemente. Tuttavia, la loro rilevazione, caratterizzazione e quantificazione sono spesso impegnative, in particolare per le interazioni a bassa affinità1.
I saggi di immunoprecipitazione sono spesso utilizzati per rilevare interazioni ad alta affinità, ma per interazioni transitorie e a bassa affinità, il rilevamento è in gran parte irrealizzabile2. Possono essere utilizzate anche tecniche di fluorescenza, ma richiedono l'aggiunta potenzialmente dirompente di etichette fluorescenti2. La crio-EM può fornire un'istantanea strutturale e una lettura d'insieme dei complessi proteici formati con un'elevata risoluzione spaziale, ma in genere richiede anche di lavorare a concentrazioni troppo basse per l'imaging di interazioni a bassa affinità. Cryo-EM comporta anche sfide relative a costi, accessibilità, preparazione del campione e tempo di analisi3.
Inoltre, la risonanza plasmonica di superficie (SPR) è diventata un modo popolare per quantificare le interazioni proteina-proteina, sebbene richieda l'immobilizzazione della proteina, che può influenzare l'equilibrio di legame e provocare velocità di attivazione variabili, riducendo così l'accuratezza della misurazione 4,5. Prevede inoltre diverse fasi di analisi prima della raccolta e dell'analisidei dati 6.
La fotometria di massa è una tecnica a singola molecola che è stata utilizzata per analizzare le interazioni proteina-proteina 5,6,7. Funziona misurando la massa di singole molecole o complessi in base alla luce che diffondono quando atterrano sulla superficie di un vetrino coprioggetti8. Le misurazioni della fotometria di massa sono state utilizzate per quantificare le affinità di legame dall'abbondanza relativa dei partner di legame e dei complessi che formano5. Tuttavia, come altre tecniche a singola molecola, la concentrazione del campione da misurare dovrebbe essere in genere inferiore a 100 nM. Se la concentrazione è maggiore, le molecole che atterrano sulla superficie del vetro si sovrapporranno spazialmente, con conseguente scarsa qualità dei dati7. Di conseguenza, le interazioni più deboli (KD ~ micromolari), che si dissociano a queste concentrazioni più basse, non possono essere misurate in modo affidabile poiché non è possibile osservare la necessaria miscela di specie non legate e legate5.
Qui, descriviamo un approccio che supera questa limitazione basato su un nuovo dispositivo di fotometria di massa microfluidica accoppiata. In particolare, un sistema microfluidico viene utilizzato in combinazione con il fotometro di massa per espandere efficacemente la gamma di interazioni che possono essere quantificate dalla fotometria di massa. La microfluidica ha dimostrato di offrire una serie di possibilità per studiare le interazioni proteina-proteina, inclusa la rapida diluizione per rilevare interazioni deboli 1,9. Il sistema qui descritto funziona diluendo rapidamente il campione fino a 10.000 volte su un chip microfluidico e facendolo scorrere immediatamente attraverso l'area di osservazione del chip, consentendo la misurazione della fotometria di massa entro 50 ms da quando le molecole hanno iniziato il processo di diluizione10. La diluizione si verifica quando il campione e il tampone vengono combinati in un miscelatore a valvola di Tesla inversa sul chip, con le relative portate delle due soluzioni che determinano la quantità di diluizione che si verifica (vedere la fase 8 del protocollo). La portata è controllabile con il software di controllo microfluidico. L'alterazione della portata può modificare la popolazione relativa della specie in quanto può influire sul numero di eventi di atterraggio sulla superficie del vetro, che è ciò che viene misurato dal fotometro di massa.
La velocità del processo è sufficientemente elevata da consentire il completamento della misurazione prima che l'integrità dell'interazione sia stata interrotta (per ulteriori dettagli, vedere anche la Discussione). Questo può essere compreso attraverso un breve sguardo alla teoria delle reazioni del primo ordine, dove 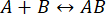 . La costante di velocità diretta (associazione) è kf, la costante di velocità all'indietro (dissociazione) è kb e la costante di dissociazione di equilibrio (KD) è definita come
. La costante di velocità diretta (associazione) è kf, la costante di velocità all'indietro (dissociazione) è kb e la costante di dissociazione di equilibrio (KD) è definita come
KD= kb/ kf
Per il legame proteico, kfè generalmente limitato dalla diffusione dei reagenti11 e quindi è ristretto all'intervallo di 106-10 7 M-1·s-1. Poiché l'intervallo di è limitato, una reazione a bassa affinità (KD~micromolari) avrà kb≈ 1 s-1. Cioè, kb= kf · KD= (106 M-1·s-1) (10-6 M) = 1 s-1, con emivita del complesso di circa 0,7 s11,12.
Il nostro sistema di esempio è il legame dell'anticorpo monoclonale IgG trastuzumab al dominio solubile del recettore IgG neonatale Fc (FcRn), che sono noti partner interagenti13. I dati pubblicati in precedenza ottenuti utilizzando la sola fotometria di massa convenzionale (cioè con la diluizione manuale dei campioni) hanno mostrato che le proteine formano più specie. I monomeri FcRn, i dimeri FcRn e le IgG non legate erano chiaramente visibili, mentre sono stati rilevati anche complessi IgG-FcRn (a pH 5,0) (a pH 5,0) ma solo con abbondanza molto bassa5. Questa osservazione solleva la questione se la formazione del complesso IgG-FcRn possa essere rilevata più chiaramente se misurata a una concentrazione più elevata. In effetti, la combinazione della fotometria di massa con un approccio accoppiato a diluizione rapida qui descritto ha fornito prove più solide della formazione di complessi da parte di un aumento delle particelle misurate.
Il protocollo di fotometria di massa e microfluidica qui descritto permette di caratterizzare la formazione di complessi con un KD fino all'intervallo micromolare. Una determinazione empirica del KD richiederà ulteriori miglioramenti sull'accuratezza del sensore di flusso, sulla stabilità della pompa, sulle variazioni truciolo-chip e sulla posizione di misurazione all'interno della finestra di osservazione, poiché tutti questi fattori influenzerebbero il tempo da quando il campione viene diluito a quello misurato.
Lo stesso approccio potrebbe essere applicato per studiare il legame tra qualsiasi proteina solubile, a condizione che abbiano pesi molecolari distinti (separati da almeno 25 kDa) che rientrino nell'intervallo adatto per l'analisi con un fotometro di massa (da 30 kDa a 6 MDa). Le intuizioni ottenute potrebbero essere utili per studi in una vasta gamma di contesti, dall'acquisizione di una comprensione meccanicistica delle funzioni cellulari alla progettazione di nuovi farmaci bioterapeutici.
Protocollo
1. Preparazione degli strumenti e avvio del software
- Accendere il fotometro di massa e lasciarlo acceso per almeno 1 ora prima di iniziare qualsiasi misurazione.
NOTA: Questo perché lo strumento deve raggiungere una temperatura costante. Non lasciare acceso il fotometro di massa per 1 ora potrebbe portare a uno spostamento di massa e, quindi, a risultati imprecisi. - Accendere il tavolo antivibrante accendendo l'alimentazione e premendo il pulsante di isolamento (sotto il fotometro di massa). Le vibrazioni limitano le prestazioni dello strumento di fotometria di massa, quindi la tabella antivibrante è importante per garantire la massima sensibilità dello strumento. Accendi la scatola microfluidica.
- Avviare il software di acquisizione dati e il software di controllo microfluidico. Accendere il compressore d'aria.
2. Preparazione di campioni proteici, tampone e soluzioni detergenti
- Aggiungere 200 ml di tampone PBS (pH 7,4) a un flacone pulito da 200 ml e 200 ml di tampone PBS (pH 5,0) a un secondo flacone da 200 ml.
NOTA: I flaconi da 200 ml dovranno essere collegati al sensore dell'unità di flusso "L" nei passaggi successivi. Utilizzare il tampone pH 7,4 PBS per la misura di taratura e il tampone pH 5,0 PBS per la misura del campione. - In una provetta da centrifuga da 0,5 mL, miscelare IgG (2 μM) e FcRn (20 μM) in un rapporto 1:10 per un volume totale di 60 μL.
- Le soluzioni stock per FcRn e IgG sono rispettivamente 91,9 μM e 13,5 μM. Preparare la reazione miscelando 9 μL di IgG madre + 13 μL di FcRn madre + 38 μL di PBS (pH 5,0, temperatura ambiente [RT]) in una provetta da centrifuga per un volume totale di 60 μL. Le concentrazioni finali per i due reagenti erano 2 μM per IgG e 20 μM per FcRn. Mantenere tutte le scorte proteiche in ghiaccio durante questo processo.
- In un'altra provetta da centrifuga da 0,5 mL, erogare 20 μl del calibrante β-amilasi a una concentrazione di 20 μM.
- Ad esempio, per preparare il calibrante β-amilasi qui utilizzato, seguire la procedura descritta in 2.3.1.1-2.3.1.2.
- Risospendere 20 mg di polvere di β-amilasi nel flaconcino in 3,57 mL di PBS (glicerolo al 5%), corrispondenti a 5,6 mg/mL e una concentrazione molare di 100 μM (poiché il peso molecolare è 56 kDa).
- Per diluire a 20 μM, combinare 200 μL del materiale da 100 μM con 800 μL di PBS (pH 7,4, RT) in una provetta da centrifuga da 1 mL.
- Ad esempio, per preparare il calibrante β-amilasi qui utilizzato, seguire la procedura descritta in 2.3.1.1-2.3.1.2.
- Incubare i campioni a RT per almeno 30 minuti.
NOTA: Iniziare a preparare il campione e calibrare le diluizioni dopo aver acceso il fotometro di massa. Per questo esperimento, i campioni sono stati incubati per 120 minuti. - In tre provette da centrifuga da 50 mL, aliquota: 50 mL di PBS (pH 7,4) - soluzione detergente 1 (CS1), 50 mL di NaOH 0,5 M - soluzione detergente 2 (CS2) e 50 mL di 100% IPA - soluzione detergente 3 (CS3).
3. Impostazione sperimentale
- Per caricare il campione e il calibratore, posizionare la provetta da centrifuga calibrante in posizione 4 e la provetta da centrifuga per campioni in posizione 5 della scatola di microfluidica (Figura 1).
- Per caricare le soluzioni detergenti, posizionare CS1, CS2 e CS3 nelle posizioni 1, 2 e 3 della scatola microfluidica (Figura 1).
NOTA: Il campione, il calibratore e le soluzioni detergenti sono collegati al multi-switch (m-switch). L'interruttore m è collegato al sensore dell'unità di flusso "S". - Avvitare il tappo che si collega alla linea del tampone sul flacone del tampone da 200 ml (pH 7,4).
NOTA: La linea tampone è collegata a una valvola di intercettazione, che è collegata al sensore dell'unità di flusso "L". La valvola di intercettazione impedisce qualsiasi effetto di sifonamento che può verificarsi quando il flusso tampone viene interrotto. Il sifonamento può portare alla contaminazione del tampone dal fluido residuo diluito che ritorna al serbatoio del tampone. - Posizionare il chip microfluidico su una piastra di preparazione.
Spingere l'altra estremità del tubo del sensore dell'unità di flusso "S" nell'"ingresso del campione" del primo canale del chip microfluidico (Figura 1). La linea di campionamento è completa. - Spingere l'altra estremità del tubo del sensore dell'unità di flusso "L" nell'"ingresso tampone" del primo canale del chip microfluidico (Figura 1). La linea del buffer è completa.
- Per raccogliere il deflusso, spingere un tubo verso l'"uscita" del primo canale nel chip microfluidico; posizionare l'altra estremità in modo che scarichi in un pallone o in un becher (Figura 1).
4. Primerizzazione del campione e delle linee del buffer con buffer
- Aprire la valvola di intercettazione manuale sulla linea del tampone.
- Nel software di controllo microfluidico, impostare la portata della linea tampone su 1000 μL/min e assicurarsi che la pressione della linea tampone non superi i 110 mbar (Figura 2). Il flusso si avvierà automaticamente nella linea del buffer (Figura 1).
NOTA: Se la pressione della linea tampone supera i 110 mbar, potrebbe essere dovuto al passaggio dell'aria attraverso il sensore di flusso. Se la pressione non torna alla normalità entro pochi secondi, c'è potenzialmente un blocco (di solito dovuto a tubi schiacciati alle connessioni). In tal caso, interrompere il flusso e controllare i collegamenti nella linea tampone, a partire dalla valvola di intercettazione. - Nel software di controllo microfluidico, selezionare la posizione 1 dell'interruttore m (corrispondente al tampone PBS), quindi impostare la portata della linea di campionamento su 8 μL/min e assicurarsi che la pressione della linea di campionamento non superi i 350 mbar. Il flusso inizierà automaticamente nella linea di campionamento (Figura 1).
- Utilizzare un puntale per pipetta (o un altro componente in plastica morbida) per applicare una leggera pressione dall'alto vicino alle bolle intrappolate nel chip per rimuovere le bolle d'aria e assicurarsi che vengano rimosse attraverso l'uscita.
NOTA: Assicurarsi di rimuovere tutte le bolle nelle sezioni "mixer" e "area di osservazione" (Figura 1) del canale in uso. Fare attenzione a non spingere troppo forte, poiché ciò potrebbe danneggiare il chip.
5. Posizionare il chip microfluidico sul fotometro di massa e trovare la messa a fuoco
- Applicare una goccia di olio da immersione per microscopio sull'obiettivo del fotometro di massa.
- Posizionare il chip microfluidico sul supporto del fotometro di massa con l'"ingresso del campione" rivolto verso l'alto e tenerlo attaccato ai morsetti dello stadio (Figura 1). Assicurarsi che tutti i collegamenti dei tubi rimangano collegati.
- Utilizzando il software di acquisizione dati, spostare il tavolino per assicurarsi che l'"Area di osservazione" del canale 1 sia allineata con l'obiettivo (Figura 1).
- Chiudere il coperchio del fotometro di massa e premere l'opzione Trova messa a fuoco per la diluizione delle gocce nel software di acquisizione dati.
- Controllare l'anello di messa a fuoco bianco nell'angolo in basso a sinistra del software di acquisizione dati (Figura 3). Gli spazi vuoti nell'anello indicano la presenza di una bolla d'aria nell'olio di immersione; Rimuoverlo aumentando il tavolino Velocità al massimo e spostando delicatamente il tavolino lateralmente.
- Al termine della messa a fuoco, attendere 2-3 minuti prima di effettuare la prima registrazione. Quindi, premere Registra per registrare una misurazione di 1 minuto e assicurarsi (osservando la misurazione) che non appaiano impurità.
NOTA: Le impurità potrebbero essere sulla superficie del vetro o nel tampone. Il valore di nitidezza nel software di acquisizione dati deve essere superiore al 4,5%.
6. Calibrazione della fotometria di massa
- Nel software di controllo microfluidico, portare l'interruttore m in posizione 4 (corrispondente al calibratore) e assicurarsi che la portata della linea di campionamento sia impostata a 8 μL/min e che la pressione della linea di campionamento non superi i 350 mbar (Figura 2).
- Il calibratore inizierà il flusso attraverso il chip. Attendere circa 1,5-2,5 minuti (o fino a quando il calibrante non viene visualizzato in modo coerente sul software di acquisizione dati). Il tempo può variare in base alla lunghezza del tubo della linea di campionamento.
- Una volta che il calibrante è visibile in modo coerente (cioè, il numero di eventi è sufficiente per una misurazione accurata della fotometria di massa), nel software di controllo microfluidico, ridurre la portata a 0,5 μL/min - il livello di diluizione target per questo esperimento.
NOTA: Iniziare con una portata maggiore riduce semplicemente il tempo necessario al calibratore per raggiungere il chip. - Assicurarsi che non ci siano "troppo poche" o "troppe" molecole che atterrano sulla superficie di misurazione (Figura 4).
- Se la densità dell'evento di atterraggio non è "ideale" (Figura 4), modificare la portata nel software di controllo microfluidico. Se la densità degli eventi è troppo bassa, aumentare la portata del campione fino a quando non si osservano eventi di atterraggio ben separati (ma non superare gli 8 μL/min). Se è troppo alto, ridurre la portata del campione (ma non scendere sotto 0,1 μL/min).
- Se il volume del calibrante si sta esaurendo nella provetta del campione, modificare la posizione dell'interruttore m sulla linea PBS pH 7.4 (posizione 1) per evitare l'iniezione di aria.
- Premere Registra ed eseguire una misurazione di 60 s. Salvare il file in una cartella scelta.
NOTA: Il software di acquisizione dati produce file con estensione .mp.
7. Pulizia della linea di campionamento e arresto del flusso
- Nel software di controllo della microfluidica, passare alla posizione 2 dell'interruttore m e modificare la pressione del campione a 800 mbar (Figura 2). Lavare il sistema con CS2 (NaOH) per 4 min.
- Passare alla posizione 3 dell'interruttore m e lavare il sistema con CS3 (IPA) per 4 min.
- Passare alla posizione 1 dell'interruttore m e lavare il sistema con CS1 (PBS) per 4 min.
- Nel software di controllo microfluidico, arrestare tutto il flusso impostando le pressioni della linea di campionamento e della linea tampone su 0 e chiudere la valvola tampone.
- Scollegare il chip dal tavolino e riposizionarlo sulla piastra di preparazione. Scollegare tutti i tubi e posizionare l'estremità del tubo del campione in un flacone di scarico. Pulire l'obiettivo con isopropanolo e salviette.
8. Misurazione del campione di fotometria di massa
- Svitare il tappo collegato al flacone del tampone da 200 ml (pH 7,4) e avvitarlo al flacone del tampone da 200 ml (pH 5,0).
- Ripeti i passaggi 3.4-5.6, ma usa il secondo canale del chip invece del primo.
- Nel software di controllo della microfluidica, portare l'interruttore m in posizione 5 (corrispondente al campione), impostare la portata della linea di campionamento su 8 μL/min e assicurarsi che la pressione della linea di campionamento non superi i 350 mbar (Figura 2).
- Ripetere i passaggi 6.2-6.4 per misurare il campione.
- Per calcolare le portate da utilizzare, assicurarsi che la differenza di piegatura della portata corrisponda al fattore di diluizione del campione desiderato. Ad esempio, per ottenere la diluizione di 2000x qui, le portate differiscono di un fattore di 2000; la portata del tampone è di 1000 μL/min e la portata del campione è di 0,5 μL/min. Al termine di un esperimento, lasciare sempre pulite le linee seguendo il protocollo di pulizia.
9. Analisi dei dati
- Una volta terminata l'acquisizione dei dati, avviare il software di analisi dei dati (Figura 5).
- Fare clic sull'icona più (+) in alto a sinistra e selezionare il file di calibrazione .mp. Il software inizierà ad analizzare il file caricato. A seconda delle dimensioni e del numero di misurazioni, l'operazione potrebbe richiedere alcuni minuti
- Non analizzare i file .mp nel software di analisi dei dati durante l'acquisizione dei dati con il software di acquisizione dati perché ciò potrebbe ridurre la qualità dei dati acquisiti.
- Premere il pulsante Crea calibrazione di massa (in basso a destra). Si aprirà una finestra di dialogo che mostra una tabella popolata con i valori di contrasto dei picchi adattati.
- Modificare i valori con i valori di massa noti per i picchi adattati (per la β-amilasi questi valori sono 56 kDa, 112 kDa e 224 kDa) e premere Salva. Il file di calibrazione appena creato (estensione .mc) apparirà sul pannello Calibrazione di massa (in basso a sinistra del software di analisi dei dati) (Figura 6).
- Fare clic sull'icona più (+) in alto a sinistra e selezionare il file di esempio .mp.
- Per creare un istogramma di massa, come mostrato nella Figura 5, passare alla scheda Analisi , selezionare la modalità Istogramma e l'opzione Grafico di massa . Regolate la larghezza del contenitore, i limiti di massa e altri parametri in base alle esigenze.
- Personalizzare ulteriormente il grafico nella scheda Figure , se lo si desidera, prima di esportare le figure e/o salvare l'intero spazio di lavoro come file .dmp.
Risultati
La fotometria di massa è stata utilizzata per misurare l'interazione tra l'anticorpo monoclonale IgG trastuzumab e il dominio solubile del recettore IgG neonatale Fc (FcRn). Una miscela 1:10 delle due proteine (a 2 μM per IgG e 20 μM per FcRn) è stata diluita manualmente a 10 nM e 20 nM in PBS, rispettivamente.
La fase di diluizione è necessaria perché la fotometria di massa, una tecnica di misurazione della massa a singola molecola, può analizzare solo campioni nell'intervallo 100 pM-100 nM. Il tentativo di misurare campioni con concentrazioni al di fuori di questo intervallo può compromettere l'accuratezza dei risultati (Figura 4). Questo esperimento non è incluso in questo protocollo, come è stato descritto in precedenza 5,6.
Negli istogrammi di massa prodotti in un esperimento di fotometria di massa, l'intensità del segnale di scattering (o "contrasto") per ogni evento di atterraggio viene tracciata sull'asse x e viene convertita in massa molecolare tramite la fase di calibrazione del contrasto di massa. Nel frattempo, l'asse y indica il numero di molecole contate con una data massa (o contrasto). Pertanto, un picco indica la presenza di una popolazione di molecole all'interno dell'intervallo di pesi molecolari mostrato sull'asse x. Il numero di eventi (conteggi) che costituiscono il picco riflette le dimensioni di quella popolazione.
La misurazione della fotometria di massa con diluizione manuale ha portato a un istogramma di massa in cui i due picchi più grandi, basati sui pesi molecolari attesi delle proteine, corrispondevano ai monomeri FcRn non legati (~50 kDa) e ai monomeri anticorpali IgG (~150 kDa) (Figura 7). Analogamente ai dati di fotometria di massa precedentemente pubblicati5, le specie non legate erano prominenti, mentre i picchi negli intervalli di massa che corrisponderebbero ai complessi erano molto meno evidenti.
L'esperimento è stato ripetuto utilizzando un sistema microfluidico a rapida diluizione per diluire la miscela alla concentrazione necessaria immediatamente prima della misurazione della fotometria di massa. Questo approccio ha permesso di rilevare ulteriori complessi a bassa affinità, che potrebbero essersi dissociati durante la fase di diluizione manuale. Per ottenere lo stesso fattore di diluizione 2000x utilizzato durante l'esperimento di diluizione manuale, la miscela di campioni 1:10 (a una concentrazione di 2 μM per IgG e 20 μM per FcRn) è stata fatta scorrere sul chip microfluidico a una velocità di 0,5 μL/min, insieme al tampone PBS (pH 5,0) a una velocità di flusso di 1 mL/min. Per garantire che le proteine non fossero degradate al momento della misurazione, IgG (2 μM) e FcRn (20 μM) sono state misurate sotto flusso con il sistema microfluidico dopo 120 minuti di incubazione dei campioni. Le misurazioni dei singoli controlli non hanno mostrato alcuna degradazione delle proteine (Figura 1 supplementare)
Per il campione sottoposto a rapida diluizione, si sono potuti nuovamente osservare i picchi corrispondenti ai monomeri FcRn (53 kDa), ai dimeri FcRn (97 kDa) e ai monomeri IgG (148 kDa). Inoltre, sono stati chiaramente osservati due picchi aggiuntivi a 196 kDa e 251 kDa, corrispondenti a complessi IgG-FcRn con stechiometrie 1:1 e 1:2 (Figura 7). Le masse previste per questi due complessi erano rispettivamente di 200 kDa e 250 kDa. La variabilità nella misurazione della massa rientra nell'errore di misurazione per il fotometro di massa utilizzato in questo studio è ±5%14 (2% per il complesso 1:1 e 0,4% per il complesso 1:2).
La presenza di questi complessi solo nel campione rapidamente diluito è coerente con l'idea che essi tendano a dissociarsi a concentrazioni più basse, ad una velocità che è veloce rispetto a un processo di diluizione manuale ma lenta rispetto al processo di diluizione rapida ottenuto con il sistema microfluidico15.
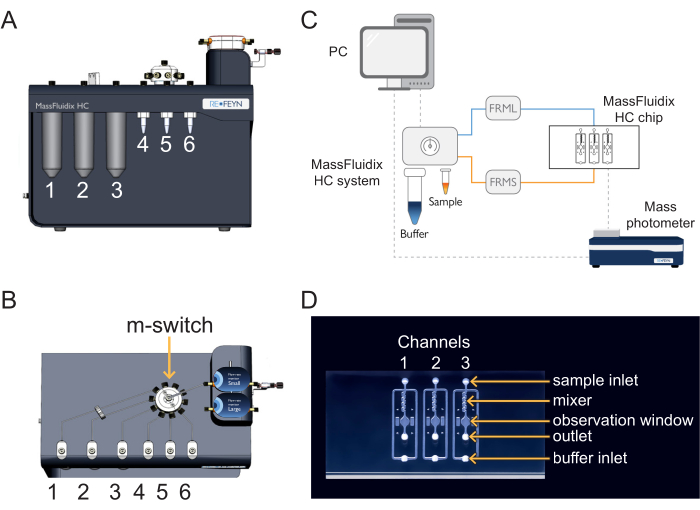
Figura 1: Combinazione di microfluidica e fotometria di massa. (A,B) La scatola di microfluidica utilizzata in questo protocollo, vista dalla parte anteriore (A) e dalla parte superiore (B). Le soluzioni detergenti sono poste nelle posizioni 1-3 e i campioni e i calibranti nelle posizioni 4-6. Tutte le soluzioni sono collegate all'm-switch, che è collegato al sensore dell'unità di flusso "Small". (C) Panoramica dell'intero sistema. Il computer (in alto a sinistra) è collegato alla scatola di microfluidica (mostrata accanto al tampone e alle provette del campione) e al fotometro di massa (in basso a destra), dove si trova il chip di microfluidica. Il monitor della portata piccola (FRMS) monitora il flusso attraverso la linea di campionamento, mentre il monitor della portata grande (FRML) monitora il flusso del tampone. Il tubo della linea del campione e del tampone si collega a un canale sul chip microfluidico, posizionato all'interno del fotometro di massa. (D) Ogni chip ha tre canali. L'FRMS è collegato all'ingresso del campione e l'FRML all'ingresso del tampone. L'area del miscelatore è il luogo in cui avviene la diluizione rapida del campione, mentre l'area di osservazione è il luogo in cui vengono eseguite le misurazioni della fotometria di massa. L'uscita è monitorata da un sensore di flusso aggiuntivo per garantire che non vi siano perdite nel chip. Fare clic qui per visualizzare una versione più grande di questa figura.
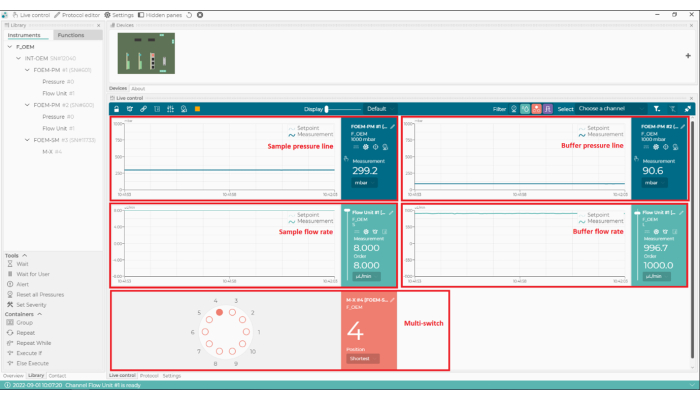
Figura 2: Il software di controllo della microfluidica. Con questo software, impostare e monitorare le portate, le linee di pressione del sistema microfluidico e la posizione del multi-switch (m-switch). Fare clic qui per visualizzare una versione più grande di questa figura.
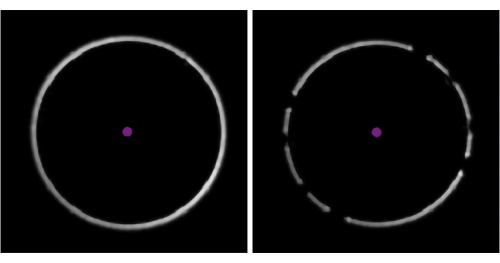
Figura 3: Identificazione della presenza di bolle nell'olio. Gli esempi del software di acquisizione dati mostrano un anello di messa a fuoco chiaro e ininterrotto (a sinistra) e un anello "rotto", che indica la presenza di bolle d'aria nell'olio di immersione (a destra). Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Esempi rappresentativi di concentrazioni di campioni in cui il numero di eventi di atterraggio di molecole è troppo poco, ideale o eccessivo. Le molecole che atterrano sulla superficie dell'area di osservazione nel chip microfluidico appariranno come macchie scure nella vista raziometrica dell'immagine di fotometria di massa. In modo ottimale, la concentrazione desiderata dovrebbe consentire una densità ideale di eventi di atterraggio durante l'acquisizione dei dati (al centro). Se la densità dell'evento di atterraggio è troppo bassa (in alto, 'troppo pochi'), non è possibile completare un'analisi statistica accurata dei dati di fotometria di massa. Se è troppo alto (in basso, "Troppi"), le molecole di atterraggio si sovrapporranno spazialmente, il che si tradurrà in una scarsa qualità dei dati. Queste immagini sono state catturate con il software di acquisizione dati utilizzando un fotometro di massa. Fare clic qui per visualizzare una versione più grande di questa figura.
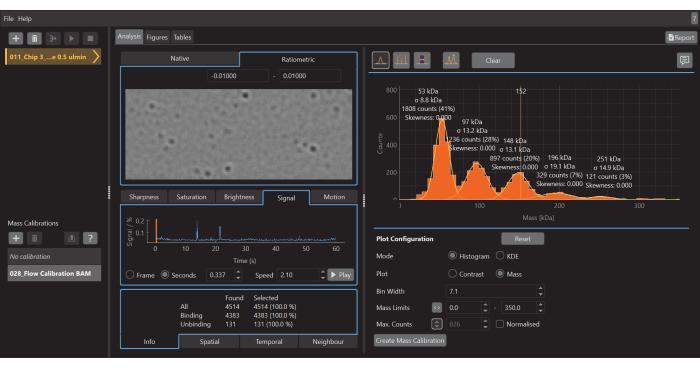
Figura 5: Una schermata del software di analisi dei dati per la fotometria di massa. Qui è possibile caricare i file .mp esportati dal software di acquisizione dati per analizzare i dati. Dai dati elaborati è possibile generare figure. Fare clic qui per visualizzare una versione più grande di questa figura.
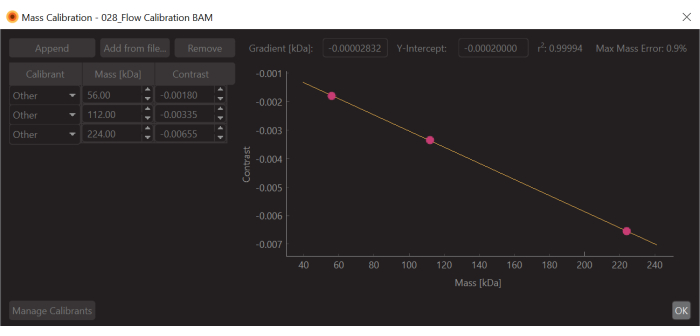
Figura 6: Una schermata della calibrazione del contrasto di massa per questo esperimento. Il calibrante utilizzato era la β-amilasi, che è nota per formare tre specie: monomero (56 kDa), dimero (112 kDa) e tetramero (224 kDa). Fare clic qui per visualizzare una versione più grande di questa figura.
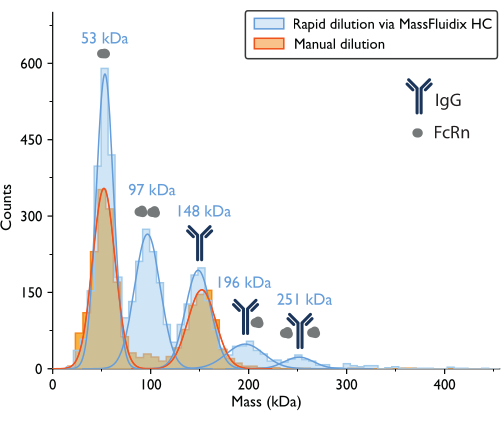
Figura 7: Gli istogrammi di massa rivelano la formazione di complessi solo dopo una rapida diluizione del campione. Gli istogrammi di massa e le corrispondenti distribuzioni gaussiane best-fit sono mostrati per le misurazioni di campioni contenenti IgG e FcRn dopo la diluizione manuale (arancione) o la diluizione rapida tramite il sistema microfluidicoHC (blu). Le etichette di massa indicano che i valori medi delle curve gaussiane si adattano agli istogrammi di massa misurati dopo una rapida diluizione. Dopo la diluizione manuale, sono stati osservati picchi corrispondenti ai monomeri FcRn (52 kDa, misurati con la diluizione manuale) e ai monomeri IgG (152 kDa). Dopo una rapida diluizione, oltre ai monomeri FcRn e IgG, sono stati chiaramente osservati anche picchi corrispondenti a dimeri FcRn e complessi IgG-FcRn con stechiometria 1:1 e 1:2. Fare clic qui per visualizzare una versione più grande di questa figura.
Dati supplementari Figura 1: Le misurazioni della fotometria di massa dei campioni di controllo dopo una rapida diluizione non mostrano alcuna degradazione delle proteine. (A) Istogramma di massa e distribuzione gaussiana best-fit per il campione di solo FcRn. La concentrazione iniziale del campione prima della diluizione rapida era di 20 μM e la velocità di flusso è stata impostata su 0,5 μL/min. Si sono potuti osservare picchi di massa corrispondenti ai monomeri di FcRn (53 kDa) e ai dimeri di FcRn (97 kDa). (B) Istogramma di massa e distribuzione gaussiana più adatta per il campione di sole IgG (Herceptin). La concentrazione iniziale del campione prima della diluizione rapida era di 2 μM e la velocità di flusso era impostata su 1 μL/min. È stato osservato un singolo picco di massa corrispondente al monomero IgG (155 kDa). Fare clic qui per scaricare questo file.
Discussione
Il protocollo qui delineato fornisce un metodo per rilevare e quantificare le interazioni proteina-proteina a bassa affinità. Utilizza un fotometro di massa accoppiato a un sistema microfluidico a rapida diluizione. La fotometria di massa è uno strumento bioanalitico senza marcatura in grado di misurare in modo affidabile la massa molecolare in soluzione per le biomolecole16, per quelle comprese tra 30 kDa e 6 MDa. Poiché la fotometria di massa è una tecnica a singola molecola che analizza i campioni uno per uno, è generalmente limitata ai campioni nell'intervallo di concentrazione 100 pM-100 nM. Al di sopra di questo intervallo, le molecole che atterrano sulla superficie del vetro si sovrapporranno spazialmente, con conseguente scarsa qualità dei dati; Al di sotto di questo intervallo, si ottengono troppo pochi dati per eseguire un'analisi robusta7. Una conseguenza importante è che può limitare lo studio delle interazioni proteiche a quelle che formano una miscela di specie legate e non legate all'interno di quell'intervallo.
Qui, abbiamo dettagliato un protocollo passo-passo per l'utilizzo di un sistema microfluidico a rapida diluizione per espandere efficacemente l'intervallo di concentrazioni del campione suscettibili di fotometria di massa. Diluendo il campione sul chip microfluidico e quindi facendolo scorrere attraverso la finestra di osservazione del rivelatore entro 50 ms, il sistema cattura i complessi presenti nel campione non diluito prima che l'equilibrio di interazione si sposti. Il campione viene continuamente consegnato al rivelatore durante le singole misurazioni. In queste condizioni, il 95% del complesso rimarrà intatto quando il campione viene misurato, anche per interazioni a bassa affinità, con un KD dell'ordine dei micromolari e tassi di dissociazione fino a 1 s-1.
Questo può essere calcolato come segue: Per una reazione 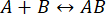 con una velocità diretta kfe una velocità all'indietro kb,
con una velocità diretta kfe una velocità all'indietro kb,
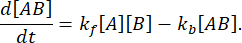
All'equilibrio, le concentrazioni di tutte e tre le specie (A, B e il complesso AB) rimangono costanti, quindi 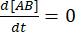 e
e 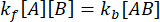 . Sotto l'ipotesi conservativa che la perturbazione (diluizione, in questo caso) possa causare la dissociazione del complesso, ma la reazione diretta (associazione) non proceda, il termine kf [A] [B] può essere trattato come trascurabile e si può fare la seguente semplificazione:
. Sotto l'ipotesi conservativa che la perturbazione (diluizione, in questo caso) possa causare la dissociazione del complesso, ma la reazione diretta (associazione) non proceda, il termine kf [A] [B] può essere trattato come trascurabile e si può fare la seguente semplificazione:
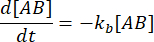
L'integrazione dà la seguente espressione per la concentrazione del complesso nel tempo dopo la perturbazione dell'equilibrio:
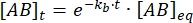
La frazione del complesso che rimane legata al tempo t dopo la perturbazione dell'equilibrio è quindi:
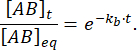
A = 50 ms, per una reazione con kb≈ 1 s-1, il limite di frazione è 0,95, o 95%11,12.
La fotometria di massa è stata utilizzata qui e in precedenza5 per studiare il legame dell'anticorpo monoclonale IgG trastuzumab al dominio solubile del FcRn. È stato riportato che i due partner di legame si legano con affinità nanomolare a pHacido 17. La fotometria di massa è stata utilizzata per valutare qualitativamente l'abbondanza dei complessi formati mentre i partner di legame erano a pH 5,0 e i campioni sono stati rapidamente diluiti attraverso un ulteriore sistema microfluidico. La procedura è stata ottimizzata per la particolare interazione proteina-proteina sulla base dei risultati precedentemente riportati5. La stessa procedura può essere utilizzata per studiare altre interazioni, a condizione che gli utenti abbiano conoscenze preliminari o ottimizzino le condizioni sperimentali per il sistema in questione, come i tamponi da utilizzare, la concentrazione iniziale della proteina, la stechiometria attesa e la quantità di incubazione necessaria per consentire all'interazione di raggiungere un equilibrio.
Quando la miscela IgG-FcRn è stata diluita manualmente, è stato difficile rilevare la presenza di complessi IgG-FcRn, anche se è noto che queste proteine interagiscono5. Questo articolo mostra che l'approccio della diluizione rapida si traduce in una quantità notevolmente maggiore di questi complessi. Per lo stesso campione, quando è stata utilizzata la diluizione rapida, sono stati entrambi chiaramente osservati complessi FcRn-IgG 1:1 e complessi FcRn-IgG 2:1. Queste differenze nella formazione di complessi dimostrano l'importanza di studiare i sistemi di interazione biomolecolare in un'ampia gamma di concentrazioni.
Inoltre, questi risultati dimostrano anche che è semplice utilizzare la microfluidica con l'analisi di singole molecole per catturare interazioni deboli, colmando una lacuna significativa nel metodo. La combinazione della microfluidica a rapida diluizione con la fotometria di massa offre vantaggi interessanti grazie ai vantaggi della fotometria di massa come tecnica analitica. Cioè, la fotometria di massa non richiede etichette, comporta una preparazione minima del campione e le misurazioni vengono eseguite in soluzione. Per questo protocollo, un altro vantaggio chiave della fotometria di massa è la sua capacità di distinguere e quantificare tutte le specie formate (a condizione che abbiano una massa distinta di >30 kDa). Ciò è in contrasto con l'SPR, ad esempio, che può misurare i tassi di legame e dislegatura ma non può fornire prontamente informazioni stechiometriche8.
Per questo protocollo, così come per gli esperimenti di fotometria di massa più in generale, sono utili diverse considerazioni. In primo luogo, la concentrazione finale della proteina dovrebbe rientrare nel limite di ciò che la fotometria di massa può misurare (100 pM-100 nM). Anche la concentrazione iniziale di incubazione dovrebbe essere all'interno dell'intervallo del sistema microfluidico (fino a 90 μM) e teorizzata essere superiore all'effettivo KD dell'interazione10. Il punto di partenza raccomandato è un rapporto di miscelazione della concentrazione 1:1 tra le specie interagenti alla concentrazione di μM. Il rapporto potrebbe quindi essere variato a 1:2, 1:5 o, come nel caso di questa interazione, 1:10. Se non ci sono informazioni precedenti sulle interazioni proteiche, l'utente dovrebbe ottimizzare l'esperimento, iniziando con un'alta concentrazione (consigliata 20 μM) per ciascun partner per determinare se l'affinità dei componenti rientra nell'intervallo di concentrazione sostenuto dal metodo presentato (cioè, si formano complessi). L'ottimizzazione potrebbe anche comportare la scelta di altre condizioni tampone per promuovere le interazioni o la titolazione di uno dei componenti di interazione per determinare il giusto rapporto di miscelazione. Una volta determinati, è possibile ottimizzare le concentrazioni e i flussi per consentire condizioni ottimali per lo studio e il metodo, ad esempio diminuendo le concentrazioni per consentire una migliore risoluzione dei picchi.
In secondo luogo, per replicare con successo questo esperimento, le impurità dovrebbero essere ridotte al minimo. Le fonti comuni di impurità che sono note per influenzare negativamente le misurazioni della fotometria di massa includono altre proteine o detriti cellulari che rimangono dopo la purificazione, tamponi non filtrati, detergenti micellatori (se presenti a una concentrazione troppo elevata) e tamponi contenenti alte concentrazioni di sale, glicerolo o altri componenti. Come discusso nel Protocollo di cui sopra, le bolle nel sistema microfluidico dovrebbero essere rimosse. Possono formarsi bolle nel sistema di tubi o se i campioni hanno un'elevata tensione superficiale e sono soggetti a formazione di schiuma. Nell'olio di immersione possono formarsi anche delle bolle, che possono essere rilevate dall'anello di messa a fuoco (Figura 3). Se le bolle non possono essere rimosse utilizzando i passaggi descritti nel protocollo, un'altra soluzione consiste nel degassare il campione utilizzando un essiccatore e una pompa a vuoto, lasciando il campione a pressione ridotta per alcuni minuti. Il vortice o l'agitazione di soluzioni proteiche altamente concentrate non è raccomandato in quanto queste azioni possono favorire la formazione di bolle.
Mentre la misurazione di una specifica interazione proteina-proteina è dimostrata qui, lo stesso protocollo può essere applicato ad altri sistemi di interazione proteina-proteina senza modifiche significative. Un'ulteriore direzione futura di questo protocollo sarebbe quella di utilizzare le misurazioni per calcolare i valori KD per i complessi identificati, come è stato descritto altrove nel contesto della fotometria di massa 5,7. Mentre gli studi precedenti utilizzavano dati provenienti da esperimenti che coinvolgevano la diluizione manuale e interazioni più forti, il principio di analisi potrebbe essere facilmente applicato in questo contesto, a condizione che vengano implementati ulteriori miglioramenti nel dispositivo microfluidico (come una maggiore precisione del sensore di flusso e stabilità della pompa).
Oltre alle interazioni proteina-proteina, è probabile che ci siano applicazioni più ampie per la fotometria di massa combinata e l'approccio microfluidico a rapida diluizione. La fotometria di massa può essere utilizzata per valutare la purezza, l'aggregazione e l'omogeneità del campione18,19; studiare l'oligomerizzazione delle proteine20, l'assemblaggio macromolecolare21 o la polimerizzazione22; e in altre aree. L'analisi della fotometria di massa si estende anche oltre le proteine; È stato utilizzato per studiare le interazioni tra acidi nucleici e proteine23, particelle virali24 e nanoparticelle25. Questo protocollo descrive quindi un'importante applicazione di un sistema microfluidico combinato di fotometria di massa: consente la misurazione diretta delle deboli interazioni proteina-proteina a livello di singole molecole e complessi. Il valore della presente applicazione è elevato, in quanto apre la possibilità di caratterizzare direttamente interazioni che sono state generalmente difficili da studiare, con rilevanza in aree terapeutiche critiche. Questo approccio combinato potrebbe anche servire come base per una gamma più ampia di indagini per campioni con concentrazioni fino a decine di micromolari.
Divulgazioni
Myndert Claasen e Zornitsa Kofinova sono dipendenti di Refeyn Ltd, che produce il fotometro di massa e il sistema microfluidico utilizzati in questo articolo. Weston Struwe è azionista e consulente di Refeyn Ltd.
Riconoscimenti
W.S. è supportato da una borsa di studio UKRI Future Leaders [MR/V02213X/1]. Il testo e la grafica del manoscritto sono stati preparati con il supporto dei membri del team di comunicazione scientifica di Refeyn (Panagiota Paganopoulou, Neus Torres Tamarit e Catherine Lichten). Ringraziamo anche il prezioso feedback di Camille Hetez, Sofia Ferreira e Matthias Langhorst.
Materiali
| Name | Company | Catalog Number | Comments |
| 2-Propanol (Isopropanol) | VWR International LLC | 20880.320 | |
| Data acquisition software | Refeyn | AcquireMP (v2022 R1) | |
| Data analysis software | Refeyn | DiscoverMP (v2022 R1) | |
| FCRN, His-Tag | Sigma | SRP0624 | |
| Herceptin (IgG) | Cambridge Bioscience | HY-P9907-1mg | |
| Mass photometer | Refeyn | TwoMP | |
| Microfluidics box | Refeyn | MassFluidix HC system | |
| Microfluidics chip | Refeyn | MassFluidix HC chip | |
| Microfluidics control software | Fluigent | OxyGEN | |
| Phosphate Buffered Saline (PBS), 1x Ultra Pure | VWR International LLC | K812 | |
| Sodium Hydroxide (NaOH) | Sigma | S2770 | |
| β-Amylase, from sweet potato | Sigma | A8781 |
Riferimenti
- Arter, W. E., Levin, A., Krainer, G., Knowles, T. P. J. Microfluidic approaches for the analysis of protein-protein interactions in solution. Biophysical Reviews. 12 (2), 575-585 (2020).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry. B. 122 (49), 11554-11560 (2018).
- Li, Z. Editorial: Methods in structural biology: Cryo-electron microscopy. Frontiers in Molecular Biosciences. 9, 1041386 (2022).
- Herling, T. W., et al. A microfluidic platform for real-time detection and quantification of protein-ligand interactions. Biophysical Journal. 110 (9), 1957-1966 (2016).
- Soltermann, F., et al. Quantifying protein-protein interactions by molecular counting with mass photometry. Angewandte Chemie International Edition. 59 (27), 10774-10779 (2020).
- Wu, D., Piszczek, G. Rapid determination of antibody-antigen affinity by mass photometry. Journal of Visualized Experiments: JoVE. 168, 61784 (2021).
- Wu, D., Piszczek, G. Measuring the affinity of protein-protein interactions on a single-molecule level by mass photometry. Analytical Biochemistry. 592, 113575 (2020).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- MassFluidix® HC system for rapid dilution via microfluidics. Available from: https://www.refeyn.com/massfluidix-hc-system (2023)
- Pollard, T. D. A guide to simple and informative binding assays. Molecular Biology of the Cell. 21 (23), 4061-4067 (2010).
- Jarmoskaite, I., AlSadhan, I., Vaidyanathan, P. P., Herschlag, D. How to measure and evaluate binding affinities. eLife. 9, e57264 (2020).
- Monnet, C., et al. Selection of IgG variants with increased FcRn binding using random and directed mutagenesis: Impact on effector functions. Frontiers in Immunology. 6, 39 (2015).
- . Refeyn TwoMP: Transforming biomolecular characterisation Available from: https://www.refeyn.com/twomp-mass-photometer (2022)
- Lai, S. -. H., Tamara, S., Heck, A. J. R. Single-particle mass analysis of intact ribosomes by mass photometry and Orbitrap-based charge detection mass spectrometry. iScience. 24 (11), 103211 (2021).
- Wu, D., Piszczek, G. Standard protocol for mass photometry experiments. European Biophysics Journal. 50 (3-4), 403-409 (2021).
- Vaughn, D. E., Bjorkman, P. J. Structural basis of pH-dependent antibody binding by the neonatal Fc receptor. Structure. 6 (1), 63-73 (1998).
- Niebling, S., et al. Biophysical screening pipeline for Cryo-EM grid preparation of membrane proteins. Frontiers in Molecular Biosciences. 9, 882288 (2022).
- Paul, S. S., Lyons, A., Kirchner, R., Woodside, M. T. Quantifying oligomer populations in real time during protein aggregation using single-molecule mass photometry. ACS Nano. 16 (10), 16462-16470 (2022).
- Schulz, L., et al. Evolution of increased complexity and specificity at the dawn of form I Rubiscos. Science. 378 (6616), 155-160 (2022).
- Malay, A. D., et al. An ultra-stable gold-coordinated protein cage displaying reversible assembly. Nature. 569 (7756), 438-442 (2019).
- Hundt, N., Cole, D., Hantke, M. F., Miller, J. J., Struwe, W. B., Kukura, P. Direct observation of the molecular mechanism underlying protein polymerization. Science Advances. 8 (35), eabm7935 (2022).
- Acharya, A., et al. Distinct RPA domains promote recruitment and the helicase-nuclease activities of Dna2. Nature Communications. 12, 6521 (2021).
- Ebberink, E. H. T. M., Ruisinger, A., Nuebel, M., Thomann, M., Heck, A. J. R. Assessing production variability in empty and filled adeno-associated viruses by single molecule mass analyses. Molecular Therapy - Methods & Clinical Development. 27, 491-501 (2022).
- Melo, L., et al. Size distributions of gold nanoparticles in solution measured by single-particle mass photometry. The Journal of Physical Chemistry B. 125 (45), 12466-12475 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon