Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Identification de cellules quiescentes dans un modèle de leucémie lymphoblastique aiguë à cellules T de poisson-zèbre à l’aide de la coloration de la prolifération cellulaire
Dans cet article
Résumé
Nous avons utilisé la coloration de prolifération cellulaire pour identifier les cellules quiescentes dans le modèle de leucémie lymphoblastique aiguë T-poisson zèbre. La coloration est conservée dans les cellules non divisées et réduite pendant la prolifération cellulaire, ce qui permet de sélectionner les cellules dormantes pour une interrogation plus approfondie. Ce protocole fournit un outil fonctionnel pour étudier l’auto-renouvellement dans le contexte de la quiescence cellulaire.
Résumé
La quiescence cellulaire est un état d’arrêt de la croissance ou de prolifération ralentie qui est décrit dans les cellules souches normales et cancéreuses (CSC). La quiescence peut protéger les CSC contre les agents chimiothérapeutiques antiprolifératifs. Dans les modèles murins de xénogreffe dérivée de patients (PDX) de leucémie aiguë lymphoblastique à cellules T (T-LLA), les cellules quiescentes sont associées à la résistance au traitement et à la souche. Les colorants de prolifération cellulaire sont des outils populaires pour le suivi de la division cellulaire. Le colorant fluorescent est ancré de manière covalente dans des groupes d’amines sur la membrane et des macromolécules à l’intérieur de la cellule. Cela permet de suivre les cellules marquées jusqu’à 10 divisions, qui peuvent être résolues par cytométrie en flux.
En fin de compte, les cellules ayant les taux de prolifération les plus élevés auront une faible rétention de colorant, car il sera dilué à chaque division cellulaire, tandis que les cellules dormantes à division plus lente auront la rétention la plus élevée. L’utilisation de colorants de prolifération cellulaire pour isoler les cellules dormantes a été optimisée et décrite dans des modèles murins T-LAL. En complément des modèles murins existants, le modèle T-ALL dérivé du poisson-zèbre rag2 :Myc offre un excellent moyen d’interroger l’auto-renouvellement dans la T-ALL en raison de la fréquence élevée des cellules souches leucémiques (LSC) et de la commodité du poisson-zèbre pour les expériences de transplantation à grande échelle.
Ici, nous décrivons le flux de travail pour la coloration des cellules T-ALL du poisson-zèbre avec un colorant de prolifération cellulaire, l’optimisation de la concentration du colorant pour les cellules de poisson-zèbre, le passage des cellules colorées avec succès in vivo et la collecte de cellules avec différents niveaux de rétention de colorant par tri de cellules vivantes d’animaux transplantés. Étant donné l’absence de fabricants de surface cellulaire bien établis pour les LSC dans la T-ALL, cette approche fournit un moyen fonctionnel d’interroger les cellules quiescentes in vivo. Pour des résultats représentatifs, nous décrivons l’efficacité de la greffe et la fréquence LSC des cellules à haute et basse rétention de colorant. Cette méthode peut aider à étudier d’autres propriétés des cellules quiescentes, notamment la réponse aux médicaments, les profils transcriptionnels et la morphologie.
Introduction
Les cellules souches adultes sont responsables de la régénération des types de cellules différenciées dans un organe donné et sont principalement présentes dansun état dormant et non divisé1,2. Par exemple, les cellules souches hématopoïétiques (CSH), qui maintiennent le sang, restent en grande partie quiescentes, et seule une petite fraction entre dans le cycle cellulaire pour s’auto-renouveler ou se différencier afin de générer des composants sanguins matures3. De même, dans les cancers, une sous-population rare de cellules appelées cellules souches cancéreuses (CSC) possède la capacité de s’auto-renouveler et est responsable du maintien à long terme de la malignité4. Les cellules souches cancéreuses existent in vivo dans un état de quiescence ou de croissance lente, ce qui peut leur permettre d’échapper aux traitements anti-prolifératifs contre le cancer5, d’échapper à l’élimination par le système immunitaire6, de réduire le stress oxydatif et d’améliorer leurs voies de réparation de l’ADN7. Même un petit nombre de CSC laissés après le traitement peut potentiellement repeupler la tumeur, entraînant une rechute du patient8. Par conséquent, la compréhension de la quiescence cellulaire est très prometteuse pour l’identification des vulnérabilités potentielles des CSC et le développement de nouvelles façons de les cibler.
Les colorants de prolifération cellulaire, tels que la coloration à l’ester succinimidylique de carboxyfluorescéine (CFSE) et ses dérivés, sont couramment utilisés pour suivre la fréquence des divisions cellulaires9. Le colorant s’infiltre à travers la membrane cellulaire et, une fois à l’intérieur de la cellule, subit une activation par des estérases intracellulaires en un produit fluorescent. Le composé fluorescent résultant est retenu à l’intérieur de la cellule par les liaisons amide covalentes formées entre la fraction succinimidyle et les groupes fonctionnels amines des protéines intracellulaires10. À chaque division cellulaire, le composé fluorescent est divisé également entre les deux cellules résultantes, provoquant une double dilution du signal. Ce colorant permet de détecter jusqu’à 10 divisions cellulaires grâce à l’analyse par cytométrie en flux11.
Cette approche a déjà été utilisée pour enrichir les populations de CSC in vitro en identifiant les populations de cellules à cycle lent avec une rétention élevée du colorant11,12. Dans la LAL-T, le CFSE a été utilisé pour suivre la croissance tumorale in vivo dans des xénogreffes dérivées de patients chez la souris. Après le marquage cellulaire et trois semaines après la greffe, l’analyse par cytométrie en flux a montré une population rare de cellules qui conservaient encore la fluorescence CFSE. Cette population était associée à la souche, à la résistance au traitement et à une grande similitude avec les cellules responsables de la rechute chez les patients13. En conséquence, ce colorant fournit un outil utile pour l’étude des phénotypes des cellules souches leucémiques (LSC) dans la LAL-T.
L’objectif du travail est d’étendre l’application du colorant de prolifération cellulaire à l’étude de la quiescence in vivo à l’aide d’un modèle T-ALL de poisson-zèbre. En particulier, le modèleT-ALL 14 du poisson-zèbre piloté par rag2 :Myc offre un excellent lieu pour l’étude de l’auto-renouvellement en raison de la fréquence élevée des LSC par rapport aux modèles murins et aux maladies humaines15. De plus, l’utilisation du poisson-zèbre permet des études de transplantation à grande échelle, qui peuvent être effectuées à un coût de soins et d’entretien beaucoup plus faible que leurs homologues souris16. Les poissons-zèbres sont également excellents pour les applications d’imagerie en direct, car les cellules tumorales marquées par fluorescence peuvent être facilement visualisées à l’aide d’un simple microscope à fluorescence pour estimer le taux de développement tumoral16.
Dans ce protocole, nous décrivons le flux de travail pour la coloration des cellules T-ALL du poisson-zèbre avec un colorant de prolifération cellulaire, suivie de la propagation in vivo des cellules colorées chez le poisson-zèbre syngénique CG1. Lors de l’apparition d’une leucémie, nous décrivons le tri des cellules qui ont conservé le colorant et leur utilisation pour une expérience ultérieure de transplantation à dilution limite afin de quantifier les taux d’auto-renouvellement des LSC. Ce protocole peut être étendu à d’autres applications, y compris le criblage in vivo de composés potentiels pour le ciblage des LSC quiescents. De plus, les cellules collectées peuvent être utilisées pour différentes analyses en aval, telles que le profilage transcriptomique, la protéomique et la métabolomique, offrant des informations uniques sur le comportement des LSC quiescentes dans la T-ALL.
Protocole
Dans ce protocole, nous utilisons des cellules T-ALL de poisson-zèbre marquées à la GFP qui ont été générées précédemment dans la souche CG1 et peuvent donc être directement injectées dans le poisson-zèbre syngénique CG115 du receveur. En bref, la leucémie a été générée par micro-injection d’ADN de rag2 :Myc et rag2 :GFP dans des embryons de poisson-zèbre CG1 unicellulaires. Les animaux ont été surveillés pour le développement d’une leucémie à partir de 3 semaines après l’injection, à l’aide de la microscopie à fluorescence. Des cellules leucémiques positives à la GFP ont été isolées par FACS et transplantées en série chez des poissons-zèbres CG1 receveurs pour générer des clones avec une fréquence élevée de LSC. Les détails de l’ensemble du protocole sont décrits par Blackburn et al.15.
Alternativement, la T-ALL primaire dérivée de rag2 :Myc peut être générée par micro-injection d’ADN d’embryons de poisson-zèbre17. La micro-injection d’ADN de rag2 :Myc avec un rapporteur fluorescent piloté par rag2 tel que la GFP peut entraîner le développement de cellules B, de cellules T et de ALL18 mixtes. Il a été précédemment vérifié que les clones de leucémie utilisés dans ce protocole étaient des T-ALL15. Toutes les procédures expérimentales impliquant le poisson-zèbre ont été examinées et approuvées par le comité institutionnel de soin et d’utilisation des animaux de l’Université du Kentucky, protocole 2019-3399.
1. Propagation des cellules T-ALL de poisson-zèbre in vivo
- Décongeler un flacon contenant 1 mL de cellules T-ALL de poisson-zèbre marquées à la GFP congelées dans un bain-marie à 37 °C en agitant doucement, jusqu’à ce qu’il soit décongelé. Une fois décongelé, transférez-le dans de la glace.
- Pipetez lentement le contenu du tube dans un tube conique de 15 ml avec 4 ml de milieu de poisson (0,9x PBS + 5% FBS) pour diluer le milieu de congélation.
- Faites tourner pendant 5 min à 2 500 × g à 4 °C. Retirez le support de la pastille. Remettre en suspension dans 0,5 mL de milieu de poisson et garder les cellules sur de la glace.
- Comptez les cellules et diluez-les si nécessaire à 100 000 cellules/5 μL.
- Anesthésie le poisson-zèbre CG1 adulte en ajoutant 200 μL d’un anesthésique à 4 mg/mL (MS-222) à 25 mL d’eau du système piscicole dans une boîte de Pétri. Tenez le poisson CG1, côté ventral vers le haut et injectez 5 à 6 μL dans l’espace intrapéritonéal (IP) à l’aide d’une seringue d’un microlitre Hamilton. Ramenez les animaux dans leurs bassins et surveillez le retour à la nage normale avant de les remettre dans le système.
REMARQUE : Des poissons-zèbres adultes, âgés de 60 jours ou plus, ont été utilisés pour les études de transplantation décrites dans ce protocole. - Surveiller la croissance de la leucémie une fois par semaine à l’aide d’un microscope à fluorescence. Voir la section 2 ci-dessous pour plus de détails sur la procédure de dépistage.
2. Prélèvement de cellules leucémiques marquées par fluorescence
- Environ 21 à 28 jours après la transplantation, anesthésier le poisson-zèbre CG1 adulte en ajoutant 200 μL (4 à 5 gouttes) d’anesthésique à 4 mg/mL à 25 mL d’eau du système piscicole dans une boîte de Pétri.
- Examinez le poisson-zèbre CG1 pour détecter la charge leucémique en évaluant le pourcentage de cellules leucémiques marquées à la GFP chez l’animal à l’aide d’un microscope épifluorescent, en utilisant le filtre approprié pour détecter la fluorescence de la GFP. La leucémie positive à la GFP est prête à être récoltée si elle s’est propagée à >70 % de l’animal.
- Euthanasier l’animal en ajoutant 1 mL d’anesthésique à 4 mg/mL dans une boîte de Pétri contenant 9 mL d’eau du système du poisson. Permettez à tous les signes de vie de cesser ; Par exemple, observez le poisson pour l’arrêt du mouvement operculaire (~5 min).
- Placez le poisson-zèbre dans une nouvelle boîte de Pétri et ajoutez 1 ml de milieu de poisson pour servir de tampon pour les cellules. À l’aide d’une nouvelle lame de rasoir, décapitez d’abord le poisson-zèbre, puis faites macérer le tissu à l’aide de la même lame de rasoir. Pipette de haut en bas pour déloger de gros amas de cellules.
- Passez les cellules à travers une crépine de 40 μm dans un tube conique de 50 mL pour les dissocier en une suspension unicellulaire. Gardez les cellules sur de la glace.
3. Optimisation de la concentration en colorant de prolifération cellulaire (CellTrace Far Red) pour la coloration et la viabilité cellulaire du poisson-zèbre
REMARQUE : Pour ce protocole, et puisque les cellules tumorales sont marquées à la GFP, nous avons utilisé un colorant de prolifération des cellules rouges lointaines (CellTrace Far Red) pour éviter le chevauchement spectral, qui sera appelé CT-FR.
- Préparez une solution de 1 mM de colorant fluorescent immédiatement avant l’utilisation en ajoutant le volume approprié de DMSO selon les recommandations du fabricant. Avoir des solutions de travail de 1 mM, 0,5 mM, 0,1 mM et 0,01 mM ; Diluer au besoin dans un milieu de poisson pour tester la viabilité cellulaire après la coloration.
- Comptez les cellules et prélevez 1 × 106 cellules dans des tubes de microcentrifugation de 1,5 ml.
- Centrifugeuse à 2 500 × g à 4 °C pendant 5 min. Retirez le liquide et remettez les cellules en suspension dans 1 mL de PBS 0,9x.
- Répartissez la suspension cellulaire entre quatre tubes de 250 μL chacun pour le traitement par CT-FR.
- Ajoutez 250 μL de PBS 0,9x pour porter le volume à 500 μL chacun avec environ 250 000 cellules/tube.
- Ajouter les quantités requises des solutions de travail décrites ci-dessus dans chacun des quatre tubes pour obtenir les concentrations finales suivantes : 1 μM, 0,5 μM, 0,1 μM et 0,05 μM. Incuber pendant 20 min à 37 °C, à l’abri de la lumière.
- Granulez les cellules en les faisant tourner à 2 500 × g pendant 5 min à température ambiante. Retirez le liquide et lavez les cellules avec 500 μL de milieu de poisson pour éliminer l’excès de colorant.
- Granulez à nouveau, retirez le liquide et mettez-le en suspension dans 25 μL de milieu de poisson. Utilisez 1 μL pour compter les cellules dans une dilution de 1:10 dans du bleu de trypan à 50 %.
- Examiner les cellules au microscope pour déterminer leur viabilité ; Les cellules vivantes ne conserveront pas le bleu trypan et apparaîtront brillantes au microscope, tandis que les cellules mortes absorberont le colorant et apparaîtront sombres. Examinez d’abord les cellules sous le canal de fluorescence approprié pour identifier les cellules tumorales, puis utilisez le canal en fond clair pour évaluer la viabilité cellulaire.
- Sélectionnez la concentration qui permet une coloration réussie tout en maintenant la viabilité cellulaire (>90 % de cellules négatives au bleu trypan).
REMARQUE : Pour les cellules T-ALL du poisson-zèbre testées dans cette expérience, une concentration de 1 μM a été utilisée pour une coloration réussie et >90 % de la viabilité cellulaire.
4. Propagation in vivo de cellules de poisson-zèbre T-ALL colorées CT-FR
- À l’aide de la concentration CT-FR optimisée de la section 3, colorez les cellules de poisson-zèbre. Laissez certaines cellules tumorales non colorées pour la génération de la tumeur T-ALL marquée à la GFP, en tant que contrôle positif à la GFP lors du tri FACS. Diluez les cellules colorées et non colorées à 50 000 cellules/5 μL pour la transplantation.
- Anesthésie le poisson-zèbre CG1 adulte en ajoutant 200 μL d’anesthésique à 4 mg/mL à 25 mL d’eau du système piscicole dans une boîte de Pétri.
- Injecter 5 μL de suspension cellulaire dans la cavité IP du poisson-zèbre comme décrit à la section 1 pour les cellules colorées et non colorées. Injectez 5 à 6 poissons-zèbres par groupe pour tenir compte de la mortalité potentielle après l’injection.
- Examinez les animaux par microscopie fluorescente sous sédation 2 fois par semaine pour le développement de la leucémie.
REMARQUE : Après ~10-14 jours, la leucémie devrait occuper environ 30-40% de ce corps. Planifiez la récolte de la leucémie et le tri ultérieur dans ce laps de temps. Attendre que la tumeur s’étende à une grande taille pourrait entraîner une plus grande dilution du colorant cellulaire fluorescent et une difficulté à détecter son signal. Envisager l’optimisation du moment de la récolte de la tumeur pour le modèle tumoral choisi. De plus, une surveillance tumorale plus fréquente est recommandée ici pour assurer la capture de la taille tumorale souhaitée.
5. Préparation au tri des cellules T-ALL de poisson-zèbre colorées au CT-FR
- Préparez les contrôles suivants pour le tri des cellules : Aucun contrôle de couleur (non coloré, non transgénique, suspension unicellulaire, provenant d’un poisson zèbre de type sauvage), cellules T-ALL marquées à la GFP et cellules de poisson-zèbre CT-FR WT nouvellement colorées (pour définir un seuil maximal de signal Far-Red).
- Préparation des contrôles non colorés et CT-FR
- Récoltez des cellules de poisson sauvage (WT) en euthanasiant un poisson-zèbre CG1 non transplanté en ajoutant 1 mL d’anesthésique à 4 mg/mL dans une boîte de Pétri contenant 9 mL d’eau du système piscicole.
- Placez le poisson-zèbre dans une nouvelle boîte de Pétri et ajoutez 1 ml de milieu de poisson pour servir de tampon pour les cellules. Utilisez une nouvelle lame de rasoir pour décapiter puis faire macérer le poisson-zèbre à l’aide de la même lame de rasoir ; Pipette de haut en bas pour déloger les gros amas de cellules.
- Passez les cellules à travers une crépine de 40 μm dans un tube conique de 50 mL pour les dissocier en une suspension unicellulaire. Gardez les cellules sur de la glace.
- Pour le contrôle CT-FR, transférez 250 000 cellules WT dans un tube de microcentrifugation et granulez les cellules par centrifugation à 2 500 × g pendant 5 min à température ambiante. Retirez le milieu de poisson et ajoutez 500 μL de FBS 0,9x PBS+ 1%.
- Ajouter 0,5 μL de la solution mère de colorant CT-FR de 1 mM pour une concentration finale de 1 μM. Pipeter doucement avec une pipette de 1 000 μL pour mélanger. Incuber à 37 °C pendant 20 min, à l’abri de la lumière.
- Retirez le liquide et lavez les cellules avec 500 μL de milieu de poisson pour éliminer l’excès de colorant.
- Granulez à nouveau les cellules par centrifugation, retirez le liquide et remettez les cellules en suspension dans 500 μL de 0,9x PBS+ 1% FBS et passez à travers un capuchon filtrant de 35 μm dans un tube FACS. Restez sur la glace.
- Préparation de la plaque donneuse
REMARQUE : La plaque donneuse est une plaque à fond en V de 96 puits dans laquelle les cellules T-ALL colorées CT-FR de poisson-zèbre transplanté seront triées. Chaque puits de la plaque contient 5 000 cellules WT CG1 non colorées dans 50 μL de milieu de poisson pour servir de support pour aider à granuler les cellules triées dans les étapes suivantes.- Comptez 500 000 cellules CG1 WT et transférez-les dans un tube conique de 15 ml.
- Diluez ce stock cellulaire à 100 cellules/μL en ajoutant 5 mL de milieu de poisson.
- Versez 50 μL de suspension cellulaire dans chaque puits de la plaque à 96 puits. Mélangez le tube de suspension cellulaire après chaque rangée pour maintenir l’homogénéité de la suspension cellulaire. Maintenir l’assiette à 4 °C jusqu’au tri.
- Préparation des échantillons de poissons transplantés
- Sélectionnez des poissons transplantés à 30-40 % de charge T-LAL. Collectez des poissons transplantés avec les cellules colorées CT-FR et les cellules tumorales GFP non colorées.
- Récoltez les cellules comme décrit à la section 2.
REMARQUE : Étant donné que l’on s’attend à ce que les cellules retenant le colorant soient présentes en faible abondance, préparez-vous à trier un grand nombre de cellules de l’échantillon CT-FR GFP. - Diluer à la concentration appropriée pour le tri ~3-5 × 106 cellules/mL dans 0,9x PBS + 1% FBS et passer à travers un capuchon filtrant de 35 μm dans un tube FACS. Ajouter du DAPI (1 mg/mL) à une dilution de 1:1 000 comme colorant vital pour exclure les cellules mortes. Restez sur la glace.
- Pour les cellules tumorales GFP non colorées, transférez 250 000 cellules dans un tube de microcentrifugation et granulez à 2 500 × g pendant 5 min à température ambiante. Retirez le média pour poissons, ajoutez 500 μL de PBS et passez-le à travers un capuchon filtrant de 35 μm dans un tube FACS. Restez sur la glace.
6. Tri
REMARQUE : Gardez tous les tubes et la plaque donneuse sur de la glace tout le temps, sauf lorsqu’ils sont utilisés pour le tri.
- Pour trier les paramètres et les portes, commencez par appliquer le contrôle de diffusion avant et latéral pour identifier les cellules d’intérêt en fonction de leur taille et éliminer les débris cellulaires. Ensuite, utilisez la hauteur de diffusion vers l’avant par rapport à la zone de diffusion vers l’avant pour exclure les cellules en double et ne conserver que les singlets. Enfin, utilisez les commandes sans couleur et unicolore pour dessiner les quadrants afin d’identifier les populations cellulaires positives à la GFP et à la CT-FR.
REMARQUE : On s’attend à ce que le témoin CT-FR soit beaucoup plus brillant que les échantillons pour le tri, car la coloration du témoin a été effectuée à l’état frais, sans divisions cellulaires ; les cellules des échantillons CT-FR GFP devraient apparaître à l’extrémité inférieure de la porte positive CT-FR. - Triez le nombre de cellules à transplanter dans chaque poisson dans un puits de la plaque donneuse.
REMARQUE : Pour cette expérience, nous avons utilisé un test de dilution limite pour estimer la fréquence des cellules souches de leucémie dans CT-FR High et CT-FR Low. Ce test a utilisé une dose de 25 cellules, transplantées chez 3 animaux, et une dose de 10 cellules, transplantées chez 10 animaux. Un test de dilution limite typique utilisant la T-ALL dérivée du poisson-zèbre rag2 :Myc impliquerait un groupe avec un nombre plus élevé de cellules (500 ou 1 000) puisque la fréquence des LSC dans ce modèle est estimée à 1 LSC dans des centaines de cellules. Cependant, dans cet essai, nous utilisons l’un des clones T-ALL précédemment décrits par Blackburn et al.15. La fréquence LSC dans l’échantillon utilisé ici est de ~1 LSC dans des dizaines de cellules ; Nous prévoyons que la leucémie sera détectée chez les animaux transplantés dans le groupe de 25 cellules. Lors de l’optimisation de ce test pour votre modèle de maladie, des précautions doivent être prises lors de la sélection du groupe de nombre de cellules le plus élevé pour assurer la greffe de la tumeur. - Après le tri, gardez la plaque sur de la glace jusqu’à ce qu’elle soit prête pour la transplantation.
REMARQUE : Les procédures de tri peuvent être stressantes et affecter la viabilité cellulaire. Avant d’exécuter l’expérience de dilution limite, une expérience pilote de tri a été réalisée. Les cellules triées ont été transplantées et le développement de la leucémie a été surveillé pour déterminer si le tri nuisait à la viabilité cellulaire. Alternativement, la viabilité cellulaire peut être évaluée en colorant les cellules triées avec du bleu de trypan et en les examinant au microscope avant la transplantation.
7. Transplantation de cellules triées à la limitation de la dilution et suivi de l’apparition de la tumeur
- Faites tourner la plaque à l’aide d’une grande centrifugeuse de paillasse à 2 500 × g à 4 °C.
- Retirez soigneusement 45 μL de surnageant de chaque puits, en laissant 5 μL de liquide derrière vous et en vous assurant d’éviter de toucher la pastille de cellule au fond du puits.
- Remettez les cellules en suspension à l’aide d’une pipette de 20 μL. Remettez en suspension 2-3 puits avec la pipette, puis à l’aide de la seringue d’un microlitre Hamilton, injectez les suspensions cellulaires de chaque puits dans le nombre souhaité de poissons-zèbres CG1 via une greffe IP. Ensuite, remettez les cellules en suspension dans le groupe de puits suivant pour éviter que les cellules ne se déstabilisent.
- Examinez le poisson-zèbre par microscopie à fluorescence sous sédation une fois par semaine pour le développement de la LAL-T pendant au moins 6 semaines.
8. Détermination de la fréquence des cellules souches de la leucémie
- Enregistrer le nombre d’animaux positifs par dose cellulaire et par groupe de traitement 6 semaines après la greffe, en considérant que les animaux présentant n’importe quelle quantité de signal GFP sont positifs. Considérez que les animaux ont une charge leucémique importante lorsque le signal GFP occupe >50 % du corps.
- Utilisez le logiciel statistique ELDA (Extreme Limiting Dilution Analysis) (http://bioinf.wehi.edu.au/software/elda/)19 pour calculer la fréquence des CSL au sein de chaque population cellulaire.
REMARQUE : Une illustration schématique du flux de travail est fournie à la figure 1.
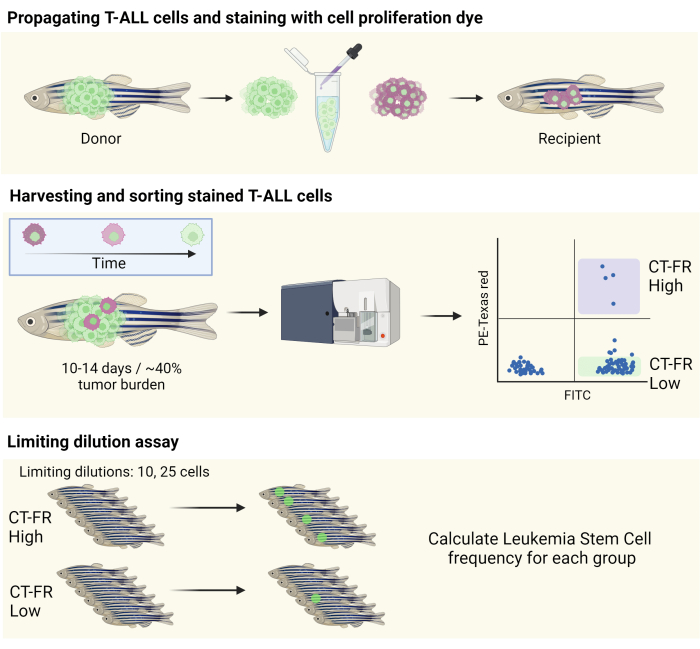
Figure 1 : Flux de travail pour l’utilisation de la coloration de suivi cellulaire pour isoler les cellules quiescentes dans le modèle T-ALL du poisson-zèbre. Illustration schématique de la coloration des cellules T-ALL du poisson-zèbre avec la coloration de prolifération cellulaire, de la propagation dans un poisson-zèbre CG1 (panneau du haut) et du tri des cellules en fonction de la rétention de la coloration de prolifération cellulaire (panneau du milieu). Les cellules collectées ont été utilisées pour un test de dilution limite afin de déterminer la fréquence des LSC (panneau inférieur). Abréviations : LAL-T = leucémie lymphoblastique aiguë à cellules T ; LSC = cellules souches leucémiques ; CT-FR = CellTrace Far Red ; FITC = isothiocyanate de fluorescéine ; PE = phycoérythrine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Nous avons suivi le protocole décrit ci-dessus pour trier les cellules qui ont conservé le colorant de prolifération cellulaire, CT-FR, et nous les avons utilisées pour un test de dilution limite (LDA) afin d’estimer la fréquence des LSC dans les populations CT-FR High et CT-FR Low. Pour définir le point de déclenchement de l’expérience de cytométrie en flux, nous avons utilisé un contrôle sans fluorophore (sans couleur) en plus des contrôles unicolores (
Discussion
Les CSL sont connues pour être résistantes aux traitements de chimiothérapie anti-prolifératifs conventionnels, et la découverte de thérapies ciblées contre ces cellules est très prometteuse pour réduire l’occurrence des rechutes et améliorer le pronostic des patients20. Des recherches antérieures ont décrit l’utilisation de colorants fluorescents de prolifération cellulaire pour identifier une petite population de cellules quiescentes associées ...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Le financement de cette recherche a été fourni par le National Cancer Institute (R37CA227656 à JSB). Cette recherche a également été financée par les ressources partagées en cytométrie en flux et en surveillance immunitaire du Markey Cancer Center (P30CA177558) de l’Université du Kentucky.
matériels
| Name | Company | Catalog Number | Comments |
| 26 G/2” micro-syringe | Hamilton | 87930 | NA |
| 35 µm filter cap FACS tubes | Falcon | 352235 | NA |
| 40 µm cell strainer | CELLTREAT | 229482 | NA |
| 96-well skirted PCR plate | Thermo Fisher Scientific | AB0800 | NA |
| Cell sorter | Sony Biotechnology | SY3200 | NA |
| CellTrace Far Red | Thermo Fisher Scientific | C34564 | NA |
| Conical tubes | VWR | 10026-078 | NA |
| DAPI | Thermo Fisher Scientific | 62248 | NA |
| DMSO | Sigma-Aldrich | D4818 | NA |
| Dulbecco'sPhosphate-buffered saline (PBS) | Caisson Labs | 22110001 | NA |
| Epifluorescence stereo microscope | Nikon | SMZ25 | NA |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | 12306C | NA |
| Fish system water | N/A | N/A | 0.03-0.05% salinity, pH 6.5-8, buffered with sodium bicarbonate |
| Microcentrifuge tubes | Thermo Fisher Scientific | C2171 | NA |
| MS-222 | Pentaire | TRS-1 | tricaine mesylate, an anesthetic |
| Petri dishes | Corning | 07-202-011 | NA |
| Razor blades | American Line | 66-0089 | NA |
| Trypan Blue | Thermo Fisher Scientific | T10282 | NA |
Références
- Reya, T., Morrison, S. J., Clarke, M. F., Weissman, I. L. Stem cells, cancer, and cancer stem cells. Nature. 414 (6859), 105-111 (2001).
- Arai, F., et al. Tie2/angiopoietin-1 signaling regulates hematopoietic stem cell quiescence in the bone marrow niche. Cell. 118 (2), 149-161 (2004).
- Li, L., Bhatia, R. Stem cell quiescence. Clin. Cancer Res. 17 (15), 4936-4941 (2011).
- Kreso, A., Dick, J. E. Evolution of the cancer stem cell model. Cell Stem Cell. 14 (3), 275-291 (2014).
- Chen, W., Dong, J., Haiech, J., Kilhoffer, M. -. C., Zeniou, M. Cancer stem cell quiescence and plasticity as major challenges in cancer therapy. Stem Cells Int. 2016, 1740936 (2016).
- Kleffel, S., Schatton, T. Tumor dormancy and cancer stem cells: Two sides of the same coin. Adv Exp Med Biol. 734, 145-179 (2013).
- Tuy, K., Rickenbacker, L., Hjelmeland, A. B. Reactive oxygen species produced by altered tumor metabolism impacts cancer stem cell maintenance. Redox Biol. 44, 101953 (2021).
- Zhou, B. -. B. S., et al. Tumour-initiating cells: Challenges and opportunities for anticancer drug discovery. Nat Rev Drug Discov. 8 (10), 806-823 (2009).
- Lyons, A. B., Blake, S. J., Doherty, K. V. Flow cytometric analysis of cell division by dilution of cfse and related dyes. Curr Protoc Cytom. 64 (1), 11-12 (2013).
- Lyons, A. B. Analysing cell division in vivo and in vitro using flow cytometric measurement of cfse dye dilution. J Immunol Methods. 243 (1-2), 147-154 (2000).
- Azari, H., Deleyrolle, L. P., Reynolds, B. A. Using carboxy fluorescein succinimidyl ester (cfse) to identify quiescent glioblastoma stem-like cells. Methods Mol Biol. 1686, 59-67 (2018).
- Deleyrolle, L. P., Rohaus, M. R., Fortin, J. M., Reynolds, B. A., Azari, H. Identification and isolation of slow-dividing cells in human glioblastoma using carboxy fluorescein succinimidyl ester (cfse). J Vis Exp. (62), e3918 (2012).
- Ebinger, S., et al. Characterization of rare, dormant, and therapy-resistant cells in acute lymphoblastic leukemia. Cancer Cell. 30 (6), 849-862 (2016).
- Langenau, D. M., et al. Myc-induced t cell leukemia in transgenic zebrafish. Science. 299 (5608), 887-890 (2003).
- Blackburn, J. S., et al. Clonal evolution enhances leukemia-propagating cell frequency in t cell acute lymphoblastic leukemia through akt/mtorc1 pathway activation. Cancer Cell. 25 (3), 366-378 (2014).
- Al-Hamaly, M. A., Turner, L. T., Rivera-Martinez, A., Rodriguez, A., Blackburn, J. S. Zebrafish cancer avatars: A translational platform for analyzing tumor heterogeneity and predicting patient outcomes. Int J Mol Sci. 24 (3), 2288 (2023).
- Blackburn, J. S., Liu, S., Langenau, D. M. Quantifying the frequency of tumor-propagating cells using limiting dilution cell transplantation in syngeneic zebrafish. J Vis Exp. (53), e2790 (2011).
- Borga, C., et al. Simultaneous b and t cell acute lymphoblastic leukemias in zebrafish driven by transgenic myc: Implications for oncogenesis and lymphopoiesis. Leukemia. 33 (2), 333-347 (2019).
- Hu, Y., Smyth, G. K. Elda: Extreme limiting dilution analysis for comparing depleted and enriched populations in stem cell and other assays. J Immunol Methods. 347 (1-2), 70-78 (2009).
- Bhojwani, D., Pui, C. -. H. Relapsed childhood acute lymphoblastic leukaemia. Lancet Oncol. 14 (6), e205-e217 (2013).
- Zon, L. I., Peterson, R. T. In vivo drug discovery in the zebrafish. Nat Rev Drug Discov. 4 (1), 35-44 (2005).
- Al-Hamaly, M. A., et al. Zebrafish drug screening identifies erlotinib as an inhibitor of wnt/β-catenin signaling and self-renewal in t-cell acute lymphoblastic leukemia. Biomed Pharmacother. 170, 116013 (2024).
- Yan, C., et al. Visualizing engrafted human cancer and therapy responses in immunodeficient zebrafish. Cell. 177 (7), 1903-1914 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon