A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הגברה וכימות HIV-1 RNA אצל אנשים שנדבקו ב-HIV עם המון ויראלי מתחת לגבול של זיהוי על ידי מבחני קלינית רגילה
In This Article
Summary
לכמת רמות של HIV-1 RNA בפלסמה רצף יחיד HIV-1 הגנום של אנשים עם המון ויראלי מתחת לגבול הגילוי (50-75 עותקים / מ"ל) קשה. כאן אנו מתארים כיצד לחלץ ולכמת רנ"א נגיפי בפלסמה בזמן אמיתי באמצעות PCR assay כי מהימן אמצעים HIV-1 RNA עד 0.3 עותקים / מ"ל וכיצד להגביר את הגנום הנגיפי על ידי רצף הגנום יחיד, ממדגמים עם המון ויראלי נמוך מאוד.
Abstract
הגברה גנים נגיפיים לכימות HIV-1-RNA של HIV-1 אנשים נגועים עם המון ויראלי מתחת לגבול של זיהוי על ידי מבחני סטנדרטית (להלן 50-75 עותקים / מ"ל) יש צורך לקבל תובנות על הדינמיקה ויראלי מארח אינטראקציות וירוס בחולים באופן טבעי לשלוט זיהום מי על טיפול משולב תרופתי (CART).
כאן אנו מתארים כיצד להגביר את הגנום הנגיפי על ידי ריצוף הגנום יחיד (פרוטוקול SGS) 13, 19, וכיצד לכמת במדויק HIV-1-RNA בחולים עם המון ויראלי נמוך (עותק יחיד (SCA) assay פרוטוקול) 12, 20.
Assay עותק יחיד הוא assay בזמן אמת PCR עם רגישות בהתאם לכמות של פלזמה להיות assayed. אם גנום נגיף יחיד מזוהה ב 7 מ"ל של פלסמה, ולאחר מכן במספר עותק RNA הוא דיווח להיות 0.3 עותקים / מ"ל. Assay יש בדיקות הבקרה הפנימית יעילות של מיצוי RNA, בקרות על הגברה אפשרי מ-DNA או זיהום. דגימות החולה נמדדים בשלושה עותקים.
יחיד ריצוף הגנום assay (SGS), כעת בשימוש נרחב נחשב ללא עבודה אינטנסיבית 3, 7, 12, 14, 15, הוא assay דילול מגביל, שבו הקצה בדילול מוצר cDNA משתרע על פני 96-טוב צלחת. על פי התפלגות פואסון, כאשר פחות מ 1 / 3 של בארות לתת מוצר, קיים סיכוי של 80% כתוצאה המוצר PCR להיות הגברה של cDNA של מולקולה בודדת. SGS יש יתרון על שיבוט של לא להיות נתון resampling ולא להיות מוטים על ידי רקומבינציה-PCR הציג 19. עם זאת, ההצלחה הגברה של SCA ו SGS תלוי בעיצוב פריימר. מבחני שניהם פותחו ב-HIV-1 תת, אבל יכול להיות מותאמים עבור תת אחרים באזורים אחרים של הגנום על ידי primers, בדיקות משתנה, סטנדרטים.
Protocol
1. הוצאה של RNA מתוך כמויות גדולות של פלסמה
סקירה של assay עותק יחיד (SCA) ו הגנום רצף אחד (SGS) פרוטוקול ניתנים איורים 1 ו -2.
- כדי להשיג 7 מ"ל של פלסמה, ספין כ 14 מ"ל של דם נאספו 15 מ"ל EDTA (לא הפרין) שפופרות לאיסוף XG 2600 במשך 15 דקות ללא בלימה. פיפטה פלזמה (לוודא כדי למנוע את שכבת מעיל באפי) לתוך צינורות 15 מ"ל.
- אם RNA הוא חילוץ עבור assay עותק יחיד (SCA), ספייק פלזמה עם 30,000 עותקים של שליטה Rous וירוס סרקומה virion פנימית (RCAS). הווירוס RCAS מוכן לפני ביצוע SCA כפי שתואר לעיל (20). בקצרה, נגיף RCAS מ supernatant תרבית תאים היא לכמת ידי פעילות RT, מדולל לריכוז סופי של 30,000 virions לכל ul 200, בדילול מלא בתרבות התקשורת RPMI רקמה עם FBS 5%, ומאוחסנים ב -80 מעלות צלזיוס aliquots להשתמש יחיד. הווירוס RCAS לא צריך להיות קפוא / מופשר לפני השימוש ב assay. RCAS הוא זינק לתוך הפלזמה המטופל לשלוט ליעילות של מיצוי את הרנ"א לדיוק של כימות PCR. מידע נוסף על RCAS ניתן למצוא בקישור הבא: http://home.ncifcrf.gov/hivdrp/RCAS/ .
- "טרום ספין" פלזמה, ב 15 מ"ל צינורות צנטריפוגה חרוטי, XG ב 2600 במשך 15 דקות להפריד את כל שומנים ופסולת הסלולר, אשר יכול להפריע וכימות רנ"א מדויק.
- לייבל סיטון Easy-Seal צנטריפוגות מבחנות עם סמן קבע לזהות מספר מדגם ואת המיקום שבו גלולה יהווה בצינור. אחרי ספין מראש, העברת פלזמה סיטון Easy-Seal צינורות צנטריפוגה ידי pipetting פלזמה ו - הימנעות מכל פסולת הסלולר ו / או שומנים על פני השטח. הקלט את נפח קלט של פלזמה.
- הוסף טריס שנאגרו מלוחים (TBS) כדי למלא נפח הנותרים של צינור סיטון-Easy החותם אל החלק התחתון של הצוואר מושחל. הקפד כי בועות נמצאים בצינור או שפופרת תקרוס ב ultracentrifuge. ודא לסמן על הצינורות פונה כלפי חוץ, כאשר הנחת צינורות לתוך הרוטור.
- חותם עם מחדש שמיש כמוסות דגימות מקום רוטור מראש מקורר T1270 צנטריפוגות Sorval וב ultracentrifuge 90SE Sorval ב XG 170,000 (50,000 סל"ד) ב 4 מעלות צלזיוס למשך 30 דקות.
- לאחר צנטריפוגה supernatant להסיר ולהוסיף 90 כיתה ul מים מולקולרית 10 ul proteinase-K (20 מ"ג / מ"ל) של גלולה ויראלי (זה לא יהיה גלוי).
- המקום צינורות אמבטיה 55 מעלות צלזיוס המים דגירה במשך 30 דקות. ודא צינורות נקבעים בזווית כך שהצד עם גלולה הוא שקוע בתוך תערובת proteinase-K והמים.
- לאחר הדגירה, זמן קצר ספין הצינור בצנטריפוגה העליון בטבלה לאסוף את כל נוזל בתחתית הצינור (כ 5 שניות) ולהוסיף 315 ul של פתרון guanidinium thiocyanate 6M ו -10 ul של גליקוגן מ"ג / מ"ל 20 (הערה: בצע המתאים הנחיות סילוק חומר מסוכן זה, לא מתערבבים עם אקונומיקה או חומצות להיפטר במיכל נפרד).
- וורטקס קלות ספין זמן קצר (כ -5 שניות). דגירה במשך 10 דקות בטמפרטורת החדר ולאחר מכן להעביר תוכן של הצינור לכל שכותרתה החדש 1.5 צינורות מ"ל RNAse צנטריפוגות חינם.
- הוספת 495 ul המולקולרי של איזופרופיל אלכוהול כיתה מערבולת כל הצינור, למשך 10 שניות ו צנטריפוגות ב XG 21,000 במשך 30 דקות בטמפרטורת החדר.
- בטל supernatant ולהוסיף 500 ul של אתנול 70%.
- וורטקס למשך 10 שניות ו צנטריפוגות ב XG 21,000 במשך 15 דקות בטמפרטורת החדר. הסר אתנול עם פיפטה העברה. ספין שוב להסיר כל אתנול שיורית עם טפטפת. האוויר יבש במשך 10 דקות. (בשביל הפרוטוקול SGS לפזר רנ"א ul 40 של 5 מ"מ טריס-HCl pH 8.0, ולהמשיך עם הפרוטוקול SGS).
- להמיס גלולה ב ul 55 של RNA-חיץ. RNA-חיץ מתקבל על ידי הוספת 10 ul DTT של 100 מ"מ ו 25 ul של Rnasin U / ul 40-965-ul טריס HCl (pH 8.0, 5 מ"מ). מניחים על קרח.
2. Assay עותק יחיד
צלחת 96-היטב מחזיקה 8 דגימות החולה כולל שני HIV סטנדרטים RCAS שולטת. פרוטוקול זה יתאר את הגדרת צלחת אחת.
- התחל על ידי הכנת 2 aliquots של 1 חיץ-RNA מ"ל שבו HIV-1 ו-RNA RCAS תעתיקי רנ"א יהיה מדולל על עקומות סטנדרטיות RNA. RNA-חיץ מתואר תחת 1.13.
- הכן דילולים על הקרח 2 צינורות צנטריפוגה מ"ל. הפוך את חצי יומן דילולים של HIV-1 תעתיקי רנ"א חיץ-RNA ע"י הוספת 25 ul של HIV-1 RNA מניות (1x10 6 עותקים / ul) עד 54 ul-RNA חיץ מתן 3x10 5 עותקים / ul.
- המשך בכך ברציפות חצי יומן דילולים (25 + 54 ul ul) עד 0.3 עותקים ul 10 (הערה: 0.3, 1, ו -3 דילולים לא יהיה חלק של עקומת סטנדרט במהלך ניתוח עבור אלה משמשים ערכים ניפוח וגם צריך להיות מוגדר dur נעלמיםניתוח ing). תמלילי RCAS הם מצויד ב 1x10 6 עותקים / ul. כמו כן, להכין חצי יומן דילולים של תעתיקי רנ"א RCAS ב-חיץ אבל עד 100 עותקים ul 10.
- הכינו את הקוקטייל RT לסינתזה cDNA כמפורט בטבלה 1. הכן RT ותגובות NRT כמפורט בטבלה 1 תוך התחשבות לפצות קצת תוספת לחשבון לכל אובדן שעלולים להתרחש במהלך ההעברה. הערה: לקוקטייל NRT, האנזים RT הוא הוחלף על ידי מים כדי לשלוט על הגברה של DNA.
הערה: זה צעד באחרונה אוטומטיות בנוסף הצלחת להקים על רובוט Qiagen (במקור קורבט). - הגדר את צלחת אופטית 96-היטב (כפי שמוצג באיור 3), על ידי הוספת 20 ul מתערובת התגובה RT היטב כל אחד. ב 8 בארות שכותרתו "NRT", להוסיף את קוקטייל ללא transcriptase הפוכה (NRT תערובת התגובה). לאחר הוספת קוקטיילים לצלחת, להוסיף 10 ul מים לבארות שכותרתו "לא שולט התבנית (NTC)". הוסף 10 ul של תמלילי HIV לבארות שכותרתו "איידס" בריכוזים שמוצג באיור 3. הוסף 10 ul של תמלילי RCAS לבארות שכותרתו "RCAS" בריכוזים פי. הוסף 10 ul המדגם החולה 3 בארות הסמוך שכותרתו "מדגם" בתרשים צלחת. הוסף 10 ul המדגם eluted את בארות שכותרתו "NRT." הוסף 5 ul המדגם באותה 5 ul מים בארות שכותרתו "בקרה פנימית".
- חותם את הצלחת לרוץ על thermocycler ב: 25 ° C למשך 15 דקות, 42 ° C במשך 40 דקות, 85 ° C למשך 10 דקות, 25 ° C במשך 30 דקות, ו - 5 ° C להחזיק.
- אמן הכן PCR מתערבב לפי טבלה 2.
- כאשר סינתזה cDNA הושלמה, לעבור לשטח המיועד באמצעות cDNA. באזור זה המיועד, להוסיף 20 ul תמהיל האב PCR היטב כל הצלחת cDNA הן איידס RCAS. חשוב לבצע שלב זה באזור המיועד, כדי למנוע זיהום אפשרי.
- צלחת ספין, חותם עם כיסוי ABI או רוש לכסות בשמיכה אופטי אופטי (שמיכה נחוץ רק עבור ABI 7700). הפעלה על רוש 480 או ABI 7700. PCR תנאים: 95 ° C למשך 10 דקות, ואז 45 מחזורים של 95 מעלות צלזיוס למשך 15 שניות, 60 ° C עבור 1 דקה. ניתוח נפרד נדרש RCAS ו-HIV. דוגמאות של התוצאות מוצגות באיור 4. השיפוע של עקומת סטנדרט תושפע התפלגות פואסון הרגיל של תעתיקי מספר נמוך להעתיק. על מנת להבטיח את השיפוע המדויק ביותר עבור עקומת סטנדרטי, מספר סטנדרטים אלה נמוך עותק (0.3, 1, ו - 3 עותקים) מוגדרים בתור "נעלמים". 17
3. יחיד לקביעת גנום
- cDNA סינתזה. הוסף 5 ul של 10 dNTPs מ"מ 5 ul של 2 מ"מ את הגן הספציפי פריימר (pol או env) אל באר צלחת 96 PCR היטב (Biorad). הוסף 40 ul של RNA חילוץ. חותם צלחת.
- לפגל RNA-התערובת thermocycler על 65 מעלות צלזיוס למשך 10 דקות.
- מערבבים את ריאגנטים לסינתזה cDNA, המפורטים בטבלה 3. לאחר השלב denaturing, מקום צלחת PCR על הקרח, מוסיפים את ul 50 של התערובת cDNA לטעום כל אחד.
- הפעל צלחת PCR על thermocycler: 45 מעלות צלזיוס במשך 50 דקות, 85 ° C במשך 10 דקות ו - 4 ° C להחזיק.
- ראשית PCR, 10 תגובות ul. מכינים את תערובת התגובה PCR במגש מגיב שימוש או primers עבור הגברה הסיבוב הראשון של P6-rt או env (ריאגנטים המפורטים בטבלה 3, primers המפורטים לוח 5).
- שימוש רב ערוצי פיפטה לוותר ul 8.0 תערובת PCR על 73 בארות על 96 גם צלחת PCR (70 דגימות 3 שולטת שלילי).
- לאחר סינתזה cDNA השלמת המהלך צלחת cDNA ו PCR המכיל תערובת PCR התגובה לאזור המיועד באמצעות cDNA. באזור המיועד, להוסיף 40 ul של טריס-HCl pH 8.0 לטעום כל cDNA להביא את נפח הסופית ul 140. הוסף ul 2.0 של cDNA לכל אחד 70 בארות 2.0 ul מים לכל אחד שולט שלילית. חותם צלחת PCR. הפעלה על thermocycler, התוכנית בלוח 4.
- Nested PCR - תגובות 20ul. מערבבים את ריאגנטים עבור PCR מקוננות במגש מגיב (ריאגנטים המפורטים בטבלה 3, primers המפורטים לוח 5).
- הוסף 18 ul תערובת PCR מקוננים על 73 בארות על צלחת 96 PCR היטב, על ידי פיפטה רב
- מדולל first PCR 01:05 ולהוסיף 2 ul בסיבוב הראשון של ה-PCR ל-PCR מקוננות. הפעל תגובת ה-PCR על thermocycler, בתנאים המפורטים בלוח 4.
- הפעל דוגמאות על ג'ל agarose 1%. על מנת להבטיח כי הרוב המכריע של המוצרים PCR הם תוצאה של מולקולה בודדת של cDNA, לא יותר מ 30% מבארות צריך להיות חיובי. התהליך כבר אוטומטית באמצעות Beckman Coulter Biomek FM רובוט כדי לטעון את תוכן הצלחות PCR ל% 1, 96 בארות E-ג'ל (Invitrogen). E-ג'לים מתנהלים במשך 7 דקות על בסיס-E. המוצרים רצף באמצעות primers המפורטים בלוח 5.
4. נציג התוצאות:
SCA:
כל שולט צריך לעבור על מנת לשקול את המדידה HIV-1 RNA הנכון. אם שליטה שלילי positiיש, יש להפעיל את צריך להתעלם בגלל זיהום אפשרי. כדי לשלוט על הפסדים של רנ"א במהלך שלב החילוץ, לפחות 50% RCAS ממוסמר בפלסמה צריך להיות התאושש. אם RCAS הממוצע <15,000 עותקים, המדגם נכשל צריך להתעלם כי כמות משמעותית של רנ"א יכול אבדו במהלך השאיבה או צעדים אחרים של הפרוטוקול. זה מתרחש כ -10% מכלל הדגימות שנבדקו החולה בשל סביר שומנים גבוהה בדם 6. ההתאוששות של הנגיף RCAS נועד כדי לשלוט על איכות החילוץ ואת היעילות של סינתזה cDNA הגברה ו-PCR. זה לא משמש הנכון HIV התאוששות מאז איידס מחויב סביר נוגדנים וחלבונים אדם אחרים מה שהופך אותו שונה במקצת מן הנגיף שבודד בתרבית רקמה. ההתאוששות של RCAS במעבדה שלנו היה בממוצע 25,716 + / - 4,057 עותקים / תערובת תגובה, או בערך 95% + / -15% של ה-RNA RCAS הוסיף 17. הבקרה (לא transcriptase הפוכה) NRT מנוהל במקביל על מנת לבדוק את ההגברה של ה-DNA. הערך NRT מופחת בכל אחד משלושת HIV-1 ערכים RNA, אז הממוצע מחושב אם מספר זה הוא פחות מאפס (הגברה של DNA עולה על הגברה של רנ"א), התוצאה היתה להיכשל צריך להתעלם כי הסיכון של הגברה מ-DNA במקום RNA. אם HIV-1 RNA מוגבר רק 1 / 3 בארות, מחדש בדיקת המדגם מומלץ לוודא הגברה נכון גם לגבי המדגם בפועל להיות assayed ולא בשל זיהום אפשרי.
אם כל שולט לעבור, הממוצע HIV-1 מספר RNA להעתיק בפלסמה יכול להיות מחושב.
דוגמה 1: אם הממוצע HIV-1 מספר עותק הוא אפס, להגביל את הגילוי מחושב על בסיס נפח של פלזמה assayed. כדוגמה, נניח 7 מ"ל של פלסמה היה assayed. הכמות הקטנה ביותר של רנ"א יכולה להיות מזוהה עם assay זה היה 1 עותק אחד בארות 0 עותקים שתי בארות אחרות נותן ממוצע של 0.33 עותקים היטב. מספר עותק הממוצע אז צריך להיות מוכפל בפקטור של 5.5 להגיע למספר להעתיק סך elution RNA (יש 10 ul היטב כל אחד, אבל נפח elution הכולל היה 55 אול). הגבול התחתון של הזיהוי ואז: 5.5 x 0.33 עותקים מחולק 7ml = 1.83 / 7 = 0.3 עותקים / מ"ל. הריכוז של HIV-1 RNA בפלזמה בדוגמה זו היתה <0.3 עותקים / מ"ל.
דוגמא 2: HIV-1 RNA הוא לזיהוי. RNA הופק מ - 7 מ"ל של פלסמה. מספר עותק הממוצע של HIV-1 RNA לכל מחושב היטב להיות 2.0 עותקים היטב. ואז להעתיק את המספר הממוצע מ"ל של פלסמה הוא 5.5 x 2.0 עותקים / 7 מ"ל = 1.6 HIV-1 RNA / מ"ל של פלסמה.
גיליון תבנית Excel המשמש לחישוב מספרים עותק ניתן לטעון מן האתר.
SGS:
אם אחד מהפקדים שלילי חיובי בטווח צריך להתעלם עקב זיהום אפשרי. מספר המוצר לרוץ כל יהיה תלוי עומס נגיפי במדגם כל מצב של המדגם. מניסיוננו הרבה שומנים או תאים פלזמה יקטין את הסיכוי של קבלת המוצר. תנאי אחסון והקפאה הקודם הפשרה של המדגם יהיה גם להשפיע על התאוששות של רנ"א מאוד. באופן כללי, כאשר עובדים עם דגימות עם המון ויראלי להלן 50 עותקים / מ"ל, המוצר (להקות על ג'ל) צפוי ב כ 1 / 3 -1 / 2 של דגימות מעובד. בהתאם הגורמים שהוזכרו לעיל, 1-5 להקות לכל צלחת יש לשקול תוצאה טובה בשל עומסים נגיפי נמוך בחולים אלו.
| סינתזה cDNA (קוקטייל RT / תגובה cDNA) | 1 תגובה | אין transcriptase הפוכה (NRT) קוקטייל / תגובה cDNA (1 תגובות) |
| מולקולרית כיתה H 2 O | 8.1 ul | 8.2 ul |
| 25 מ"מ MgCl 2 | 6 ul | 6 ul |
| 25 dNTPs mM | 0.6 ul | 0.6 ul |
| 100 מ"מ DTT | 0.2 ul | 0.2 ul |
| Hexamers אקראית (0.1 UG / ul) | 1.5 ul | 1.5 ul |
| מאגר 10X TaqMan | 3.0 ul | 3.0 ul |
| Rnasin (40 U / UL) | 0.5 ul | 0.5 ul |
| Strategene RT (200 U / ul) | 0.1 ul | אל תוסיף |
| סך הכל | 20 ul | 20 ul |
| לדוגמא RNA | 10 ul | 10 ul |
| סך נפח | 30 ul | 30 ul |
1. טבלת התגובה mixtures לסינתזה cDNA ב Assay עותק יחיד.
| PCR מאסטר מיקס | 1 תגובה | Primers |
| H 2 O | 15.1 ul | HIV קדימה פריימר 5'-CATGTTTTCAGCATTATCAGAAGGA-3 " |
| מאגר 10X PCR זהב | 2.0 ul | HIV פריימר הפוך 5'-TGCTTGATGTCCCCCCACT-3 " |
| 25 מ"מ MgCl 2 | 2.0 ul | HIV-Probe5'FAM CCACCCCACAAGATTTAAACACCATGCTAA-טמרה 3 " |
| * פריימר קדימה (100 אממ) | 0.3 ul | |
| * פריימר הפוך (100 אממ) | 0.3 ul | RCAS קדימה Primer5'-GTCAATAGAGAGAGGGATGGACAAA-3 " |
| * בדיקה (100 אממ) | 0.05 ul | RCAS הפוך Primer5'-TCCACAAGTGTAGCAGAGCCC-3 " |
| TaqGold (5 U / ul) | 0.25 ul | RCAS Probe 5'FAM-TGGGTCGGGTGGTCGTGCC טמרה-3 " |
| סך הכל | 20.0 ul |
2. טבלה PCR מאסטר לערבב primers עבור Assay עותק יחיד.
| cDNA קוקטייל / cDNA התגובות | 1 RNA מדגם | קוקטייל ראשון PCR | צלחת 1 (75 תגובות) | קוקטייל מקונן PCR | צלחת 1 (75 תגובות) |
| 10X RT חוצץ (Invitrogen) | 10 ul | 10X PCR חיץ (Invitrogen) | 75 ul | חיץ 10X-PCR | 150 ul |
| 25 מ"מ MgCl 2 | 20 ul | 50 מ"מ MgSO 4 | 30 ul | 50 מ"מ MgSO 4 | 60 ul |
| 0.1M DTT | 1 ul | 10 dNTPs mM | 15 ul | 10 dNTPs mM | 30 ul |
| RNase ללא מים | 17.5 ul | 50 primers UM (EA) | 3 אול | 50 primers UM (EA) | 6 ul |
| Rnase-Out (Invitrogen) | 1 ul | פלטינום תקי היי Fi אנזימים (Invitrogen) | 6 ul | פלטינום תקי היי Fi אנזימים (Invitrogen) | 12 ul |
| כתב עילי III (200 U / ul) (Invitrogen) | 0.5 ul | מולקולרית כיתה מים | 468 ul | מולקולרית כיתה מים | 1086 ul |
טבלה 1. CDNA ותערובות PCR עבור רצף הגנום יחיד.
| P6-RT-PCR תוכנית 1 | Env 1 PCR התוכנית |
| 1. 94 ° C למשך דקה 2 | 1. 94 ° C למשך דקה 2 |
| 2. 94 ° C למשך 30 שניות | 2 0.94 מעלות צלזיוס למשך 30 שניות |
| 2 0.94 מעלות צלזיוס למשך 30 שניות | 3. 52 ° C למשך 30 שניות |
| 4. 72 ° C עבור 1 דקה 30 שניות | 4. 72 ° C למשך דקה 1 |
| 5. עבור מס '2, 44 מחזורים | 5. עבור מס '2, 44 מחזורים |
| 6. 72 ° C במשך 3 דקות | 6. 72 ° C במשך 3 דקות |
| 7. 4 ° C להחזיק | 7. 4 ° C להחזיק |
| P6-RT-PCR 2 תוכנית | Env 2 PCR התוכנית |
| 1. 94 מעלות צלזיוס למשך 2 דקות | 1. 94 מעלות צלזיוס למשך 2 דקות |
| 2. 94 ° C למשך 30 שניות | 2. 94 ° C למשך 30 שניות |
| 3. 55 ° C למשך 30 שניות | 3. 56 ° C למשך 30 שניות |
| 4. 72 ° C למשך דקה 1 | 4. 72 ° C למשך 45 שניות |
| 5. עבור מס '1, 40 (41 מחזורים בסך הכל) | 5. עבור מס '1, 40 (41 מחזורים בסך הכל) |
| 6. 72 ° C במשך 3 דקות | 6. 72 ° C במשך 3 דקות |
| 7. 4 ° C להחזיק | 7. 4 ° C להחזיק |
טבלה 4. Thermocycler התנאים Assay יחיד רצף הגנום.
| תגובה | תחל | רצף |
| P6-RT cDNA | 3500 - | 5 "CTATTAAGTATTTTGATGGGTCATAA 3" |
| cDNA env | E115- | 5'AGAAAAATTCCCCTCCACAATTAA 3 " |
| P6-RT 1. PCR | 3500 - | 5 "CTATTAAGTATTTTGATGGGTCATAA 3" |
| P6-RT 1. PCR | 1849 + | 5 "GATGACAGCATGTCAGGGAG 3" |
| P6-RT2.PCR | 1870 + | 5 "GAGTTTTGGCTGAGGCAATGAG 3" |
| P6-RT 2.PCR | 3410 - | 5 "CAGTTAGTGGTATTACTTCTGTTAGTGCTT 3" |
| env 1. PCR | E115- | 5'AGAAAAATTCCCCTCCACAATTAA 3 " |
| env 1. PCR | E20 + | 5'GGGCCACACATGCCTGTGTACCCACAG 3 " |
| env 2. PCR | E30 + | 5'GTGTACCCACAGACCCCAGCCCACAAG3 " |
| env 2. PCR | E125- | 5'CAATTTCTGGGTCCCCTCCTGAGG 3 " |
| P6-RT רצף | 2030 + | 5'TGTTGGAAATGTGGAAAGGAAGGAC 3 " |
| P6-RT רצף | 2600 + | 5'ATGGCCCAAAAGTTAAACAATGGC3 " |
| P6-RT רצף | 2610 - | 5'TTCTTCTGTCAATGGCCATTGTTTAAC3 " |
| P6-RT רצף | 3330 - | 5'TTGCCCAATTCAATTTTCCCACTAA 3 " |
| env רצף | E30 + | 5'GTGTACCCACAGACCCCAGCCCACAAG3 " |
| env רצף | E125- | 5'CAATTTCTGGGTCCCCTCCTGAGG 3 " |
לוח 5. Primers עבור Assay יחיד רצף הגנום.
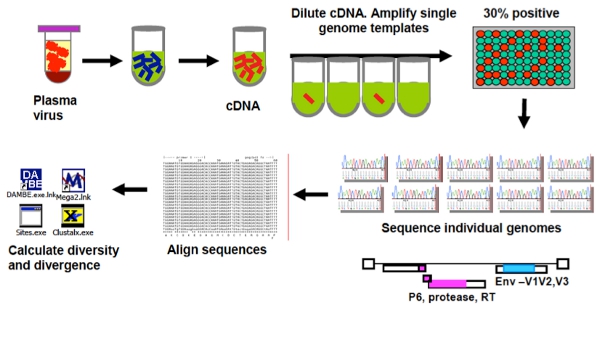
באיור 1. סקירה של Assay לקביעת גנום לחרוך (SGS).
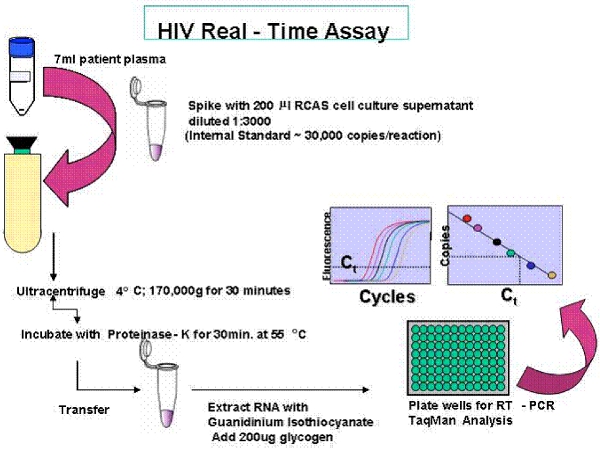
איור 2. סקירה של Assay העתק יחיד (SCA).
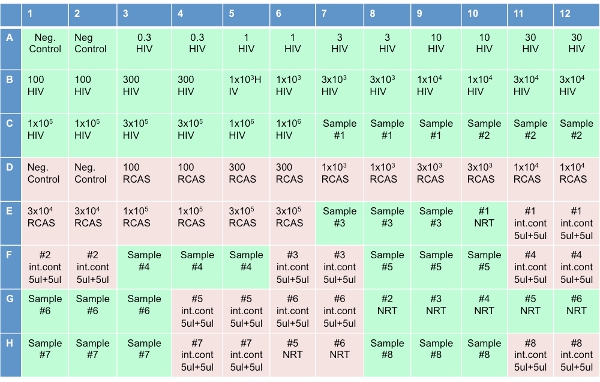
איור 3. פלייט הגדרת עבור Assay העתק יחיד.
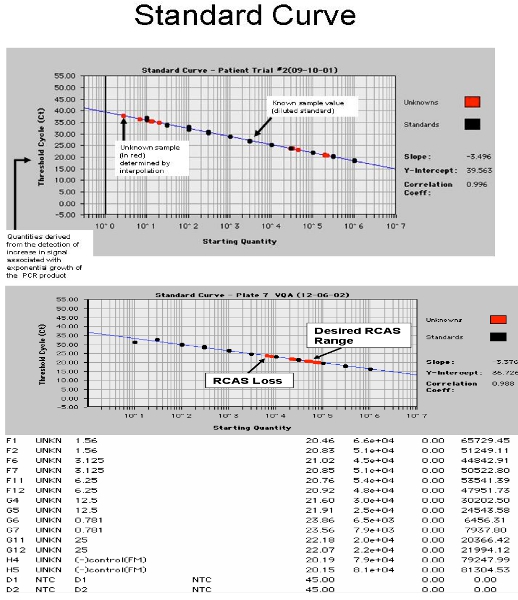
איור 4. צילום מסך של ABI 7700. א 'מראה את HIV-1 עקומת רנ"א רגיל עם דגימות החולה ב מראה עקומה RCAS רגיל עם דגימות החולה ממוסמר.
Discussion
HIV-1 אנשים נגועים על טיפול משולב תרופתי (CART) או שמטבע הדברים לשלוט יש המון זיהום נגיפי נמוך מאוד, בדרך כלל סביב 1 מ"ל לכל עותק של 4 פלזמה, 11, 12, 17, 18. המון נגיפי בחולים עם שליטה טבעית, לעתים קרובות תנודות סביב להגדיר אדם נקודה 1, 2, 17. מבחני הרגישות של המתוארים כאן...
Disclosures
אין ניגודי אינטרסים הכריז.
Acknowledgements
המחברים מבקשים להודות חולים שהשתתפו במחקרים רבים של HIV-1.
JMC היה פרופסור מחקר של האגודה האמריקנית לסרטן בתמיכת קרן קירבי FM.
Materials
| Name | Company | Catalog Number | Comments |
| שם מגיב | חברה | מספר קטלוגי | תגובות |
| Hexamers אקראיים (500 UG / ml) | Promega | C118A | |
| TaqMan חיץ | Applied Biosystems | 4304441 | |
| RNAsin (40 U / ul) | Promega | N211B | |
| האנזים RT (200 U / ul) | Strategene | 600107-51 | |
| Amplitaq זהב פולימראז ה-DNA | Applied Biosystems | 4311814 | 6-pack |
| Amplitaq 10XGold PCR חיץ II | Applied Biosystems | 4311814 | מגיע עם האנזים |
| Magnesiumcloride 25 מ"מ | Applied Biosystems | 4311814 | מגיע עם האנזים |
| טריס-1M HCl חיץ, pH 8.0, 5 מ"מ | Invitrogen | AM9855G | |
| כתב עילי III הפוך האנזים transcriptase, 200 U / ul | Invitrogen | 18080-044 | |
| כתב עילי III חיץ 10X | Invitrogen | מגיע עם האנזים | |
| DTT 100 מ"מ | Invitrogen | מגיע עם האנזים | |
| פלטינום תקי DNA פולימראז נאמנות גבוהה 5 U / ul | Invitrogen | 11304-102 | |
| 10X נאמנות גבוהה PCR הצפת | Invitrogen | 11304-102 | מגיע עם האנזים |
| Proteinase K-20 מ"ג / מ"ל | Ambion | 2546 | |
| Guanidinium Thiocyanate פתרון 6M | FlukaBiochemica | 50983 | |
| גליקוגן 20 מ"ג / מ"ל | רוש | 10901393001 | |
| Isopropanol | סיגמא אולדריץ | 19516 | |
| אתנול 70% | סיגמא אולדריץ | E702-3 | |
| מים כיתה מולקולרית | Gibco / Invitrogen | 10977-015 | |
| dNTPs (25 mmol כל אחת) | Bioline | BIO-39025 |
| שם Euipment | חברה | מספר קטלוגי | תגובות |
| Easy צנטריפוגה חותם צינורות (16x73mm) | סיטון מדעי | 6041 | |
| לבן Delrin כתרים | סיטון מדעי | 4020 | Caps עבור צינורות חותם קל |
| Tube Rack עבור Bell-Top Tubes Optiseal, 8.9 מ"ל | Beckman | 361642 | |
| הנהג Hex ½ פתיחת inc | סיטון מדעי | 4013 | |
| כובע tupe הסרת | סיטון מדעי | 4014 | |
| 5 pipettes סרולוגיות מ"ל | שחקן המשנה | 4051 | |
| Pipetboy | כל | ||
| 15 מ"ל pipettes העברה | ISC Bioexpress | P-5005-7 | |
| 5.8 מ"ל pipettes העברת Dispossable, טיפ מצוין | VWR | 14670-329 | |
| 15 צנטריפוגות צינורות מ"ל | בז | ||
| 1 מ"ל צינורות צנטריפוגה | כל | ||
| טריס חיץ טבליות מלוחים | סיגמא אולדריץ | T5030-100TAB | |
| Ultracentrifuge ואת הרוטור | Sorval | 90SE ו-T-1270 | |
| 96-PCR גם צלחות | Biorad | HSS-9601 | |
| 50 מ"ל מאגר מגיב | שחקן המשנה | 4870 | |
| Microamp אופטי 96-היטב צלחת תגובה | Applied Biosystems | N801-0560 | |
| צלחת אופטית מכסה | Applied Biosystems | 4333183 | |
| MicroAmp Pad דחיסה אופטי | Applied Biosystems | 4312639 | |
| Microsealסרט | Biorad | MSA-5001 | |
| 2 מ"ל צינורות צנטריפוגה | כל | ||
| 1% agarose ג'לים (E-ג'ל, Invitrogen) | Invitrogen | G-7008-01 | |
| E-הבסיס (Invitrogen) | Invitrogen | EB-M03 |
EDTA אוסף צינורות כל מותג
References
- Amara, R. R., Villinger, F., Altman, J. D., Lydy, S. L., O'Neil, S. P., Staprans, S. I., Montefiori, D. C., Xu, Y., Herndon, J. G., Wyatt, L. S. Control of a mucosal challenge and prevention of AIDS by a multiprotein DNA/MVA vaccine. Vaccine. 20, 1949-1955 (2002).
- Dinoso, J., Kim, S., Blankson, J., Siliciano, R. F. Viral Dynamics of Elite Suppressors in HIV-1 Infection. Conference on Retroviruses and Opportunistic Infections. , (2008).
- Dinoso, J. B., Kim, S. Y., Wiegand, A. M., Palmer, S. E., Gange, S. J., Cranmer, L., O'Shea, A., Callender, M., Spivak, A., Brennan, T., Kearney, . Treatment intensification does not reduce residual HIV-1 viremia in patients on highly active antiretroviral therapy. Proc. Natl. Acad. Sci. U.S.A. 106, 9403-9408 (2009).
- Doria-Rose, N. A., Klein, R. M., Manion, M. M., O'Dell, S., Phogat, A., Chakrabarti, B., Hallahan, C. W., Migueles, S. A., Wrammert, J., Ahmed, R., Nason, M., Wyatt, R. T., Mascola, J. R., Connors, M. Frequency and phenotype of human immunodeficiency virus envelope-specific B cells from patients with broadly cross-neutralizing antibodies. J. Virol. 83, 188-199 (2009).
- Dornadula, G., Zhang, H., Uitert, B. V. a. n., Stern, J., Livornese, L., Ingerman, M. J., Witek, J., Kedanis, R. J., Natkin, J., DeSimone, J., Pomerantz, R. J. Residual HIV-1 RNA in blood plasma of patients taking suppressive highly active antiretroviral therapy. JAMA. 282, 1627-1632 (1999).
- Gandhi, R. T., Zheng, L., Bosch, R. J., Chan, E. S., Margolis, D. M., Read, S., Kallungal, B., Palmer, S., Medvik, K., Lederman, M. M., Alatrakchi, N., Jacobson, J. M., Wiegand, A., Kearney, M., Coffin, J. M., Mellors, J. W., Eron, J. J. The effect of raltegravir intensification on low-level residual viremia in HIV-infected patients on antiretroviral therapy: a randomized controlled trial. PLoS medicine. 7, (2010).
- Gay, C., Dibben, O., Anderson, J. A., Stacey, A., Mayo, A. J., Norris, P. J., Kuruc, J. D., Salazar-Gonzalez, J. F., Li, H., Keele, B. F., Hicks, C., Margolis, D., Ferrari, G., Haynes, B., Swanstrom, R. Cross-sectional detection of acute HIV infection: timing of transmission, inflammation and antiretroviral therapy. PLoS One. 6, 19617-19617 (2011).
- Hatano, H., Delwart, E. L., Norris, P. J., Lee, T. H., Dunn-Williams, J., Hunt, P. W., Hoh, R., Stramer, S. L., Linnen, J. M., McCune, J. M., Martin, J. N., Busch, M. P., Deeks, S. G. Evidence for persistent low-level viremia in individuals who control human immunodeficiency virus in the absence of antiretroviral therapy. J. Virol. 83, 329-335 (2009).
- Havlir, D. V., Bassett, R., Levitan, D., Gilbert, P., Tebas, P., Collier, A. C., Hirsch, M. S., Ignacio, C., Condra, J., Gunthard, H. F., Richman, D. D., Wong, J. K. Prevalence and predictive value of intermittent viremia with combination hiv therapy. JAMA. 286, 171-179 (2001).
- Jordan, M. R., Kearney, M., Palmer, S., Shao, W., Maldarelli, F., Coakley, E. P., Chappey, C., Wanke, C., Coffin, J. M. Comparison of standard PCR/cloning to single genome sequencing for analysis of HIV-1 populations. J Virol Methods. 168, 114-120 (2010).
- Kaufmann, D. E., Kavanagh, D. G., Pereyra, F., Zaunders, J. J., Mackey, E. W., Miura, T., Palmer, S., Brockman, M., Rathod, A., Piechocka-Trocha, A., Baker, B., Zhu, B., Gall, S. L. e., Waring, M. T., Ahern, R., Moss, K., Kelleher, A. D. Upregulation of CTLA-4 by HIV-specific CD4+ T cells correlates with disease progression and defines a reversible immune dysfunction. Nat Immunol. 8, 1246-1254 (2007).
- Kearney, M., Maldarelli, F., Shao, W., Margolick, J. B., Daar, E. S., Mellors, J. W., Rao, V., Coffin, J. M., Palmer, S. HIV-1 Population Genetics and Adaptation in Newly Infected Individuals. J. Virol. 83, 2715-2727 (2008).
- Kearney, M., Palmer, S., Maldarelli, F., Shao, W., Polis, M. A., Mican, J., Rock-Kress, D., Margolick, J. B., Coffin, J. M., Mellors, J. W. Frequent polymorphism at drug resistance sites in HIV-1 protease and reverse transcriptase. AIDS. 22, 497-501 (2008).
- Kearney, M., Spindler, J., Shao, W., Maldarelli, F., Palmer, S., Hu, S. L., Lifson, J. D., Kewal Ramani, V. N., Mellors, J. W., Coffin, J. M., Ambrose, Z. Genetic diversity of simian immunodeficiency virus encoding HIV-1 reverse transcriptase persists in macaques despite antiretroviral therapy. Journal of Virology. 85, 1067-1076 (2011).
- Keele, B. F., Giorgi, E. E., Salazar-Gonzalez, J. F., Decker, J. M., Pham, K. T., Salazar, M. G., Sun, C., Grayson, T., Wang, S., Li, H., Wei, X., Jiang, C., Kirchherr, J. L., Gao, F., Anderson, J. A., Ping, L. H., Swanstrom, R. Identification and characterization of transmitted and early founder virus envelopes in primary HIV-1 infection. Proc. Natl. Acad. Sci. U.S.A. , 105-7552 (2008).
- Liu, S. L., Rodrigo, A. G., Shankarappa, R. G., Learn, H., Hsu, L., Davidov, O., Zhao, L. P., Mullins, J. I. HIV quasispecies and resampling. Science. 273, 415-416 (1996).
- Mahalanabis, M., Jayaraman, P., Miura, T., Pereyra, F., Chester, E. M., Richardson, B., Walker, B., Haigwood, N. L. Continuous viral escape and selection by autologous neutralizing antibodies in drug-naive human immunodeficiency virus controllers. J. Virol. 83, 662-672 (2009).
- Migueles, S. A., Connors, M. The Role of CD4(+) and CD8(+) T Cells in Controlling HIV Infection. Curr. Infect. Dis. Rep. 4, 461-467 (2002).
- Palmer, S., Kearney, M., Maldarelli, F., Halvas, E. K., Bixby, C. J., Bazmi, H., Rock, D., Falloon, J., Davey, R. T., Dewar, R. L., Metcalf, J. A., Hammer, S., Mellors, J. W., Coffin, J. M. Multiple, linked human immunodeficiency virus type 1 drug resistance mutations in treatment-experienced patients are missed by standard genotype analysis. J. Clin. Microbiol. 43, 406-413 (2005).
- Palmer, S., Wiegand, A. P., Maldarelli, F., Bazmi, H., Mican, J. M., Polis, M., Dewar, R. L., Planta, A., Liu, S., Metcalf, J. A., Mellors, J. W., Coffin, J. M. New real-time reverse transcriptase-initiated PCR assay with single-copy sensitivity for human immunodeficiency virus type 1 RNA in plasma. J. Clin. Microbiol. 41, 4531-4536 (2003).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved