È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Amplificazione e quantificazione di HIV-1 RNA in soggetti affetti da HIV con una carica virale inferiore il limite di rilevamento da parte di Standard saggi clinici
In questo articolo
Riepilogo
Quantificare i livelli di HIV-1 RNA nel plasma e sequenziamento singolo HIV-1 genomi di individui con una carica virale al di sotto del limite di rilevabilità (50-75 copie / ml) è difficile. Qui si descrive come estrarre e quantificare l'RNA virale plasmatico usando un test PCR in tempo reale che misura affidabile HIV-1 RNA fino a 0,3 copie / ml e come amplificare genomi virali mediante sequenziamento del genoma unico, da campioni con una carica virale molto bassa.
Abstract
Amplificando i geni virali e la quantificazione di HIV-1 RNA di HIV-1 individui infetti con una carica virale al di sotto del limite di rilevabilità da test standard (sotto 50-75 copie / ml) è necessario per ottenere informazioni alla dinamica virale e le interazioni ospite del virus in pazienti che naturalmente controllare l'infezione e coloro che sono in terapia antiretrovirale combinata (CART).
Qui si descrivono come amplificare genomi virali da sequenziamento del genoma singolo (il protocollo SGS) 13, 19 e come quantificare con precisione HIV-1 RNA nei pazienti con bassa carica virale (la singola copia saggio (SCA) protocollo) 12, 20.
Il singolo-copia saggio è un real-time PCR con sensibilità a seconda del volume di plasma essere analizzati. Se un singolo genoma del virus viene rilevato in 7 ml di plasma, quindi il numero di copie di RNA è segnalato per essere 0,3 copie / ml. Il test è un test di controllo interno per l'efficienza di estrazione di RNA, e controlli per l'amplificazione del DNA o da possibili contaminazioni. I campioni dei pazienti sono misurati in triplice copia.
Il singolo test di sequenziamento del genoma (SGS), oggi largamente utilizzati e considerati non-lavoro intensivo 3, 7, 12, 14, 15, è un test di diluizione limite, in cui endpoint diluito prodotto cDNA si estende su una da 96 pozzetti piatto. Secondo una distribuzione di Poisson, quando meno di 1 / 3 dei pozzi danno prodotto, c'è una probabilità 80% della risultante prodotto di PCR è di amplificazione da una singola molecola di cDNA. SGS ha il vantaggio sulla clonazione di non essere sottoposto a ricampionamento e di non essere prevenuto mediante PCR-introdotto ricombinazione 19. Tuttavia, il successo di amplificazione di SCA e SGS dipendono dal disegno primer. Entrambi i saggi sono stati sviluppati per l'Hiv-1 sottotipo B, ma può essere adattato per altri sottotipi e in altre regioni del genoma di primer cambiando, sonde, e standard.
Protocollo
1. Estrazione di RNA da grandi volumi di plasma
Una panoramica dei test singola copia (SCA) e il singolo sequenziamento del genoma (SGS) protocollo sono forniti nelle figure 1 e 2.
- Per ottenere 7 ml di plasma, rotazione di circa 14 ml di sangue raccolte in 15 ml di EDTA (non eparina) tubi di raccolta a 2.600 xg per 15 minuti senza freni. Pipettare plasma (avendo cura di evitare lo strato del buffy coat) in provette da 15 ml.
- Se l'RNA è stato estratto per il saggio singola copia (SCA), punta il plasma con 30.000 copie del virione Virus Rous Sarcoma di controllo interno (RCA). Il virus RCA è pronto prima di eseguire SCA come descritto in precedenza (20). In breve, virus RCA da supernatante di coltura cellulare viene quantificato attività RT, diluito ad una concentrazione finale di 30.000 virioni per 200 ul, diluito in mezzi di coltura RPMI con il 5% FBS, e conservati a -80 ° C in aliquote uso singola. Il virus RCA non devono essere congelati / scongelati prima di utilizzare nel test. RCA è drogata nel plasma del paziente per controllare l'efficienza di estrazione dell'RNA e per la precisione di quantificazione PCR. Maggiori informazioni sulla RCA si possono trovare al seguente link: http://home.ncifcrf.gov/hivdrp/RCAS/ .
- "Pre-spin" al plasma, in 15 provette ml, a 2.600 xg per 15 minuti per estrarre le lipidi e detriti cellulari, che possono interferire con accurata quantificazione RNA.
- Etichetta Facile Seton-Seal provette con un pennarello indelebile per identificare il numero del campione e la posizione in cui il pellet si forma nel tubo. Dopo la pre-spin, il trasferimento al plasma ad una facile Seton-Seal provette pipettando il plasma ed evitando qualsiasi detriti cellulari e / o di lipidi in superficie. Registrare il volume di ingresso di plasma.
- Aggiungi Tris soluzione salina tamponata (TBS) per riempire il volume residuo della Seton-Easy tubo Seal alla parte inferiore del collo filettato. Assicurarsi che bolle presenti nel tubo o il tubo crollerà nel ultracentrifuga. Assicurarsi che il marchio sui tubi è rivolto verso l'esterno quando si posiziona i tubi nel rotore.
- Tenuta con tappi riutilizzabili e campioni luogo in un pre-raffreddata Sorval T1270 rotore e centrifugare in ultracentrifuga Sorval 90SE a 170.000 xg (50.000 rpm) a 4 ° C per 30 min.
- Dopo la centrifugazione rimuovere surnatante e aggiungere 90 ul di acqua grado molecolare e 10 ul-proteinasi K (20 mg / ml) al pellet virale (non sarà visibile).
- Tubi posto in un bagno d'acqua ° 55 C e incubare per 30 minuti. Assicurarsi che i tubi sono impostati in maniera tale che il lato con il pellet è immersa nel proteinasi-K e d'impasto.
- Dopo l'incubazione, poco girare il tubo in una centrifuga da tavolo per raccogliere tutto il liquido sul fondo della provetta (circa 5 secondi) e aggiungere 315 ul di soluzione di tiocianato 6M guanidinio e 10 ul di 20 mg / ml di glicogeno (Nota: attenersi appropriato direttive di eliminazione di questo materiale pericoloso, non mescolare con candeggina o acidi e smaltire in un contenitore separato).
- Vortex leggermente e girare a breve (per circa 5 secondi). Incubare per 10 minuti a temperatura ambiente e poi trasferire il contenuto di ciascun tubo per una nuova etichetta da 1,5 ml provette RNAse gratuito.
- Aggiungere 495 ul di molecolare alcool isopropilico per ogni tipo a tubi vortex per 10 secondi e centrifugare a 21000 xg per 30 minuti a temperatura ambiente.
- Gettare il surnatante e aggiungere 500 ul di etanolo 70%.
- Vortex per 10 secondi e centrifugare a 21000 xg per 15 minuti a temperatura ambiente. Eliminare l'etanolo con una pipetta di trasferimento. Girare di nuovo e rimuovere eventuali residui di etanolo con una pipetta. Aria secca per 10 minuti. (Per il protocollo SGS sciogliere RNA in 40 ul di 5 mM Tris-HCl pH 8.0, e continuare con il protocollo SGS).
- Sciogliere a pellet in 55 ul di RNA-buffer. RNA-buffer si ottiene aggiungendo 10 ul di 100 mM DTT e 25 ul di 40 U / ul Rnasin a 965 ul Tris-HCl (pH 8.0, 5 mm). Mettere in ghiaccio.
2. Il test a copia singola
Un piatto da 96 pozzetti contiene 8 campioni di pazienti inclusi sia l'HIV e standard RCA e controlli. Questo protocollo verrà descritto il set-up di un piatto unico.
- Inizia preparazione di 2 aliquote di 1 ml di RNA-tampone in cui l'HIV-1 RNA e RCA trascrizioni di RNA sarà diluito per le curve standard di RNA. RNA-buffer è descritta sotto 1.13.
- Preparare diluizioni sul ghiaccio nei tubi ml 2 centrifuga. Fare la metà-log diluizioni di HIV-1 RNA trascritti in RNA-tampone aggiungendo 25 ul di HIV-1 RNA magazzino (1x10 6 copie / ul) a 54 ul-RNA tampone dando 3x10 5 copie / ul.
- Continuare facendo consecutivi a metà log diluizioni (25 ul + 54 ul) fino a 0,3 copie per 10 ul (nota: 0,3, 1 e 3 diluizioni non sarà una parte della curva standard durante l'analisi di questi sono utilizzati come valori di estrapolazione e deve essere impostato durante incogniteING analisi). Trascrizioni RCA sono stoccate a 1x10 6 copie / ul. Allo stesso modo, preparare mezza log diluizioni di trascrizioni RCA in RNA-tampone, ma fino a 100 copie per 10 ul.
- Preparare il cocktail RT per la sintesi del DNA come indicato nella Tabella 1. Preparare RT e reazioni NRT elencati nella tabella 1 tenendo a mente di fare un piccolo extra per tenere conto di eventuali perdite che possono verificarsi durante il trasferimento. Nota: per il cocktail NRT, l'enzima RT è sostituito da acqua per il controllo per l'amplificazione da DNA.
Nota: questo passaggio è stato recentemente automatizzato oltre alla piastra istituiti su Robot Qiagen (originariamente Corbett). - Impostare il ottica 96-pozzetti (come mostrato nella Figura 3), aggiungendo 20 ul di miscela di reazione RT in ciascun pozzetto. In 8 pozzetti etichetta "TSN", aggiungere il cocktail senza la trascrittasi inversa (miscela di reazione NRT). Dopo aver aggiunto il cocktail al piatto, aggiungere 10 ul di acqua ai pozzi etichetta "Nessun controllo template (NTC)". Aggiungere 10 ul di trascrizioni HIV ai pozzi etichettato come "HIV" in concentrazioni mostrato nella Figura 3. Aggiungere 10 ul di trascrizioni RCA per pozzi etichetta "RCA" in concentrazioni secondo. Aggiungere 10 ul di campione del paziente nei 3 pozzetti adiacenti etichettato come "campione" sul diagramma piatto. Aggiungere 10 ul del campione eluito ai pozzetti etichetta "TSN". Aggiungere 5 ul dello stesso campione e 5 ul di acqua ai pozzi etichetta "controllo interno".
- Sigillare la piastra ed eseguito su termociclatore a: 25 ° C per 15 minuti, 42 ° C per 40 minuti, 85 ° C per 10 minuti, 25 ° C per 30 minuti, e 5 ° C in attesa.
- Preparare maestro PCR mix secondo la Tabella 2.
- Quando la sintesi del cDNA è stata completata, spostarsi in un'area designata per l'utilizzo di cDNA. In questa zona designata, aggiungere 20 ul del master mix PCR in ciascun pozzetto della piastra di cDNA sia per l'HIV e RCA. E 'importante eseguire questo passaggio in una zona designata per evitare ogni possibile contaminazione.
- Spin piatto, tenuta con coperchio ABI o Roche ottico e coprire con una coperta ottico (coperta necessaria solo per ABI 7700). Eseguiti su Roche 480 o ABI 7700. Condizioni di PCR: 95 ° C per 10 minuti, quindi 45 cicli di 95 ° C per 15 secondi, 60 ° C per 1 min. Un'analisi separata è richiesta per RCA e HIV. Esempi di risultati sono mostrati in Figura 4. La pendenza della curva standard sarà influenzato da una distribuzione normale di Poisson di trascrizioni a basso numero di copie. Al fine di garantire la pendenza più accurato per la curva standard, i bassi standard del numero di copie (0,3, 1 e 3 copie) sono impostati come "incognite." 17
3. Singolo Genome Sequencing
- sintesi del DNA. Aggiungere 5 ul di 10 mM dNTPs e 5 ul del 2 mM gene specifico primer (pol o ENV) a un pozzo in un pozzo 96 piastra PCR (Biorad). Aggiungere 40 ul di RNA estratto. Guarnizione della piastra.
- Denaturare l'RNA miscela in termociclatore a 65 ° C per 10 min.
- Mescolare i reagenti per la sintesi di cDNA, elencati nella Tabella 3. Dopo il passo denaturazione, luogo piastra PCR in ghiaccio, aggiungete i 50 ul della miscela cDNA di ogni campione.
- Esegui piastra PCR su termociclatore: 45 ° C per 50 min, 85 ° C per 10 minuti e 4 ° premuto C.
- In primo luogo PCR, 10 reazioni ul. Preparare la miscela di reazione PCR in un vassoio dei reagenti utilizzando i primer per il primo giro di amplificazione p6-rt o env (reagenti elencati nella tabella 3, primer elencati nella tabella 5).
- Utilizzando una pipetta multicanale erogare 8,0 ul di miscela di PCR a 73 pozzi in 96 pozzetti piastra PCR (70 campioni e 3 controlli negativi).
- Dopo la sintesi del DNA è stata completata la movimentazione delle lamiere cDNA e PCR contenente miscela di reazione PCR in un'area designata per l'utilizzo di cDNA. Nella zona, aggiungere 40 ul di Tris-HCl pH 8.0 ad ogni campione di cDNA portando il volume finale a 140 ul. Aggiungi ul 2.0 su cDNA a ciascuno dei 70 pozzi e 2,0 ul di acqua a ciascuno dei controlli negativi. Seal PCR piatto. Esegui il termociclatore, programma in tabella 4.
- Nested PCR - Reazioni 20 ul. Mescolare i reagenti per la nested PCR nel vassoio reagenti (reagenti elencati nella tabella 3, primer elencati nella tabella 5).
- Aggiungere 18 ul di miscela nested PCR a 73 pozzetti su una piastra 96 pozzetti PCR, con pipetta multicanale
- Diluire prima PCR 1:5 e aggiungere 2 ul di primo turno al nested PCR PCR. Esegui reazione PCR sul termociclatore, alle condizioni elencate nella Tabella 4.
- Esegui campioni su un gel di agarosio all'1%. Per garantire che la maggior parte dei prodotti di PCR sono il risultato di una singola molecola di cDNA, non oltre il 30% dei pozzi dovrebbe essere positiva. Il processo è stato automatizzato mediante il Beckman Coulter Biomek FM robot per caricare il contenuto delle piastre PCR per una riduzione dell'1%, 96 pozzetti E-gel (Invitrogen). E-gel sono correre per 7 minuti a E-base. Prodotti sequenza usando primer elencati nella Tabella 5.
4. Rappresentante dei risultati:
SCA:
Tutti i comandi devono passare al fine di considerare l'HIV-1 RNA di misura corretta. Se un controllo negativo è positive, la corsa dovrebbe essere ignorato a causa della possibile contaminazione. Al fine di controllare le perdite di RNA durante la fase di estrazione, almeno il 50% della RCA a spillo nel plasma dovrebbero essere recuperati. Se la media RCA è <15.000 copie, il campione non riesce e deve essere ignorata, perché una notevole quantità di RNA avrebbero potuto essere persi durante l'estrazione o altre fasi del protocollo. Ciò si verifica in circa il 10% di tutti i campioni dei pazienti testati probabilmente a causa di alto contenuto di lipidi plasmatici 6. Il recupero del virus RCA ha lo scopo di controllo per la qualità della estrazione e l'efficienza della sintesi del DNA e PCR. Non è utilizzato per correggere per l'HIV dal recupero HIV è probabilmente legato alle immunoglobuline e di altre proteine umane che lo rende leggermente diverso da virus isolati da colture di tessuti. Il recupero della RCA nel nostro laboratorio è stato in media 25.716 + / - 4.057 copie / miscela di reazione, o circa il 95% + / -15% del RNA RCA aggiunto 17. Il (non trascrittasi inversa) NRT controllo viene eseguito in parallelo al fine di testare per l'amplificazione di DNA. Il valore NRT è sottratto da ciascuno dei tre valori di HIV-1 RNA, la media è calcolata e se questo numero è inferiore a zero (l'amplificazione del DNA supera l'amplificazione di RNA), il risultato non e deve essere ignorata perché del rischio di amplificazione del DNA piuttosto che da RNA. Se l'HIV-1 RNA è solo amplificato da 1 / 3 pozzi, una nuova misurazione il campione si consiglia di assicurarsi che l'amplificazione è vero per il campione vero di essere analizzati e non a causa di un possibile contaminante.
Se tutti i controlli passano, la media di HIV-1 numero di copie di RNA nel plasma può essere calcolato.
Esempio 1: se la media di HIV-1 numero di copie è zero, il limite di rilevabilità è calcolato in base al volume del plasma analizzato. Per fare un esempio, diciamo 7 ml di plasma è stato analizzato. La più piccola quantità di RNA che avrebbero potuto essere rilevati con questo test sarebbe stato 1 copia in uno dei pozzi e 0 copie negli altri due pozzi con una media di 0,33 copie per bene. Il numero medio di copia deve poi essere moltiplicato per un fattore pari a 5,5 per arrivare al numero di copie totali nel eluizione RNA (vi è di 10 ul di ogni bene, ma il volume di eluizione totale era di 55 ul). Il limite inferiore di rilevamento è quindi: 5,5 x 0,33 copie diviso per 7 ml = 1.83 / 7 = 0,3 copie / ml. La concentrazione di HIV-1 RNA nel plasma in questo esempio è stato <0,3 copie / ml.
Esempio 2: HIV-1 RNA è rilevabile. L'RNA è stato estratto da 7 ml di plasma. Il numero di copie medio di HIV-1 RNA per pozzetto è calcolato per essere 2,0 copie per bene. Poi il numero di copie medio per ml di plasma è di 5,5 x 2,0 copie / 7 ml = 1,6 HIV-1 RNA / ml di plasma.
Un foglio di modello di Excel utilizzato per calcolare il numero di copie possono essere scaricati dal sito web.
SGS:
Se uno dei controlli negativi è positivo il percorso dovrebbe essere ignorato a causa della possibile contaminazione. Il numero di prodotti da ogni corsa dipende dalla carica virale in ogni campione e la condizione del campione. Nella nostra esperienza un sacco di lipidi nel plasma o cellule si riducono le possibilità di ottenere dei prodotti. Memorizzazione di condizioni e di congelamento e scongelamento prima del campione influenzerà anche il recupero di RNA notevolmente. In generale, quando si lavora con campioni con carica virale sotto le 50 copie / ml, il prodotto (bande su gel), è prevedibile in circa 1 / 3 -1 / 2 dei campioni trattati. A seconda dei fattori di cui sopra, 1-5 bande per piastra deve essere considerato un buon risultato a causa del basso carico virale in questi pazienti.
| sintesi del DNA (RT Cocktail / reazione cDNA) | 1 di reazione | Nessun trascrittasi inversa (NRT) cocktail / reazione cDNA (1 di reazione) |
| Molecolari di grado H 2 O | 8,1 ul | 8,2 ul |
| 25 mM MgCl 2 | 6 ul | 6 ul |
| 25 mM dNTP | 0,6 ul | 0,6 ul |
| 100 mM DTT | 0,2 ul | 0,2 ul |
| Esameri casuali (0,1 ug / ul) | 1,5 ul | 1,5 ul |
| 10X TaqMan tampone A | 3,0 ul | 3,0 ul |
| Rnasin (40 U / uL) | 0,5 ul | 0,5 ul |
| Strategene RT (200 U / ul) | 0,1 ul | Non aggiungere |
| Totale | 20 ul | 20 ul |
| Campione di RNA | 10 ul | 10 ul |
| Volume totale | 30 ul | 30 ul |
Tabella 1. Reazione MixtUres per la sintesi del DNA in analisi a copia singola.
| PCR Master Mix | 1 di reazione | Primer |
| H 2 O | 15,1 ul | HIV Forward primer 5'-CATGTTTTCAGCATTATCAGAAGGA-3 ' |
| 10X PCR buffer Oro | 2,0 ul | HIV Primer inversione 5'-TGCTTGATGTCCCCCCACT-3 ' |
| 25 mM MgCl 2 | 2,0 ul | HIV-Probe5'FAM CCACCCCACAAGATTTAAACACCATGCTAA TAMRA-3 ' |
| * Primer Forward (100 um) | 0,3 ul | |
| * Primer Reverse (100 um) | 0,3 ul | RCA avanti Primer5'-GTCAATAGAGAGAGGGATGGACAAA-3 ' |
| * Sonda (100 um) | 0,05 ul | RCA Reverse Primer5'-TCCACAAGTGTAGCAGAGCCC-3 ' |
| TaqGold (5 U / ul) | 0,25 ul | RCA sonda 5'FAM-TGGGTCGGGTGGTCGTGCC TAMRA-3 ' |
| Totale | 20,0 ul |
Tabella 2. PCR master mix e primer per analisi a copia singola.
| cDNA cocktail / cDNA reazioni | 1 RNA campione | In primo luogo PCR cocktail | 1 piastra (75 reazioni) | Nested PCR cocktail | 1 piastra (75 reazioni) |
| 10X RT buffer (Invitrogen) | 10 ul | 10X PCR buffer (Invitrogen) | 75 ul | 10X PCR buffer di | 150 ul |
| 25 mM MgCl 2 | 20 ul | 50 mM MgSO 4 | 30 ul | 50 mM MgSO 4 | 60 ul |
| 0.1M DTT | 1 ul | 10 mM dNTP | 15 ul | 10 mM dNTP | 30 ul |
| RNasi-free acqua | 17,5 ul | 50 primer uM (bis) | 3 ul | 50 primer uM (bis) | 6 ul |
| RNAsi-Out (Invitrogen) | 1 ul | Platinum Taq Hi Fi enzima (Invitrogen) | 6 ul | Platinum Taq Hi Fi enzima (Invitrogen) | 12 ul |
| Apice III (200 U / ul) (Invitrogen) | 0,5 ul | Molecolari acqua di grado | Ul 468 | Molecolari acqua di grado | 1086 ul |
Tabella 1. CDNA e miscele PCR per Genome Sequencing unico.
| P6-1 RT PCR programma | Busta 1 PCR programma |
| 1. 94 ° C per 2 minuti | 1. 94 ° C per 2 minuti |
| 2. 94 ° C per 30 secondi | 2 0,94 ° C per 30 secondi |
| 2 0,94 ° C per 30 secondi | 3. 52 ° C per 30 secondi |
| 4. 72 ° C per 1 minuto e 30 secondi | 4. 72 ° C per 1 minuto |
| 5. Vai alla # 2, 44 cicli | 5. Vai alla # 2, 44 cicli |
| 6. 72 ° C per 3 minuti | 6. 72 ° C per 3 minuti |
| 7. 4 ° C tenere | 7. 4 ° C tenere |
| P6-RT 2 PCR programma | Env 2 PCR programma |
| 1. 94 ° C per 2 minuti | 1. 94 ° C per 2 minuti |
| 2. 94 ° C per 30 secondi | 2. 94 ° C per 30 secondi |
| 3. 55 ° C per 30 secondi | 3. 56 ° C per 30 secondi |
| 4. 72 ° C per 1 minuto | 4. 72 ° C per 45 secondi |
| 5. Vai alla # 1, 40 (41 cicli in totale) | 5. Vai alla # 1, 40 (41 cicli in totale) |
| 6. 72 ° C per 3 minuti | 6. 72 ° C per 3 minuti |
| 7. 4 ° C tenere | 7. 4 ° C tenere |
Tabella 4. Termociclatore condizioni per il singolo test Genome Sequencing.
| Reazione | Primer | Sequenza |
| P6-RT cDNA | 3500 - | 5 'CTATTAAGTATTTTGATGGGTCATAA 3' |
| cDNA env | E115- | 5'AGAAAAATTCCCCTCCACAATTAA 3 ' |
| P6-RT 1. PCR | 3500 - | 5 'CTATTAAGTATTTTGATGGGTCATAA 3' |
| P6-RT 1. PCR | 1849 + | 5 'GATGACAGCATGTCAGGGAG 3' |
| P6-RT2.PCR | 1870 + | 5 'GAGTTTTGGCTGAGGCAATGAG 3' |
| P6-RT 2.PCR | 3410 - | 5 'CAGTTAGTGGTATTACTTCTGTTAGTGCTT 3' |
| env 1. PCR | E115- | 5'AGAAAAATTCCCCTCCACAATTAA 3 ' |
| env 1. PCR | E20 + | 5'GGGCCACACATGCCTGTGTACCCACAG 3 ' |
| env 2. PCR | E30 + | 5'GTGTACCCACAGACCCCAGCCCACAAG3 ' |
| env 2. PCR | E125- | 5'CAATTTCTGGGTCCCCTCCTGAGG 3 ' |
| P6-RT sequenziamento | 2030 + | 5'TGTTGGAAATGTGGAAAGGAAGGAC 3 ' |
| P6-RT sequenziamento | 2600 + | 5'ATGGCCCAAAAGTTAAACAATGGC3 ' |
| P6-RT sequenziamento | 2610 - | 5'TTCTTCTGTCAATGGCCATTGTTTAAC3 ' |
| P6-RT sequenziamento | 3330 - | 5'TTGCCCAATTCAATTTTCCCACTAA 3 ' |
| env sequenziamento | E30 + | 5'GTGTACCCACAGACCCCAGCCCACAAG3 ' |
| env sequenziamento | E125- | 5'CAATTTCTGGGTCCCCTCCTGAGG 3 ' |
Primer Tabella 5. Per il singolo test Genome Sequencing.
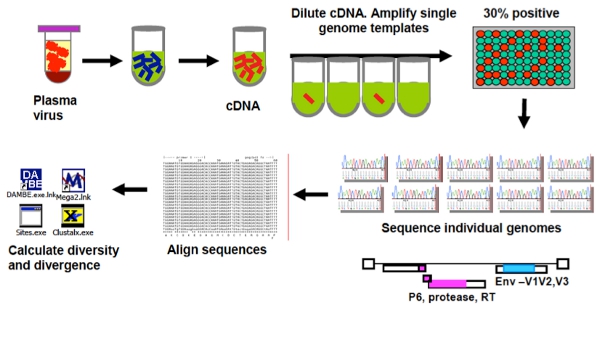
Figura 1. Panoramica del Genome Sequencing Assay Singe (SGS).
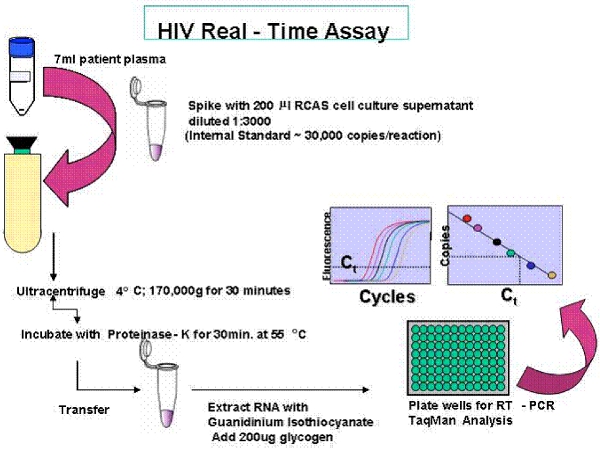
Figura 2. Panoramica del saggio a copia singola (SCA).
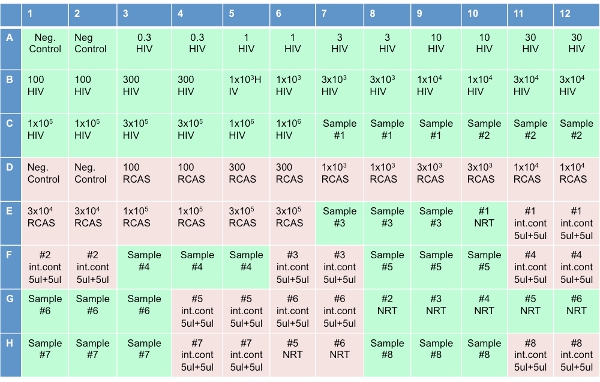
Figura 3. Piastra set-up per l'analisi a copia singola.
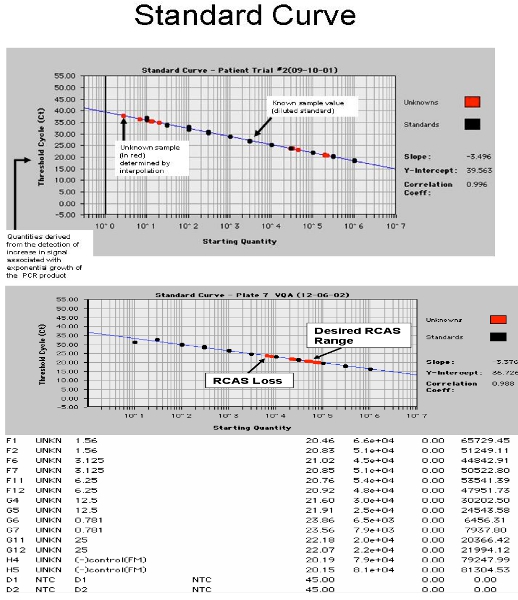
Figura 4. Schermata da ABI 7700. A. mostra il virus HIV-1 RNA con curva standard campioni di pazienti e B. mostrando curva standard RCA con campioni di pazienti a spillo.
Discussione
HIV-1 individui infetti in trattamento antiretrovirale combinata (CART) o che naturalmente controllare l'infezione hanno carica virale molto bassa, in genere circa 1 copia per ml di plasma 4, 11, 12, 17, 18. Carica virale nei pazienti con controllo naturale, spesso oscillare intorno ad un singolo set di punto 1, 2, 17. La sensibilità del test è descritto nel presente documento dipende in larga misura il volume di ingresso di plasma. In generale, abbiamo lavorato con 7 ml di plasma, ma i risul...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Gli autori desiderano ringraziare i pazienti che hanno partecipato a numerosi studi di HIV-1.
JMC era un professore di ricerca della American Cancer Society con il sostegno della Fondazione FM Kirby.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti |
| Esameri random (500 ug / ml) | Promega | C118A | |
| Taqman tampone A | Applied Biosystems | 4304441 | |
| RNAsin (40 U / ul) | Promega | N211B | |
| RT enzima (200 U / ul) | Strategene | 600107-51 | |
| AmpliTaq Gold DNA polimerasi | Applied Biosystems | 4311814 | 6-Pack |
| AmpliTaq 10XGold tampone PCR II | Applied Biosystems | 4311814 | viene fornito con l'enzima |
| Magnesiumcloride 25 mM | Applied Biosystems | 4311814 | viene fornito con l'enzima |
| 1M Tris-HCl, pH 8,0, 5 mM | Invitrogen | AM9855G | |
| Apice III enzima trascrittasi inversa, 200 U / ul | Invitrogen | 18080-044 | |
| Apice III tampone 10X | Invitrogen | viene fornito con l'enzima | |
| DTT 100 mM | Invitrogen | viene fornito con l'enzima | |
| Platinum Taq DNA polimerasi ad alta fedeltà 5 U / ul | Invitrogen | 11304-102 | |
| 10X High Fidelity PCR Buffer | Invitrogen | 11304-102 | viene fornito con l'enzima |
| Proteinasi K-20 mg / ml | Ambion | 2546 | |
| Guanidinio soluzione Tiocianato 6M | FlukaBiochemica | 50983 | |
| Glicogeno 20 mg / ml | Roche | 10901393001 | |
| Isopropanolo | Sigma-Aldrich | 19516 | |
| Etanolo al 70% | Sigma-Aldrich | E702-3 | |
| Molecolare acqua grado | Gibco / Invitrogen | 10977-015 | |
| dNTPs (25 mmol ognuno) | Bioline | BIO-39025 |
| Nome del Euipment | Azienda | Numero di catalogo | Commenti |
| Facile Seal provette da centrifuga (16x73mm) | Seaton scientifico | 6041 | |
| Bianco Delrin corone | Seaton scientifico | 4020 | Tappi per le Tubi Seal Facile |
| Rack tubo per Optiseal Bell-Top Tubi, 8,9 ml | Beckman | 361642 | |
| Hex conducente ½ apertura inc | Seaton scientifico | 4013 | |
| Tappo rimozione TUPE | Seaton scientifico | 4014 | |
| 5 ml pipette sierologiche | Costar | 4051 | |
| Pipetboy | qualsiasi | ||
| 15 ml di trasferimento pipette | ISC Bioexpress | P-5005-7 | |
| 5,8 ml di pipette di trasferimento Dispossable, punta fine | VWR | 14670-329 | |
| Provette da 15 ml per centrifuga | Falco | ||
| 1 ml centrifuga tubi | qualsiasi | ||
| Tampone Tris compresse Saline | Sigma-Aldrich | T5030-100TAB | |
| Ultracentrifuga e rotore | Sorval | 90SE e T-1270 | |
| 96-PCR e piastre | Biorad | HSS-9601 | |
| 50 ml di Reagente serbatoio | Costar | 4870 | |
| Microampere ottico 96-pozzetti di reazione | Applied Biosystems | N801-0560 | |
| Piatto ottico copre | Applied Biosystems | 4333183 | |
| MicroAmp Pad compressione ottica | Applied Biosystems | 4312639 | |
| MicrosealUn film | Biorad | MSA-5001 | |
| 2 ml centrifuga tubi | Qualsiasi | ||
| 1% gel di agarosio (E-gel, Invitrogen) | Invitrogen | G-7008-01 | |
| E-base (Invitrogen) | Invitrogen | EB-M03 |
EDTA tubi di raccolta di qualsiasi marca
Riferimenti
- Amara, R. R., Villinger, F., Altman, J. D., Lydy, S. L., O'Neil, S. P., Staprans, S. I., Montefiori, D. C., Xu, Y., Herndon, J. G., Wyatt, L. S. Control of a mucosal challenge and prevention of AIDS by a multiprotein DNA/MVA vaccine. Vaccine. 20, 1949-1955 (2002).
- Dinoso, J., Kim, S., Blankson, J., Siliciano, R. F. Viral Dynamics of Elite Suppressors in HIV-1 Infection. Conference on Retroviruses and Opportunistic Infections. , (2008).
- Dinoso, J. B., Kim, S. Y., Wiegand, A. M., Palmer, S. E., Gange, S. J., Cranmer, L., O'Shea, A., Callender, M., Spivak, A., Brennan, T., Kearney, . Treatment intensification does not reduce residual HIV-1 viremia in patients on highly active antiretroviral therapy. Proc. Natl. Acad. Sci. U.S.A. 106, 9403-9408 (2009).
- Doria-Rose, N. A., Klein, R. M., Manion, M. M., O'Dell, S., Phogat, A., Chakrabarti, B., Hallahan, C. W., Migueles, S. A., Wrammert, J., Ahmed, R., Nason, M., Wyatt, R. T., Mascola, J. R., Connors, M. Frequency and phenotype of human immunodeficiency virus envelope-specific B cells from patients with broadly cross-neutralizing antibodies. J. Virol. 83, 188-199 (2009).
- Dornadula, G., Zhang, H., Uitert, B. V. a. n., Stern, J., Livornese, L., Ingerman, M. J., Witek, J., Kedanis, R. J., Natkin, J., DeSimone, J., Pomerantz, R. J. Residual HIV-1 RNA in blood plasma of patients taking suppressive highly active antiretroviral therapy. JAMA. 282, 1627-1632 (1999).
- Gandhi, R. T., Zheng, L., Bosch, R. J., Chan, E. S., Margolis, D. M., Read, S., Kallungal, B., Palmer, S., Medvik, K., Lederman, M. M., Alatrakchi, N., Jacobson, J. M., Wiegand, A., Kearney, M., Coffin, J. M., Mellors, J. W., Eron, J. J. The effect of raltegravir intensification on low-level residual viremia in HIV-infected patients on antiretroviral therapy: a randomized controlled trial. PLoS medicine. 7, (2010).
- Gay, C., Dibben, O., Anderson, J. A., Stacey, A., Mayo, A. J., Norris, P. J., Kuruc, J. D., Salazar-Gonzalez, J. F., Li, H., Keele, B. F., Hicks, C., Margolis, D., Ferrari, G., Haynes, B., Swanstrom, R. Cross-sectional detection of acute HIV infection: timing of transmission, inflammation and antiretroviral therapy. PLoS One. 6, 19617-19617 (2011).
- Hatano, H., Delwart, E. L., Norris, P. J., Lee, T. H., Dunn-Williams, J., Hunt, P. W., Hoh, R., Stramer, S. L., Linnen, J. M., McCune, J. M., Martin, J. N., Busch, M. P., Deeks, S. G. Evidence for persistent low-level viremia in individuals who control human immunodeficiency virus in the absence of antiretroviral therapy. J. Virol. 83, 329-335 (2009).
- Havlir, D. V., Bassett, R., Levitan, D., Gilbert, P., Tebas, P., Collier, A. C., Hirsch, M. S., Ignacio, C., Condra, J., Gunthard, H. F., Richman, D. D., Wong, J. K. Prevalence and predictive value of intermittent viremia with combination hiv therapy. JAMA. 286, 171-179 (2001).
- Jordan, M. R., Kearney, M., Palmer, S., Shao, W., Maldarelli, F., Coakley, E. P., Chappey, C., Wanke, C., Coffin, J. M. Comparison of standard PCR/cloning to single genome sequencing for analysis of HIV-1 populations. J Virol Methods. 168, 114-120 (2010).
- Kaufmann, D. E., Kavanagh, D. G., Pereyra, F., Zaunders, J. J., Mackey, E. W., Miura, T., Palmer, S., Brockman, M., Rathod, A., Piechocka-Trocha, A., Baker, B., Zhu, B., Gall, S. L. e., Waring, M. T., Ahern, R., Moss, K., Kelleher, A. D. Upregulation of CTLA-4 by HIV-specific CD4+ T cells correlates with disease progression and defines a reversible immune dysfunction. Nat Immunol. 8, 1246-1254 (2007).
- Kearney, M., Maldarelli, F., Shao, W., Margolick, J. B., Daar, E. S., Mellors, J. W., Rao, V., Coffin, J. M., Palmer, S. HIV-1 Population Genetics and Adaptation in Newly Infected Individuals. J. Virol. 83, 2715-2727 (2008).
- Kearney, M., Palmer, S., Maldarelli, F., Shao, W., Polis, M. A., Mican, J., Rock-Kress, D., Margolick, J. B., Coffin, J. M., Mellors, J. W. Frequent polymorphism at drug resistance sites in HIV-1 protease and reverse transcriptase. AIDS. 22, 497-501 (2008).
- Kearney, M., Spindler, J., Shao, W., Maldarelli, F., Palmer, S., Hu, S. L., Lifson, J. D., Kewal Ramani, V. N., Mellors, J. W., Coffin, J. M., Ambrose, Z. Genetic diversity of simian immunodeficiency virus encoding HIV-1 reverse transcriptase persists in macaques despite antiretroviral therapy. Journal of Virology. 85, 1067-1076 (2011).
- Keele, B. F., Giorgi, E. E., Salazar-Gonzalez, J. F., Decker, J. M., Pham, K. T., Salazar, M. G., Sun, C., Grayson, T., Wang, S., Li, H., Wei, X., Jiang, C., Kirchherr, J. L., Gao, F., Anderson, J. A., Ping, L. H., Swanstrom, R. Identification and characterization of transmitted and early founder virus envelopes in primary HIV-1 infection. Proc. Natl. Acad. Sci. U.S.A. , 105-7552 (2008).
- Liu, S. L., Rodrigo, A. G., Shankarappa, R. G., Learn, H., Hsu, L., Davidov, O., Zhao, L. P., Mullins, J. I. HIV quasispecies and resampling. Science. 273, 415-416 (1996).
- Mahalanabis, M., Jayaraman, P., Miura, T., Pereyra, F., Chester, E. M., Richardson, B., Walker, B., Haigwood, N. L. Continuous viral escape and selection by autologous neutralizing antibodies in drug-naive human immunodeficiency virus controllers. J. Virol. 83, 662-672 (2009).
- Migueles, S. A., Connors, M. The Role of CD4(+) and CD8(+) T Cells in Controlling HIV Infection. Curr. Infect. Dis. Rep. 4, 461-467 (2002).
- Palmer, S., Kearney, M., Maldarelli, F., Halvas, E. K., Bixby, C. J., Bazmi, H., Rock, D., Falloon, J., Davey, R. T., Dewar, R. L., Metcalf, J. A., Hammer, S., Mellors, J. W., Coffin, J. M. Multiple, linked human immunodeficiency virus type 1 drug resistance mutations in treatment-experienced patients are missed by standard genotype analysis. J. Clin. Microbiol. 43, 406-413 (2005).
- Palmer, S., Wiegand, A. P., Maldarelli, F., Bazmi, H., Mican, J. M., Polis, M., Dewar, R. L., Planta, A., Liu, S., Metcalf, J. A., Mellors, J. W., Coffin, J. M. New real-time reverse transcriptase-initiated PCR assay with single-copy sensitivity for human immunodeficiency virus type 1 RNA in plasma. J. Clin. Microbiol. 41, 4531-4536 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon