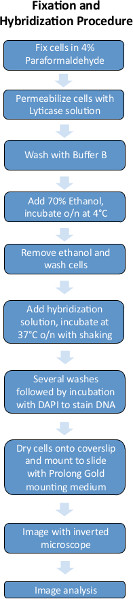
Method Article
הדמיה וניתוח של מולקולות ה-mRNA באמצעות הקרינה
In This Article
Summary
פרוטוקול זה מתאר הליך ניסיוני לביצוע פלואורסצנטי באתר הכלאה (דגים) לספירת mRNAs בתאים בודדים ברזולוציה של מולקולה בודדת.
Abstract
הקרינה הכלאה באתרו (FISH) השיטה מאפשרת לאדם לזהות את חומצות גרעין בסביבת הסלולר המקומית. כאן אנו מספקים פרוטוקול לשימוש בדגים כדי לכמת את מספר mRNAs בתאי שמרים בודדים. ניתן לגדל תאים בכל מצב של עניין ולאחר מכן תיקנו ועשו חדיר. כתוצאה מכך, deoxyoligonucleotides יחידה תקוע מרובים מצומדות לצבעי ניאון משמש לתייג ולדמיין mRNAs. הקרינה עקיפה מוגבלת ממולקולות ה-mRNA יחידה היא לכמת באמצעות אלגוריתם ספוט זיהוי כדי לזהות ולספור את מספר mRNAs לכל תא. בעוד שיטות הכימות הסטנדרטיות יותר של כתמים צפוניים, RT-PCR וmicroarrays ביטוי גנים לספק מידע על mRNAs הממוצעת באוכלוסייה בתפזורת, דגים מאפשרים גם הספירה והלוקליזציה של mRNAs אלה בתאים בודדים ברזולוציה מולקולה בודדת.
Introduction
באמצעות שימוש בטכניקות מדידה בכמות גדולה, שלא ניתן לassay מספר התעתיקים או פעילות תעתיק בתוך תאים בודדים 1. באמצעות חלבונים פלואורסצנטיים המונעים על ידי יזמים בעלי עניין ככתבים של ביטוי גנים יכולים לטפל בבעיה זו במידה מסוימת, אך הזמן הנדרש לחלבוני ניאון להתקפל מטשטש דינמיקה מוקדמת. חלבוני ניאון חיים ארוכים גם לא יכולים לדווח על חיי mRNA. שיטת הדגים יכולה לשמש כדי mRNA assay במהלך מחזור החיים המלא שלו, מהתחלת שעתוק בגרעין להבשלה וריקבון שלאחר מכן בתאים בודדים, עם רזולוציה של מולקולה בודדת.
המקורי בניסויים באתר המאפשר הדמיה חומצות גרעין המשמשות בדיקות RNA radiolabeled לחקור רצפי דנ"א. אלה כללו הדמיה דנ"א ריבוזומלי בשחלות של צפרדע Xenopus laevis 2 וה-DNA בלווין ברקמת עכבר 3. הניאון ראשון באתרו experiment משמש מולקולת RNA מסומנת בfluorophore לחקור רצפי דנ"א מסוימים 4. היישום הראשון של בדיקות ניאון המאפשרים הדמיה RNA באתרו היה ההדמיה של ביטוי גנים אקטין בתרבית רקמת שריר עוף 5. לאחרונה, בשמרי ניצנים, דגים נעשו שימוש כדי לחקור את התנודות בשעתוק במהלך מחזור חילוף החומרים השמרים 6, הדעיכה של mRNAs במהלך התקדמות במחזור תא 7, ולוקליזציה המרחבי של תעתיקי mRNA במהלך מיטוזה 8. דגים כבר בשימוש בשמרים כדי להראות שתנודות מתואמות בגנים constitutively עיבד, המהווים יותר ממחצית מכל הגנים השמרים, נובעות מייזום שעתוק מתואם 9. במינים שאינם שמרים, דגים כבר משמשים לזיהוי סמנים בתאי גזע במעי העכבר 10 ולקבוע כי penetrance שלמה של גורלות סלולריים יכולה לנבוע מתנודות אקראיות בביטוי גנים געוברי elegans 11.
שיטת הדגים המתוארות כאן פועלת על ידי ההכלאה שכותרתו צבע, בדיקות דנ"א חד גדילי mRNA להודעות. תאים הם צילמו וmRNAs נספרות באמצעות אלגוריתם זיהוי מקום. יכולות להיות שנוצרו בדיקות יחידה התקועות עם סינתיסייזר ולאחר מכן ה-DNA שכותרתו (המכונה כאן בדיקות זינגר) או הורה מסחרי כמו בדיקות שכותרתו מראש (בדיקות Stellaris) 12,13. הבדל עיקרי בין בדיקות זינגר וStellaris הוא כי בדיקות זינגר הן ארוכות יותר (~ 50 נ"ב) וכותרתו הן רב ואילו בדיקות Stellaris הן קצרות (~ 20 נ"ב) עם תווית אחת בלבד לכל בדיקה, כפי שתוארה על ידי ראג' ואח' 14. בנוסף, גישת Stellaris שימושים רבות יותר לבדיקות גנטית מזה של זינגר (~ 30 לעומת 5 בדיקות בגן, בהתאמה). להלן אנו מספקים פרוטוקול שמתאר את השימוש בכל אחד מסוגי הבדיקה. בסעיף 2, אנו מספקים פרוטוקול לתיוג בדיקות המכילות thymidine האמין allyl wה-i סיי צבע שנבחר. סקירה של צעדי החישוב הנדרשים כדי לזהות כתמים בודדים mRNA היא אמורה בסעיף 7.
Protocol
איורים 1 ו -2 הם שרטוטים של הפרוצדורות הדגים וצנרת ניתוח תמונה המשמשת לכימות תמונות דגים.
1. פתרונות כדי להכין
* פתרונות להלן הם לשימוש עם בדיקות זינגר. אם באמצעות בדיקות Stellaris, להחליף "לפוראמיד 40%" עם "לפוראמיד 10%" בשניהם חיץ הכלאה והצפה לשטוף. שינויים נוספים במאגר ההכלאה כאשר באמצעות בדיקות Stellaris הם (1) סולפט 1 גרם Dextran להוסיף ו( 2) לא כולל 10 מ"ג ssDNA.
חיץ B
8 מ"ל 1 M KH 2 PO 4
41.5 מ"ל 1M K 2 HPO 4
109.3 גרם סורביטול
מאגר spheroplasting
890 ב 'מאגר μl
VRC μl 100
10 μl 25,000 U / מיליליטר Lyticase
2 β-mercaptoethanol μl
<מאגר הכלאה> חזק (נפח מ"ל, סופי 10)
10 מ"ג א ' tRNA coli
10 מ"ג ssDNA *
100 מניות μl 200 מ"מ VRC
40 μl של 50 מ"ג / מיליליטר BSA
מיליליטר 20X SSC 1
4 40% לפוראמיד מ"ל *
המים nuclease חינם (לנפח סופי 10 מ"ל)
1 סולפט * Dextran גרם
* מאגר ההכלאה נשמרות ב0.5 aliquots מ"ל ב -20 ° C לנוחות.
שטוף חוצץ (מ"ל, נפח סופי 50)
5 מ"ל 20X SSC
% 20 מ"ל 40 לפוראמיד *
המים nuclease חינם (לנפח סופי 50 מ"ל)
תיוג הצפת
1.06 נתרן קרבונט גרם
מים 100 מ"ל DEPC
ה-pH 9
2. בדיקה תיוג (בדיקות זינגר בלבד)
אנו להשיג בדיקות אלה בסינתזה בתוך הבית באמצעות מנגנון סינתזת oligonucleotide ABI. Typicהברית, 4-5 ~ 50 נ"ב oligonucleotides מסונתז שהם הומולוגיים לגן של עניין, החלפת thymidine אמין allyl במשך כמה thymidines מרווחים לפחות 8, רצוי 10 + נ"ב זה מזה. בגלל רגישותם לאוזון, אנו עובדים במתקן ללא אוזון בעת שימוש בצבעי CY.
- השג ~ 5 בדיקות וresuspend במי μl 100 - ריכוזים לבדוק על Nanodrop.
- תלוי כמה בדיקות / גנים, לשלב כולל של 10 oligonucleotides מיקרוגרם / גן (למשל, אם יש 5 / בדיקות גנטיות, אז רוצים 2 מיקרוגרם / חללית).
- השתמש בעמודות QIAquick לטהר בדיקות על פי פרוטוקול ערכת הסרת נוקלאוטיד QIAquick.
- הוסף 10 כרכי המאגר PN לסך נפח של בדיקות ותמהיל משולבים.
- החל מדגם לעמודת QIAquick - אם נפח כולל הוא יותר מ 750 μl, ספין למטה פעמיים באמצעות מחצית מנפח בכל ספין
- לתת לעמוד במשך דקות 1.
- צנטריפוגה 1 דקות ב 6000 סל"ד.
- לשטוף עם 750 PE המאגר μl.
- סרכזת1 דקות ב 6000 סל"ד.
- השלך לזרום ועמודה מחדש ספין 1 דקות ב13,000 סל"ד לייבוש.
- טור QIAquick מקום בצינור microcentrifuge החדש וה-DNA elute עם 50 H 2 O μl - ודא H-pH O 2 הוא בתוך 7.0 ו -8.5 וממוקם ישירות על הממברנה.
- לתת לעמוד במשך דקות 1.
- צנטריפוגה 1 דקות ב13,000 סל"ד elute ה-DNA.
- Lyophilize DNA ב 45 ° C.
- Resuspend גלולה ב10 חיץ תיוג μl ולהוסיף לצינור של צבע בלי לגעת בצבע.
- חזור עם חיץ תיוג μl 10 אחר.
- וורטקס ומסובב את הצינור של צבע וה-DNA.
- לכסות את הצינורות בנייר אלומיניום ולשמור בחושך בRT O / N לתייג.
- חזור על פרוטוקול ערכת הסרת נוקלאוטיד QIAquick. להופיע עם שני הבדלים:
- הוספה 200 חיץ PN μl לבדיקות ולשים את בדיקות שכותרתו דרך 2x עמודות.
- בצע 3 שוטף עם החיץ PE כדי לשטוף את כל צבע פנוי לפני elution. - After elution להשיג באמצעות ריכוז NanoDrop. יעילות התיוג היא בדרך כלל ~ 0.25 pmol / ng של גדילי דנ"א יחיד.
3. הכנת coverslip
- coverslips מקום בשקופיות בפלזמה-להתקשט ואקום קאמרי (http://www.plasmapreen.com/) (הקרוב יותר למרכז טוב יותר).
- שים תא ואקום במיקרוגל ולוודא שהוא אטום.
- הפעילו משאבה הראשונה לאחר מכן הפעל את הוואקום רק פעם אחת משאבה הוא התחיל.
- הפעל מיקרוגל, ולהפסיק 5 שניות לאחר שהפלזמה היא גלויה לעין.
- כבה את הוואקום ואז לשאוב.
- משוך את תא ואקום ולהסיר coverslips עם מלקחיים (אלה שנפלו יש לנקות שוב).
- מקום Coverslips צד ניקה ב12 גם צלחות.
4. נוהל קיבעון
- לגדול שמרים לOD 600 של כ 0.1-0.2 בתקשורת מינימאלית. 10 מ"ל של תאים מניב מספיק עבורr ~ 10 הכלאות נפרדות.
- הוסף 37% פורמלדהיד נפח 1/10 באופן ישיר לתקשורת הצמיחה (10 מ"ל של התרבות + 37% פורמלדהיד 1 מ"ל) ולתת לשבת במשך 45 דקות.
- לשטוף 2x עם B 1 מ"ל קר כקרח הצפת בצינור microcentrifuge (יכול להסתובב ב13,000 סל"ד דקות 1).
- הוסף 1 מ"ל של חיץ spheroplasting.
- לדגור על 37 מעלות צלזיוס למשך 15 דקות. בדקו כל כמה תאים תחת מיקרוסקופ דקות עד שרוב התאים הם שחורים (כלומר, לא בשלב בהיר).
- לשטוף 2x B עם המאגר הקר כקרח, ספינינג במהירות נמוכה (~ 3,500 סל"ד).
- הוסף 1 מ"ל של 70% EtOH, resuspend בעדינות ולהשאיר למשך הלילה ב 4 ° C (ניתן לאחסן לזמן בלתי מוגבל ב -20 ° C).
5. נוהל הכלאה
- הכן את פתרון ההכלאה: עד 100 μl של חיץ הכלאה, להוסיף 1-3 μl של חללית, ואז מערבולת ו צנטריפוגות. בדיקות זינגר להשתמש 8-10 כולל ng לכל סט בדיקה (כלומר לכל גן). יש לנו צילמו עד 3 גנים בו זמנית עם 3 אונטערשיידתווית בדיקה שונה שממפה קובעת.
הקפד לחמם את פתרון ההכלאה לטמפרטורת חדר לפני פתיחתו.
לבדיקות Stellaris, מומלץ להתחיל 4 תגובות הכלאה נפרדות על ידי הוספת μl 1 כל אחד מ1:10, 1:20, 1:50 ו1:100 דילולים של בדיקות עובדים כדי לראות איזה מהם הוא אופטימלי. דילולים עבודה של בדיקות Stellaris ערוכים חיץ הכלאה.
- צנטריפוגה (את כל הצעדים הבאים; 3,500 סל"ד; 5 דקות) את התאים הקבועים (למשל 200 μl) ולשאוב ממנו את האתנול.
- resuspend עדינות בחיץ 1 מיליליטר שטיפה המכיל את אותו אחוז לפוראמיד כחיץ ההכלאה. בואו לעמוד במשך 2-5 דקות.
- צנטריפוגה מדגם וחיץ לשטוף לשאוב, ואז מוסיף פתרון הכלאה. דגירה בחושך עם רעד עדין, O / N ב 37 ° C.
הערה: ההליך הבא הוא עבור יישום תאים / הדמיה על coverslips. עבור washing / תאי הדמיה בצלחות 96-כן, כולל פתרון חלופי תגובתי חמצן מיני נבלות לראות http://www.biosearchtech.com/stellarisprotocols.
- למחרת, או מראש, נקי (ראה הליך) ולטפל בתלושים לכסות עם 150 0.01% פולי-L-ליזין μl למשך 5 דקות. לשאוב, לתת להתייבש, לשטוף 3x עם DH 2 O ולתת להתייבש.
- בבוקר, להוסיף 1 מ"ל של חיץ לשטוף לדוגמה, resuspend בעדינות, צנטריפוגות ולשאוב, אז resuspend ב 1 מ"ל נוסף של חיץ לשטוף ולדגור על 37 מעלות צלזיוס למשך 30 דקות.
- לשטוף עם 2X SSC + 0.1% דקות טריטון X-100 ב RT 15 על שייקר.
- קח הרכבה בינונית ממקפיא כדי לאפשר לו לבוא לRT לפני ההרכבה.
- לשטוף עם 1X SSC בדקות RT 15 על שייקר.
- לדלל DAPI לתוך PBS (0.1 מיקרוגרם / מ"ל סופי) ותאי resuspend ב 150 μl.
- פתרון מקום אל ניקה / פולי-lys-גידתלושי טד לכסות בצלחת 12 היטב, לפחות 30 דקות באין מפריע.
- סור פתרון (אולי אתה מניח אותו על פיסת כיסוי חילוף כגיבוי) ולשטוף 3x עם 1 מיליליטר 1X PBS.
- הנח 3 μl של להאריך הרכבה בינונית זהב לשקופית (Invitrogen P36934). לעשות את זה אחת בכל פעם, אם יש לך תלושי לכסות מרובים כדי למנוע התייבשות של הרכבה בינונית.
- הוסף אתנול ~ 0.5 מ"ל לcoverslip בצלחת 12 היטב, להסיר להחליק את המכסה והאוויר היבש בעת החזקה עם פינצטה.
- הנח לכסות להחליק תא בצד למטה על הרכבה בינונית ולתת להקשיח כמה שעות או O / N בחושך.
- לאטום את הקצוות עם לק ולהמשיך להדמיה.
6. הדמיה של תאים עם סקירה כללית מיקרוסקופ הפוכה אולימפוס IX-81
- לרכישת תמונה אנו מנצלים תוכנה (אינטליגנטי-imaging.com) Slidebook ו,, מטרת שמן 1.45 NA TIRFM 100X. לGFP סדרתי, DAPI, Cy3, Cy3.5 וCy5 הדמיה: מערכות סינון Chroma (ראה חומרים כימיים).
- השתמש במסנן DAPIכדי למצוא ולמקד את התאים.
- להגדיר את זה כנקודת התייחסות.
- קח Z-מחסנית של תמונות סביב נקודת ההתייחסות שבו המרחק הכולל הוא 5 מיקרומטר עם גודל 0.2 מיקרומטר (25 מטוסים). חזור על פעולה עבור כל אחד מערוצי צבע (קבועה בכל בדיקה, כלומר).
- יצוא כמו TIFF 16 ביט לניתוח תמונה.
7. סקירת ניתוח תמונה (בדיקות זינגר)
להלן אנו מספקים קווי המתאר של שיטות חישוביות אנו משתמשים לניתוח תמונות דגים בMATLAB. פונקציות MATLAB הרלוונטיות שמשו הם בסוגריים בצד ימין. האלגוריתמים והספים מכוונים כעת לנתונים מבדיקות בסגנון זינגר. באמצעות בדיקות בסגנון Stellaris ידרוש קצת הסתגלות, במיוחד לסינון השלב האחרון (7.8).
זיהוי תא 15
- תאים נפרדים מרקע באמצעות עולמי על סף DAPI תמונות 16 [graythresh].
- זהה את הגרעינים באמצעות פונקצית מקסימום מורחבת [imextendedmax].
- תאי מגזר באמצעות גרעינים כזרעים לאלגוריתם פרשת מים [קו פרשת מים].
מצא את נקודות בכל אחד מערוצי הקרינה
- לבצע שינוי tophat לנרמל את הרקע ולשפר את יחס אות לרעש [bwmorph].
- מצא את מקסימום כדי לזהות את השכבה בפוקוס (במטוס-z) על כל מקום [imregionalmax].
- סנן כתמים פוטנציאליים תוך שימוש במודל מתאים ליניארי לגרדיאנט רדיאלי.
עוצמת כתם מדוד ואותות בדיקה בודדות לעומת מרובים סינון
- Fit פרופיל Gaussian 2D למקום בשכבה בפוקוס זוהתה בעבר ובעוצמת אומדן 17.
- לסנן את הנקודות תורפה בשימוש בסף (המבוסס על היסטוגרמה). לבדיקות מסוג הזמר הזה הוא חשוב, אבל הוא פחות מכך לבדיקות Stellaris.
- לספור נקודות בכל אחד מתאים.
תוצאות
איור 3 מציג היסטוגרמות טיפוסיים מחושבים מתמונות דגים ומשמשים כדי לקבוע את מספר mRNAs המצויים בתאים בודדים. יתרון חשוב של מיקרוסקופיה מבוסס כימות RNA הוא שאדם יכול להשיג מידע על הלוקליזציה של תמלילים. לדוגמה, השתמשנו בדגים כדי לזהות mRNAs בתאים בודדים עם אלל מושרה CBF1 (איור 4). כי מולקולות mRNA רבות נמצאות באתר של שעתוק, אנו מסוגלים לזהות את נוכחותו ואת המיקום של אתרי שעתוק בתוך הגרעין.
על ידי ניצול צבעים שונים כדי mRNAs התווית של גנים שונים, אפשר לכמת מיני mRNA מרובים באותם תאים. כדי להמחיש זאת, תאי שמרים הודגרו בנוכחות α-הפקטור וסורביטול. FUS1 שעתוק (Quasar670 צבע, אדום) הנגרם על ידי α-גורם. שעתוק STL1 (Quasar 570 צבע, ירוק) הוא מושרה על ידי עליות בosmolarity תאי ( איור 5). איור 4 הוא דוגמה של דג עם בדיקות זינגר. איור 5 הוא דוגמה של דג עם בדיקות Stellaris.
איור 1. סכמטי של דגי הליך ניסיוני. לחץ כאן לצפייה בדמות גדולה.

איור 2. סכמטי של צינור ניתוח תמונה. הנקודות הסופיות נקבעות בדמות הימנית ביותר.
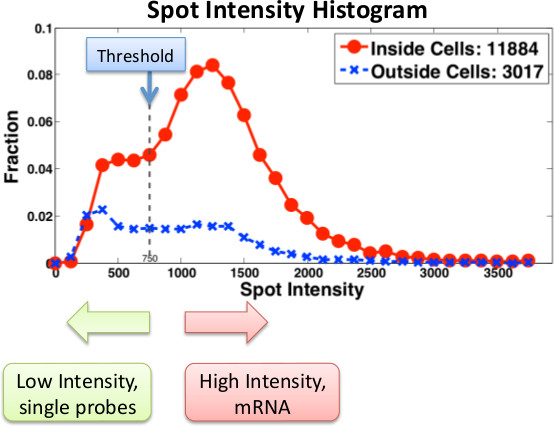
איור 3. היסטוגרמה של עוצמות מקום עבור גן מסוים באמצעות בדיקות זינגר . עוצמות בדיקה מחושבות מבפנים (אדום) ו( הכחולים) של תאים חיצוניים. כתמים בעצימות נמוכים הם או רעש או בדיקות יחידה. הודעות ה-mRNA אמיתיות מסומנות עם בדיקות מרובות. בעת שימוש בבדיקות Stellaris, בדיקות יחידה הן פחות לזיהוי וכך הספים והסינון חייבים להיות בהתאם.
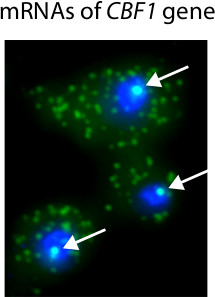
איור 4. נציג תוצאות של הליך דגי זמרת. בניסוי זה, שעתוק CBF1 מופעל עם אמרגן מושרה 18. הגרעינים הם מוכתמים כחולים עם DAPI. MRNAs CBF1 מתויגות עם בדיקות Cy3 שכותרת. חצים לבנים להדגיש את נוכחותו של CBF1 אתרי שעתוק בגרעין. תעתיקי mRNA יחיד גלויים בציטופלסמה.
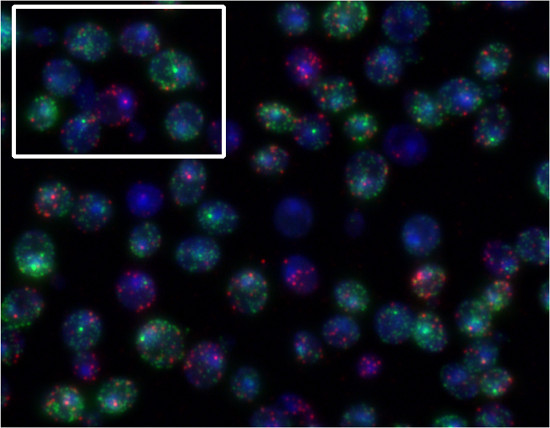
איור 5. נציג תוצאות של הליך דגי Stellaris. תאי שמרי מאטה בו זמנית נחשפו ל -30 α-גורם ng / ml 0.75 מ 'וסורביטול למשך 10 דקות ובו זמנית נבדקו עבור FUS1 (Quasar 670, אדום) וSTL1 (Quasar 570, ירוק) תעתיקים. בתיבה המסומנת, אנחנו יכולים לראות רק תא אחד מגיב לפרומון (אתר תחילת FUS1, אדום) ועוד בעיקר מגיבים לסורביטול (אתר תחילת STL1, ירוק).
Discussion
נכון להיום, דגים כבר שיטת תפוקה נמוכה בעיקר. השימוש בCy3, Cy3.5, וCy5 צבעים מגביל את מספר גנים אחד יכול לחקור בתאים בודדים עד שלוש בכל פעם. כמה בדיקות נוספות שפותחו (Stellaris), אך במספר הבדיקות להבחין עדיין ברוב שבע. כדי לעקוף מגבלה זו, אסטרטגיות תיוג קומבינטורית באמצעות fluorophores מרובים שמשו כדי ליצור ברקודים למיני mRNA שונים 19,20. לאחרונה, לובק וקאי משמשים Barcoding האופטי ולכמת רפאים 32 מינים שונים בו זמנית עם דגים בתאי שמרים רווקים בן 19. מגבלה אחת של גישה קומבינטורית האחרונה זו היא שהיא דורשת שימוש במיקרוסקופ סופר רזולוציה. הניתוח הדרוש כדי להבחין בין בדיקות המינימרקטים הוא גם די מורכב.
מצאנו כי Cy3 וCy3.5 עדיף על Cy5 לניסויי דגים. אחת המגבלות של הצבע Cy5 הוא הרגישות שלולphotobleaching. עם זאת, Stellaris פיתחה לאחרונה Cy5 ריאנטים המפורסמים כעמיד יותר בפני photobleaching, ועשוי להקל על הבעיה טכנית הזאת. כמו כן, ראוי לציין כי דגים היא שיטה יקרה ליישום והבדיקות שזינגר והן Stellaris בדרך כלל עולות $ 700 - $ 1000 לכל סט בדיקה, אם כי מחירים עבור בדיקות זמינות מסחרי צריכים להקטין בעתיד. חוסך של חומרים כימיים ותיוג יעיל מביא בדיקות זינגר עד לטווח נמוך יותר במחיר.
אחד האתגרים הטכניים העיקריים היא ההפרדה של נקודות בודדות לעומת בדיקה מרובות, אשר דורשת יישום של אלגוריתמים מתוחכמים הקובעים את המקום. זה יכול לקחת בדיקה ידנית נרחבת לכוון פרמטרים ניתוח תמונה למערכי ניסוי ספציפיים. מתווה של הצינור החישובית שלנו עם פונקציות MATLAB רלוונטיות היא אמורה בסעיף 7 לפרוטוקול. בעיה זו היא להקל במידה מסוימת על ידי בדיקות Stellaris שבו יש רק תווית אחת לבדיקה. לפיכך דורש colocalization של בדיקות מרובות כדי לראות את אות.
כי דגים מחייבים תיקון תאים, זה לא להקל על תאים בודדים מעקב לאורך זמן. בעבר, השתמשנו בנתוני תמונת מצב דגים כדי לשחזר את הדינמיקה של ביטוי גנים בשמרי אוכלוסיות אופניים מטבולית בודדות 6. רכיבה על אופניים מטבולי הוא ציין בתרבויות טרום מורעבות, רציפות, ומאופיין בתנודות קולקטיביות אוכלוסייה רחבה בצריכת חמצן. תנודות אלו קשורים עם תנודות הגנום של תמלילים המתרחשים למחצית מכל הגנים השמרים בשלבים של צריכת חמצן שונים. אנחנו ביקש לקבוע אם רכיבה על אופניים מטבולי היו קיימת בתרבויות שמרים הרציפות unsynchronized. אם קיים, תמלילים, כי הם אנטי קורלציה באוכלוסיות סינכרוני צריכים גם להיות אנטי מתואמים בunsynchronized תאים בודדים, ולהיפך לתעתיקי קורלציה.
אף אוזן גרון "> כדי לשחזר את הדינמיקה של ייצור ה-mRNA בזמן, את נתוני תמונת המצב הנצפים חייבים להיות לעומת מה שמצפה ממודל של ההתנהגות הבסיסית. ישנן מגבלות תיאורטיות לכאשר כאלה" צלמו "נתוני ביטוי גנים יכול לשמש כדי לקבוע ניתן להבחין בדינמיקת ביטוי גנים המשמשת כבסיס ואילו סוגים של דגמים 21. לנתוני מחזור חילוף חומרים, ולא ישירות מראה נוכחות של תנודות זמניות, מדידות סטטיסטיות יושמו על מנת לבסס כי אכן תא תכנית תנודתית אוטונומית בקנה אחד עם microarray בתפזורת מדידות.Disclosures
המחברים מצהירים שאין להם אינטרסים כלכליים מתחרים.
Acknowledgements
מחקר זה נתמך על ידי מענקים לGM046406 (DB) ועל ידי המכון הלאומי למדעי רפואה כללי מרכז לביולוגיה כמותית (GM071508). RSM מכיר במימון מNSF בוגר מחקר המלגה. MNM נתמך על ידי מלגת לואיס-סיגלר. ברצוננו להודות לחברים במעבדה בוטשטיין לדיונים מועילים ולשעבר חברי אלגרה פטי וניקולאי Slavov על תרומתם לפרויקט מחזור חילוף חומרים. אנו מודים לדניאל וZenklusen רוברט זינגר להשגתנו התחילה עם שיטת FISH.
Materials
| Name | Company | Catalog Number | Comments |
| Vanadyl Ribonucleoside Complex | NEB | S1402S | |
| Lyticase | Sigma | L5263 | |
| E. coli tRNA | Roche | 1010954001 | |
| BSA (RNase free) | Ambion | ||
| Beta-mercapt–thanol | Fisher | 03446l | |
| DAPI, dilactate | Sigma | D9564 | |
| PBS 10X (RNase free) | Ambion | AM9624 | |
| Triton X-100 | Shelton Scientific | ||
| Dextran sulfate | Sigma | D6001 | Or equivalent |
| Saline-sodium citrate (SSC) 20X | VWR | 82021-484 | |
| Formamide (deionized) | Ambion | AM9342 | |
| Nuclease-free water | Ambion | AM9932 | |
| Alpha-D-glucose | Sigma | 158968 | For GLOX solution |
| 1 M Tris-HCl, pH 8.0 | Ambion | AM9855G | |
| 100% Ethanol | |||
| Glucose oxidase | Sigma | G0543 | For GLOX solution |
| Catalase | Sigma | C3155 | For GLOX solution |
| Concanavalin A | MP Biomedicals | 150710 | |
| Polylysine (0.01%) | Sigma | P8920 | |
| Coverslips | Warner Instruments | Cs-18R15 | |
| Prolong Gold Mounting Medium | Invitrogen | P36934 | |
| QIAquick Nucleotide Removal Kit | QIAGEN | 28304 | |
| FISH Probes | Biosearch Technologies | Custom order for your desired mRNA sequence | |
| Glass bottom 96-well plates | Nunc | 265300 | Alternative to coverslips |
| 12-well plates | BD Falcon | 351143 | |
| Cy3, Cy3.5, Cy5 dyes | GE Healthcare | monofunctional NHS-ester | |
| EQUIPMENT | |||
| Plasma-Preen I Cleaner | Terra Universal | 9505-00 | Controller (Cat #9505-17 optional) |
| Vacuum Pump | Alcatel | 205SDMLAM | For operating Plasma-Preen |
| Widefield Fluorescence Microscope | Olympus | IX81 | Or equivalent |
| 100X objective | Olympus | 1-UB617R | |
| Light Source | X-Cite | XCT 10-A | Or equivalent |
| Filter Sets | Chroma | U-NSP100V2-SPR, U-NSP101V2-SPR, U-NSP102V2-SPR, U-NSP103V2-SPR,U-NSP104V2-SPR. | |
| Cooled CCD or EMCCD Camera | Hamamatsu | C4742-98-24ER | |
References
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of single RNA transcripts in situ. Science. 280, 585-590 (1998).
- Gall, J. G., Pardue, M. L. Formation and Detection of Rna-DNA Hybrid Molecules in Cytological Preparations. Proceedings of the National Academy of Sciences of the United States of America. 63, 378 (1969).
- Jones, K. W. Chromosomal and Nuclear Location of Mouse Satellite DNA in Individual Cells. Nature. 225, 912 (1970).
- Bauman, J. G., Wiegant, J., Borst, P., van Duijn, P. A new method for fluorescence microscopical localization of specific DNA sequences by in situ hybridization of fluorochromelabelled RNA. Exp Cell Res. 128, 485-490 (1980).
- Singer, R. H., Ward, D. C. Actin gene expression visualized in chicken muscle tissue culture by using in situ hybridization with a biotinated nucleotide analog. Proc. Natl. Acad. Sci. U.S.A. 79, 7331-7335 (1982).
- Silverman, S. J., et al. Metabolic cycling in single yeast cells from unsynchronized steady-state populations limited on glucose or phosphate. Proc. Natl. Acad. Sci. U.S.A. 107, 6946-6951 (2010).
- Trcek, T., Larson, D. R., Moldon, A., Query, C. C., Singer, R. H. Single-molecule mRNA decay measurements reveal promoter- regulated mRNA stability in yeast. Cell. 147, 1484-1497 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Mol Cell. 2, 437-445 (1998).
- Gandhi, S. J., Zenklusen, D., Lionnet, T., Singer, R. H. Transcription of functionally related constitutive genes is not coordinated. Nat. Struct. Mol. Biol. 18, 27-34 (2011).
- Itzkovitz, S., et al. Single-molecule transcript counting of stem-cell markers in the mouse intestine. Nature Cell Biology. 14, 106-U193 (2012).
- Raj, A., Rifkin, S. A., Andersen, E., van Oudenaarden, A. Variability in gene expression underlies incomplete penetrance. Nature. 463, 913-U984 (2010).
- Raj, A., Tyagi, S. Detection of individual endogenous RNA transcripts in situ using multiple singly labeled probes. Methods Enzymol. 472 (10), 365-386 (2010).
- Trcek, T., et al. Single-mRNA counting using fluorescent in situ hybridization in budding yeast. Nature Protocols. 7, 408-419 (2012).
- Raj, A., vanden Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5, 877-879 (2008).
- Otsu, N. A Tlreshold Selection Method from Gray-Level Histograms. IEEE Transactions on Systems, Man and Cybernetics. 9, 62-66 (1979).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophys J. 82, 2775-2783 (2002).
- McIsaac, R. S., et al. Fast-acting and nearly gratuitous induction of gene expression and protein depletion in Saccharomyces cerevisiae. Molecular biology of the cell. 22, 4447-4459 (2011).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nature Methods. 9, 743-U159 (2012).
- Levsky, J. M., Shenoy, S. M., Pezo, R. C., Singer, R. H. Single-cell gene expression profiling. Science. 297, 836-840 (2002).
- Wyart, M., Botstein, D., Wingreen, N. S. Evaluating Gene Expression Dynamics Using Pairwise RNA FISH Data. Plos Computational Biology. 6, (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved