Method Article
可视化和分析的mRNA分子,使用荧光
摘要
本协议描述的实验过程进行荧光杂交(FISH)技术在单细胞单分子分辨率计数的mRNA。
摘要
荧光原位杂交(FISH)方法允许一个原生细胞环境检测核酸。在这里,我们提供了一个协议,使用鱼量化的单个酵母细胞中的基因数目。细胞可以生长在任何条件下的利益,然后固定和透水。随后,多个共轭的荧光染料的单链脱氧寡核苷酸被用于标记和可视化的mRNA。从单个mRNA分子的衍射极限的荧光定量使用点检测算法,识别和计算每个细胞的基因数量。虽然北部印迹,RT-PCR和基因表达微阵列提供了更标准的量化方法在大量人口的平均的mRNA上的信息,FISH有利于这些mRNA的计数和定位在单细胞单分子分解。
引言
使用批量的测量技术,它是无法测定的转录或转录活性的数量,单电池1内。使用发起人的利益驱动,记者对基因表达的荧光蛋白可以在一定程度上解决这个问题,,但是荧光蛋白折叠所需要的时间掩盖早期动力学。长寿命的荧光蛋白质也不能报告mRNA的寿命。 FISH方法可用于检测mRNA的在其整个生命周期中,在细胞核中的转录起始,随后以单核细胞的成熟和衰减,与单分子分解。
原来在原位实验的可视化核酸用放射性标记的RNA探针,来探测DNA元素。其中包括可视化核糖体DNA在卵巢的青蛙非洲爪蟾和卫星DNA在小鼠组织3。第一荧光原位的experiment用荧光团标记的探测特定的DNA序列4的RNA分子。可视化的RNA 原位荧光探针的首次应用是在肌动蛋白基因表达的可视化鸡肌肉组织培养5。最近,在芽殖酵母,鱼已经被用于研究转录的振荡在酵母代谢周期6期间 ,衰减的mRNA在细胞周期进程7,和8在有丝分裂过程中的mRNA转录物的空间定位。 FISH已被用于在酵母中的组成型转录的基因,从而构成一半以上的所有的酵母基因,互不相关的波动产生的不相关的转录起始9。在非酵母物种,鱼类,已被用来识别在小鼠肠干细胞标记10,并决定细胞命运的不完全外显C.从随机的基因表达的波动可以导致线虫胚胎11。
对FISH这里描述的方法的工作原理是染料标记的单链DNA探针杂交的mRNA消息。细胞成像和基因使用抽查检测算法计算。可以产生的单链探针,用DNA合成仪,然后标记(这里所指的作为歌手探针)或命令市售的预标记的探针(斯特拉瑞斯探头)12,13。歌手的Stellaris探针之间的主要区别是,在歌手探针长度(〜50 bp)的多标记而拉吉等人所描述的每个探针,只有一个标签(〜20 bp)的与短的Stellaris探针14。此外,在Stellaris方法是使用更多的歌手(30比5〜每个基因探针),分别比每个基因探针。下面我们提供一种协议,它描述了使用两种不同的探针。在第2节中,我们提供了一个协议标签氨基烯丙基含胸苷探头瓦特第i个选定的CY染料。需要确定单基因点的计算步骤概述第7。
研究方案
图1和2的的FISH实验程序和用于量化FISH图像的图像分析管道的原理图。
1。准备的解决方案
*下面的解决办法是使用歌手探头。如果使用的Stellaris探头,更换"40%甲酰胺","10%甲酰胺"的杂交缓冲液和洗涤液。其他杂交缓冲液使用的Stellaris探头时:(1)加1克葡聚糖硫酸酯和(2)不包括10毫克单链DNA。
缓冲液B
"1 M8毫升KH 2 PO 4
41.5毫升1M K 2 HPO 4
109.3克山梨醇
Spheroplasting缓冲区
890μL缓冲液B
将100μl(VRC)
10微升25,000 U /毫升Lyticase的
2微升β-巯基乙醇
杂交缓冲液(10毫升,最终卷)
10毫克E.大肠杆菌的tRNA
10毫克单链*
100微升200毫米的VRC股票
40微升50毫克/毫升牛血清白蛋白
1毫升20X SSC
4毫升40%甲酰胺*
不含核酸酶的水(10毫升的最终体积)
1克葡聚糖硫酸酯*
*杂交缓冲液可以保持在0.5毫升分装,在-20℃下,以方便使用。
洗涤缓冲液(50毫升,最终卷)
5毫升20X SSC
20毫升40%甲酰胺*
不含核酸酶的水(50毫升的最终体积)
标记缓冲
1.06克钠碳酸
100毫升DEPC水
pH为9
2。探针标记(歌手探头)
我们获得这些探头由内部使用ABI寡核苷酸合成装置合成。炎热的典型盟友,4-5〜50 bp的寡核苷酸的合成是同源的感兴趣的基因,代烯丙基氨基间隔至少为8,优选为10 +沸点除了几个胸苷胸苷。由于其对臭氧的敏感度,我们的工作在使用时无臭氧设施CY染料。
- 获取〜5探头,悬浮在100μl水 - 检查浓度的NanoDrop。
- 根据多少探头/基因,共10微克寡核苷酸/基因结合( 例如 ,如果有5个探头/基因,然后想2微克/探针)。
- 使用QIAQUICK柱净化探针的核苷酸去除试剂盒QIAQUICK协议。
- 添加10卷缓冲PN总成交量组合探头和混合。
- 申请样品到QIAQUICK列 - 如果总量大于750微升,两次降速使用量的一半在每个旋
- 静置1分钟。
- 在6000转离心1分钟。
- 洗净用750微升缓冲区PE。
- 离心分离1分钟,以6,000 rpm。
- 处理流经重新旋柱1分钟13,000转干。
- 将QIAQUICK柱在新的离心管和洗脱DNA与50微升H 2 O -确保H 2 O pH值在7.0和8.5,是直接放在膜。
- 静置1分钟。
- 以13,000 rpm离心1分钟洗脱DNA。
- 冷冻干燥的DNA在45℃下
- 在10微升标记缓冲液和悬浮颗粒管染料,没有触摸染料。
- 重复与其他10微升标记缓冲。
- 涡管染料与DNA和自旋向下。
- 用铝箔盖管,并保持在黑暗RT O / N标签。
- 重复核苷酸移除套件QIAQUICK协议。有两个不同执行:
- 加入200微升PN缓冲探头和把标记探针通过列的2倍。
- 执行洗涤3次以独立的染料洗去洗脱前用缓冲液PE。 - AF洗脱后获得通过的NanoDrop浓度。标记率通常是〜0.25 pmol /纳克的单链DNA。
3。盖玻片准备
- 将盖玻片等离子体梳理的真空室( http://www.plasmapreen.com/ )(接近中心的更好)的幻灯片。
- 在微波和真空室,确保密封。
- 开启油泵,然后打开真空只有一次泵启动。
- 打开微波炉,停5秒后血浆可见。
- 然后关闭真空泵。
- 拉出的真空室,并用镊子除去盖玻片(下降需要再次清洁)。
- 的地方在12孔板中的盖玻片清洗侧。
4。固定程序
- 在基本培养基中生长的酵母的OD 600为0.1-0.2左右。 10毫升的细胞产生足够的FOR〜10个独立的杂交。
- 加入1/10体积的37%的甲醛直接生长介质(文化+ 10毫升1毫升37%的甲醛),让坐在45分钟。
- 洗2倍与1毫升冰冷的缓冲液B(1分钟可以旋转在13,000 rpm)离心管中。
- 添加1毫升spheroplasting缓冲区。
- 在37°C孵育15分钟。检查细胞每隔几分钟,直到大部分细胞在显微镜下是黑色的( 即不相亮)。
- 洗净用冰冷的缓冲液B的2倍,在低转速(每分钟3500转)旋转。
- 加入1毫升70%乙醇,轻轻重悬,并留下过夜,4°C(可以无限期地储存在-20°C)。
5。杂交过程
- 准备杂交液:100μl的杂交缓冲液中,加入1-3微升探针,然后涡流离心。歌手探头使用8-10纳克合计每一套探针( 即每个基因)。我们已经成像的同时与3个不同的3个基因ferent探针集。
可以肯定地暖杂交液至室温后再打开它。
对于Stellaris探头的,它是建议启动4个独立的杂交反应,通过添加各1μl1:10,1:20,1:50和1:100稀释的探针,看看哪一个是最佳的。在杂交缓冲液稀释的Stellaris探头准备工作。
- 离心机(后面所有步骤; 3500转,5分钟)固定细胞( 例如 200微升),吸走乙醇。
- 轻轻悬浮在1毫升洗涤缓冲液包含相同的杂交缓冲液中甲酰胺为百分比。静置2-5分钟。
- 离心样品抽吸洗涤缓冲液,然后加入杂交液。孵育在黑暗中轻轻摇动,O / N,37°C。
注:以下程序申请/成像细胞盖玻片上。对于W灰化/成像细胞在96孔板,包括替代活性氧清除剂溶液看到http://www.biosearchtech.com/stellarisprotocols的。
- 第二天, 或提前 ,干净的(见程序)和治疗盖玻片加入150μl0.01%的聚-L-赖氨酸5分钟。吸,让干燥,洗用DH 2 O 3倍,让干。
- 在早晨,在样品中加入1毫升洗涤缓冲液,轻轻悬浮,离心,吸出,然后重悬在另1毫升的洗涤缓冲液和孵育在37℃下30分钟。
- 洗净2X SSC + 0.1%的Triton X-100在室温下15分钟摇床。
- 以安装介质的冰柜,允许它拿出来RT安装前。
- 洗净用1X SSC在室温下15分钟摇床。
- 稀释DAPI PBS(0.1微克/毫升决赛)和150微升悬浮细胞。
- 到清洁/聚-LYS-珍稀的地方解决方案泰德盖玻片上,在12孔板中,至少30分钟原状。
- 删除溶液(您可以将其放置在一个备用盖滑作为备份)和1毫升1X PBS洗3倍。
- 将3微升到幻灯片延长黄金的安装介质(Invitrogen公司P36934)。在同一时间做这一块,如果你有多个盖玻片,避免干燥的安装介质。
- 〜0.5毫升乙醇添加盖玻片12孔板,而持有用镊子取出盖玻片和空气干燥。
- 放置盖玻片单元侧上安装介质,并让变硬几个小时或O / N在黑暗中。
- 封边指甲油和进行成像。
6。细胞成像奥林巴斯IX-81倒置显微镜概述
- 对于图像采集我们利用Slidebook的(智能imaging.com)软件和100倍,1.45 NA,TIRFM油目标。对于串口GFP,DAPI,CY3,CY3.5和Cy5的成像:色度滤波器套(见试剂)的。
- 使用DAPI过滤器发现并关注细胞。
- 设置此作为参考点。
- 径的Z堆叠的参考点周围的图像的总距离是用0.2微米的尺寸(25面)5μM。重复每种染料通道( 即每个探头套)。
- 出口为16位TIFF图像分析。
7。图像分析概述(歌手探头)
下面我们提供的大纲计算鱼在MATLAB图像分析方法,我们使用的。有关MATLAB函数使用括号在右边。目前调整的算法和阈值歌手型探头的数据。使用的Stellaris式探针,将需要一些调整,尤其是最后的过滤工序(7.8)。
细胞识别15
- 独立的细胞从背景中使用全局阈值[上DAPI影像16 graythresh]。
- 确定原子核使用极大扩展功能[IM] extendedmax。
- 分部作为种子细胞利用细胞核的分水岭算法[分水岭]。
寻找点在每个荧光通道
- 执行高帽转型正常化背景和改善信噪比[BWMORPH]。
- 查找极大值,以确定每个点在焦点层(在z平面)[IMREGIONALMAX]。
- 过滤潜在的点使用的线性拟合模型的径向渐变。
的测量光斑强度和过滤单个与多个探头信号
- 安装一个二维高斯分布在先前确定的重点层,估计强度17点。
- 筛选出的薄弱点,用一个阈值(基于直方图)。歌手类型探头,这是很重要的,但是较少的Stellaris探头。
- 计算在每个单元格中的点。
结果
图3显示了典型的直方图计算FISH图像,用于确定存在于单个细胞的mRNA的数量。显微镜基于RNA定量的一个重要优势是可以得到成绩单的本地化信息。例如,我们使用FISH,以确定在单个细胞中的mRNA与可诱导的CBF1等位基因( 图4)。许多mRNA分子,因为在网站上的转录本,我们是在细胞核内能够识别的转录位点的存在和位置。
通过利用不同的染料来标记不同的基因表达,可以量化多个在相同的细胞中的mRNA种类。为了证明这一点,酵母α-因子和山梨糖醇的存在下温育细胞。 α-因子诱导FUS1的转录(Quasar670染料,红色)。的转录STL1(类星体570染料,绿色)是诱发细胞外渗透压增加( 图5)。 图4是一个示例图5是一个示例的Stellaris探针的FISH的FISH与歌手探针的。
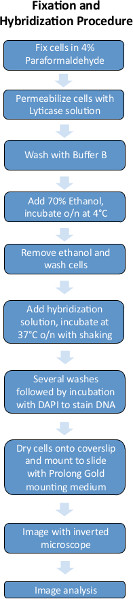
图1。鱼实 验过程示意图 点击此处查看大图 。

图2。图像分析管道的示意图 。最右边的图中确定的最终点。
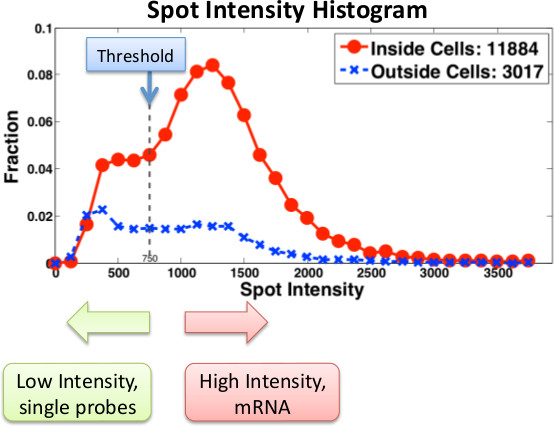
图3。现货强度的直方图为一个特定的基因歌手探头 。探头强度计算内外(红色)和外(蓝色)的细胞。低强度的斑点噪声或单探头。房地产mRNA的消息都标有多个探头。使用Stellaris探针的时,单探头检测到,因此必须作相应调整的阈值和滤波。
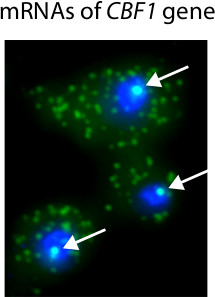
图4。代表歌手鱼过程的结果 。在这个实验中,CBF1转录激活与诱导型启动子18。细胞核呈蓝色用DAPI,,CBF1基因标签与Cy3标记的探针。白色箭头突出CBF1转录位点存在于细胞核内。单一的mRNA转录物是在细胞质中可见。
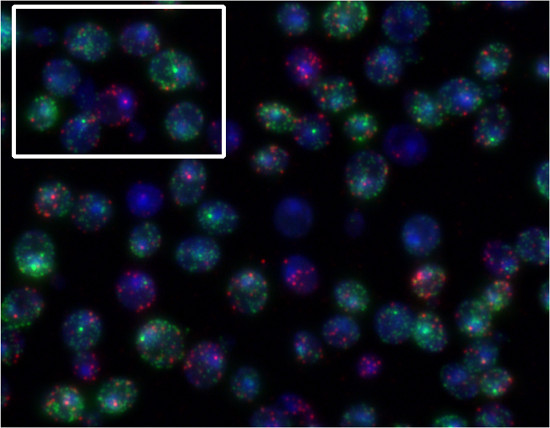
图5。代表性的结果的Stellaris FISH程序 。 MATa的酵母细胞,同时暴露至30毫微克/毫升的α-因子和0.75M的山梨糖醇10分钟,并同时为的FUS1(类星体670,红色)和STL1(类星体570,绿)的成绩单探测。在突出显示的框中,然后,我们可以看到只响应一个单元格的信息素(FUS1起始位点,红色)和其他主要响应山梨糖醇(STL1起始位点,绿色)。
讨论
至目前为止,鱼主要是低通量的方法。 Cy3标记,CY3.5,和Cy5染料的使用数限制在单个细胞中的基因,可以调查三个一时间。已经开发了一些额外的探针(斯特拉瑞斯),但仍然是可区分的探针数量至多为7。为了克服这个限制,使用多个荧光基团的组合的标记策略已被用于创建条形码的不同的mRNA物种19,20。最近,吕贝克和蔡用光学和光谱条形码单个酵母细胞FISH在19到32个不同的物种,同时量化。最近这一次的组合方法的一个限制是它需要使用超高分辨率显微镜。需要区分条形码探针分析也相当复杂。
我们已经发现,Cy3和CY3.5 Cy5的FISH实验优选。 Cy5染料的局限性之一是它的灵敏度光漂白。然而,最近的Stellaris开发的Cy5的变种,广告更耐漂白,可能缓解这一技术问题。还值得注意的是鱼是一种昂贵的方法来实现,而歌手的Stellaris探头通常花费$ 700 - $ 1,000每一套探针,虽然市售探头的价格应在未来减少。备用试剂和有效的标记带来歌手探头下降到较低的价格范围。
主要技术挑战之一是分离的单个与多个探针斑点,这需要实施复杂点确定算法。这可能需要大量的人工审核调整图像分析具体的实验设置的参数。提供该协议的第7条中有关MATLAB函数的计算管道的轮廓。由Stellaris探头只有这个问题有所缓解每一个标签探头。因此,它需要多个探头共存看到信号。
因为的FISH必须固定细胞,但不便于跟踪单个细胞随着时间的推移。以前,我们用FISH快照数据重建的动态中基因表达的个体的代谢循环的酵母群6。预饥饿,连续培养代谢循环中观察到的特点是广泛人口的集体振荡氧的消耗。这些振荡都与全基因组所有酵母基因的一半发生在不同阶段氧气消耗的成绩单振荡。我们寻求代谢循环,以确定是否存在不同步的连续酵母培养。如果存在,成绩单是反相关,在同步人群也应该是反相关的不同步的单细胞,反之亦然相关转录。
耳鼻喉科">为了重构动态mRNA的生产时间,所观察到的快照数据必须从相关的行为的模型预期相比有理论的限制时,基因表达数据的"快照"可以被用来确定相关基因表达的动态和种机型可以区分21。循环代谢数据,而不是直接显示时间振荡的存在,统计测量实施,以证明确实是有一个细胞自主的振荡程序符合散装芯片测量。披露声明
作者宣称,他们有没有竞争经济利益。
致谢
这项研究得到了补助GM046406(DB)和国立普通医学科学的定量生物学中心(GM071508)。 RSM承认从NSF的研究生研究奖学金资助。 MNM支持由刘易斯西格勒奖学金。我们希望,承认成员博茨泰因实验室的有益讨论和前成员阿莱格拉PETTI和尼古拉Slavov循环代谢工程所作出的贡献。我们感谢丹尼尔Zenklusen和罗伯特歌手让我们开始与鱼方法。
材料
| Name | Company | Catalog Number | Comments |
| Vanadyl Ribonucleoside Complex | NEB | S1402S | |
| Lyticase | Sigma | L5263 | |
| E. coli tRNA | Roche | 1010954001 | |
| BSA (RNase free) | Ambion | ||
| Beta-mercapt–thanol | Fisher | 03446l | |
| DAPI, dilactate | Sigma | D9564 | |
| PBS 10X (RNase free) | Ambion | AM9624 | |
| Triton X-100 | Shelton Scientific | ||
| Dextran sulfate | Sigma | D6001 | Or equivalent |
| Saline-sodium citrate (SSC) 20X | VWR | 82021-484 | |
| Formamide (deionized) | Ambion | AM9342 | |
| Nuclease-free water | Ambion | AM9932 | |
| Alpha-D-glucose | Sigma | 158968 | For GLOX solution |
| 1 M Tris-HCl, pH 8.0 | Ambion | AM9855G | |
| 100% Ethanol | |||
| Glucose oxidase | Sigma | G0543 | For GLOX solution |
| Catalase | Sigma | C3155 | For GLOX solution |
| Concanavalin A | MP Biomedicals | 150710 | |
| Polylysine (0.01%) | Sigma | P8920 | |
| Coverslips | Warner Instruments | Cs-18R15 | |
| Prolong Gold Mounting Medium | Invitrogen | P36934 | |
| QIAquick Nucleotide Removal Kit | QIAGEN | 28304 | |
| FISH Probes | Biosearch Technologies | Custom order for your desired mRNA sequence | |
| Glass bottom 96-well plates | Nunc | 265300 | Alternative to coverslips |
| 12-well plates | BD Falcon | 351143 | |
| Cy3, Cy3.5, Cy5 dyes | GE Healthcare | monofunctional NHS-ester | |
| EQUIPMENT | |||
| Plasma-Preen I Cleaner | Terra Universal | 9505-00 | Controller (Cat #9505-17 optional) |
| Vacuum Pump | Alcatel | 205SDMLAM | For operating Plasma-Preen |
| Widefield Fluorescence Microscope | Olympus | IX81 | Or equivalent |
| 100X objective | Olympus | 1-UB617R | |
| Light Source | X-Cite | XCT 10-A | Or equivalent |
| Filter Sets | Chroma | U-NSP100V2-SPR, U-NSP101V2-SPR, U-NSP102V2-SPR, U-NSP103V2-SPR,U-NSP104V2-SPR. | |
| Cooled CCD or EMCCD Camera | Hamamatsu | C4742-98-24ER | |
参考文献
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of single RNA transcripts in situ. Science. 280, 585-590 (1998).
- Gall, J. G., Pardue, M. L. Formation and Detection of Rna-DNA Hybrid Molecules in Cytological Preparations. Proceedings of the National Academy of Sciences of the United States of America. 63, 378 (1969).
- Jones, K. W. Chromosomal and Nuclear Location of Mouse Satellite DNA in Individual Cells. Nature. 225, 912 (1970).
- Bauman, J. G., Wiegant, J., Borst, P., van Duijn, P. A new method for fluorescence microscopical localization of specific DNA sequences by in situ hybridization of fluorochromelabelled RNA. Exp Cell Res. 128, 485-490 (1980).
- Singer, R. H., Ward, D. C. Actin gene expression visualized in chicken muscle tissue culture by using in situ hybridization with a biotinated nucleotide analog. Proc. Natl. Acad. Sci. U.S.A. 79, 7331-7335 (1982).
- Silverman, S. J., et al. Metabolic cycling in single yeast cells from unsynchronized steady-state populations limited on glucose or phosphate. Proc. Natl. Acad. Sci. U.S.A. 107, 6946-6951 (2010).
- Trcek, T., Larson, D. R., Moldon, A., Query, C. C., Singer, R. H. Single-molecule mRNA decay measurements reveal promoter- regulated mRNA stability in yeast. Cell. 147, 1484-1497 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Mol Cell. 2, 437-445 (1998).
- Gandhi, S. J., Zenklusen, D., Lionnet, T., Singer, R. H. Transcription of functionally related constitutive genes is not coordinated. Nat. Struct. Mol. Biol. 18, 27-34 (2011).
- Itzkovitz, S., et al. Single-molecule transcript counting of stem-cell markers in the mouse intestine. Nature Cell Biology. 14, 106-U193 (2012).
- Raj, A., Rifkin, S. A., Andersen, E., van Oudenaarden, A. Variability in gene expression underlies incomplete penetrance. Nature. 463, 913-U984 (2010).
- Raj, A., Tyagi, S. Detection of individual endogenous RNA transcripts in situ using multiple singly labeled probes. Methods Enzymol. 472 (10), 365-386 (2010).
- Trcek, T., et al. Single-mRNA counting using fluorescent in situ hybridization in budding yeast. Nature Protocols. 7, 408-419 (2012).
- Raj, A., vanden Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5, 877-879 (2008).
- Otsu, N. A Tlreshold Selection Method from Gray-Level Histograms. IEEE Transactions on Systems, Man and Cybernetics. 9, 62-66 (1979).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophys J. 82, 2775-2783 (2002).
- McIsaac, R. S., et al. Fast-acting and nearly gratuitous induction of gene expression and protein depletion in Saccharomyces cerevisiae. Molecular biology of the cell. 22, 4447-4459 (2011).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nature Methods. 9, 743-U159 (2012).
- Levsky, J. M., Shenoy, S. M., Pezo, R. C., Singer, R. H. Single-cell gene expression profiling. Science. 297, 836-840 (2002).
- Wyart, M., Botstein, D., Wingreen, N. S. Evaluating Gene Expression Dynamics Using Pairwise RNA FISH Data. Plos Computational Biology. 6, (2010).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。