Method Article
Visualisierung und Analyse von mRNA Moleküle mittels Fluoreszenz
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt ein experimentelles Verfahren für die Durchführung Fluorescence In situ Hybridisierung (FISH) zum Zählen mRNAs in einzelnen Zellen auf Einzel-Molekül-Auflösung.
Zusammenfassung
Die Fluoreszenz in situ Hybridisierung (FISH) erlaubt es, Nukleinsäuren in der nativen zellulären Umgebung zu erfassen. Hier bieten wir ein Protokoll für die Verwendung von FISH, um die Anzahl von mRNAs in einzelnen Hefezellen zu quantifizieren. Die Zellen können in einem Zustand von Interesse angebaut werden und dann fixiert und durchlässig gemacht. Anschließend werden mehrere einzelne Desoxyoligonukleotiden konjugiert verwendeten Fluoreszenzfarbstoffe zur Markierung und zu visualisieren mRNAs. Beugungsbegrenzte Fluoreszenz von einzelnen mRNA-Moleküle wird unter Verwendung eines Spot-Algorithmus zu identifizieren und zählen die Anzahl der mRNAs pro Zelle. Während die mehr Standard-Verfahren zur Quantifizierung von Northern Blots, RT-PCR und Genexpression-Microarrays Informationen über durchschnittliche mRNAs in der Bulk-Bevölkerung stellen, erleichtert FISH sowohl die Zählung und Lokalisation dieser mRNAs in einzelnen Zellen auf Einzel-Molekül-Auflösung.
Einleitung
Mit bulk Messtechniken, ist es nicht möglich, die Anzahl der Test-Transkripte oder transkriptionelle Aktivität in einzelnen Zellen 1. Mit fluoreszierenden Proteinen durch Promotoren von Interesse als Reporter der Genexpression angetrieben können dieses Problem in gewissem Umfang anzugehen, aber die Zeit für fluoreszierende Proteine zu falten erforderlich verdeckt frühen Dynamik. Langlebige fluoreszierende Proteine können auch nicht berichten mRNA Lebenszeiten. Die FISH-Methode kann zum Testen mRNA während seines gesamten Lebenszyklus genutzt werden, von Transkriptionsinitiation in den Zellkern, um nachfolgende Reifung und Zerfall in einzelne Zellen, mit Einzel-Molekül-Auflösung.
Das Original in situ Experimente zur Visualisierung von Nukleinsäuren verwendet radioaktiv markierte RNA-Sonden mit DNA-Elementen untersuchen. Dazu gehörten Visualisierung ribosomalen DNA in den Eierstöcken der Frosch Xenopus laevis 2 und Satelliten-DNA in Mausgewebe 3. Die erste Fluoreszenz in situ Knowführungsform verwendet ein RNA-Molekül mit einem Fluorophor markiert Sonde DNA-Sequenzen, insbesondere 4. Die erste Anwendung von fluoreszierenden Sonden zur Visualisierung RNA in situ war die Visualisierung von Aktin Genexpression in Hühnermuskel Gewebekulturen 5. In jüngerer Zeit in Hefezellen, FISH wurde verwendet, um Schwingungen in der Transkription zu untersuchen während der Hefe Stoffwechselkreislauf 6, der Zerfall der mRNAs während des Zellzyklus 7 und räumliche Lokalisierung der mRNA-Transkripte der Mitose 8. Fisch hat in Hefe verwendet worden, um zu zeigen, dass unkorrelierte Schwankungen konstitutiv transkribierten Gene, die mehr als die Hälfte aller Hefegenen bilden, von unkorrelierten Transkriptionsinitiation 9 entstehen. In Nicht-Hefe-Arten, hat FISH verwendet worden, um Stammzell-Marker in der Mausdarm 10 zu identifizieren und zu bestimmen, dass unvollständige Penetranz Zellschicksale kann von stochastischen Schwankungen Genexpression in C. führenelegans Embryonen 11.
Die FISH hier beschriebene Verfahren arbeitet durch Hybridisierung Farbstoff markierte, einzelsträngige DNA-Sonden, um Nachrichten mRNA. Zellen abgebildet werden und mRNAs gezählt mit einer Spot-Detektion Algorithmus. Einzelsträngige Sonden können mit einem DNA-Synthesizer erzeugt werden und dann mit (hier bezeichnet als Singer Sonden) oder bestellt kommerziell als pre-markierten Sonden (Stellaris Sonden) 12,13. Ein wesentlicher Unterschied zwischen dem Sänger und Stellaris Sonden ist, dass die Sänger Sonden mehr (~ 50 bp) und sind multi-markierten Sonden während die Stellaris (~ 20 bp) mit nur einem Etikett pro Sonde, wie Raj et al beschrieben sind kurz 14. Darüber hinaus verwendet der Stellaris Ansatz viele weitere Sonden pro Gen als die von Singer (~ 30 gegenüber 5 Sonden pro Gen, respectively). Nachfolgend wir ein Protokoll, das die Verwendung eines der Art der Sonde beschrieben. In Abschnitt 2 stellen wir ein Protokoll für die Kennzeichnung Amino-allyl Thymidin enthaltenden Sonden wit einem Farbstoff Cy gewählt. Eine Übersicht über die Rechenschritte erforderlich, um einzelne mRNA-Spots zu identifizieren ist in Abschnitt 7 vorgesehen.
Protokoll
Abbildungen 1 und 2 sind schematische Darstellungen der FISH experimentellen Verfahren und Bildanalyse zur Quantifizierung Pipeline FISH Bildern verwendet.
1. Lösungen bereiten
* Die folgenden Lösungen sind für den Einsatz mit Singer-Sonden. Bei Verwendung von Stellaris Sonden ersetzen "40% Formamid" mit "10% Formamid" sowohl Hybridisierung und Waschpuffer. Zusätzliche Änderungen an Hybridisierungspuffer bei Verwendung Stellaris Sonden sind (1) 1 g Dextransulfat und (2) nicht enthalten 10 mg ssDNA.
Puffer B
8 ml 1 M KH 2 PO 4
41,5 ml 1M K 2 HPO 4
109,3 g Sorbit
Spheroplasting Buffer
890 ul Puffer B
100 ul VRC
10 ul 25.000 U / ml Lyticase
2 ul β-Mercaptoethanol
Hybridisierungspuffer (10 ml, Endvolumen)
10 mg E. coli tRNA
10 mg ssDNA *
100 ul 200 mM VRC Lager
40 ul von 50 mg / ml BSA
1 ml 20X SSC
4 ml 40% Formamid *
Nuclease Free Water (bis 10 ml Endvolumen)
1 g Dextransulfat *
* Hybridisierungspuffer kann in 0,5 ml Aliquots bei -20 ° C werden der Einfachheit halber beibehalten.
Waschpuffer (50 ml, Endvolumen)
5 ml 20X SSC
20 ml 40% Formamid *
Nuclease Free Water (bis 50 ml Endvolumen)
Markierungspuffer
1,06 g Sodium Carbonate
100 ml DEPC Wasser
pH 9
2. Probe-Kennzeichnung (Singer Probes Only)
Wir erhalten diese Sonden durch in-house-Synthese unter Verwendung eines ABI Oligonucleotidsynthese Gerät. TypicAlly werden 4-5 ~ 50 bp Oligonukleotide synthetisiert, die homolog zu dem Gen von Interesse, substituierende Aminosäure-allyl Thymidin mehrere Thymidinen Abstand von mindestens 8, vorzugsweise 10 + bp auseinander. Aufgrund ihrer Empfindlichkeit gegenüber Ozon, arbeiten wir in einer Ozon-Anlage kostenlos bei der Verwendung von CY Farbstoffe.
- Erhalten ~ 5 Sonden und resuspendieren in 100 ul Wasser - check Konzentrationen auf Nanodrop.
- Je nachdem, wie viele Sonden / Gen verbinden insgesamt 10 ug Oligonukleotide / Gen (zB wenn haben 5 Sonden / Gen wollen, dann 2 ug / Sonde).
- Verwenden QIAquick Spalten Sonden nach QIAquick Nucleotide Removal Kit Protocol reinigen.
- In 10 Bänden Buffer PN, um die Lautstärke des kombinierten Sonden und Mix insgesamt.
- Bewerben Probe QIAquick Säule - wenn Gesamtvolumen größer als 750 ul, Spin-down zweimal mit der Hälfte des Volumens in jedem Spin
- Lassen Sie stehen für 1 min.
- Zentrifuge 1 min bei 6.000 Umdrehungen pro Minute.
- Waschen mit 750 ul Puffer PE.
- Zentrifuge1 min bei 6.000 Umdrehungen pro Minute.
- Entsorgen Durchströmung und Re-Spin-Säule für 1 min bei 13.000 rpm zu trocknen.
- Platz QIAquick Säule in neue Mikrozentrifugenröhrchens und eluieren DNA mit 50 ul H 2 O - Stellen Sie sicher, H 2 O pH innerhalb 7,0 und 8,5 und liegt direkt am Membran platziert.
- Lassen Sie stehen für 1 min.
- Säule für 1 min bei 13.000 rpm, um DNA zu eluieren.
- Lyophilisiert DNA bei 45 ° C
- Pellet in 10 ul Markierungspuffer und Rohr von Farbstoff hinzuzufügen, ohne den Farbstoff.
- Wiederholen Sie mit einem anderen 10 ul Markierungspuffer.
- Vortex und Spin-Down Tube Farbstoff und DNA.
- Röhrchen abdecken mit Alufolie aus und halten in Dunkeln bei RT O / N zu bezeichnen.
- Wiederholen QIAquick Nucleotide Removal Kit Protocol. Führen Sie mit zwei Unterschieden:
- In 200 ul Puffer PN, Sonden und legte markierten Sonden durch Spalten 2x.
- Führen Sie 3 Wäschen mit Puffer PE, um abzuwaschen ungebunden Farbstoff vor der Elution. - After Elution erhalten Konzentration über nanodrop. Die Kennzeichnung Effizienz ist in der Regel ~ 0,25 pmol / ng einzelner DNA.
3. Coverslip Vorbereitung
- Platz Deckgläser auf Objektträger in Plasma-putzen Vakuumkammer ( http://www.plasmapreen.com/ ) (die näher am Zentrum, desto besser).
- Setzen Vakuumkammer in Mikrowelle und sicherstellen, dass es verschlossen ist.
- Schalten Sie Pumpe zuerst, dann auf Vakuum nur einmal drehen Pumpe gestartet wird.
- Schalten Sie Mikrowelle und stoppen 5 sec nach Plasma sichtbar ist.
- Schalten Sie dann Vakuum pumpen.
- Ziehen Sie Vakuumkammer und entfernen Deckgläser mit einer Pinzette (diejenigen, die gefallen muss wieder gereinigt werden).
- Platz Deckgläser gereinigt Seite nach oben in 12-Well-Platten.
4. Fixation Vorgehensweise
- Wachsen Hefe bis zu einer OD 600 von ca. 0,1-0,2 in minimal Medien. 10 ml von Zellen liefert genug for ~ 10 separate Hybridisierungen.
- 1/10 Volumen 37% Formaldehyd direkt an den Wachstums-Medien (10 ml Kultur + 1 ml 37% Formaldehyd) und lassen Sie sich für 45 min.
- Waschen Sie 2x mit 1 ml eiskaltem Puffer B in einem Mikrozentrifugenröhrchen (kann bei 13.000 rpm für 1 min drehen).
- 1 ml Spheroplasting Puffer.
- Bei 37 ° C für 15 min. Prüfen Zellen alle paar Minuten unter dem Mikroskop, bis die meisten Zellen schwarz (dh nicht-Phase hell) sind.
- 2x Waschen mit eiskaltem Puffer B, Schleudern bei niedriger Geschwindigkeit (~ 3.500 rpm).
- 1 ml 70% EtOH, resuspendieren vorsichtig und lassen Sie über Nacht bei 4 ° C (unbegrenzt bei -20 ° C).
5. Hybridisierung Vorgehensweise
- Bereiten Sie die Hybridisierungslösung: zu 100 ul Hybridisierungspuffer, in der Regel 1-3 ul Sonde, dann Wirbel und Zentrifuge. Singer Sonden verwenden 8-10 ng Gesamt pro Sonde Set (dh pro Gen). Wir haben bis zu 3 Gene gleichzeitig mit 3 ver abgebildetschiedenen markierten Sonde setzt.
Achten Sie darauf, die Hybridisierung Lösung auf Raumtemperatur erwärmen, bevor Sie es öffnen.
Für Stellaris Sonden, ist es empfehlenswert, 4 separate Hybridisierungsreaktionen durch Zugabe von 1 ul jedes von 1:10, 1.20, 1:50 und 1:100 Verdünnungen von Sonden arbeiten, um zu sehen, welche optimal ist gestartet. Arbeiten Verdünnungen von Stellaris Sonden in Hybridisierungspuffer vorbereitet.
- Zentrifuge (für alle folgenden Schritte, 3.500 rpm, 5 min) die fixierten Zellen (z. B. 200 ul) und saugen das Ethanol entfernt.
- Vorsichtig in 1 ml Waschpuffer, die den gleichen Prozentsatz Formamid als Hybridisierungspuffer enthält resuspendieren. Lassen Sie stehen für 2-5 min.
- Zentrifuge Probe und absaugen Waschpuffer, dann fügen Hybridisierungslösung. Im Dunkeln unter leichtem Schütteln, O / N bei 37 ° C
Hinweis: Die folgende Prozedur ist für die Anwendung / Bildgebung von Zellen auf Deckgläsern. Für wVeraschen / Bildgebung von Zellen in 96-Well-Platten, einschließlich anderer reaktiver Sauerstoffspezies Fängerlösung siehe http://www.biosearchtech.com/stellarisprotocols.
- Am nächsten Tag, oder im Voraus, sauber (siehe Arbeitsgang) einsetzen und behandeln Deckgläschen mit 150 ul 0,01% Poly-L-Lysin für 5 min. Absaugen, trocknen lassen, 3x mit dH 2 O und trocknen lassen.
- In der Früh, 1 ml Waschpuffer auf die Probe, sanft resuspendieren, Zentrifuge und absaugen, dann in einem anderen 1 ml Waschpuffer und Inkubation bei 37 ° C für 30 min resuspendieren.
- Waschen mit 2X SSC + 0,1% Triton X-100 bei RT 15 min auf Schüttler.
- Nehmen Sie den Einbau Medium aus der Tiefkühltruhe, damit sie kommen bis RT vor der Montage.
- Waschen mit 1X SSC bei RT 15 min auf Schüttler.
- Verdünnen DAPI in PBS (0,1 ug / ml final) und Zellen in 150 ul.
- Platz Lösung auf gereinigt / Poly-lys-TREAten Deckgläschen in 12-Well-Platte, mindestens 30 min ungestört.
- Lösung entfernen (Sie können es auf einem Ersatz-Deckglas als Backup zu platzieren) und waschen mit 1 ml 1X PBS 3x.
- Platz 3 ul verlängern Goldmontageecken Medium auf einem Objektträger (Invitrogen P36934). Tun Sie dies ein zu einer Zeit, wenn Sie mehrere Deckgläser zu vermeiden Trocknen des Eindeckmediums haben.
- In ~ 0,5 ml Ethanol in 12-Well-Platte Deckglas, entfernen Sie das Deckglas und der Luft trocknen lassen, während mit einer Pinzette.
- Zeigen Deckglas Zell-Seite nach unten auf Eindeckmediums und lassen verhärten mehrere Stunden oder O / N in der Dunkelheit.
- Seal die Ränder mit Nagellack und fahren Bildgebung.
6. Imaging von Zellen mit Olympus IX-81 Inverses Mikroskop Übersicht
- Für die Bildaufnahme nutzen wir Slidebook (intelligent-imaging.com) Software und ein 100X, 1,45 NA, TIRFM Öl-Objektiv. Für die serielle GFP, DAPI, Cy3, Cy5 und Cy3.5 imaging: Chroma-Filter-Sets (siehe Reagenzien).
- Verwenden DAPI-Filterzu finden und zu konzentrieren Zellen.
- Setzen Sie diese als Bezugspunkt.
- Werfen Sie einen z-Stapel von Bildern rund um den Bezugspunkt, wo die Gesamtstrecke beträgt 5 uM mit 0,2 uM Größe (25 Flugzeuge). Wiederholen Sie dies für jeden Farbstoff Kanal (dh jede Sonde set).
- Export als 16-Bit-TIFF für die Bildanalyse.
7. Bildanalyse Übersicht (Singer Probes)
Im Folgenden geben wir einen Überblick über Berechnungsmethoden verwenden wir zur Analyse FISH Bilder in MATLAB. Die relevanten MATLAB-Funktionen verwendet werden, auf der rechten eingeklammert. Die Algorithmen und Schwellenwerte sind derzeit für Daten aus Singer-Sonden abgestimmt. Mit Stellaris-Sonden erfordert einige Anpassungen, insbesondere auf die endgültige Filterung Schritt (7.8).
Zellidentifikation 15
- Separate Zellen aus dem Hintergrund mit einem globalen Schwellwert auf DAPI Bilder 16 [graythresh].
- Identifizieren Kerne mit erweiterten Maxima-Funktion [imextendedmax].
- Segment Zellen mit Kernen als Keime für eine Wasserscheide-Algorithmus [Wendepunkt].
Finden Sie wichtige Punkte in jeder Fluoreszenzkanal
- Führen tophat Transformation Hintergrund normalisieren und verbessern Signal-Rausch-Verhältnis [bwmorph].
- Finden Maxima, die In-Fokus-Schicht (in der z-Ebene) für jeden Punkt zu identifizieren [imregionalmax].
- Filtern Potenzial Flecken mit einem linearen Modell fit zu einem radialen Farbverlauf.
Measure Spotintensität und Filter einzigen vs mehrere Sondensignale
- Fit ein 2D-Gauß-Profil an die Stelle in der zuvor identifizierten In-Fokus-Schicht und Schätzung Intensität 17.
- Filtern Sie Schwachstellen mit einer Schwelle (basierend auf Histogramm). Für Singer-Sonden das ist wichtig, aber es ist weniger für Stellaris Sonden.
- Graf Flecken in jeder Zelle.
Ergebnisse
Abbildung 3 zeigt typische Histogramme von FISH Bilder berechnet und verwendet, um die Anzahl der mRNAs in einzelnen Zellen zu bestimmen. Ein wichtiger Vorteil der Mikroskopie-basierte RNA Quantifizierung ist, dass man Informationen über die Lokalisation der Transkripte zu erhalten. Zum Beispiel haben wir FISH auf mRNAs in einzelnen Zellen mit einem induzierbaren CBF1 Allel (Abbildung 4) zu identifizieren. Da viele mRNA Moleküle am Ort der Transkription sind, sind wir in der Lage, das Vorhandensein und die Lage der Transkription Stellen innerhalb des Kerns zu identifizieren.
Durch die Verwendung unterschiedlicher Farbstoffe zur Kennzeichnung mRNAs von verschiedenen Genen, kann man mehrere mRNA-Spezies in den gleichen Zellen zu quantifizieren. Um dies zu demonstrieren, wurden Hefe-Zellen in Gegenwart von α-Faktor und Sorbitol inkubiert. FUS1 Transkription (Quasar670 Farbstoff, rot) wird durch α-Faktor induziert. STL1 Transkription (Quasar 570 Farbstoff, grün) wird durch Erhöhung der extrazellulären Osmolarität induziert ( Abbildung 5). Abbildung 4 ist ein Beispiel von FISH-Sonden mit den Singer. Abbildung 5 ist ein Beispiel von FISH-Sonden mit den Stellaris.
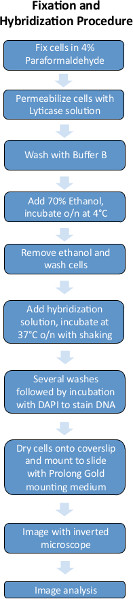
Abbildung 1. Schematische Darstellung der FISH experimentelle Verfahren. Klicke hier, um eine größere Abbildung anzuzeigen .

Abbildung 2. Schematische Darstellung der Bildanalyse-Pipeline. Die letzten Punkte werden in der rechten Figur bestimmt.
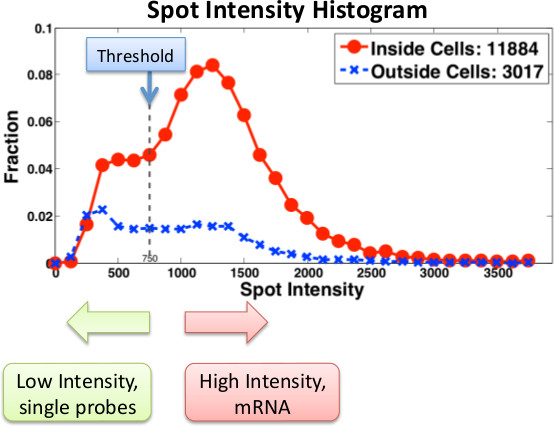
Abbildung 3. Histogramm Spotintensitäten für ein bestimmtes Gen mit Singer Sonden . Probe Intensitäten sowohl innerhalb (rot) und außerhalb (blau) Zellen berechnet. Niedrige Intensität Flecken sind entweder Rauschen oder einzelne Sonden. Echter mRNA Nachrichten werden mit mehreren Sonden markiert. Bei der Verwendung von Stellaris-Sonden sind Sonden einzelne weniger nachweisbar und damit die Schwellen und die Filterung entsprechend angepasst werden müssen.
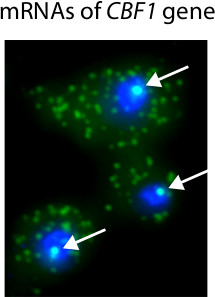
Abbildung 4. Repräsentative Ergebnisse von Singer FISH-Verfahren. In diesem Experiment wird CBF1 Transkription mit einem induzierbaren Promotor 18 aktiviert. Zellkerne sind blau angefärbt mit DAPI. CBF1 mRNAs mit Cy3-markierten Sonden sind verschlagwortet. Weiße Pfeile markieren die Anwesenheit von CBF1 Transkription Websites in den Zellkern. Einzel-mRNA-Transkripte sind im Zytoplasma.
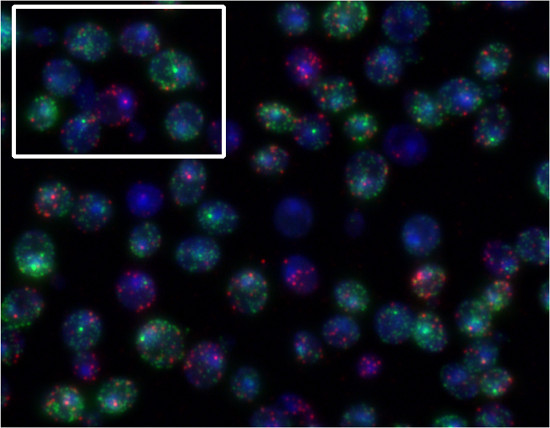
Abbildung 5. Repräsentative Ergebnisse von Stellaris FISH-Verfahren. MATa Hefezellen wurden gleichzeitig 30 ng / ml α-Faktor und 0,75 M Sorbitol für 10 Minuten ausgesetzt und gleichzeitig sondiert FUS1 (Quasar 670, rot) und STL1 (Quasar 570, grün) Transkripte. In dem markierten Feld können wir sehen, eine Zelle reagiert nur auf das Pheromon (FUS1 Startstelle, rot) und ein anderer überwiegend Reaktion auf Sorbit (STL1 Startstelle, grün).
Diskussion
Bis heute hat FISH in erster Linie ein Low-Durchsatz-Methode. Die Verwendung von Cy3, Cy3.5, Cy5 Farbstoffen und begrenzt die Anzahl der Gene in einem einzelnen Zellen zu drei untersuchen können zu einem Zeitpunkt. Einige zusätzliche Sonden entwickelt (Stellaris), aber die Anzahl der unterscheidbaren Sonden noch höchstens sieben. Um diese Einschränkung zu umgehen, wurden kombinatorische Kennzeichnung Strategien mit mehreren Fluorophoren verwendet worden, um Barcodes für verschiedene mRNA-Spezies 19,20 erstellen. Zuletzt verwendet Lübeck und Cai optische und spektrale barcoding zu 32 verschiedene Arten gleichzeitig zu quantifizieren mit FISH in einzelnen Hefezellen 19. Eine Einschränkung dieser jüngsten kombinatorischen Ansatzes ist es erfordert die Verwendung von Super-Resolution-Mikroskopie. Die Analyse erforderlich, um die Barcode-Sonden unterscheiden ist auch sehr komplex.
Wir haben festgestellt, dass Cy3 und Cy5 Cy3.5 vorzuziehen, für FISH Experimente sind. Eine der Einschränkungen des Cy5-Farbstoff ist seine Empfindlichkeitzum Bleichen. Allerdings hat kürzlich Stellaris Cy5 Varianten, die als resistenter gegen Ausbleichen beworben werden entwickelt und können diese technische Frage zu lindern. Es auch erwähnenswert, dass Fisch ist ein teures Verfahren zu implementieren und dass sowohl Sänger und Stellaris Sonden in der Regel kosten $ 700 - $ 1.000 pro Sonde gesetzt, obwohl die Preise für kommerziell erhältliche Sonden in der Zukunft verringern sollte. Sparing von Reagenzien und effiziente Markierung bringt Singer Sonden bis in den unteren Bereich im Preis.
Eines der größten technischen Herausforderungen ist die Trennung von einzelnen gegen mehrere Sonde Flecken, die die Umsetzung von anspruchsvollen spot-Bestimmung Algorithmen erfordert. Dies kann umfangreiche manuelle Überprüfung der Bildanalyse Parameter für bestimmte Versuchsanordnungen abzustimmen. Ein Überblick über unsere Computational Pipeline mit relevanten MATLAB-Funktionen wird in Abschnitt 7 des Protokolls zur Verfügung gestellt. Dieses Problem wird durch die Stellaris etwas Sonden, die nur gelindert ein Etikett pro Sonde. Es erfordert daher die Kolokalisation von mehreren Sonden, um ein Signal zu sehen.
Da FISH erfordert Festsetzung Zellen, es nicht ermöglichen Tracking einzelnen Zellen über die Zeit. Früher verwendeten wir FISH Snapshot-Daten, um die Dynamik der Genexpression in einzelnen metabolisch Radfahren Hefepopulationen 6 rekonstruieren. Metabolic Radfahren wird in Pre-verhungert, kontinuierliche Kulturen beobachtet, und wird von der Bevölkerung-weiten kollektiven Schwingungen in Sauerstoffverbrauch gekennzeichnet. Diese Schwingungen werden mit Genom-weiten Schwingungen Transkripte, die für die Hälfte aller Hefegenen treten in verschiedenen Phasen der Sauerstoffverbrauch verbunden. Wir versuchten zu bestimmen, ob metabolische Radfahren war in synchronisierten kontinuierliche Hefekulturen. Falls vorhanden, sollten Transkripte, die in synchronen Bevölkerung anti-korreliert sind auch anti-korrelierte in einzelnen Zellen nicht synchronisiert, und umgekehrt für korrelierte Transkripte.
ent "> Zur Dynamik der mRNA-Produktion in der Zeit zu rekonstruieren, müssen die beobachteten Snapshot-Daten zu dem, was von einem Modell des zugrunde liegenden Verhalten erwartet verglichen werden. Es gibt theoretische Einschränkungen, wenn diese" Schnappschüsse "der Genexpression Daten können verwendet werden, um festzustellen, die zugrunde liegenden Genexpression Dynamik und die Arten von Modellen 21 unterschieden werden können. Für die metabolische Zyklus Daten, anstatt direkt zeigt das Vorhandensein von zeitlichen Oszillationen wurden statistische Messungen durchgeführt, um zu belegen, dass es tatsächlich eine Zelle autonomen oszillierende Programm mit Bulk-Microarray Messungen.Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen konkurrieren.
Danksagungen
Diese Forschung wurde unterstützt durch Zuschüsse GM046406 (DB) und dem National Institute of General Medical Sciences Center for Quantitative Biology (GM071508) unterstützt. RSM räumt Mittel aus dem NSF Graduate Research Fellowship. MNM wird durch eine Lewis-Sigler Fellowship unterstützt. Wir möchten den Mitgliedern des Botstein Labor für hilfreiche Diskussionen und der ehemaligen Mitglieder Allegra Petti und Nikolai Slavov für ihre Beiträge zur metabolischen Zyklus Projekt bestätigen. Wir danken Daniel Zenklusen und Robert Singer für uns immer begann mit der FISH-Methode.
Materialien
| Name | Company | Catalog Number | Comments |
| Vanadyl Ribonucleoside Complex | NEB | S1402S | |
| Lyticase | Sigma | L5263 | |
| E. coli tRNA | Roche | 1010954001 | |
| BSA (RNase free) | Ambion | ||
| Beta-mercapt–thanol | Fisher | 03446l | |
| DAPI, dilactate | Sigma | D9564 | |
| PBS 10X (RNase free) | Ambion | AM9624 | |
| Triton X-100 | Shelton Scientific | ||
| Dextran sulfate | Sigma | D6001 | Or equivalent |
| Saline-sodium citrate (SSC) 20X | VWR | 82021-484 | |
| Formamide (deionized) | Ambion | AM9342 | |
| Nuclease-free water | Ambion | AM9932 | |
| Alpha-D-glucose | Sigma | 158968 | For GLOX solution |
| 1 M Tris-HCl, pH 8.0 | Ambion | AM9855G | |
| 100% Ethanol | |||
| Glucose oxidase | Sigma | G0543 | For GLOX solution |
| Catalase | Sigma | C3155 | For GLOX solution |
| Concanavalin A | MP Biomedicals | 150710 | |
| Polylysine (0.01%) | Sigma | P8920 | |
| Coverslips | Warner Instruments | Cs-18R15 | |
| Prolong Gold Mounting Medium | Invitrogen | P36934 | |
| QIAquick Nucleotide Removal Kit | QIAGEN | 28304 | |
| FISH Probes | Biosearch Technologies | Custom order for your desired mRNA sequence | |
| Glass bottom 96-well plates | Nunc | 265300 | Alternative to coverslips |
| 12-well plates | BD Falcon | 351143 | |
| Cy3, Cy3.5, Cy5 dyes | GE Healthcare | monofunctional NHS-ester | |
| EQUIPMENT | |||
| Plasma-Preen I Cleaner | Terra Universal | 9505-00 | Controller (Cat #9505-17 optional) |
| Vacuum Pump | Alcatel | 205SDMLAM | For operating Plasma-Preen |
| Widefield Fluorescence Microscope | Olympus | IX81 | Or equivalent |
| 100X objective | Olympus | 1-UB617R | |
| Light Source | X-Cite | XCT 10-A | Or equivalent |
| Filter Sets | Chroma | U-NSP100V2-SPR, U-NSP101V2-SPR, U-NSP102V2-SPR, U-NSP103V2-SPR,U-NSP104V2-SPR. | |
| Cooled CCD or EMCCD Camera | Hamamatsu | C4742-98-24ER | |
Referenzen
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of single RNA transcripts in situ. Science. 280, 585-590 (1998).
- Gall, J. G., Pardue, M. L. Formation and Detection of Rna-DNA Hybrid Molecules in Cytological Preparations. Proceedings of the National Academy of Sciences of the United States of America. 63, 378 (1969).
- Jones, K. W. Chromosomal and Nuclear Location of Mouse Satellite DNA in Individual Cells. Nature. 225, 912 (1970).
- Bauman, J. G., Wiegant, J., Borst, P., van Duijn, P. A new method for fluorescence microscopical localization of specific DNA sequences by in situ hybridization of fluorochromelabelled RNA. Exp Cell Res. 128, 485-490 (1980).
- Singer, R. H., Ward, D. C. Actin gene expression visualized in chicken muscle tissue culture by using in situ hybridization with a biotinated nucleotide analog. Proc. Natl. Acad. Sci. U.S.A. 79, 7331-7335 (1982).
- Silverman, S. J., et al. Metabolic cycling in single yeast cells from unsynchronized steady-state populations limited on glucose or phosphate. Proc. Natl. Acad. Sci. U.S.A. 107, 6946-6951 (2010).
- Trcek, T., Larson, D. R., Moldon, A., Query, C. C., Singer, R. H. Single-molecule mRNA decay measurements reveal promoter- regulated mRNA stability in yeast. Cell. 147, 1484-1497 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Mol Cell. 2, 437-445 (1998).
- Gandhi, S. J., Zenklusen, D., Lionnet, T., Singer, R. H. Transcription of functionally related constitutive genes is not coordinated. Nat. Struct. Mol. Biol. 18, 27-34 (2011).
- Itzkovitz, S., et al. Single-molecule transcript counting of stem-cell markers in the mouse intestine. Nature Cell Biology. 14, 106-U193 (2012).
- Raj, A., Rifkin, S. A., Andersen, E., van Oudenaarden, A. Variability in gene expression underlies incomplete penetrance. Nature. 463, 913-U984 (2010).
- Raj, A., Tyagi, S. Detection of individual endogenous RNA transcripts in situ using multiple singly labeled probes. Methods Enzymol. 472 (10), 365-386 (2010).
- Trcek, T., et al. Single-mRNA counting using fluorescent in situ hybridization in budding yeast. Nature Protocols. 7, 408-419 (2012).
- Raj, A., vanden Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5, 877-879 (2008).
- Otsu, N. A Tlreshold Selection Method from Gray-Level Histograms. IEEE Transactions on Systems, Man and Cybernetics. 9, 62-66 (1979).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophys J. 82, 2775-2783 (2002).
- McIsaac, R. S., et al. Fast-acting and nearly gratuitous induction of gene expression and protein depletion in Saccharomyces cerevisiae. Molecular biology of the cell. 22, 4447-4459 (2011).
- Lubeck, E., Cai, L. Single-cell systems biology by super-resolution imaging and combinatorial labeling. Nature Methods. 9, 743-U159 (2012).
- Levsky, J. M., Shenoy, S. M., Pezo, R. C., Singer, R. H. Single-cell gene expression profiling. Science. 297, 836-840 (2002).
- Wyart, M., Botstein, D., Wingreen, N. S. Evaluating Gene Expression Dynamics Using Pairwise RNA FISH Data. Plos Computational Biology. 6, (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten