Method Article
מיזוג במונחים מוחלטים ובאופן יחסי PCR כמותי נתונים לכמת STAT3 אחוי Variant תעתיקים
In This Article
Summary
Tandem splicing events occur at sites less than 12 nucleotides apart. Quantifying ratios of such splice variants is feasible using an absolute quantitative PCR approach. This manuscript describes how splice variants of the gene STAT3, in which two splicing events results in Serine-701 inclusion/exclusion and α/β C-termini, can be quantified.
Abstract
Human signal transducer and activator of transcription 3 (STAT3) is one of many genes containing a tandem splicing site. Alternative donor splice sites 3 nucleotides apart result in either the inclusion (S) or exclusion (ΔS) of a single residue, Serine-701. Further downstream, splicing at a pair of alternative acceptor splice sites result in transcripts encoding either the 55 terminal residues of the transactivation domain (α) or a truncated transactivation domain with 7 unique residues (β). As outlined in this manuscript, measuring the proportions of STAT3's four spliced transcripts (Sα, Sβ, ΔSα and ΔSβ) was possible using absolute qPCR (quantitative polymerase chain reaction). The protocol therefore distinguishes and measures highly similar splice variants. Absolute qPCR makes use of calibrator plasmids and thus specificity of detection is not compromised for the sake of efficiency. The protocol necessitates primer validation and optimization of cycling parameters. A combination of absolute qPCR and efficiency-dependent relative qPCR of total STAT3 transcripts allowed a description of the fluctuations of STAT3 splice variants' levels in eosinophils treated with cytokines. The protocol also provided evidence of a co-splicing interdependence between the two STAT3 splicing events. The strategy based on a combination of the two qPCR techniques should be readily adaptable to investigation of co-splicing at other tandem splicing sites.
Introduction
לטווח קצר (טנדם) שחבור חלופי, שבו אתרי acceptor או תורם חלופיים נמצאים בסמיכות זה לזה, נפוץ אצל יונקים 1, חסרי חוליות 2 וצמחים 3. ההערכה היא כי 20% מהגנים יונקים מכילים אתרי השחבור אלטרנטיבה 2-12 נוקלאוטידים מזה 4. רבים מהאתרים הללו הם זה מזה 3 נוקלאוטידים ולגרום הכללה או אי הכללה של קודון יחיד. קיימת מחלוקת לגבי טבעו של רגולציה שחבור באתרים אלה 5,6 עם כמה להתווכח שההבדלים מוטיב שחבור שלנו כה עדין, כי הבחירה היא 7 סטוכסטיים, בעוד שאחרים להסיק רגולציה המבוססת על סגוליות רקמות 8.
מבחר אחוי אתר טנדם כבר נתח חצי כמותית באמצעות אלקטרופורזה נימים שונה 7, ו ג'ל אלקטרופורזה ברזולוציה גבוהה 8. RNA-seq (רצף RNA) קורא יכול לשמש כדי לכמת את יחסי שחבור בכל אחויאֲתַר. בדרך זו, נתוני RNA-seq ספקו תובנה הסדרת אתרי שחבור טנדם 9. זה גם אפשר חיזוי של יחסים וריאנט אחוי צפויים המבוססים על מוטיב נוקלאוטיד 10. רוב הדגש על שחבור הכולל או כולל קודון יחיד כבר על אתרי השחבור acceptor יותר נפוצה המתרחשים במקביל, המכונים NAGNAGs (כש- n = כל נוקלאוטיד).
תורם טנדם אתרי שחבור אלטרנטיביים כוללים או לא כוללים קודון יחיד (מוטיב הכרת GYNGYN, שבו Y = פירימידין) הם נפוצים פחות מאשר acceptors טנדם. מתמר אותות activator של שעתוק 3 (STAT3) הוא גן מפתח העוברים שחבור חלופי תורם טנדם 1,11. אתרי השחבור התורם טנדם להצטרף אקסונים 21 ו -22 ו לגרום הכללת או אי הכללת קודון עבור סרין-701 (S או ΔS בהתאמה) 1,11. אתרי acceptor אלטרנטיבה Downstream (למעט 40 נוקלאוטידים זה מזה) אקסונים הצטרפותו 22 ו23A / תוצאה ב ב הכללת 55 או שאריות מסוף של תחום transactivation (α) או תחום transactivation קטום עם 7 שאריות ייחודי C-terminal (β) 11. לכן, ארבע גרסות אחוי אפשריות.
חלבון STAT3 הוא גורם שעתוק אינטגרטור אות מרכזי סוגי תאים רבים 12 וכאשר מוטצית ההפעלה המכוננת שלה תורמת פנוטיפים סרטן כמה (הנסקרת תוך התייחסות 13). איוב תסמונת, הפרעת כשל חיסוני מאופיינת ברמות גבוהות של IgE, נגרמת גם על ידי מוטציות STAT3 (הנסקרת תוך התייחסות 14). תפקידים ברורים עבור α STAT3 וחלבוני גרסת β אחוי כבר שתוארו לעיל 15. בתחילה, STAT3 β היה נהוג לחשוב לפעול באופן דומיננטי שלילי 16, להכעיס פעילות התעתיק של α STAT3, אבל ופועלם הציע יש לו גני המטרה עצמאיים 17 . למרות התחכום של שחבור טנדם, יש סיבה להאמין היעדרותם ונוכחותם של סרין-701 (Ser701) השפעות פונקציה. לא זו בלבד Ser701 בסמיכות טירוזין-705 (שאריות פוספורילציה הפעלת STAT3 18), אך מחקר שנערך לאחרונה מצביע על כך STAT3 S ו- ΔS הגירסות אחוי הן הכרחיות כדאיות של לימפומה תאי B הגדול המפוזר מכורת STAT3 (DLBLCL) תאים 19. הרלוונטיות הביולוגית נשארה כדי להיחקר. בהתחשב בכך רכב גרסת אחוי יכול להשפיע על תפקוד, אנחנו השתדלנו לגלות אם היחס היה מוטרד על ידי גירוי ציטוקין אאוזינופילים.
אנחנו בתחילה ניסו לנתח את הקשר בין שני האירועים שחבור באמצעות ספציפי PCR עבור α STAT3 וגירסאות אחוי β, ואחריו המחשוף של מוצרים עם הגבלה אנזים מסוים עבור גרסאות אחוי S, AfeI. צפיפות של מוצרים מצוינים הכללת Ser701 היה roughly פי עשרה יותר נפוץ מאשר אי עשייתו (ΔS) בשני α STAT3 ו β (מידע לא מוצג). עם זאת, גישה וכמותיות זה לא היה לשחזור מספיק, ולא ניתן היה להשתמש ביעילות כדי למדוד את כל אחוי ארבע גרסאות בו זמנית. כדי לנתח פרופורציות של כל אחד הגירסות הארבע אחוי, היה צורך להקים פרוטוקול כמוני PCR (qPCR) שהניב (מבחנים כמה במדגם נתון) טכניים חזקים משכפל.
QPCR יחסית מסתמכת על השוואה של גן של עניין לגן רגיל או משק הידוע להתבטא ברמה מסוימת 20 ומתאים כאשר הגן של ריבית גן המשק הם מוגברים עם יעילות דומה. גדילי כפול (DS) ניאון ה- DNA מחייבת (cyanine) לצבוע נקשר amplicons PCR 21, ואחרי מספר מסוים של מחזורי, הגברה מספיק התרחשה על הקרינה כדי להתגלות. ככל שעולה הרמה הראשונית הגבוהה יותרלפרוטוקול, מחזור הסף הנמוך (CT) ערך. מאז הריכוז של הכנות cDNA שונה, צריך להשוות את הריכוז של התמליל עם הריכוז של תעתיק הידוע להתבטא ברמה עקבית בכל הדגימות, כמו glucuronidase-β (GUSB) ב אאוזינופילים 22.
qPCR יחסית היא לא ריאלית עבור רצפים דומים מאוד, כפי שניתן לראות גרסות אחוי נובע שחבור טנדם. התנאים המחמירים הנדרשים כדי להגביר את גרסאות אחוי במיוחד לגרום יעילות ירד. במקום זאת, כימות מוחלטת חייבת לשמש 23. לשם כך עליהם הכנת עקומת סטנדרט עם ריכוזים ידועים לפרוטוקול השחבור של עניין, והבטחת תנאי PCR לייעל סגולי 24. כפי שתואר, נתוני qPCR מוחלטים ויחסים עבור גן מסוים ניתן למזג להודיע הבנה של ביטוי הגנים בפיזור לסוג תא מסוים, בSTAT3 במקרה זה ב אאוזינופילים מגורה שונה 25.
בזאת, כימות גרסת אחוי STAT3 מתואר מתוך הציפייה כי השיטה ניתן להתאים מחקרים ממוקדים של אירועי שחבור טנדם אחרים. אופטימיזציה היה תהליך ממושך, שבו כמה זוגות פריימר בריכוזים שונים חזרות רבות של פרמטרי אופניים נבדקו במשך כמה חודשים. התכונות העיקריות של הפרוטוקול האלה מהוות הוכחה סגולית פריימר וכימות מבוסס על עקומות סטנדרטיות עם ריכוזים ידועים של וריאנטים אחוי. qPCR יחסית בשיתוף הוכיחה מועילה עבור היישום שלנו אבל אין צורך.
Protocol
הערה: אאוזינופילים דם היקפיים התקבלו ללא פרטים מזהים בהסכמה עם פרוטוקול שאושר (# 2013-1570) על ידי אוניברסיטת ויסקונסין-מדיסון המרכז למדעי הבריאות מוסדיים המועצה לביקורת. חתום הסכמה מדעת מן התורם הושגה לשימוש של כל דגימה במחקר.
1. יצירת פלסמידים כתקנים תבנית
- צור פריימרים עבור amplicon פורש הן אתרי השחבור STAT3, עם 5'- KpnI ו NheI אתרי הגבלה (5'-extensions) לפי לוח 1 א.
הערה: KpnI ו NheI אתרים נבחרו כדי לאפשר מוסיף להיות ligated לתוך וקטור PET-28a שונה עם kanamycin התנגדות 25,26 אתר KpnI נוסף לתוך אתר השיבוט המרובה.. - שימוש אאוזינופילים תרם טרי, להכין כפולות דגימות של 2.5 x 10 מ"ל 6 תאים / במדיום תרבית תאים (רוזוול פארק Memorial המכון (RPMI) 1640 בינוני). דגירה עבור שעה 1 ב 37 מעלות צלזיוס מתחת 5% CO 2 לחות ו -10%.
- כן משלימים (ג) דנ"א מדגימה (לפחות 1 x 10 6 תאים לכל בהכנה) לפי הוראות יצרן באמצעות מיצוי RNA וערכות סינתזת cDNA.
- מ cDNA התבנית מוכנה בשלב 1.2.1, להגביר אחוי STAT3 וריאנטים באמצעות הגברת PCR לפי לוח 2 א.
- פתור amplicons ב 2% agarose טריס-אצטט-ethylenediaminetetraacetate (TAE) ג'ל 27. להקות ובלו מן ג'ל ולטהר בהתאם להוראות ערכת כריתת ג'ל.
- Amplicons קטוף פלסמיד עם אנזימי הגבלה מתאימים (סקירה של הגבלה בהתייחס 27), באמצעות כרכים, פעמי דגירה וטמפרטורות המומלץ על ידי ספק אנזים; ולטהר כמו בשלב 1.4. ולקשור amplicons מוגבל לתוך פלסמיד בתנאים מומלץ ספק.
- כן המשך שלילירול (DNA פלסמיד מוגבלת מבלי להוסיף אבל עם האנזים) ו כביקורת חיובית (פלסמיד מוגבל עם התנגדות kanamycin). בצע טרנספורמציות פלסמיד רמת E. המוסמכת coli DH5α 28 תערובות קשירה באמצעות 27.
- דגירה טרנספורמציה E. coli ב 1 מ"ל לוריא-מרק בינוני (LB) (מוכן כמו 27 הורה) רעד במשך שעה 1 ב 37 מעלות צלזיוס. transformants מורחים על צלחות LB המכיל 50 מיקרוגרם / מ"ל kanamycin ו לדגור על 37 מעלות צלזיוס למשך הלילה.
- פיק מספר מושבות מן α STAT3 וצלחות STAT3 β באמצעות קיסמים סטרילי 27. העברת כל 2 מ"ל LB. לגדול תרבויות הלילה ב 37 מעלות צלזיוס חממה רועדת.
- לטהר פלסמידים מתרבויות חיידקי ידי ביצוע ההוראות המופיעות ערכת טיהור DNA. פלסמידים חנות ב C ° -20 או -80.
- פלסמידים רצף מכמה מושבות על ידי הכנת 20 תגובות רצף μl. 3 פיפטהמאגר התגובה רצף μl, 2 μl תערובת התגובה רצף, 12 μl מים ultrapure, 1 פלסמיד μl כתבנית ו פריימר רצף 2 μl.
הערה: אם אתה משתמש PET-28a וקטור, להשתמש רצף פריימר 5'-TAA TAC GAC TCA CTA TAG GGG-3 ".- להעריך electrophoretograms של פלסמידים קידוד כל גרסה: Sα, Sβ, ΔSα ו ΔSβ 25.
- ריכוז מדוד של פלסמיד דנ"א טהור ng / μl. אם קריאה ספיגה ב 260 ננומטר באמצעות ספקטרופוטומטר, לחשב ריכוז ה- DNA באמצעות החוק בר-למברט 29:
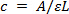
איפה c = ריכוז, A = ספיגת, ε (מקדם הכחדה) = 0.02 (מיקרוגרם / מ"ל) -1 ס"מ -1 עבור פעמיים גדילי דנ"א ו- L = אורך הדרך של אור (בדרך כלל 1 ס"מ). - באמצעות המשקולות המולקולריות של בבוקר cDNA גרסת אחוי STAT3plicons ואת וקטור, לחשב את מספר עותק לכל μl.
הערה: משקל מולקולרי לכל זוג בסיס (נ"ב) נאמדת 650 Da.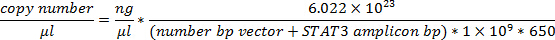
2. ניתוח פריימר וספציפי המוחלט qPCR
- כן 1 ב 10 סדרת דילול של פלסמידים STAT3, עם ~ 10 8 כדי 10 3 עותקים לכל μl (20 μl) באמצעות מי ultrapure. ריכוז מדוד של המרוכזים ביותר (~ 10 8 עותקים לכל μl, ראה שלב 1.11).
- השתמש פלסמידים בדילול להכין ארבע תערובות "היעד הלא" (כלומר., STAT3 ΔSβ בקרה שלילית המכילים ריכוזים שווים של STAT3 Sα, ΔSα, Sβ אבל לא ΔSβ) בשעה 10 6 עותקים לכל μl כל. להכין תערובת "יעד" עם ריכוזים שווים של כל ארבע תבניות כל 10 6 עותקים לכל μl.
- כן פתרון זוג פריימרים עבור כל חלופה אחוי (ראה סעיף ב 1 טבלה ותרשים 1) על ידי ביצוע ~ 60 μl של פריימרים כל בכל 7 ריכוז מיקרומטר (ריכוז סופי במדגם יהיה 560 ננומטר).
- לדלל DNA פולימרז / תמהיל לצבוע מחייב dsDNA 7: 5 עם H 2 O. טהור
- הגדרת Assay 1 צלחת 96-היטב PCR כמוצג תבנית (לוח 3).
- להוסיף 21 μl מדולל DNA פולימרז / dsDNA מחייב שילוב צבע, אז 2 μl של תערובת פריימר 2 μl של תבנית (לפי טבלה 2 ב). במקום תבנית, להוסיף 2 μl מסוננים H 2 O ל אין שליטת תבנית (NTC) היטב. הגדרת תגובות בשני עותקים להעריך דירות 30.
- חותם qPCR צלחת עם כיסוי צנטריפוגות דבק במשך 5 דקות ב 1200 XG ב 12 ° C.
- אם אתה משתמש בתוכנה רשום על חומרים, להגדיר את הניסוי כמתואר.
- הפעל מכונת הכנס qPCR אטום צלחת qPCR. לחץ על קובץ &# 62; חדש> הבא> בחר "גלאי חדש", לבחור כתב, מרווה ולספק שם. הגדרה "גלאי חדש" עבור כל גרסה אחוי.
- בחרו כעת להגדיר סטנדרטים כפי שיתבקש בדף הגדרת פלייט לדוגמא, הבטיחו כמויות מוזנות לתוך השולחן ונבחרים גלאים מתאימים. לחץ על סיום.
- גדר אופניים לפי לוח 2 ב. ודא כי קרינת קריאה (כדי לקבוע CT) מתרחש במהלך שלב C 72 ° של כל מחזור. בחר הפעלה.
- כאשר הריצה תושלם, לחץ על תוצאות כרטיסייה> עלילת הגברת הכרטיסייה. הערכת עלילת הגברה פלט להעריך איכות נתונים (לחפש עלילת מעריכים, כמו באיור 2).
הערה: ודא מגרשי הגברה הם בעיקר מעריכים כי סף המחזור ניתן להגדיר בחלק המעריכים של העלילה. ודא NTCs אינו מוגבר נקודות סטנדרטיות אחרות להפגין ערך ה- CT נמוך עם ΔRn גבוה. - כדי expאורט תוצאות ל תוכנת גיליון אלקטרוני, לחץ על קובץ> ייצוא> תוצאות.
3. הערכה ספציפית Assay המוחלט qPCR ו נשנים
- הֲדִירוּת
- השתמש בנתוני משלב 2.7.5 לחשב סטיית התקן של CT (מחזור סף קרינה) ערכים.
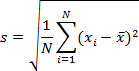
איפה N = מספר הדגימות (2 אם נעשה בשני עותקים),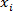 = מדגם של CT ו-
= מדגם של CT ו- 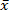 = מדגם Ct מתכוון. בדוק כי סטיית התקן של ערכי ה- CT של כפילויות הוא ≤0.2. בדוק כי ערכי ה- CT בכל אך בארות NTC הם קטנים מ -38.
= מדגם Ct מתכוון. בדוק כי סטיית התקן של ערכי ה- CT של כפילויות הוא ≤0.2. בדוק כי ערכי ה- CT בכל אך בארות NTC הם קטנים מ -38. - אם ההדירות פרמטרי רכיבת אופטימיזציה עניים או על ידי זמן חישול להגדיל או להקטין.
- השתמש בנתוני משלב 2.7.5 לחשב סטיית התקן של CT (מחזור סף קרינה) ערכים.
- תחושת יומן עותק מגרשתאה (כפי שנקבע בשלב 1.12 ו מוכן בשלב 2.1) לעומת ערך Ct כדי ליצור עקומת סטנדרט; מניב את המשוואה הבאה:
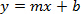
איפה y הוא Ct, m הוא שיפוע, x הוא יומן (מספר עותק) ו- B הוא הערך ליירט.
הערה: ערך R 2 של רגרסיה ליניארית יהיה ≥0.95 עבור נתונים טובים. - חישוב יעיל הגברה באמצעות עקומת סטנדרט ואת המשוואה:
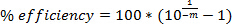
הערה: כוון יעיל ≥75%. - חישוב סגולי מן assay qPCR 1 באמצעות הנוסחא של מדי:
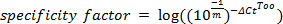
איפה Δ Ct מדי target = Ct - יעד שאינו Ct, המתאר את ההבדל במספר מחזור סף מגבר ספציפי שאינו ספציפיlification
הערה: ספציפי יעלה ארבעה סדרי הגודל (כלומר, גורם סגולי ≥4.).- אם סגולי הוא עני, לייעל ידי הורדת ריכוז פריימר. הגדרת מבחנים (כמתואר בשלבי 2.5-2.7) עם ריכוזי פריימר סופיים הנעים בין 100 ננומטר עד 500 ננומטר.
4. ביצוע מבחנים יחסית qPCR
- לטפל בתאים עם ציטוקינים כדי לקדם את התמלול.
- לקבלת אאוזינופילים תרם טרי, להכין כפולות דגימות של 2.5 x 10 מ"ל 6 תאים / במדיום תרבית תאים (RPMI 1640 בינוני) ולטפל עם 50 ng / ml interleukin-3 (IL3) ו / או 50 ng / α גורם הגידול נמק מ"ל ( TNFα) לתקופות של 3, 6, 9 ו -20 שעות ב 37 מעלות צלזיוס מתחת 5% CO 2 ולחות 10%.
- כן cDNA ממדגמים אאוזינופילים (לפחות 1 x 10 6 תאים לכל בהכנה) לפי הוראות יצרן באמצעות ערכות סינתזת מיצוי RNA ו cDNA.
- כן דילולי סדרה של שני aliquots cDNA המדגם (שליטה או דוגמאות המנוחות), עם מגוון דילול מ "1" ל "1 1024" דילול.
- עבור דילול 1 ב 4, פיפטה 1 cDNA μl ו -3 μl מים ultrapure.
- עבור דילול 1 ב 16, פיפטה 1 μl של 1 ב 4 דילול 3 μl מים ultrapure, וכו '
- הגדרת PCR בצלחת 96-היטב עם 25 תגובות μl כמו השלבים המתוארים 2.3-2.5 אבל עם כמה ריכוזי פריימר עבור STAT3 פאן ו GUSB (ראה תבנית בטבלה 4).
- הוסף "הגלאי החדש" עבור GUSB ובצע את הפעולות 2.6-2.7, באמצעות הפרמטרים אופניים המתואר 2c טבלה.
- צור עקומות סטנדרטיות (ראה צעדים 3.2 ו -3.3) כדי לקבוע אילו ריכוזים פריימר לגרום יעילות הגברה 95-100%.
- צלחת גדר בעזרת דגימות cDNA מוכנות (מ שלב 4.2) וריכוזי פריימר אופטימיזציה(ראה לוח 2 ג עבור פרמטרים אופניים, ריאגנטים ולוח 5 96-היטב תבנית צלחת).
- בצע שלבים 2.6-2.7. assay חוזר לפחות פעם ולבדוק את איכות נתונים.
- חשבתי את ערך ה- CT הממוצע של מדגם הכפול Cts עבור STAT3 שניהם GUSB גן המשק.
- השג ערכי ה- CT Δ עבור כל דגימה.
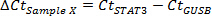
- לייעד מדגם המנוחה (יש בדרך כלל הריכוז הנמוך ביותר של תמליל של עניין) כמו לדוגמא 0. Ct חישוב ΔΔ 31 עבור כל דגימה (המיועד כאן מדגם X):
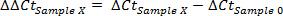
- חישוב-גידול של פי עבור כל דגימה:
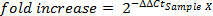
- שחזור
- חשבתי את המקדם השונה (CV) של מספר עותק פאן STAT3 ו GUSB.
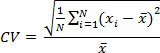
איפה N = מספר הדגימות, = מספר העותק המחושב של המדגם = מדגם מתכוונים. בדקו שקורות חיים של כל דגימה (מבחנים מרובים) הוא ≤10%.
- חשבתי את המקדם השונה (CV) של מספר עותק פאן STAT3 ו GUSB.
5. ניתוח מוחלט qPCR נתונים עבור דגימות ידועות
- השוואת ערכי ה- CT שהושגו ביחס qPCR (כמתואר בשלב 4.9) עבור GUSB גן המשק מנת להבטיח ריכוזי cDNA 'הדגימות הם בתוך סדר גודל אחד.
- לדלל cDNA בהתאם (למשל, אם המדגם 1 יש ערך CT של ~ 17.5, ו ערכי ה- CT "דוגמאות אחרות הן ~ 21, מדגם 1 הוא בערך 2 (21-17.5) = 11 פעמים יותר מרוכז בצע 1:. דילול 1 עם מים ultrapure בתוך אותו טווח מבלי לדלל יותר מהנדרש).
הערה: ריכוזי התחלה שונים יהיה בהרבה הטיה בניתוח רגרסיה ליניארית (שלב 4.12).
- לדלל cDNA בהתאם (למשל, אם המדגם 1 יש ערך CT של ~ 17.5, ו ערכי ה- CT "דוגמאות אחרות הן ~ 21, מדגם 1 הוא בערך 2 (21-17.5) = 11 פעמים יותר מרוכז בצע 1:. דילול 1 עם מים ultrapure בתוך אותו טווח מבלי לדלל יותר מהנדרש).
- הגדרת assay qPCR המוחלט של דגימות. כן צלחת תבנית 2a assay עבור Sα ו Sβ לפי טבלה 6; ולהכין 2b assay עבור ΔSα ו ΔSβ לפי לוח 7. ביצוע מבחני כמתואר בשלבים 2.6-2.7.3 (ולוח 2b). חזור על מבחנים לפחות פעם אחת.
- בדוק נתוני איכות הייצוא כמתואר בשלבים 2.7.4-2.7.5.
- הגדרה צלחת היטב מוחלט 96 qPCR עם 25 תגובות μl באמצעות פריימרים STAT3 פאן (פריימרים ב 1c שולחן, תבנית בטבלה 8) ב 400 מיקרומטר ו שילוב של כל ארבעת פלסמידים או פלסמיד יחיד בריכוזים כוללים רשומים.
הערה: יעילות לא משנה עבור מוחלט qPCR, אבל מיטוב היעילות של פריימרים אלו חשובה ביחס qPCR (שלב 4). - חותם qPCR צלחת עם כיסוי צנטריפוגות דבק במשך 5 דקות ב 1200 XG ב 12 ° C.
- הגדרה "גלאי חדש" עבור מחבת- STAT3 כמו צעדים 2.7.1-2.7.3.
- הגדרת האופניים בשביל פאן STAT3 לפי לוח 2 ג (אותם תנאים כמו qPCR יחסית). ודא כי קרינת קריאה (כדי לקבוע CT) מתרחש במהלך שלב C 72 ° של כל מחזור. assay חוזר לפחות פעם אחת כדי להבטיח שחזור.
- בדוק נתוני איכות יצוא (ראה צעדים 2.7.4-2.7.5).
- אם מגרשי הגברה אינם מעריכי (ראה איור 2), לדלל cDNA מדגם (1:10) וחזור assay כמתואר (שלב 5.7).
- באמצעות המשוואה של עקומת סטנדרט (ראה 3.2, איור 3) ואת ערכי ה- CT של דגימות, לחשב את מספר עותק של כל חלופה אחוי ושל STAT3 פאן במדגם אחד.
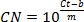
- יומן מגרש (ערכי מספר העותק מוחלטים qPCR המתקבלים עבור STAT3 פאן) vs יומן (ערכי מספר עותק qPCR מוחלטים מצטברים עבור STAאחוי T3 גרסאות).
הערה: ערכי רגרסיה ליניארית אידיאלית ושיפוע היו שניהם להיות ~ 1. - חישוב דירות (שלב 3.1) ואת שחזור (שלב 4.13).
6. מיזוג במונחים מוחלטים ובאופן יחסי qPCR נתונים
- הכפל את משקל הגרסה עם מתקפלת עליית STAT3 הכוללת לחשב מתקפלים גידול של כל גרסה אחוי.
- חשב את סטיית התקן (ראה שלב 3.1.1) של ערכים מוחלטים ויחסיים לתת דין וחשבון על התפשטות של שגיאה. לקבוע סטיית התקן של מדידה (SEM), כפי שמוצג:
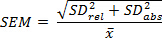
תוצאות
נתוני qPCR באיכות טובים יהיו ליצור עלילת הגברה sigmoidal (איור 2 א), מסמל גידול מעריכי תמלילי במהלך הרכיבה. הנוכחות של תבנית יותר מדי יכולה לגרום לרקע קרינה גבוהה, כלומר קו בסיס הולם היא הוקמה בשנת המחזורים הראשונים. אם הנתונים אינם מספקים עקומה מעריכית (איור 2b), אופטימיזציה נוספת יש צורך (המפורטים צעדים 3.1 ו -3.4). לקבלת מידע נוסף אודות תוצאות תקלות qPCR, להתייחס התייחסות 32. עקומות התקן שנוצרו עבור פלסמידים כיל תבנית תציינה את היעילות של הגברה (עקום עבור STAT3 Sα שמוצג באיור 3). יעילות בין 83 ו -95% נצפתה בתנאים שתוארו. המשוואה עבור סגוליות (שלב 3.4) מניחה יעילות שווה, המהווה 25 סביר, אז את הספציפיותהוא צפוי להיות גדול יותר מאשר גורם סגולי מרמז.
על מנת להעריך את congruency של רמות STAT3, הערכים המוחלטים של כל אחת מארבע גרסאות אחוי נמדדו כמו גם רמת STAT3 הכל, פריימרים באמצעות האחרונים הגברה באזור המשותף לארבע גרסאות אחוי (איור 4). באופן אידיאלי רגרסיה ליניארית (אינדיקציה קורלציה) ושיפוע (יחס של STAT3 פאן וריאנטים אחוי סיכם) צריכים גם להיות קרוב ל -1.
נתוני qPCR המוחלטים מוצגים תרשימי עוגה להראות את הפרופורציות של הארבעה וריאנטים אחוי לאורך זמן שלאחר גירוי עם ציטוקינים (איור 5 א). נח אאוזינופילים (0 hr) מאופיין באחוז הקטן של STAT3 Sα, למרות וריאנט זה היה תמיד הנפוץ ביותר. הכפלת חלק של וריאנטים אחוי β STAT3 (S ^6; + ΔSβ) על ידי השבר של וריאנטים אחוי STAT3 ΔS (ΔSα + ΔSβ) באופן עקבי נתן ערך נמוך מהערך מוקלט באופן ניסיוני עבור ΔSβ. אם אירועי השחבור היו עצמאיים, ניתן היה לצפות כי הכפלת שבריר ΔS וריאנטים ידי השבריר של וריאנטים β ייתן ערך שמסכים עם הערך נקבע באופן ניסיוני. רמות גבוהות יותר של ΔSβ מהצפוי מאירועי שחבור עצמאיים מצביעות על הטית שחבור-שיתוף קיימת.
מיזוג נתונים qPCR מוחלט ויחסי הראו כי רמות של כל גרסאות אחוי STAT3 הגדילה גירוי פוסט עם ציטוקינים IL3 ו TNFα, עם רמות השיא hr 6 שלאחר גירוי (איור 5 ב '- ה). במשך שלוש של גרסות הארבע אחוי, רמות התמליל היו בערך 3 פעמים גבוהות יותר IL3 + TNFα אאוזינופילים מטופלים (6 שעות) לעומת אאוזינופילים בתקשורתבאותו נקודת זמן. רמות STAT3 Sα היו גבוהים פי 3.5 ב IL3 + TNFα מטופלים אאוזינופילים לעומת אאוזינופילים התקשורת בנקודת זמן זו. אי הוודאות הגדולה ביותר (סטיית תקן גדולה של מדידה) נתפסה ΔSβ (5e איור), הכולל את החלקיק הקטן של STAT3 סך כל הדגימות. הדבר לא היה מפתיע, כמו רמות נמוכות קשורות עם ערכי ה- CT גבוהים. הדורשים יותר מחזורים כדי להגיע מחזור סף יהיה מתחם הוודאות עקב מחזור ל-מחזור וריאציה היעילות של הגברה.
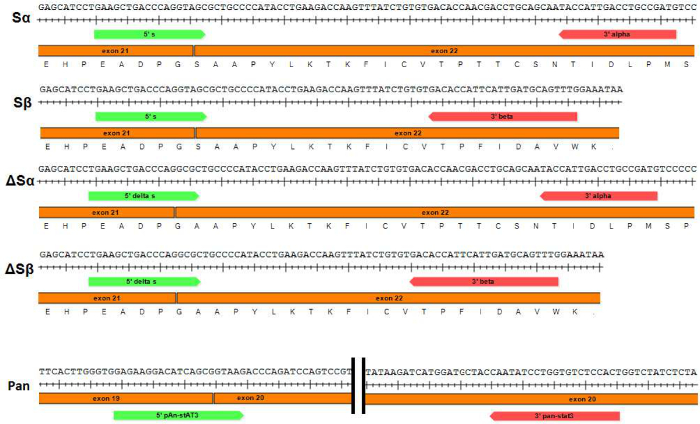
איור 1: סכמטי של זוגות פריימר המשמש לביצוע qPCR של וריאנטים אחוי STAT3 והפאן-STAT3 Primers משמשים כדי להגביר כל ספציפי של וריאנטים אחוי STAT3 (Sα, Sβ, ΔSα ו ΔSβ בהתאמה) s הם.hown. פריימרים קדמיים (STAT3 "S" ו "ΔS") משתרעים על פני הצומת בין אקסונים 21 ו 22. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
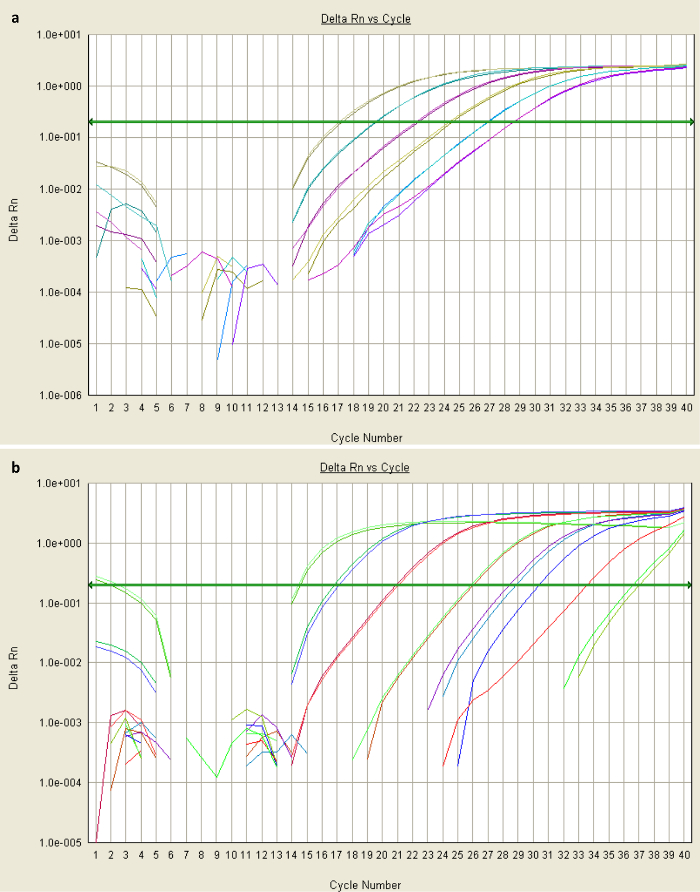
איור 2:. מגרשי הגברה של נתוני qPCR (א) מגרש הגברת sigmoidal פירושו הגברה אמינה. נתונים אלה התקבלו qPCR של שני דילולים סדרתי של פלסמיד המכיל STAT3 Sα, כאשר כל זוג של קווים צבעוניים המייצגים את רמות הקרינה של דגימות בדילול כפולים במשך 40 מחזורים (ציר x). המדגם המרוכז ביותר (ירוק-האפור) היה מוגבר באופן מספק על ידי מחזור 17 (עם dsDNA מחייב לצבוע פרופורציונאלי קרינה, המוצג על ציר ה- y) יעלה את ערך קרינת הסף (baseline כפי שמוצג חץ ירוק). ערך ה- CT שלה יהיה 17. (ב) ללא מעריכים מגרשים מראים כי סף רקע הקרינה לא הוקם כראוי המחזורים הראשונים. זה יכול להיות בשל נוכחותם של מעכב, או פערים מרוכז תבנית או פריימרים. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
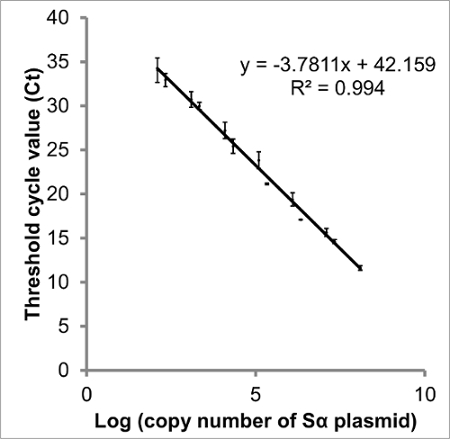
איור 3: עקומת סטנדרט של log (עותק מספר STAT3 Sα) vs Ct קיים קשר ליניארי בין יומן של כל מספר עותק של גרסה אחוי STAT3 מחזור סף (CT).. יצירת עקומת סטנדרט מ- DNA פלסמיד מחק את cDNA של דגימות ובכךמספק מדידה טובה יותר מאשר עקום שנוצר amplicons בדילול PCR. הנתונים המוצגים הם ערכי ה- CT המתקבל qPCR של שני דילולים סדרתי של פלסמיד המכיל STAT3 Sα. מתוך העקומה הזו, בהווה מספר העותק בכל מדגם ניתן אינטרפולציה, ויעילות הגברה מחושבת (83.9%). למרות מיירט y- פחות לשחזור מ המדרון, ליירט מרמז 42.2 מחזורים יהיה צורך להיות בטוח לא DNA היעד הוא ההווה. ברי שגיאה מצביעים SEM, n = 3. עקומות להשוואה נבנו עבור STAT3 Sβ, ΔSα ו ΔSβ (לא מוצג). נא ללחוץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
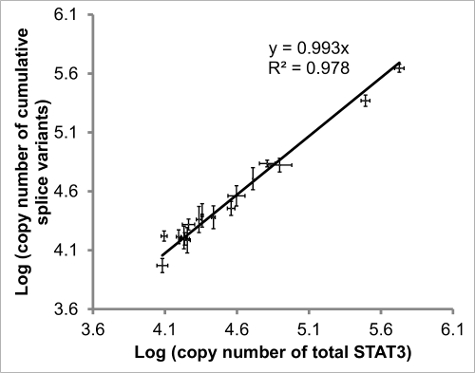
איור 4: השוואה של פאן לכמת STAT3 vs גרסאות אחוי STAT3 מצטבר. הרגרסיה של אחוי STAT3 הוסיף וריאנטים vs STAT3 הכולל צריך המדרון (יחס של פאן כדי מצטבר) ו- R 2 ערך (קורלציה) קרוב ל -1 ערכים מ -17 דגימות (אאוזינופילים DLBCL) כלול. ברי שגיאה מצביעים SEM של קביעות x-אל-y, n ≥ 2 עבור כל אחד. איור מותאם התייחסות 20. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.

איור 5:. רמות גרסת אחוי STAT3 נעו במהלך טיפול ציטוקינים (א) תרשימי עוגה המציין באחוזים של כל גרסה STAT3 אחויב אאוזינופילים במהלך הטיפול עם IL3 ו TNFα. (ב - ה) שינויים גרסאות אחוי STAT3 ב אאוזינופילים שטופלו שילובים שונים של ציטוקינים, שהיא נמדדת על ידי שילוב נתונים qPCR יחסי ומוחלט STAT3 Sα (ב), Sβ (ג), ΔSα (ד), ו ΔSβ רמות (ה). נע לאורך זמן גירוי פוסט. רמות בתחילה גדלו והגיעו לשיא 6 שעות-גירוי פוסט. השילוב TNFα + IL3 שהושרו ביטוי גבוה של כל גרסאות אחוי ארבעה STAT3 מ IL3 לבד. SEM מחושב עבור כל נקודת נתונים והיוותה התפשטות של שגיאה. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
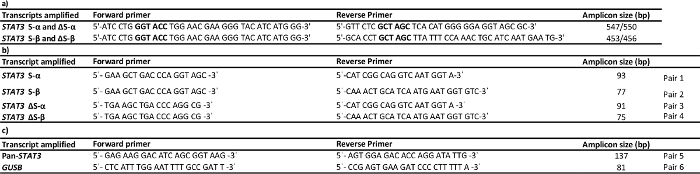
שולחן 1: Primers המשמש הגברה (א), מוחלט (ב) ו- יחסית (ג) PCR כמוני. פריימרים שיבוט יש רצפי הגבלה 5'-סיומות עבור חיתוך יעיל. KpnI ו NheI אתרי הגבלה מסומנים באותיות מודגשות. אנא לחץ כאן כדי להציג גרסה גדולה יותר של השולחן הזה.
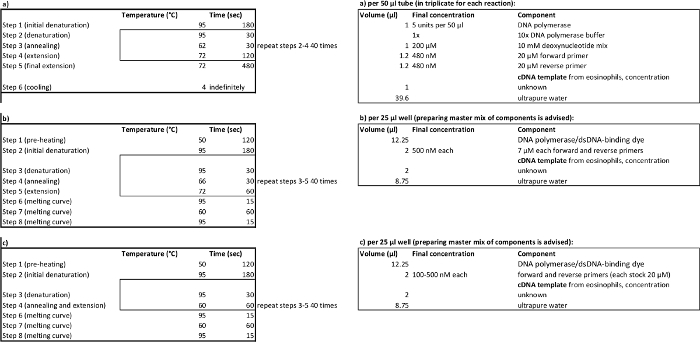
טבלת 2. פרמטרי אופני PCR (משמאל) כרכים מגיבים (מימין) הגברה (א), ביחס (ב), מוחלט (ג) PCR כמוני. אנא לחץ כאן כדי להציג גרסה גדולה יותר של השולחן הזה.
3 / 54473tbl3.jpg "/>
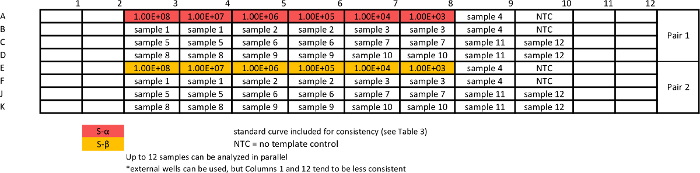
טבלה 3:. תבנית עבור assay כיול פלסמיד מוחלט qPCR assay זה יש צורך להעריך שחזור ויעילות, כמו גם יצירת עקומות סטנדרטיות שממנו ניתן לשרבב נתונים. תערובות "היעד הלא" תיתנה אומדן של סגולי. אופטימיזציה עשוי להיות נחוץ כדי להשיג עקביות. אנא לחץ כאן כדי להציג גרסה גדולה יותר של השולחן הזה.
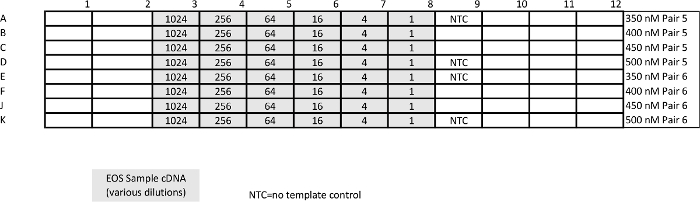
לוח 4:. תבנית ביחס qPCR (STAT3 פאן ומשק הגן GUSB) assay כיול בניגוד מוחלט qPCR, הנקודה של assay זה היא לקבוע תנאים בהם יעילות הגברה הוא ~ 100%."Target =" tp_upload / 54,473 / 54473tbl4large.jpg _ blank "> אנא לחץ כאן כדי להציג גרסה גדולה יותר של השולחן הזה.
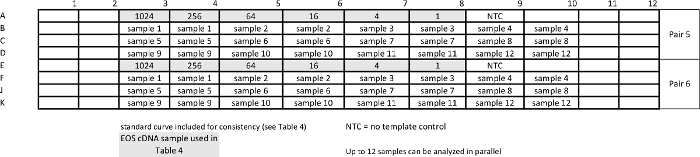
לוח 5:.. תבנית עבור assay מדגם ביחס qPCR למדידת גן STAT3 ומשק פאן GUSB עקומה סטנדרטי חוזרים יחד עם דגימות על מנת להבטיח יעילות דומה של המבחנים אנא לחץ כאן כדי להציג גרסה גדולה יותר של השולחן הזה.
לוח 6: תבנית עבור assay מדגם מוחלט qPCR למדידת גרסאות S עקומות סטנדרטיות חוזרים יחד עם דגימות כדי להבטיח EFF דומה. iciency של המבחנים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של השולחן הזה.
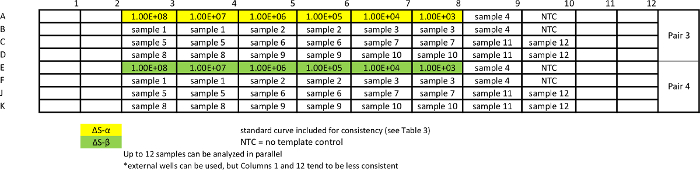
לוח 7:.. תבנית עבור assay מדגם המוחלט qPCR למדידת גרסות ΔS עקומה סטנדרטי חוזרים יחד עם דגימות על מנת להבטיח יעילות דומה של המבחנים אנא לחץ כאן כדי להציג גרסה גדולה יותר של השולחן הזה.
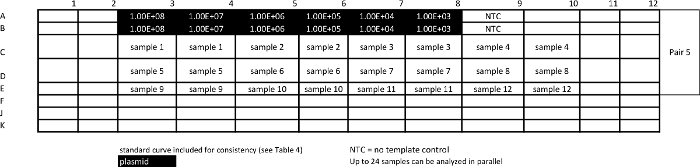
לוח 8: תבנית עבור assay מדגם מוחלט qPCR למדידת STAT3 פאן.large.jpg "target =" _ blank "> אנא לחץ כאן כדי להציג גרסה גדולה יותר של השולחן הזה.
Discussion
פתחנו פרוטוקול זה על מנת להעריך את הרמות ופרופורציות של תמלילי גרסת אחוי STAT3 ב אאוזינופילים ותאי לימפומה וללמוד אם גירוי ציטוקינים השפיע על הרמות ופרופורציות. STAT3 היא בעלת עניין מיוחד בגלל הפונקציונליות pleiotropic ולא בטוח שלה, עם דיווחים סותרים על אם הוא מתנהג כמו מדכאי oncoprotein או גידולים סרטניים (הנסקרת תוך התייחסות 33). הבדלי α STAT3 ו β אחוי גרסת הפונקציה התאפיינו בעבר 34,35, והפרוטוקול שלנו הקל ניתוח עקום למטה / מחדש ביטוי מרמז צורך היחס אופטימלי של S ו- ΔS תמלילי 19.
כימות מדויק של וריאנטים אחוי ברורים יקל חקירות נוספות של הנוגעים פונקצית STAT3 הטרוגנית אחוי רכב גרסה. פרוטוקול משלב נתוני qPCR מוחלטים ויחסים, המשלב את היכולת של abהמומס qPCR לחישוב יחסי מידות גרסה אחוי, ויחסית qPCR למדוד שינויים בביטוי STAT3 הכוללת. גישה זו מאפשרת לאדם להבחין בהבדלים דקים ברצף ובמקביל למדוד יחסי שחבור בשני אתרי אחוי חלופיים יותר מזה 50 נוקלאוטידים. קביעת היחס בין אירועי השחבור בנפרד לא הניבה את הממצא המרשים כי הטית שיתוף שחבור קיימת כזה כי רמות ΔSβ גבוהות מצפוי אם שימושים של שני אתרים הם איחו אקראי 25.
אנושות, מוחלט qPCR עם השימוש של עקומות כיול פלסמיד מאפשר כימות (ביעילות תת אופטימלית) של וריאציות אחוי שתוצאתן רצפים דומים מאוד. אנו צופים assay qPCR שחבור עדין רומן יארך חודשים כדי לייעל. שלבים עיקריים בפיתוח assay הם פרי יצירתה של פלסמידים STAT3 שבשימוש בתהליך יצירת עקומות סטנדרטיות עבור מוחלט qPCR; באופן ניסיוניקביעת רצפי פריימר אופטימליים ופרמטרי אופניים כדי להבטיח ספציפי שחזור; ושילוב של נתונים qPCR היחסי הנגזר לכימות ביחס הביטוי הפאן STAT3 לביטוי GAPDH. המתאם של מספר עותק לכמת ידי פאן STAT3 לעומת כימות מצטבר (Sα + ΔSα + Sβ + ΔSβ) מראה כי פרוטוקול מייצר תוצאות אמינות.
הערת אזהרה של הטכניקה היא תהליך האימות הנרחב. יש צורך להעריך השתנות תוך assay (דירות), השתנות interassay (שחזור) וספציפי. הפרוטוקול מתאר דרכים להשיג תפוקות מספריות עבור פרמטרים אלה. אנו רואים יעילים ≥75%, גורם סגולי ≥4, מקדם שונה (שחזור) ≤10% וסטיית תקן CT (דירות) ≤0.2 ספים מתאימים כמו 30. מוטציות או מחיקות של חומצות אמינו רצף STAT3 1-690 לא תהיינה discoveאדום על ידי פרוטוקול זה, למרות שהם עשויים להשפיע על יחסי שחבור. פרופורציות תמליל עלולות להיות לא פרופורציונליות proteoform פרופורציות 36.
מאז דגימות שונות כמויות התחלתי של cDNA הכולל, qPCR המוחלט מתאים השוואת מספרי עותק של וריאנטים אחוי בתוך מדגם אבל לא להשוואה בין מדגם אלא אם מצמיד עם qPCR ביחס באמצעות גן משק הוקם. השיטה המתוארת תואמת את הנחיות MIQE qPCR עבור שחזור 30. PCR פרמטרים אופניים וריכוזי פריימר ייתכן שיהיה צורך להיות שונה כדי לקבל נתונים לשחזור אם משמש ציוד אחר. סגולי מושלם אינו אפשרי ללא יעילות להתפשר באופן דרסטי, אבל היעד היה מוגבר באופן יעיל יותר על ידי יותר מ ארבעה סדרי הגודל.
לינארית DNA מוגבר יותר בקלות מאשר מעגלי. אם הפלסמיד חלופי אינו מספק עקומות סטנדרטיות משביע רצון (R 2 <; 0.95), שקול ליניאריזציה של פלסמיד על ידי הגבלה באתר אחד לפני כימות. אופטימיזציה qPCR היא קריטית להשגת נתונים באיכות טובה (איור 1). רוב פרוטוקולי qPCR להסתמך על אופניים דו-שלבים, ומכונות מותאמות בהתאם. חימום לא אחיד של בלוק החימום עלול להחריף ב אופניים בן שלושה שלבים, תורם דירות עניות. מבחני יש להקים בתנאים סטריליים עם טיפים פיפטה מסנן מים ultrapure, אידיאלי במנדף זרימה למינרית ייעודי. בגלל מזהמים יכולים להוביל לתוצאות לא עקביות, מבחני יש להגדיר בתנאים סטריליים עם טיפים פיפטה מסנן מים ultrapure, אידיאלי במנדף זרימה למינרית ייעודי. לקבלת מידע נוסף בנושא אופטימיזציה של qPCR, עיין בוסטין et al. 32
כימות STAT3 עלולה להוביל לתובנה רבה יותר בכמה הקשרים. STAT3 אוטומטית מסדיר 37 ביטוי משלו, ואת הפרוטוקול המתואר לעיל עשוי לעזורכדי להבהיר אם היחס בין גרסאות אחוי STAT3 לתרום להסדרת לולאת המשוב החיובי הזה. הפרוטוקול יכול לשמש כדי לחקור משמרות ביחסי גרסה אחוי כפי שנצפה בתאים בצפיפויות שונות 38 או במהלך פיתוח: זה ידוע כי שינויי יחס STAT3 α / β ברמת החלבון במהלך hematopoiesis 16. Sundin et al. נמצא כי שחבור מוטה פולימורפיזם נוקלאוטיד בודד רצפי האינטרונים של אקסון 12 ב STAT3 של חולה עם תסמונת איוב 39. זה מתקבל על הדעת כי אחד מני רבים SNPs נוכח אינטרונים בין אקסון 21 ו -22, או אקסון 22 ו -23 עשויים לתרום יחסי שחבור של ΔS / S ו α / β בהתאמה. את assay יכול לשמש כדי לכמת תמלילי STAT3 בתאים סרטניים, שבו מוטציות או שינויים ברגולציה שחבור עשוי להציג את ההטיה לתהליך השחבור 40. מוטציות בגורמי שחבור (כמו SF3B1), כמו observאד בשנת התסמונת המיאלודיספלסטית 41 עשוי גם להוביל לשינויים שניתן למדוד על ידי פרוטוקול זה.
במובן רחב יותר, גישה זו מזהה באופן ספציפי שיתוף העמותה שחבור, אשר אינו ריאלי עם RNA-seq קונבנציונאלי, ולא סטנדרטי qPCR. בעוד תופעת שחבור אקסון סותרים מדגימה תיאום של החלטות שחבור, שיתוף עמותת אירועי שחבור אחרים לא כבר המבוססת על מחקר. שיטה חלופית תארה לאחרונה, שבו RNA-seq שונה כדי לחקור cDNA באורך מלא, מציעה אירועי שחבור רחוק יותר שיתוף תלוי מאשר סבר עד כה 42.
STAT3 מכיל אתר שחבור טנדם התורם. אתרי מקבל אחוי טנדם הם תכופים יותר 43 ואת עקרונות הפרוטוקול המתואר יכולים לשמש כנקודה מוצאת לפיתוח מבחנים לגילוי מקרי של שחבור NAGNAG ואירועי שחבור אחרים בתוך 200 נוקלאוטידים. poten אחריישומים tial כוללים כימות של צירוף מקרים של הבדלים רצף עדינים אחרים, כמו indels או פולימורפיזם נוקלאוטיד כפול / משולש 44.
Disclosures
The authors have nothing to disclose.
Acknowledgements
המחברים מבקשים להודות ה- National Institutes of Health-NHLBI עבור גרנט פרויקט תוכנית על תפקידה של אאוזינופילים Airway דלקת Remodeling: P01HL088584 (PI: נ Jarjour), ואוניברסיטת מרכז הסרטן ויסקונסין קארבון והמחלקה לרפואה למימון בין החומות. אנו מודים דאגלס אניס לשיבוט וריאנטים STAT3 ארבע.
Materials
| Name | Company | Catalog Number | Comments |
| MJ Research PTC-200 Thermal Cycler | GMI | N/A | Used for standard PCR |
| 7500 Real-Time PCR System | Applied Biosystems | N/A | qPCR machine |
| GS-6R | Beckman Coulter | N/A | centrifuge for 96-well plates |
| Nanodrop 2000 sprectrophotometer | ThermoFisher Scientific | N/A | |
| RPMI-1640 medium | Sigma Aldrich | R8758 | cell culture medium |
| PfuTurbo DNA Polymerase | Agilent Technologies | 600410 | DNA polymerase for standard PCR |
| KpnI | New England Biosciences | R0142S | |
| NheI | New England Biosciences | R0131S | |
| SYBR Green PCR Master Mix | Qiagen | 330523 | qPCR, DNA polymerase/dsDNA-binding dye mix |
| Rneasy Mini Kit | Qiagen | 74204 | RNA extraction kit |
| SuperScript III First-Strand Synthesis System | Invitrogen (ThermoFisher Scientific) | 18080-044 | cDNA synthesis kit |
| Primers | Integrated DNA Technology | N/A | |
| NEBuffer 1.1 | New England Biosciences | B7201S | |
| GenePure LE Agarose | ISC BioExpress | E-3120-500 | component of TAE gel |
| Pipettors | Major lab suppliers (MLS) | N/A | |
| Filter pipette tips | Neptune Scientific | BT10XL, BT20, BT200 | |
| EU One Piece Thin Wall Plate | MidSci | ABI7501 | |
| ThermalSeal A Sealing Film | MidSci | TSA-100 | 96 well plate seal |
| pET-Elmer (variant of pET-28a) | Novagen; modified in Mosher lab | N/A | Details in PMID: 20947497 |
| Wizard Plus SV Minipreps DNA purification system | Promega | A1460 | Plasmid purification |
| BigDye Terminator v3.1 Cycle Sequencing Kit | ThermoFisher Scientific | 4337455 | Sequencing kit |
| QIAEX II Gel Extraction kit | Qiagen | 20021 | Amplicon purification |
| DH5α competent cells | ThermoFisher Scientific | 18265-017 | available from several providers, see PMID: 2162051 |
| kanamycin | Research Products International Corp. | K22000-5.0 | |
| Tris base | ThermoFisher Scientific | BP152-5 | component of TAE buffer |
| Acetic acid, glacial | ThermoFisher Scientific | A38C-212 | component of TAE buffer |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma Chemical Company (Sigma Aldrich) | E-5134 | component of TAE buffer |
| Bacto Tryptone | BD Biosciences | 211705 | component of Luria Broth |
| Bacto Yeast extract, technical | BD Biosciences | 288620 | component of Luria Broth |
| Sodium chloride | ThermoFisher Scientific | S271-10 | component of Luria Broth |
| Sodium hydroxide | ThermoFisher Scientific | SS255-1 | component of Luria Broth |
| Bacto Agar | BD Biosciences | 214010 | component of Luria Broth plate |
| Lasergene SeqBuilder | DNASTAR | Figure 1 generated using Lasergene SeqBuilder software version 12.2.0 (DNASTAR) |
References
- Hiller, M., et al. Phylogenetically widespread alternative splicing at unusual GYNGYN donors. Genome Biol. 7 (7), R65 (2006).
- Treacy, M. N., et al. Twin of I-POU: a two amino acid difference in the I-POU homeodomain distinguishes an activator from an inhibitor of transcription. Cell. 68 (3), 491-505 (1992).
- Schindler, S., et al. Alternative splicing at NAGNAG acceptors in Arabidopsis thaliana SR and SR-related protein-coding genes. BMC Genomics. 9, 159 (2008).
- Szafranski, K., Kramer, M. It's a bit over, is that ok? The subtle surplus from tandem alternative splicing. RNA Biol. 12 (2), 115-122 (2015).
- Wang, M., et al. Alternative splicing at GYNNGY 5' splice sites: more noise, less regulation. Nucleic Acids Res. 42 (22), 13969-13980 (2014).
- Hiller, M., Platzer, M. Widespread and subtle: alternative splicing at short-distance tandem sites. Trends Genet. 24 (5), 246-255 (2008).
- Tsai, K. W., Lin, W. C. Quantitative analysis of wobble splicing indicates that it is not tissue specific. Genomics. 88 (6), 855-864 (2006).
- Tadokoro, K., et al. Frequent occurrence of protein isoforms with or without a single amino acid residue by subtle alternative splicing: the case of Gln in DRPLA affects subcellular localization of the products. J Hum Genet. 50 (8), 382-394 (2005).
- Bradley, R. K., Merkin, J., Lambert, N. J., Burge, C. B. Alternative splicing of RNA triplets is often regulated and accelerates proteome evolution. PLoS Biol. 10 (1), e1001229 (2012).
- Zheng, C. L., Fu, X. D., Gribskov, M. Characteristics and regulatory elements defining constitutive splicing and different modes of alternative splicing in human and mouse. RNA. 11 (12), 1777-1787 (2005).
- Schaefer, T. S., Sanders, L. K., Nathans, D. Cooperative transcriptional activity of Jun and Stat3 beta, a short form of Stat3. Proc Natl Acad Sci U S A. 92 (20), 9097-9101 (1995).
- Waitkus, M. S., et al. Signal integration and gene induction by a functionally distinct STAT3 phosphoform. Mol Cell Biol. 34 (10), 1800-1811 (2014).
- Srivastava, J., DiGiovanni, J. Non-canonical Stat3 signaling in cancer. Mol Carcinog. , (2015).
- Holland, S. M., et al. STAT3 mutations in the hyper-IgE syndrome. N Engl J Med. 357 (16), 1608-1619 (2007).
- Maritano, D., et al. The STAT3 isoforms alpha and beta have unique and specific functions. Nat Immunol. 5 (4), 401-409 (2004).
- Hevehan, D. L., Miller, W. M., Papoutsakis, E. T. Differential expression and phosphorylation of distinct STAT3 proteins during granulocytic differentiation. Blood. 99 (5), 1627-1637 (2002).
- Yoo, J. Y., Huso, D. L., Nathans, D., Desiderio, S. Specific ablation of Stat3beta distorts the pattern of Stat3-responsive gene expression and impairs recovery from endotoxic shock. Cell. 108 (3), 331-344 (2002).
- Stahl, N., et al. Choice of STATs and other substrates specified by modular tyrosine-based motifs in cytokine receptors. Science. 267 (5202), 1349-1353 (1995).
- Zheng, M., et al. A mix of S and DeltaS variants of STAT3 enable survival of activated B-cell-like diffuse large B-cell lymphoma cells in culture. Oncogenesis. 4, 184 (2016).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Zipper, H., Brunner, H., Bernhagen, J., Vitzthum, F. Investigations on DNA intercalation and surface binding by SYBR Green I, its structure determination and methodological implications. Nucleic Acids Res. 32 (12), e103 (2004).
- Kelly, E. A., Liu, L. Y., Esnault, S., Quinchia Johnson, B. H., Jarjour, N. N. Potent synergistic effect of IL-3 and TNF on matrix metalloproteinase 9 generation by human eosinophils. Cytokine. 58 (2), 199-206 (2012).
- Whelan, J. A., Russell, N. B., Whelan, M. A. A method for the absolute quantification of cDNA using real-time PCR. J Immunol Methods. 278 (1-2), 261-269 (2003).
- Too, H. P. Real time PCR quantification of GFRalpha-2 alternatively spliced isoforms in murine brain and peripheral tissues. Brain Res Mol Brain Res. 114 (2), 146-153 (2003).
- Turton, K. B., Annis, D. S., Rui, L., Esnault, S., Mosher, D. F. Ratios of Four STAT3 Splice Variants in Human Eosinophils and Diffuse Large B Cell Lymphoma Cells. PloS One. 10 (5), e0127243 (2015).
- Maurer, L. M., et al. Extended binding site on fibronectin for the functional upstream domain of protein F1 of Streptococcus pyogenes. J Biol Chem. 285 (52), 41087-41099 (2010).
- Ausubel, F. M., et al. . Current protocols in molecular biology. , (1987).
- Grant, S. G., Jessee, J., Bloom, F. R., Hanahan, D. Differential plasmid rescue from transgenic mouse DNAs into Escherichia coli methylation-restriction mutants. Proc Natl Acad Sci U S A. 87 (12), 4645-4649 (1990).
- Kocsis, L., Herman, P., Eke, A. The modified Beer-Lambert law revisited. Phys Med Biol. 51 (5), N91-N98 (2006).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin Chem. 55 (4), 611-622 (2009).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Res. 29 (9), e45 (2001).
- Bustin, S. A. . A-Z of quantitative PCR. , (2004).
- Zhang, H. F., Lai, R. STAT3 in cancer-friend or foe. Cancers (Basel). 6 (3), 1408-1440 (2014).
- Caldenhoven, E., et al. STAT3beta, a splice variant of transcription factor STAT3, is a dominant negative regulator of transcription. J Biol Chem. 271 (22), 13221-13227 (1996).
- Zammarchi, F., et al. Antitumorigenic potential of STAT3 alternative splicing modulation. Proc Natl Acad Sci U S A. 108 (43), 17779-17784 (2011).
- Liu, Y., Beyer, A., Aebersold, R. On the Dependency of Cellular Protein Levels on mRNA Abundance. Cell. 165 (3), 535-550 (2016).
- Narimatsu, M., et al. Tissue-specific autoregulation of the stat3 gene and its role in interleukin-6-induced survival signals in T cells. Mol Cell Biol. 21 (19), 6615-6625 (2001).
- Szafranski, K., et al. Physiological state co-regulates thousands of mammalian mRNA splicing events at tandem splice sites and alternative exons. Nucleic Acids Res. 42 (14), 8895-8904 (2014).
- Sundin, M., et al. Novel STAT3 mutation causing hyper-IgE syndrome: studies of the clinical course and immunopathology. J Clin Immunol. 34 (4), 469-477 (2014).
- Oltean, S., Bates, D. O. Hallmarks of alternative splicing in cancer. Oncogene. 33 (46), 5311-5318 (2014).
- Boultwood, J., Dolatshad, H., Varanasi, S. S., Yip, B. H., Pellagatti, A. The role of splicing factor mutations in the pathogenesis of the myelodysplastic syndromes. Adv Biol Regul. 54, 153-161 (2014).
- Tilgner, H., et al. Comprehensive transcriptome analysis using synthetic long-read sequencing reveals molecular co-association of distant splicing events. Nat Biotechnol. 33 (7), 736-742 (2015).
- Hiller, M., et al. Widespread occurrence of alternative splicing at NAGNAG acceptors contributes to proteome plasticity. Nat Genet. 36 (12), 1255-1257 (2004).
- Rosenfeld, J. A., Malhotra, A. K., Lencz, T. Novel multi-nucleotide polymorphisms in the human genome characterized by whole genome and exome sequencing. Nucleic Acids Res. 38 (18), 6102-6111 (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved