Method Article
La fusion absolue et relative des données quantitatives PCR pour Quantifier STAT3 Splice Variant Transcriptions
Dans cet article
Résumé
Tandem splicing events occur at sites less than 12 nucleotides apart. Quantifying ratios of such splice variants is feasible using an absolute quantitative PCR approach. This manuscript describes how splice variants of the gene STAT3, in which two splicing events results in Serine-701 inclusion/exclusion and α/β C-termini, can be quantified.
Résumé
Human signal transducer and activator of transcription 3 (STAT3) is one of many genes containing a tandem splicing site. Alternative donor splice sites 3 nucleotides apart result in either the inclusion (S) or exclusion (ΔS) of a single residue, Serine-701. Further downstream, splicing at a pair of alternative acceptor splice sites result in transcripts encoding either the 55 terminal residues of the transactivation domain (α) or a truncated transactivation domain with 7 unique residues (β). As outlined in this manuscript, measuring the proportions of STAT3's four spliced transcripts (Sα, Sβ, ΔSα and ΔSβ) was possible using absolute qPCR (quantitative polymerase chain reaction). The protocol therefore distinguishes and measures highly similar splice variants. Absolute qPCR makes use of calibrator plasmids and thus specificity of detection is not compromised for the sake of efficiency. The protocol necessitates primer validation and optimization of cycling parameters. A combination of absolute qPCR and efficiency-dependent relative qPCR of total STAT3 transcripts allowed a description of the fluctuations of STAT3 splice variants' levels in eosinophils treated with cytokines. The protocol also provided evidence of a co-splicing interdependence between the two STAT3 splicing events. The strategy based on a combination of the two qPCR techniques should be readily adaptable to investigation of co-splicing at other tandem splicing sites.
Introduction
Courte portée (tandem) d'épissage alternatif, où les sites accepteurs ou donneurs de remplacement sont à proximité les uns des autres, est fréquente chez les mammifères, les invertébrés 1 2 et 3 plantes. On estime que 20% des gènes de mammifères contiennent des sites d'épissage alternatif de 2-12 nucléotides en dehors 4. Beaucoup de ces sites sont 3 nucléotides à part et donnent lieu à l'inclusion ou l'exclusion d'un seul codon. Il y a un débat sur la nature de la réglementation de l' épissage sur ces sites 5,6 avec certains estimant que les différences épissage motifs sont si subtiles que la sélection est stochastique 7, tandis que d' autres en déduisent une réglementation fondée sur la spécificité des tissus 8.
Tandem sélection de site d'épissage a été analysé semi-quantitativement par électrophorèse capillaire modifiée 7, et le gel à haute résolution électrophorèse 8. ARN-Seq (séquençage de l'ARN) lit peut être utilisé pour quantifier les ratios d'épissage à chaque épissuresite. De cette façon, les données de l' ARN-Seq a donné un aperçu de la réglementation des sites d'épissage tandem 9. Il a également permis la prédiction des rapports variants d'épissage attendus sur la base nucléotidique motif 10. La plupart de l'accent mis sur l'épissage qui inclut ou exclut un seul codon a été mis sur les survenant plus fréquemment tandem accepteur sites d'épissage, appelés NAGNAGs (où N = nucleotide quelconque).
sites d'épissage alternatifs Tandem donateurs incluant ou excluant un seul codon (GYNGYN motif de reconnaissance, où Y = pyrimidine) sont moins fréquentes que les accepteurs en tandem. Transducteur de signal et activateur de la transcription 3 (STAT3) est un gène clé subissant tandem donneur épissage alternatif 1,11. Les sites de tandem donneur d'épissage rejoindre exons 21 et 22 et se traduisent par l'inclusion ou l' exclusion du codon pour Serine-701 (S ou ΔS respectivement) 1,11. en aval des sites alternatifs d'accepteur (40 nucléotides espacées les unes des autres) de jonction des exons 22 et23a / b en raison de l'inclusion soit des 55 résidus terminaux du domaine de transactivation (de α) ou un domaine de transactivation tronqué avec 7 résidus C-terminaux uniques (ß) 11. Par conséquent, quatre variants d'épissage sont possibles.
La protéine STAT3 est un facteur majeur de la transcription et l' intégrateur de signal dans de nombreux types cellulaires 12 et lorsqu'ils sont mutés son activation constitutive contribue à plusieurs phénotypes cancéreux (revue dans la référence 13). Syndrome de Job, un trouble de l' immunodéficience caractérisée par des niveaux élevés d'IgE, est également causée par des mutations dans STAT3 (revue en référence 14). Rôles distincts pour α STAT3 et protéines variantes β d'épissage ont été décrites précédemment 15. Dans un premier temps , STAT3 β a été pensé pour agir d'une manière négative dominante 16, antagoniser l' activité transcriptionnelle de STAT3 de α, mais les travaux ultérieurs suggéré qu'il a des gènes cibles indépendants 17 . Malgré la subtilité du tandem épissage, il y a lieu de croire que l'absence ou la présence de sérine-701 fonction (Ser701) des influences. Non seulement est Ser701 à proximité de Tyrosine-705 (le résidu phosphorylée dans l' activation de STAT3 18), mais une étude récente suggère que les variants d'épissage STAT3 S et ΔS sont tous deux nécessaires pour la viabilité du lymphome STAT3-addicted Diffuse Grand cellules B (DLBLCL) 19 cellules. La pertinence biologique reste à explorer. Étant donné que la composition de variant d'épissage pourrait influer sur la fonction, nous nous sommes efforcés de découvrir si le rapport a été perturbée par la stimulation des cytokines dans les éosinophiles.
Nous avons d' abord tenté d'explorer le lien entre les deux événements d'épissage en utilisant une PCR spécifique pour α STAT3 et variants d'épissage β, suivie d' un clivage de produits avec une enzyme de restriction spécifique pour les variants d'épissage S AFEI. Densitométrie des produits a indiqué l'inclusion de Ser701 a été roughly dix fois plus fréquente que son omission (ΔS) dans les deux α STAT3 et β (données non présentées). Cependant, cette approche semi-quantitative n'a pas été suffisamment reproductible, et ne pouvait être utilisé efficacement pour mesurer les quatre variants d'épissage simultanément. Pour analyser les proportions de chacun des quatre variants d'épissage, il était nécessaire d'établir une PCR (qPCR) protocole quantitatif qui a donné serrés techniques (plusieurs essais d'un échantillon donné) réplique.
QPCR relative repose sur la comparaison d'un gène d'intérêt à un gène standard ou le ménage connu pour être exprimé à un niveau particulier 20 et est appropriée lorsque le gène d'intérêt et le gène de ménage sont amplifiés avec une efficacité similaire. Un brin (ds) fluorescent (cyanine) , un colorant de double liaison à l' ADN se lie aux amplicons PCR 21, et après un certain nombre de cycles, une amplification suffisante pour la fluorescence est produite pour être détectable. Plus le niveau initialde la transcription, plus le cycle seuil (Ct) valeur. Puisque la concentration de préparations d' ADNc diffère, il faut comparer la concentration de la transcription avec la concentration d'une transcription connue pour être exprimée à un niveau constant dans tous les échantillons, comme glucuronidase-β (GUSB) en éosinophiles 22.
qPCR relative est impossible pour des séquences très similaires, comme le montre les variants d'épissage résultant d'un épissage en tandem. Les conditions strictes requises pour amplifier spécifiquement les variantes d'épissage conduisent à une diminution du rendement. Au lieu de cela, la quantification absolue doit être utilisé 23. Cela implique la préparation d' une courbe standard avec des concentrations connues de la transcription épissé d'intérêt, et les conditions de PCR assurant optimiser la spécificité 24. Comme décrit, les données absolues et relatives qPCR pour un gène particulier peuvent être fusionnés pour informer la compréhension de l'expression du gène dans un type cellulaire particulier,ce cas STAT3 dans les éosinophiles diversement stimulées 25.
Ici, STAT3 variant d'épissage de quantification est décrit dans l'espoir que le procédé peut être adapté à des études ciblées en tandem d'autres événements d'épissage. L'optimisation est un processus long, où plusieurs paires d'amorces à diverses concentrations et de nombreuses itérations de paramètres de cycle ont été testés au cours de quelques mois. Les principales caractéristiques du protocole sont la validation de la spécificité primaire et quantification basée sur les courbes standard avec des concentrations connues des variants d'épissage. qPCR relative en collaboration se sont révélées utiles pour notre application mais pas nécessaire.
Protocole
NOTE: éosinophiles du sang périphérique ont été reçues sans identifier l'information en accord avec un protocole approuvé (# 2013-1570) par l'Université de Wisconsin-Madison Centre des sciences de la santé Institutional Review Board. Signé le consentement éclairé du donneur a été obtenu pour l'utilisation de chaque échantillon dans la recherche.
1. Création d'Plasmides comme normes de modèle
- Créer des amorces pour un amplicon comprenant les deux sites d'épissage STAT3, avec 5 ' des sites Kpnl et Nhel restriction (et des extensions 5') selon le Tableau 1a.
REMARQUE: Les sites Kpnl et Nhel ont été choisies pour permettre à des inserts à ligaturer dans un vecteur pET-28a modifié avec résistance à la kanamycine 25,26 site Kpnl avait été ajouté dans le site de clonage multiple.. - En utilisant les éosinophiles fraîchement donnés, préparer des échantillons en double de 2,5 x 10 6 cellules / ml dans un milieu de culture cellulaire (Roswell Park Memorial 'Institut (RPMI) 1640). Incuber pendant 1 heure à 37 ° C sous 5% de CO2 et 10% d' humidité.
- Préparer (c) un ADN complémentaire à partir d' échantillons (au moins 1 x 10 6 cellules par préparation) selon les instructions du fabricant en utilisant l'extraction de l' ARN et des kits de synthèse d' ADNc.
- À partir du modèle d' ADNc préparé à l' étape 1.2.1, amplifier STAT3 variants d' épissage en utilisant une amplification PCR selon le tableau 2a.
- Resolve amplicons dans 2% d' agarose Tris-acétate-éthylènediaminetétraacétate (TAE) gel 27. bandes d'accise du gel et purifient selon gel instructions du kit d'excision.
- Amplicons coupées et plasmide avec des enzymes de restriction appropriées (vue d'ensemble de la restriction en référence 27), en utilisant des volumes, des temps d'incubation et les températures recommandées par le fournisseur d'enzyme; et purifier comme dans l'étape 1.4. Ligaturer amplicons restreintes dans le plasmide dans des conditions de fournisseurs recommandés.
- Préparer une suite négativerol (ADN plasmidique restreint sans insert mais avec ligase) et un contrôle positif (plasmide sans restriction avec résistance à la kanamycine). Effectuer des transformations plasmidiques standard de E. compétente coli DH5a 28 utilisant Les mélanges de ligature 27.
- Incuber transformé E. coli dans 1 ml Luria-Broth (LB) (préparé selon les instructions 27) agitation pendant 1 heure à 37 ° C. transformants Spread sur des plaques LB contenant 50 pg / ml de kanamycine et incuber à 37 ° C pendant la nuit.
- Choisissez plusieurs colonies à partir des α STAT3 et plaques STAT3 ß en utilisant des cure - dents stériles 27. Transfert chacun 2 ml LB. Cultivez cultures durant la nuit à 37 ° C dans un incubateur à agitation.
- On purifie les plasmides à partir de cultures bactériennes en suivant les instructions fournies dans le kit de purification d'ADN. plasmides Conserver à -20 ou -80 ° C.
- plasmides de séquence de plusieurs colonies en préparant 20 réactions de séquençage ul. pipette 3un tampon de réaction de séquençage ul, 2 ul de mélange réactionnel de séquençage, de l'eau ultrapure 12 ul, 1 ul de plasmide comme matrice et 2 pi amorce de séquençage.
NOTE: Si vous utilisez vecteur pET-28a, utiliser une amorce de séquençage 5'-TAA TAC GAC TCA CTA TAG GGG-3 '.- Évaluer électrophorétogrammes de plasmides codant pour chaque variante: Sα, Sβ, ΔSα et ΔSβ 25.
- Mesurer la concentration de l'ADN plasmidique pur en ng / ul. Si la lecture de l' absorbance à 260 nm en utilisant un spectrophotomètre, calculer la concentration d'ADN en utilisant la loi de Beer-Lambert 29:
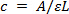
Où c = concentration, A = absorbance, ε (coefficient d'extinction) = 0,02 (pg / ml) -1 cm -1 pour l' ADN et L = longueur de trajet double brin de lumière (généralement 1 cm). - En utilisant les poids moléculaires de STAT3 variant d' épissage d' ADNc amplicons et le vecteur, calculer le nombre de copies par ml.
NOTE: Le poids moléculaire par paire de bases (pb) est estimée à 650 Da.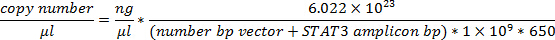
2. Analyse Primer Spécificité pour Absolute qPCR
- Préparer 1 à 10 série de dilutions de plasmides STAT3, avec 10 ~ 8 à 10 copies par 3 ul (20 ul) avec de l' eau ultrapure. Concentration de la mesure de la plupart concentrées (~ 10 8 copies par ml, voir l' étape 1.11).
- Utilisez plasmides dilué pour préparer quatre mélanges "non-cibles" (ie., STAT3 ΔSβ contrôle négatif contenant des concentrations égales de STAT3 Sα, ΔSα, Sβ mais pas ΔSβ) à 10 6 copies par ml chacun. Préparer un mélange "cible" avec des concentrations égales de tous les quatre modèles chacun à 10 6 copies par ml.
- Préparer la solution de paire d'amorcess pour chaque variante d'épissure (voir le tableau 1b et à la figure 1) en faisant ~ 60 ul d'amorces , chacun à une concentration de 7 uM (concentration finale dans l' échantillon sera de 560 nM).
- Diluer l' ADN polymérase / dsDNA liaison colorant mélange 7: 5 avec pur H 2 O.
- Mettre en place l' essai 1 dans une plaque à 96 puits par PCR comme indiqué dans le modèle (tableau 3).
- Ajouter 21 ul dilué l' ADN polymérase / dsDNA liaison colorant mélange, puis 2 pi de mélange d' amorces et 2 pi de modèle (selon le tableau 2b). Au lieu de modèle, ajoutez 2 pl filtrés H 2 O à la commande sans modèle (NTC) ainsi. Mettre en place des réactions en double afin d' évaluer la répétabilité 30.
- plaque Seal qPCR avec couvercle adhésif et centrifuger pendant 5 min à 1200 g à 12 ° C.
- Si vous utilisez des logiciels répertoriés sur les matériaux, mis en place l' expérience comme décrit.
- Allumez la machine qPCR et insert étanche plaque qPCR. Cliquez sur Fichier &# 62; Nouveau> Suivant> Sélectionnez "Nouveau détecteur", choisissez reporter, désaltérante et fournir le nom. Mettre en place un "nouveau détecteur" pour chaque variante d'épissage.
- Sélectionnez Suivant et mettre en place des normes comme demandé sur la page de la plaque de l'échantillon Set Up, assurant des quantités sont entrées dans la table et des détecteurs appropriés sont sélectionnés. Cliquez sur Terminer.
- Mettre en place le cyclisme selon le tableau 2b. Faire en sorte que la fluorescence de la lecture (pour déterminer Ct) se produit au cours de l'étape C à 72 ° de chaque cycle. Sélectionnez Exécuter.
- Lorsque l'exécution est terminée, cliquez sur le> onglet parcelle Amplification onglet Résultats. Évaluer parcelle d'amplification de sortie pour évaluer la qualité des données (chercher une parcelle exponentielle, comme dans la figure 2).
NOTE: Assurez-parcelles d'amplification sont essentiellement exponentielle et que le seuil de cycle peut être fixé à la partie exponentielle de la parcelle. Veiller à CTN ne sont pas amplifiées et d'autres points standard démontrent une faible valeur Ct avec un haut ARn. - Pour export résultats au logiciel de feuille de calcul, cliquez sur Fichier> Exporter> Résultats.
3. Évaluation absolue qPCR Assay Spécificité et répétabilité
- Répétabilité
- Utilisez les données de l' étape 2.7.5 pour calculer l' écart type de Ct (cycle seuil pour fluorescence) valeurs.
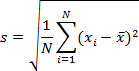
Où N = nombre d'échantillons (2 si elle est faite en double exemplaire),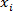 = Ct et de l' échantillon
= Ct et de l' échantillon 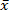 = Échantillon Ct signifie. Vérifiez que l'écart type des valeurs Ct de doublons est ≤0.2. Vérifiez que les valeurs de Ct dans tous les puits , mais NTC sont plus petits que 38.
= Échantillon Ct signifie. Vérifiez que l'écart type des valeurs Ct de doublons est ≤0.2. Vérifiez que les valeurs de Ct dans tous les puits , mais NTC sont plus petits que 38. - Si la répétabilité est mauvais paramètres optimize de cyclisme en augmentant ou en diminuant le temps de recuit.
- Utilisez les données de l' étape 2.7.5 pour calculer l' écart type de Ct (cycle seuil pour fluorescence) valeurs.
- Plot copie du journal engourdieer (comme déterminé à l' étape 1.12 et préparé à l' étape 2.1) vs valeur Ct pour créer une courbe standard; donnant l'équation suivante:
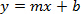
Où y est Ct, m est la pente, x est log (nombre de copies) et b est la valeur d'interception.
NOTE: La R 2 valeur de la régression linéaire sera ≥0.95 de bonnes données. - Calculer l'efficacité d'amplification en utilisant la courbe standard et l'équation:
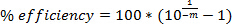
NOTE: Viser l'efficience ≥75%. - Calculer la spécificité de qPCR test 1 en utilisant la formule de trop:
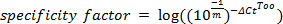
Où Δ Ct Trop = Ct cible - Ct non-cible, décrivant la différence dans le nombre de cycle seuil pour ampli spécifique et non spécifiquelification
NOTE: Spécificité devrait dépasser quatre ordres de grandeur (ie, le facteur de spécificité ≥4.).- Si la spécificité est faible, d'optimiser en abaissant la concentration de l'amorce. Mettre en place des essais (comme décrit dans les étapes 2,5-2,7) avec des concentrations d'amorces finales allant de 100 à 500 nm.
4. Exécution relative dosages qPCR
- Traiter les cellules avec Cytokines pour favoriser la transcription.
- Pour les éosinophiles fraîchement donnés, préparer des échantillons en double de 2,5 x 10 6 cellules / ml dans un milieu de culture cellulaire (RPMI 1640) et traiter avec 50 ng / ml d' interleukine-3 (IL3) et / ou 50 ng / ml de facteur α de nécrose tumorale ( TNFa) pendant des périodes de 3, 6, 9 et 20 heures à 37 ° C sous 5% de CO2 et 10% d' humidité.
- Préparer l' ADNc à partir d' échantillons d'éosinophiles (au moins 1 x 10 6 cellules par préparation) selon les instructions du fabricant en utilisant les kits d'extraction d'ARN et synthèse d'ADNc.
- Préparer des dilutions en série de deux aliquotes échantillon d'ADNc (contrôle ou échantillons au repos), avec une dilution allant de "1" à la dilution "1 en 1024".
- Pour la dilution 1 à 4, la pipette 1 pi d'ADNc et 3 pi d'eau ultrapure.
- Pour la dilution de 1 sur 16, pipette 1 pl de la 1 à 4 dilution et de l' eau ultrapure 3 pi, etc.
- Mettre en place la PCR en plaque de 96 puits avec 25 ul de réactions que les étapes décrites 2.3-2.5 mais avec plusieurs concentrations d' amorces pour STAT3 pan- et GUSB (voir modèle dans le tableau 4).
- Ajouter "nouveau détecteur" pour GUSB et suivez les étapes 2,6-2,7, en utilisant les paramètres de cycle décrits dans le tableau 2c.
- Générer des courbes standard (voir les étapes 3.2 et 3.3) pour déterminer les concentrations d'amorces aboutissent à 95-100% d'efficacité d'amplification.
- Mettre en place la plaque avec des échantillons préparés d'ADNc (de l'étape 4.2) et des concentrations d'amorces optimisées(voir le tableau 2c pour les paramètres de cyclisme, les réactifs et le tableau 5 96 puits modèle de plaque).
- Effectuer les étapes 2,6-2,7. Refaire le test au moins une fois et de vérifier la qualité des données.
- Calculer la valeur Ct moyenne de l' échantillon en double Cts pour les deux STAT3 et GUSB de gène de ménage.
- Obtenir des valeurs Δ Ct pour chaque échantillon.
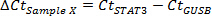
- Désigner échantillon de repos (a généralement plus faible concentration de transcrit d'intérêt) que l' échantillon 0. Calculer Ct 31 pour chaque échantillon (ici désigné comme échantillon X) ΔΔ:
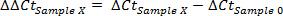
- Calculer fois d'augmentation pour chaque échantillon:
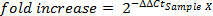
- Reproductibilité
- Calculer le coefficient de variation (CV) du nombre de copies pour pan- STAT3 et GUSB.
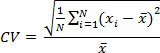
Où N = nombre d'échantillons, = calculé le nombre de copies de l' échantillon et = moyenne de l' échantillon. Vérifier que CV de chaque échantillon (plusieurs essais) est ≤10%.
- Calculer le coefficient de variation (CV) du nombre de copies pour pan- STAT3 et GUSB.
5. Analyse de Absolute qPCR données pour les échantillons inconnus
- Comparer les valeurs Ct obtenues en rapport qPCR (tel que décrit à l' étape 4.9) pour GUSB de gène de ménage pour assurer les concentrations d' ADNc de échantillons sont dans un ordre de grandeur.
- Diluer ADNc en conséquence (par exemple, si l' échantillon 1 a une valeur Ct de ~ 17,5, et les valeurs de Ct d' autres échantillons de sont ~ 21, l' échantillon 1 est à peu près 2 (21-17.5) = 11 fois plus concentré Effectuez un 1: 1. Dilution avec eau ultrapure dans la même gamme sans diluer plus que nécessaire).
NOTE: Considérablement différentes concentrations de départ seront biais de l'analyse de régression linéaire (étape 4.12).
- Diluer ADNc en conséquence (par exemple, si l' échantillon 1 a une valeur Ct de ~ 17,5, et les valeurs de Ct d' autres échantillons de sont ~ 21, l' échantillon 1 est à peu près 2 (21-17.5) = 11 fois plus concentré Effectuez un 1: 1. Dilution avec eau ultrapure dans la même gamme sans diluer plus que nécessaire).
- Mettre en place absolue test qPCR d'échantillons. Préparer dosage plaque de modèle de 2a pour Sα et Sβ selon le tableau 6; et préparer dosage 2b pour ΔSα et ΔSβ selon le tableau 7. Effectuer des essais comme décrit dans les étapes 2.6-2.7.3 (et le tableau 2b). Répéter les essais au moins une fois.
- Vérifiez la qualité des données et l'exportation comme décrit dans les étapes 2.7.4-2.7.5.
- Mettre en place absolue qPCR plaque de 96 puits avec 25 ul de réactions en utilisant des amorces pan- STAT3 (les amorces dans le tableau 1c, modèle dans le tableau 8) à 400 uM et un mélange des quatre plasmides ou un plasmide unique à des concentrations totales inscrites.
NOTE: L'efficacité n'a pas d'importance pour qPCR absolue, mais l'optimisation de l'efficacité de ces amorces est important pour qPCR par rapport (étape 4). - plaque Seal qPCR avec couvercle adhésif et centrifuger pendant 5 min à 1200 g à 12 ° C.
- Mettre en place "nouveau détecteur" pour pan- STAT3 comme dans les étapes 2.7.1-2.7.3.
- Mettre en place le vélo pour pan- STAT3 selon le tableau 2c (mêmes conditions que par rapport qPCR). Faire en sorte que la fluorescence de la lecture (pour déterminer Ct) se produit au cours de l'étape C à 72 ° de chaque cycle. Refaire le test au moins une fois pour assurer la reproductibilité.
- Vérifiez la qualité des données et l'exportation (voir les étapes 2.7.4-2.7.5).
- Si les parcelles d'amplification ne sont pas exponentielle (voir la figure 2), diluer l' ADNc de l' échantillon (1:10) et répéter dosage comme décrit (étape 5.7).
- En utilisant l'équation de la courbe standard (voir 3.2, Figure 3) et les valeurs Ct des échantillons, calculer le nombre de copies de chaque variant d'épissage et de STAT3 pan- dans chaque échantillon.
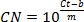
- Journal de la parcelle (qPCR valeurs du nombre de copies absolues obtenues pour pan- STAT3) vs log (qPCR valeurs du nombre de copies absolues cumulées pour STAT3 variants d' épissage).
NOTE: régression linéaire Idéal et la pente des valeurs seraient tous deux ~ 1. - Calculer la répétabilité (étape 3.1) et la reproductibilité (étape 4.13).
6. La fusion absolue et relative des données qPCR
- Multipliez la proportion de la variante avec le STAT3 pli-augmentation totale pour calculer un facteur d'augmentation de chaque variant d'épissage.
- Calculer les écarts-types (voir l'étape 3.1.1) des valeurs absolues et relatives pour tenir compte de la propagation de l'erreur. Déterminer l'erreur de mesure standard (SEM), comme indiqué:
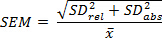
Résultats
De bonnes données qPCR de qualité vont générer une courbe d'amplification sigmoïde (Figure 2a), ce qui signifie augmentation exponentielle dans les transcriptions au cours du cyclisme. La présence de trop de modèle peut se traduire par un fond de fluorescence élevée, ce qui signifie une base de référence inappropriée est établie dans les premiers cycles. Si les données ne fournissent pas une courbe exponentielle (Figure 2b), une optimisation plus poussée est nécessaire (décrite dans les étapes 3.1 et 3.4). Pour de plus amples informations sur la résolution des résultats de qPCR, reportez - vous à la référence 32. Les courbes d' étalonnage obtenues pour les plasmides modèle d'étalonnage indiquent l'efficacité de l' amplification (courbe pour STAT3 Sα représenté sur la figure 3). Efficacités entre 83 et 95% ont été observées dans les conditions décrites. L'équation pour la spécificité (étape 3.4) suppose égale efficacité, ce qui est peu probable 25, de sorte que la spécificitéest susceptible d'être plus grande que le facteur de spécificité indique.
Afin d'évaluer la congruence des niveaux STAT3, les valeurs absolues de chacune des quatre variants d'épissage ont été mesurés, ainsi que le niveau de STAT3 totale, celle - ci en utilisant des amorces d' amplification d' une région commune aux quatre variantes d'épissage (figure 4). Idéalement , la régression linéaire (indication de corrélation) et la pente (rapport de STAT3 pan- à des variants d'épissage additionnées) devraient tous deux être proche de 1.
Les données absolues qPCR sont présentées comme des graphiques circulaires pour montrer les proportions des quatre variants d'épissage au fil du temps post-stimulation avec des cytokines (figure 5a). Éosinophiles repos (0 h) avaient la plus faible proportion de STAT3 Sα, bien que cette variante était toujours le plus abondant. Multipliant la fraction de STAT3 variants d'épissage β (S ^6; + ΔSβ) par la fraction des variants d'épissage STAT3 ΔS (+ ΔSα ΔSβ) a donné une valeur constante inférieure à la valeur expérimentale enregistrée pour ΔSβ. Si les événements d'épissage étaient indépendants, on pourrait penser que la multiplication fraction de ΔS variantes par la fraction de variantes ß donnerait une valeur qui correspond à la valeur déterminée expérimentalement. Des niveaux plus élevés de ΔSβ que prévu des événements d'épissage indépendants suggère un biais co-épissage existe.
Fusion des données qPCR absolue et relative a démontré que les niveaux de tous les variants d'épissage de STAT3 augmenté post-stimulation avec des cytokines IL3 et TNFa, avec des niveaux pic 6 h post-stimulation (Figure 5b - e). Pour trois des quatre variants d'épissage, les niveaux de transcription étaient environ 3 fois plus élevé chez les éosinophiles traités IL3 + TNFa (6 h) par rapport à éosinophiles dans les médiasau même point de temps. Les niveaux STAT3 Sα étaient 3,5 fois plus élevé dans IL3 + TNFa éosinophiles traités par rapport à éosinophiles dans les médias à ce point de temps. La plus grande incertitude ( la plus grande erreur de mesure standard) a été observée dans ΔSβ (Figure 5e), qui comprend la plus petite fraction du total STAT3 dans tous les échantillons. Ce ne fut pas surprenant que les niveaux inférieurs sont associés à des valeurs Ct plus élevées. Exiger plus de cycles pour atteindre le cycle de seuil composé incertitude due à la variation du cycle à cycle dans l'efficacité de l'amplification.
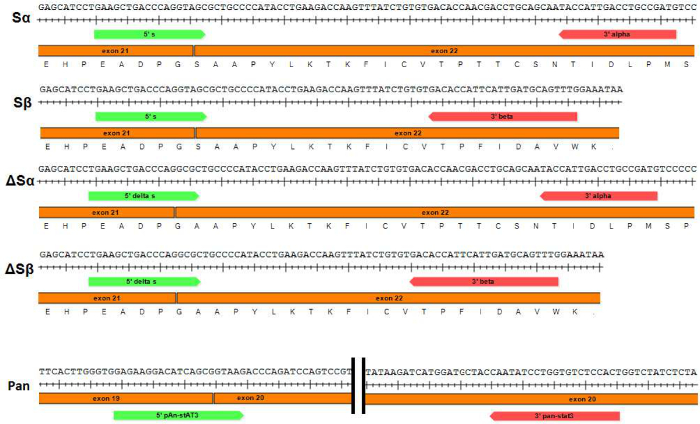
Figure 1: Représentation schématique des paires d'amorces utilisées pour effectuer la qPCR des variants d'épissage STAT3 et pan-STAT3 Les amorces utilisées pour amplifier spécifiquement chacune des variantes d'épissage STAT3 (Sα, Sβ, et ΔSα ΔSβ respectivement) s.hown. Amorces Forward (STAT3 "S" et "ΔS") couvrent la jonction entre les exons 21 et 22. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
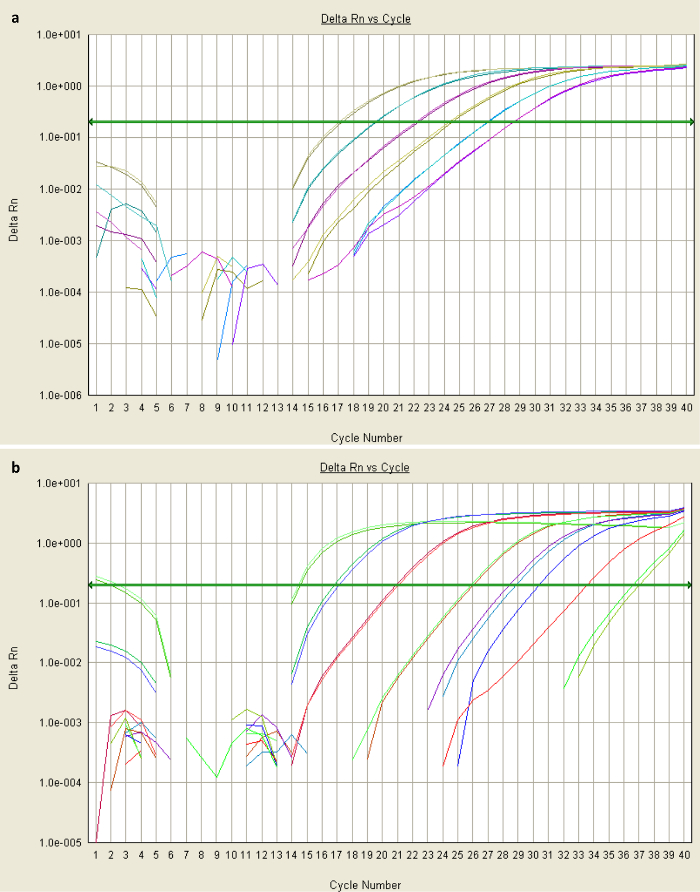
Figure 2:. Parcelles d'amplification de données qPCR (a) des parcelles d'amplification sigmoïde des moyens d' amplification fiable. Ces données ont été obtenues à partir qPCR de deux dilutions en série de plasmide contenant STAT3 Sα, chaque paire de lignes de couleur représentant les niveaux de fluorescence des échantillons en double dilution au cours de 40 cycles (axe des x). L'échantillon le plus concentré (gris-vert) a été suffisamment amplifié par cycle de 17 (avec colorant proportionnel à fluorescence, montré sur l'axe des y dsDNA liaison) pour dépasser la valeur de fluorescence de seuil (BaseliNE présentée comme flèche verte). Sa valeur Ct serait 17. (b) des parcelles non-exponentielles suggèrent que le seuil d' arrière-plan de fluorescence n'a pas été correctement mis en place dans les premiers cycles. Cela pourrait être dû à la présence d'un inhibiteur, ou un modèle hautement concentré ou amorces. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
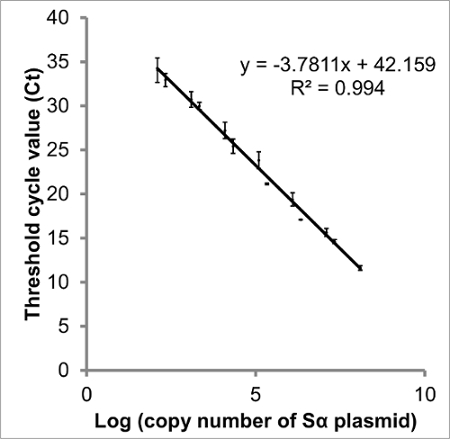
Figure 3: Courbe standard de journal (nombre de copies de STAT3 Sα) vs Ct Il existe une relation linéaire entre le log du nombre de copies et le seuil du cycle de chaque variant d'épissage STAT3 (Ct).. Création d'une courbe standard à partir d'ADN plasmidique imite l'ADNc d'échantillons et doncfournit une meilleure mesure qu'une courbe créée à partir de amplicons PCR dilué. Les données présentées sont des valeurs Ct obtenues à partir de qPCR de deux dilutions successives de plasmide contenant STAT3 Sα. A partir de cette courbe, le nombre de copies présent dans chaque échantillon peut être interpolée, et l'efficacité d'amplification calculée (83,9%). Bien que intersections de y sont moins reproductibles que la pente, l'interception suggère 42,2 cycles seraient nécessaires pour être certain pas d' ADN cible est présente. Les barres d'erreur indiquent SEM, n = 3. courbes comparables ont été construits pour STAT3 Sβ, ΔSα et ΔSβ (non représenté). S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
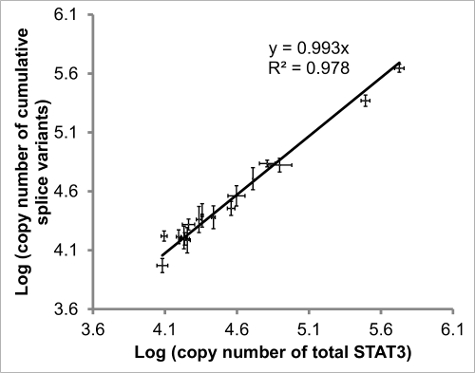
Figure 4: Comparaison des pan- quantifiée STAT3 vs variantes cumulatifs STAT3 d'épissage. La régression des ajouté épissage STAT3 variantes vs STAT3 totale devrait avoir la pente (rapport de pan- à cumulative) et R valeur 2 (corrélation) proche de 1. Les valeurs de 17 échantillons (éosinophiles et DLBCL) compris. Les barres d'erreur représentent la SEM de X à Y déterminations, n ≥ 2 pour chacun. Figure adapté de la référence 20. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 5:. Les niveaux de variants d'épissage STAT3 ont fluctué au cours du traitement de la cytokine (a) Les graphiques circulaires indiquant les pourcentages de chaque variante STAT3 d'épissuredes éosinophiles pendant le traitement par l'IL3 et TNFa. (B - e) Variation des variants d'épissage de STAT3 dans les éosinophiles traitées avec différentes combinaisons de cytokines, mesurées en combinant les données relatives et absolues qPCR Sα (b), Sβ (c), ΔSα (d), et ΔSβ (e) niveaux STAT3. fluctué au fil du temps post-stimulation. Niveaux d'abord augmenté, atteignant un sommet de 6 heures après la stimulation. La combinaison IL3 + TNFa a suscité une expression plus élevée de tous les variants d'épissage quatre STAT3 que IL3 seul. SEM calculée pour chaque point de données comptables pour la propagation de l' erreur. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
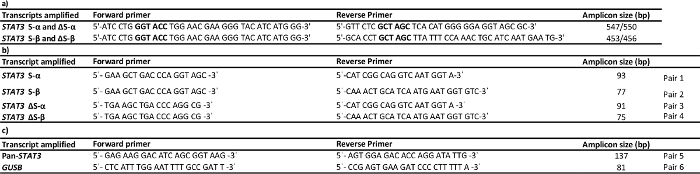
Tableau 1: Les amorces utilisées pour l' amplification (a), absolu (b) et relatif (c) PCR quantitative. Amorces de clonage ont des séquences de restriction et 5'-extensions pour une coupe efficace. Sites de restriction Kpnl et Nhel sont indiqués en gras. S'il vous plaît cliquer ici pour voir une version plus grande de cette table.
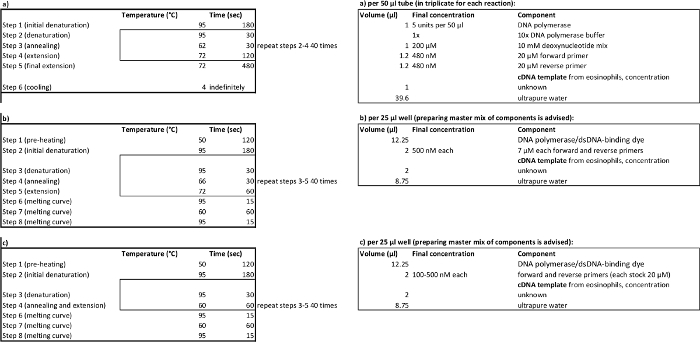
Tableau 2. Paramètres de cyclisme PCR ( à gauche) et des volumes de réactifs ( à droite) pour l' amplification (a), relative (b), absolue (c) PCR quantitative. S'il vous plaît cliquer ici pour voir une version plus grande de cette table.
3 / 54473tbl3.jpg "/>
Tableau 3:. Modèle pour absolue qPCR test d'étalonnage plasmidique Ce test est nécessaire pour évaluer la reproductibilité et l' efficacité, ainsi que de générer des courbes standard à partir de laquelle interpoler des données. Les mélanges "non-cibles" donnera une estimation de la spécificité. L' optimisation peut être nécessaire pour assurer la cohérence. S'il vous plaît cliquer ici pour voir une version plus grande de cette table.
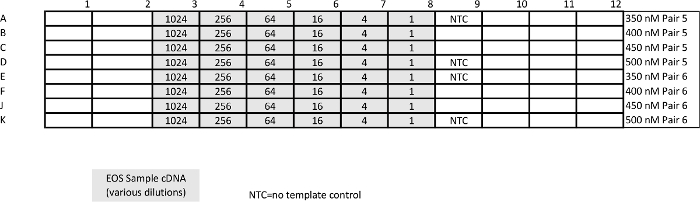
Tableau 4:. Modèle de qPCR relative (STAT3 pan- et d' entretien ménager gène GUSB) d'essai d'étalonnage Contrairement qPCR absolue, le point de ce test est de déterminer les conditions dans lesquelles l' efficacité d'amplification est ~ 100%.tp_upload / 54473 / 54473tbl4large.jpg "target =" _ blank "> S'il vous plaît cliquer ici pour voir une version plus grande de cette table.
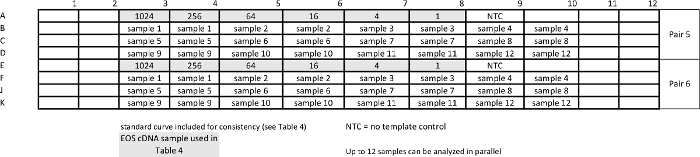
Tableau 5:.. Modèle de rapport analyse de l' échantillon qPCR pour mesurer pan- STAT3 et gène de ménage GUSB courbes standard sont répétées avec des échantillons pour assurer une efficacité comparable des essais S'il vous plaît cliquer ici pour voir une version plus grande de cette table.
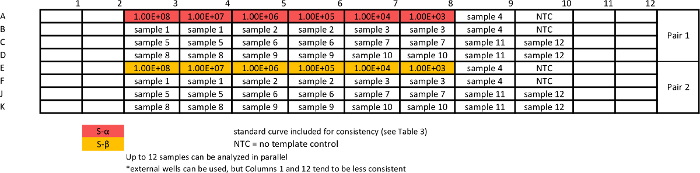
Tableau 6: Modèle d'absolu dosage de l' échantillon qPCR pour mesurer les variantes S courbes standard sont répétées avec des échantillons pour assurer eff comparable. cacité des tests. S'il vous plaît cliquer ici pour voir une version plus grande de cette table.
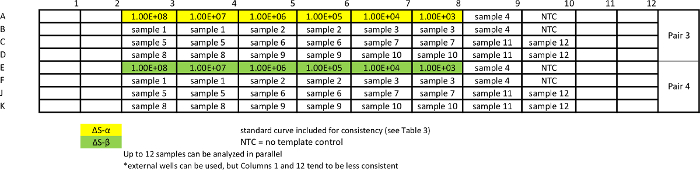
Tableau 7:.. Modèle pour absolue dosage de l' échantillon qPCR pour mesurer les variantes ΔS courbes standard sont répétées avec des échantillons pour assurer une efficacité comparable des essais S'il vous plaît cliquer ici pour voir une version plus grande de cette table.
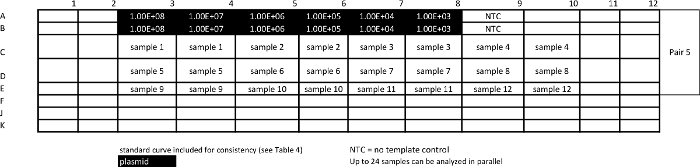
Tableau 8: Modèle d'absolu dosage de l' échantillon qPCR pour mesurer STAT3 pan-.large.jpg "target =" _ blank "> S'il vous plaît cliquer ici pour voir une version plus grande de cette table.
Discussion
Nous avons développé ce protocole afin d'évaluer les niveaux et les proportions de STAT3 variant d'épissage transcrits dans les éosinophiles et les cellules de lymphome et d'apprendre si la stimulation de cytokine a affecté les niveaux et les proportions. STAT3 est d' un intérêt particulier en raison de sa fonctionnalité pléiotropique et incertaine, avec des rapports contradictoires quant à savoir si elle agit comme une oncoprotéine ou suppresseur de tumeur dans le cancer (examiné en référence 33). Les différences de STAT3 α et β variant d'épissage fonction avaient été précédemment caractérisé 34,35, et notre protocole facilité une analyse knock-down / ré-expression qui suggère un besoin pour un rapport optimal entre S et ΔS transcriptions 19.
quantification précise des variants d'épissage distincts facilitera de nouvelles enquêtes de relation fonction de STAT3 hétérogène pour épissure composition variant. Le protocole intègre les données absolues et relatives qPCR, combinant la capacité de absoluté qPCR pour calculer des proportions variant d'épissage, et par rapport qPCR pour mesurer les changements dans l'expression globale de STAT3. Cette approche permet de distinguer des différences subtiles dans l'ordre et en même temps la mesure des rapports d'épissage sur deux sites d'épissage alternatif plus de 50 nucléotides d'intervalle. Déterminer les rapports des événements d'épissage individuellement aurait pas donné la conclusion remarquable qu'un biais co-épissage existait de telle sorte que les niveaux ΔSβ sont plus élevés que prévu si les utilisations des deux sites sont épissés au hasard 25.
Critique, absolue qPCR en utilisant des courbes d'étalonnage de plasmides permet une quantification (avec une efficacité sous-optimale) des variations d'épissage qui donnent lieu à des séquences très similaires. Nous nous attendons à un épissage subtil dosage qPCR roman devrait prendre environ deux mois pour optimiser. Les étapes clés dans le développement de tests sont la création de plasmides de STAT3 utilisés pour générer des courbes standard pour qPCR absolue; expérimentalementla détermination des séquences d'amorces optimales et paramètres de cycle pour assurer la spécificité et la reproductibilité; et l'intégration des données relatives qPCR dérivée de la quantification pan-STAT3 expression relative à l'expression de GAPDH. La corrélation du nombre de copies quantifiées par la quantification par rapport à STAT3 pan- cumulative (Sα + ΔSα + + Sβ ΔSβ) montre que le protocole donne des résultats fiables.
Une mise en garde de la technique est le vaste processus de validation. Il est nécessaire d'évaluer la variabilité intra-essai (répétabilité), la variabilité inter-essai (reproductibilité) et la spécificité. Le protocole décrit des façons d'obtenir des sorties numériques pour ces paramètres. Nous avons estimé que l' efficacité ≥75%, le facteur de spécificité ≥4, coefficient de variation (reproductibilité) ≤10% et Ct écart - type (répétabilité) ≤0.2 des seuils appropriés 30. Des mutations ou délétions dans la séquence d'acides aminés 1-690 STAT3 ne seront pas découveRouge par ce protocole, même si elles peuvent influencer les rapports d'épissage. Proportions de transcrits pourraient ne pas être proportionnelle à proteoform proportions 36.
Puisque les échantillons ont des quantités différentes à partir d'ADNc totale, qPCR absolue est adapté pour comparer le nombre de copies de variants d'épissage dans un échantillon, mais pas pour la comparaison inter-échantillon à moins couplé avec le parent qPCR utilisant un gène de ménage établi. La méthode décrite est conforme aux directives MIQE qPCR pour la reproductibilité 30. peut-être besoin d'être modifié pour obtenir des données reproductibles si un autre appareil est utilisé PCR paramètres de cycle et les concentrations d'amorces. spécificité parfaite est impossible sans compromettre l'efficacité de façon drastique, mais la cible a été amplifié plus efficacement par plus de quatre ordres de grandeur.
ADN linéaire est plus facilement amplifié que circulaire. Si un autre plasmide ne fournit pas des courbes standards satisfaisants (R 2 <; 0,95), considèrent linéariser le plasmide par restriction à un seul site avant quantification. Optimisation de qPCR est crucial pour obtenir des données de bonne qualité (Figure 1). La plupart des protocoles qPCR reposent sur deux étapes du vélo, et les machines sont optimisées en conséquence. chauffage non uniforme du bloc de chauffage peut être exacerbée en trois étapes à vélo, ce qui contribue à une mauvaise répétabilité. Les essais doivent être mis en place dans des conditions stériles avec des pointes de pipette et filtre l'eau ultrapure, idéalement dans une hotte à flux laminaire dédié. Parce que les contaminants peuvent conduire à des résultats incohérents, des essais doivent être mis en place dans des conditions stériles avec des pointes de pipette et filtre l'eau ultrapure, idéalement dans une hotte à flux laminaire dédié. Pour plus d' informations sur l' optimisation qPCR, reportez - vous à Bustin et al. 32
Quantifier STAT3 peut conduire à une plus grande perspicacité dans un certain nombre de contextes. STAT3 auto-régule sa propre expression 37, et le protocole décrit ci - dessus peut aiderd'élucider si les rapports de variants d'épissage de STAT3 contribuent à la régulation de cette boucle de rétroaction positive. Le protocole peut être utilisé pour étudier les changements dans les rapports de variants d'épissage comme observé dans des cellules à des densités différentes 38 ou au cours du développement: il est connu que α / ratio β des STAT3 changements au niveau protéique au cours de l' hématopoïèse 16. Sundin et al. découvert qu'un polymorphisme nucléotidique simple épissage biaisée intronique de l' exon 12 dans STAT3 d'un patient avec le syndrome de Job 39. Il est concevable que l'un des nombreux SNP présents dans les introns entre l'exon 21 et 22, ou de l'exon 22 et 23 peuvent contribuer à des rapports d'épissage de ΔS / S et α / β, respectivement. Le test peut être utilisé pour quantifier la transcription STAT3 dans les cellules cancéreuses, où des mutations ou des changements dans la régulation de l' épissage peuvent introduire un biais dans le processus d'épissage 40. Des mutations dans les facteurs d'épissage (comme SF3B1), comme observed dans les syndromes myélodysplasiques 41 peut également conduire à des changements qui peuvent être mesurés par ce protocole.
Plus largement, cette approche détecte spécifiquement co-association dans l'épissage, ce qui est impossible avec les classiques ARN-Seq, ni norme qPCR. Bien que le phénomène de l'épissage mutuellement exclusifs exon démontre la coordination des décisions d'épissage, le co-association d'autres événements d'épissage n'a pas été bien documenté. Une autre méthode récemment décrite, dans laquelle l' ARN-Seq a été modifié de manière à interroger ADNc pleine longueur, suggère des événements d'épissage éloignés sont plus co-dépendants qu'on ne le pensait 42.
STAT3 contient un site d'épissage donneur en tandem. Accepteur des sites d'épissage tandem sont plus fréquentes 43 et les principes du protocole décrit pourraient servir de point de départ pour le développement de tests pour la détection de coïncidence de NAGNAG épissage et d' autres événements d'épissage à moins de 200 nucléotides. Autres potenapplications tiels comprennent la quantification de coïncidence d'autres différences de séquences subtiles, comme indels ou doubles , triples polymorphismes / nucléotidiques 44.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
Les auteurs tiennent à remercier les National Institutes of Health-NHLBI pour la subvention de projet Programme sur le rôle des éosinophiles dans Airway Inflammation et remodelage: P01HL088584 (PI: N. Jarjour), et l'Université du Wisconsin Carbone Cancer Center et Département de médecine pour le financement intra-muros. Nous remercions Douglas Annis pour le clonage des quatre variantes de STAT3.
matériels
| Name | Company | Catalog Number | Comments |
| MJ Research PTC-200 Thermal Cycler | GMI | N/A | Used for standard PCR |
| 7500 Real-Time PCR System | Applied Biosystems | N/A | qPCR machine |
| GS-6R | Beckman Coulter | N/A | centrifuge for 96-well plates |
| Nanodrop 2000 sprectrophotometer | ThermoFisher Scientific | N/A | |
| RPMI-1640 medium | Sigma Aldrich | R8758 | cell culture medium |
| PfuTurbo DNA Polymerase | Agilent Technologies | 600410 | DNA polymerase for standard PCR |
| KpnI | New England Biosciences | R0142S | |
| NheI | New England Biosciences | R0131S | |
| SYBR Green PCR Master Mix | Qiagen | 330523 | qPCR, DNA polymerase/dsDNA-binding dye mix |
| Rneasy Mini Kit | Qiagen | 74204 | RNA extraction kit |
| SuperScript III First-Strand Synthesis System | Invitrogen (ThermoFisher Scientific) | 18080-044 | cDNA synthesis kit |
| Primers | Integrated DNA Technology | N/A | |
| NEBuffer 1.1 | New England Biosciences | B7201S | |
| GenePure LE Agarose | ISC BioExpress | E-3120-500 | component of TAE gel |
| Pipettors | Major lab suppliers (MLS) | N/A | |
| Filter pipette tips | Neptune Scientific | BT10XL, BT20, BT200 | |
| EU One Piece Thin Wall Plate | MidSci | ABI7501 | |
| ThermalSeal A Sealing Film | MidSci | TSA-100 | 96 well plate seal |
| pET-Elmer (variant of pET-28a) | Novagen; modified in Mosher lab | N/A | Details in PMID: 20947497 |
| Wizard Plus SV Minipreps DNA purification system | Promega | A1460 | Plasmid purification |
| BigDye Terminator v3.1 Cycle Sequencing Kit | ThermoFisher Scientific | 4337455 | Sequencing kit |
| QIAEX II Gel Extraction kit | Qiagen | 20021 | Amplicon purification |
| DH5α competent cells | ThermoFisher Scientific | 18265-017 | available from several providers, see PMID: 2162051 |
| kanamycin | Research Products International Corp. | K22000-5.0 | |
| Tris base | ThermoFisher Scientific | BP152-5 | component of TAE buffer |
| Acetic acid, glacial | ThermoFisher Scientific | A38C-212 | component of TAE buffer |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma Chemical Company (Sigma Aldrich) | E-5134 | component of TAE buffer |
| Bacto Tryptone | BD Biosciences | 211705 | component of Luria Broth |
| Bacto Yeast extract, technical | BD Biosciences | 288620 | component of Luria Broth |
| Sodium chloride | ThermoFisher Scientific | S271-10 | component of Luria Broth |
| Sodium hydroxide | ThermoFisher Scientific | SS255-1 | component of Luria Broth |
| Bacto Agar | BD Biosciences | 214010 | component of Luria Broth plate |
| Lasergene SeqBuilder | DNASTAR | Figure 1 generated using Lasergene SeqBuilder software version 12.2.0 (DNASTAR) |
Références
- Hiller, M., et al. Phylogenetically widespread alternative splicing at unusual GYNGYN donors. Genome Biol. 7 (7), R65 (2006).
- Treacy, M. N., et al. Twin of I-POU: a two amino acid difference in the I-POU homeodomain distinguishes an activator from an inhibitor of transcription. Cell. 68 (3), 491-505 (1992).
- Schindler, S., et al. Alternative splicing at NAGNAG acceptors in Arabidopsis thaliana SR and SR-related protein-coding genes. BMC Genomics. 9, 159 (2008).
- Szafranski, K., Kramer, M. It's a bit over, is that ok? The subtle surplus from tandem alternative splicing. RNA Biol. 12 (2), 115-122 (2015).
- Wang, M., et al. Alternative splicing at GYNNGY 5' splice sites: more noise, less regulation. Nucleic Acids Res. 42 (22), 13969-13980 (2014).
- Hiller, M., Platzer, M. Widespread and subtle: alternative splicing at short-distance tandem sites. Trends Genet. 24 (5), 246-255 (2008).
- Tsai, K. W., Lin, W. C. Quantitative analysis of wobble splicing indicates that it is not tissue specific. Genomics. 88 (6), 855-864 (2006).
- Tadokoro, K., et al. Frequent occurrence of protein isoforms with or without a single amino acid residue by subtle alternative splicing: the case of Gln in DRPLA affects subcellular localization of the products. J Hum Genet. 50 (8), 382-394 (2005).
- Bradley, R. K., Merkin, J., Lambert, N. J., Burge, C. B. Alternative splicing of RNA triplets is often regulated and accelerates proteome evolution. PLoS Biol. 10 (1), e1001229 (2012).
- Zheng, C. L., Fu, X. D., Gribskov, M. Characteristics and regulatory elements defining constitutive splicing and different modes of alternative splicing in human and mouse. RNA. 11 (12), 1777-1787 (2005).
- Schaefer, T. S., Sanders, L. K., Nathans, D. Cooperative transcriptional activity of Jun and Stat3 beta, a short form of Stat3. Proc Natl Acad Sci U S A. 92 (20), 9097-9101 (1995).
- Waitkus, M. S., et al. Signal integration and gene induction by a functionally distinct STAT3 phosphoform. Mol Cell Biol. 34 (10), 1800-1811 (2014).
- Srivastava, J., DiGiovanni, J. Non-canonical Stat3 signaling in cancer. Mol Carcinog. , (2015).
- Holland, S. M., et al. STAT3 mutations in the hyper-IgE syndrome. N Engl J Med. 357 (16), 1608-1619 (2007).
- Maritano, D., et al. The STAT3 isoforms alpha and beta have unique and specific functions. Nat Immunol. 5 (4), 401-409 (2004).
- Hevehan, D. L., Miller, W. M., Papoutsakis, E. T. Differential expression and phosphorylation of distinct STAT3 proteins during granulocytic differentiation. Blood. 99 (5), 1627-1637 (2002).
- Yoo, J. Y., Huso, D. L., Nathans, D., Desiderio, S. Specific ablation of Stat3beta distorts the pattern of Stat3-responsive gene expression and impairs recovery from endotoxic shock. Cell. 108 (3), 331-344 (2002).
- Stahl, N., et al. Choice of STATs and other substrates specified by modular tyrosine-based motifs in cytokine receptors. Science. 267 (5202), 1349-1353 (1995).
- Zheng, M., et al. A mix of S and DeltaS variants of STAT3 enable survival of activated B-cell-like diffuse large B-cell lymphoma cells in culture. Oncogenesis. 4, 184 (2016).
- Livak, K. J., Schmittgen, T. D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) Method. Methods. 25 (4), 402-408 (2001).
- Zipper, H., Brunner, H., Bernhagen, J., Vitzthum, F. Investigations on DNA intercalation and surface binding by SYBR Green I, its structure determination and methodological implications. Nucleic Acids Res. 32 (12), e103 (2004).
- Kelly, E. A., Liu, L. Y., Esnault, S., Quinchia Johnson, B. H., Jarjour, N. N. Potent synergistic effect of IL-3 and TNF on matrix metalloproteinase 9 generation by human eosinophils. Cytokine. 58 (2), 199-206 (2012).
- Whelan, J. A., Russell, N. B., Whelan, M. A. A method for the absolute quantification of cDNA using real-time PCR. J Immunol Methods. 278 (1-2), 261-269 (2003).
- Too, H. P. Real time PCR quantification of GFRalpha-2 alternatively spliced isoforms in murine brain and peripheral tissues. Brain Res Mol Brain Res. 114 (2), 146-153 (2003).
- Turton, K. B., Annis, D. S., Rui, L., Esnault, S., Mosher, D. F. Ratios of Four STAT3 Splice Variants in Human Eosinophils and Diffuse Large B Cell Lymphoma Cells. PloS One. 10 (5), e0127243 (2015).
- Maurer, L. M., et al. Extended binding site on fibronectin for the functional upstream domain of protein F1 of Streptococcus pyogenes. J Biol Chem. 285 (52), 41087-41099 (2010).
- Ausubel, F. M., et al. . Current protocols in molecular biology. , (1987).
- Grant, S. G., Jessee, J., Bloom, F. R., Hanahan, D. Differential plasmid rescue from transgenic mouse DNAs into Escherichia coli methylation-restriction mutants. Proc Natl Acad Sci U S A. 87 (12), 4645-4649 (1990).
- Kocsis, L., Herman, P., Eke, A. The modified Beer-Lambert law revisited. Phys Med Biol. 51 (5), N91-N98 (2006).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin Chem. 55 (4), 611-622 (2009).
- Pfaffl, M. W. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Res. 29 (9), e45 (2001).
- Bustin, S. A. . A-Z of quantitative PCR. , (2004).
- Zhang, H. F., Lai, R. STAT3 in cancer-friend or foe. Cancers (Basel). 6 (3), 1408-1440 (2014).
- Caldenhoven, E., et al. STAT3beta, a splice variant of transcription factor STAT3, is a dominant negative regulator of transcription. J Biol Chem. 271 (22), 13221-13227 (1996).
- Zammarchi, F., et al. Antitumorigenic potential of STAT3 alternative splicing modulation. Proc Natl Acad Sci U S A. 108 (43), 17779-17784 (2011).
- Liu, Y., Beyer, A., Aebersold, R. On the Dependency of Cellular Protein Levels on mRNA Abundance. Cell. 165 (3), 535-550 (2016).
- Narimatsu, M., et al. Tissue-specific autoregulation of the stat3 gene and its role in interleukin-6-induced survival signals in T cells. Mol Cell Biol. 21 (19), 6615-6625 (2001).
- Szafranski, K., et al. Physiological state co-regulates thousands of mammalian mRNA splicing events at tandem splice sites and alternative exons. Nucleic Acids Res. 42 (14), 8895-8904 (2014).
- Sundin, M., et al. Novel STAT3 mutation causing hyper-IgE syndrome: studies of the clinical course and immunopathology. J Clin Immunol. 34 (4), 469-477 (2014).
- Oltean, S., Bates, D. O. Hallmarks of alternative splicing in cancer. Oncogene. 33 (46), 5311-5318 (2014).
- Boultwood, J., Dolatshad, H., Varanasi, S. S., Yip, B. H., Pellagatti, A. The role of splicing factor mutations in the pathogenesis of the myelodysplastic syndromes. Adv Biol Regul. 54, 153-161 (2014).
- Tilgner, H., et al. Comprehensive transcriptome analysis using synthetic long-read sequencing reveals molecular co-association of distant splicing events. Nat Biotechnol. 33 (7), 736-742 (2015).
- Hiller, M., et al. Widespread occurrence of alternative splicing at NAGNAG acceptors contributes to proteome plasticity. Nat Genet. 36 (12), 1255-1257 (2004).
- Rosenfeld, J. A., Malhotra, A. K., Lencz, T. Novel multi-nucleotide polymorphisms in the human genome characterized by whole genome and exome sequencing. Nucleic Acids Res. 38 (18), 6102-6111 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon