A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
עיצוב ניסיוני עבור לייזר Microdissection RNA-seq: לקחים מניתוח התפתחות תירס ליף
In This Article
Summary
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Abstract
גנים עם תפקידים חשובים בפיתוח לעתים קרובות יש מרחבית ו / או דפוסי ביטוי מוגבלים בזמן. לעתים קרובות תמלילי גנים אלה אינם מזוהים או אינם מזוהים כפי שבאו לידי הביטוי באופן דיפרנציאלי (DE) בניתוחי transcriptomic איברי צמח כולו. לייזר Microdissection RNA-seq (LM RNA-seq) הוא כלי רב עוצמה כדי לזהות גנים הם DE בתחומים התפתחותיים ספציפיים. עם זאת, הבחירה של תחומים הסלולר microdissect ולהשוות, ואת הדיוק של microdissections הם קריטיים להצלחת הניסויים. הנה, שתי דוגמאות להמחיש שיקולי תכנון ניסויים transcriptomics; ניתוח RNA-seq LM לזהות גנים דה לאורך ציר הפרוקסימלי-דיסטלי עלה תירס, וכן בניסוי השני כדי לזהות גנים הם DE ב liguleless1-R (lg1-R) מוטציות לעומת wild-type. מרכיבים מרכזיים שתרמו להצלחת הניסויים הללו פורטו היסטולוגית ב סיכלת tu המנתח של האזור להיות מנותחת, מבחר primordia עלה בשלבי התפתחותיים מקבילים, השימוש ציוני דרך מורפולוגיים לבחור אזורים עבור microdissection, ו microdissection של תחומים למדוד בדייקנות. מאמר זה מספק פרוטוקול מפורט לניתוח תחומי התפתחותיים ידי LM RNA-seq. הנתונים המוצגים כאן להמחיש עד כמה את האזור שנבחר עבור microdissection ישפיע על התוצאות שהושגו.
Introduction
העלה התירס הוא מודל אידיאלי לחקור היווצרות שדות התפתחותית במהלך המורפוגנזה, שכן יש בה גבול ברור בין הלהב לבין הנדן כי ניתן לנתיחה גנטית (איור 1 א). במהלך השלבים המוקדמים של פיתוח עלה, להקה ליניארי של תאים קטנים יותר, הלהקה preligule (PLB), subdivides העלה primordium לתחומים מראש להב וטרום-נדן. Ligule שולי דמוי auricles משולש לפתח מן PLB (איור 1 א ', ג', ד). מסך גנטי זיהה מוטציות המשבשות את גבול להב-נדן. לדוגמה, liguleless1 רצסיבי (lg1) מוטציות למחוק את ligule ו auricles 1, 2, 3, 4 (איור 1 ב). באתרו הכלאה גילתה כי תמליל lg1 מצטבר על PLB ומתפתחים ligule, מה שהופך אותו סמן מצוין לפיתוח ligule 5, 6 (איור 1E).
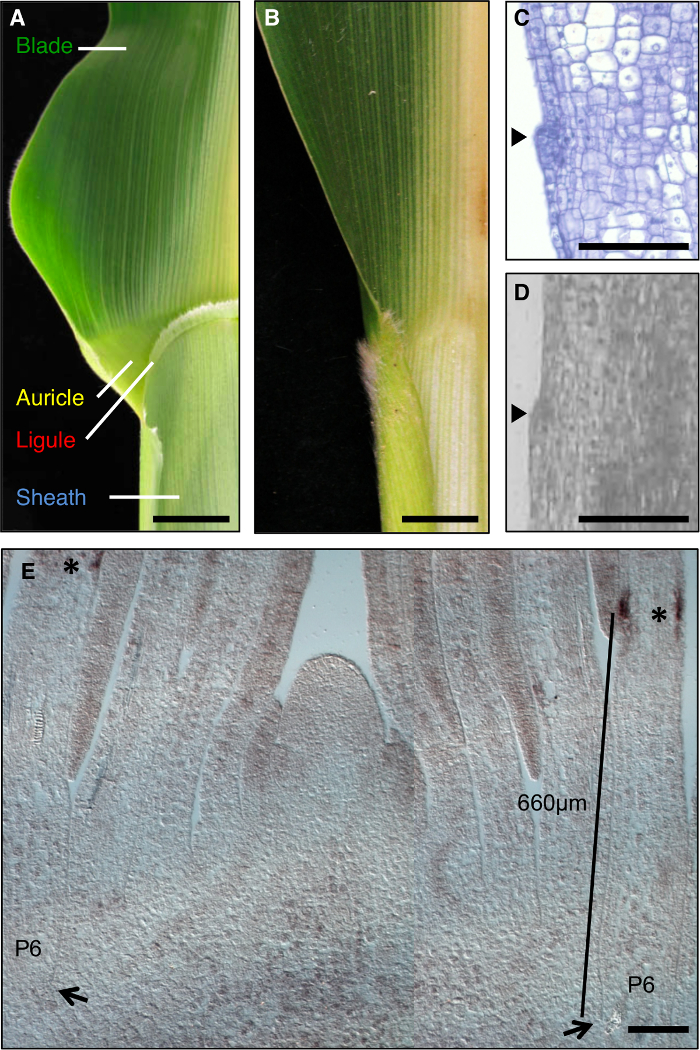
איור 1: wild-type ו liguleless1-R עלי תירס. (א) גבול Blade-נדן באזור עלה wild-type הבוגרת מראים מבני ligule ו אפרכסת. (ב) באזור הגבול Blade-נדן היעדרות מראה עלה בוגרת liguleless1-R של מבנים ligule ו אפרכסת. עלים ב A ו- B קוצצו במחצית לאורך midrib. (ג) אורך קטע דרך primordium עלה wild-type. לדוגמא תעובד ומוכתם לניתוח היסטולוגית. Ligule הייזום ניכר כבליטה הבולטת מהמטוס של העלה (ראש החץ). (ד) כת אורכיתיון דרך primordium עלה wild-type. לדוגמא תעובד עבור LM כמתואר בטקסט. ראש החץ מציין ייזום ligule. (E) lg1 הכלאה באתרו של חתך לאורך לרוחב איפקס לירות. כוכביות מצביעים הצטברות תמליל lg1 בבית PLB של primordium עלה P6. החצים מצביעים על בסיס של primordium P6. בר מציין מדידה מהבסיס של primordium אל PLB. ברי סולם ב A ו- B = 20 מ"מ. ברים בקנה מידה לספירה = 100 מיקרומטר. נתון זה יש הבדל בין התייחסות 6 (אגודה האמריקנית זכויות יוצרים של ביולוגי צמח). אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
במחקר זה, LM RNA-seq הועסק לזהות חבילה של הגנים באים לידי ביטוי באופן דיפרנציאלי (DE) בבית ביחס גבול להב-נדן לחלקים אחרים של primordium עלה ל IDE גני ntify כי הם DE ב lg1-R מוטציות ביחס אחי wild-type. LM RNA-seq היא שיטה לכימות הצטברות תמליל בתאים ספציפיים או תחומים הסלולר 7. מערכות LM לשלב לייזר מיקרוסקופ עם מצלמה דיגיטלית. רקמה מחולקת היא רכובה על שקופיות נצפית מבעד למיקרוסקופ. תוכנת LM כוללת בדרך כלל כלי ציור המאפשרים למשתמש להתוות בכל אזור שנבחר עבור microdissection. קיצוצי הליזר לאורך הקו, ואת הרקמה שנבחרו הזניקו את השקופית לתוך צינור התלוי מעל השקופית. LM מאפשר למשתמש microdissect תחומים מדויק, כולל שכבות תאים ספציפיים יחיד, גם תאי 8, 9. RNA ניתן לחלץ אז מרקמת microdissected. בהמשך לכך, מרכיב-Seq RNA מנצל רצף של הדור הבא רצף ספריות cDNA שנוצר מן RNA חילוץ 10,= "Xref"> 11.
יתרונות עיקריים של רנ"א-seq LM הם היכולת לכמת הצטברות תמליל בתחומים מוגדרים במדויק ואת היכולת גטסבי הגדול transcriptome כולו 7 זמנית. הטכניקה מתאימה במיוחד חיטוט אירועים התפתחותי מוקדם שבי את האזור של עניין הוא לעתים קרובות מיקרוסקופי. מחקרים קודמים מנוצל LM בשילוב עם הטכנולוגיה microarray ללמוד תהליכים התפתחותיים בצמחים 9, 12, 13. יש RNA-seq היתרון של לכימות תמלילים על פני טווח דינמי רחב, כוללים גנים נמוכים הביע, ומידע רצף לפני אינו נדרש 10, 11. יתר על כן, LM RNA-seq יש הפוטנציאל להדגיש גנים חשובים מבחינה התפתחותית שעשויות מוחמצות מסכי mutagenesis בשל יתירות גנטית או לערוך קטלני של האובדן-של-מוטצית פונקציה.
מבחינה התפתחותית גנים חשובים, כגון sheath1 צר (ns1) ו cotyledon2 בצורת כוס (cuc2), לעתים קרובות יש דפוסי ביטוי ספציפי אחד בלבד או מספר תאים 17, 18, 19, 20. הרבים באים לידי ביטוי רק במהלך שלבי התפתחות מוקדמים ולא באיבר הבוגר. כאשר איברים שלמים או תחומים גדולים מנותחים, תמלילי תא ספציפי אלה הם בדילול וייתכן שלא להתגלות יותר ניתוחים קונבנציונליים. על ידי התרת ניתוחים של תחומים מוגדרים במדויק, LM RNA-seq מאפשר גני הרקמות ספציפיות אלה כדי להיות מזוהים לכימות.
גורמים מכריעים בהצלחתה של הניסויים שתוארו כאן היו ניתוח היסטולוגית יסודי שהנחה מבחר הבמה המושלמת ההתפתחותית המתאימה לניתוח, ו measureme המדויקNT של תחומי רקמות תאים עבור LM. כדי להבטיח כי לדומיינים מקבילים נדגמו לכל משכפל, רקמות נאסף primordia עלה באותו שלב התפתחותי התחומים microdissected נמדדו ביחס ציוני דרך מורפולוגיים כגון ligule המתעוררים (איור 2). זה ידוע כי גנים מסוימים מתבטאים שיפוע מהקצה לבסיס של העלה. באמצעות מדידת תחומים מדויקים, וריאציה בשל דגימה ממקומות שונים לאורך הציר הפרוקסימלי-דיסטלי עלה הוחזק על (איור 3 א) מינימום. על ידי microdissecting תחומים של באותו גודל, וריאציה בשל דיפרנציאלי דילול של תמלילי תא ספציפי הופחת גם (איור 3B). סעיפים אורכים לרוחב של הקודקוד לירות שמשו כל microdissections. אלה הם חלקים כי הם בניצב לציר midrib-שוליים (איור 4). באמצעות סעיפים בלבד הכוללים את SAM מבטיח כי אזורים לרוחב מקבילים שלprimordia עלה מנותחים.
בדגימות מעובדים מחולקים עבור LM, סימן מורפולוגיים הראשון של תולדת ligule הוא בליטה בצד adaxial עקב חלוקות תא periclinal באפידרמיס adaxial (1D איור, איור 2). לעניין זה נקבע כי ligule המתעוררים יכול להיות מזוהה באופן מהימן לפי primordia עלה שלב פלסטוכרון 7. היינו מעוניינים גנים הביעו באזור ligule כולו, כולל ligule המתעוררים והתאים מייד דיסטלי שיהוו את האפרכסת. על מנת להבטיח כי בחירות רקמות מקבילות נעשו, גבשושית ligule שמשה כציון דרך מורפולוגיים מלבנים 100 מיקרומטר מרוכזים על בליטת ligule נבחרה LM (איור 2 א, 2 ב). מלבנים בגודל שווה של שבשבת טרום-נדן מראש נבחרו מאותו primordia עלה.
ניתוח של צמחים מוטנטים liguleless הציג challe שונהnge; מוטציות lg1-R לא יוצרים ligule, ולכן תכונה מורפולוגית זה לא יכול לשמש כדי לבחור את האזור עבור LM. במקום זאת, התחום של הצטברות תמליל lg1 ב primordia עלה wild-type נקבע, וכן באזור שיקיף תחום זה הוגדר. ניתוחים ראשוניים אלה בוצעו על השתילים מאותו שתילה כמו שימשו בסופו של דבר, מאז מחקרים קודמים הראו כי מיקומו של PLB משתנה בהתאם לתנאי הגידול. באתרו הכלאה ציינו כי תמלילי lg1 להצטבר primordia עלה PLB של P6 (איור 1E). בחרנו מיקרומטר תחום 400-900 מהבסיס של primordia עלה שהקיף את תחום ביטוי lg1 (מלבנים סגולים, איור 2 א) וכבש אזורים המקבילים אלה מן wild-type וצמחי lg1-R. כדי למזער וריאציה בתנאי הרקע וצמיחה גנטי כאשר משווים transcripהצטברות t ב lg1-R וצמחים בר-סוג, תוך הפרדת משפחות של מוטציות ואחי wild-type שמשה.
Protocol
הערה: תקן רקמות לניתוח היסטולוגית באותו הזמן כי רקמה הוא קבוע עבור LM. בדוק סעיפים מוכתמים עבור תכונות מורפולוגיות כי תדרכנה מאוחר LM. בהשוואת מוטציה wild-type, לבצע הכלאה באתרו או immunolocalization להגדיר את התחום שבו הגן של עניין מתבטא (ב lg1 במקרה זה).
קיבוע רקמות 1. ועיבוד
- לגדול דירות של שתילי תירס עד שבועות ישנים בתנאים סטנדרטיים 6.
- לנתח apices לירות עבור חלקים לרוחב (איור 4).
- שתיל ובלו רק מתחת לקו הקרקע.
- באמצעות סכין גילוח, להסיר פרוסות דקות מבסיס הגבעול (חתכי 1, איור 4 א) עד אליפסה של culm המוקפת אחד או שני עלים בוגרים גלוי (איור 4B).
- הפוך להורדת ריבית נוספת כ 10 מ"מ מעל הבסיס (לחתוך 2, איור 4 א).קטע 10 מ"מ זה יכיל את SAM ו primordia העלים הצעירים.
- סובב את קטע 10 מ"מימ ולכן הבסיס הוא פונה כלפי מעלה. בשני חתכים מקבילים לצייר לרוחב כך פרוס מ"מ רקמת 2-3 מתקבל עבה (חתכים 3, ו -4, איור 4B). מחק את שני חלקים החיצוניים ולשמר את הפרוסה המרכזת קיבעון והטבעה.
הערה: עלים חיצוניים עשויים להיות מטופחים וזנוח.
- תקן רקמות תהליך להטבעה.
- ודא כי כל החומרים לשמש בשלבים הבאים חופשי RNase. פנקו את הפתרונות עם diethyl pyrocarbonate (DEPC) (1 מ"ל DEPC לליטר פתרון. דגירה לילה עם רעד מדי פעם, החיטוי). כלי זכוכית אופים בתנור ב 200 מעלות צלזיוס ומעלה במשך לפחות 6 שעות ולטפל כלי פלסטיק עם פתרון טיהור RNase.
- יום 1: לטבול בחתכי רקמה ב ~ 10 מ"ל של תיקון של פארמר (3: 1 אתנול: חומצה אצטית) ב בקבוקון זכוכית על הקרח. אחרי הכל הדגימות כבר גזורות, להחיל vacuuמ 'כדי להסיר בועות אוויר וחדירה לעזרת מקבע. חזק תחת ואקום במשך 10 דקות ולאחר מכן לשחרר את הוואקום לאט. החלף מקבע ו דגירה על 4 מעלות צלזיוס לילה עם רעד עדין.
- יום 2: דגירה בסדרת הפתרונות הבאים, ~ 10 מ"ל כל אחת, 1 hr כל, כל עם רעד עדין; 85% אתנול ב 4 ° C, 95% אתנול ב 4 ° C, אתנול 100% ב 4 ° C, 100% אתנול ב 4 ° C, 100% אתנול ב 4 ° C, 1: 1 אתנול: xylenes בטמפרטורת החדר, xylenes 100% בטמפרטורת החדר, 100 xylenes% בטמפרטורת החדר.
הערה: xylenes רעיל על ידי מגע ושאיפה. העבודה במנדף ולהשתמש בכפפות מתאימות. - להוסיף רקמת פרפין הטבעה כדורי בינוני עד כמחצית היקף xylenes דגירה לילה בטמפרטורת החדר עם רעד עדין.
- יום 3: מעביר את הבקבוקון עד 60 ° C בתנור עד הכדורים להמס. יוצק את הפתרון ולהחליף עם מדיום הטבעת רקמות מומסות טרי. שינוי פעמים הבינוניות יותרבמהלך היום.
- יום 4: רקמת שינוי הטבעה בינונית פעם בבוקר. חזור אל 60 ° C בתנור עד אחר הצהריים.
- בלוקים יצוקים
- מניח תבניות הטבעה על צלחת חמה של תחנת הטבעת רקמות. שימוש במלקחיים להעביר את דגימות רקמה על תבניות הטבעה עם המשטח החתוך כלפי מטה. הוספת כספים את התבנית עם פרפין המומס ומניח את טבעת ההטבעה על גבי התבנית. מעבירים לצלחת קר עד פרפין יש הקרושה. אחסן את אבני פרפין ב 4 מעלות צלזיוס בכלי אטום עם ג'ל סיליקה.
2. חתך וחלק כנה
- חותכים 10 מיקרומטר חלקים על microtome 25.
- בדוק סרטים ולבחור חלקים החציוני. סעיפי חציון הם אלה הכוללים את SAM, אשר נראה כמו כיפה של תאים המוקפים primordia עלה.
- סעיפי הר בשקופיות.
- מקום שקופית מתאימים LM (או RNase חינם אואפוי) על 42 מעלות צלזיוס להחליק חם ולהחיל כמה טיפות של תמיסת אתנול 50% כדי לכסות את השקופית.
- Float מדורי פתרון אתנול עד בסעיפים הרחיבו.
הערה: צפים חלקים על פתרון אתנול ולא מים שומר RNA במצב זירז צמצום השפלה RNA. - הטה שקופיות ולהסיר פתרון אתנול עודף ידי שאיפה עם טפטפת העברת הפנויה. השתמש מגבונים ללא סיבים כדי פתיל משם כל פתרון אתנול נוסף.
- מגלשות יבשות ב 42 מעלות צלזיוס במשך כמה שעות או לילה. חנות מחליקה על 4 מעלות צלזיוס בכלי אטום עם ג'ל סיליקה.
- Deparaffinize מחליק ביום שימוש.
- כן שלוש צנצנות הזכוכית Coplin המכילות; 100% xylenes (xylenes I), 100% xylenes (xylenes II), ו -100% אתנול (~ 50 מ"ל של כל פתרון).
- בעזרת מלקחיים נקיים להעביר שקופיות, לטבול שקופיות xylenes לי למשך 2 דקות, xylenes שנייה למשך 2 דקות, ו 100% אתנול 1 דקות.
- Drain החליקes על מגבונים ללא סיבים ואוויר יבש בטמפרטורת החדר.
Microdissection 3. בלייד, דוגמאות Ligule נדן מ פלסטוכרון 7 ליף primordia
- אבטח את השקופיות על הבמה של מיקרוסקופ LM. השתמש חמש או שש שקופיות עבור כל לשכפל, ניצול חמישה סעיפים לכל שקופית.
הערה: רקמות איגום עבור לשכפל יחיד מתוארת באיור 5. - בדוק שקופיות ולזהות חמשת הסעיפים החציוני ביותר על כל שקופית, באמצעות איפקס SAM כנקודת התייחסות מרכזית.
הערה: ניתן לעשות זאת בהגדלה נמוכה, בדרך כלל מטרת 5X מספיקה. - באמצעות אובייקטיבי 10X או 20X, לזהות את המיקום של ligule על primordium עלה פלסטוכרון 7 של כל קטע. Ligule יהיה גלוי כבליטה הבולטת ממשטח adaxial של primordium עלה. סמן עמדה זו באמצעות כלי הציור של תוכנת LM; בחר את סמל העיפרון, להזיז את הסמן positi המתאיםעל ולחץ וגרור את העכבר כדי לצייר.
הערה: מטרת 10X או 20X מתאימה זה בשלבים הבאים. בעת שימוש בסעיפים לרוחב, שני הצדדים של כל primordium עלה יהיו נוכחים בכל (איור 2 א) בסעיף. - באמצעות הכלי השליט ואת כלי ציור המלבניים, למדוד 100 מיקרומטר מלבנים גבוהים מרוכזים על ligule של כל מקטע (מלבנים אדומים, איור 2 א, 2 ב). אלה יהיו מדגם "Ligule".
- כדי להשתמש בכלי השליט; בחר בסמל שליט, להזיז את הסמן לקצה אחד של האובייקט כדי להימדד, לחץ וגרור כדי למדוד את האובייקט. אורכו של השליט יוצג על המסך.
- כדי לצייר מלבן; בחר בסמל מלבן, להזיז את הסמן לנקודה כי יהיה בפינה אחת של המלבן, לחץ וגרור כדי לצייר מלבן בגודל המתאים. לחלופין, בחרו בכלי ציור קו ישר לצייר ארבעה קווים ישרים.
- מדוד 100 מיקרומטר מלבנים ממוקמים 50 מיקרומטר מעל ומתחת המלבן "Ligule".
הערה: אלה יהיו "בלייד" ו "נדן" דגימות, בהתאמה (ירוק וכחול מלבנים, איור 2 א, 2 ב). בהתבסס על נתונים היסטולוגית שלנו, מלבן 100 מיקרומטר מקיף באזור ligule כולו. חלקים בגודל שווה של להב נדן נבחרו על מנת להבטיח כי כמויות דומות של רקמות נאספו עבור כל אחד. מרווחים של 50 מיקרומטר שימשו כדי לוודא ששום רקמה באזור ligule כלול בשוגג microdissections להב או נדן.
- Microdissect נמדד מלבנים (איור 2 ד - 2F) 7, 8, 9, איסוף Ligule, דגימות להב נדן צינורות נפרדים. השתמש בפונקציה לחתוך לייזר לחתוך את קטע רקמה לאורך קו המתאר של התחום הנבחר. השתמש CATAפונקציה פולטת כדי להניע את המלבן של רקמות את השקופית לתוך המכסה של הצינור (איור 2 ד - 2F).
Microdissection 4. של בלייד, Ligule נדן Adaxial עוריות דוגמאות מן פלסטוכרון 7 ליף primordia
- בחר קטעים ולהשתמש בכלי בסרגל למדוד 100 מיקרומטר מגזרים גבוהים מרוכזים על ligule פלסטוכרון 7, כמפורט בסעיף 3 (לעיל).
- בחר את תאי אפידרמיס adaxial רק של כל 100 מיקרומטר "בלייד" גבוה במגזר "נדן" (בחירות ירוקות והכחולות, איור 2 ג) על ידי בהתוויית עם כלי הציור. האפידרמיס הוא שכבת התאים החיצונית; בצד adaxial הוא הקרוב ביותר אל SAM.
- למדגם "Ligule", לבחור רק את התאים של בליטה ligule המתעוררים כמפורט בסעיף 3.3 (מבחר אדום, איור 2C).
- באזורים נבחרים Microdissect, איסוף Blade, Ligule ושייה האפידרמיס דגימות צינורות נפרדים, כמתואר בסעיף 3.5.
Microdissection 5. פלסטוכרון 6 ליף primordia מ lg1-R ואחים wild-type
- לגדול תוך הפרדת משפחות של מוטציה (lg1-R) וצמחי בר-סוג.
- תקן ותהליך לירות apices עבור LM, כמפורט בסעיפים 1.2-1.4. תקן wild-type ו מוטציה לירות apices צלוחיות נפרדות על מנת לשמור אותם נפרדים. דוגמאות מאותו השתילה צריכות להיות קבועות ומעובד הכלאה באתרה.
- לקבוע היכן lg1 הוא עבד ב אחי wild-type, על ידי ביצוע lg1 הכלאה באתרו 6, 26, 27. נמדד מצבה של הצטברות תמליל lg1 מבסיס העלה primordium בדגימות מרובות (איור 1E).
- בהתבסס על נתונים כולים באתרו, לבחור חלק פרימו עלהrdium המקיף את האזור שבו lg1 הוא עבד. במקרה זה, 400-900 מיקרומטר מבסיס primordia עלה פלסטוכרון 6 (מלבנים סגולים, איור 2 א).
- Microdissect שנבחרו חלק primordia עלה, כמתואר בסעיף 3.5, איסוף lg1-R ודוגמאות wild-type צינורות נפרדים.
6. החל RNA הפקת ההצפה
- החל 50 μl חיץ מיצוי RNA לרקמות microdissected והמשך עם מיצוי RNA. המשך עם מיצוי RNA, הגברת RNA, בניית ספרייה, רצף וניתוח ביואינפורמטיקה כמתואר התייחסות 6.
תוצאות
באמצעות סכימת LM התווה באיור 2, כ 1,000,000-1,500,000 מיקרומטר 2 של רקמות נאסף עבור כל לשכפל כל-תא-שכבות LM (איור 5), ו -200,000 מיקרומטר 2 לכל לשכפל עבור LM האפידרמיס adaxial. כ 2,500,000 מיקרומטר 2 של רקמות נאספות עבור כל לשכפל LM של lg1-R ו-...
Discussion
עיצוב ניסיוני מהווה גורם קריטי בניסויים seq-RNA. שיקולים עיקריים הם התחום המדויק (הים) ו לשלב ההתפתחותי (ים) להיות מנותחים, ומה השוואות תיעשינה. זה חיוני כדי לחשוב במונחים של השוואות, מאז הפלט הוא בדרך כלל רשימה של גנים אשר עברו דה בין שניים או יותר תנאים. כמו עם כל הניסויי?...
Disclosures
The authors have nothing to disclose.
Acknowledgements
המחברים מודים ס הייק עבור נמשכת, שיתוף פעולה ועידוד דיונים על פיתוח ligule. עבודה זו נתמכת על ידי הקרן הלאומית למדע מענקים MCB 1052051 ו IOS-1,848,478.
Materials
| Name | Company | Catalog Number | Comments |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials - 22 ml | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 mm x 15 mm x 5 mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60 °C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
References
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved