JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
레이저 이외에 Microdissection RNA-서열에 대한 실험 디자인 : 옥수수 잎 개발의 분석 교훈
요약
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
초록
개발에 중요한 역할 유전자는 자주 공간적 및 / 또는 시간적으로 제한된 발현 패턴이 있습니다. 종종 이러한 유전자 성적 증명서가 감지되지 않거나 차별적으로 전체 식물 기관의 transcriptomic 분석에서 (DE) 표시로 확인되지 않습니다. 레이저 이외에 Microdissection RNA-SEQ (LM RNA-SEQ)는 특정 발달 영역에서 DE이다 유전자를 식별 할 수있는 강력한 도구입니다. 그러나, 셀룰러 도메인의 선택은 microdissect과 비교하고 microdissections의 정확도는 실험의 성공에 매우 중요한다. 여기서 두 가지 예 transcriptomics 실험에 대한 디자인 고려 사항을 설명; RNA-서열 분석은 옥수수 잎 근위 말단부 축을 따라 DE되는 유전자를 동정하는 LM 및 두번째 실험에서 liguleless1 DE-R (LG1-R)이다 유전자를 동정 할 돌연변이는 야생형에 비해. 이 실험의 성공에 기여한 핵심 요소는 조직 학적 및시에 설명 된TU 하이브리드 분석 할 수있는 영역 분석, 동등한 발달 단계에서 잎 primordia의 선택, 형태 학적 랜드 마크의 사용은 미세 절제에 대한 영역을 선택하고 정확하게 측정 영역의 미세 절제합니다. 이 논문은 LM RNA-서열에 의해 개발 도메인 분석을위한 상세한 프로토콜을 제공합니다. 여기에 제시된 데이터는 미세 절제 선택 영역이 얻은 결과에 영향을 미칠 것입니다 방법을 보여줍니다.
서문
이 유전자 해부 (그림 1A)에 의무가 블레이드와 외피 사이에 뚜렷한 경계가 같이 옥수수 잎, 형태 형성 과정 개발 분야의 형성을 연구하는 이상적인 모델이다. 잎 발달 작은 셀 선형 대역의 초기 단계 동안, preligule 대역 (PLB)은, 미리 블레이드 프리 시스 도메인에 리프 primordium을 세분화. 프린지 같은 잎혀와 삼각 이개는 PLB (그림 1A, C, D)에서 개발. 유전 화면은 블레이드 시스 경계를 방해 돌연변이를 확인했다. 예를 들면, 열성 liguleless1 (LG1) 돌연변이는 잎혀 귓바퀴 및 1, 2, 3, 4 (도 1b)을 삭제. 현장에서 하이브리드 LG1 성적은 P에서 축적 것으로 나타났다LB와 잎혀 개발 5, 6 (그림 1E)에 대한 그것에게 우수한 마커를 만들고, 잎혀 신흥.
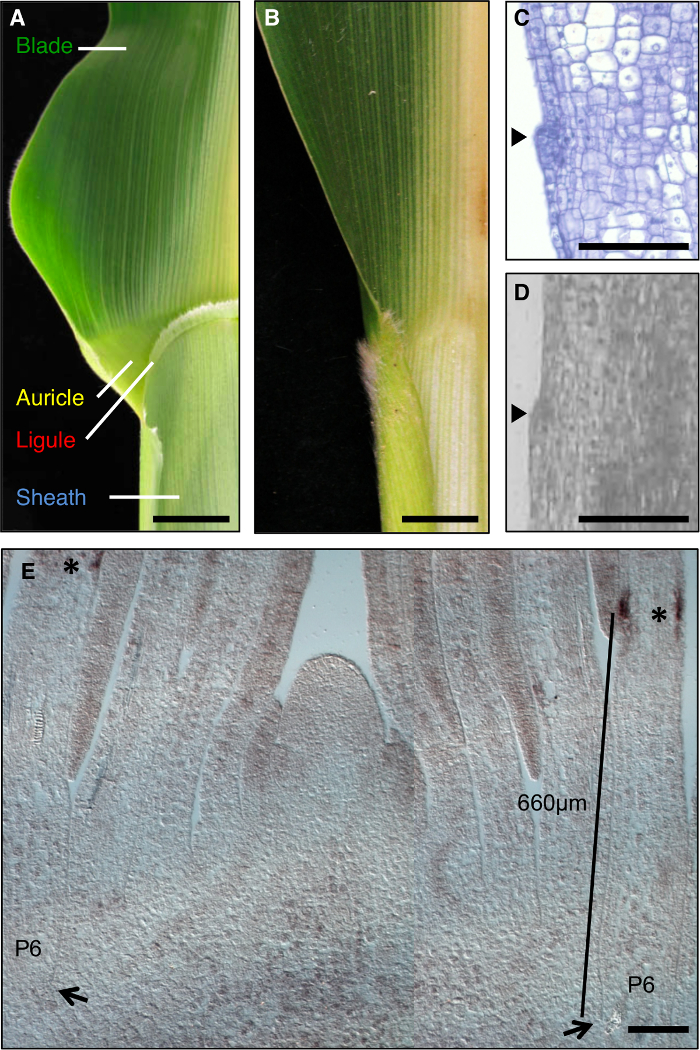
그림 1 : 야생 형 및 liguleless1-R 옥수수 잎. 잎혀와 귓바퀴 구조를 나타내는 성숙한 야생형 리프 (A) 블레이드 시스 경계 영역. (B) 잎혀와 귓바퀴 구조의 성숙 liguleless1-R 잎 보여주는 부재의 블레이드 - 시스 경계 지역. A와 B의 잎은 주맥을 따라 반으로 잘라되었습니다. 야생형 잎 primordium 내지 (C) 종단면도. 샘플 처리 및 조직 학적 분석을 위해 염색되었다. 개시 잎혀 리프 (화살촉)의 평면으로부터 돌출 된 돌기로서 명백하다. (D) 세로 분파야생형 잎 primordium을 통해 이온. 본문에 설명 된 바와 같이 샘플 LM 위해 처리되었다. 화살촉은 잎혀를 시작 나타냅니다. 촬영 정점 측면 종단면의 현장 하이브리드에서 (E) LG1. 별표는 P6 잎 primordium의 PLB에서 LG1 성적 증명서 축적을 나타냅니다. 화살표는 P6의 primordium의 기반을 나타냅니다. 바는 PLB에 primordium의 기지에서 측정을 나타냅니다. A와 B의 스케일 바는 20mm를 =. CE에서 스케일 바 = 100 μm의. 이 수치는 참조 6 (식물 생물학의 저작권 미국 사회)에서 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
이 연구에서, LM RNA-SEQ은 리프 primordium 및 IDE 다른 부분에 블레이드 시스 경계에 대하여 차등 발현되는 유전자 세트 (DE)를 식별하는데 사용 된 야생 형 형제에 대해 LG1-R 돌연변이에 드입니다 ntify 유전자. LM RNA-SEQ 특정 세포 또는 세포 영역 (7)에 전사 축적을 정량하는 방법이다. LM 시스템은 레이저와 디지털 카메라로 현미경을 결합한다. 단면 조직 슬라이드에 장착 현미경을 통해 볼 수 있습니다. LM 소프트웨어는 일반적으로 사용자가 미세 절제를 위해 선택된 영역을 간략하게 설명 할 수 있도록 그리기 도구가 포함되어 있습니다. 라인을 따라 레이저 절단, 및 선택된 조직 슬라이드를 발사 슬라이드 위에 매달린 튜브로되어있다. LM은 사용자가 특정 세포층 심지어 단 전지 (8, 9)를 포함하여 정확한 도메인을 microdissect 수있다. RNA는 다음 microdissected 조직으로부터 추출 될 수있다. 이어서, RNA-SEQ 성분이 추출 된 RNA로부터 생성 된 cDNA를 10 라이브러리 시퀀스 차세대 시퀀싱을 이용하여,= "외부 참조"> 11.
LM-RNA의 서열의 주요 장점은 정확하게 정의 된 영역에 축적 전사를 정량화하는 능력과 동시에 전 사체 7을 프로파일하는 능력이다. 이 기술은 관심 영역은 종종 마이크로 일찍 개발 이벤트 프로빙에 특히 적합하다. 이전 연구 LM 식물 9, 12, 13의 발달 과정을 연구하기 위해 마이크로 어레이 기술과 함께 이용했다. RNA-SEQ 저 발현 된 유전자를 포함하는 폭 넓은 동적 범위에 걸쳐 사체 정량의 장점을 가지며, 이전 시퀀스 정보 (11) (10)를 요구하지 않는다. 또한, LM RNA-서열 인해 외 손실 유전자 중복 또는의 치사에 돌연변이 화면에서 놓칠 수있다 발달 중요 유전자를 강조 할 수있는 잠재력을 가지고있다기능 돌연변이.
협 sheath1 (NS1) 및 컵 형상 cotyledon2 (cuc2)와 같은 중요한 발달 유전자는 종종 단지 하나의 특이 적 발현 패턴 또는 몇 셀 17, 18, 19, 20가있다. 대부분은 초기 개발 단계에서가 아닌 성숙한 장기로 표현된다. 전체 기관 또는 대형 도메인이 분석되면,이 세포 특이 성적 증명서는 희석 등 기존의 분석에서 검출되지 않을 수 있습니다. 정확하게 정의 된 도메인의 분석을 가능하게함으로써, LM RNA-서열이 조직 특이 적 유전자 식별 및 정량 할 수 있습니다.
여기에 설명 된 실험의 성공에 결정적 요인은 분석을 위해 적절한 발달 단계 및 도메인 선택 안내 철저한 조직 학적 분석 및 정확한 방식 측정했다LM에 대한 세포 조직 도메인의 NT를. 해당하는 도메인이 모든 복제에 대한 샘플링되었는지 확인하려면 조직은 동일한 발달 단계에서 잎 primordia에서 수집하고 microdissected 도메인은 신흥 잎혀 (그림 2)와 같은 형태의 랜드 마크를 기준으로 측정 하였다. 어떤 유전자가 잎의베이스 끝 부분에서 그라데이션 표현되는 것이 알려져있다. 인해 잎 근위 말단부 축을 따라 상이한 위치에서 샘플링 정확한 도메인 변동을 측정하여 최소 (도 3a)에 유지 하였다. 같은 크기의 영역을 microdissecting으로 인한 변화는 셀 특정 성적 증명서의 희석도 (그림 3B) 감소 차등합니다. 촬영 정점의 측면 길이 섹션은 모든 microdissections 사용 하였다. 이 (그림 4) 주맥 마진 축에 수직 섹션이다. 샘 만 포함 섹션을 사용하여의 동등한 측면 영역을 보장합니다잎 primordia 분석된다.
처리 및 LM에 대한 단면 샘플에서, 잎혀 가지의 첫 번째 형태 학적 기호 인해 향축 표피 (그림 1D, 그림 2)에서 periclinal 세포 분열에 향축 측에 범프이다. 신흥 잎혀 신뢰성 plastochron 7 단계 잎 primordia에서 식별 할 수 있다고 판단 하였다. 우리는 새로운 잎혀와 귓바퀴를 형성 할 것이다 즉시 말단 세포를 포함한 전체 잎혀 영역에서 발현 유전자에 관심이 있었다. 등가 조직의 선택이 이루어되었는지 확인하기 위해서, 범프 잎혀는 랜드 마크 형태로 사용하고, 범프 잎혀 중심 100㎛의 사각형은 LM (도 2A, 2B)으로 선정 하였다. 사전 블레이드 및 사전 시스의 동등한 크기의 사각형은 같은 잎 primordia에서 선정되었다.
liguleless 돌연변이 식물의 분석은 다른 challe 발표NGE; LG1-R 돌연변이 따라서이 형태 학적 특징 LM의 지역을 선택하는 데 사용할 수없는 잎혀을 형성하지 않는다. 대신, 야생형 잎 primordia에서 LG1 사체 축적 영역을 결정하고,이 영역을 포함 할 영역을 정의 하였다. 최종 분석에 사용 된 바와 같이 이전의 연구는 PLB의 위치가 성장 조건에 따라 변화하는 것이 도시 이후 이러한 예비 분석은, 동일한 모종 심기에서 수행 하였다. 현장에서 하이브리드 LG1 성적 증명서가 PLB P6의 잎 primordia (그림 1E)에 축적 것으로 나타났다. 우리는 LG1 표현의 도메인 (보라색 사각형, 그림 2A)를 포위하고 야생 형 및 LG1-R의 식물이 해당하는 지역을 촬영 한 잎 primordia의 기지에서 도메인 400-900 μm의를 선택했습니다. transcrip 비교할 때 유전 적 배경과 성장 조건의 변화를 최소화하기 위해LG1-R 및 야생형 식물 돌연변이 및 야생형 형제 가족 편석에 t 축적 하였다.
프로토콜
주 : 조직 LM 고정되는 동시에, 조직 학적 분석을 위해 조직을 수정. 나중에 LM을 안내 할 것입니다 형태 학적 기능에 대한 스테인드 섹션을 검사합니다. 야생형 변이체와 비교하면, (이 경우 LG1)에 관심있는 유전자를 발현하는 도메인을 정의하는 계내 혼성화 또는의 면역에서 수행한다.
1. 조직의 고정 및 처리
- 표준 조건 (6)에서 이전 2 주 옥수수 모종의 아파트를 성장.
- 측면 부분 (그림 4)에 대한 촬영 정점을 해부하다.
- 단지 토양 라인 아래 소비세 종묘.
- 면도날을 사용하여 하나 또는 두 개의 성숙한 잎에 의해 둘러 쌓여 줄기의 타원형이 표시 (그림 4B)가 될 때까지 줄기 (인하 1,도 4A)의 기지에서 얇은 조각을 제거합니다.
- 다른 컷베이스 위의 약 10 mm의 (도 4a 2 컷)합니다.이 10mm 세그먼트는 SAM과 어린 잎 primordia 포함됩니다.
- 기본이 위를 향하도록 10mm 세그먼트를 켭니다. 조직 두께 2~3mm의 조각이 얻어 지도록 두 삭감 횡축으로 평행 확인 (상처 3, 4,도 4b). 바깥 쪽 두 부분을 취소하고 고정 및 매립에 대한 중앙 슬라이스를 유지합니다.
참고 : 외부 잎을 손질하고 폐기 할 수있다.
- 내장에 대한 조직과 프로세스를 수정합니다.
- 의 RNase 무료로 모든 자료는 다음 단계에서 사용할 수 있는지 확인합니다. (. 솔루션의 리터 당 1 ml의 DEPC 가끔 흔들림 및 오토 클레이브와 하룻밤 품어), 디 에틸 피로 카보네이트 (DEPC)와 솔루션을 취급합니다. 구워 200 ° C의 오븐에 유리하거나 적어도 6 시간 동안 높은과의 RNase의 오염 제거 용액 플라스틱 제품을 취급합니다.
- 1 일 : 얼음에 유리 바이알에 (아세트산 : 1 에탄올 3) ~ 농부의 수정 10 ㎖를에 조직 조각을 담가. 모든 샘플을 해부 한 후, vacuu 적용m 공기 거품과 정착액의 원조 침투를 제거합니다. 10 분 후 천천히 진공을 해제 진공 하에서 만요. 정착액을 교체하고 부드러운 흔들림과 함께 밤새 4 ° C에서 품어.
- 주 2 : 솔루션의 다음 시리즈에 품어 ~ 10 ml를 각각 1 시간마다, 부드러운 흔들어 모든; 4 ° C, 4 ° C, 4 ° C, 4 ° C, 4 ° C에서 100 % 에탄올 100 % 에탄올 100 % 에탄올 95 % 에탄올, 1에서 85 % 에탄올 : 1 에탄올 : 실온에서 자일 렌, 실온에서 100 % 크실렌, 실온에서 100 % 크실렌.
참고 : 크실렌 접촉 및 흡입 독성. 흄 후드에서 작업하고 적절한 장갑을 사용합니다. - 자일 렌의 약 절반 볼륨 중간 알약을 포함 파라핀 조직을 추가하고 부드러운 흔들림 실온에서 밤새 품어.
- 주 3 : 펠릿이 녹아 때까지 60 ° C 오븐에 유리 병을 전송합니다. 솔루션을 붓고 신선한 용융 조직 포함 매체로 대체합니다. 매체 두 번 이상 변경하루 동안.
- 4 일 : 아침에 한 번 매체를 삽입 변경 조직. 오후까지 60 ° C 오븐으로 돌아갑니다.
- 캐스트 블록
- 조직 삽입 역의 핫 플레이트에 삽입 금형을 놓습니다. 절단 된면이 아래로 향하게하여 삽입 금형에 조직 샘플을 전송하는 집게를 사용합니다. 녹은 파라핀 몰드를 상부 및 상기 몰드의 상부에 매립하여 환을 놓는다. 파라핀이 응고 될 때까지 냉각 판에 전송합니다. 실리카 젤 밀폐 용기에 4 ° C에서 파라핀 블록을 저장합니다.
2. 단면과 준비를 밀어
- 마이크로톰 (25)에 10 μm의 섹션을 잘라.
- 리본을 검사하고 중간 섹션을 선택합니다. 중간 부분은 잎 primordia으로 둘러싸인 세포의 돔으로 표시되는 SAM을 포함하는 것들이다.
- 슬라이드에 마운트 부분.
- LM에 적합한 장소 슬라이드 (하나의 RNase 무료 또는구운) 42 °에 따뜻한 슬라이드 및 슬라이드를 충당하기 위해 50 %의 에탄올 용액 몇 방울을 적용 C.
- 섹션이 확장 될 때까지 에탄올 용액에 섹션을 플로트.
참고 : 에탄올 용액보다는 물에 섹션을 부동하는 RNA의 분해를 감소시키는 침전 된 상태에서 RNA를 유지합니다. - 슬라이드를 기울여 일회용 전송 피펫으로 흡인하여 과량의 에탄올 용액을 제거합니다. 추가 에탄올 용액을 멀리 심지에 보풀이없는 와이프를 사용합니다.
- 42 ° C에서 건조 슬라이드 몇 시간 또는 하룻밤. 스토어 실리카겔과 함께 밀폐 용기에 4 ° C에서 슬라이드.
- Deparaffinize 사용 당일에 슬라이드.
- 포함 3 개의 유리 코 플린 항아리를 준비; 100 % 크실렌 (크실렌 I), 100 % 크실렌 (크실렌 II), 100 % 에탄올 (~ 각 용액 50 mL)을 첨가 하였다.
- 깨끗한 집게를 사용하면 2 분, 1 분 동안 100 % 에탄올, 슬라이드를 전송 나는 2 분 동안 크실렌에 슬라이드를 담가, 자일 렌 II합니다.
- 배수 하락실온에서 보풀이없는 와이프와 공기 건조에 말이지.
Plastochron 7 잎 Primordia에서 블레이드, 잎혀와 칼집 샘플 3. 이외에 Microdissection
- LM 현미경의 무대에 슬라이드를 고정합니다. 슬라이드에 5 섹션을 이용하여, 각 복제에 대 한 대여섯 슬라이드를 사용합니다.
주 : 단일 복제 풀링 조직은도 5에 도시되어있다. - 슬라이드를 검사하고 중앙 기준점으로 SAM 정점을 사용하여 각 슬라이드에 다섯 대부분의 중간 부분을 식별합니다.
참고 :이 낮은 배율에서 수행 할 수 있습니다, 일반적으로 5 배의 목적은 충분하다. - 또는 10X 20X 대물를 사용하여, 각 부의 plastochron 7 리프 primordium에 잎혀의 위치를 식별한다. 잎혀는 잎 primordium의 향축 표면에서 돌출 범프로 볼 수 있습니다. LM 소프트웨어의 그리기 도구를 사용하여이 위치를 표시; 연필 아이콘을 선택하고 적절한 positi로 커서를 이동및 클릭하고 그릴 마우스를 드래그합니다.
참고 : 10 배 또는 20 배의 목적은이 이후의 단계에 적합합니다. 측면부를 사용할 때, 각각의 리프 primordium의 양측은 각각의 섹션 (도 2A)으로 존재할 것이다. - 눈금자 도구 직사각형 그리기 도구를 사용하여, 100 ㎛의 각각의 섹션의 중심 잎혀 높은 사각형 (적색 직사각형,도 2A, 2B)를 측정한다. 이들은 "잎혀"샘플 될 것입니다.
- 눈금자 도구를 사용하여; 눈금자 아이콘을 선택하여 측정 대상의 일단에 커서를 이동 대상물을 측정하기 위해 클릭하고 드래그. 눈금자의 길이는 화면에 표시한다.
- 사각형을 그리려면; 사각형 아이콘을 선택, 사각형의 한쪽 모서리 수 클릭하고 적절한 크기의 사각형을 그립니다 드래그하는 지점으로 커서를 이동합니다. 또한, 직선 그리기 도구를 선택하고 네 개의 직선을 그립니다.
- 측정 (10)0 ㎛의 직사각형 50㎛의은 "잎혀"사각형의 상하 위치.
참고 :이은 "블레이드"와 "칼집"샘플, 각각 (녹색 및 파랑 사각형, 그림 2A, 2B)을 할 것이다. 우리 학적 데이터에 기초하여, 100㎛의 직사각형 전체 잎혀 영역을 포함한다. 블레이드와 시스의 동등한 크기의 부분은 조직의 유사한 양의 각 수집되었는지 확인하기 위해 선택되었다. 50 ㎛, 스페이서는 더 잎혀 지역 조직이 실수로 날 또는 외장 microdissections에 포함되지 않도록하기 위해 사용되었다.
- 별도의 튜브에 잎혀, 블레이드 앤 시스 샘플을 수집 7, 8, 9, - Microdissect 측정 사각형 (2 층도 2D). 선택한 도메인의 윤곽을 따라 조직 섹션을 통해 절단 레이저 절단 기능을 사용하십시오. CATA를 사용하여pult 기능은 슬라이드 오프 튜브 (- 2 층 그림 2D)의 뚜껑에 조직의 사각형을 추진한다.
Plastochron 7 잎 Primordia에서 블레이드, 잎혀와 칼집 향축 표피 샘플 4. 이외에 Microdissection
- 섹션을 선택하고 섹션 3 (위)에 설명 된대로 plastochron 7 잎혀을 중심으로 100 μm의 높은 부분을 측정하는 눈금자 도구를 사용합니다.
- 드로잉 도구로 요약하여 각 100 μm의 높은 "블레이드"와 "칼집"세그먼트 (녹색 및 파랑 선택, 그림 2C) 만 향축 표피 세포를 선택합니다. 표피는 외부 전지 층이고; 향축면은 SAM에 가장 가까운 하나입니다.
- 3.3 절 (빨간색 선택, 그림 2C)에 설명 된대로 "잎혀"샘플의 경우, 새로운 잎혀 범프의 셀을 선택합니다.
- 블레이드, 잎혀와 시어 수집 Microdissect 선택된 영역,3.5 절에 설명 된대로 번째는, 별도의 튜브에 샘플을 표피.
LG1-R 및 야생 형 형제 자매에서 Plastochron 6 잎 Primordia 5. 이외에 Microdissection
- 돌연변이 (LG1-R) 및 야생형 식물의 가족을 분리하는 성장.
- 섹션 1.2-1.4에 설명 된대로, LM에 대한 정점을 수정하고 프로세스 촬영. 별도의 그들을 유지하기 위해 야생 형 및 별도의 유리 병에 돌연변이 촬영 정점을 수정합니다. 같은 재배에서 샘플은 고정 및 현장 하이브리드에 대해 처리해야한다.
- LG1은 계내 혼성화 6, 26, 27 (LG1)을 수행하여, 야생형 형제의 전사 위치를 결정한다. 여러 샘플 (그림 1E)의 잎 primordium의 기지에서 LG1 성적 증명서 축적의 위치를 측정한다.
- 동일계 하이브리드 화 데이터에 기초하여, 리프 프리모 부분을 선택할rdium 그 LG1이 전사되는 영역을 포함한다. 이 경우, 6 plastochron 리프 primordia (보라색 사각형,도 2a)의베이스 400-900 μm의.
- 3.5 절에 설명 된대로 Microdissect 별도의 튜브에 LG1-R 및 야생형 샘플을 수집, 잎 primordia의 부분을 선택했습니다.
6. RNA 추출 버퍼 적용
- microdissected 조직에 50 μl의 RNA 추출 버퍼를 적용하고 RNA 추출을 진행합니다. 참조 6에 설명 된대로 RNA 추출, RNA 증폭, 도서관 건설, 시퀀싱 및 생물 정보학 분석을 계속합니다.
결과
약 1,000,000-1,500,000 μm의이 조직의 각각에 대해 수집 된 그림 2에 나와있는 LM 방식을 사용하면 모든 세포 층 LM에 (그림 5)를 복제하고, 20 μm의 2 당은 향축 표피 LM에 대한 복제합니다. 각각 LG1-R 및 야생형 잎 primordia의 LM에 복제 약 2,500,000 μm의 조직이 수집되었다. 선형 RNA 증폭의 두 라운드는 각 복제에 대 한 RNA의 마이...
토론
실험 설계는 RNA-서열 실험에서 중요한 요소이다. 중요한 고려 사항은 정확한 도메인 (들) 및 발달 단계 (들)을 분석하는 것과 비교가 이루어질 것이다이다. 출력은 두 개 이상의 상태 사이 DE되는 유전자의 목록은 전형적 이래로, 비교의 관점에서 생각하는 것이 중요하다. 모든 실험에서와 같이, 한 번에 단 하나의 변수를 변경하는 것이 중요하다. 다른 리프 도메인을 비교할 때, 예를 들어, 동일한 ?...
공개
The authors have nothing to disclose.
감사의 말
저자는 협업을 진행하고 잎혀 개발에 대한 토론을 자극하기위한 S. 메르 루사 감사합니다. 이 작품은 국립 과학 재단 (National Science Foundation) 교부금 MCB 1,052,051 및 IOS-1848478에 의해 지원됩니다.
자료
| Name | Company | Catalog Number | Comments |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials - 22 ml | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 mm x 15 mm x 5 mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60 °C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
참고문헌
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유