Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Experimentelles Design für Laser Mikrodissektion RNA-Seq: Lehren aus einer Analyse von Maisblattentwicklung
In diesem Artikel
Zusammenfassung
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Zusammenfassung
Gene eine wichtige Rolle bei der Entwicklung haben häufig räumlich und / oder zeitlich beschränkten Expressionsmuster. Oft werden diese Gentranskripten werden nicht erkannt oder nicht als differentiell exprimierte (DE) in transkriptomischen Analysen von ganzen Pflanzenorgane identifiziert. Laser Mikrodissektion RNA-Seq (LM RNA-Seq) ist ein leistungsfähiges Werkzeug, um Gene zu identifizieren, die DE sind in spezifischen Entwicklungsdomänen. Die Auswahl der zellulären Domänen jedoch auf microdissect und zu vergleichen, und die Genauigkeit der Mikrodissektionen sind entscheidend für den Erfolg der Experimente. Hier sind zwei Beispiele zeigen Design-Überlegungen für Transkriptomik Experimente; a LM RNA-seq Analyse Gene zu identifizieren , die Gene entlang der Maisblatt proximal-distalen Achse, und ein zweites Experiment DE sind zu identifizieren , die in DE liguleless1-R (LG1-R) sind Mutanten im Vergleich zum Wildtyp. Wesentliche Elemente, die zum Erfolg dieser Versuche beigetragen wurden histologische und in si detaillierttu Hybridisierung der Region Analysen analysiert werden, die Auswahl der Blattprimordien in äquivalenten Entwicklungsstadien, die Verwendung von morphologischen Sehenswürdigkeiten Regionen auszuwählen , für die Mikrodissektion und Mikrodissektion von genau gemessen Domänen. Dieses Papier ein detailliertes Protokoll für die Analyse der Entwicklungs-Domänen durch LM RNA-Seq. Die hier präsentierten Daten veranschaulichen, wie die Region für die Mikrodissektion ausgewählt wird, die erhaltenen Ergebnisse beeinflussen.
Einleitung
Die Maisblatt ist ein ideales Modell die Bildung von Entwicklungsfelder während der Morphogenese zu untersuchen, da es eine deutliche Grenze zwischen der Klinge und Hülle aufweist , die zu genetischen Dissektion (1A) zugänglich ist. Während der frühen Stadien der Blattentwicklung, einem linearen Band von kleineren Zellen, die preligule Band (PLB), unterteilt die Blatt primordium in vorge Klinge und pre-Mantel-Domänen. Ein saumartig ligule und Dreiecks Vorhöfe entwickeln sich aus der PLB (1A, C, D). Genetische Screens identifiziert Mutationen, die die Klingenscheide Grenze unterbrechen. Zum Beispiel, rezessiv liguleless1 (LG1) Mutationen löschen Sie die ligule und Vorhöfe 1, 2, 3, 4 (1B). In - situ - Hybridisierung zeigte , dass LG1 Transkript sammelt sich an der PLB und Schwellen ligule, es eine ausgezeichnete Marker für ligule Entwicklung 5, 6 (Abbildung 1E) zu machen.
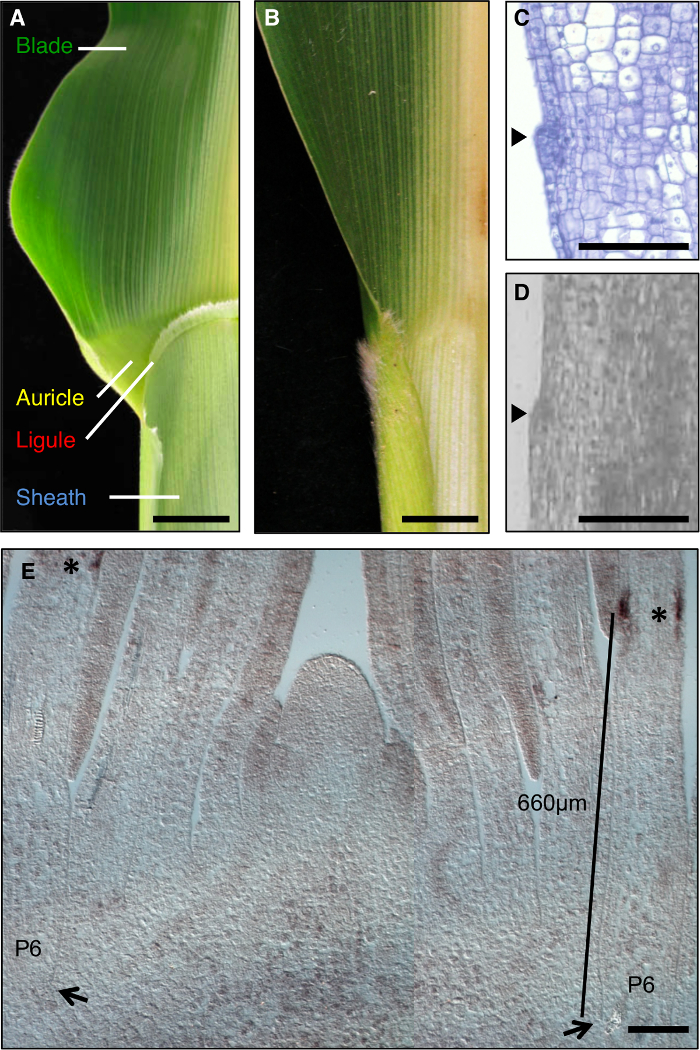
Abbildung 1: Wildtyp und liguleless1-R Maisblätter. (A) Blattscheide Randbereich des reifen Blatt Wildtyp zeigt ligule und Ohrmuschel Strukturen. (B) Blattscheide Grenzbereich von reifen liguleless1-R Blatt zeigt Abwesenheit von ligule und Ohrmuschel Strukturen. Blätter in der A und B wurden in zwei Hälften entlang der Mittelrippe geschnitten. (C) Längsschnitt durch Wildtyp - Blatt primordium. Probe wurde für die histologische Analyse verarbeitet und gefärbt. Der initiierende ligule ist offensichtlich, wie eine Unebenheit von der Ebene des Blattes vorsteht (Pfeilspitze). (D) Längs SekteIon durch Wildtyp-Blatt primordium. Probe wurde für LM verarbeitet, wie im Text beschrieben. Pfeilspitze zeigt ligule initiieren. (E) LG1 in - situ - Hybridisierung von Spross seitlichen Längsschnitt. Sternchen zeigen LG1 Transkriptakkumulation an der PLB des P6 Blatt primordium. Die Pfeile zeigen die Basis von P6 primordium. Balken zeigt Messung von der Basis des primordium zum PLB. Maßstabsbalken in A und B = 20 mm. Maßstabsbalken in CE = 100 & mgr; m. Diese Zahl wurde von Referenz 6 (Urheberrecht American Society of Plant Biologists) modifiziert. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
In dieser Studie wurde LM RNA-Seq verwendet eine Reihe von Genen zu identifizieren, die differentiell exprimierte (DE) an der Messerscheide Grenze relativ zu anderen Teilen des Blatt primordium und ide ntify Gene , die DE in LG1-R - Mutanten im Vergleich zu Wildtyp - Geschwister sind. LM RNA-Seq ist ein Verfahren der 7 Transkriptakkumulation in spezifischen Zellen oder zellulären Domänen zu quantifizieren. LM-Systeme kombinieren einen Laser und ein Mikroskop mit einer Digitalkamera. Geteilter Gewebe wird auf Schlitten montiert und durch das Mikroskop betrachtet. Die LM-Software enthält in der Regel Zeichenwerkzeuge, die dem Benutzer ermöglichen, alle ausgewählten Region für die Mikrodissektion zu skizzieren. Die Laserschnitte entlang der Linie, und das ausgewählte Gewebe wird die Folie und in ein Rohr über dem Objektträger aufgehängt katapultiert. LM ermöglicht es dem Benutzer präzise Domains microdissect, einschließlich spezifischer Zellschichten und sogar einzelne Zellen 8, 9. RNA kann dann aus dem microdissected Gewebe extrahiert werden. Anschließend wird die RNA-Seq Komponente der nächsten Generation Sequenzierung Sequenzierung von cDNA - Bibliotheken erzeugt aus der extrahierten RNA 10 verwendet,= "xref"> 11.
Die wichtigsten Vorteile von LM RNA-seq sind die Fähigkeit , Transkriptakkumulation in genau definierten Bereichen und die Fähigkeit zum Profil des gesamten Transkriptom gleichzeitig 7 zu quantifizieren. Die Technik eignet sich besonders für frühe Entwicklungs Ereignisse Sondieren, wo die Region von Interesse ist oft mikroskopisch. Frühere Studien haben LM mit Microarray - Technologie in Kombination verwendet , um Entwicklungsprozesse in Pflanzen 9, 12, 13 studieren. RNA-Seq hat den Vorteil der Transkripte in einem breiten Dynamikbereich, einschließlich niedrig exprimierten Genen Quantifizieren und vor Sequenzinformation ist nicht erforderlich , 10, 11. Darüber hinaus hat LM RNA-Seq das Potenzial ihrer Entwicklung wichtige Gene hervorzuheben, die durch genetische Redundanz in Mutagenese-sehen werden oder zu Letalität der Verlust-of-Funktion Mutante.
Entwicklungs wichtige Gene, wie schmal sheath1 (NS1) und becherförmigen cotyledon2 (CUC2), haben oft spezifische Expressionsmuster von nur einem oder wenigen Zellen 17, 18, 19, 20. Viele von ihnen sind in der frühen Entwicklungsstadien und nicht in der reifen Organ nur zum Ausdruck gebracht. Wenn ganze Organe oder große Domänen analysiert werden, wobei diese zellspezifische Transkripte werden verdünnt und kann nicht in konventionelleren Analysen nachgewiesen werden. Durch Analysen von genau definierten Domänen ermöglicht, LM RNA-Seq ermöglicht diese gewebespezifischen Gene identifiziert und quantifiziert werden.
Entscheidende Faktoren für den Erfolg der hier beschriebenen Experimente wurden eine gründliche histologische Analyse, die Auswahl der geeigneten Entwicklungsstadium und Domäne für die Analyse geführt und präzise measurement von Zell-Gewebe-Domänen für LM. Um sicherzustellen , dass äquivalente Domänen für alle Wiederholungs abgetastet wurden, Gewebe wurde aus Blattprimordien auf der gleichen Entwicklungsstadium gesammelt und die microdissected Domänen wurden relativ gemessen zu morphologischen Orientierungspunkte wie die Schwellen ligule (Abbildung 2). Es ist bekannt, dass einige Gene in einem Gradienten von der Spitze bis zur Basis des Blattes exprimiert werden. Durch die Messung präzise Domänen Variation aufgrund der Abtastung von verschiedenen Stellen entlang des Blatt proximal-distalen Achse auf ein Minimum (3A) gehalten werden . Von Domänen der gleichen Größe microdissecting Variation aufgrund unterschiedlicher Verdünnung von zellspezifische Transkripte wurde ebenfalls reduziert werden (3B). Seitliche Längsschnitte des Spross wurden für alle Mikrodissektionen verwendet. Dies sind Bereiche , die senkrecht zur Mittelrippe-margin Achse (Abbildung 4). Mit nur Abschnitte, die die SAM umfassen wird sichergestellt, dass äquivalente Seitenbereiche vonBlattprimor analysiert.
In Proben verarbeitet und LM, der erste morphologischen Zeichen von ligule Auswuchs geschnitten ist eine Beule auf der oberseits aufgrund perikline Zellteilungen in den adaxial Epidermis (1D, 2). Es wurde festgestellt, dass die Schwellen ligule zuverlässig bei Plastochron 7 Stadium Blattanlagen identifiziert werden konnten. Wir waren daran interessiert, Gene in der gesamten Region ligule ausgedrückt, einschließlich der Schwellen ligule und die Zellen unmittelbar distal, die die Ohrmuschel bildet. Um sicherzustellen , dass Selektionen äquivalente Gewebe hergestellt wurden, wurde die ligule Bump als morphologische Wahrzeichen verwendet und eine 100 & mgr; m Rechteck auf der ligule Bump zentriert wurde LM (2A, 2B) ausgewählt. Equivalent große Rechtecke vorge Klinge und pre-Mantel wurden aus den gleichen Blattprimordien ausgewählt.
Die Analysen der liguleless mutierten Pflanzen präsentiert eine andere challenge; LG1-R - Mutanten bilden keine ligule, daher ist diese morphologische Funktion nicht verwendet werden, um die Region für LM auszuwählen. Stattdessen wurde die Domäne von LG1 Transkriptakkumulation in Wildtyp - Blattanlagen bestimmt, und eine Region , die diese Domäne wurde definiert umfassen würde. Diese vorläufigen Analysen wurden auf Sämlingen aus demselben Anpflanzung durchgeführt, wie für die endgültige Analyse verwendet wurden, da frühere Arbeiten haben gezeigt, dass der Ort des PLB variiert je nach Wachstumsbedingungen abhängig. In - situ - Hybridisierung angegeben , dass LG1 - Transkripte in der PLB von P6 Blattanlagen (Abbildung 1E) akkumulieren. Wir haben für Sie eine Domäne 400-900 & mgr; m von der Basis des Blattes primordia, die die Domäne von LG1 Ausdruck umfasste (lila Rechtecke, 2A) und erfasst diese äquivalenten Regionen von Wildtyp und LG1-R Pflanzen. Zur Minimierung der Variation in der genetischen Hintergrund und Wachstumsbedingungen bei einem Vergleich TRANSCRIPt Akkumulation in LG1-R und Wildtyp - Pflanzen, Aussortieren Familien von Mutanten und Wildtyp - Geschwister wurden verwendet.
Protokoll
HINWEIS: Fix Gewebe für die histologische Analyse zur gleichen Zeit, dass Gewebe für LM fixiert ist. Gefärbten Abschnitte für morphologische Merkmale, die später LM führen wird. Wenn Mutante Wildtyp - Vergleich, führen in - situ - Hybridisierung oder Immunolokalisation die Domäne zu definieren , in denen das Gen von Interesse (in diesem Fall LG1) exprimiert wird.
1. Gewebefixierung und Verarbeitung
- Wachsen Wohnungen von Maiskeimlinge bis zwei Wochen alt unter Standardbedingungen 6.
- Sezieren shoot Scheiteln zur seitlichen Abschnitte (Abbildung 4).
- Excise Sämling knapp unterhalb der Bodenlinie.
- Mit einer Rasierklinge, entfernen dünne Scheiben von der Basis des Stiels (Schnitte 1, 4A) , bis ein Oval von culm umgeben von ein oder zwei reifen Blätter sichtbar ist (4B).
- Legen Sie einen anderen Schnitt etwa 10 mm über dem Boden (2 geschnitten, 4A).Das 10 mm-Segment wird die SAM und jungen Blattanlagen enthalten.
- Drehen Sie das 10 mm-Segment, so dass die Basis nach oben zeigt. Machen Sie zwei Schnitte zur Querachse parallel , so dass ein Stück Gewebe 2-3 mm Dicke erhalten wird (Schnitte 3 und 4, 4B). Entsorgen Sie die beiden äußeren Teile und behalten die zentrale Scheibe zur Fixierung und Einbettung.
HINWEIS: Äußere Blätter abgeschnitten und entsorgt werden können.
- Fix Gewebe und Verfahren zum Einbetten.
- Sicherzustellen, dass alle Materialien, die in nachfolgenden Schritten verwendet werden, sind RNase frei. Behandeln Sie Lösungen mit Diethylpyrocarbonat (DEPC) (1 ml DEPC pro Liter Lösung. Inkubieren über Nacht mit gelegentlichem Schütteln und Autoklaven). Bake Glas in einem Ofen bei 200 ° C oder höher für mindestens 6 Stunden und Behandlung von Kunststoff-ware mit RNase Dekontaminationslösung.
- Tag 1: Tauchen Gewebescheiben in ~ 10 ml Bauern Fix (3: 1 Ethanol: Essigsäure) in Glasfläschchen auf dem Eis. Nachdem alle Proben präpariert wurden, gelten VACUUm, um Luftblasen und Hilfe Eindringen von Fixiermittel zu entfernen. Halten unter Vakuum für 10 Minuten dann langsam das Vakuum freizugeben. Ersetzen Fixiermittel und Inkubation bei 4 ° C über Nacht unter leichtem Schütteln.
- Tag 2: Inkubieren in der folgenden Reihe von Lösungen, ~ 10 ml, 1 h jeweils alle unter leichtem Schütteln; 85% Ethanol bei 4 ° C, 95% Ethanol bei 4 ° C, 100% Ethanol bei 4 ° C, 100% Ethanol bei 4 ° C, 100% Ethanol bei 4 ° C, 1: 1 Ethanol: Xylole bei Raumtemperatur, 100% Xylole bei Raumtemperatur, 100% Xylole bei Raumtemperatur.
HINWEIS: Xylenes giftig sind durch Berührung und Einatmen. Die Arbeit in einer Abzugshaube und geeignete Handschuhe verwenden. - In Paraffin Gewebe Einbettmediums Pellets auf ungefähr die Hälfte von Xylolen und Inkubation über Nacht bei Raumtemperatur unter leichtem Schütteln.
- Tag 3: Übertragen Sie die Fläschchen auf 60 ° C heißen Ofen, bis die Pellets zu schmelzen. Abgießen Lösung und ersetzen mit frischen geschmolzenen Gewebeeinbettungsmedium. Ändern Sie die Medium zwei weitere Malewährend des Tages.
- Tag 4: Gewebe ändern Medium einmal morgens einzubetten. Zurück zu 60 ° C warmen Ofen bis zum Nachmittag.
- Gussblöcke
- Legen Sie Einbettschälchen auf heißer Platte von Gewebeeinbettung Station. Verwenden einer Pinzette, um die Gewebeproben in die Einbettformen zu übertragen mit der Schnittfläche nach unten zeigt. Top die Form mit geschmolzenem Paraffin und legen die Einbettung Ring an der Oberseite der Form. Übertragung auf eine kalte Platte, bis Paraffin erstarrt ist. Speichern der Paraffinblöcke bei 4 ° C in einem luftdichten Behälter mit Kieselgel.
2. Sectioning und Vorbereitung Schieben
- Schnitt 10 um Abschnitte auf einem Mikrotom 25.
- Untersuchen Sie Bänder und wählen mittlere Abschnitte. Median Abschnitte sind solche, die die SAM umfassen, die durch Blattprimor umgeben als Kuppel der Zellen angezeigt.
- Berg Abschnitte auf Folien.
- Die Objektträger, die geeignet sind für LM (entweder RNase frei odergebacken) auf 42 ° C wärmer gleiten und einige Tropfen 50% igen Ethanollösung gelten die Folie abzudecken.
- Float Abschnitte auf Ethanollösung, bis die Abschnitte erweitert.
HINWEIS: Floating Abschnitte auf Ethanollösung statt Wasser hält RNA-Abbau-RNA in einer gefällten Zustand zu reduzieren. - Kippschiebers und überschüssiges Ethanollösung durch Absaugen mit einem Einweg-Transferpipette. Verwenden Sie fusselfreien Tüchern zusätzliche Ethanollösung Feuchtigkeit ab.
- Trockenobjektträger bei 42 ° C für mehrere Stunden oder über Nacht. Store Objektträger bei 4 ° C in einem luftdichten Behälter mit Kieselgel.
- Entparaffinieren gleitet am Tag der Verwendung.
- Bereiten Sie drei Glas Färbetrog enthält; 100% Xylole (Xylole I), 100% Xylole (Xylole II) und 100% Ethanol (~ 50 ml von jeder Lösung).
- Mit sauberen Pinzette zu übertragen Dias, tauchen Dias in Xylolen ich für 2 min, Xylole II für 2 min und 100% Ethanol für 1 min.
- Ablass rutschtees auf fusselfreien Tüchern und Luft bei Raumtemperatur trocknen.
3. Mikrodissektion von Blade, Blatthäutchen und Mantel Proben aus Plastochron 7 Blatt Primordia
- Sichern Sie sich die Folien auf der Bühne von LM Mikroskop. Verwenden Sie fünf oder sechs Folien für jede Wiederholung, unter Verwendung von fünf Abschnitte pro Folie.
HINWEIS: Gewebe für ein einzelnes Replikat Pooling ist in Abbildung 5 dargestellt. - Untersuchen Sie Dias und identifizieren die fünf mittlere Abschnitte auf jeder Folie, die SAM-Spitze als zentraler Bezugspunkt.
HINWEIS: Dies kann bei geringer Vergrößerung durchgeführt werden, in der Regel ein 5X Ziel ausreichend ist. - Mit 10X oder 20X Ziel, identifizieren die Position des ligule auf dem Plastochron 7 Blatt primordium jedes Abschnitts. Die ligule wird als Bump sichtbar sein aus der adaxial Oberfläche des Blattes primordium vorsteht. Markieren Sie diese Position mit dem Zeichenwerkzeug der LM-Software; Wählen Sie das Stift-Symbol, um den Cursor auf die entsprechende positi bewegenauf und klicken Sie auf und ziehen Sie die Maus zu ziehen.
HINWEIS: Ein 10X oder 20X Ziel ist geeignet für diese und die nachfolgenden Schritte. Wenn Seitenabschnitte verwendet, werden die beiden Seiten jedes Blattes primordium in jedem Abschnitt (2A) zugegen sein. - Mit dem Lineal - Werkzeug und die rechteckige Zeichenwerkzeug, messen 100 & mgr; m hohe Rechtecke zentriert auf dem ligule jedes Abschnitts (rote Rechtecke, 2A, 2B). Diese werden die "Blatthäutchen" Probe sein.
- Um das Lineal-Werkzeug; Wählen Sie das Lineal-Symbol, um den Cursor auf ein Ende des Objekts bewegen gemessen werden, klicken Sie auf und ziehen Sie das Objekt zu messen. Die Länge des Lineals wird auf dem Bildschirm angezeigt.
- So zeichnen Sie ein Rechteck; Wählen Sie das Rechteck-Symbol, um den Cursor auf einen Punkt zu bewegen, die eine Ecke des Rechtecks sein wird, klicken Sie auf und ziehen Sie ein Rechteck in der entsprechenden Größe zu zeichnen. Alternativ können Sie das Zeichenwerkzeug gerade Linie und vier geraden Linien zeichnen.
- Maßnahme 100 um Rechtecke 50 positioniert, um oberhalb und unterhalb des "Blatthäutchen" Rechteck.
HINWEIS: Diese werden die "Blade" und "Mantel" Proben, bzw. (grüne und blaue Rechtecke, 2A, 2B) sein. Auf der Grundlage unserer histologischen Daten umfasst ein 100 & mgr; m Rechteck den gesamten ligule Region. Equivalent große Portionen von Klinge und Hülle wurden ausgewählt, um sicherzustellen, daß ähnliche Mengen von Gewebe für jede gesammelt. Abstandshalter von 50 um wurden verwendet, um sicherzustellen, dass kein Bereich ligule Gewebe wird in den Schaufel oder Hülle Mikrodissektionen versehentlich eingeschlossen.
- Microdissect gemessen Rechtecke (2D - 2F) 7, 8, 9, sammeln Blatthäutchen, Klinge und Mantel Proben in getrennten Röhren. Verwenden Sie die Laser-Cut-Funktion durch den Gewebeschnitt entlang der Kontur der ausgewählten Domäne zu schneiden. Verwenden Sie die Catapult Funktion das Rechteck von Gewebe aus der Folie und in den Deckel des Rohres (- 2F 2D) zu treiben.
4. Mikrodissektion von Blade, Blatthäutchen und Mantel adaxial Epidermal Proben von Plastochron 7 Blatt Primordia
- Wählen Sie Abschnitte und verwenden Sie das Lineal zu messen 100 & mgr; m hohe Segmente zentriert auf dem Plastochron 7 ligule, wie in Abschnitt 3 (siehe oben) beschrieben.
- Wählen Sie nur die adaxial epidermalen Zellen von jeweils 100 & mgr; m hoch "Blade" und "Mantel" Segment (grün und blau Auswahlen, 2C) , indem sie mit dem Zeichenwerkzeug umreißt. Die Epidermis ist die äußere Zellschicht; die adaxial Seite ist der am nächsten zum SAM.
- Für die "Blatthäutchen" Probe, wählen Sie nur die Zellen des entstehenden ligule Beule , wie in Abschnitt 3.3 (rote Auswahl, 2C) beschrieben.
- Microdissect ausgewählten Regionen, sammeln Blade, Blatthäutchen und Sheath Epidermis Proben in getrennten Röhren, wie in Abschnitt 3.5 beschrieben.
5. Mikrodissektion von Plastochron 6 Blatt Primordia von LG1-R und Wildtyp - Geschwister
- Wachsen Familien von mutierten Aussortieren (LG1-R) und Wildtyp - Pflanzen.
- Fix und Prozess schießen Scheiteln für LM, wie 1,2-1,4 in Abschnitten beschrieben. Fix Wildtyp und Mutante schießen Scheiteln in separaten Fläschchen, um sie getrennt zu halten. Proben aus dem gleichen Pflanzungs sollte für die in situ - Hybridisierung fixiert und verarbeitet werden.
- Bestimmen Sie, wo LG1 in Wildtyp - Geschwister transkribiert wird, von LG1 in - situ - Hybridisierung Durchführung 6, 26, 27. Messen die Position von LG1 Transkriptakkumulation von der Basis des Blatt primordium in mehreren Proben (Abbildung 1E).
- Basierend auf in - situ - Hybridisierungsdaten, wählen Teil des Blattes primordium daß umfasst die Region , in der LG1 transkribiert wird. In diesem Fall 400-900 & mgr; m von der Basis des Plastochron 6 Blattanlagen (lila Rechtecke, 2A).
- Microdissect Teil des Blattanlagen ausgewählt, wie in Abschnitt 3.5, sammeln LG1-R und Wildtyp - Proben in getrennten Röhren.
6. Wenden RNA Extraction Buffer
- Bewerben 50 ul RNA-Extraktionspuffer zu microdissected Gewebe und gehen mit RNA-Extraktion. Fahren Sie mit dem RNA - Extraktion, die RNA - Amplifikation, Bibliothekskonstruktion, Sequenzierung und Bioinformatik - Analyse , wie in Bezug auf 6 beschrieben.
Ergebnisse
Die LM - Schema in Abbildung 2 wurde für jede Wiederholung gesammelt 1,000,000-1,500,000 um 2 von Gewebe etwa skizziert Verwendung in der all-Zell-Schichten LM (Abbildung 5), und 200.000 & mgr; m 2 pro für die adaxial Epidermis LM replizieren. Etwa 2,5 Millionen um 2 von Gewebe gesammelt wurde für jede in der LM von LG1-R und Wildtyp - Blattanlagen replizieren. Zwei Runden von linearen RNA-Amplifikation er...
Diskussion
Experimentelles Design ist ein kritischer Faktor in RNA-seq Experimenten. Wichtige Überlegungen sind die genaue Domain (s) und des Entwicklungsstadiums (en) analysiert werden, und welche Vergleiche gemacht werden. Es ist von entscheidender Bedeutung im Hinblick auf die Vergleiche zu denken, da die Ausgabe typischerweise eine Liste von Genen ist, die zwischen zwei oder mehr conditions de sind. Wie bei allen Experimenten ist es wichtig, nur eine Variable zu einer Zeit zu ändern. Zum Beispiel, wenn verschiedene Blatt Dom...
Offenlegungen
The authors have nothing to disclose.
Danksagungen
Die Autoren danken S. Hake für die Zusammenarbeit im Gange und Diskussionen über ligule Entwicklung zu stimulieren. Diese Arbeit wird von der National Science Foundation Grants MCB 1052051 und IOS-1848478 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials - 22 ml | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 mm x 15 mm x 5 mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60 °C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
Referenzen
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten