Method Article
שיטת הפרעות RNA בתתיווך אלקטרופולציה באודונה
In This Article
Summary
אנו מספקים פרוטוקול מפורט להפרעת רנ"א מתווכת אלקטרופורציה בחרקים מסדר אודונטה (שפיריות וסכרים) באמצעות הסכר הכחול זנב (Ischnura senegalensis: Coenagironidae: Zygoptera) ואתשפירית הרחפן pied (פסאודותמיס זונאטה: Libellulidae: אניסופטרה).
Abstract
שפיריות ועלמות (סדר Odonata) מייצגים את אחד החרקים אבותינו ביותר עם מטמורפוזה, שבו הם משנים את בית הגידול שלהם, מורפולוגיה, והתנהגות דרסטית מן הזחלים מימיים למבוגרים יבשתיים / אוויריים ללא שלב pupal. למבוגרים באודונטה יש ראיית צבעים מפותחת והם מראים מגוון מדהים של צבעי גוף ודפוסים על פני מינים, שלבים ומינים. בעוד מחקרים אקולוגיים והתנהגותיים רבים על Odonata נערכו, מחקרים גנטיים מולקולריים היו נדירים בעיקר בשל הקושי ליישם ניתוח תפקודי גנים על Odonata. לדוגמה, הפרעה RNA (RNAi) הוא פחות יעיל Odonata, כפי שדווח ב Lepidoptera. כדי להתגבר על בעיה זו, הקמנו בהצלחה שיטת RNAi בשילוב עם אלקטרופורציה in vivo. כאן אנו מספקים פרוטוקול מפורט הכולל וידאו של שיטת RNAi בתיווך אלקטרופורציה כדלקמן: הכנת זחלים, זיהוי מינים, הכנת פתרון dsRNA / siRNA ומחטי הזרקה, הרדמה קרה כקרח של זחלים, הזרקת dsRNA / siRNA, אלקטרופוריה vivo, וגידול אישי עד הופעת מבוגרים. שיטת RNAi בתתיווך אלקטרופורציה ישימה הן עלמה (תת-הזמנה זיגופטרה) והן על שפיריות (תת-הזמנה אניסופטרה). בפרוטוקול זה, אנו מציגים את השיטות עבור הסכר הכחול זנב Ischnura סנגלנסיס (Coenagrionidae) כדוגמה של מינים damselfly ואת רחפן pied שפירית Pseudothemis זונטה (Libellulidae) כדוגמה נוספת של מינים שפירית. כדוגמאות מייצגות, אנו מראים את התוצאות של RNAi מיקוד מלנין סינתזה גן multicopper אוקסידאז 2. שיטת RNAi זו תאפשר הבנה של פונקציות גנים שונות המעורבות במטהמורפוזה, מורפוגנזה, היווצרות תבנית צבע ותכונות ביולוגיות אחרות של Odonata. יתר על כן, פרוטוקול זה עשוי להיות ישים בדרך כלל על אורגניזמים שאינם מודל שבו RNAi הוא פחות יעיל בדיכוי גנים בשל חוסר היעילות וחדירה נמוכה.
Introduction
שפיריות ועלמים (הסדר Odonata) הם בין הקבוצות אבות אבות ביותר של חרקים המציגים "מטמורפוזה"1,2. על ידי מטמורפוזה, הם משנים את בית הגידול שלהם, מורפולוגיה, והתנהגות דרסטית מן הזחלים הימיים למבוגרים יבשתיים /אוויריים 3. למבוגרים Odonata יש ראיית צבע מפותחת ומייצגים מגוון מדהים של צבעי גוף ודפוסים על פני מינים,שלבים, ומינים 3,4,5. בעוד מחקרים אקולוגיים והתנהגותיים רבים על Odonataנערכו 6,7 , מחקריםגנטייםמולקולריים נבלמו בעיקר על ידי הקושי ליישם ניתוח תפקודי גנים על Odonata.
שיטת הפרעת RNA קונבנציונלית (RNAi), שבה מוזרק RNA כפול תקע (dsRNA) כדי לדכא אתהפונקציה של הגן של עניין 8, התברר להיות יעיל חרקים Odonata9, כפי שדווח חרקים Lepidopteran10. מצד שני, דיווחים קודמים הראו כי RNAi בתיווך אלקטרופולציה יעילה במין Lepidopteran, במיוחדברקמות אפידרמיס 11,12,13. לאחרונה מצאנו כי RNAi בתיווך אלקטרופורציה עובד ביעילות שפירית זעירה Nannophya פיגמאיה (Libellulidae: אניסופטרה)9, אבל N. פיגמאיה הוא מין נדיר יחסית ולכן אינו מתאים למחקרים גנטיים מולקולריים.
רוב מיני אודונטה מסווגים לאף אחד משני תתי-ההזמנות, זיגופטרה (עלמה) או אניסופטרה (שפיריות אמיתיות)3. כאן התמקדנו בסנגלנסיס של אישנורה (Coenagrionidae; איור 1A) כנציג המין הזיגופטראני ורחפן הפייד שפירית פסאודותמיס זונאטה (Libellulidae; איור 1B) כמין אניסופטראני מייצג. שני המינים הם בין מינים Odonata הנפוצים ביותר בבריכות טבעיות ועירוניות ביפן, כולל אלה בעיר Tsukuba, ואנחנו יכולים לאסוף זחלים רבים של שני המינים בתחום. לאחרונה הקמנו מערכת גידול מעבדה עבור זחלים בודדים של I. סנגלנסיס, אשר אפשר ניטור מתמשך של פיתוח ומורפוגנזה של הזחלים Odonataבפירוט 14.
בדו"ח זה, אנו מספקים שיטה מעודנת ופרוטוקול וידאו עבור RNAi מתווך אלקטרופולציה ב I. סנגלנסיס ו P. zonata. ביפן, I. סנגלנסיס ו Ischnura asiatica, אשר קרובים גנטית, נמצאים לעתיםקרובות סימפאטית 15, והם קשים להבחיןזחלים 16. אנו מתארים גם כיצד ניתן להבדיל בין שני מינים של אישנורה באמצעות פולימורפיזם באורך שבר הגבלה (RFLP).
להערכת האפקטיביות של RNAi מתווך אלקטרופולציה, אנו בוחרים multicopper oxidase 2 גן (MCO2; הידוע גם בשם laccase2) כגן יעד מייצג, על חשבון פנוטיפ גלוי של צבע קוטיקל חיוור עם הפלת ביטויהגן 9. MCO2 ידוע כחיוני להחשכה של האפידרמיס במגוון מיני חרקים17,18.
Protocol
הערה: הערכה הכוללת של הפרוטוקולים מוצגת איור 1.
1. הכנת זחלים של שפיריות או עלמה.
- לאסוף זחלים בשדה באמצעות רשת יד.
הערה: אני. זחלי סנגלנסיס נצמדים לעתים קרובות לצמחי מים צפים על פני המים, בעוד זחלי P. zonata לעתים קרובות להישאר בין המלטה עלים בתחתית. בעיר Tsukuba, הזחלים instar הסופי של I. סנגלנסיס נמצאים בעיקר ממרץ עד יוני, ואלה של P. zonata ממאי עד יוני. I. זחלי סנגלנסיס ניתן לגדל מביצים במעבדה14, אבל הזחלים שנאספו בתחום יש שיעור הצלחה גבוה יותר בבירור של הופעת מבוגרים. - זיהוי מינים על ידי פולימורפיזם אורך שבר הגבלה (RFLP) של מוצרים מוגברים PCR.
הערה: שלב זה נחוץ רק אם המין קשה לזהות מן המראה של הזחלים, כמו I. סנגלנסיס.- החזק אחד הזימים caudal של זחל באמצעות מדפים. לאחר מכן, הזחל נופל מהגיל caudal שלה עצמו (גרימת אוטוטומיה).
הערה: לזחלים של עלמה זיגופטרנית יש בדרך כלל שלושה זימים קאודאליים (איור 1A). כאשר הם מותקפים על ידי טורף, הם יכולים להוריד את הזימים caudal שלהם לברוח. אם הזימים הקוודאליים אינם זמינים, חלק מהרגל מנותחת. - שים אחד caudal זימ לתוך 100 μL של פתרון PBS [0.8% NaCl, 0.02% KCl, 0.115% Na2HPO4, ו 0.02% KH2PO4 (w /v)] ו homogenize עם מערבל יד באמצעות עלה הפקדה.
- סובב כלפי מטה את התערובת ב- 5,000 x g למשך 10 שניות, ונושא 0.5 μL של הגברה על-טבעית ל- PCR באמצעות פולימראז DNA פריימרים כדי להגביר את המאריך הפנימי מתמלל 1 (ITS1) אזור של ה-DNA הגרעיני.
הערה: בצע את ה- PCR בהתאם לפרוטוקול היצרן. השילוב של ITS-F0 (5'- GGA AAG ATG GCC AAA CTT GA -3') ו 5.8S-AS1 (5'- GCC GGC CCT CAG G -3') פריימרים יכולים להגביר את אזור ITS1 כמעט בכל מיני אודונטה19. - הדגירה את התערובת של 1 μL של מוצר PCR מוגברת, 0.3 μL של 10x M Buffer, 0.1 μL של אנזים הגבלת DraI, ו 1.6 μL של מים ב 37 °C (69 °F) עבור 1 שעות.
הערה: בחר את אנזימי ההגבלה הטובים ביותר, בהתאם לשילוב של מינים. אם אין אנזימי הגבלה המתאימים לזיהוי מינים, אשר על ידי ריצוף DNA. - לטעון 2 μL של המוצרים עם טעינת צבע על 2% ג'ל אגרוז ולהפעיל אלקטרופורזה.
הערה: קשה לזהות מינים בעת שימוש 1% או 1.5% ג'ל אגרוז. - בדוק את דפוס האלקטרופורזה כדי לזהות את המין (איור 2).
הערה: תבניות RFLP הן תלויות מינים ואוכלוסייה. באוכלוסיית Tsukuba, I. asiatica יש להקה גדולה של 400-500 bp, ואילו I. senegalensis יש להקה גדולה של כ 200 bp ורצועה נוספת של פחות מ 100 bp (ראש חץ איור 2). אזור ITS1 קיים בעותקים מרובים בגנום, ובמין אישי, פולימורפיזם מיקרו-סטלי בתוך אזור ITS1 קיים לעתים קרובות באותו אדם, המשפיע על התבנית של RFLPs.
- החזק אחד הזימים caudal של זחל באמצעות מדפים. לאחר מכן, הזחל נופל מהגיל caudal שלה עצמו (גרימת אוטוטומיה).
- מאחור את הזחלים שנאספו במעבדה עד לשימוש עבור RNAi.
- מניחים כל זחל בנפרד לתוך כל באר של צלחת 12 באר עם כ 3 מ"ל של מים.
הערה: I. זחלי סנגלנסיס חייבים להישמר בנפרד כי הם לעתים קרובות קניבליזציה אחד את השני, בעוד P. זחלים זונטה יכול להישמר בקבוצה כי הם לעתים רחוקות קניבליזציה. - להאכיל I. זחלי סנגלנסיס עם שרימפס מלח ארטמיה כל יום ו P. זונטה זחלים עם תולעי דם ו / או תולעי Tubifex לפחות פעמיים בשבוע עד שהם גדלים לשלב ההתפתחותי המתאים עבור RNAi.
הערה: האכלה תכופה היא קריטית כדי להגדיל את שיעור ההצלחה של הופעת מבוגרים. - לשנות את המים ברגע שהוא הופך מלוכלך עם צואה או שאריות.
הערה: שינוי מים תכופים חשוב גם כדי להגדיל את שיעור ההצלחה של הופעת מבוגרים. - שפטו את השלב ההתפתחותי המתאים עבור RNAi.
הערה: הזחלים instar הסופי של I. סנגלנסיס ניתן לסווג לתוך חמישה שלביםהתפתחותיים 14,20. השלב הראשון (שלב א', לפני שהכנפיים מתחילות להתרחב) או השלב השני (שלב ב') של זחלי instar הסופיים מתאים לניסויי RNAi, כאשר בוחנים את הפנוטיפ הברור של RNAi לאחר הופעת מבוגרים (ראו דיון). ב P. zonata, הזחלים instar הסופי לפני הרחבת הכנף (המקביל לשלב A של I. סנגלנסיס) שימשו במחקר זה.
- מניחים כל זחל בנפרד לתוך כל באר של צלחת 12 באר עם כ 3 מ"ל של מים.
2. הכנת פתרון dsRNA / siRNA ומחטי הזרקה עבור RNAi.
הערה: בחר RNA מפריע קטן (siRNA) (שלב 2.1) או RNA דו-תקע (dsRNA) (שלב 2.2) כפתרון עבור RNAi.
- הכנת פתרון siRNA
- עיצוב siRNA באמצעות siDirect תוכנית גירסה 2.0 (http://sidirect2.rnai.jp/)21,בעקבותההנחיות שדווחו בעבר חרקים Lepidopteran22.
- השג siRNA מסונתז מסחרית.
הערה: הרצפים של siRNA מיקוד גן MCO2 של I. סנגלנסיס בשימוש במחקר זה היו כדלקמן: 5'- GCA CUU UCC UCC AUC AAU AUA -3' עבור גדיל חוש ו 5'- UAU UGA UAA AAA AAA GUG CUC -3' עבור גדיל antisense. כשליטה שלילית, הרצפים של siRNA מיקוד חלבון פלואורסצנטי ירוק משופר (EGFP) הגן היו כדלקמן: 5 '- GCA UCA AGG AGG ACU ACA AGA -3' עבור גדיל חוש ו 5'- UUG AAG UUC UUG AUG CCG -3' עבור גדילantisense 11. - לדלל את siRNA ל 100 מיקרומטר עם חיץ הזרקה [100 mM CH3COOK, 2 מ"ג מ"ג (CH3COO)2, 30 mM HEPES-KOH, pH 7.4]22.
- יש לאחסן ב-80 מעלות צלזיוס עד לשימוש.
- הכנת פתרון dsRNA.
- בחר אזור 300-400 bp עבור dsRNA באמצעות תוכנית primer3 גירסה 4.1.0 (http://bioinfo.ut.ee/primer3/)23 ולעצב את ערכות פריימר.
- השג ערכות פריימר מסונתזות מסחרית.
הערה: כדי לייצר תבניות עבור סינתזת dsRNA, ערכות פריימר בשימוש במחקר זה היו כדלקמן: (5'- GCC TGT CAG CTT CTT CC -3' עבור פריימר קדימה ו 5'- GGT GTC TGG CGG ACA ACT AT -3' עבור פריימר הפוך) עבור גנים MCO2 של I. senegalensis (IsMCO2, גישה לא. LC589180) ו (5'- CCG CAC ACA CTA TTA AA -3' עבור פריימר קדימה ו 5'- GGA GGA TTC CTT CAT CGA CA -3' עבור פריימר הפוך) עבור גנים MCO2 של P. zonata (PzMCO2, הצלחה לא. LC589179). כשליטה שלילית, ערכת פריימר עבור dsRNA מיקוד β-lactamase (בלה)גן על וקטור שיבוט היו (5'- CTA TGT GGC GCG GTA TTA T -3' עבור פריימר קדימה ו 5'- CAG AAG TGG TCC TGC AAC T-3' עבור פריימר הפוך). - לחלץ RNA מן הזחלים Odonata באמצעות ערכת מיצוי RNA זמין מסחרית ולבצע סינתזה cDNA באמצעות תעתיק הפוך על פי פרוטוקול היצרן.
הערה: ניתן להשתמש גם בספריית cDNA המוכנה לניתוח רצף RNA. - הגבר את רצפי המטרה באמצעות cDNA מסונתז ואת ערכת פריימר מעוצב לשבט אותם לתוך וקטור שיבוט באמצעות רצועות מסחריות על פי פרוטוקול היצרן.
- להפוך את הפלסמיד לתאים מוסמכים E. coli ולאסוף מושבה אחת לאחר הדגירה לילה.
- PCR-הגבר את אזור ההוספה באמצעות פריימרים בווקטור.
- אשר את הרצף המשוכפל על-ידי רצף סנגר.
- PCR-להגביר את הכנס באמצעות פריימרים וקטוריים המכילים את רצף מקדם פולימראז T724, 25.
הערה: במחקר זה, פריימרים הבאים שימשו: T7-F (5' - TAA TAC GAC TCA CTA תג GGA GAC תג TCA TAT GGA T - 3 ') ו T7-R (5'- TAA TAC GAC TCA תג CTA GGA GGG GGG GGG ATC CGA T - 3') 25. - לטהר את המוצר PCR באמצעות ערכת טיהור PCR על פי פרוטוקול היצרן, לרחף את ה-DNA עם 50 μL של מים מזוקקים, ולרכז את פתרון ה-DNA אלוט כ 10 μL באמצעות אידוי צנטריפוגלי.
- לסנתז dsRNA על ידי תמלול במבחנה על פי פרוטוקול היצרן. השתמש בסך הכל של 1000 ננוגרם תבנית DNA ו dsRNA מסונתז elute עם 100 μL של מאגר אלוטיון.
- למדוד את הריכוז של dsRNA באמצעות ספקטרופוטומטר ולאשר את האיכות של dsRNA על ידי אלקטרופורזה על 1.5% ג'ל אגרוז.
הערה: ניתן לראות רצועה אחת כאשר סינתזת dsRNA מצליחה. - לדלל dsRNA ל 1000 ng/μL עם מאגר אלוטיון ולאחסן ב -20 °C (60 °F) עד השימוש.
- הכנת מחטי הזרקה
- משוך נימי זכוכית באמצעות מושך מחט זכוכית.
- מניחים את קצה נימי משך על סרט דבק דו צדדי ולשבור את קצה נימי עם מדפים.
- תן את נימי למזרק.
הערה: קל יותר לטפל נימי אם אתה לובש כפפות ניטריל. - טען תמיסת siRNA/dsRNA ל נימי מוכן.
הערה: אין להשתמש בנימי שוב ושוב מכיוון שטיפ נימי מוזרק עלול להיות סתום בעפר מכיוון שהזחלים שנאספו בשדה חיו בבוץ.
3. הזרקת siRNA/dsRNA
הערה: ההליך שונה במקצת עבור damselflies (שלב 3.1) שפיריות (שלב 3.2).
- הזרקה לזחל זיגופטרני (Damselfly) (למשל, I. סנגלנסיס).
- הרדמה של זחל מכוסה בנייר רטוב על קרח כתוש למשך 50-70 שניות (איור 3A-C).
הערה: משך ההרדמה הקרה כקרח תלוי במצב הזחל; אם הזחל מתחיל לנוע לאחר 70 שניות של הרדמה קרה כקרח, מוחלות 70 שניות נוספות של הרדמה קרה כקרח. עבור זחלים כי לעתים רחוקות לנוע (למשל, P. zonata), הליך זה אינו חיוני. - צרף שני פינים משני צידי הפרותורקס ותקן את הזחל לעמוד קבוע (למשל, חתיכת קלקר) (איור 3D).
הערה: התאימו את המיקום ואת מספר הפינים, בהתאם למקום ההזרקה. - למתוח את הממברנה הבין-מגזרית בין קטע הבטן 7 ו -8 עבור RNAi בבטן או בין פרותורקס וסינתורקס (mesothorax מותך metathorax) עבור RNAi בבית החזה באמצעות ידיים ומדפים.
- יש למתוח את הממברנה הבין-מגזרית ביד.
- הכנס את קצה נימי מוכן לתוך הממברנה הבין-מגזרית המתוחה (איור 3D, 3F).
- להזריק 1 μL של פתרון siRNA / dsRNA.
- הרדמה של זחל מכוסה בנייר רטוב על קרח כתוש למשך 50-70 שניות (איור 3A-C).
- הזרקה לזחל אניסופטראן (שפירית) (למשל, פ. זונטה).
- מנגבים את המים מפני הזחל במגבת נייר.
- במידת הצורך, צרפו שני סיכות משני צידי הפרותורקס וסדרו את הזחל לעמוד קבוע (למשל, חתיכת קלקר) (איור 3H).
הערה: התאימו את המיקום ואת מספר הפינים, בהתאם למקום ההזרקה. במקרה של P. zonata, אין זה חיוני כדי לתקן את הזחלים עם סיכות בשלב זה. - למתוח את הממברנה הבין-מגזרית בין קטע הבטן הרביעית וה-5 ביד.
- לעשות חור קטן עם מחט בסדר קרום בין מגזרי בין קטע 4 ו 5 קטע הבטן.
הערה: הליך זה נחוץ עבור מינים רבים אניסופטראן (שפירית) כי הממברנה הבין-מגזרית קשה מדי להכניס נימי זכוכית ישירות. - הכנס את קצה נימי מוכן לתוך החור המוכן (איור 3I).
הערה: כפי שמוצג באיזור 3H, חלק מהצד הגבי של הזחלים תואם לצד הגחוני של המבוגר, כך שהפנוטיפ מופיע באופן גחוני כשמתייחסים אליו כפי שמוצג באיזור 3H-J. - להזריק 1 μL של פתרון siRNA / dsRNA.
4. ב vivo אלקטרופורציה
- במידת הצורך, להוסיף סיכות נוספות כדי לתקן את הזחל לעמוד קבוע (למשל, חתיכת קלקר).
- בעקבות הזריקה של פתרון siRNA / dsRNA, להחיל שתי טיפות של ג'ל אולטרסאונד על פני השטח הזחל באמצעות מדפים.
- מניחים אלקטרודות על ג'ל אולטרסאונד, עם האלקטרודה החיובית בצד מוזרק עם פתרון siRNA / dsRNA ואלקטרודה שלילית בצד הנגדי (איור 3E, 3G, 3J).
הערה: אין לגעת ישירות באפידרמיס של הזחל כדי למנוע את השפעת הצריבה של אלקטרופורציה. - צור פולסים של 10 פעמים אלקטרופוזיה (280 ms/s כל אחד) באמצעות אלקטרופורטור.
הערה: התאימו את המתח של אלקטרופורציה, בהתאם למינים, לשלבים ולרקמות. במחקר זה, 25 V הוחל על I. סנגלנסיס ו 45 V כדי P. zonata. - לנגב את הג'ל הנותר על פני השטח עם מגבת נייר.
- שמור את הזחלים המטופלים נחים על מגבת נייר רטובה במשך יום אחד בערך להחלמה ולהעביר אותם למקרה גידול ביום שלמחר.
5. ניתוח פנוטיפי ספציפי לאתר
- שמור I. senegalensis בנפרד בצלחת פטרי (5 ס"מ קוטר) המכיל כ 10 מ"ל של מים פיסת מגבת נייר, ולשמור P. zonata בכלוב פלסטיק עם רשת חד פעמית לא ארוגים (כלוב אקלוסיה14).
- עבור I. senegalensis, לאחר שהזחלים מפסיקים לאכול, להעביר אותם בנפרד לתוך כלוב פלסטיק עם רשת חד פעמית לא ארוגה.
הערה: אין לשים שני זחלים או יותר באותו כלוב. אחרת, הם יהיו קניבליזציה אחד את השני מיד לאחר שהם הופכים למבוגרים. - לאחר הופעתו של המבוגר, להתבונן ולצלם את הפנוטיפ סביב האזור שבו האלקטרודה החיובית הונחה עבור אלקטרופורציה.
הערה: הפנוטיפ מופיע רק במדבקות. פנוטיפים הם לעתים קרובות קשה לזהות מיד לאחר הופעתה עקב פיגמנטציה שלם. - על מנת לבחון את היעילות של RNAi (למשל, כמותי RT-PCR), אם הפנוטיפ גלוי, לנתח את האזור ולהשוות אותו עם האזור ללא פנוטיפ.
הערה: הרמות של פנוטיפ RNAi, כלומר גודל ומיקום של decolorization קוטיקל, לעתים קרובות להפגין שונות ניכרת בין אנשים (ראה איור 4). - שמור את המבוגרים הגיחים ב 100% EtOH לניתוחים עתידיים.
הערה: צבע הגוף של מינים Odonata נמוג במהירות לאחר המוות, ולכן חשוב לאחסן אותם באתנול לפני שהם מתים. אתנול לפעמים מביעות את החרקים, במקרים כאלה שהחרקים קפואים לפני שהם מתים.
תוצאות
יישמו את הפרוטוקול לעיל על RNAi מתווך אלקטרופולציה המכוונת לגנים MCO2 וגנים שליליים ( EGFP עבור siRNA ו bla for dsRNA) (i) בבטנו של I. senegalensis (איור 4), (ii) בבית החזה של I. סנגלנסיס (איור 5), ו-(iii) בבטנו של פ. זונטה (איור 6). תוצאות ניסויי RNAi מסוכמות בטבלה 1. בגלל siRNA טעון שלילית / dsRNA משולב רק לתוך תאים טעונים חיובית, פנוטיפים RNAi נצפו ברחבי האזור שבו האלקטרודה החיובית הונחה עבור אלקטרופורציה.
גם ב-I. סנגלנסיס וגם ב-פ. זונטה, עיכוב פיגמנטציה מלנין (כלומר, שחור, חום, וחום אדמדם) הופיע כתמים סביב האזור שבו האלקטרודה החיובית הונחה (ראשי חץ לבנים וקווים מנוקדיםבאיור 4 , איור 5, ו איור 6) כאשר MCO2 RNAi בוצע בשילוב עם אלקטרופורציה (טבלה 1), כפי שדווח בעבר ב- N. פיגמאיה9. לעומת זאת, לא נצפו השפעות פנוטיפיות סביב אתר האלקטרופולציה כאשר הוזרק גן הבקרה (EGFP siRNA או bla dsRNA) (איור 4, איור 5 ואיור 6, טבלה 1). בנוסף, להזרקת הגן MCO2 ללא אלקטרופולציה לא הייתה השפעה עלפיגמנטציה למבוגרים( איור 4 , טבלה 1), מה שמצביע על כך שאלקטרופורציה חיונית ל- RNAi באודונה. יש לציין כי הצבעים הכחולים, הירוקים והצהובים אינם מושפעים מהגן RNAi של MCO2 המעורב בסינתזת מלנין ב קוטיקל, המשקפים באופן סביר את העובדה כי צבעי גוף אלה מיוחסים גרגרי פיגמנט נוכח בתאי האפידרמיס הנראים דרך קוטיקלשקוף 26. כפי שמוצג איור 4, לא הוכרו הבדלים פנוטיפיים יוצאי דופן בין האנשים הכפופים לטיפול siRNA וטיפול dsRNA, ואילו שונות ניכרת בגודל ובמיקום של פנוטיפ RNAi נצפתה בקרב אנשים שונים הנתונים לאותו טיפול RNAi (לדוגמה, השוו שתי דוגמאות של IsMCO2 dsRNA איור 4).
כדי לקבוע את השלב ההתפתחותי המתאים ביותר לטיפול ב-RNAi, השווינו את ההשלכות הפנוטיפיות של הטיפול ב-RNAi בחמישה שלבים מורפולוגיים (שלב A-E) בכוכב הזחל הסופי של I. senegalensis (איור 7A). עיכוב של פיגמנטציה מלנין הנגרמת על ידי MCO2 RNAi נצפתה בכל המבוגרים הגיחים כאשר מוזרק בשלבים A ו- B (איור 7C). כאשר מוזרק בשלבים C ו- D, דיכוי של פיגמנטציה מלנין נצפתה בהחלט אצל כמה מבוגרים הגיח, אבל מבוגרים אחרים הפגינו צבע חריג שנגרם על ידי פצעים (איור 7B).
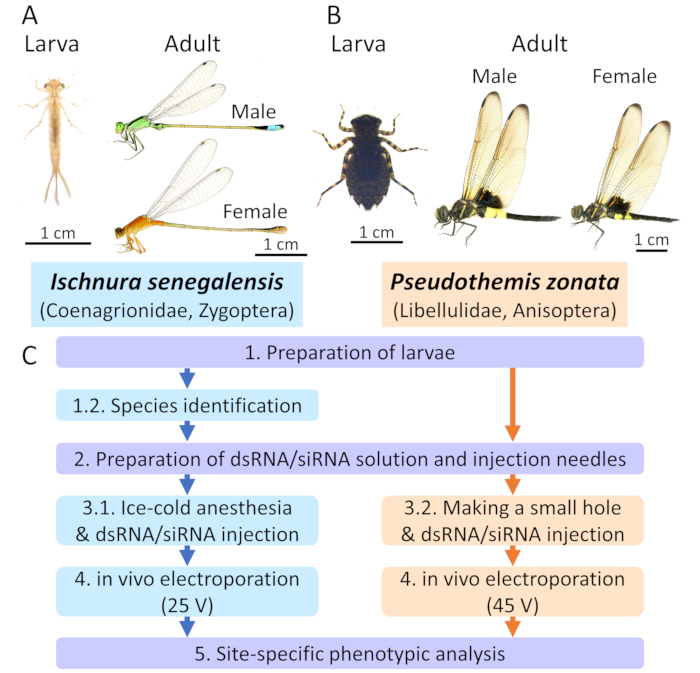
איור 1: שיטות RNAi בניווך אלקטרופולציה באודונה. א. ישנורה סנגלנסיס (קונגיריון) כמין עלמה מייצג. ב. פסאודותמיס זונטה (Libellulidae) כמין שפירית מייצג. ג. הערכה הכוללת של הפרוטוקולים. קופסאות כחולות וכתום מציינות את הפרוטוקולים עבור I. סנגלנסיס ו P. zonata, בהתאמה. הקופסאות הסגולות מציינות את הפרוטוקולים הנפוצים החלים על שני המינים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
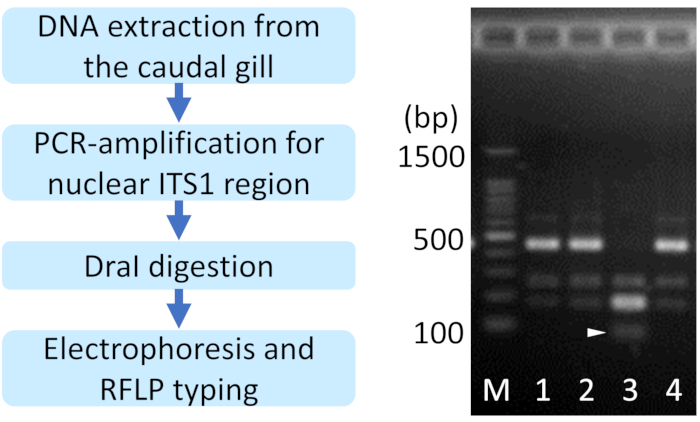
איור 2: תוצאה מייצגת של זיהוי מינים מבוססי אורך שברהגבלה (RFLP) עבור מינים של אישינורה. ראש החץ מציין את הלהקה הספציפית ל-I.סנגלנסיס. 1, 2, 4: I. asiatica, 3: I. סנגלנסיס, ז: סמן סולם זוג 100 בסיסים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 3: שיטת RNAi בתתיווך אלקטרופולציה באודונה. איי-סי. הרדמה קרה כקרח של סנגלנסיס I.. ראשי חץ מצביעים על זחל. א. לשים זחל על קרח כתוש עם נייר רטוב. ב. תצוגה מוגדלת של זחל על קרח. ג. זחל מכוסה בנייר רטוב על קרח. די-ג'י. שיטת RNAi עבור I. סנגלנסיס. די. הזרקה לבית החזה. אי, אי. אלקטרופורציה בבית החזה. פ. הזרקה לתוך הבטן. ג'י. אלקטרופורציה על הבטן. סי-ג'יי. שיטת RNAi עבור פ. זונטה. ה. עושה חור קטן בבטן. אני, אני, אני. הזרקה לתוך הבטן. ג'יי ג'יי. אלקטרופורציה על הבטן. חצים מציינים את הנקודה של עשיית חור או זריקה. +, -: אלקטרודות צד חיוביות/שליליות. המספרים מצביעים על קטע הבטן. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
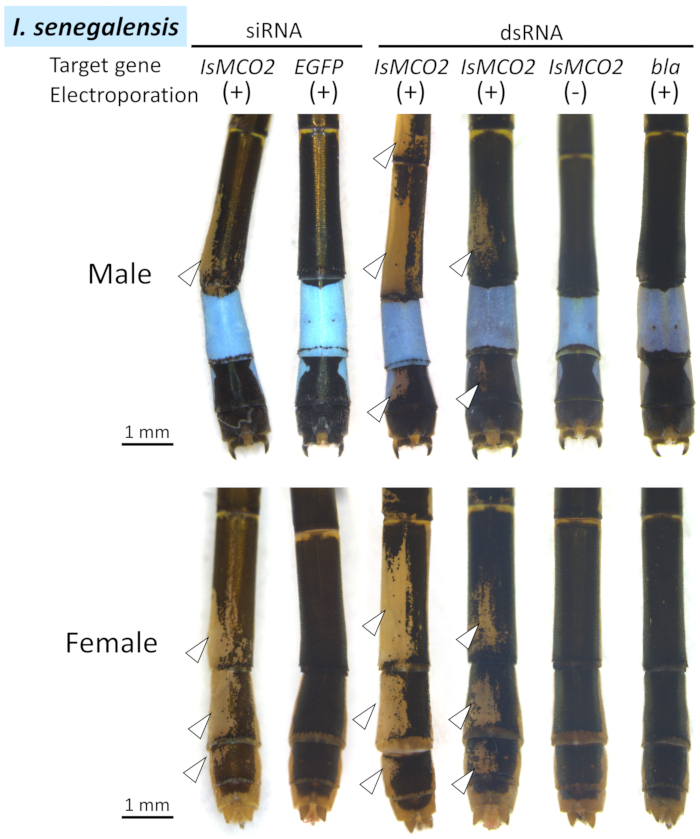
איור 4: נופים דורסלים של פנוטיפים RNAi על הבטן של I. סנגלנסיס. ראשי חץ לבנים מציינים את האזורים של מלניזציה מודחקת. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
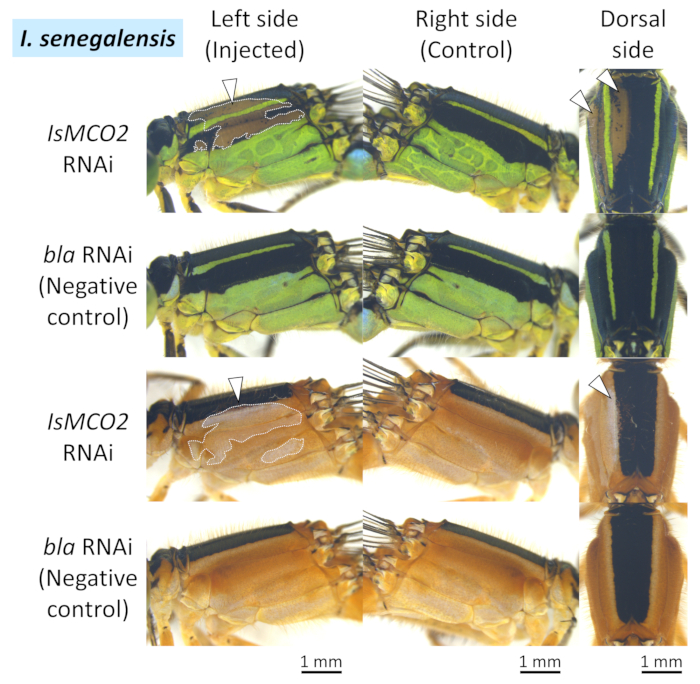
איור 5: נופים רב-ממדיים ודורסיים של פנוטיפים של RNAi על בית החזה של סנגלנסיס. ראשי חץ לבנים וקווים מנוקדים מציינים את האזורים של פיגמנטציה מודחקת. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
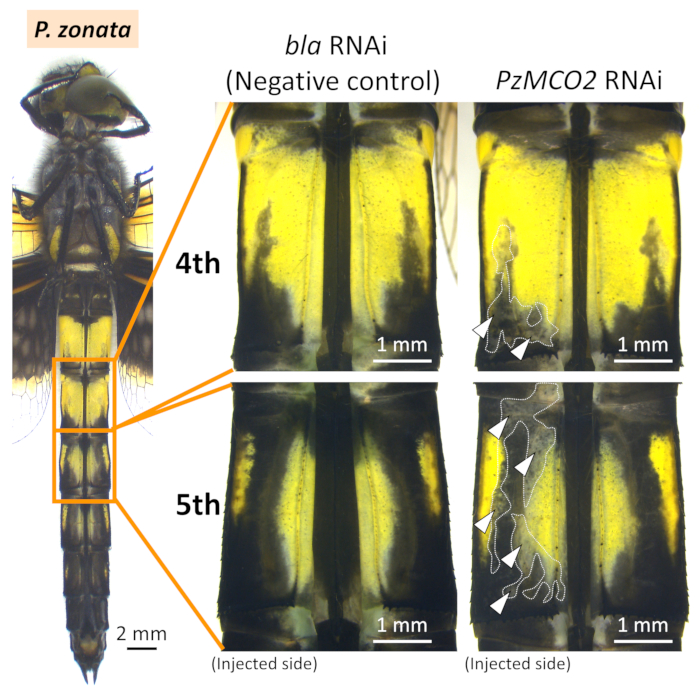
איור 6: נופים גחונים של פנוטיפים של RNAi בבטנו של פ. זונטה. ראשי חץ לבנים וקווים מנוקדים מציינים את האזורים של מלניזציה מודחקת. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
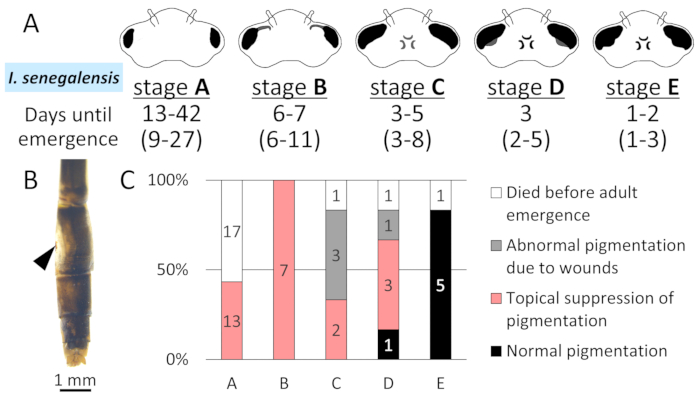
איור 7: שלב תלוי IsMCO2 RNAi אפקטים במהלך instar הזחל הסופי של I.senegalensis. א. שינויים מורפולוגיים בעיניים מורכבות בחמישה שלבים מורפולוגיים (שלב A-E) ומספר הימים להופעת מבוגרים במחקר זה. המספרים בסוגריים הם מדוח14 הקודם. ב. פיגמנטציה לא תקינה עקב פצעים. ראש החץ מציין אתר אלקטרופוזיה. ג. ההשפעה של RNAi בחמישה שלבים מורפולוגיים על פיגמנטציה למבוגרים ב I. סנגלנסיס. המספר על הרף מציין את מספר האנשים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
| מינים | סנגלנסיס | פ. זונטה | |||||||||
| אזור מוזרק | הבטן | בית החזה | הבטן | ||||||||
| siRNA/dsRNA | siRNA (תב"א | dsRNA (תב"א) | dsRNA (תב"א) | dsRNA (תב"א) | |||||||
| גן יעד | ISMCO2 | EGFP (EGFP) | ISMCO2 | ISMCO2 | בלה | ISMCO2 | בלה | פזמקו2 | פזמקו2 | בלה | |
| אלקטרופורציה (אלקטרופורציה) | + | + | + | - | + | + | + | + | - | + | |
| זחלים מוזרקים | 22 | 25 | 30 | 6 | 53 | 12 | 20 | 17 | 5 | 9 | |
| מבוגרים הגיחו | 7 | 6 | 13 | 4 | 40 | 11 | 14 | 11 | 2 | 5 | |
| מבוגרים עם פחות אזורים פיגמנטיים (יחס) | 7 (100%) | 0 (0%) | 13 (100%) | 0 (0%) | 0 (0%) | 10 (91%) | 0 (0%) | 11 (100%) | 0 (0%) | 0 (0%) | |
שולחן 1. ההשפעה של RNAi על פיגמנטציה למבוגרים ב I. סנגלנסיס ו P. zonata. תוצאות בשלב א' מוצגות ב- I. סנגלנסיס. IsMCO2: אוקסידאז multicopper 2 גן של I. סנגלנסיס, EGFP: גן חלבון פלואורסצנטי ירוק משופרת, בלה : גן בטא לקטמאז מ pGEM-T וקטור קל, PzMCO2: אוקסידאז multicopper 2 גן של P. zonata. התוצאות עבור גנים הבקרה מייצגים את המספר הכולל של ניסויים המחברים ערכו עד כה.
Discussion
קטלניות של טיפול RNAi
מצאנו שהקטלניות של הטיפול ב-RNAi תלויה מאוד בהיסטוריית גידול הזחלים של אודונטה. הזחלים זמן קצר לאחר האיסוף בתחום הם בדרך כלל בריאים ומציגים שיעורי תמותה נמוכים לאחר הטיפול RNAi מתווך אלקטרופורציה. לעומת זאת, הזחלים שגודלו במעבדה במשך תקופה ארוכה (למשל, חודש אחד) נוטים לסבול מ שיעורי הצלחה נמוכים של הופעת מבוגרים. ב- I. senegalensis, במקום הזחלים שנאספו בשטח, הזחלים שגודלו במעבדה מביציםיכולים לשמש 14, אך שיעורי ההצלחה של RNAi באמצעות הזחלים שגודלו במעבדה נוטים להיות נמוכים משמעותית (אנשים רבים מתו במהלך מטמורפוזה) מאלה המשתמשים בזחלים שנאספו בשדה. בנוסף, הזנת זחל תכופה וגידול מים נקיים חשובים להגדלת שיעורי ההצלחה של הופעת מבוגרים והפחתת הקטלניות של הטיפול RNAi.
יעילות הטיפול ב- RNAi
כפי שתואר לעיל, רמות הפנוטיפ של RNAi, כלומר גודל ומיקום של דה-קולוריזציה קוטיקל, לעתים קרובות הפגינו שונות ניכרת בין אנשים נתון לאותו טיפול RNAi (למשל, איור 4), אבל רמות החדירה פנוטיפית נראה שונה להפליא בין המינים Odonata. האזורים הפנוטיפיים שנצפו היו גדולים ובולטים יותר ב- I. senegalensis (איורים 4-5) מאשר ב P. zonata (איור 6) ו N. פיגמאה9. הבדל זה יכול להיות בגלל עובי של קוטיקל על פני השטח הזחל, בהתחשב בכך קוטיקל של I. סנגלנסיס הוא דק יותר מאשר קוטיקל של P. zonata ו N. פיגמאיה). ככל שבדקנו, לא הוכר הבדל ברור בין ההשפעות של siRNA ו- dsRNA (איור 4, טבלה 1).
שלב התפתחותי מתאים עבור RNAi
יש לציין כי בימוי זחל נאות חשוב לביצוע RNAi ביעילות. עיכוב של פיגמנטציה למבוגרים נגרם על ידי MCO2 RNAi לפני שלב D (כ 3 ימים לפני הופעת מבוגרים), אשר עולה בקנה אחד עם הדו"ח הקודם על N. פיגמאה9. פנוטיפים RNAi שנצפו כאשר מוזרק בשלבים C ו- D היו פחות בולטים מאלה שטופלו בשלבים A ו- B, אשר מצביעים על כך שלבים C ו- D עשוי להיות מאוחר מדי כדי לדכא מספיק את ביטוי הגן. העיתוי המתאים לטיפול RNAi תלוי בעיתוי של ביטוי גנים, וגן MCO2 מציג ביטוי גבוה חולףבמהלך הופעת מבוגרים 9, כמו חרקיםאחרים 17,18. ב stinkbug Plautia stali, RNAi נוקאאוט של גן MCO2 נצפתה מהיום 4 ואילך לאחרהזרקה 27, אשר עולה בקנה אחד עם התוצאות הנוכחיות.
המחקר הקודם שלנו על I. senegalensis הראה כי, לאחר השלב B, ימים להופעת מבוגרים להפגין וריאציה קטנה יחסית בין רוב הזחלים instar הסופי, מה שמרמז כי השלב B עשוי להתאים את תחילת התהליך לקראת הופעת מבוגרים, לאחר מכן התהליכים ההתפתחותיים עבור מטמורפוזה להמשיך באופן קידומתומתואמת 14. חריגות מורפולוגיות שנגרמו על ידי פצעים נצפו לעתים קרובות כאשר הזחלים טופלו RNAi בשלבים C ו- D (איור 7B, 7C). זה עשוי להיות קשור עם התקדמות דרמטית של מטמורפוזה בשלבים אלה, מה שמרמז כי טיפול RNAi יש להימנע מהשלב C ועל. לסיכום, אנו ממליצים כי זחלי instar הסופי בשלב A או B (או בשלב לפני כנפי הזחל להרחיב באופן משמעותי) יש להשתמש עבור ניסויים RNAi.
שימושיות ועליונות של שיטת RNAi בתתיווך אלקטרופולציה
RNAi קונבנציונאלי היא שיטה ניסיונית פשוטה ורבת עוצמה, אבל כמהשושלת חרקים כמו פרפרים 10,כניפות 28 ו שפיריות 9 להפגין יעילות RNAi נמוכה, אשר הקמת ניתוח תפקוד גנים הוא אתגר גדול. במחקר זה, מצאנו כי RNAi מתווך אלקטרופורציה יכול לגרום לדיכוי גנים מקומיים שפיריות עם יעילות של כמעט 100%, לפחות באפידרמיס, אם מטופלים בשלבים התפתחותיים מתאימים (טבלה 1). לאחרונה, נוקאאוטים גנים מבוססי CRISPR/Cas9 הוחלו בהצלחה על מגוון חרקים, ומספקים כלי גנטי מולקולרי רב עוצמה עבוראורגניזמים שאינם מודל 29. כאן, לעומת זאת, אנו מציינים כי CRISPR / Cas9 הוא בהחלט נהדר אבל שיטת RNAi בתתיווך אלקטרופולציה עשויה להיות עדיפה על CRISPR / Cas9 בכמה מובן.
ראשית, בשיטת RNAi בתתיווך אלקטרופורציה, אזור הגוף שבו מופיעים פנוטיפים RNAi ניתן לשלוט בקלות באופן ניסיוני על ידי המיקום של האלקטרודה החיובית על אלקטרופולציה. בנוסף, מאז האזור שבו ביטוי הגנים מודחק מוגבל סביב האזור שבו האלקטרודה החיובית הוצבה, פנוטיפים RNAi ניתן להשוות בקלות עם פנוטיפים שליטה זה לצד זה באותו אדם. שנית, בהשוואה לשיטת CRISPR/Cas9 שבה יש לגדל ביצים מוזרקות לבגרות כדי לצפות בפנוטיפים של הנוקאאוט, ה- RNAi התווך באלקטרופורציה עדיף גם בחנוטיפים של הפלת הגנים בדרך כלל בזמן קצר בהרבה. לדוגמה, זה לוקח שלושה עד ארבעה חודשים עבור I. סנגלנסיס ו שנה עד שנתיים עבור P. zonata מביציםלמבוגרים 14,30. עם זאת, על מנת לצפות פנוטיפים RNAi בתוך האפידרמיס למבוגרים, זה לוקח פחות מחודש מהזרקת dsRNA לזחלי instar הסופי בשלב B להופעה למבוגרים הן I. סנגלנסיס ו P. zonata (איור 7). שלישית, שיטת RNAi בתתיווך אלקטרופורציה כרוכה בהזרקת dsRNA לזחלים גדולים, וזה קל יותר ממיקרו-ג'ינג'י לביצים זעירות הנדרשות לשיטת CRISPR/Cas9. בנוסף, RNAi בתתיווך אלקטרופורציה חל על מיני חרקים שהביצים שזה עתה הטילו קשה לאסוף. לדוגמה, נקבות פ. זונאטה מטילות ביצים על צמחים צפים על פני המים במהלך הטיסה, ולכן קשה לאסוף את ביציהן הן בשטח והן במעבדה. לפיכך, אנו מצפים כי פרוטוקול זה עשוי להיות ישים בדרך כלל אורגניזמים שאינם מודל שבו השיטה RNAi קונבנציונאלי לא עובד ביעילות.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
אנו מודים למינורו מוריאמה על ייעוץ ותומך טכניים, בן הירוטה וריוטארו סוזוקי על איסוף זחלי אודונאטה, ומיזא שיניה על הערות מועילות על כתב היד. עבודה זו נתמכה על ידי JSPS KAKENHI גרנט מספרים JP18J21561 ללכת JP18H02491, JP18H04893, JP19H03287, ו JP20H04936 כדי RF.
Materials
| Name | Company | Catalog Number | Comments |
| 12-well plate | Violamo | VTC-P12 | For rearing larvae before injection |
| Brine shrimp eggs | JAPAN PET DESIGN | 4975677012396 | |
| Calibrated micropipette (1-5 µL) | Drummond | 2-000-001-90 | |
| Deposit pestle 1.5 mL | Thermo Fisher Scientific | K749520-0090 | |
| Digital high defenition microscope camera | Leica Microsystems | MC170HD | |
| Disposable non-woven mesh | HAKUGEN EARTH | DSC-105A | |
| DNA Ligation Kit Ver.2.1 | Takara Bio | 6022 | |
| DraI | Takara Bio | 1037A | |
| Electrode (1mmφ) | NEPAGENE | CUY650P1 | |
| Electroporator | CellProduce | Cure-gene | |
| Forceps | KOWA Forceps Industry | K-10 No1 | |
| Glass needle puller | NARISHIGE | PN-3 | |
| Hand mixer | AS ONE | 1-229-02 | |
| Hand net | HOGA | IS33-3B | |
| HEPES | FUJIFILM Wako Pure Chemical Corporation | 346-01373 | For injection buffer |
| Insect pin capitate No. 3 | Shiga Konchu Fukyusha | N230 | For fixing larvae |
| KCl | FUJIFILM Wako Pure Chemical Corporation | 163-03545 | For PBS |
| KH2PO4 | FUJIFILM Wako Pure Chemical Corporation | 169-04245 | For PBS |
| KOAc | FUJIFILM Wako Pure Chemical Corporation | 160-03175 | For injection buffer |
| KOH | FUJIFILM Wako Pure Chemical Corporation | 168-21815 | For injection buffer |
| Laboratory Jack (150x150) | AS ONE | 1-4642-11 | For adjusting the position of the manipulator |
| LOGIQLEAN Gel for Ultrasound Hard type | GE Healthcare | 2369385 | |
| Manipulator | Muromachi Kikai Co., Ltd. | SJ-1 | For adjusting the position of the injector and the capillary |
| MEGAscript RNAi kit | Thermo Fisher Scientific | AM1626 | |
| Mg(OAc)2 | FUJIFILM Wako Pure Chemical Corporation | 130-00095 | For injection buffer |
| Na2HPO4 | FUJIFILM Wako Pure Chemical Corporation | 197-02865 | For PBS |
| NaCl | FUJIFILM Wako Pure Chemical Corporation | 191-01665 | For PBS |
| Petri dish (5 cm diameter) | Iwaki | 1010-060 | For rearing injected larvae |
| Pneumatic Injector | NARISHIGE | IM-12 | |
| pT7Blue T-Vector | Novagen | 69820 | |
| QIAquick PCR Purification Kit | QIAGEN | 28106 | |
| RNAiso Plus | FUJIFILM Wako Pure Chemical Corporation | 9109 | |
| RNeasy Mini Kit | QIAGEN | 74106 | |
| Shiga micro insect pin with stainless headless | Shiga Konchu Fukyusha | N251 | For making a small hole |
| Stereoscopic microscope | Leica Microsystems | S8APO | |
| SuperScript IV Reverse Transcriptase | Thermo Fisher Scientific | 18090010 | |
| TaKaRa Ex Taq | Takara Bio | RR001B | For PCR-amplification from plasmid |
| Tks Gflex DNA Polymerase | Takara Bio | R060B | For PCR-amplification from caudal gill |
References
- Misof, B., et al. Phylogenomics resolves the timing and pattern of insect evolution. Science. 346 (6210), 763-767 (2014).
- Wipfler, B., et al. Evolutionary history of Polyneoptera and its implications for our understanding of early winged insects. Proceedings of the National Academy of Sciences of the United States of America. 116 (8), 3024-3029 (2019).
- Corbet, P. S. . Dragonflies Behavior and Ecology of Odonata. , (1999).
- Futahashi, R. Color vision and color formation in dragonflies. Current Opinion in Insect Science. 17, 32-39 (2016).
- Futahashi, R. Diversity of UV reflection patterns in Odonata. Frontiers in Ecology and Evolution. 8 (201), (2020).
- Córdoba-Aguilar, A. . Dragonflies and damselflies. Model organisms for ecological and evolutionary research. , (2008).
- Bybee, S., et al. Odonata (dragonflies and damselflies) as a bridge between ecology and evolutionary genomics. Frontiers in Zoology. 13 (46), (2016).
- Linz, D. M., Clark-Hachtel, C. M., Borràs-Castelles, F., Tomoyasu, Y. Larval RNA interference the red flour beetle, Tribolium castaneum. Journal of Visualized Experiments. 92, 52059 (2014).
- Okude, G., et al. Electroporation-mediated RNA interference reveals a role of multicopper oxidase 2 gene in dragonfly’s cuticular pigmentation. Applied Entomology and Zoology. 53 (3), 379-387 (2017).
- Terenius, O., et al. RNA interference in Lepidoptera: An overview of successful and unsuccessful studies and implications for experimental design. Journal of Insect Physiology. 57, 231-245 (2011).
- Ando, T., Fujiwara, H. Electroporation-mediated somatic transgenesis for rapid functional analysis in insects. Development. 140, 454-458 (2013).
- Nishikawa, H., et al. A genetic mechanism for female-limited Batesian mimicry in Papilio butterfly. Nature Genetics. 47 (4), 405-409 (2015).
- Osanai-Futahashi, M., et al. Positional cloning of a Bombyx pink-eyed white egg locus reveals the major role of cardinal in ommochrome synthesis. Heredity. 116 (2), 135-145 (2016).
- Okude, G., Futahashi, R., Tanahashi, M., Fukatsu, T. Laboratory rearing system for Ischnura senegalensis (Insecta: Odonata) enables detailed description of dragonfly’s larval development and morphogenesis. Zoological Science. 34 (5), 386-397 (2017).
- Ozono, A., Kawashima, I., Futahashi, R. . Dragonflies of Japan (3rd edition). , (2017).
- Ozono, A., Kawashima, I., Futahashi, R. . The Handbook of Japanese Aquatic Insects. Volume 3: Dragonfly larvae. , (2019).
- Arakane, Y., Noh, M. Y., Asano, T., Kramer, K. J. Tyrosine metabolism for insect cuticle pigmentation and sclerotization. Extracellular Composite Matrices in Arthropods. , 165-220 (2016).
- Asano, T., et al. Mini-review an insect-specific system for terrestrialization: Laccase-mediated cuticle formation. Insect Biochemistry and Molecular Biology. 108, 61-70 (2019).
- Futahashi, R., Okude, G., Sugimura, M., Ugai, S. Interspecific hybrids in Japanese Odonata. Tombo. 60, 1-49 (2018).
- Okude, G., Fukatsu, T., Futahashi, R. Interspecific crossing between blue-tailed damselflies Ischnura elegans and I. senegalensis in the laboratory. Entomological Science. 23 (2), 165-172 (2020).
- Naito, Y., Yoshimura, J., Morishita, S., Ui-Tei, K. siDirect 2.0: updated software for designing functional siRNA with reduced seed-dependent off-target effect. BMC Bioinformatics. 10, 392 (2009).
- Yamaguchi, J., Mizoguchi, T., Fujiwara, H. siRNAs induce efficient RNAi response in Bombyx mori embryos. PLoS ONE. 6 (9), e25469 (2011).
- Untergasser, A., et al. Primer3--new capabilities and interfaces. Nucleic Acids Research. 40 (15), e115 (2012).
- Futahashi, R. Whole-mount in situ hybridization of sectioned tissues of species hybrids to detect cis-regulatory changes in gene expression pattern. Methods in Molecular Biology. 772, 319-328 (2011).
- Matsuura, Y., Kikuchi, Y., Miura, T., Fukatsu, T. Ultrabithorax is essential for bacteriocyte development. Proceedings of the National Academy of Sciences of the United States of America. 112 (30), 9376-9381 (2015).
- Henze, M. J., Lind, O., Wilts, B. D., Kelber, A. Pterin-pigmented nanospheres create the colours of the polymorphiic damselfly Ischnura elegans. Journal of The Royal Society Interface. 16, 20180785 (2019).
- Nishide, Y., et al. Diversity and function of multicopper oxidase genes in the stinkbug Plautia stali. Scientific Reports. 10 (1), 3464 (2020).
- Christiaens, O., Swevers, L., Smagghe, G. DsRNA degradation in the pea aphid (Acyrthosiphon pisum) associated with lack of response in RNAi feeding and injection assay. Peptides. 53, 307-314 (2014).
- Sun, D., Guo, Z., Liu, Y., Zhang, Y. Progress and prospects of CRISPR/Cas systems in insects and other arthropods. Frontiers in Physiology. 8, 608 (2017).
- Miyakawa, K. A study of the life-history of Pseudothemis zonata (Burm.) (Odonata, Libellulidae) II. Immature stage. Kontyû. 37 (4), 409-422 (1969).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved