Method Article
Méthode de brouillage de l’ARN à base d’électroporation à Odonata
Dans cet article
Résumé
Nous fournissons un protocole détaillé pour l’interférence de l’ARN à base d’électroporation chez les insectes de l’ordre Odonata (libellules et demoiselles) à l’aide du damselfly à queue bleue(Ischnura senegalensis: Coenagironidae: Zygoptera) et de la libellule écumoire pied(Pseudothemis zonata: Libellulidae: Anisoptera).
Résumé
Les libellules et les demoiselles (ordre Odonata) représentent l’un des insectes les plus ancestraux avec métamorphose, dans lequel ils changent radicalement d’habitat, de morphologie et de comportement, des larves aquatiques aux adultes terrestres/aériens sans stade pupal. Les adultes odonata ont une vision des couleurs bien développée et montrent une diversité remarquable dans les couleurs du corps et les motifs entre les sexes, les étapes et les espèces. Bien que de nombreuses études écologiques et comportementales sur Odonata aient été menées, les études génétiques moléculaires ont été rares principalement en raison de la difficulté d’appliquer l’analyse fonctionnelle des gènes à Odonata. Par exemple, l’interférence d’ARN (RNAi) est moins efficace dans l’Odonata, comme rapporté dans le Lépidoptère. Pour surmonter ce problème, nous avons réussi à établir une méthode RNAi combinée avec l’électroporation in vivo. Ici, nous fournissons un protocole détaillé comprenant une vidéo de la méthode RNAi à base d’électroporation comme suit : préparation des larves, identification des espèces, préparation de la solution dsRNA/siRNA et aiguilles d’injection, anesthésie glacée des larves, injection de dsRNA/siRNA, électroporation in vivo et élevage individuel jusqu’à l’émergence adulte. La méthode RNAi à base d’électroporation s’applique à la fois aux demoiselles (sous-commande Zygoptera) et aux libellules (sous-commande Anisoptera). Dans ce protocole, nous présentons les méthodes de la digue à queue bleue Ischnura senegalensis (Coenagrionidae) comme un exemple d’espèces de demoiselles et la libellulidae pseudothemis zonata (Libellulidae) comme un autre exemple d’espèces de libellules. À titre d’exemples représentatifs, nous montrons les résultats de l’ARNi ciblant le gène de synthèse de la mélanine multicopper oxidase 2. Cette méthode RNAi facilitera la compréhension des diverses fonctions génétiques impliquées dans la métamorphose, la morphogenèse, la formation de motifs de couleur, et d’autres caractéristiques biologiques d’Odonata. En outre, ce protocole peut être généralement applicable aux organismes non modèles dans lesquels l’ARNi est moins efficace dans la suppression des gènes en raison de l’inefficacité et de la faible pénétration.
Introduction
Libellules et demoiselles (l’ordre Odonata) sont parmi les groupes les plus ancestraux d’insectes qui présentent une « métamorphose »1,2. Par métamorphose, ils modifient radicalement leur habitat, leur morphologie et leur comportement, des larves aquatiques aux adultes terrestres/aériens3. Les adultes odonata ont une vision des couleurs bien développée et représentent une diversité remarquable dans les couleurs du corps et les motifs entre les sexes, les étapes et lesespèces 3,4,5. Alors que de nombreuses études écologiques et comportementales sur Odonata ont étémenées 6,7, les études génétiques moléculaires ont été entravées principalement par la difficulté d’appliquer l’analyse fonctionnelle des gènes à Odonata.
La méthode conventionnelle d’interférence d’ARN (RNAi), dans laquelle l’ARN double-échoué (dsRNA) est injecté pour supprimer la fonction du gène d’intérêt8,s’est avérée inefficace dans les insectes d’Odonata9,comme rapporté dans les insectes lépidoptères10. D’autre part, des rapports précédents ont suggéré que l’ARNi à base d’électroporation est efficace chez les espèces de lépidoptères, en particulier dans les tissus épidermiques11,12,13. Nous avons récemment constaté que le RNAi à base d’électroporation fonctionne efficacement dans la petite libellule Nannophya pygmaea (Libellulidae: Anisoptera)9, mais N. pygmaea est une espèce relativement rare et donc ne convient pas aux études génétiques moléculaires.
La plupart des espèces d’Odonata sont classées dans l’une ou l’autre des deux sous-commandes, Zygoptera (demoiselles) ou Anisoptera (vraies libellules)3. Ici, nous nous sommes concentrés sur le damselfly à queue bleue Ischnura senegalensis (Coenagrionidae; Figure 1A) en tant qu’espèce représentative de Zygopteran et la libellulidae de libellulidae de libellulidae de pied écrémant ; Figure 1B) en tant qu’espèce anisopteran représentative. Les deux espèces sont parmi les espèces odonata les plus communes dans les étangs naturels et urbains au Japon, y compris ceux de la ville de Tsukuba, et nous pouvons recueillir de nombreuses larves des deux espèces dans le champ. Récemment, nous avons mis en place un système d’élevage en laboratoire pour les larves individuelles d’I. senegalensis, qui a permis un suivi continu du développement et de la morphogenèse des larves d’Odonataen détail 14.
Dans ce rapport, nous fournissons une méthode raffinée et un protocole vidéo pour l’RNAi à 20 h 20 dans I. senegalensis et P. zonata. Au Japon, I. senegalensis et Ischnura asiatica, qui sont génétiquement proches, sont souvent trouvéssympathiquement 15, et ils sont difficiles à distinguer chez les larves16. Nous décrivons également comment deux espèces d’Ischnura peuvent être distinguées par le polymorphisme de longueur de fragment de restriction (RFLP).
Pour évaluer l’efficacité du gène RNAi à base d’électroporation, nous sélectionnons le gène multicopper oxidase 2 (MCO2; également connu sous le nom de laccase2) comme gène cible représentatif, en raison du phénotype visible de la couleur cuticule plus pâle au moment du renversement de l’expression du gène9. MCO2 est connu pour être essentiel pour assombrir l’épiderme dans une variété d’espèces d’insectes17,18.
Protocole
REMARQUE : Le schéma global des protocoles est indiqué à la figure 1.
1. Préparation des larves de libellules ou de demoiselles.
- Recueillir les larves sur le terrain à l’aide d’un filet à main.
REMARQUE : Les larves d’I. senegalensis s’accrochent souvent aux plantes aquatiques flottant à la surface de l’eau, tandis que les larves de P. zonata restent souvent parmi la litière des feuilles au fond. Dans la ville de Tsukuba, les larves finales de L’I. senegalensis se trouvent principalement de mars à juin, et celles de P. zonata de mai à juin. I. les larves de sénégalensis peuvent être élevées à partird’œufs dans le laboratoire 14, mais les larves recueillies sur le terrain ont le taux de réussite nettement plus élevé de l’émergence des adultes. - Identification des espèces par polymorphisme de longueur de fragment de restriction (RFLP) des produits amplifiés par PCR.
REMARQUE : Cette étape n’est nécessaire que si l’espèce est difficile à identifier à partir de l’apparition des larves, comme dans I. senegalensis.- Tenez l’une des branchies caudales d’une larve à l’aide de forceps. Puis, la larve tombe de sa branchie caudale elle-même (causant l’autotomie).
REMARQUE : Les larves de demoiselles zygopteran ont habituellement trois branchies caudales (figure 1A). Lorsqu’ils sont attaqués par un prédateur, ils peuvent enlever leurs propres branchies caudales pour s’échapper. Si la branchie caudale n’est pas disponible, une partie de la jambe est disséquée. - Mettez une branchie caudale dans 100 μL de solution PBS [0,8% NaCl, 0,02% KCl, 0,115% Na2HPO4, et 0,02% KH2PO4 (w/v)] et homogénéisez avec un mélangeur à main à l’aide d’un pilon de dépôt.
- Faites tourner le mélange à 5 000 x g pendant 10 secondes et soumettez 0,5 μL du supernatant à l’amplification PCR à l’aide de polymélase d’ADN et d’amorce pour amplifier la région interne transcrite de l’espacement 1 (ITS1) de l’ADN nucléaire.
REMARQUE : Effectuez le PCR selon le protocole du fabricant. La combinaison des amorce ITS-F0 (5'- GGA AAG ATG GCC AAA CTT GA -3') et 5.8S-AS1 (5'- GCC GGC CCT CAG CCA G -3') peut amplifier la région ITS1 dans presque toutes les espèces odonata19. - Incuber le mélange de 1 μL de produit amplifié par PCR, de 0,3 μL de tampon de 10 x M, de 0,1 μL d’enzyme de restriction DraI et de 1,6 μL d’eau à 37 °C pendant 1 h.
REMARQUE : Sélectionnez les meilleures enzymes de restriction, selon la combinaison des espèces. S’il n’y a pas d’enzymes de restriction adaptées à l’identification des espèces, confirmer par séquençage de l’ADN. - Chargez 2 μL des produits avec le colorant de chargement sur le gel d’agarose de 2% et exécutez l’électrophoresis.
REMARQUE : Il est difficile d’identifier les espèces lorsqu’on utilise du gel d’agarose de 1 % ou 1,5 %. - Vérifiez le modèle d’électrophorèse pour identifier l’espèce( Figure 2).
REMARQUE : Les modèles de RFLP dépendent des espèces et de la population. Dans la population de Tsukuba, I. asiatica a une bande importante de 400-500 bp, tandis que I. senegalensis a une bande importante d’environ 200 bp et une bande supplémentaire de moins de 100 bp (une pointe de flèche dans la figure 2). La région ITS1 existe en plusieurs copies dans le génome, et chez les espèces d’Ischnura, le polymorphisme microsatellite dans la région ITS1 est souvent présent chez le même individu, affectant le modèle des RFLPs.
- Tenez l’une des branchies caudales d’une larve à l’aide de forceps. Puis, la larve tombe de sa branchie caudale elle-même (causant l’autotomie).
- Élever les larves recueillies en laboratoire jusqu’à ce qu’elles soient utilisées pour l’ARNi.
- Placez chaque larve séparément dans chaque puits d’une plaque de 12 puits avec environ 3 mL d’eau.
REMARQUE : Les larves d’I. senegalensis doivent être conservées individuellement parce qu’elles se cannibalisent fréquemment les unes les autres, tandis que les larves de P. zonata peuvent être conservées en groupe parce qu’elles cannibalisent rarement. - Nourrir les larves d’I. senegalensis avec des crevettes saumures Artemia tous les jours et des larves de P. zonata avec des vers sanguins et/ou des vers Tubifex au moins deux fois par semaine jusqu’à ce qu’elles se développent au stade de développement approprié pour l’ARNi.
REMARQUE : L’alimentation fréquente est essentielle pour augmenter le taux de réussite de l’émergence des adultes. - Changez l’eau dès qu’elle devient sale avec des excréments ou des restes.
REMARQUE : Des changements fréquents d’eau sont également importants pour augmenter le taux de réussite de l’émergence des adultes. - Jugez de l’étape de développement appropriée pour RNAi.
REMARQUE : Les larves finales de l’I. senegalensis peuvent être classées en cinq stades dedéveloppement 14,20. Le premier stade (stade A, avant que les ailes commencent à se développer) ou le deuxième stade (stade B) des larves finales du stade est adapté aux expériences rnai, lorsqu’on considère le phénotype clair de l’ARNi après l’émergence adulte (voir Discussion). Dans P. zonata, les larves finales du stade avant l’expansion des ailes (correspondant au stade A de I. senegalensis)ont été utilisées dans cette étude.
- Placez chaque larve séparément dans chaque puits d’une plaque de 12 puits avec environ 3 mL d’eau.
2. Préparation de la solution dsRNA/siRNA et des aiguilles d’injection pour RNAi.
REMARQUE : Sélectionnez soit un petit ARN interférant (siRNA) (étape 2.1) ou un ARN à double brin (dsRNA) (étape 2.2) comme solution pour l’ARNi.
- Préparation de la solution siRNA
- Concevoir siRNA en utilisant siDirect version du programme 2.0 (http://sidirect2.rnai.jp/)21,suivant les lignes directrices précédemment rapportées chez les insectes lépidoptères22.
- Obtenez du siRNA synthétisé commercialement.
REMARQUE: Les séquences de siRNA ciblant le gène MCO2 de I. senegalensis utilisés dans cette étude étaient les suivantes: 5'- GCA CUU UCC GUU AUC AUC AUA -3' pour le brin de sens et 5'- UAU UAA CGG AAA GUG CUC -3' pour le brin antisens. Comme un contrôle négatif, les séquences de siRNA ciblant la protéine fluorescente verte améliorée (EGFP) gène étaient les suivantes: 5'- GCA UCA AGG UGA ACU UCA AGA -3' pour le brin de sens et 5'- UUG AAG UUC ACC UUG AUG CCG -3' pour le brin antisens11. - Diluer le siRNA à 100 μM avec tampon d’injection [100 mM CH3COOK, 2 mM Mg (CH3COO)2, 30 mM HEPES-KOH, pH 7,4]22.
- Conserver à -80 °C jusqu’à utilisation.
- Préparation de la solution dsRNA.
- Sélectionnez une région de 300-400 bp pour dsRNA en utilisant primer3 version du programme 4.1.0 (http://bioinfo.ut.ee/primer3/)23 et concevoir les ensembles d’amorce.
- Obtenez des ensembles d’amorce synthétisés commercialement.
REMARQUE: Pour produire des modèles pour la synthèse dsRNA, les ensembles d’amorce utilisés dans cette étude étaient les suivants: (5'- GCC TGT CAG CTT TGT CTT CC -3' pour l’amorce avant et 5'- GGT GTC TGG CGG ACA ACT AT -3' pour l’amorce inverse) pour les gènes MCO2 d’I. senegalensis (IsMCO2, accession no. LC589180) et (5'- CCG CAC AGC TCA TTA TTC AA -3' pour l’amorce avant et 5'- GGA GGA TGA TTT CAT CGA CA -3' pour l’amorce inverse) pour les gènes MCO2 de P. zonata (PzMCO2, accession no. LC589179). Comme un contrôle négatif, l’amorce définie pour le ciblage dsRNA β-lactamase (bla)gène sur le vecteur de clonage ont été (5'- CTA TGT GGC GCG GTA T -3' pour l’amorce avant et 5'- CAG AAG TGG TCC TGC TAC T -3' pour l’amorce inverse). - Extraire l’ARN des larves d’Odonata à l’aide d’un kit d’extraction d’ARN disponible dans le commerce et effectuer la synthèse de l’ADN en utilisant la transcriptase inverse selon le protocole du fabricant.
REMARQUE : La bibliothèque cDNA préparée pour l’analyse du séquençage de l’ARN peut également être utilisée. - Amplifiez les séquences cibles à l’aide de l’ADNC synthétisé et de l’amorce conçue et clonez-les dans le vecteur de clonage à l’aide d’une ligase commerciale selon le protocole du fabricant.
- Transformez le plasmide en cellules compétentes e. coli et prenez une seule colonie après l’incubation de la nuit.
- PCR-amplifier la région d’insertion à l’aide d’amorce sur le vecteur.
- Confirmez la séquence clonée par séquençage Sanger.
- PCR-amplifier l’insert à l’aide d’amorces vectorielles contenant la séquence de promoteur de polymésase T724, 25.
REMARQUE : Dans cette étude, les amorces suivantes ont été utilisées : T7-F (5' - TAA TAC GAC TCA CTA TAG GGA GAC TAG TCA TAT GGA T - 3') et T7-R (5'- TAA TAC GAC TCA CTA TAG GGA GAC GGG ATC CGA T - 3') 25. - Purifiez le produit PCR à l’aide du kit de purification PCR selon le protocole du fabricant, évacuez l’ADN avec 50 μL d’eau distillée et concentrez la solution d’ADN élutée à environ 10 μL à l’aide d’un évaporateur centrifuge.
- Synthétiser le dsRNA par transcription in vitro selon le protocole du fabricant. Utilisez un total de 1000 ng d’ADN modèle et dsRNA synthétisé elute avec 100 μL de tampon d’élitution.
- Mesurer la concentration de dsRNA à l’aide d’un spectrophotomètre et confirmer la qualité de l’ARNd par électrophorèse sur un gel d’agarose de 1,5 %.
REMARQUE : Une seule bande peut être vue lorsque la synthèse dsRNA est réussie. - Diluer le dsRNA à 1000 ng/μL avec tampon d’élitution et conserver à -20 °C jusqu’à utilisation.
- Préparation d’aiguilles d’injection
- Tirez un capillaire en verre à l’aide d’un tire-aiguille en verre.
- Placez la pointe du capillaire tiré sur un ruban adhésif à double face et brisez la pointe du capillaire avec des forceps.
- Réglez le capillaire sur un injecteur.
REMARQUE : Il est plus facile de manipuler le capillaire si vous portez des gants de nitrile. - Chargez la solution siRNA/dsRNA au capillaire préparé.
REMARQUE : N’utilisez pas le capillaire à plusieurs reprises puisque la pointe du capillaire injecté peut être obstruée par la saleté parce que les larves recueillies dans le champ vivaient dans la boue.
3. injection de siRNA/dsRNA
REMARQUE : La procédure est légèrement différente pour les demoiselles (étape 3.1) et les libellules (étape 3.2).
- Injection à une larve Zygopteran (damselfly) (par exemple, I. senegalensis).
- Anesthésier une larve recouverte d’un papier humide sur de la glace concassée pendant 50 à 70 secondes (figure 3A-C).
REMARQUE : La durée de l’anesthésie glacée dépend de l’état de la larve; si la larve commence à se déplacer après 70 secondes d’anesthésie glacée, 70 secondes supplémentaires d’anesthésie glacée sont appliquées. Pour les larves qui se déplacent rarement (p. ex., P. zonata),cette procédure n’est pas essentielle. - Fixez deux broches des deux côtés du prothorax et fixez la larve à un support fixe (p. ex., un morceau de polystyrène) (figure 3D).
REMARQUE : Ajustez la position et le nombre d’épingles, selon le lieu d’injection. - Étirez la membrane interse segmentale entre le 7ème et le 8ème segment abdominal pour RNAi dans l’abdomen ou entre le prothorax et le synthorax (mésothorax fusionné et métathorax) pour RNAi dans le thorax utilisant des mains et des forceps.
- Gardez la membrane interse segmentale tendue à la main.
- Insérez la pointe du capillaire préparé dans la membrane interse segmentale étirée (Figure 3D, 3F).
- Injecter 1 μL de solution siRNA/dsRNA.
- Anesthésier une larve recouverte d’un papier humide sur de la glace concassée pendant 50 à 70 secondes (figure 3A-C).
- Injection à une larve anisopteran (libellule) (p. ex., P. zonata).
- Essuyer l’eau de la surface larvaire à l’l’l’insu d’une serviette en papier.
- Si nécessaire, fixez deux broches des deux côtés du prothorax et fixez la larve à un support fixe (p. ex., un morceau de polystyrène) (figure 3H).
REMARQUE : Ajustez la position et le nombre d’épingles, selon le lieu d’injection. Dans le cas de P. zonata, il n’est pas essentiel de fixer les larves avec des broches à cette étape. - Étirez la membrane interse segmentale entre le 4ème et le 5ème segment abdominal à la main.
- Faire un petit trou avec une aiguille fine dans la membrane interse segmentale entre le 4ème et le 5ème segment abdominal.
REMARQUE : Cette procédure est nécessaire pour de nombreuses espèces d’anisopteran (libellule) car la membrane interse segmentale est trop difficile à insérer directement dans un capillaire en verre. - Insérez la pointe du capillaire préparé dans le trou préparé (figure 3I).
REMARQUE : Comme le montre la figure 3H,une partie du côté dorsale des larves correspond au côté ventral de l’adulte, de sorte que le phénotype apparaît ventrally lorsqu’il est traité comme indiqué dans la figure 3H-J. - Injecter 1 μL de solution siRNA/dsRNA.
4. électroporation in vivo
- Au besoin, ajouter plus d’épingles pour fixer la larve à un support fixe (p. ex., un morceau de polystyrène).
- Après l’injection de la solution siRNA/dsRNA, appliquez deux gouttelettes de gel à ultrasons sur la surface larvaire à l’aide de forceps.
- Placez les électrodes sur le gel à ultrasons, avec l’électrode positive sur le côté injectée avec la solution siRNA/dsRNA et une électrode négative sur le côté opposé (Figure 3E, 3G, 3J).
REMARQUE : Ne touchez pas directement à l’épiderme de la larve pour éviter l’effet brûlant de l’électroporation. - Générer 10 fois des impulsions d’électroporation (280 ms/s chacune) à l’aide d’un électroporateur.
REMARQUE : Ajustez la tension de l’électroporation en fonction de l’espèce, des stades et des tissus. Dans cette étude, 25 V ont été appliqués à I. senegalensis et 45 V à P. zonata. - Essuyer le reste du gel sur la surface avec une serviette en papier.
- Gardez les larves traitées au repos sur une serviette en papier humide pendant environ un jour pour la récupération et transférez-les dans un étui d’élevage le lendemain.
5. Analyse phénotypique spécifique au site
- Gardez I. senegalensis individuellement dans une boîte de Pétri (5 cm de diamètre) contenant environ 10 mL d’eau et un morceau de papier essuie-tout, et gardez P. zonata dans une cage en plastique avec un maillage jetable non tissé (cage d’éclosion14).
- Pour I. senegalensis, après que les larves cessent de manger, déplacez-les individuellement dans une cage en plastique avec un maillage jetable non tissé.
REMARQUE : Ne mettez pas deux larves ou plus dans la même cage. Sinon, ils se cannibaliseront immédiatement après être devenus adultes. - Après l’émergence de l’adulte, observez et photographiez le phénotype autour de la région où l’électrode positive a été placée pour l’électroporation.
REMARQUE : Le phénotype n’apparaît que dans les patchs. Les phénotypes sont souvent difficiles à reconnaître immédiatement après l’émergence en raison d’une pigmentation incomplète. - Afin d’examiner l’efficacité de l’ARNi (p. ex., RT-PCR quantitatif), si le phénotype est visible, disséquer la région et la comparer à la région sans phénotype.
REMARQUE : Les niveaux de phénotype de l’ARNi, à savoir la taille et l’emplacement de la décoloration cuticule, présentent souvent des variations considérables entre les individus (voir la figure 4). - Gardez les adultes émergés dans 100% EtOH pour les analyses futures.
REMARQUE : La couleur corporelle des espèces d’Odonata s’estompe rapidement après la mort, il est donc important de les stocker dans de l’éthanol avant qu’elles ne meurent. L’éthanol décolore parfois les insectes, dans de tels cas que les insectes sont congelés avant de mourir.
Résultats
Nous avons appliqué le protocole ci-dessus aux ARN à base d’électroporation ciblant le gène MCO2 et les gènes de contrôle négatifs(EGFP pour siRNA et bla pour dsRNA) (i) dans l’abdomen de I. senegalensis (Figure 4), (ii) dans le thorax de I. senegalensis (Figure 5), et (iii) dans l’abdomen de P. zonata (Figure 6). Les résultats des expériences RNAi sont résumés dans le tableau 1. Puisque le siRNA/dsRNA chargé négativement est incorporé seulement dans les cellules positivement chargées, des phénotypes de RNAi ont été observés autour de la région où l’électrode positive a été placée pour l’électroporation.
Dans I. senegalensis et P. zonata, l’inhibition de la pigmentation de la mélanine (c.-à-d. noir, brun et brun rougeâtre) est apparue dans les parcelles de la région où l’électrode positive a été placée (pointes de flèche blanches et lignes pointillées dans la figure 4, figure 5et figure 6) lorsque l’ARNI MCO2 a été réalisée en combinaison avec l’électroporation (tableau 1), comme indiqué précédemment dans N. pygmaea9. En revanche, aucun effet phénotypique n’a été observé autour du site d’électroporation lorsque le gène de contrôle a été injecté(EGFP siRNA ou bla dsRNA) (Figure 4,Figure 5 , et Figure 6, Tableau 1). De plus, l’injection du gène MCO2 sans électroporation n’a eu aucun effet sur la pigmentation adulte (figure 4, tableau 1), ce qui indique que l’électroporation est essentielle pour l’ARNi à Odonata. Il convient de noter que les colorations bleues, vertes et jaunes ne sont pas affectées par le gène RNAi de MCO2 qui est impliqué dans la synthèse de mélanine dans la cuticule, qui reflètent vraisemblablement le fait que ces couleurs corporelles sont attribuées aux granules pigmentaires présents dans les cellules épidermiques qui sont visibles par la cuticule transparente26. Comme le montre la figure 4, aucune différence phénotypique remarquable n’a été observée entre les personnes soumises au traitement par arRNA et au traitement par dsARN, tandis que des variations considérables de taille et d’emplacement du phénotype RNAi ont été observées chez différentes personnes soumises au même traitement RNAi (par exemple, comparer deux exemples d’ARNd IsMCO2 à la figure 4).
Pour déterminer le stade de développement le plus approprié pour le traitement RNAi, nous avons comparé les conséquences phénotypiques du traitement RNAi à cinq stades morphologiques (stade A-E) dans le stade larvaire final de I. senegalensis (figure 7A). L’inhibition de la pigmentation de mélanine provoquée par MCO2 RNAi a été observée dans tous les adultes émergés une fois injectés aux étapes A et B (figure 7C). Lorsqu’on l’injecte aux stades C et D, la suppression de la pigmentation de mélanine a certainement été observée chez certains adultes émergés, mais d’autres adultes ont montré une coloration anormale causée par des blessures (figure 7B).
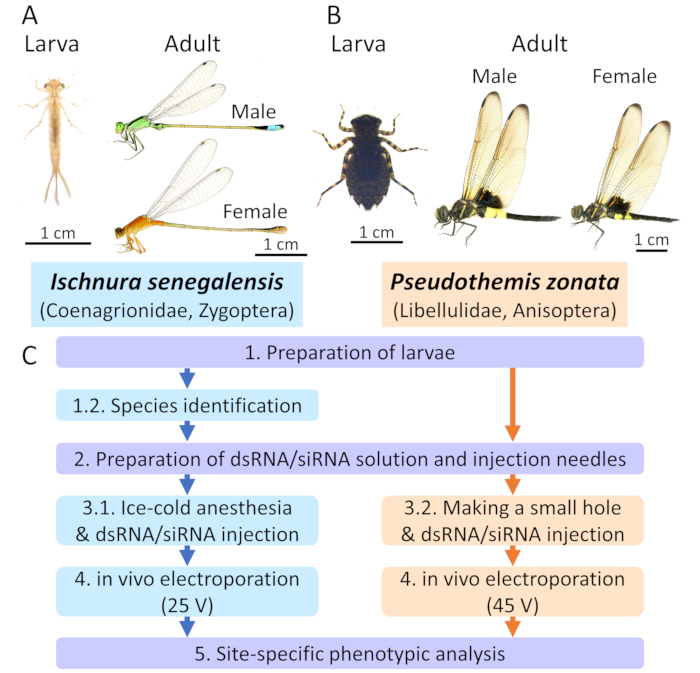
Figure 1 :Méthodes RNAi à médiatisation par électroporation à Odonata. R. Ischnura senegalensis (Coenagirionidae) en tant qu’espèce représentative de damselfly. B. Pseudothemis zonata (Libellulidae) en tant qu’espèce représentative de libellule. C. C. Le schéma global des protocoles. Les boîtes bleues et oranges indiquent les protocoles pour I. senegalensis et P. zonata,respectivement. Les boîtes violettes indiquent les protocoles communs appliqués aux deux espèces. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
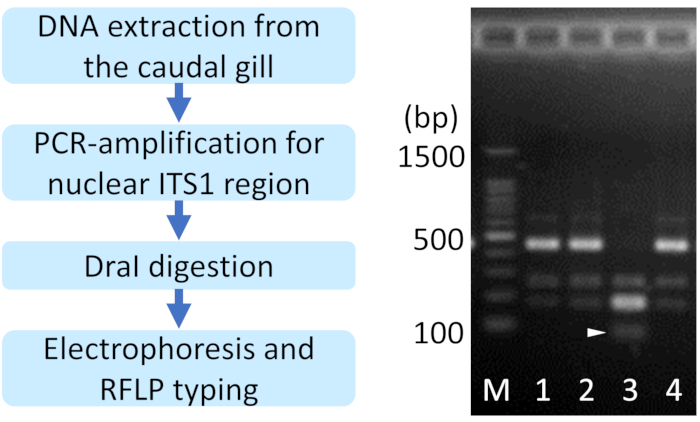
Figure 2 :Résultat représentatif de l’identification des espèces basées sur le polymorphisme de la longueur des fragments de restriction (RFLP) pour les espèces d’Ischnura. Arrowhead indique la bande spécifique i. senegalensis. 1, 2, 4: I. asiatica, 3: I. senegalensis, M: marqueur échelle paire de 100 base. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 3 :Méthode RNAi à médiatisation par électroporation dans Odonata. A-C. Anesthésie glacée d’I. senegalensis. Les pointes de flèche indiquent une larve. R. Mettre une larve sur de la glace concassée avec un papier mouillé. B. Vue agrandie d’une larve sur la glace. C. C. Une larve couverte d’un papier humide sur la glace. D-G. Méthode RNAi pour I. senegalensis. D. D. Injection dans le thorax. E. E. Électroporation du thorax. F. F. Injection dans l’abdomen. G. Électroporation sur l’abdomen. H-J. Méthode RNAi pour P. zonata. H. Faire un petit trou sur l’abdomen. Je. Injection dans l’abdomen. J. J. Électroporation sur l’abdomen. Les flèches indiquent l’intérêt de faire un trou ou une injection. +, -: Électrodes latérales positives/négatives. Les chiffres indiquent le segment abdominal. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
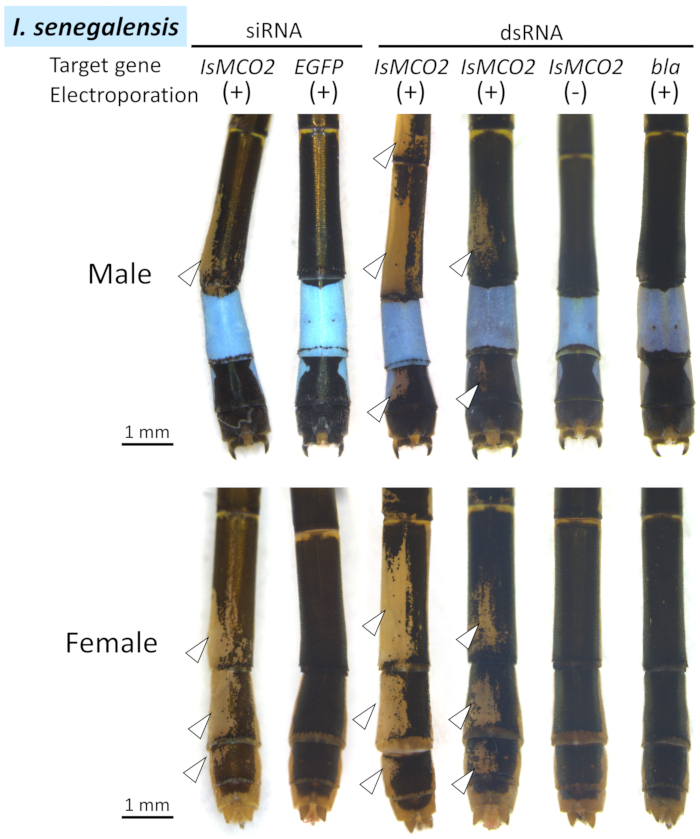
Figure 4: Vues dorsales des phénotypes RNAi sur l’abdomen de I. senegalensis. Les pointes de flèche blanches indiquent les régions de mélanine supprimée. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
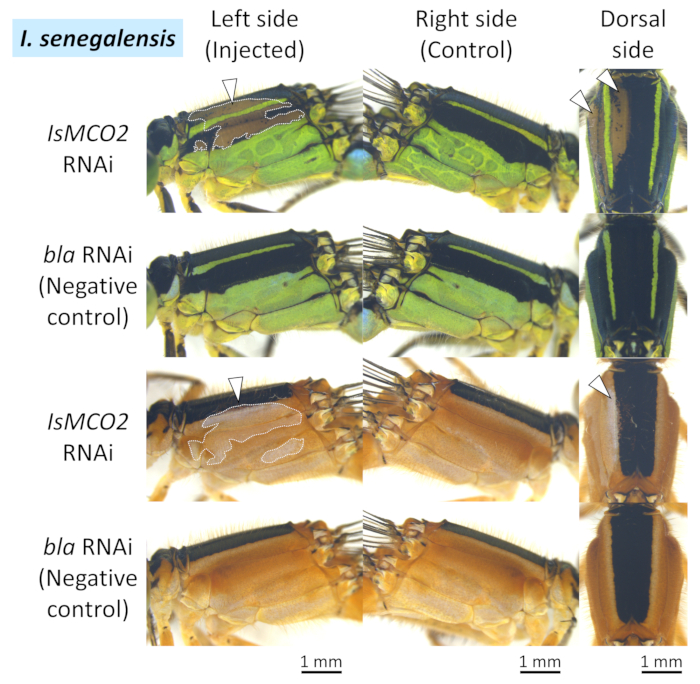
Figure 5: Vues latérales et dorsales des phénotypes RNAi sur le thorax d’I. senegalensis. Les pointes de flèche blanches et les lignes pointillées indiquent les régions de pigmentation supprimée. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
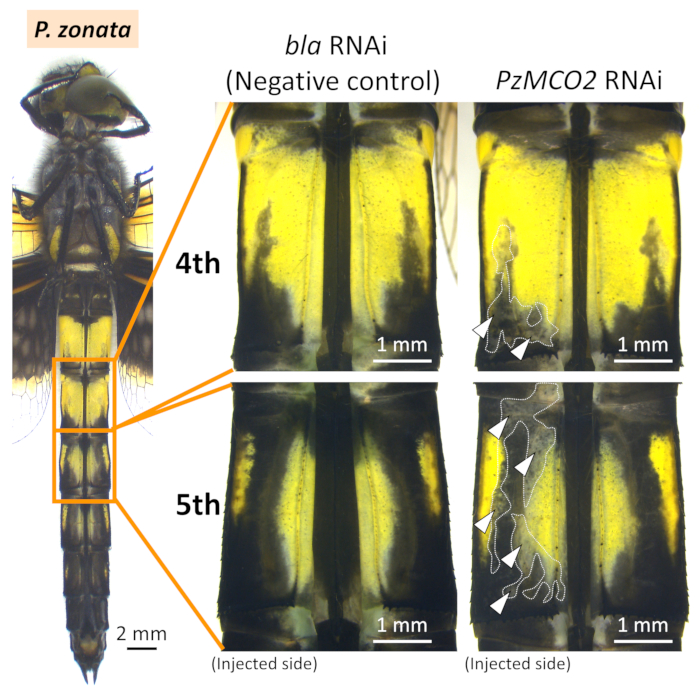
Figure 6: Vues ventrales des phénotypes RNAi dans l’abdomen de P. zonata. Les pointes de flèche blanches et les lignes pointillées indiquent les régions de mélanine supprimée. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
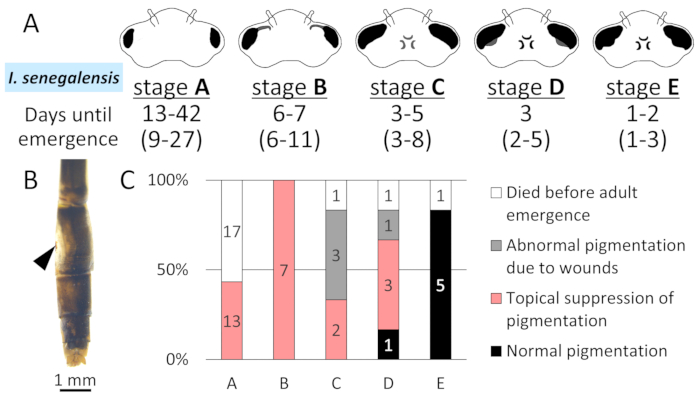
Figure 7: Effets isMCO2 RNAi dépendants de l’étape lors de l’image larvaire finale d’I.senegalensis. R. Changements morphologiques dans les yeux composés à cinq stades morphologiques (stade A-E) et le nombre de jours à l’émergence adulte dans cette étude. Les nombres entre parenthèses proviennent du rapport précédent14. B. Pigmentation anormale due aux blessures. Arrowhead indique le site d’électroporation. C. C. L’effet de RNAi à cinq étapes morphologiques sur la pigmentation adulte dans I. senegalensis. Le numéro sur la barre indique le nombre de personnes. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
| Espèces | I.senegalensis | P.zonata | |||||||||
| Région injectée | Abdomen | Thorax ( Thorax ) | Abdomen | ||||||||
| siRNA/dsRNA | Siarn | Dsrna | Dsrna | Dsrna | |||||||
| Gène cible | IsMCO2 (en) | EGFP (egfp) | IsMCO2 (en) | IsMCO2 (en) | Bla | IsMCO2 (en) | Bla | PzMCO2 PzMCO2 | PzMCO2 PzMCO2 | Bla | |
| Électroporation | + | + | + | - | + | + | + | + | - | + | |
| Larves injectées | 22 | 25 | 30 | 6 | 53 | 12 | 20 | 17 | 5 | 9 | |
| Adultes émergés | 7 | 6 | 13 | 4 | 40 | 11 | 14 | 11 | 2 | 5 | |
| Adultes ayant des régions moins pigmentées (ratio) | 7 (100%) | 0 (0%) | 13 (100%) | 0 (0%) | 0 (0%) | 10 (91%) | 0 (0%) | 11 (100%) | 0 (0%) | 0 (0%) | |
Tableau 1. L’effet de RNAi sur la pigmentation adulte dans I. senegalensis et P. zonata. Les résultats au stade A sont présentés dans I. senegalensis. IsMCO2: gène oxidase multicopper 2 d’I. senegalensis, EGFP: Gène amélioré des protéines fluorescentes vertes, bla: gène bêta lactamase de pGEM-T Easy Vector, PzMCO2: oxidase multicopper 2 gène de P. zonata. Les résultats des gènes de contrôle représentent le nombre total d’expériences que les auteurs ont menées à ce jour.
Discussion
Létalité du traitement RNAi
Nous avons constaté que la létalité du traitement RNAi dépend fortement de l’histoire de l’élevage et de l’état des larves d’Odonata. Peu de temps après la collecte sur le terrain, les larves sont généralement en bonne santé et présentent de faibles taux de mortalité après le traitement par électroporation. En revanche, les larves élevés en laboratoire pendant une longue période (p. ex., un mois) ont tendance à souffrir de faibles taux de réussite de l’émergence des adultes. Dans I. senegalensis, au lieu des larves recueillies sur le terrain, les larves élevés en laboratoire à partir d’œufspeuvent être utilisées 14, mais les taux de réussite des ARNi utilisant les larves élevés en laboratoire ont tendance à être considérablement inférieurs (de nombreuses personnes sont mortes pendant la métamorphose) que celles utilisant les larves recueillies sur le terrain. En outre, l’alimentation larvaire fréquente et l’élevage de l’eau propre sont importants pour augmenter les taux de réussite de l’émergence des adultes et réduire la létalité du traitement rnai.
Efficacité du traitement RNAi
Comme décrit ci-dessus, les niveaux de phénotype RNAi, à savoir la taille et l’emplacement de la décoloration cuticule, présentaient souvent des variations considérables entre les individus soumis au même traitement RNAi (p. ex., figure 4),mais les niveaux de pénitence phénotypique semblent être remarquablement différents entre les espèces odonata. Les régions phénotypique observées étaient plus grandes et plus importantes en I. senegalensis (figures 4-5) que dans P. zonata (figure 6) et N. pygmaea9. Cette différence peut être due à l’épaisseur de la cuticule sur la surface larvaire, considérant que la cuticule de I. senegalensis est plus mince que la cuticule de P. zonata et N. pygmaea). En ce qui nous concerne, aucune différence claire n’a été reconnue entre les effets du siRNA et de l’ARNd (figure 4, tableau 1).
Étape de développement appropriée pour RNAi
Il convient de noter qu’une mise en scène larvaire appropriée est importante pour effectuer rnai efficacement. L’inhibition de la pigmentation adulte a été causée par l’ARNi MCO2 avant le stade D (environ 3 jours avant l’émergence de l’adulte), ce qui est conforme au rapport précédent sur N. pygmaea9. Les phénotypes RNAi observés lorsqu’ils ont été injectés aux stades C et D étaient moins visibles que ceux traités aux stades A et B, ce qui indique que les stades C et D peuvent être trop tard pour supprimer suffisamment l’expression du gène. Le moment approprié pour le traitement d’ARNi dépend du moment de l’expression des gènes, et le gène MCO2 présente une expression transitoirement élevée pendant l’émergenceadulte 9, comme chez d’autresinsectes 17,18. Dans le stinkbug Plautia stali, RNAi knockdown du gène MCO2 a été observé à partir du jour 4 après injection27, ce qui est compatible avec les résultats actuels.
Notre étude précédente sur I. senegalensis a montré qu’après le stade B, les jours d’émergence des adultes présentent une variation relativement faible parmi la majorité des larves finales du stade, ce qui suggère que le stade B peut correspondre à l’apparition du processus vers l’émergence des adultes, après quoi les processus de développement de la métamorphose se déroulent d’une manière préfixéeet coordonnée 14. Des anomalies morphologiques causées par des blessures étaient souvent observées lorsque les larves étaient traitées par RNAi aux stades C et D (figure 7B, 7C). Ceci est susceptible d’être associé à une progression dramatique de la métamorphose pendant ces étapes, suggérant que le traitement de RNAi devrait être évité du stade C et ainsi de suite. En résumé, nous recommandons que les larves finales du stade A ou B (ou au stade précédant l’expansion significative des ailes larvaires) soient utilisées pour des expériences d’ARNi.
Utilité et supériorité de la méthode RNAi à médiatisation par électroporation
Le RNAi conventionnel est une méthode expérimentale simple et puissante, mais certaines lignées d’insectes comme les papillons10,pucerons 28 et libellules9 présentent une faible efficacité RNAi, pour lequel l’établissement de l’analyse de la fonction génétique est un défi majeur. Dans cette étude, nous avons constaté que l’ARNi à base d’électroporation peut induire une suppression génétique locale chez les libellules avec une efficacité de près de 100 %, du moins en épiderme, si elle est traitée à des stades de développement appropriés (tableau 1). Récemment, les knockouts génétiques basés sur CRISPR/Cas9 ont été appliqués avec succès à une variété d’insectes, fournissant un outil génétique moléculaire puissant pour les organismes non modèles29. Ici, cependant, nous faisons souligner que CRISPR/Cas9 est certainement grand mais la méthode RNAi électroporation-ment médiatisée peut être supérieure à CRISPR/Cas9 à certains égards.
Tout d’abord, dans la méthode RNAi à base d’électroporation, la région du corps où apparaissent les phénotypes RNAi peut être facilement contrôlée expérimentalement par la position de l’électrode positive lors de l’électroporation. En outre, puisque la région où l’expression du gène est supprimée est limitée autour de la région où l’électrode positive a été placée, les phénotypes RNAi peuvent être facilement comparés aux phénotypes de contrôle côte à côte chez le même individu. Deuxièmement, par rapport à la méthode CRISPR/Cas9 dans laquelle les œufs injectés doivent être élevés à l’âge adulte pour observer les phénotypes knockout, le RNAi à base d’électroporation est supérieur en ce que les phénotypes knockdown gène peut généralement être observé dans un temps beaucoup plus court. Par exemple, il faut de trois à quatre mois pour I. senegalensis et un à deux ans pour P. zonata des oeufs aux adultes14,30. Cependant, afin d’observer les phénotypes RNAi dans l’épiderme adulte, il faut moins d’un mois de l’injection de dsRNA dans les larves finales de stade B à l’émergence adulte pour I. senegalensis et P. zonata (figure 7). Troisièmement, la méthode RNAi à base d’électroporation implique l’injection d’ARNd dans de grandes larves, ce qui est plus facile que la microinjection dans les œufs minuscules nécessaires à la méthode CRISPR/Cas9. De plus, l’ARNi à 20 œufs 2012 à 20 p. 100 est applicable aux espèces d’insectes dont les œufs nouvellement pondus sont difficiles à recueillir. Par exemple, les femelles de P. zonata pondent des œufs sur des plantes flottantes à la surface de l’eau pendant le vol, et il est donc difficile de recueillir leurs œufs à la fois sur le terrain et en laboratoire. Par conséquent, nous nous attendons à ce que ce protocole puisse être généralement applicable aux organismes non modèles dans lesquels la méthode rnai conventionnelle ne fonctionne pas efficacement.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Minoru Moriyama pour ses conseils techniques et ses soutiens, Bin Hirota et Ryutaro Suzuki pour la collecte des larves d’Odonata, et Misa Shinya pour les commentaires utiles sur le manuscrit. Ce travail a été soutenu par JSPS KAKENHI Grant Numbers JP18J21561 à GO et JP18H02491, JP18H04893, JP19H03287, et JP20H04936 à RF.
matériels
| Name | Company | Catalog Number | Comments |
| 12-well plate | Violamo | VTC-P12 | For rearing larvae before injection |
| Brine shrimp eggs | JAPAN PET DESIGN | 4975677012396 | |
| Calibrated micropipette (1-5 µL) | Drummond | 2-000-001-90 | |
| Deposit pestle 1.5 mL | Thermo Fisher Scientific | K749520-0090 | |
| Digital high defenition microscope camera | Leica Microsystems | MC170HD | |
| Disposable non-woven mesh | HAKUGEN EARTH | DSC-105A | |
| DNA Ligation Kit Ver.2.1 | Takara Bio | 6022 | |
| DraI | Takara Bio | 1037A | |
| Electrode (1mmφ) | NEPAGENE | CUY650P1 | |
| Electroporator | CellProduce | Cure-gene | |
| Forceps | KOWA Forceps Industry | K-10 No1 | |
| Glass needle puller | NARISHIGE | PN-3 | |
| Hand mixer | AS ONE | 1-229-02 | |
| Hand net | HOGA | IS33-3B | |
| HEPES | FUJIFILM Wako Pure Chemical Corporation | 346-01373 | For injection buffer |
| Insect pin capitate No. 3 | Shiga Konchu Fukyusha | N230 | For fixing larvae |
| KCl | FUJIFILM Wako Pure Chemical Corporation | 163-03545 | For PBS |
| KH2PO4 | FUJIFILM Wako Pure Chemical Corporation | 169-04245 | For PBS |
| KOAc | FUJIFILM Wako Pure Chemical Corporation | 160-03175 | For injection buffer |
| KOH | FUJIFILM Wako Pure Chemical Corporation | 168-21815 | For injection buffer |
| Laboratory Jack (150x150) | AS ONE | 1-4642-11 | For adjusting the position of the manipulator |
| LOGIQLEAN Gel for Ultrasound Hard type | GE Healthcare | 2369385 | |
| Manipulator | Muromachi Kikai Co., Ltd. | SJ-1 | For adjusting the position of the injector and the capillary |
| MEGAscript RNAi kit | Thermo Fisher Scientific | AM1626 | |
| Mg(OAc)2 | FUJIFILM Wako Pure Chemical Corporation | 130-00095 | For injection buffer |
| Na2HPO4 | FUJIFILM Wako Pure Chemical Corporation | 197-02865 | For PBS |
| NaCl | FUJIFILM Wako Pure Chemical Corporation | 191-01665 | For PBS |
| Petri dish (5 cm diameter) | Iwaki | 1010-060 | For rearing injected larvae |
| Pneumatic Injector | NARISHIGE | IM-12 | |
| pT7Blue T-Vector | Novagen | 69820 | |
| QIAquick PCR Purification Kit | QIAGEN | 28106 | |
| RNAiso Plus | FUJIFILM Wako Pure Chemical Corporation | 9109 | |
| RNeasy Mini Kit | QIAGEN | 74106 | |
| Shiga micro insect pin with stainless headless | Shiga Konchu Fukyusha | N251 | For making a small hole |
| Stereoscopic microscope | Leica Microsystems | S8APO | |
| SuperScript IV Reverse Transcriptase | Thermo Fisher Scientific | 18090010 | |
| TaKaRa Ex Taq | Takara Bio | RR001B | For PCR-amplification from plasmid |
| Tks Gflex DNA Polymerase | Takara Bio | R060B | For PCR-amplification from caudal gill |
Références
- Misof, B., et al. Phylogenomics resolves the timing and pattern of insect evolution. Science. 346 (6210), 763-767 (2014).
- Wipfler, B., et al. Evolutionary history of Polyneoptera and its implications for our understanding of early winged insects. Proceedings of the National Academy of Sciences of the United States of America. 116 (8), 3024-3029 (2019).
- Corbet, P. S. . Dragonflies Behavior and Ecology of Odonata. , (1999).
- Futahashi, R. Color vision and color formation in dragonflies. Current Opinion in Insect Science. 17, 32-39 (2016).
- Futahashi, R. Diversity of UV reflection patterns in Odonata. Frontiers in Ecology and Evolution. 8 (201), (2020).
- Córdoba-Aguilar, A. . Dragonflies and damselflies. Model organisms for ecological and evolutionary research. , (2008).
- Bybee, S., et al. Odonata (dragonflies and damselflies) as a bridge between ecology and evolutionary genomics. Frontiers in Zoology. 13 (46), (2016).
- Linz, D. M., Clark-Hachtel, C. M., Borràs-Castelles, F., Tomoyasu, Y. Larval RNA interference the red flour beetle, Tribolium castaneum. Journal of Visualized Experiments. 92, 52059 (2014).
- Okude, G., et al. Electroporation-mediated RNA interference reveals a role of multicopper oxidase 2 gene in dragonfly’s cuticular pigmentation. Applied Entomology and Zoology. 53 (3), 379-387 (2017).
- Terenius, O., et al. RNA interference in Lepidoptera: An overview of successful and unsuccessful studies and implications for experimental design. Journal of Insect Physiology. 57, 231-245 (2011).
- Ando, T., Fujiwara, H. Electroporation-mediated somatic transgenesis for rapid functional analysis in insects. Development. 140, 454-458 (2013).
- Nishikawa, H., et al. A genetic mechanism for female-limited Batesian mimicry in Papilio butterfly. Nature Genetics. 47 (4), 405-409 (2015).
- Osanai-Futahashi, M., et al. Positional cloning of a Bombyx pink-eyed white egg locus reveals the major role of cardinal in ommochrome synthesis. Heredity. 116 (2), 135-145 (2016).
- Okude, G., Futahashi, R., Tanahashi, M., Fukatsu, T. Laboratory rearing system for Ischnura senegalensis (Insecta: Odonata) enables detailed description of dragonfly’s larval development and morphogenesis. Zoological Science. 34 (5), 386-397 (2017).
- Ozono, A., Kawashima, I., Futahashi, R. . Dragonflies of Japan (3rd edition). , (2017).
- Ozono, A., Kawashima, I., Futahashi, R. . The Handbook of Japanese Aquatic Insects. Volume 3: Dragonfly larvae. , (2019).
- Arakane, Y., Noh, M. Y., Asano, T., Kramer, K. J. Tyrosine metabolism for insect cuticle pigmentation and sclerotization. Extracellular Composite Matrices in Arthropods. , 165-220 (2016).
- Asano, T., et al. Mini-review an insect-specific system for terrestrialization: Laccase-mediated cuticle formation. Insect Biochemistry and Molecular Biology. 108, 61-70 (2019).
- Futahashi, R., Okude, G., Sugimura, M., Ugai, S. Interspecific hybrids in Japanese Odonata. Tombo. 60, 1-49 (2018).
- Okude, G., Fukatsu, T., Futahashi, R. Interspecific crossing between blue-tailed damselflies Ischnura elegans and I. senegalensis in the laboratory. Entomological Science. 23 (2), 165-172 (2020).
- Naito, Y., Yoshimura, J., Morishita, S., Ui-Tei, K. siDirect 2.0: updated software for designing functional siRNA with reduced seed-dependent off-target effect. BMC Bioinformatics. 10, 392 (2009).
- Yamaguchi, J., Mizoguchi, T., Fujiwara, H. siRNAs induce efficient RNAi response in Bombyx mori embryos. PLoS ONE. 6 (9), e25469 (2011).
- Untergasser, A., et al. Primer3--new capabilities and interfaces. Nucleic Acids Research. 40 (15), e115 (2012).
- Futahashi, R. Whole-mount in situ hybridization of sectioned tissues of species hybrids to detect cis-regulatory changes in gene expression pattern. Methods in Molecular Biology. 772, 319-328 (2011).
- Matsuura, Y., Kikuchi, Y., Miura, T., Fukatsu, T. Ultrabithorax is essential for bacteriocyte development. Proceedings of the National Academy of Sciences of the United States of America. 112 (30), 9376-9381 (2015).
- Henze, M. J., Lind, O., Wilts, B. D., Kelber, A. Pterin-pigmented nanospheres create the colours of the polymorphiic damselfly Ischnura elegans. Journal of The Royal Society Interface. 16, 20180785 (2019).
- Nishide, Y., et al. Diversity and function of multicopper oxidase genes in the stinkbug Plautia stali. Scientific Reports. 10 (1), 3464 (2020).
- Christiaens, O., Swevers, L., Smagghe, G. DsRNA degradation in the pea aphid (Acyrthosiphon pisum) associated with lack of response in RNAi feeding and injection assay. Peptides. 53, 307-314 (2014).
- Sun, D., Guo, Z., Liu, Y., Zhang, Y. Progress and prospects of CRISPR/Cas systems in insects and other arthropods. Frontiers in Physiology. 8, 608 (2017).
- Miyakawa, K. A study of the life-history of Pseudothemis zonata (Burm.) (Odonata, Libellulidae) II. Immature stage. Kontyû. 37 (4), 409-422 (1969).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon