A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בדיקת כרומוזומים של עוברים אנושיים טרום השרשה באמצעות מדיום תרבית משומש: איסוף דגימות וניתוח פלואידיות כרומוזומליות
* These authors contributed equally
In This Article
Erratum Notice
Summary
המחקר הנוכחי מדווח על פרוטוקול לבדיקת כרומוזומים של עוברים אנושיים המשתמש במדיום תרבית משומש, המונע ביופסיה של עוברים ומאפשר זיהוי כרומוזום פלואידי באמצעות NGS. המאמר הנוכחי מציג את ההליך המפורט, כולל הכנת מדיום תרבית, הגברה של גנום שלם (WGA), הכנת ספריית ריצוף הדור הבא (NGS) וניתוח נתונים.
Abstract
בהפריה חוץ גופית קלינית (IVF), השיטה הרווחת עבור PGT-A דורשת ביופסיה של כמה תאים מהטרופקטודרם (TE). זוהי השושלת היוצרת את השליה. שיטה זו, לעומת זאת, דורשת מיומנויות מיוחדות, היא פולשנית, וסובלת מתוצאות חיוביות ושליליות שגויות מכיוון שמספרי הכרומוזומים ב- TE ומסת התא הפנימית (ICM), המתפתחת לעובר, אינם תמיד זהים. NICS, טכנולוגיה הדורשת ריצוף של דנ"א המשתחרר למדיום התרבית הן מ-TE והן מ-ICM, עשויה להציע מוצא לבעיות אלה, אך הוכחה בעבר כבעלת יעילות מוגבלת. המחקר הנוכחי מדווח על הפרוטוקול המלא של NICS, הכולל שיטות דגימה בינוניות בתרבית, הגברה של גנום שלם (WGA) והכנת ספריות, וניתוח נתוני NGS באמצעות תוכנת ניתוח. בהתחשב בזמני ההקפאה השונים במעבדות עוברים שונות, לאמבריולוגים יש שתי שיטות לאיסוף מדיום תרבית עוברים שניתן לבחור על פי התנאים בפועל של מעבדת הפריה חוץ גופית.
Introduction
טכנולוגיות רבייה בסיוע (ARTs) שימשו יותר ויותר לטיפול באי פוריות. עם זאת, שיעור ההצלחה של ART, כגון הפריה חוץ גופית, היה מוגבל, ושיעור אובדן ההריון גבוה משמעותית מזה של האוכלוסייה הרגילה1. הגורם העיקרי לבעיות אלה הוא הפרעות כרומוזומליות, הקיימות בדרך כלל בעוברים אנושיים טרום השרשה2. PGT-A היא שיטה יעילה לבדיקת עוברים לאיזון כרומוזומלי לפני השרשה 3,4. כמה מחקרים הוכיחו כי PGT-A יכול להפחית את שיעור ההפלות ולשפר את שיעור ההריון 5,6,7,8. עם זאת, PGT-A דורש מומחיות טכנית מורכבת הדורשת הכשרה וניסיון ספציפיים. הליך ביופסיית העובר הפולשני עלול גם לגרום נזק לעוברים9. מחקרים הראו כי ביופסיית בלסטומרים יכולה לעכב את ההתפתחות הבאה, ומספר הביופסיות עשוי להשפיע על שיעורי ההשתלה10. למרות שנושא הבטיחות הביולוגית ארוכת הטווח של ביופסיית עוברים עדיין לא נבדק ביסודיות בבני אדם, מחקרים בבעלי חיים הראו את ההשפעות השליליות שלה על התפתחות העובר11,12,13.
דיווחים קודמים הצביעו על כך שכמויות זעירות של חומרי דנ"א הופרשו למדיום התרבית במהלך התפתחות העובר, ונעשו מאמצים לבצע בדיקת כרומוזומים מקיפה (CCS) באמצעות תרבית עוברים משומשת 14,15,16,17,18. עם זאת, שיעורי הגילוי והדיוק של הבדיקות לא עמדו בדרישות לשימוש קליני נרחב. המחקר הנוכחי דיווח על שיפור בבדיקת NICS להגדלת שיעורי האיתור, כמו גם את הדיוק של מבחן NICS19. בשנים האחרונות, נוזל בלסטוקוele (BF) נחקר כמדגם אנליטי של PGT-A זעיר פולשני. עם זאת, שיעור ההגברה המוצלחת של הגנום כולו והדנ"א הניתן לזיהוי בדגימות נוזל בלסטוציסט נע בין 34.8% ל-82%20,21,22. נפח ה-BF המדווח במחקרים שונים נע בין 0.3 nL ל-1 μL. לאור הכמות הנמוכה של DNA ב- BF, ניתן להגדיל את כמות ה- DNA ללא תאים על ידי ערבוב נוזל בלסטוציסט ומדיום תרבית כדי לשפר את שיעור ההצלחה ואת עקביות האיתור. Kuznyetsov et al.23 ו-Li et al.24 טיפלו בזונה פלוצ'ידה בלייזר ושחררו נוזל בלסטוציסט למדיום התרבית כדי לשפר את הכמות הכוללת של דנ"א עוברי, וקצב ההגברה של דגימות בינוניות/BF משולבות לאחר WGA היה 100% ו-97.5%, בהתאמה. Jiao et al.25 השיגו גם שיעור הצלחה של 100% הגברה באמצעות אותה שיטה.
המחקר הנוכחי מדווח על פרוטוקול מפורט הכולל הכנת דגימות מדיה משומשות, הכנת NGS וניתוח נתונים. על ידי הסרה זהירה של תאי קומולוס מביציות, המחקר הנוכחי ביצע הזרקת זרע יחיד תוך ציטופלזמית (ICSI) ותרבית בלסטוציסט. היום, 4 ימים 5/יום 6 בילה בינוני נאסף להכנת ספריית WGA ו- NGS. על ידי שימוש בטכנולוגיית NICS, המחקר הנוכחי ייעל את שלבי הכנת ספריית WGA ו- NGS תוך כ -3 שעות והשיג תוצאות CCS באופן לא פולשני תוך כ -9 שעות.
Protocol
אישור אתי התקבל מוועדת האתיקה של בית החולים השלישי של אוניברסיטת פקין.
1. הכנה
הערה: החומרים והציוד הדרושים מפורטים בטבלת החומרים.
- ריאגנטים
- חימום מקדים ושיווי משקל (מאוזן) של 20-30 מיקרוליטר מדיום גמטה/מדיום דישון ומדיום תרבית שלב מחשוף/בלסטוציסט (מכוסה בשמן מינרלי) והיאלורונידאז (בצינור מכוסה) ב-37°C, 5%CO2 ו-5% O2 באינקובטור טרי-גז לילה לפני השימוש.
- מחממים מראש את ההיאלורונידאז ל-37°C על משטח עבודה במכסה אדים.
- הכינו מאגר ויטריפיקציה וריאגנטים לאיסוף דגימות בהתאם להוראות היצרן.
- כלים
- הכינו פיפטות איסוף והעברה של דגימות (קוטר פנימי של ~200 עד 250 מיקרומטר), פיפטות דנודציה/חשפניות (קוטר פנימי של ≥150 מיקרומטר, ~130-140 מיקרומטר, ו~120 מיקרומטר), ופיפטות לשטיפה (קוטר פנימי של ~150 מיקרומטר) על ידי משיכת פיפטות פסטר מזכוכית ליצירת קצוות עדינים פתוחים מלוטשים באש.
הערה: ניתן לרכוש ישירות את הפיפטות המשמשות לאיסוף/העברת דגימות, דנודציה ושטיפה. ניתן לרכוש ישירות גם את מחטי האחיזה ומחטי ההזרקה.
- הכינו פיפטות איסוף והעברה של דגימות (קוטר פנימי של ~200 עד 250 מיקרומטר), פיפטות דנודציה/חשפניות (קוטר פנימי של ≥150 מיקרומטר, ~130-140 מיקרומטר, ו~120 מיקרומטר), ופיפטות לשטיפה (קוטר פנימי של ~150 מיקרומטר) על ידי משיכת פיפטות פסטר מזכוכית ליצירת קצוות עדינים פתוחים מלוטשים באש.
2. פרוטוקול 1: איסוף דוגמאות
- טיפול מקדים בקומפלקס ביציות-קורונה-קומולוס (OCCCs) לפני העיכול עם היאלורונידאז
- השיגו גירוי שחלתי בעזרת הורמון מגרה זקיק (FSH) ותכשירי גונדוטרופין בגיל המעבר האנושי (hMG). כאשר זקיק העופרת הוא >18 מ"מ, השתמש ב- 10,000 IU של גונדוטרופין כוריוני (hCG) להבשלה סופית של הביציות.
- בצע שאיבת ביציות 36 שעות לאחר זריקת טריגר. הרימו והעבירו ביציות לצלחות תרבית רקמה עם 2.5 מ"ל m-HTF שחומם מראש מכוסה בשמן מינרלי.
- העבירו במהירות את OCCCs לבאר המרכזית של צלחת תרבית איברים המכילה 1 מ"ל של מדיום הפריה באמצעות פיפטת העברה ולאחר מכן לדגור עם הביציות ב 37 ° C ב 5% CO 2 ו 5% O 2 אינקובטור במשך2-4 שעות.
- לעכל OCCCs עם hyaluronidase על ידי הוספת 1 מ"ל של 37 ° C מחומם מראש hyaluronidase (80 IU / מ"ל) לבאר המרכזית של צלחת תרבית איברים המכילה OCCCs (שלב 2.1.3). יש לשמור על הריכוז הסופי של היאלורונידאז על 40 IU/mL ולערבב היטב.
- לדגור OCCCs על פלטפורמה תרמית 37 ° C במשך 2 דקות. התבוננו בשינויים תחת מיקרוסקופ כל 30 שניות עד שנותרו רק 1-2 שכבות של תאי גרנולוסה.
- דנודציה של תאי גרנולוזה
- מעבירים במהירות את ה-OCCCs המעוכלים בצלחת התרבית לטיפול בביציות ומכסים בשמן מינרלי בכל באר.
- התבוננו בתאי הגרנולוזה המופרדים תחת מיקרוסקופ. שאפו בעדינות ושחררו את הביציות 5 פעמים כדי להסיר שאריות תאי גרנולוזה סביב הביציות.
- חזור על השלב הקודם ב -3 הבארות הנותרות כדי להסיר לחלוטין את תאי הגרנולוסה.
הערה: ניתן לבצע את השלבים הנ"ל (2.1-2.3) בהתאם לפעולה השגרתית של כל מעבדה.
- הערכת הביצית
- להעריך את השלמות של הסרת תאי גרנולוזה באמצעות מיקרוסקופ. אם לא ניתן היה להסיר את התאים לחלוטין, אז שימור של 5 או פחות תאי גרנולוזה מקובל בשלב זה.
הערה: אם תאי קומולוס עדיין מחוברים לביצית, ניתן להסיר את השאריות מאוחר יותר ביום 3 לפני העברת העובר ממדיום תרבית בשלב המחשוף למדיום תרבית בשלב הבלסטוציסט.
- להעריך את השלמות של הסרת תאי גרנולוזה באמצעות מיקרוסקופ. אם לא ניתן היה להסיר את התאים לחלוטין, אז שימור של 5 או פחות תאי גרנולוזה מקובל בשלב זה.
- לאחר ביצוע הזרקת זרע תוך ציטופלזמית (ICSI)26, מעבירים את הביציות למיקרו-טיפות בינוניות בתרבית עוברים במחשוף של 20-30 מיקרו-ליטר (ביצית אחת מתאימה למיקרו-טיפה אחת) באמצעות פיפטות העברה ודוגרות באינקובטור של 37°C, 5% CO 2 ו-5% O2.
- רשום את היום של ICSI כיום 0. בדקו את העוברים והניקוד על פי סדנת הקונצנזוס באיסטנבול בנושא הערכת עוברים של יום 1 להפריה (כ-18 שעות), יום 2 (כ-45 שעות) ויום 3 (כ-68 שעות) למחשוף עוברים27.
- שטיפת עוברים
- הכינו 20-30 מיקרו-ליטר של מיקרו-טיפות בינוניות בתרבית בלסטוציסט לכל עובר מכוסה בשמן מינרלי בצלחות תרבית רקמה ביום 2 באינקובטור של 37°C, 5% CO 2 ו-5% O2.
- הכינו עוד שלוש מיקרו-טיפות מכוסות בשמן מינרלי, ותייגו את כלי תרבית הרקמה החדשים לשטיפה מס' 1-3.
- העבירו את 3 העוברים של היום לטיפות הכביסה. שאפו בעדינות ושחררו את העוברים 3 פעמים בכל טיפה באמצעות פיפטות דנודציה.
הערה: הליך זה יכול גם לסייע בהסרת שאריות התאים הגרגיריים המחוברים לעובר. - התבוננו והעריכו את העוברים תחת מיקרוסקופ ביום השלישי לפני שהתווך השתנה ממדיום תרבית בשלב המחשוף למדיום תרבית בלסטוציסט לצורך ניקוד מורפולוגי. אם תאי קומולוס עדיין היו מחוברים לעובר, פיפטה מתאימה למעלה ולמטה בתרבית בלסטוציסט אחרת מחוממת מראש ומאוזנת טיפה בינונית מכוסה בשמן מינרלי עם פיפטה חשפנית עד שתאי הקומולוס הוסרו לחלוטין.
הערה: היה צורך להסיר לחלוטין את כל תאי הקומולוס המחוברים ביום 3 לפני העברת העובר מהצלחת הבינונית של תרבית שלב המחשוף לצלחת המדיום של תרבית שלב הבלסטוציסט. כל תאי הקומולוס הנותרים יפריעו לניתוח הסופי ויתנו תוצאות שליליות כוזבות.
- שתי אפשרויות לקולקציית מדיום תרבות
הערה: המרכז להפריה חוץ גופית יכול לבחור באחת משתי שיטות לאיסוף מדיום תרבות בהתבסס על המשאבים, הדרישות וההעדפות של המרכז.- אפשרות 1: שטיפת עוברים ותרבית
הערה: אפשרות זו מיועדת למעבדות IVF המבצעות ויטריפיקציה בבוקר היום החמישי.- מעבירים את העובר למיקרו-טיפות מחוממות מראש (37°C) של מדיום תרבית, ושוטפים בעדינות כל עובר באופן סדרתי ב-3 מיקרו-טיפות על ידי פיפטינג ביום 4 אחר הצהריים.
- מעבירים כל עובר למיקרו-טיפה יחידה ייחודית שחוממה מראש (37°C) של מדיום תרבית לאיסוף דגימה. נפחה של טיפה אחת של מדיום תרבית אינו יכול לעלות על 25 מיקרוליטר.
- בצע תרבית עובר בלסטוציסט ביום 5/יום 6 ב 37 ° C, 5% CO 2, ו 5% O2.
- אפשרות 2: שטיפת עוברים ותרבית
הערה: אפשרות זו מיועדת למעבדות IVF המבצעות ויטריפיקציה ביום 5 אחר הצהריים או ביום 6.- מעבירים את העובר למיקרו-טיפות שחוממו מראש (37°C) בתרבית בינונית של 10-15 μL, ושוטפים בעדינות כל עובר באופן סדרתי ב-3 מיקרו-טיפות על ידי פיפטינג ביום החמישי.
- מעבירים כל עובר למיקרו-טיפה יחידה ייחודית שחוממה מראש (37°C) של מדיום תרבית לאיסוף דגימה. נפחה של טיפה אחת של מדיום תרבית אינו יכול לעלות על 15 מיקרוליטר.
- בצע תרבית עובר בלסטוציסט ביום 5/יום 6 ב 37 ° C ו 5% CO2.
- אפשרות 1: שטיפת עוברים ותרבית
- אוסף דוגמאות
- כוונן בעדינות את ה- ICM במרחק ניכר מנקודת היעד של קרן הלייזר, המתמקדת בצומת התא של הטרופקטודרם כדי ליצור חור קטן בטרופקטודרם כדי לשחרר את הנוזל מחלל הבלסטוקול. לאחר מכן עוברים עוברים לתמיסת הקפאה לשימור בהקפאה בהתאם לתהליך הקונבנציונלי.
- העבר מדיום תרבית מכל עובר מתורבת לתוך צינור PCR ללא RNase / DNase המכיל 5 μL של חיץ ליזה התא.
- לאסוף את אותה כמות של מדיום תרבית מבלי לשמש לתרבית עוברים כביקורת שלילית. יש להקפיא את כל הדגימות שנאספו מיד בחנקן נוזלי ולאחר מכן לאחסן בטמפרטורה של -80°C לאחר שנאספו עד לבדיקת NICS.
- בצע ויטריפיקציה כמתואר בפרוטוקול.
3. פרוטוקול 2: בניית הספרייה
- תרבות בינונית ליזה
- לדלל 1 μL של שליטה חיובית (10 ng gDNA אנושי) עם 199 μL של מדיום תרבית טרי. מערבבים היטב וצנטריפוגות את הצינור לזמן קצר (200 x גרם במשך 5 שניות).
- העבר 10 μL של יום 5-יום 6 מדיום תרבית בלסטוציסטה, שליטה חיובית מדוללת, ומדיום תרבית טרי לצינורות PCR חדשים של 0.2 מ"ל.
- הוסף 1 μL של תערובת אנזים MT לכל צינור PCR וערבב היטב על ידי פיפטינג וצנטריפוגה מיד במשך 2-3 שניות ב 200 x גרם.
- שים את שפופרת ה- PCR משלב 3.1.3 בתחנת הכנה לדוגמה של NICS שחוממה מראש והפעל את תוכנית הליזיס כדלקמן: 10 דקות ב- 75 °C; 4 דקות ב 95 ° C; להחזיק ב 22 ° C.
הערה: תחנת ההכנה לדוגמה דומה למכונת PCR רגילה.- לחץ על סמל Lysis כדי להיכנס למסך ההתקנה.
- בחר Tube עבור מצב בקרה; קלט 10 μL עבור נפח דגימה; בחר מופעל עבור בקרת Hotlid והזן 105 ° C עבור הטמפרטורה. בחר לא עבור השהיה בסג הראשון. לחץ על אישור כדי להמשיך.
- המתן עד שזמן השהייה יופיע --:-- :---, המציין את סוף התוכנית ולאחר מכן לחץ על עצור כדי לסיים את התוכנית.
- הפסק את התוכנית לאחר השלמת התהליך. המשך לשלב הבא באופן מיידי.
- הכנה טרום ספרייתית
- הפשירו את החיץ Pre-Lib ל-RT. ערבבו היטב על ידי פיפטינג וצנטריפוגה מיד במשך 2-3 שניות ב 200 x גרם.
- הכינו תערובת אב לתגובה קדם-ספרייתית באופן הבא: הוסיפו 2 μL של תערובת אנזימים Pre-Lib ל-60 μL של Pre-Lib Buffer, ערבבו את התגובה ביסודיות וצנטריפוגה קצרה.
- הוסף 60 μL של תערובת תגובה קדם-ספרייתית לכל דגימה בינונית שטופלה מראש מהשלב הקודם. מערבבים היטב על ידי פיפטינג וצנטריפוגה מיד במשך 2-3 שניות ב 200 x גרם.
- מקם את שפופרת ה-PCR משלב 3.2.3 בתחנת ההכנה לדוגמה והפעל את תוכנית קדם-ספריה באופן הבא: 95°C למשך 2 דקות; 12 מחזורים של 15 °C עבור 40 s, 22 °C עבור 40 s, 33 °C עבור 30 s, 65 °C עבור 30 s, 72 °C עבור 40 s, 95 °C עבור 10 s ו 63 °C עבור 10 שניות; והחזק בטמפרטורה של 4°C.
- לחץ על סמל Pre_Lib כדי להיכנס למסך ההתקנה.
- בחר Tube עבור מצב בקרה; קלט 70 μL עבור נפח מדגם; בחר מופעל עבור בקרת מכסה חם והזן 105 ° C עבור הטמפרטורה. בחר לא עבור השהיה בסג הראשון. לחץ על אישור כדי להמשיך.
- המתן עד שזמן השהייה יופיע --:-- :---, המציין את סוף התוכנית, ולחץ על עצור כדי לסיים את התוכנית.
- הפסק את התוכנית עם השלמת התהליך. המשך לשלב הבא באופן מיידי.
- הכנת הספרייה
- הפשירו את מאגר הספרייה ל-RT. ערבבו היטב על ידי פיפטינג וצנטריפוגה מיד במשך 2-3 שניות ב 200 x גרם.
- הכינו תערובת אב לתגובת הספרייה באופן הבא: הוסיפו 1.6 מיקרוליטר של תערובת אנזימי הספרייה ל-60 מיקרוליטר של מאגר הספרייה, ערבבו את התגובה ביסודיות וצנטריפוגה קצרה.
- הוסף 60 μL של תערובת תגובת ספרייה ו- 2 μL של פריימר ברקוד לכל מוצר קדם-ספרייה משלב 3.2.3. מערבבים היטב את התגובה וצנטריפוגה קצרה.
- מקם את שפופרת ה- PCR משלב 3.2.3 במחזור התרמי והפעל את תוכנית הכנת הספרייה באופן הבא: 94 ° C למשך 30 שניות; 17 מחזורים של 94 ° C עבור 25 שניות, 62 ° C עבור 30 שניות, ו 72 ° C עבור 45 שניות); ולאחר מכן להחזיק ב 4 °C (75 °F).
- לחץ על סמל Lib_Prep כדי להיכנס למסך ההתקנה.
- בחר Tube עבור מצב בקרה; קלט 130 μL עבור נפח מדגם; בחר מופעל עבור בקרת Hotlid והזן 105 ° C עבור הטמפרטורה המתאימה. בחר לא עבור השהיה בסג הראשון. לחץ על אישור כדי להמשיך.
- המתן עד שזמן השהייה יופיע --:-- :---, המציין את סוף התוכנית, ולחץ על עצור כדי לסיים את התוכנית.
- טיהור הספרייה
- הסר את Magbeads מהאחסון בטמפרטורה של 2-8°C למשך 20 דקות לפחות לפני שלב הטיהור. מערבבים ומערבבים את ה-Magbeads במשך 20 שניות. הוציאו מספיק חרוזים לשלב הטיהור לתוך צינור מיקרוצנטריפוגה חדש בנפח 1.5 מ"ל וחרוזים חמים ל-RT.
- הוסיפו 1x Magbeads לכל ספרייה. מערבבים על ידי פיטינג למעלה ולמטה ≥10 פעמים ודגרים ב-RT במשך 5 דקות.
הערה: לדוגמה, הוסף 100 μL של Magbeads ל- 100 μL של דוגמת ספרייה. - לאחר הדגירה, צנטריפוגו את הצינור לזמן קצר והניחו על מעמד מגנטי.
- המתן כ-5 דקות עד שהפתרון יתבהר. תוך שמירה על הצינור על המעמד המגנטי, בזהירות לשאוף את הפתרון ולהשליך.
- הוסף 200 μL של 80% אתנול מוכן טרי לצינור. דוגרים ב-RT במשך 30 שניות ומסירים בזהירות את הסופרנטנט. חזור על הפעולה פעם נוספת.
- הסר את האתנול לחלוטין ככל האפשר. יבשו באוויר את החרוזים על המעמד המגנטי למשך כ-5-10 דקות ב-RT.
- הסר את הצינור מהמעמד המגנטי, הוסף 17.5 מיקרוליטר של חיץ אלוציה, וסובב את הצינור כדי להשהות מחדש את החרוזים. צנטריפוגו את הצינור לזמן קצר ודגרו ב-RT למשך 5 דקות.
- הניחו את הצינור על המעמד המגנטי והמתינו עד שהתמיסה תתבהר. בזהירות להעביר 15 μL supernatant לצינור חדש.
- כימות הספרייה
- כמת ספריות מטוהרות באמצעות פלואורומטר בהתאם למדריך למשתמש של ערכות בדיקת dsDNA HS של קיוביט28. התשואה של הספריות נעה בין ~ 15 ל 300 ng.
- איגום ספריות
- השתמש ב- 10 ננוגרם מכל דגימת ספרייה לצורך איגום.
- רצף
- עיין במדריך למשתמש של רצף (15027617 v01)29.
- רצפי ספרייה מטוהרים של 50 bp בקצה אחד על הפלטפורמה הניבו כ-2 מיליון קריאות עבור כל דגימה, והומלץ על עומק רצף של 0.03 ×.
- ניתוח נתונים
- הזן את שם המשתמש והסיסמה בדף הכניסה
- לאחר הכניסה למערכת, לחץ על ניתוח ויופיע דף חדש. לחץ על צור שליחה תחת הכרטיסיה NICS-A. לאחר מכן, בחר NGS עבור הפלטפורמה, בחר תאגיד, בחר NICSInst עבור מגיב, הזן את פרטי הפרויקט בתיבה תחת מזהה פרויקט, הגדר את העדפות הניתוח והעלה את הקבצים. לאחר שכל קבצי הרצף הועלו בהצלחה, לחץ על שלח כדי להתחיל בניתוח (איור 3A).
- לחץ על הצג שליחות כדי להציג את רשימת הפרויקטים שנשלחו. לאחר השלמת הניתוח, מצב הפרויקט יהפוך ל'הושלם' ולחצן 'הצג' יופיע בשדה הדוח. לחץ על הלחצן Show כדי להציג את טבלת הסיכום של ניתוח NICS (איור 3B).
- לחץ על הלחצן יצא דוח כדי לשמור את הדוחות (איור 3C).
הערה: שלושה סוגי קבצים ייוצאו עבור כל ניתוח. קובץ גרפי הכולל את כל תרשימי וריאציית מספר ההעתקה (CNV) עבור כל כרומוזום וגנום שלם, אשר יאוחסן תחת תיקיית "גרף"; גיליון אלקטרוני המכיל את פרטי QC לדוגמה של הפעלת ניתוח זו; קובץ מסמך המכיל את דוחות NICS המותאמים אישית על ידי המשתמש; וגיליון אלקטרוני המכיל את פרטי הסיכום לדוגמה של הפעלת ניתוח זו.
תוצאות
המחקר הנוכחי יישם את השיטה המוצעת על מטופל. אישור IRB והסכמה מדעת התקבלו לפני יישום ניתוח NICS. המחקר הנוכחי השיג 6 בלסטוציסטים מחולים וביצע NICS על כל 6 העוברים ביום 4 עד יום 5 בינוני. הפרעות כרומוזומים שנגרמו על ידי טרנסלוקציה מאוזנת של ההורים התגלו בחמישה כרומוזומים עם בדיקת NICS; לכן, לא ניתן היה ...
Discussion
שינויים ופתרון בעיות
אם תוצאות NICS מזוהמות בחומרים גנטיים של ההורים, יש לוודא שכל תאי הקומולוס-קורונה רדיאטה מוסרים ולוודא כי ICSI מבוצע לצורך הפריה. נמנעים מאחסון בינוני לא תקין או מתהליכי הכנת תבניות, מה שעלול לפגוע בדנ"א. חלל העבודה טוהר ביסודיות עם ריאגנטים לטיהור...
Disclosures
Yaxin Yao, Jieliang Ma, Jing Wang ו- Sijia Lu הם עובדים של Yikon Genomics Co., Ltd.
Acknowledgements
המחברים רוצים להודות ל-Shiping Bo ול-Shujie Ma על עזרתם בניתוח נתוני NGS. מימון: עבודה זו נתמכה על ידי התוכנית הלאומית למחקר ופיתוח מפתח (מענק מס' 2018YFC1003100).
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
References
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T., Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid?. Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- . Qubit dsDNA HS Assay Kit Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015)
- . Miseq system use guide Available from: https://support.illumina.com/downloads/miseq_system (2016)
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Erratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021. Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
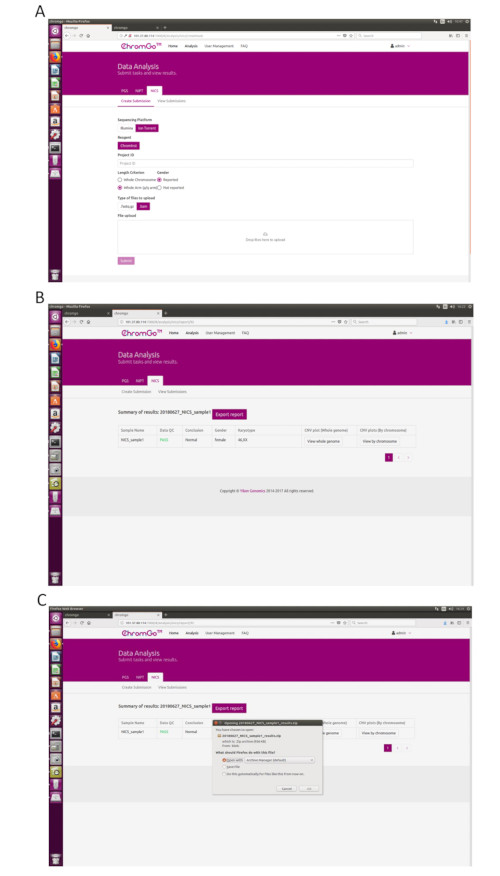
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
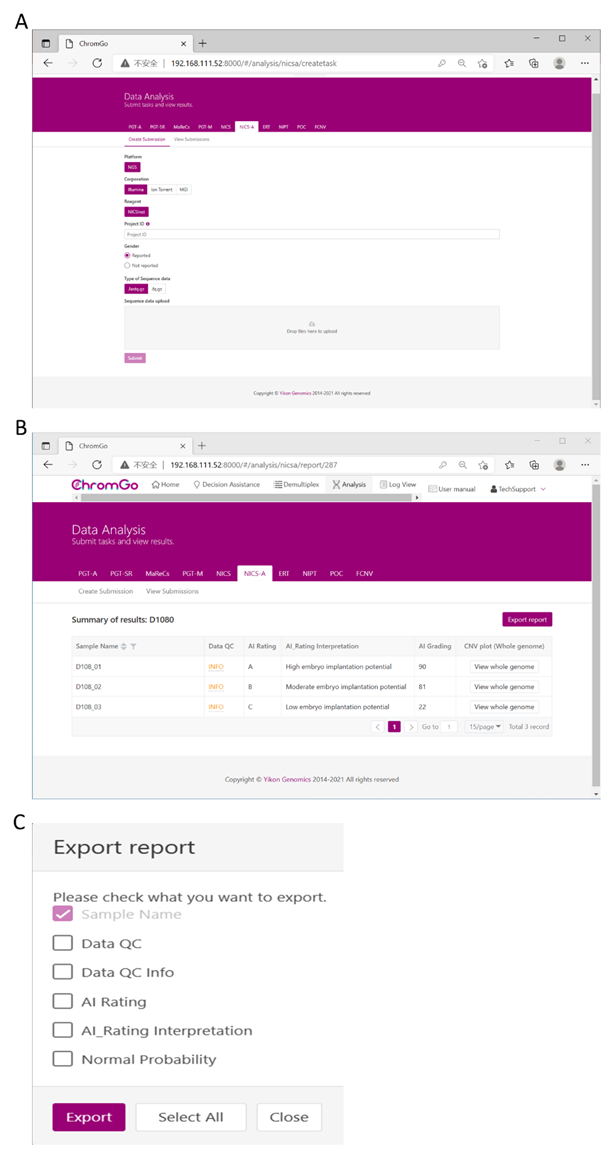
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved