Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Хромосомный скрининг преимплантационных эмбрионов человека с использованием отработанной питательной среды: забор образцов и анализ хромосомной плоидности
* Эти авторы внесли равный вклад
В этой статье
Erratum Notice
Резюме
В настоящем исследовании представлен протокол хромосомного скрининга эмбрионов человека, использующий отработанную питательную среду, что позволяет избежать биопсии эмбриона и позволяет идентифицировать хромосомную плоидию с помощью NGS. В настоящей статье подробно описана процедура, включающая подготовку питательной среды, полногеномную амплификацию (WGA), подготовку библиотеки секвенирования нового поколения (NGS) и анализ данных.
Аннотация
При клиническом экстракорпоральном оплодотворении (ЭКО) преобладающий метод ПГТ-А требует биопсии нескольких клеток трофэктодермы (ТЭ). Это линия, которая формирует плаценту. Этот метод, однако, требует специальных навыков, является инвазивным и страдает от ложноположительных и отрицательных результатов, потому что число хромосом в ТЭ и внутренняя клеточная масса (ВКМ), которая развивается в плод, не всегда совпадают. NICS, технология, требующая секвенирования ДНК, которая высвобождается в питательную среду как из TE, так и из ICM, может предложить выход из этих проблем, но ранее было показано, что она имеет ограниченную эффективность. В настоящем исследовании представлен полный протокол NICS, который включает в себя методы отбора проб питательных сред, полногеномную амплификацию (WGA) и подготовку библиотеки, а также анализ данных NGS с помощью аналитического программного обеспечения. Учитывая разное время криоконсервации в разных лабораториях эмбрионов, у эмбриологов есть два метода сбора питательной среды для эмбрионов, которые могут быть выбраны в соответствии с фактическими условиями лаборатории ЭКО.
Введение
Вспомогательные репродуктивные технологии (ВРТ) все чаще используются для лечения бесплодия. Тем не менее, показатели успешности ВРТ, таких как ЭКО, были ограничены, а частота потери беременности значительно выше, чем в нормальной популяции1. Основной причиной этих проблем являются хромосомные аномалии, которые обычно встречаются у преимплантационных эмбрионов человека2. ПГТ-А является эффективным методом скрининга эмбрионов на хромосомный баланс перед имплантацией 3,4. Некоторые исследования доказали, что ПГТ-А может снизить частоту абортов и улучшить частоту наступления беременности 5,6,7,8. Тем не менее, PGT-A требует сложных технических знаний, которые требуют специальной подготовки и опыта. Инвазивная процедура биопсии эмбриона также потенциально может привести к повреждению эмбрионов9. Исследования показали, что биопсия бластомеров может препятствовать последующему развитию, а количество биопсированных ТЭ может влиять на частоту имплантации10. Несмотря на то, что долгосрочная проблема биобезопасности биопсии эмбриона еще не была тщательно оценена на людях, исследования на животных показали ее негативное влияние на развитие эмбриона11,12,13.
В предыдущих сообщениях указывалось, что следовые количества материалов ДНК секретировались в питательной среде во время развития эмбриона, и были предприняты усилия по проведению комплексного хромосомного скрининга (CCS) с использованием отработанной питательной средыэмбрионов 14,15,16,17,18. Тем не менее, частота выявления и точность тестов не соответствовали требованиям для широкого клинического использования. В настоящем исследовании сообщалось об улучшении анализа NICS для повышения частоты обнаружения, а также точности теста NICS19. В последние годы бластоцельная жидкость (БФ) изучается в качестве аналитического образца малоинвазивной ПГТ-А. Однако доля успешной полногеномной амплификации и детектируемой ДНК в образцах жидкости бластоцисты колеблется от 34,8% до 82%20,21,22. Объем БФ, о котором сообщалось в различных исследованиях, колеблется от 0,3 нл до 1 мкл. Учитывая низкое количество ДНК в БФ, можно увеличить количество внеклеточной ДНК путем смешивания бластоцистной жидкости и питательной среды для улучшения успешности и последовательности обнаружения. Кузнецов и др.23 и Li et al.24 обрабатывали zona pellucida лазером и выпускали бластоцистную жидкость в питательную среду для улучшения общего количества эмбриональной ДНК, а скорость амплификации комбинированных образцов среды/БФ после WGA составила 100% и 97,5% соответственно. Jiao et al.25 также получили 100% успешность амплификации, используя тот же метод.
В настоящем исследовании представлен подробный протокол, включающий подготовку проб отработанных сред, подготовку NGS и анализ данных. Путем тщательного удаления кумулюсных клеток из ооцитов, в настоящем исследовании была проведена интрацитоплазматическая инъекция одного сперматозоида (ИКСИ) и культивирование бластоцисты. Для подготовки библиотек WGA и NGS был собран материал 4-го дня 5-го дня и 6-го дня. Используя технологию NICS, настоящее исследование оптимизировало этапы подготовки библиотек WGA и NGS примерно за 3 ч и получило результаты CCS неинвазивным способом примерно за 9 ч.
протокол
Этическое разрешение было получено от Комитета по этике Третьей больницы Пекинского университета.
1. Подготовка
ПРИМЕЧАНИЕ: Необходимые материалы и оборудование перечислены в таблице материалов.
- Реагентов
- Предварительно нагрейте и уравновесите (сбалансированные) 20-30 мкл среды гаметы/среды для оплодотворения и питательной среды на стадии расщепления/бластоцисты (покрытой минеральным маслом) и гиалуронидазы (в плотно закрытой пробирке) при температуре 37 °C, 5% CO 2 и 5% O2 в инкубаторе Tri-gas в течение ночи перед использованием.
- Гиалуронидазу разогреть до 37 °C на рабочей поверхности в вытяжном шкафу.
- Приготовьте буфер для витрификации и реагенты для отбора проб в соответствии с инструкциями производителя.
- Инструменты
- Подготовьте пипетки для отбора и переноса проб (внутренний диаметр ~200–250 мкм), пипетки для денудации/стриппинга (внутренний диаметр ≥150 мкм, ~130–140 мкм и ~120 мкм) и пипетки для промывки (внутренний диаметр ~150 мкм) путем вытягивания стеклянных пипеток Пастера для получения открытых тонких наконечников, отполированных огнем.
ПРИМЕЧАНИЕ: Пипетки, используемые для сбора/передачи проб, денудации и промывки, можно приобрести напрямую. Фиксирующие иглы и инъекционные иглы также можно приобрести напрямую.
- Подготовьте пипетки для отбора и переноса проб (внутренний диаметр ~200–250 мкм), пипетки для денудации/стриппинга (внутренний диаметр ≥150 мкм, ~130–140 мкм и ~120 мкм) и пипетки для промывки (внутренний диаметр ~150 мкм) путем вытягивания стеклянных пипеток Пастера для получения открытых тонких наконечников, отполированных огнем.
2. Протокол 1: Отбор проб
- Предварительная обработка комплекса ооцит-корона-кучевые облака (OCCCs) перед расщеплением гиалуронидазой
- Стимуляция яичников осуществляется как препаратами фолликулостимулирующего гормона (ФСГ), так и препаратами менопаузального гонадотропина человека (ХМГ). Когда ведущий фолликул составляет >18 мм, используйте 10 000 МЕ хорионического гонадотропина (ХГЧ) для окончательного созревания ооцитов.
- Забор ооцитов проводят через 36 ч после триггерного укола. Соберите и перенесите ооциты в чашки для культивирования тканей с 2,5 мл предварительно подогретого m-HTF, покрытого минеральным маслом.
- Быстро перенесите OCCC в центральную лунку чашки для культивирования органов, содержащую 1 мл среды для оплодотворения, с помощью пипетки для переноса, а затем инкубируйте с ооцитами при 37 °C в инкубаторе 5% CO 2 и 5% O 2 в течение2-4 ч.
- Расщепляют OCCC с гиалуронидазой, добавляя 1 мл предварительно подогретой гиалуронидазы 37 °C (80 МЕ/мл) в центральную лунку чашки для культивирования органов, содержащую OCCC (шаг 2.1.3). Конечную концентрацию гиалуронидазы поддерживать на уровне 40 МЕ/мл и тщательно перемешать.
- Инкубируйте OCCC на термической платформе с температурой 37 °C в течение 2 минут. Наблюдайте за изменениями под микроскопом каждые 30 с до тех пор, пока не останется только 1-2 слоя гранулезных клеток.
- Денудация гранулезных клеток
- Быстро перенесите переваренные OCCC в чашку для культивирования для обработки ооцитов и залейте минеральным маслом в каждую лунку.
- Понаблюдайте за отделенными гранулезными клетками под микроскопом. Осторожно аспирируйте и отпустите ооциты 5 раз, чтобы удалить остаточные гранулезные клетки вокруг ооцитов.
- Повторите предыдущий шаг в оставшихся 3 лунках, чтобы полностью удалить гранулезные клетки.
ПРИМЕЧАНИЕ: Вышеуказанные шаги (2.1-2.3) могут быть выполнены в соответствии с обычной работой каждой лаборатории.
- Оценка яйцеклетки
- Оцените полноту удаления гранулезных клеток с помощью микроскопа. Если клетки не удалось удалить полностью, то в настоящее время допустимо сохранение 5 или менее гранулезных клеток.
ПРИМЕЧАНИЕ: Если кумулюсные клетки все еще прикреплены к ооциту, остаток может быть удален позже на 3-й день, прежде чем эмбрион будет перенесен из питательной среды стадии расщепления в питательную среду стадии бластоцисты.
- Оцените полноту удаления гранулезных клеток с помощью микроскопа. Если клетки не удалось удалить полностью, то в настоящее время допустимо сохранение 5 или менее гранулезных клеток.
- После проведения интрацитоплазматической инъекции сперматозоида (ИКСИ)26 переносят ооциты в микрокапли культуры расщепления эмбрионов объемом 20-30 мкл (один ооцит соответствует одной микрокапле) с помощью трансферных пипеток и инкубируют в инкубаторе с температурой 37 °C, 5% CO 2 и 5% O2.
- Запишите день ИКСИ как день 0. Проверьте эмбрионы и наберите баллы в соответствии со Стамбульским консенсусным семинаром по оценке эмбрионов на 1-й день для оплодотворения (примерно 18 ч), 2-й день (примерно 45 ч) и 3-й день (примерно 68 ч) для расщепления эмбрионов27.
- Промывание эмбрионов
- На 2-й день в инкубаторе с температурой 37 °C, 5%CO2 и 5% O2 , для каждого эмбриона, покрытого минеральным маслом, в чашках для культивирования тканей подготовить 20-30 мкл среды для культивирования бластоцисты.
- Подготовьте еще три микрокапли, покрытые минеральным маслом, и пометьте новые посуды для культуры тканей для мытья No 1-3.
- Перенесите на 3 день эмбрионы в промывочные микрокапли. Осторожно аспирируйте и выпускайте эмбрионы 3 раза в каждой капле с помощью денудационных пипеток.
ПРИМЕЧАНИЕ: Эта процедура также может помочь удалить остаточные гранулярные клетки, прикрепленные к эмбриону. - Наблюдайте и оценивайте эмбрионы под микроскопом на 3-й день до того, как среда была изменена с питательной среды стадии расщепления на питательную среду бластоцисты для морфологической оценки. Если кумулюсные клетки все еще прикреплены к эмбриону, соответствующим образом пипетка вверх и вниз в другой предварительно разогретой и уравновешенной средней капле культуры бластоцисты, покрытой минеральным маслом, с помощью стриппер-пипетки до тех пор, пока кучевые клетки не будут полностью удалены.
ПРИМЕЧАНИЕ: Все прикрепленные кумулюсные клетки должны были быть полностью удалены на 3-й день, прежде чем эмбрион был перенесен из питательной среды стадии расщепления в культуральную пластину стадии бластоцисты. Любые оставшиеся кумулюсные клетки будут вмешиваться в окончательный анализ и давать ложноотрицательные результаты.
- Два варианта сбора питательных сред
ПРИМЕЧАНИЕ: Центр ЭКО может выбрать один из двух методов сбора питательной среды в зависимости от ресурсов, потребностей и предпочтений центра.- Вариант 1: Промывка эмбрионов и культивирование
ПРИМЕЧАНИЕ: Этот вариант предназначен для лабораторий ЭКО, которые проводят витрификацию утром на 5-й день.- Пересадить эмбрион в предварительно подогретые (37 °C) микрокапли питательной среды и аккуратно промыть каждый эмбрион поочередно в 3 микрокаплях пипетированием на 4-й день после полудня.
- Пересадить каждый эмбрион в уникальную предварительно подогретую (37 °C) одну микрокаплю питательной среды для забора образца. Объем одной капли питательной среды не может превышать 25 мкл.
- Проводят культивирование эмбриона бластоцисты на 5-й день/6-й день при 37 °C, 5% CO2 и 5%O2.
- Вариант 2: Промывка эмбрионов и культивирование
ПРИМЕЧАНИЕ: Этот вариант предназначен для лабораторий ЭКО, которые проводят витрификацию на 5-й день после обеда или на 6-й день.- Переносят эмбрион в предварительно подогретые (37 °С) микрокапли питательной среды по 10-15 мкл и осторожно промывают каждый эмбрион поочередно в 3 микрокаплях пипетированием на 5-й день.
- Пересадить каждый эмбрион в уникальную предварительно подогретую (37 °C) одну микрокаплю питательной среды для забора образца. Объем одной капли питательной среды не может превышать 15 мкл.
- Проводят культивирование эмбриона бластоцисты на 5-й день/6-й день при 37 °C и 5% CO2.
- Вариант 1: Промывка эмбрионов и культивирование
- Сбор образцов
- Осторожно отрегулируйте ICM на значительном расстоянии от целевой точки лазерного луча, который фокусируется на клеточном соединении трофэктодермы, чтобы создать небольшое отверстие в трофэктодерме для высвобождения жидкости из полости бластоцеля. Затем эмбрионы перемещаются в замораживающий раствор для криоконсервации в соответствии с традиционным процессом.
- Перенос питательной среды с каждого культивируемого эмбриона в ПЦР-пробирку без РНКазы/ДНКазы, содержащую 5 мкл буфера для лизиса клеток.
- Соберите такое же количество питательной среды, не используя ее для культивирования эмбрионов в качестве отрицательного контроля. Все собранные образцы немедленно замораживают в жидком азоте, а затем хранят при температуре −80 °C после сбора до проведения анализа NICS.
- Выполните витрификацию, как описано в протоколе.
3. Протокол 2: Строительство библиотек
- Лизис питательной среды
- Разбавляют 1 мкл положительного контроля (10 нг гДНК человека) 199 мкл свежей питательной среды. Тщательно перемешайте и быстро центрифугируйте пробирку (200 x g в течение 5 с).
- Пересадить 10 мкл 5-дневной 6-дневной питательной среды бластоцисты, разбавленной положительной контрольной и свежей питательной среды в новые пробирки для ПЦР по 0,2 мл.
- Добавьте 1 мкл смеси ферментов МТ в каждую пробирку для ПЦР и тщательно перемешайте с помощью пипетирования и центрифуги в течение 2-3 с при 200 x g.
- Поместите пробирку (пробирки) для ПЦР из шага 3.1.3 в предварительно нагретую станцию подготовки образцов NICS и запустите программу лизиса следующим образом: 10 мин при 75 °C; 4 мин при 95 °C; держать при 22 °C.
ПРИМЕЧАНИЕ: Станция подготовки образцов сравнима со стандартным ПЦР-аппаратом.- Щелкните значок Lysis , чтобы перейти на экран настройки.
- Выберите Tube (Трубка) для параметра Control mode (Режим управления); вход 10 мкл для объема образца; выберите Вкл для управления Hotlid и введите 105 °C в качестве температуры. Выберите «Нет» для паузы на первом шаге. Нажмите кнопку ОК, чтобы продолжить.
- Подождите, пока в поле Время пребывания не отобразится -- :--:-, указывающее на окончание программы, а затем нажмите кнопку Остановить , чтобы завершить программу.
- Остановите программу после завершения процесса. Немедленно переходите к следующему шагу.
- Предбиблиотечная подготовка
- Разморозьте буфер Pre-Lib до RT. Тщательно перемешайте пипеткой и немедленно центрифугируйте в течение 2-3 с при 200 x g.
- Приготовьте мастер-смесь для пребиблиотечной реакции следующим образом: добавьте 2 мкл смеси ферментов Pre-Lib к 60 мкл буфера Pre-Lib, тщательно перемешайте реакцию и кратковременно центрифугируйте.
- Добавьте 60 мкл пребиблиотечной реакционной смеси в каждый предварительно обработанный образец среды, полученный на предыдущем этапе. Тщательно перемешать пипеткой и сразу же центрифугировать в течение 2-3 с при 200 х г.
- Поместите пробирки для ПЦР, показанные на шаге 3.2.3, в станцию подготовки образцов и запустите программу предварительной библиотеки следующим образом: 95 °C в течение 2 мин; 12 циклов по 15 °C в течение 40 с, 22 °C в течение 40 с, 33 °C в течение 30 с, 65 °C в течение 30 с, 72 °C в течение 40 с, 95 °C в течение 10 с и 63 °C в течение 10 с; и держать при температуре 4 °C.
- Щелкните значок Pre_Lib , чтобы перейти на экран настройки.
- Выберите Tube (Трубка) для параметра Control mode (Режим управления); вход 70 мкл для объема образца; выберите On (Вкл.) для параметра Hot lid control (Управление горячей крышкой) и введите 105 °C в качестве температуры. Выберите «Нет» для паузы на первом шаге. Нажмите кнопку ОК, чтобы продолжить.
- Подождите, пока в поле Время пребывания не отобразится -- :--:-, указывающее на окончание программы, и нажмите кнопку Остановить, чтобы завершить программу.
- Остановите программу после завершения процесса. Немедленно переходите к следующему шагу.
- Подготовка библиотеки
- Разморозьте библиотечный буфер до RT. Тщательно перемешайте пипеткой и немедленно центрифугируйте в течение 2-3 с при 200 x g.
- Приготовьте мастер-смесь для библиотечной реакции следующим образом: добавьте 1,6 мкл библиотечной ферментной смеси к 60 мкл библиотечного буфера, тщательно перемешайте реакцию и быстро центрифугируйте.
- Добавьте 60 мкл библиотечной реакционной смеси и 2 мкл праймера штрих-кода к каждому предбиотечному продукту, начиная с шага 3.2.3. Тщательно перемешайте реакцию и ненадолго центрифугируйте.
- Поместите пробирку (пробирки) для ПЦР из шага 3.2.3 в амплификатор и запустите программу подготовки библиотеки следующим образом: 94 °C в течение 30 с; 17 циклов 94 °C в течение 25 с, 62 °C в течение 30 с и 72 °C в течение 45 с); а затем выдержать при температуре 4 °C.
- Щелкните значок Lib_Prep , чтобы перейти на экран настройки.
- Выберите Tube (Трубка) для параметра Control mode (Режим управления); вход 130 мкл для объема образца; выберите Вкл для управления Hotlid и введите 105 °C для соответствующей температуры. Выберите «Нет» для паузы на первом шаге. Нажмите кнопку ОК, чтобы продолжить.
- Подождите, пока в поле Время пребывания не отобразится -- :--:-, указывающее на окончание программы, и нажмите кнопку Остановить, чтобы завершить программу.
- Очистка библиотеки
- Выньте Magbeads из хранилища при температуре 2-8 °C не менее чем за 20 минут до этапа очистки. Перемешайте и перемешайте Magbeads в течение 20 с. Выдавите достаточное количество гранул для этапа очистки в новую микроцентрифужную пробирку объемом 1,5 мл и нагрейте гранулы до RT.
- Добавьте 1 Магбусину в каждую библиотеку. Перемешайте пипеткой вверх и вниз ≥10 раз и инкубируйте при RT в течение 5 мин.
ПРИМЕЧАНИЕ: Например, добавьте 100 мкл магнитных шариков к 100 мкл библиотечного образца. - После инкубации ненадолго центрифугируйте пробирку и поместите на магнитную подставку.
- Подождите примерно 5 минут, пока раствор не станет прозрачным. Держа пробирку на магнитной подставке, осторожно аспирируйте раствор и выбросьте.
- Добавьте в пробирку 200 мкл свежеприготовленного 80% этанола. Инкубируют при РТ в течение 30 с и осторожно удаляют надосадочную жидкость. Повторите еще раз.
- Удалите этанол как можно полнее. Высушите шарики на воздухе на магнитной подставке в течение примерно 5-10 минут при RT.
- Снимите пробирку с магнитной подставки, добавьте 17,5 мкл элюирующего буфера и вкрутите трубку, чтобы снова взвесить шарики. Кратковременно центрифугируют пробирку и инкубируют при RT в течение 5 мин.
- Поместите пробирку на магнитную подставку и подождите, пока раствор не станет прозрачным. Осторожно перелейте 15 мкл надосадочной жидкости в новую пробирку.
- Количественная оценка библиотеки
- Количественная оценка очищенных библиотек с помощью флуориметра в соответствии с руководством пользователя наборов для анализа кубитов dsDNA HS28. Ресурс библиотек колеблется от ~15 до 300 нг.
- Объединение библиотек в пул
- Используйте 10 нанограммов каждого библиотечного образца для объединения.
- Секвенирование
- Обратитесь к руководству пользователя по виртуализации (15027617 v01)29.
- Очищенные библиотечные последовательности 50.н. на одном конце платформы давали около 2 миллионов прочтений для каждого образца, и было рекомендовано 0,03 × глубины секвенирования.
- Анализ данных
- Введите имя и пароль пользователя на странице входа
- После входа в систему нажмите «Анализ » и появится новая страница. Нажмите кнопку Create Submission (Создать отправку ) на вкладке NICS-A. Затем выберите NGS для платформы, выберите корпорацию, выберите NICSInst для реагента, введите информацию о проекте в поле под идентификатором проекта, задайте параметры анализа и загрузите файлы. После того, как все файлы секвенирования будут успешно загружены, нажмите кнопку Submit (Отправить ), чтобы начать анализ (рисунок 3A).
- Нажмите Просмотреть отправленные проекты, чтобы отобразить список отправленных проектов. После завершения анализа статус проекта изменится на Завершен, а в поле отчета появится кнопка Показать. Нажмите кнопку Показать, чтобы просмотреть сводную таблицу анализа NICS (рисунок 3B).
- Нажмите кнопку Export report , чтобы сохранить отчеты (рисунок 3C).
ПРИМЕЧАНИЕ: Для каждого анализа будут экспортированы три типа файлов. Графический файл, включающий в себя все графики вариаций числа копий (CNV) для каждой хромосомы и всего генома, который будет храниться в папке «graph»; электронную таблицу, содержащую примеры сведений о контроле качества этого выполнения анализа; файл документа, содержащий настраиваемые пользователем отчеты NICS; и электронную таблицу, содержащую образец сводной информации этого выполнения анализа.
Результаты
В настоящем исследовании предложенный метод применялся к пациенту. Одобрение IRB и информированное согласие были получены до применения анализа NICS. В настоящем исследовании было получено 6 бластоцист от пациенток и проведена NICS на всех 6 эмбрионах на 4-5 сутки. Хромосомные аномалии, вызва...
Обсуждение
Модификации и устранение неполадок
Если результаты NICS загрязнены родительским генетическим материалом, убедитесь, что все лучевые клетки кучевой короны удалены, и убедитесь, что для оплодотворения проводится ИКСИ. Это позволяет избежать неправильного хранени...
Раскрытие информации
Ясинь Яо, Цзелян Ма, Цзин Ван и Сицзя Лу являются сотрудниками компании Yikon Genomics Co., Ltd.
Благодарности
Авторы выражают благодарность Шипин Бо (Shiping Bo) и Шуцзе Ма (Shujie Ma) за помощь в анализе данных NGS. Финансирование: работа выполнена при поддержке Национальной программы ключевых исследований и разработок (грант No 2018YFC1003100).
Материалы
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
Ссылки
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T., Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid?. Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- . Qubit dsDNA HS Assay Kit Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015)
- . Miseq system use guide Available from: https://support.illumina.com/downloads/miseq_system (2016)
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Erratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021. Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
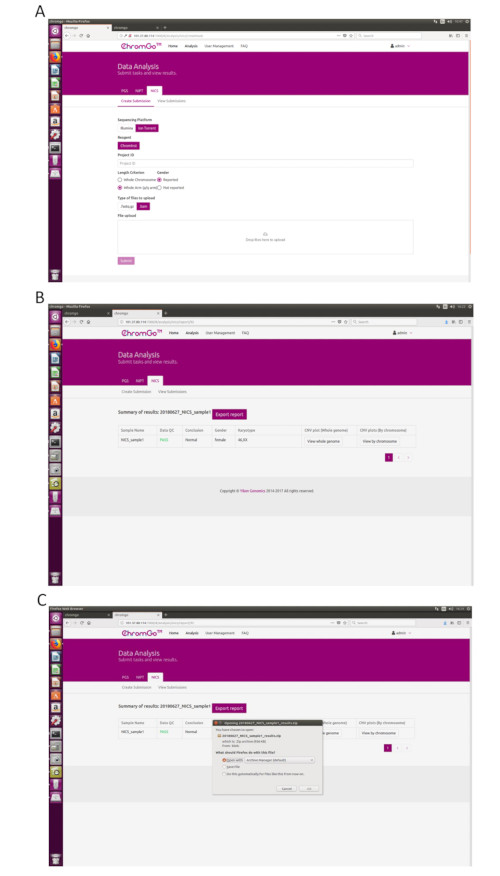
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
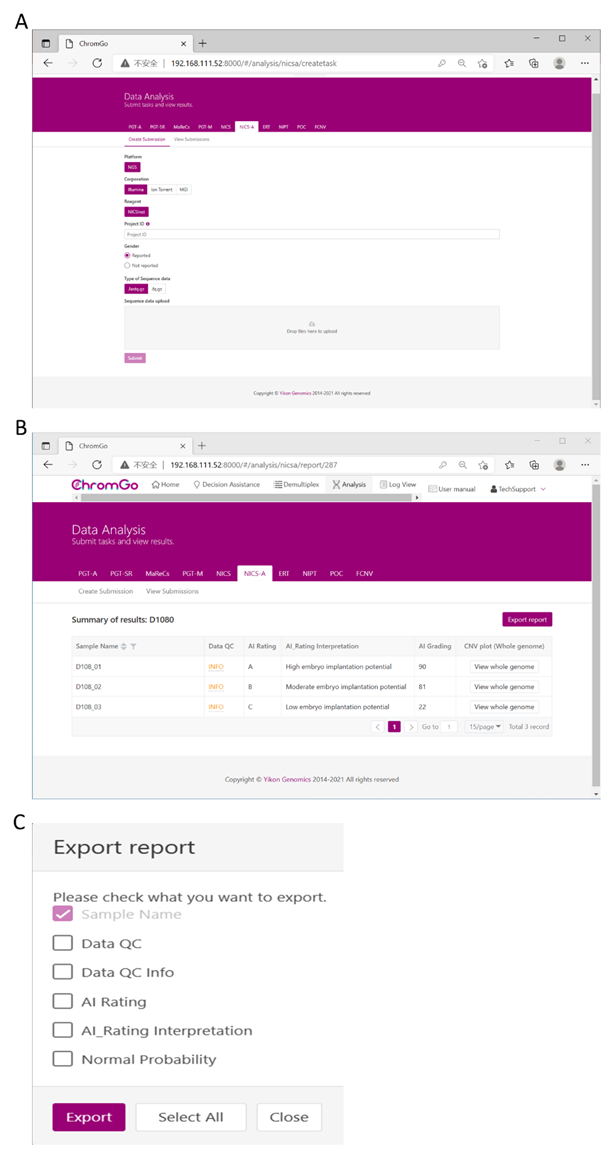
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены