É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Triagem Cromossômica de Embriões Humanos Pré-implantacionais Utilizando Meio de Cultura Usado: Coleta de Amostras e Análise de Ploidia Cromossômica
* Estes autores contribuíram igualmente
Neste Artigo
Erratum Notice
Resumo
O presente estudo relata um protocolo de triagem cromossômica de embriões humanos que utiliza meio de cultura usado, o que evita a biópsia embrionária e permite a identificação de ploidia cromossômica usando NGS. O presente artigo apresenta o procedimento detalhado, incluindo a preparação do meio de cultura, amplificação do genoma completo (WGA), preparação da biblioteca de sequenciamento de próxima geração (NGS) e análise dos dados.
Resumo
Na fertilização clínica in vitro (FIV), o método predominante para PGT-A requer biópsia de algumas células do trofectoderma (TE). Esta é a linhagem que forma a placenta. Esse método, no entanto, requer habilidades especializadas, é invasivo e sofre de falsos positivos e negativos porque os números cromossômicos no ET e a massa celular interna (MCI), que se desenvolve no feto, nem sempre são os mesmos. O NICS, uma tecnologia que requer o sequenciamento do DNA liberado no meio de cultura tanto do TE quanto do ICM, pode oferecer uma saída para esses problemas, mas já demonstrou ter eficácia limitada. O presente estudo relata o protocolo completo do NICS, que inclui métodos de amostragem em meio de cultura, amplificação do genoma completo (WGA) e preparação de bibliotecas, e análise de dados NGS por software de análise. Considerando os diferentes tempos de criopreservação em diferentes laboratórios de embriões, os embriologistas possuem dois métodos de coleta de meio de cultura embrionário que podem ser selecionados de acordo com as condições reais do laboratório de FIV.
Introdução
As tecnologias de reprodução assistida (TARA) têm sido cada vez mais utilizadas para o tratamento da infertilidade. No entanto, a taxa de sucesso da TARV, como a FIV, tem sido limitada, e a taxa de perda gestacional é significativamente maior do que a da população normal1. A principal causa desses problemas são as anormalidades cromossômicas, comumente existentes em embriões humanos pré-implantacionais2. A PGT-A é um método eficaz de triagem de embriões para equilíbrio cromossômico antes da implantação 3,4. Alguns estudos comprovaram que a PGT-A pode reduzir a taxa de aborto e melhorar a taxa de gravidez 5,6,7,8. No entanto, a PGT-A requer conhecimentos técnicos complexos que exigem treinamento e experiência específicos. O procedimento de biópsia invasiva de embriões também poderia potencialmente causar danos aos embriões9. Estudos têm demonstrado que a biópsia de blastômeros pode dificultar o desenvolvimento subsequente, e o número de TEs biopsiados pode afetar as taxas de implante10. Embora a questão da biossegurança a longo prazo da biópsia embrionária ainda não tenha sido completamente avaliada em humanos, estudos em animais têm mostrado suas influências negativas no desenvolvimento embrionário11,12,13.
Relatos anteriores indicaram que traços de materiais de DNA foram secretados para o meio de cultura durante o desenvolvimento embrionário, e esforços têm sido feitos para realizar triagem cromossômica abrangente (CCS) usando meio de cultura embrionário usado 14,15,16,17,18. No entanto, as taxas de detecção e a acurácia dos testes não atenderam aos requisitos para uso clínico extensivo. O presente estudo relatou uma melhora no ensaio NICS para aumentar as taxas de detecção, bem como a acurácia do teste NICS19. Nos últimos anos, o líquido blastocelêmico (BF) tem sido estudado como uma amostra analítica de PGT-A minimamente invasiva. No entanto, a proporção de sucesso na amplificação do genoma amplo e no DNA detectável em amostras de fluido de blastocisto varia de 34,8% a 82%20,21,22. O volume de AM relatado em vários estudos varia de 0,3 nL a 1 μL. Tendo em vista a baixa quantidade de DNA no FS, é possível aumentar a quantidade de DNA livre de células misturando fluido de blastocisto e meio de cultura para melhorar a taxa de sucesso e a consistência da detecção. Kuznyetsov et al.23 e Li et al.24 trataram a zona pelúcida com laser e liberaram líquido blastocisto no meio de cultura para melhorar a quantidade total de DNA embrionário, e a taxa de amplificação das amostras combinadas meio/FS após WGA foi de 100% e 97,5%, respectivamente. Jiao et al.25 também obtiveram 100% de sucesso na amplificação utilizando o mesmo método.
O presente estudo relata um protocolo detalhado que inclui a preparação de amostras de mídia gasta, preparação de NGS e análise de dados. Ao remover cuidadosamente as células do cumulus dos ovócitos, o presente estudo realizou injeção intracitoplasmática de espermatozoides únicos (ICSI) e cultura de blastocisto. O meio gasto do dia 4 ao dia 5/dia 6 foi coletado para a preparação da biblioteca WGA e NGS. Usando a tecnologia NICS, o presente estudo simplificou as etapas de preparação da biblioteca WGA e NGS em aproximadamente 3 h e obteve resultados de ECC de forma não invasiva em aproximadamente 9 h.
Protocolo
A permissão ética foi obtida do Comitê de Ética do Terceiro Hospital da Universidade de Pequim.
1. Preparo
NOTA: Os materiais e equipamentos necessários estão listados na Tabela de Materiais.
- Reagentes
- Pré-aquecer e equilibrar (balanceado) 20-30 μL de meio gameta/meio de fertilização e meio de cultura de clivagem/blastocisto (coberto com óleo mineral) e hialuronidase (em um tubo bem tampado) a 37 °C, 5% CO 2 e 5% O2 em uma incubadora Tri-gás durante a noite antes do uso.
- Pré-aqueça a hialuronidase a 37 °C numa superfície de trabalho num exaustor.
- Preparar tampão de vitrificação e reagentes de coleta de amostras de acordo com as instruções do fabricante.
- Ferramentas
- Prepare pipetas de coleta e transferência de amostras (diâmetro interno de ~200 a 250 μm), pipetas de desnudamento/stripper (diâmetro interno de ≥150 μm, ~130-140 μm e ~120 μm) e pipetas para lavagem (diâmetro interno de ~150 μm) puxando pipetas de vidro Pasteur para gerar pontas finas abertas polidas a fogo.
NOTA: As pipetas utilizadas para coleta/transferência de amostras, desnudamento e lavagem podem ser adquiridas diretamente. As agulhas de retenção e as agulhas de injeção também podem ser compradas diretamente.
- Prepare pipetas de coleta e transferência de amostras (diâmetro interno de ~200 a 250 μm), pipetas de desnudamento/stripper (diâmetro interno de ≥150 μm, ~130-140 μm e ~120 μm) e pipetas para lavagem (diâmetro interno de ~150 μm) puxando pipetas de vidro Pasteur para gerar pontas finas abertas polidas a fogo.
2. Protocolo 1: Coleta de amostras
- Pré-tratamento do complexo oócito-corona-cumulus (CCOC) antes da digestão com hialuronidase
- Conseguir a estimulação ovariana com preparações de hormônio folículo estimulante (FSH) e gonadotrofina menopausal humana (hMG). Quando o folículo condutor estiver >18 mm, use 10.000 UI de gonadotrofina coriônica (hCG) para maturação final do ovócito.
- Realizar a recuperação do ovócito 36 h após o disparo do gatilho. Captar e transferir ovócitos para placas de cultura de tecidos com 2,5 mL de m-HTF pré-aquecido coberto por óleo mineral.
- Transferir rapidamente os CCOCs para o poço central de uma placa de cultura de órgãos contendo 1 mL de meio de fertilização usando uma pipeta de transferência e, em seguida, incubar com os ovócitos a 37 °C em uma estufa de 5% CO 2 e 5% O 2 por2-4 h.
- Digerir OCCCs com hialuronidase adicionando 1 mL de hialuronidase pré-aquecida a 37 °C (80 UI/mL) ao poço central de uma placa de cultura de órgãos contendo OCCC (etapa 2.1.3). Manter a concentração final de hialuronidase a 40 UI/ml e misturar bem.
- Incubar os CCOC numa plataforma térmica a 37 °C durante 2 minutos. Observe as mudanças ao microscópio a cada 30 s até restarem apenas 1-2 camadas de células da granulosa.
- Denudação de células da granulosa
- Transfira rapidamente os OCCCs digeridos na placa de cultura para manuseio de ovócitos e cubra com óleo mineral em cada poço.
- Observe as células da granulosa separadas ao microscópio. Aspirar suavemente e liberar os ovócitos 5 vezes para remover as células residuais da granulosa ao redor dos ovócitos.
- Repita o passo anterior nos 3 poços restantes para remover completamente as células da granulosa.
NOTA: As etapas acima (2.1-2.3) podem ser realizadas de acordo com a operação de rotina de cada laboratório.
- Avaliação do ovócito
- Avaliar a completude da remoção de células da granulosa utilizando um microscópio. Se as células não puderam ser completamente removidas, então a retenção de 5 ou menos células da granulosa é aceitável neste momento.
NOTA: Se as células do cumulus ainda estiverem aderidas ao ovócito, o remanescente pode ser removido mais tarde no dia 3 antes que o embrião seja transferido do meio de cultura em estágio de clivagem para o meio de cultura em estágio de blastocisto.
- Avaliar a completude da remoção de células da granulosa utilizando um microscópio. Se as células não puderam ser completamente removidas, então a retenção de 5 ou menos células da granulosa é aceitável neste momento.
- Após a realização da injeção intracitoplasmática de espermatozoides (ICSI)26, transferir os ovócitos para microgotículas de meio de cultura embrionário de clivagem de 20-30 μL (um ovócito corresponde a uma microgota) usando pipetas de transferência e incubar em estufa a 37 °C, 5% CO 2 e 5% O2.
- Registre o dia da ICSI como dia 0. Verifique os embriões e a pontuação de acordo com o workshop de consenso de Istambul sobre avaliação embrionária do dia 1 para fertilização (aproximadamente 18 h), dia 2 (aproximadamente 45 h) e dia 3 (aproximadamente 68 h) para clivagem embrionária27.
- Lavagem de embriões
- Preparar 20-30 μL de microgotículas de meio de cultura de blastocisto para cada embrião coberto com óleo mineral em placas de cultura de tecidos no dia 2 em estufa a 37 °C, 5% CO 2 e 5% O2.
- Prepare mais três microgotículas cobertas com óleo mineral e rotule as novas placas de cultura de tecidos para lavagem nº 1-3.
- Transfira os embriões do dia 3 para as microgotículas de lavagem. Aspirar suavemente e liberar os embriões 3 vezes em cada gota usando pipetas de desnudamento.
NOTA: Este procedimento também pode ajudar a remover as células granulares residuais ligadas ao embrião. - Observar e avaliar os embriões ao microscópio no 3º dia antes da troca do meio de cultura em estágio de clivagem para o meio de cultura de blastocisto para escore morfológico. Se as células do cumulus ainda estivessem aderidas ao embrião, pipetar apropriadamente para cima e para baixo em outra gota de meio de cultura de blastocisto pré-aquecido e equilibrado coberta com óleo mineral com uma pipeta stripper até que as células do cumulus fossem completamente removidas.
NOTA: Todas as células do cumulus anexadas tiveram que ser removidas completamente no dia 3 antes que o embrião fosse transferido da placa do meio de cultura em estágio de clivagem para a placa do meio de cultura em estágio de blastocisto. Quaisquer células cumulus remanescentes interferirão na análise final e darão resultados falsos negativos.
- Duas opções de coleta de meio de cultura
NOTA: O centro de fertilização in vitro pode escolher um dos dois métodos para coleta de mídia de cultura com base nos recursos, demandas e preferências do centro.- Opção 1: Lavagem e cultura de embriões
NOTA: Esta opção é para laboratórios de fertilização in vitro que realizam vitrificação na manhã do dia 5.- Transferir o embrião para microgotículas pré-aquecidas (37 °C) de meio de cultura e lavar suavemente cada embrião em série em 3 microgotículas por pipetagem no dia 4 à tarde.
- Transferir cada embrião para uma única microgota pré-aquecida (37 °C) de meio de cultura para coleta de amostras. O volume de uma única gota de meio de cultura não pode exceder 25 μL.
- Realizar cultura de embriões de blastocisto no dia 5/dia 6 a 37 °C, 5% CO 2 e 5% O2.
- Opção 2: Lavagem e cultura de embriões
NOTA: Esta opção é para laboratórios de fertilização in vitro que realizam vitrificação no dia 5 à tarde ou dia 6.- Transferir o embrião para microgotículas pré-aquecidas (37 °C) de 10-15 μL de meio de cultura e lavar suavemente cada embrião em série em 3 microgotículas por pipetagem no dia 5.
- Transferir cada embrião para uma única microgota pré-aquecida (37 °C) de meio de cultura para coleta de amostras. O volume de uma única gota de meio de cultura não pode exceder 15 μL.
- Realizar cultura embrionária de blastocisto no dia 5/dia 6 a 37 °C e 5% CO2.
- Opção 1: Lavagem e cultura de embriões
- Coleta de amostras
- Ajuste suavemente o MCI a uma distância considerável do ponto alvo do feixe de laser, que se concentra na junção celular do trofectoderma para gerar um pequeno orifício no trofectoderma para liberar o fluido da cavidade do blastocoel. Em seguida, os embriões são movidos para solução de congelamento para criopreservação de acordo com o processo convencional.
- Transferir o meio de cultura de cada embrião cultivado para um tubo de PCR livre de RNase/DNase contendo 5 μL de tampão de lise celular.
- Coletar a mesma quantidade de meio de cultura sem ser utilizado para cultura de embriões como controle negativo. Congelar imediatamente todas as amostras coletadas em nitrogênio líquido e, em seguida, armazenar a -80 °C após serem coletadas até serem submetidas ao ensaio NICS.
- Realizar vitrificação conforme descrito no protocolo.
3. Protocolo 2: Construção de bibliotecas
- Lise do meio de cultura
- Diluir 1 μL de controle positivo (10 ng de gDNA humano) com 199 μL de meio de cultura fresco. Misture bem e centrifugar o tubo brevemente (200 x g por 5 s).
- Transferir 10 μL do meio de cultura de blastocisto dia 5 dias 6, controle positivo diluído e meio de cultura fresco para novos tubos de PCR de 0,2 mL.
- Adicionar 1 μL de MT Enzyme Mix a cada tubo de PCR e misturar cuidadosamente por pipetagem e centrifugação imediatamente por 2-3 s a 200 x g.
- Coloque o(s) tubo(s) de PCR da etapa 3.1.3 em uma estação de preparação de amostra NICS pré-aquecida e execute o programa de lise da seguinte forma: 10 min a 75 °C; 4 min a 95 °C; manter a 22 °C.
NOTA: A estação de preparação de amostras é comparável a uma máquina de PCR padrão.- Clique no ícone Lysis para entrar na tela de configuração.
- Selecione Tubo para o modo de controle; entrada de 10 μL para o volume da amostra; selecione Ativado para controle Hotlid e insira 105 °C para a temperatura. Selecione Não para Pausar no primeiro seg. Clique em OK para continuar.
- Aguarde até que Remain time mostre --:-- :--, que indica o final do programa e, em seguida, clique em Parar para encerrar o programa.
- Pare o programa após a conclusão do processo. Prossiga para a próxima etapa imediatamente.
- Preparação Pré-Biblioteca
- Descongele o buffer pré-lib para RT. Misture completamente pipetando e centrifugando imediatamente por 2-3 s a 200 x g.
- Prepare uma mistura mestra para a reação pré-biblioteca da seguinte forma: adicione 2 μL de Pre-Lib Enzyme Mix a 60 μL de Pre-Lib Buffer, misture a reação completamente e centrifugue brevemente.
- Adicionar 60 μL de mistura de reacção pré-biblioteca em cada amostra de meio pré-tratado do passo anterior. Misture bem por pipetagem e centrifugação imediatamente por 2-3 s a 200 x g.
- Coloque o(s) tubo(s) de PCR da etapa 3.2.3 na Estação de Preparação de Amostra e execute o programa de pré-biblioteca da seguinte maneira: 95 °C por 2 min; 12 ciclos de 15 °C por 40 s, 22 °C por 40 s, 33 °C por 30 s, 65 °C por 30 s, 72 °C por 40 s, 95 °C por 10 s e 63 °C por 10 s; e manter a 4 °C.
- Clique no ícone Pre_Lib para entrar na tela de configuração.
- Selecione Tubo para o modo de controle; entrada de 70 μL para o volume da amostra; selecione Ativado para controle da tampa quente e insira 105 °C para a temperatura. Selecione Não para Pausar no primeiro seg. Clique em OK para continuar.
- Aguarde até que Remain time mostre --:-- :--, que indica o final do programa, e clique em Parar para encerrar o programa.
- Pare o programa quando o processo estiver concluído. Prossiga para a próxima etapa imediatamente.
- Preparação da Biblioteca
- Misture bem pipetando e centrifugando imediatamente por 2-3 s a 200 x g.
- Prepare uma mistura mestra para a reação da biblioteca da seguinte forma: adicione 1,6 μL de mistura enzimática de biblioteca a 60 μL de tampão de biblioteca, misture a reação completamente e centrifuja brevemente.
- Adicionar 60 μL de mistura de reação de biblioteca e 2 μL de Primer de código de barras a cada produto de pré-biblioteca a partir da etapa 3.2.3. Misture bem a reação e centrifugue brevemente.
- Colocar o(s) tubo(s) de PCR do passo 3.2.3 no termociclador e executar o programa de preparação da biblioteca da seguinte forma: 94 °C durante 30 s; 17 ciclos de 94 °C por 25 s, 62 °C por 30 s e 72 °C por 45 s); e depois segure a 4 °C.
- Clique no ícone Lib_Prep para entrar na tela de configuração.
- Selecione Tubo para o modo de controle; entrada de 130 μL para o volume da amostra; selecione Ativado para controle Hotlid e insira 105 °C para a temperatura correspondente. Selecione Não para Pausar no primeiro seg. Clique em OK para continuar.
- Aguarde até que Remain time mostre --:-- :--, que indica o final do programa, e clique em Parar para encerrar o programa.
- Purificação da Biblioteca
- Remova as magbeads do armazenamento a 2-8 °C durante pelo menos 20 minutos antes da etapa de purificação. Vórtice e misture os Magbeads por 20 s. Distribua contas suficientes para a etapa de purificação em um novo tubo de microcentrífuga de 1,5 mL e contas quentes para RT.
- Adicione 1x Magbeads em cada biblioteca. Misture pipetando para cima e para baixo ≥10 vezes e incube em TR por 5 min.
Observação : por exemplo, adicione 100 μL de Magbeads a 100 μL de amostra de biblioteca. - Após a incubação, centrifugar brevemente o tubo e colocar em um suporte magnético.
- Aguarde aproximadamente 5 min até que a solução fique clara. Ao manter o tubo no suporte magnético, aspirar cuidadosamente a solução e descartar.
- Adicionar 200 μL de etanol 80% recém-preparado ao tubo. Incubar em RT por 30 s e remover cuidadosamente o sobrenadante. Repita mais uma vez.
- Retire o etanol o mais completamente possível. Seque ao ar as contas no suporte magnético por aproximadamente 5-10 min no RT.
- Retire o tubo do suporte magnético, adicione 17,5 μL de tampão de eluição e vomite o tubo para ressuspender as contas. Centrifugar brevemente o tubo e incubar em TR por 5 min.
- Coloque o tubo no suporte magnético e aguarde até que a solução fique clara. Transfira cuidadosamente o sobrenadante de 15 μL para um novo tubo.
- Quantificação da biblioteca
- Quantificar bibliotecas purificadas usando o fluorômetro de acordo com o guia do usuário dos kits de ensaio qubit dsDNA HS28. O rendimento das bibliotecas varia de ~15 a 300 ng.
- Agrupamento de bibliotecas
- Use 10 nanogramas de cada amostra de biblioteca para agrupamento.
- Seqüenciamento
- Consulte o guia do usuário de sequenciamento (15027617 v01)29.
- Sequências de bibliotecas purificadas de 50 pb em uma única extremidade na plataforma produziram aproximadamente 2 milhões de leituras para cada amostra, e uma profundidade de sequenciamento de 0,03 × foi recomendada.
- Análise de dados
- Digite o nome e a senha dos usuários na página de login
- Depois de fazer login no sistema, clique em Análise e uma nova página aparecerá. Clique em Criar envio na guia NICS-A. Em seguida, escolha NGS para a plataforma, selecione corporação, escolha NICSInst para o reagente, insira as informações do projeto na caixa em ID do projeto, defina as preferências de análise e carregue os arquivos. Depois que todos os arquivos de sequenciamento forem carregados com êxito, clique em Enviar para iniciar a análise (Figura 3A).
- Clique em Exibir envios para mostrar a lista de projetos enviados. Quando a análise estiver concluída, o status de um projeto se tornará Concluído e um botão Mostrar aparecerá no campo de relatório. Clique no botão Mostrar para exibir a tabela de resumo da análise NICS (Figura 3B).
- Clique no botão Exportar relatório para salvar os relatórios (Figura 3C).
Observação : três tipos de arquivos serão exportados para cada análise. Um arquivo gráfico que inclui todos os gráficos de variação do número de cópias (CNV) para cada cromossomo e genoma inteiro, que serão armazenados na pasta "gráfico"; uma planilha que contém os detalhes do QC de exemplo dessa execução de análise; um arquivo de documento que contém os relatórios NICS personalizados pelo usuário; e uma planilha que contém as informações de resumo de exemplo dessa execução de análise.
Resultados
O presente estudo aplicou o método proposto em um paciente. A aprovação do IRB e o consentimento informado foram obtidos antes da aplicação da análise NICS. O presente estudo obteve 6 blastocistos de pacientes e realizou NICS em todos os 6 embriões no meio do dia 4 ao dia 5. Anormalidades cromossômicas causadas pela translocação balanceada dos pais foram detectadas em cinco cromossomos com o ensaio NICS; portanto, não puderam ser utilizados para transferência (Figura 4A-E...
Discussão
Modificações e solução de problemas
Se os resultados do NICS estiverem contaminados com materiais genéticos parentais, certifique-se de que todas as células cumulus-corona radiata sejam removidas e certifique-se de que a ICSI seja realizada para fertilização. Evitam-se processos inadequados de armazenamento de meios ou preparação de moldes, o que pode degradar o DNA. O espaço de trabalho foi cuidadosamente purificado com reagentes de descontaminação DNase e RNase....
Divulgações
Yaxin Yao, Jieliang Ma, Jing Wang e Sijia Lu são funcionários da Yikon Genomics Co., Ltd.
Agradecimentos
Os autores gostariam de agradecer a Shiping Bo e Shujie Ma por sua assistência na análise de dados NGS. Financiamento: este trabalho foi apoiado pelo Programa Nacional de Pesquisa e Desenvolvimento (Processo Nº 2018YFC1003100).
Materiais
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
Referências
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T., Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid?. Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- . Qubit dsDNA HS Assay Kit Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015)
- . Miseq system use guide Available from: https://support.illumina.com/downloads/miseq_system (2016)
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Erratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021. Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
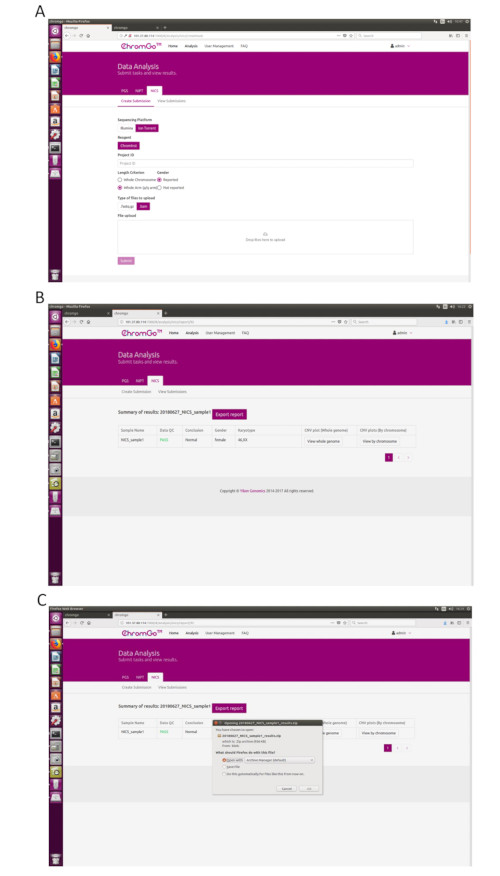
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
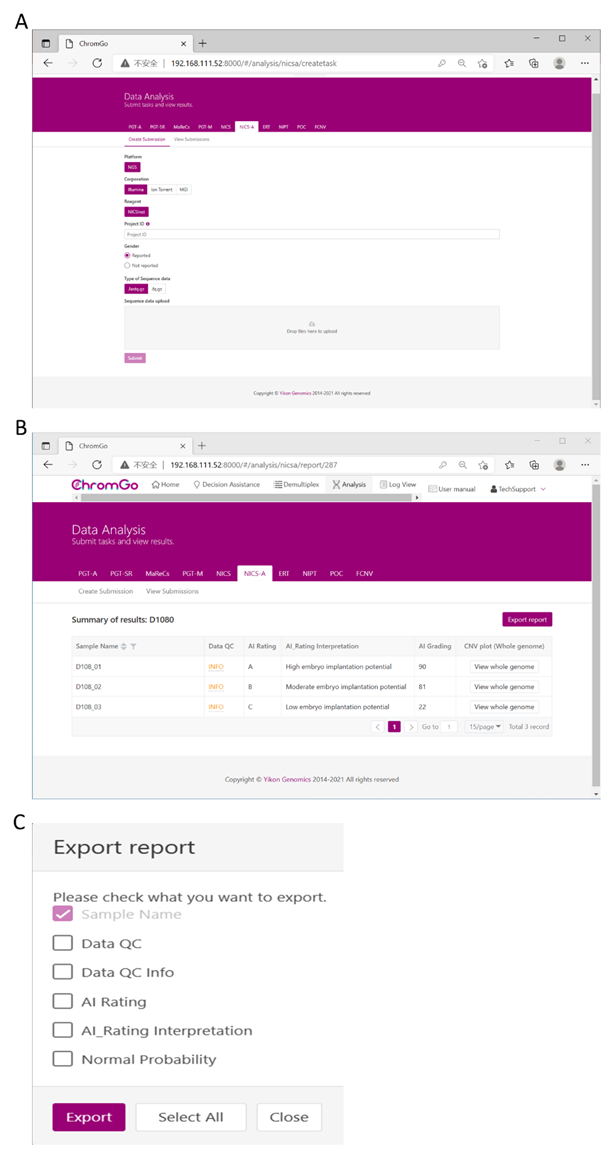
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados