このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
使用済み培地を用いたヒト着床前胚の染色体スクリーニング:サンプル採取と染色体倍数性解析
* これらの著者は同等に貢献しました
Erratum Notice
要約
本研究は、使用済み培地を使用したヒト胚の染色体スクリーニングのプロトコルを報告し、胚生検を回避し、NGSを使用して染色体倍数性を同定できるようにします。本稿では、培地の調製、全ゲノム増幅(WGA)、次世代シーケンシング(NGS)ライブラリの調製、データ解析など、詳細な手順を紹介します。
要約
臨床体外受精(IVF)では、PGT-Aの一般的な方法では、栄養外胚葉(TE)からのいくつかの細胞の生検が必要です。これが胎盤を形成する系統です。しかし、この方法は専門的な技術が必要で、侵襲的であり、TEの染色体数と胎児に成長する内部細胞塊(ICM)が常に同じであるとは限らないため、偽陽性と偽陰性に悩まされます。NICS は、TE と ICM の両方から培地に放出された DNA のシーケンシングを必要とする技術であり、これらの問題に対する解決策を提供する可能性がありますが、有効性は限定的であることが以前に示されていました。本研究では、培地サンプリング法、全ゲノム増幅(WGA)およびライブラリ調製、および解析ソフトウェアによるNGSデータ解析を含むNICSの完全なプロトコルを報告します。胚検査室によって凍結保存時間が異なることを考慮し、胚培養士は、体外受精検査室の実際の状況に応じて選択できる胚培養培地を採取するための2つの方法を持っています。
概要
生殖補助医療(ART)は、不妊症の治療にますます使用されています。しかし、体外受精などのARTの成功率は限られており、流産率は正常集団よりも有意に高くなっています1。これらの問題の主な原因は染色体異常であり、着床前のヒト胚に一般的に存在します2。PGT-Aは、着床前に胚の染色体バランスをスクリーニングする効果的な方法です3,4。いくつかの研究は、PGT-Aが中絶率を減らし、妊娠率を改善できることを証明しています5,6,7,8。ただし、PGT-Aには、特定のトレーニングと経験を必要とする複雑な技術的専門知識が必要です。侵襲的な胚生検手順も、胚に損傷を与える可能性があります9。研究によると、割球生検はその後の発生を妨げる可能性があり、生検されたTEの数は着床率に影響を与える可能性があります10。胚生検の長期的なバイオセーフティの問題は、ヒトではまだ完全に評価されていませんが、動物実験では、胚の発生に悪影響を与えることが示されています11,12,13。
これまでの報告では、胚発生時に培地に微量のDNA物質が分泌されることが報告されており、使用済み胚培養液14,15,16,17,18を用いて包括的染色体スクリーニング(CCS)を行う取り組みが行われています。しかし、検出率と検査の精度は、広範な臨床使用の要件を満たしていません。本研究は、NICS検査の検出率と精度を向上させるためのNICSアッセイの改善を報告した19。近年、低侵襲PGT-Aの分析サンプルとして割球体液(BF)が研究されています。しかし、胚盤胞液サンプル中のゲノムワイド増幅と検出可能なDNAの割合は、34.8%から82%の範囲です20,21,22。さまざまな研究で報告されているBFの量は、0.3 nLから1 μLの範囲です。BF中のDNA量が少ないため、胚盤胞液と培地を混合することで無細胞DNAの量を増やすことができ、検出の成功率と一貫性を向上させることができます。クズニェツォフ他図23およびLiら24は、透明帯をレーザーで処理し、胚盤胞液を培地に放出して胚性DNAの総量を改善し、WGA後の培地/BFサンプルを組み合わせた増幅率は、それぞれ100%および97.5%であった。Jiao et al.25 も、同じ方法を用いて 100% の増幅成功率を得ました。
本研究では、使用済み培地サンプルの調製、NGSの調製、およびデータ分析を含む詳細なプロトコルを報告します。本研究では、卵母細胞を卵母細胞から慎重に除去し、細胞質内単精子注入(ICSI)と胚盤胞培養を行いました。4日目から5日目/6日目に使用した培地をWGAおよびNGSライブラリ調製のために回収した。NICS技術を用いることで、WGAおよびNGSライブラリ調製ステップを約3時間で合理化し、約9時間で非侵襲的にCCS結果を得た。
プロトコル
北京大学第三病院の倫理委員会から倫理的許可を取得しました。
1. 事前準備
注意: 必要な材料と機器は 、材料表にリストされています。
- 試薬
- 20-30μLの配偶子培地/受精培地、切断/胚盤胞段階の培養培地(鉱物油で覆われている)とヒアルロニダーゼ(しっかりとキャップされたチューブ内)を37°C、5%CO2および5%O2で、使用前にトライガスインキュベーターで一晩予温し、平衡化(バランス)する。
- ヒアルロニダーゼをドラフト内の作業面で37°Cに予熱する。
- ガラス化緩衝液とサンプル採取試薬は、メーカーの指示に従って調製してください。
- ツール
- サンプル採取・移送用ピペット(内径~200〜250μm)、脱皮/ストリッパーピペット(内径≥150μm、~130-140μm、~120μm)、洗浄用ピペット(内径~150μm)をガラス製パスツールピペットを引っ張って準備し、ファイヤーポリッシュ仕上げのオープンファインチップを生成します。
注:サンプルの採取/移送、脱皮、洗浄に使用するピペットは、直接購入できます。保持針と注射針は直接購入することもできます。
- サンプル採取・移送用ピペット(内径~200〜250μm)、脱皮/ストリッパーピペット(内径≥150μm、~130-140μm、~120μm)、洗浄用ピペット(内径~150μm)をガラス製パスツールピペットを引っ張って準備し、ファイヤーポリッシュ仕上げのオープンファインチップを生成します。
2.プロトコル1:サンプル収集
- ヒアルロニダーゼによる消化前の卵母細胞-コロナ-卵丘複合体(OCCC)の前処理
- 卵胞刺激ホルモン(FSH)とヒト更年期ゴナドトロピン(hMG)製剤の両方で卵巣刺激を達成します。鉛卵胞が>18 mmの場合、最終的な卵母細胞の成熟に10,000 IUの絨毛性ゴナドトロピン(hCG)を使用します。
- トリガーショットの36時間後に卵母細胞回収を行います。卵母細胞を拾い上げ、鉱物油で覆われた2.5 mLの予熱されたm-HTFで組織培養皿に移します。
- トランスファーピペットを使用してOCCCを1mLの受精培地を含む臓器培養皿の中央ウェルに迅速に移し、5%CO2および5%O2インキュベーターで37°Cで卵母細胞と2〜4時間インキュベートします。
- OCCCを含む臓器培養皿の中央ウェルに、37°Cで予熱したヒアルロニダーゼ(80 IU/mL)1 mLを添加して、ヒアルロニダーゼでOCCCを消化する(ステップ2.1.3)。ヒアルロニダーゼの最終濃度を40 IU/mLに保ち、十分に混合する。
- OCCCを37°Cのサーマルプラットホームで2分間インキュベートします。顆粒膜細胞の1〜2層のみが残るまで、30秒ごとに顕微鏡で変化を観察します。
- 顆粒膜細胞の脱皮
- 消化したOCCCを培養皿に迅速に移し、卵母細胞を取り扱うために、各ウェルで鉱油で覆います。
- 分離した顆粒膜細胞を顕微鏡で観察します。卵母細胞を5回静かに吸引して放出し、卵母細胞の周囲に残っている顆粒膜細胞を除去します。
- 残りの3つのウェルで前のステップを繰り返して、顆粒膜細胞を完全に除去します。
注:上記の手順(2.1〜2.3)は、各ラボの日常的な操作に従って実行できます。
- 卵母細胞の評価
- 顕微鏡を使用して顆粒膜細胞除去の完全性を評価します。細胞を完全に除去できなかった場合は、現時点では5個以下の顆粒膜細胞の保持が許容されます。
注:卵丘細胞がまだ卵母細胞に付着している場合は、胚が切断段階の培養液から胚盤胞段階の培養液に移される前に、3日目の後半に残骸を取り除くことができます。
- 顕微鏡を使用して顆粒膜細胞除去の完全性を評価します。細胞を完全に除去できなかった場合は、現時点では5個以下の顆粒膜細胞の保持が許容されます。
- 細胞質内精子注入(ICSI)26を行った後、トランスファーピペットを用いて卵母細胞を20〜30μLの切断胚培養培地の微小液滴(1つの卵母細胞は1つの微小液滴に相当)に移し、37°C、5%CO2および5%O2インキュベーターでインキュベートする。
- ICSIの日を0日目として記録します。受精の1日目(約18時間)、2日目(約45時間)、および胚の卵割の3日目(約68時間)の胚評価に関するイスタンブールコンセンサスワークショップに従って、胚を確認し、スコアを確認します27。
- 胚洗浄
- 2日目に37°C、5%CO2および5%O2インキュベーターで、組織培養皿に鉱油で覆われた各胚に対して20〜30μLの胚盤胞培養培地の微小液滴を調製する。
- さらに3つの微小液滴を鉱物油で覆い、洗浄用新しい組織培養皿にラベルを付けます。
- 3日目の胚を洗浄用微小液滴に移します。脱皮ピペットを使用して、各液滴に胚を3回静かに吸引し、放出します。
注:この手順は、胚に付着した残留顆粒細胞を除去するのにも役立ちます。 - 3日目に顕微鏡で胚を観察し、評価し、形態学的スコアリングのために培地を切断段階の培養培地から胚盤胞培養培地に変更した。卵丘細胞がまだ胚に付着している場合は、卵丘細胞が完全に除去されるまで、鉱油で覆われた別の予熱および平衡化された胚盤胞培養液滴でストリッパーピペットで適切に上下にピペットします。
注:胚を切断段階の培養培地プレートから胚盤胞段階の培養培地プレートに移す前に、付着したすべての卵丘細胞を3日目に完全に除去する必要がありました。残っている積雲セルは、最終分析に干渉し、偽陰性の結果を生じます。
- 培地回収の2つの選択肢
注:体外受精センターは、センターのリソース、需要、および好みに基づいて、培地収集の2つの方法のいずれかを選択できます。- オプション1:胚の洗浄と培養
注:このオプションは、5日目の朝にガラス化を行うIVFラボ用です。- 胚を予熱した(37°C)培養液の微小液滴に移し、4日目の午後にピペッティングにより、各胚を3つの微小液滴で連続して静かに洗浄します。
- 各胚を、事前に温めた(37°C)単一の微小液滴の培地に移し、サンプルを採取します。培地の1液滴の容量は25μLを超えることはできません。
- 胚盤胞胚培養を5日目/6日目に37°C、5%CO2、および5%O2で実施する。
- オプション2:胚の洗浄と培養
注:このオプションは、5日目の午後または6日目にガラス化を行う体外受精ラボ用です。- 胚を10〜15μLの培養培地の予熱(37°C)マイクロドロップレットに移し、5日目にピペッティングにより、各胚を3つのマイクロドロップレットで連続的に静かに洗浄します。
- 各胚を、事前に温めた(37°C)単一の微小液滴の培地に移し、サンプルを採取します。培地の1液滴の容量は15μLを超えることはできません。
- 胚盤胞胚培養を5日目/6日目に37°C、5%CO2で実施する。
- オプション1:胚の洗浄と培養
- サンプルコレクション
- 栄養外胚葉の細胞接合部に焦点を合わせて栄養外胚葉に小さな穴を開けて胚盤胞腔から液体を放出するレーザービームのターゲットポイントからかなりの距離でICMを静かに調整します。次に、胚を凍結液に移し、従来のプロセスに従って凍結保存します。
- 培養した各胚から、5 μLの細胞溶解バッファーを含むRNase/DNaseフリーPCRチューブに培養液を移します。
- ネガティブコントロールとして胚培養に使用せずに、同量の培地を採取します。採取したサンプルは直ちに液体窒素で凍結し、採取後は-80°CでNICSアッセイにかけるまで保存してください。
- プロトコルに記載されているようにガラス化を行います。
3. 議定書2:図書館建設
- 培地溶解
- 1 μL のポジティブコントロール(10 ng ヒト gDNA)を 199 μL の新鮮培地で希釈します。完全に混合し、チューブを短時間遠心分離します(200 x g で5秒間)。
- 5日目から6日目の胚盤胞培養培地10 μL、希釈したポジティブコントロール、および新鮮な培養培地を新しい0.2 mLのPCRチューブに移します。
- 各PCRチューブに1 μLのMT酵素ミックスを加え、ピペッティングで完全に混合し、すぐに200 x gで2〜3秒間遠心分離します。
- ステップ 3.1.3 の PCR チューブを予熱した NICS サンプル調製ステーションに入れ、次のように溶解プログラムを実行します。 75°Cで10分間。95°Cで4分。22°Cで保持します。
注:サンプル調製ステーションは、標準的なPCRマシンに匹敵します。- Lysisアイコンをクリックして、セットアップ画面に入ります。
- 制御モードに チューブ を選択します。サンプル量は10 μLです。ホットリッド制御(Hotlid control)に オン(On )を選択し、温度(temperature)に 105 °C と入力します。最初のセグメントで一時停止するために [いいえ ] を選択します。「 OK 」をクリックして次に進みます。
- [残り時間] にプログラムの終了を示す --:--:-- が表示されるまで待ってから、[停止] をクリックしてプログラムを終了します。
- プロセスが完了したら、プログラムを停止します。すぐに次の手順に進みます。
- ライブラリ前準備
- Pre-Lib Buffer を RT に解凍します。 ピペッティングで十分に混合し、すぐに 200 x g で 2-3 秒間遠心分離します。
- 2 μL の Pre-Lib Enzyme Mix を 60 μL の Pre-Lib バッファーに加え、反応液を完全に混合し、短時間遠心分離します。
- 前のステップで前処理した各培地サンプルに60 μLのプリライブラリ反応ミックスを加えます。ピペッティングで完全に混合し、すぐに200 x gで2〜3秒間遠心分離します。
- ステップ 3.2.3 の PCR チューブをサンプル調製ステーションにセットし、次のようにプレライブラリプログラムを実行します。 95°Cで2分間。15°Cで40秒、22°Cで40秒、33°Cで30秒、65°Cで30秒、72°Cで40秒、95°Cで10秒、63°Cで10秒の12サイクル。4°Cで保持します。
- Pre_Libアイコンをクリックして、セットアップ画面に入ります。
- 制御モードに チューブ を選択します。サンプル量の場合は70 μLを入力します。ホットリッドコントロールで オン を選択し、温度に105°Cと入力します。最初のセグメントで一時停止するために [いいえ ] を選択します。「 OK 」をクリックして次に進みます。
- [残り時間] にプログラムの終了を示す --:--:--- が表示されるまで待ち、[停止] をクリックしてプログラムを終了します。
- プロセスが完了したら、プログラムを停止します。すぐに次の手順に進みます。
- ライブラリの準備
- ライブラリバッファーをRTに融解し、ピペッティングで完全に混合し、すぐに200 x gで2〜3秒間遠心分離します。
- ライブラリー反応用のマスターミックスを調製するには、1.6 μLのLibrary Enzyme Mixを60 μLのライブラリーバッファーに加え、反応液を十分に混合し、短時間遠心分離します。
- ステップ 3.2.3 の各プレライブラリ製品に、60 μL のライブラリ反応ミックスと 2 μL のバーコードプライマーを加えます。反応液を十分に混合し、短時間遠心分離します。
- 手順3.2.3のPCRチューブをサーマルサイクラーに入れ、ライブラリ調製プログラムを次のように実行します:94°Cで30秒。94°Cで25秒、62°Cで30秒、72°Cで45秒の17サイクル。その後、4°Cで保持します。
- Lib_Prepアイコンをクリックして、セットアップ画面に入ります。
- 制御モードに チューブ を選択します。サンプル量は130μL。Hotlid control(ホットリッド制御)で On を選択し、対応する温度に105°Cと入力します。最初のセグメントで一時停止するために [いいえ ] を選択します。「 OK 」をクリックして次に進みます。
- [残り時間] にプログラムの終了を示す --:--:--- が表示されるまで待ち、[停止] をクリックしてプログラムを終了します。
- ライブラリの精製
- 浄化のステップの前に少なくとも20分の2-8 °Cで貯蔵からMagbeadsを取除いて下さい。マグビーズを20秒間ボルテックスして混ぜます。精製ステップに必要な量のビーズを新しい 1.5 mL 微量遠心チューブに分注し、温めて RT に注入します。
- 各ライブラリーにマグビーズを1つ追加します。ピペッティングで≥10回混合し、室温で5分間インキュベートします。
注:例えば、100 μLのライブラリサンプルに100 μLのMagbeadsを加えます。 - インキュベーション後、チューブを短時間遠心分離し、磁気スタンドに置きます。
- 溶液が明らかになるまで約5分間待ちます。チューブを磁気スタンドに置いたまま、溶液を慎重に吸引して廃棄します。
- 調製したばかりの80%エタノール200μLをチューブに加えます。室温で30秒間インキュベートし、上清を慎重に除去します。もう一度繰り返します。
- エタノールをできるだけ完全に取り除きます。磁気スタンド上のビーズをRTで約5〜10分間風乾します。
- チューブを磁気スタンドから取り外し、17.5 μLの溶出バッファーを添加し、チューブをボルテックスしてビーズを再懸濁します。チューブを短時間遠心分離し、室温で5分間インキュベートします。
- チューブを磁気スタンドに置き、溶液が透明になるまで待ちます。上清15μLを新しいチューブに慎重に移します。
- ライブラリの定量
- 量子ビットdsDNA HSアッセイキット28のユーザーガイドに従って、蛍光光度計を使用して精製されたライブラリを定量します。ライブラリの収量は~15〜300ngの範囲です。
- ライブラリー・プーリング
- 各ライブラリーサンプルを10ナノグラムでプーリングに用います。
- シークエンシング
- シーケンシングユーザーガイド(15027617 v01)29を参照してください。
- プラットホーム上で片末端に 50 bp の精製ライブラリ配列を添加すると、各サンプルで約 200 万回のリードが得られ、シーケンシング深度は 0.03 × が推奨されました。
- データ解析
- ログインページにユーザーの名前とパスワードを入力します
- システムにログインした後、[ 分析 ]をクリックすると、新しいページが表示されます。[NICS-A] タブの [ 提出書類の作成 ] をクリックします。次に、プラットフォームに NGS 、corporation、試薬に NICSInstを選択し、Project IDの下のボックスにプロジェクト情報を入力し、分析設定を行い、ファイルをアップロードします。すべてのシーケンシングファイルが正常にアップロードされたら、[ Submit ]をクリックして解析を開始します(図3A)。
- [ 提出物の表示 ] をクリックして、提出済みプロジェクトの一覧を表示します。分析が完了すると、プロジェクトのステータスが「完了」になり、レポートフィールドに「表示」ボタンが表示されます。 [Show ](表示)ボタンをクリックすると、NICS分析の概要表が表示されます(図3B)。
- [ レポートのエクスポート ] ボタンをクリックして、レポートを保存します(図 3C)。
注: 解析ごとに 3 種類のファイルがエクスポートされます。各染色体と全ゲノムのすべてのコピー数多型(CNV)プロットを含むグラフィックファイルで、「グラフ」フォルダーに保存されます。この分析実行のサンプルQCの詳細を含むスプレッドシート。ユーザーがカスタマイズした NICS レポートを含むドキュメントファイル。この分析実行のサンプル・サマリー情報を含むスプレッドシート。
結果
本研究では、提案手法を患者に適用した。NICS分析の適用前にIRBの承認とインフォームドコンセントが得られました。本研究では、患者から6つの胚盤胞を取得し、培地4日目から5日目に6つの胚すべてにNICSを実施しました。両親の平衡転座によって引き起こされる染色体異常は、NICSアッセイで5つの染色体で検出されました。そのため、転写には使用できませんでした(図4A-E
ディスカッション
変更とトラブルシューティング
NICSの結果が親の遺伝物質で汚染されている場合は、すべての積丘-コロナラジアータ細胞が除去されていることを確認し、受精のためにICSIが行われていることを確認してください。DNAを分解する可能性のある不適切な培地保管やテンプレート調製プロセスを回避します。作業空間はDNaseおよびRNase除染試薬で徹底的に浄化され?...
開示事項
Yaxin Yao氏、Jieliang Ma氏、Jing Wang氏、Sijia Lu氏は、Yikon Genomics Co., Ltd.の従業員です。
謝辞
著者らは、NGSデータ解析に協力してくれたShiping Bo氏とShujie Ma氏に感謝します。資金提供:この研究は、国家重点研究開発プログラム(助成金番号2018YFC1003100)の支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 1.5 mL EP tube, 0.2 mL PCR tube | Axygen | MCT-150-C, PCR-02-C | DNase/RNase free, Low Binding PCR tubes and 1.5 mL micro-centrifuge tubes are recommended. |
| 10 µL, 200 µL, 1000 µL DNase /RNase Free Tips | Axygen | T-300-R-S, T-200-Y-R-S, T-1000-B-R-S | This can be replaced by other brand/For sample transfer |
| 100 % ethanol | Sinopharm Chemical | 10009218 | This can be replaced by other brand/For DNA library purification |
| Barcode Primer1-48 | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| BD Falcon Organ Culture Dish, Sterile | BD Bioscience | 363037 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes (Easy Grip) , Sterile | BD Bioscience | 353001 | This can be replaced by other brand/For embryo culture |
| BD Falcon Tissue culture Dishes, Sterile | BD Bioscience | 353002 | This can be replaced by other brand/For embryo culture |
| Cell Lysis Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| Cell Lysis Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| ChromGo software | Yikon Genomics | Data analysis | |
| CMPure Magbeads | Yikon Genomics | Reagent in NICSInst library preparation kit | For library purification |
| Cryotop open systerm | KITAZATO BioPharma | 81110 | This can be replaced by other brand/For embryo vitrification |
| Distill water | Yikon Genomics | Reagent in NICSInst library preparation kit | To dissolve DNA |
| ES (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| HOLDNIG | ORIGIO | MPH-MED-35 | This can be replaced by other brand/For ICSI |
| Hyaluronidase solution, 80 U/mL | SAGE | ART4007-A | This can be replaced by other brand/Digest oocyte-corona-cumulus complex |
| ICSI | ORIGIO | MPH-35-35 | This can be replaced by other brand/For ICSI |
| Illumina MiSeq® System | Illumina | SY-410-1001 | For library sequencing |
| Incubator | Labotect | Inkubator C16 | This can be replaced by other brand/For embryo culture |
| Library buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Library Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For library amplificaton |
| Magnetic Stand | DynaMagTM-2 | 12321D | For library purification |
| Microscope | OLYMPUS | 1X71 | This can be replaced by other brand/For embryo observation |
| Mini-centrifuge | ESSENSCIEN | ELF6 | For separation |
| MT Enzyme Mix | Yikon Genomics | Reagent in NICSInst library preparation kit | For culture medium pre-treatment |
| NICSInst library preparation kit | Yikon Genomics | KT1000800324 | Whole genome amplification and library construction |
| NICSInst Sample Prep Station | Yikon Genomics | ME1001003 | Amplificate DNA |
| Nunc IVF 4-Well Dish | Thermo Scientific | 144444 | This can be replaced by other brand/For embryo washing and blastocyst culture |
| Pasteur Pipette | Oirgio | MXL3-IND-135 | This can be replaced by other brand/For embryo tansfer |
| Pasteur pipettes | ORIGIO | PP-9-1000 | This can be replaced by other brand/For IVF laboratory |
| Pre-Lib Buffer | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Pre-Lib Enzyme | Yikon Genomics | Reagent in NICSInst library preparation kit | Pre-library preparation |
| Qubit® 3.0 Fluorometer | Thermo Scientific | Q33216 | For library quantification |
| Quinn's Advantage Blastocyst Medium | SAGE | ART-1029 | For embryo blastocyst stage culture |
| Quinn's Advantage Cleavage Medium | SAGE | ART-1026 | This can be replaced by other brand/For embryo cleavage stage culture |
| Quinn's Advantage Fertilization Medium | SAGE | ART-1020 | This can be replaced by other brand/For oocyte and sperm fertilization |
| Quinn's Advantage m-HTF Medium with HEPES | SAGE | ART-1023 | This can be replaced by other brand/For embryo clutrure |
| Quinn's Advantage SPS Serum protein Substitute Kit | SAGE | ART-3010 | This can be replaced by other brand/To denude the oocyte |
| Quinn's Advantage Tissue culture mineral oil | SAGE | ART-4008P | This can be replaced by other brand/To cover the culture medium |
| STRIPPER TIPS | ORIGIO | MXL3-IND-135 | This can be replaced by other brand/For denudating granulosa cells |
| Vitrification Cryotop Open systerm | KIZTAZATO | 81111 | This can be replaced by other brand/For embryo vitrification |
| Vitrification kit | KITAZATO BioPharma | VT101 | This can be replaced by other brand/For embryo vitrification |
| Vortexer | Qilinbeier | DNYS8 | Sample mix |
| VS (Vitrification kit) | KITAZATO BioPharma | Reagent inVitrification kit | This can be replaced by other brand/For embryo vitrification |
| ZILOS-tk Laser System | Hamilton Thorne | CLASS 1 laser | This can be replaced by other brand/For artificial blastocoele collapse |
参考文献
- Barlow, P. Early pregnancy loss and obstetrical risk after in-vitro fertilization and embryo replacement. Human Reproduction. 3 (5), 671-675 (1988).
- Munne, S. Chromosome abnormalities and their relationship to morphology and development of human embryos. Reproductive BioMedicine Online. 12 (2), 234-253 (2006).
- Harton, G. L. Diminished effect of maternal age on implantation after preimplantation genetic diagnosis with array comparative genomic hybridization. Fertility and Sterility. 100 (6), 1695-1703 (2013).
- Hodes-Wertz, B. Idiopathic recurrent miscarriage is caused mostly by aneuploid embryos. Fertility and Sterility. 98 (3), 675-680 (2012).
- Keltz, M. D. Preimplantation genetic screening (PGS) with Comparative genomic hybridization (CGH) following day 3 single cell blastomere biopsy markedly improves IVF outcomes while lowering multiple pregnancies and miscarriages. Journal of Assisted Reproduction and Genetics. 30 (10), 1333-1339 (2013).
- Scott, R. T. Blastocyst biopsy with comprehensive chromosome screening and fresh embryo transfer significantly increases in vitro fertilization implantation and delivery rates: a randomized controlled trial. Fertility and Sterility. 100 (3), 697-703 (2013).
- Forman, E. J. In vitro fertilization with single euploid blastocyst transfer: a randomized controlled trial. Fertility and Sterility. 100 (1), 100-107 (2013).
- Yang, Z. Selection of single blastocysts for fresh transfer via standard morphology assessment alone and with array CGH for good prognosis IVF patients: results from a randomized pilot study. Molecular Cytogenetics. 5 (1), 24 (2012).
- Cimadomo, D. The Impact of Biopsy on Human Embryo Developmental Potential during Preimplantation Genetic Diagnosis. BioMed Research International. 2016, 7193075 (2016).
- Scott, R. T., Upham, K. M., Forman, E. J., Zhao, T., Treff, N. R. Cleavage-stage biopsy significantly impairs human embryonic implantation potential while blastocyst biopsy does not: a randomized and paired clinical trial. Fertility and Sterility. 100 (3), 624-630 (2013).
- Wu, Y. Blastomere biopsy influences epigenetic reprogramming during early embryo development, which impacts neural development and function in resulting mice. Cellular and Molecular Life Sciences. 71 (9), 1761-1774 (2014).
- Zhao, H. C. Aberrant epigenetic modification in murine brain tissues of offspring from preimplantation genetic diagnosis blastomere biopsies. Biology of Reproduction. 89 (5), 117 (2013).
- Zeng, Y. Preimplantation genetic diagnosis (PGD) influences adrenal development and response to cold stress in resulting mice. Cell and Tissue Research. 354 (3), 729-741 (2013).
- Palini, S. Genomic DNA in human blastocoele fluid. Reproductive BioMedicine Online. 26 (6), 603-610 (2013).
- Gianaroli, L. Blastocentesis: a source of DNA for preimplantation genetic testing. Results from a pilot study. Fertility and Sterility. 102 (6), 1692-1699 (2014).
- Stigliani, S., Anserini, P., Venturini, P. L., Scaruffi, P. Mitochondrial DNA content in embryo culture medium is significantly associated with human embryo fragmentation. Human Reproduction. 28 (10), 2652-2660 (2013).
- Stigliani, S. Mitochondrial DNA in Day 3 embryo culture medium is a novel, non-invasive biomarker of blastocyst potential and implantation outcome. Molecular Human Reproduction. 20 (12), 1238-1246 (2014).
- Wu, H. Medium-Based Noninvasive Preimplantation Genetic Diagnosis for Human α-Thalassemias-SEA. Medicine. 94 (12), e669 (2015).
- Xu, J. Noninvasive chromosome screening of human embryos by genome sequencing of embryo culture medium for in vitro fertilization. Proceedings of the National Academy of Sciences. 113 (42), 11907-11912 (2016).
- Capalbo, A. Diagnostic efficacy of blastocoel fluid and spent media as sources of DNA for preimplantation genetic testing in standard clinical conditions. Fertility and Sterility. 110 (5), 870-879 (2018).
- Tobler, K. J. Blastocoel fluid from differentiated blastocysts harbors embryonic genomic material capable of a whole-genome deoxyribonucleic acid amplification and comprehensive chromosome microarray analysis. Fertility and Sterility. 104 (2), 418-425 (2015).
- Magli, M. C. Preimplantation genetic testing: polar bodies, blastomeres, trophectoderm cells, or blastocoelic fluid?. Fertility and Sterility. 105 (3), 676-683 (2016).
- Kuznyetsov, V. Evaluation of a novel non-invasive preimplantation genetic screening approach. PLoS One. 13 (5), e0197262 (2018).
- Li, P. Preimplantation Genetic Screening with Spent Culture Medium/Blastocoel Fluid for in Vitro Fertilization. Scientific Reports. 8 (1), 9275 (2018).
- Jiao, J. Minimally invasive preimplantation genetic testing using blastocyst culture medium. Human Reproduction. 34 (7), 1369-1379 (2019).
- Palermo, G. D. Births after intracytoplasmic injection of sperm obtained by testicular extraction from men with nonmosaic Klinefelter's syndrome. New England Journal of Medicine. 338 (9), 588-590 (1998).
- Alpha Scientists in Reproductive, M., & Embryology, E. S. I. G. o. The Istanbul consensus workshop on embryo assessment: proceedings of an expert meeting. Human Reproduction. 26 (6), 1270-1283 (2011).
- . Qubit dsDNA HS Assay Kit Available from: https://www.thermofisher.com/order/catalog/product/Q32851?ICID=search-product (2015)
- . Miseq system use guide Available from: https://support.illumina.com/downloads/miseq_system (2016)
- Lane, M. Ability to detect aneuploidy from cell free DNA collected from media is dependent on the stage of development of the embryo. Fertility and Sterility. 108 (3), (2017).
- Rubio, C. Multicenter prospective study of concordance between embryonic cell-free DNA and trophectoderm biopsies from 1301 human blastocysts. American Journal of Obstetrics and Gynecology. 223 (5), 751-751 (2020).
- Rubio, C. Embryonic cell-free DNA versus trophectoderm biopsy for aneuploidy testing: concordance rate and clinical implications. Fertility and Sterility. 112 (3), 510-519 (2019).
- Lledo, B. Consistent results of non-invasive PGT-A of human embryos using two different techniques for chromosomal analysis. Reproductive BioMedicine Online. 42 (3), 555-563 (2021).
- Kuznyetsov, V. Minimally Invasive Cell-Free Human Embryo Aneuploidy Testing (miPGT-A) Utilizing Combined Spent Embryo Culture Medium and Blastocoel Fluid -Towards Development of a Clinical Assay. Scientific Reports. 10 (1), 7244 (2020).
Erratum
Formal Correction: Erratum: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis
Posted by JoVE Editors on 10/01/2021. Citeable Link.
An erratum was issued for: Chromosome Screening of Human Preimplantation Embryos by Using Spent Culture Medium: Sample Collection and Chromosomal Ploidy Analysis. The Protocol and Representaive Results sections were updated.
In the Protocol, step 3.8.2 was updated from:
After logging into the system, click Create Submission under the NICS tab. Then, select the sequencing platform, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
to:
After logging into the system, click Create Submission under the NICS-A tab. Then, choose NGS for the platform, select corporation, choose ChromInst for the reagent, enter the project information in the box under Project ID, set the analysis preferences and upload the files. Once all sequencing files are successfully uploaded, click Submit to start the analysis (Figure 3A).
In the Representative Results, Figure 3 was updated from:
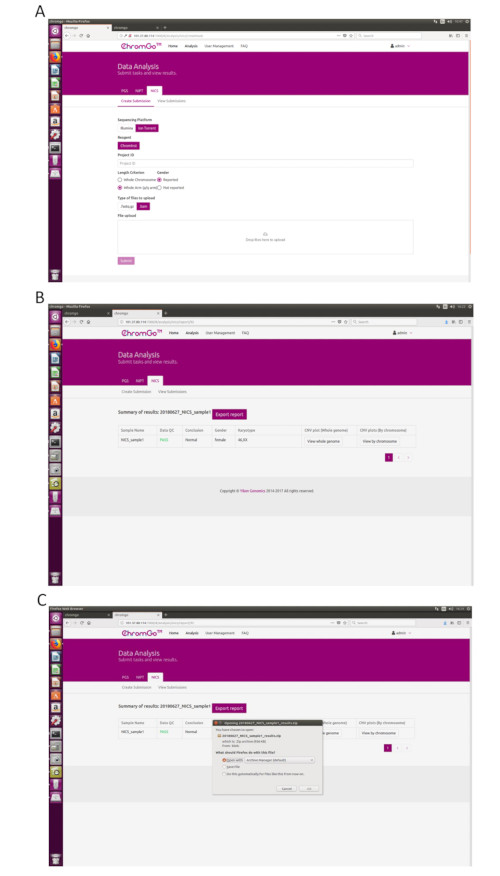
Figure 3. Data Analysis. (A) The page of Create Submission. There are different options for the user application. For sequencing platform, users can choose Illumina or Ion Torrent. For analysis criterion, there are two length detection resolution for selection, the whole chromosome and whole arm level. The users also can choose whether the mosaicism or gender information is reported. Finished the above parameter setting,click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. For Ion Torrent platform, choose files with an extension of bam. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; Conclusion: Indicates whether the NICS analysis is normal or abnormal, "N/A" indicates no conclusive result is available; Gender: If the user chooses to report the sex information, this column will appear in the summary table; Karyotype: Shows the analysis results; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; CNV plot (By Chromosome): View the CNV profiles of each chromosome. (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. Select Save File in the appearing dialog window and then click OK. The reports will be saved to the Download folder of the computer. Please click here to view a larger version of this figure.
to:
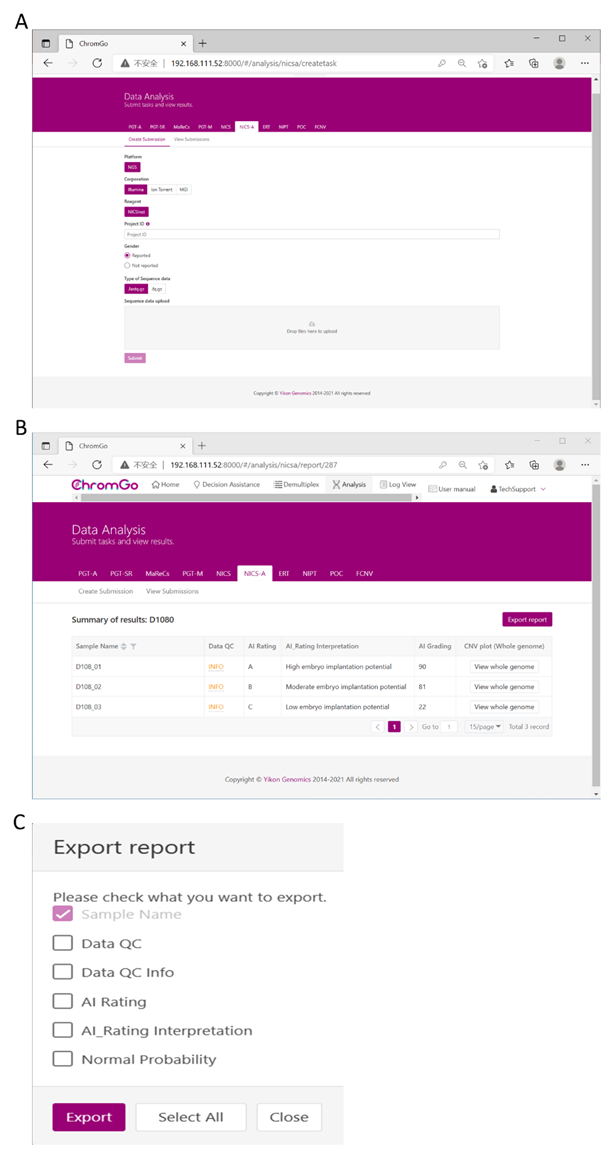
Figure 3. Data Analysis. (A) There are different options for the user application. For sequencing platform corporation, users can choose Illumina, Ion Torrent or MGI. The users can choose whether the gender information is reported. Finished the above parameter setting, click on the box under File upload and choose the appropriate sequencing files to upload. For Illumina, choose the files with an extension of fastq.gz. Click Submit to start the analysis after successfully upload. (B) The view of summary table. The summary table consists of following information: Sample Name: The name of each NICS sample is listed; Data QC: Indicates whether the sequencing file passes the QC for NICS analysis; AI Rating: The rating (A, B or C) for each NICS sample; AI_Rating Interpretation: Evaluation of embryo implantation potential; AI Grading: The score for each NICS sample; CNV plot (Whole Genome): View the CNV profiles of all chromosomes; (C) The Save Report Page. Click Export report button next to the Summary of Results. Select the information you want to show on the final report and click Export. The reports will be saved to the Download folder of your computer. Please click here to view a larger version of this figure.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved