A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
אפיון הדמיית החזר אינטרפרומטרי של חלקיק יחיד של שלפוחיות חוץ-תאיות בודדות ודינמיקה של אוכלוסייה
In This Article
Summary
פרוטוקול זה מציג הדמיית החזר אינטרפרומטרי של חלקיק יחיד המיועדת למדידות רב-שכבתיות ומקיפות של גודל שלפוחיות חוץ-תאיות (EV), ספירת EV, פנוטיפ EV וקולוקליזציה של סמן ביולוגי EV.
Abstract
שלפוחיות חוץ-תאיות (EVs) הן שלפוחיות בגודל ננומטר עם שכבת שומנים דו-שכבתית המופרשות על ידי רוב התאים. EVs נושאים מספר רב של מולקולות ביולוגיות שונות, כולל חלבון, שומנים, DNA ו-RNA, ומניחים שהם מקלים על תקשורת תא לתא ברקמות ואיברים מגוונים. לאחרונה, רכבים חשמליים משכו תשומת לב משמעותית כסמנים ביולוגיים לאבחון וחומרים טיפוליים למחלות שונות. שיטות רבות פותחו לאפיון EV. עם זאת, לשיטות הנוכחיות לניתוח EV יש מגבלות שונות. לפיכך, פיתוח שיטות יעילות ואפקטיביות לבידוד ואפיון EV נותר אחד הצעדים המכריעים עבור תחום מחקר חדשני זה ככל שהוא מתבגר. כאן, אנו מספקים פרוטוקול מפורט המתאר חיישן הדמיית החזר אינטרפרומטרי של חלקיק יחיד (SP-IRIS), כשיטה המסוגלת לזהות ולאפיין EVs ממקורות ביולוגיים לא מטוהרים ורכבים חשמליים מטוהרים על ידי מתודולוגיות אחרות. ניתן להשתמש בטכניקה מתקדמת זו למדידות מרובות רמות ומקיפות לניתוח גודל EV, ספירת EV, פנוטיפ EV ולוקליזציה של סמנים ביולוגיים.
Introduction
שלפוחיות חוץ-תאיות (EVs) הן שלפוחיות ממברנה בגודל ננומטר ממקור תאי הניתנות לבידוד מנוזלים ביולוגיים רבים, כולל דם, חלב אם, רוק, שתן, מרה, מיץ לבלב ונוזלי מוח וצפק. גזירת EVs מתרחשת באמצעות שלושה מנגנונים עיקריים: אפופטוזיס, שחרור באמצעות היתוך של גופים רב-שלפוחיתיים עם קרום הפלזמה, ובליבינג של קרום הפלזמה1. עדויות להעברת EV של רכיבי תא תורם לתאים ורקמות שכנים או רחוקים מצביעות על כך שאריזות סגורות ממברנה אלה עשויות למלא תפקידים חשובים במפלי איתות פרקריניים כמו גם למרחקים ארוכים או אנדוקריניים 1,2,3. מכיוון ש-EVs יכולים לספק תמונת מצב של הפנוטיפ של התא, הפוטנציאל לשימוש בהם ככלי אבחון וטיפול לטיפול במחלות שונות הפך לתחום מחקר פעיל 4,5,6,7,8.
פותחו שיטות רבות שמטרתן אפיון EV 9,10,11,12,13. רוב השיטות הללו מספקות מידע ייחודי ובעל ערך על אוכלוסיות של רכבים חשמליים בעיקר בכמויות גדולות. בעוד שתת-קבוצה של טכניקות אלה יכולה לספק פרטים לגבי חומרים בתוך או על רכבים חשמליים בודדים, יכולות להיות מגבלות לאפיון רכבים חשמליים ברמת EV יחיד. לדוגמה, ניתן להשתמש במיקרוסקופ אימונו-אלקטרונים כדי להבין EVs בודדים והרכבם, אך טכניקה זו היא בעלת תפוקה נמוכה, מוגבלת מאוד ביכולתה לשמש לתיאור דינמיקה של אוכלוסייה, ודורשת פיתוח שיטות משמעותיות14.
לאחרונה, פיתוח ומסחור של טכניקת חיישן הדמיית החזר אינטרפרומטרי של חלקיק יחיד (SP-IRIS), באמצעות פלטפורמת ExoView, פתח אפיון EV אינדיבידואלי באמצעות שיטת איסוף נתונים אוטומטית שגרתית ופשוטה. הליבה של טכנולוגיה זו היא השבב, שכבה כפולה Si/SiO2 בגודל 1 ס"מ על 1 ס"מ, המאפשרת מדידה אינטרפרומטרית של ננו-חלקיקים ביולוגיים בודדים. השבב מעובד במיקרו-מערך של נקודות נוגדנים פונקציונליות בודדות, המאפשר זיהוי מרובה של עד שישה סוגי לכידה שונים. שבבים סטנדרטיים כוללים את סמני הטטרה-ספנין הנפוצים (CD81, CD63 ו-CD9) ללכידה במהלך שלב הדגירה, והמשתמש יכול להוסיף נקודות לכידה מותאמות אישית נוספות כדי לבודד אוכלוסיות נפרדות של רכבים חשמליים נפרדים מהטטרה-ספנינים. לאחר שלב הדגירה, כל נקודת לכידה קשרה אליה רכבים חשמליים רבים המבטאים את הסמן המתאים. לאחר מכן ניתן פשוט לשטוף, לייבש ולסרוק את ה-EVs שנלכדו בקורא כדי לכמת את גודל השלפוחיות הקשורות לנקודת הלכידה בין 50-200 ננומטר כדי לתת התפלגות גודל משוקללת באמצעות SP-IRIS15. המערכת מציעה גם שלושה ערוצי זיהוי פלואורסצנטיים לתיוג חיסוני של הרכבים החשמליים שנלכדו, ומספקת הן את עוצמת הפלואורסצנט הממוצעת, שאינה מוגבלת על ידי הגודל כגון מדידות SP-IRIS, והן היבטי קולוקליזציה עבור כל כתם פלואורסצנטי. זה מאפשר למשתמש להגדיר אוכלוסיות של EVs בודדים על סמך תצוגה של ארבעה סמנים ביולוגיים שונים לכל EV (לכידה בתוספת שלוש תוויות אימונו-פלואורסצנטיות). המערכת יכולה לחרוג ממדידת חלבונים על פני השטח עם אימונופלואורסצנטיות, שכן פרוטוקול מטען אופציונלי מאפשר למשתמש לחקור חלבונים פנימיים של ה-EVs שנלכדו ואפיטופים לומינליים של סמני פני השטח המשתרעים על פני הממברנה, כמו גם מאפשר למשתמש לבדוק את תקינות ממברנת EV. במאמר זה, אנו מספקים פרוטוקול מפורט המתאר את הצעדים הדרושים להשגת נתונים עקביים לגבי גודל ומספר EV, עם עד ארבעה סמנים ביולוגיים שונים ברמת EV אחת על אוכלוסיות גדולות של EVs. ניתן להשתמש בטכניקה זו הן על נוזלים ביולוגיים לא מעובדים והן על EVs מבודדים באמצעות מספר רב של טכניקות, כגון אולטרה-צנטריפוגה, אולטרה-סינון, חומרים משקעים, לכידת חיסון, מיקרופלואידיקה וכרומטוגרפיה של אי הכללת גודל.
הפרוטוקול המתואר להלן משתמש בשלפוחיות חוץ-תאיות (EV) שמקורן במדיה של תרבית תאים HEK 293 ומסרום העכבר בשיטת בידוד מבוססת16. הפרוטוקול יושם על נוזלים ביולוגיים רבים אחרים, מדיום תרבית תאים ושלפוחיות חוץ-תאיות מטוהרות המבודדות מנוזלים ביולוגיים. פרוטוקול זה מחולק להליך של יומיים עם זרימת העבודה עבור ניסוי טיפוסי המוצג באיור 1.
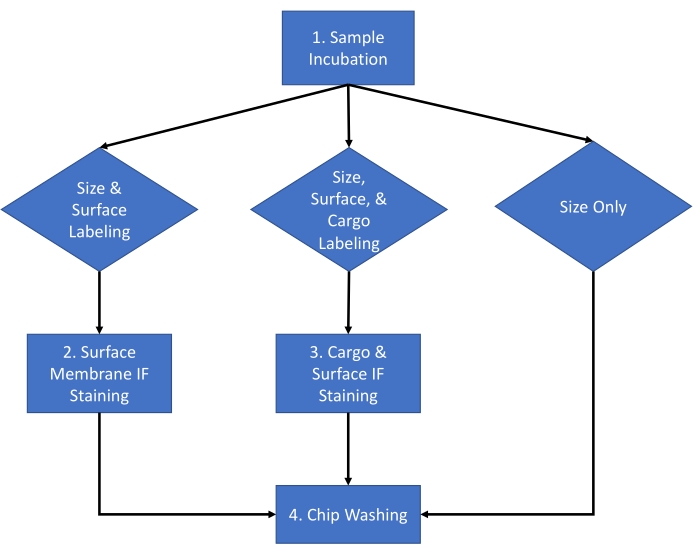
איור 1: זרימת עבודה של בדיקה. זרימת עבודה של בדיקה לבחירת סוג הניתוח שיושלם עבור המדגם בין גודל לספירה, ספירת גודל וכתמי משטח, וספירת גודל וצביעת מטען. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Access restricted. Please log in or start a trial to view this content.
Protocol
דגימות סרום נאספו מעכברים על פי ועדות מוסדיות לטיפול ושימוש בבעלי חיים (IACUC) בפרוטוקול המרכז הרפואי של אוניברסיטת קנזס (KUMC). השימוש בדגימות ביולוגיות אלה בניסויים אלה אושר גם על ידי KUMC.
1. הכנת דגימה (יום 1)
- קבע את ריכוז EV באמצעות מעקב אחר ננו-חלקיקים או טכניקה מקבילה.
- לדלל את הדגימה עם תמיסת הדגירה לריכוז של 5 x 107-5 x 108 EVs/mL; יש צורך במינימום של 50 מיקרוליטר.
הערה: עבור דגימות שבהן ריכוז ה-EV אינו ידוע, ניתן להשתמש בחלבון הכולל ב-1 מיקרוגרם/מ"ל כמדד חלופי. אם הדגימה צפויה להיות בריכוז נמוך, בצע לפחות דילול של 1:1 בתמיסת הדגירה לפני הטעינה. - הניחו צלחת 24 בארות על משטח ישר, ללא רעידות ותנועות גסות.
הערה: כדי לשפר את הניגודיות, ניתן להניח דף נייר לבן מתחת לצלחת. - הוסיפו מים לאזורים שמסביב לבארות (איור 2).
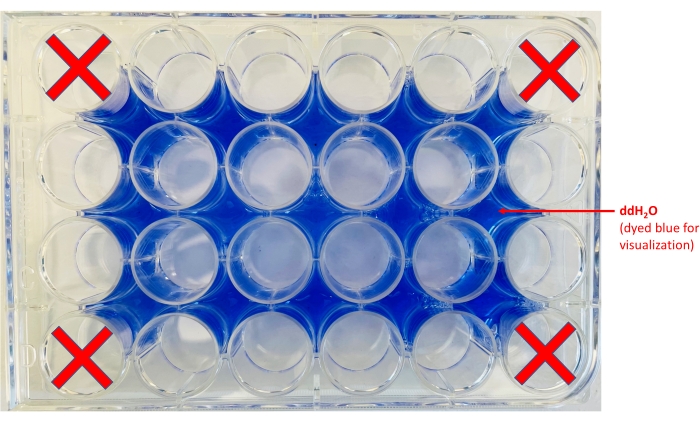
איור 2: פריסת לוחות 24 בארות. מוצגות המיקומים שבהם יש לצרוך ddH2O (צבע כחול נוסף למטרות הדמיה בלבד) והבארות שבהן יוחזקו השבבים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
2. הכנה וסריקה מוקדמת של שבבים
- הסר את הצלחת האטומה של 48 בארות המכילה צ'יפס מהמקרר 4-8 מעלות צלזיוס ואפשר להם להגיע לטמפרטורת החדר (~15 דקות) לפני פתיחת האטם.
הערה: זה חיוני כדי למנוע עיבוי על השבבים שעלול לפגוע בכתמים. אם נצפה עיבוי על פני השטח בעת הוצאת השבב מהאריזה, המתן זמן רב יותר לפני הסרת אחרים. - המשך לשלב 8 כדי לאחזר את הצ'אק כדי להתכונן לריצת טרום-הסריקה (איור 3).
הערה: נתוני הסריקה המוקדמת ישמשו לזיהוי חלקיקים הניתנים לזיהוי בנקודות הלכידה לפני הדגירה עם הדגימה, כך שניתן יהיה להסיר אותם במהלך שלב הניתוח.

איור 3: תמונה של התפסנית המשמשת לטעינת השבב לתוך המכונה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
- בעזרת פינצטה, הסר את מספר השבבים הרצוי (שבב אחד לכל דגימה) מצלחת 48 הבארות וטען את השבבים ישירות לתוך הצ'אק לריצת הסריקה. לאחר שכל השבבים המיועדים לשימוש לניסוי נטענו ונסרקו מראש כמתואר בשלב 8, המשך לשלב 2.4.
הערה: בעת טיפול בשבבים, הקפד לא לגעת בריבועים במרכז, מכיוון שנקודות לכידת הנוגדנים נמצאות באזור זה וייפגעו אם הפינצטה תיגע בהן (איור 4).

איור 4: שבב וטיפול נכון בשבב. (A) קו מקווקו צהוב מציין את מיקום הנוגדנים הנקודים, או את הצד הפונקציונלי של השבב. מזהה השבב ממוקם מתחת לקו ("58"). האיור מראה גם טיפול נכון. (ב) מדגים טיפול לא הולם בשבב. (C) צד לא פונקציונלי של השבב. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
- הנח כל שבב סרוק מראש כשהמשטח הפונקציונלי פונה כלפי מעלה לתוך צלחת 24 הבארות שהוכנה מראש. ניתן לזהות בקלות את המשטח הפונקציונלי על ידי המספור הברור, רשתות היישור ושלוש הקופסאות השחורות באמצע. הצד הלא פונקציונלי הוא משטח אחיד כסוף שטוח.
- השתמש בפינצטה עדינה כדי להבטיח שכל שבב מרוכז בתוך הבאר (איור 5).
הערה: מרכוז השבב בתוך הבאר הוא קריטי מכיוון שאם השבב נוגע בדופן הבאר ניתן לנדף את הדגימה לאחר הטעינה.

איור 5: הדגמה של מיקום נכון של שבבים בבאר. (A) יש להציב שבבים באמצע הבאר, ללא פינות הנוגעות בצידי הבאר. (ב) תיאור של מיקום לא נכון של השבב, כאשר הפינות נוגעות בצידי הבאר. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
3. טעינה ודגירה של השבב והדגימה
- פיפטה 35 מיקרוליטר מהדגימה המוכנה (משלב 1) על השבב.
הערה: היזהר לא להוסיף בועות או לגעת בשבב עם קצה הפיפטה, מכיוון שהוא עלול למנוע פיזור אחיד של הדגימה על השבב או לפגוע בכתמי הנוגדנים על השבב. הדגימה צריכה להתפשט על פני כל משטח השבב. במקרה שהדגימה מנדפת מהשבב במהלך הטעינה, הוסף דגימה נוספת לבאר עד לנפח של 250 מיקרוליטר או הוסף תמיסת דגירה עד 250 מיקרוליטר ושים לב לגורם הדילול שהשתנה עבור אותה דגימה. - אטום את הצלחת באמצעות סרט הצלחת הכלול בערכה כדי למנוע אידוי של הדגימה.
- דגרו את הדגימה/שבב למשך הלילה (~16 שעות) בצלחת האטומה בטמפרטורת החדר באזור נקי מרעידות או תנועה.
הערה: לאחר הדגירה של הלילה, המשתמש יבחר באפשרות המתאימה ליום השני לשאלת המחקר שלו. אם רוצים רק את הגודל והספירה של EV, המשך לשלב 4 (יום 2). אם יש צורך בניתוח סמני שטח מרובים, דלג לשלב 5 (יום 2).
4. קביעת גודל וספירת EV (יום 2)
- הוסף 1 מ"ל של תמיסה A בצד של כל באר המכילה שבב, הקפד לא להוסיף ישירות את התמיסה על השבב או לשרוט את השבב עם קצה הפיפטה.
- הנח את הצלחת על שייקר מסלולי המסתובב במהירות ~500 סל"ד למשך 3 דקות בטמפרטורת החדר.
הערה: אם השבבים מקשקשים על הצלחת, הפחיתו מיד את המהירות כך שהנוזל מסתחרר אך אין רעש. - הסר 750 מיקרוליטר נוזל. הימנע מהטיית הצלחת במהלך הסרת נוזלים כדי למנוע ייבוש מקרי של השבב.
- הוסף 750 מיקרוליטר של תמיסה B בטכניקה המתוארת בשלב 4.1 ונער ב-~500 סל"ד למשך 3 דקות בטמפרטורת החדר.
- חזרו על שלבים 4.3-4.4 פעמיים נוספות ובסך הכל שלוש כביסות עם תמיסה B.
- בתום הכביסה האחרונה, הסר 750 מיקרוליטר מהתמיסה, והשאיר 250 מיקרוליטר של תמיסה B בבאר.
- מוסיפים 750 מיקרוליטר מים מזוקקים כפולים (ddH2O) ומנערים בחום של ~500 סל"ד למשך 3 דקות בטמפרטורת החדר.
- ממלאים צלחת פטרי בגודל 10 ס"מ ב -50 מ"ל ddH2O ומעבירים צ'יפס אחד בכל פעם מהבאר לתבשיל בעזרת פינצטה.
הערה: הקפד להעביר את השבב אופקית וודא שהוא לא מתייבש. ניתן לכבס עד שמונה שבבים לפני החלפת ה-ddH2O. - ב-ddH2O, החזיקו את השבב בקצוות בעזרת פינצטה, וסובבו בצלחת במשך שלוש סיבובים כדי להסיר את הפסולת.
- הסר את השבב בזווית של 45° מחוץ למים והנח את השבב על נייר סופג כשמזהה השבב פונה כלפי מעלה (איור 6).
הערה: צ'יפס מוכן כעת לקריאה. דלג לשלב 8 לסריקה בקורא SP-IRIS.

איור 6: דרך נכונה להוציא את השבב ממי ddH2O בזווית של 45°. (A) מבט מלמעלה ו-(B) מבט מהצד המדגים את הזווית שבה יש להסיר את השבב. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
5. הכנת תמיסת נוגדנים (יום 2)
- הוסף 300 מיקרוליטר של תמיסת חסימה לכל שבב לתוך צינור בגודל מתאים (0.5 עד 3 מ"ל).
- הוסף 0.6 מיקרוליטר נוגדן לכל 300 מיקרוליטר של התמיסה החוסמת. מערבבים בעדינות על ידי הקשה על הצינור וסיבוב מהיר.
הערה: נוגדנים ל-CD9, CD81 ו-CD63 או עכברים CD9, CD81 ו-CD63 כלולים בערכות המתאימות. אם רוצים נוגדנים מצומדים פלואורסצנטיים אחרים, קבע את ריכוז הצביעה האופטימלי באמצעות טיטרציה טיפוסית בטווח של 0.1 עד 10 מיקרוגרם/מ"ל עם טעינת דגימה קבועה כפי שהיית עושה עבור ציטומטריית זרימה.
6. קביעת גודל, ספירה ופנוטיפ EV עם צביעה אימונו-פלואורסצנטית
- הוסף 1 מ"ל של תמיסה A בצד של כל באר המכילה שבב, הקפד לא להוסיף ישירות את התמיסה על השבב או לשרוט את השבב עם קצה הפיפטה.
- הנח את הצלחת על שייקר מסלולי המסתובב במהירות ~500 סל"ד למשך 3 דקות בטמפרטורת החדר.
הערה: אם השבבים מקשקשים על הצלחת, הפחיתו מיד את המהירות כך שהנוזל מסתחרר אך אין רעש. - הסר 750 מיקרוליטר מהנוזל. הימנע מהטיית הצלחת במהלך הסרת נוזלים כדי למנוע ייבוש מקרי של השבב.
- הוסף 750 מיקרוליטר של תמיסה A באמצעות הטכניקה המתוארת בשלב 6.1 ונער ב-~500 סל"ד למשך 3 דקות בטמפרטורת החדר.
- חזור על שלבים 6.3 ו-6.4 פעמיים בסך הכל שלוש כביסות עם פתרון A.
- בתום הכביסה האחרונה, הסר 750 מיקרוליטר מהתמיסה, והשאיר 250 מיקרוליטר של תמיסה A בבאר.
הערה: אם המשתמש מעוניין בכתמי מטען, המשך לשלב 7 בשלב זה. אם לא, המשך לשלב 6.7 - הוסף 250 מיקרוליטר של תמיסת הנוגדנים (שלב 5) לכל שבב בבאר. מכסים את הצלחת בנייר כסף להגנה מפני אור ומנערים למשך שעה אחת על שייקר המסלול בטמפרטורת החדר.
- הוסף 500 מיקרוליטר של תמיסה A, כך שהנפח הכולל לבאר הוא ~1,000 מיקרוליטר.
- הסר מיד 750 מיקרוליטר והוסף 750 מיקרוליטר של תמיסה A ונער ב-~500 סל"ד על שייקר המסלול למשך 3 דקות בטמפרטורת החדר.
- הסר 750 מיקרוליטר מהתמיסה והוסף 750 מיקרוליטר של תמיסה B ונער ב-~500 סל"ד למשך 3 דקות בטמפרטורת החדר.
- חזור על שלב 6.10 פעמיים נוספות בסך הכל שלוש כביסות.
- מוסיפים 750 מיקרוליטר מים מזוקקים כפולים (ddH2O) ומנערים בחום של ~500 סל"ד למשך 3 דקות בטמפרטורת החדר.
- ממלאים צלחת פטרי בגודל 10 ס"מ ב -50 מ"ל ddH2O ומעבירים צ'יפס אחד מהבאר לתבשיל בעזרת פינצטה.
הערה: הקפד להעביר את השבב אופקית וודא שהוא לא מתייבש. ניתן לשטוף עד שמונה צ'יפס לפני ההחלפה ל-ddH2O טרי. - ב-ddH2O, החזיקו את השבב בקצוות בעזרת פינצטה, וסובבו בצלחת במשך שלוש סיבובים כדי להסיר פסולת.
- הסר את השבב בזווית של 45° מהמים והנח את השבב על נייר סופג כשמזהה השבב פונה כלפי מעלה (איור 6).
הערה: צ'יפס מוכן כעת לקריאה. דלג לשלב 8 (איסוף נתונים) לקבלת הוראות כיצד להגדיר את השבבים לסריקה בקורא.
7. מכתים מטען אופציונלי
הערה: פרוטוקול זה מאפשר תיוג בו-זמני של סמנים פנימיים ומשטחיים.
- עם 250 מיקרוליטר של תמיסה A שנותרו בכל באר, הוסף 250 מיקרוליטר של תמיסה C לכל באר.
- הנח מיד על שייקר המסלול והגדר ל-~200 סל"ד למשך 10 דקות בדיוק.
הערה: בשלב זה ובשלב 7.8 להלן, גם תזמון וגם מהירות טלטול איטית יותר הם קריטיים; ודא שהמהירות נותנת רק מערבולת איטית בבארות והשתמש בטיימר. - לאחר 10 דקות של דגירה עם תמיסה C, הוסף 500 מיקרוליטר של תמיסה A לכל באר.
- הסר מיד 750 מיקרוליטר והוסף 750 מיקרוליטר של תמיסה A ונער ב-~500 סל"ד על שייקר המסלול למשך 3 דקות בטמפרטורת החדר.
- חזור על שלב 7.4 פעמיים נוספות בסך הכל שלוש כביסות.
- בתום הכביסה האחרונה, הסר 750 מיקרוליטר מהתמיסה, והשאיר ~ 250 מיקרוליטר מהתמיסה בבאר עם השבב.
- הוסף 250 מיקרוליטר של תמיסה D לכל באר.
- הנח מיד על שייקר המסלול המוגדר ל-~200 סל"ד למשך 10 דקות בדיוק.
- לאחר 10 דקות של דגירה עם תמיסה C, הוסף 500 מיקרוליטר של תמיסה A לכל באר.
- הסר מיד 750 מיקרוליטר והוסף 750 מיקרוליטר של תמיסה A ונער ב-~500 סל"ד על שייקר המסלול למשך 3 דקות בטמפרטורת החדר.
- חזור על שלב 7.10 פעמיים בסך הכל שלוש כביסות.
- הסר 750 מיקרוליטר מהתמיסה לאחר הכביסה האחרונה וחזור לשלב 6.7 לעיל לצביעה והשלמת פרוטוקול הבדיקה.
8. איסוף נתונים
הערה: ההליך לאיסוף נתונים מהשבבים באמצעות ExoView R100 הוא אוטומטי ואינו דורש קלט משתמש. הוראות מפורטות ניתן למצוא במדריך למשתמש ובסרטון המתאים לטעינת נושא השבבים, או "צ'אק", ורכישת נתונים17.
- הפעל את פלטפורמת אפיון ה-EV באמצעות שני מתגי ההפעלה בחלק האחורי של המכשיר, ולאחר מכן הפעל את תוכנת הסורק על ידי לחיצה כפולה על סמל שולחן העבודה.
הערה: כאשר תוכנת הסורק מופעלת, הקורא יוגדר אוטומטית למסך הבית ולאחר מכן יבקש מהמשתמש "פתח את הדלת לטעינת שבבים". - פתח את הדלת בחזית הקורא על ידי הרמת הידית הכסופה. זה יוציא את הבמה, ויאפשר למשתמש גישה לצ'אק ולהגדיר סריקה חדשה בתוכנה.
- זהה את המיקום בו המשתמש מעוניין לשמור את הנתונים על ידי לחיצה על תיקיית השמירה ובחירת מיקום רצוי לשמירת הנתונים.
הערה: לעתים קרובות שימושי לתת למיקום השמירה שם תובנה כמו שם הניסוי, ולאחר מכן ליצור שתי תיקיות משנה, אחת לסריקות מוקדמות ואחת לסריקות שלאחר סריקות. זה מקל על מציאת הנתונים והתאמתם במהלך הניתוח. - כדי לסרוק את השבבים, אתר את קבצי השבבים במחשב הבקרה.
הערה: קבצי שבב הם מפות של פריסת נקודת הנוגדנים על השבב בשימוש ומאפשרים לסורק לדעת היכן לסרוק על כל שבב. בכל ערכה יש מפתח USB עם קבצי השבבים לשבבים בפנים. יש לשמור אותם במיקום במחשב הבקרה עם קבלת הערכה, שם המשתמשים יכולים למצוא אותם באופן מהימן. - טען את השבבים על הצ'אק כשהמספר על השבבים פונה לידית הצ'אק, ולאחר מכן מהתפריט הנפתח של השבב במחשב, בחר כל שבב מרשימת קבצי השבבים והנח אותו במיקום התואם המתאים בצ'אק הווירטואלי בתוכנת הסורק. לאחר השלמתו, תופיע הנחיה על המסך המבקשת מהמשתמש למקם את הצ'אק על הבמה.
- הנח את הצ'אק הטעון על הבמה; היישור המגנטי על הצ'אק יעביר אותו אוטומטית למיקום הנכון על הבמה, ולאחר מכן לחץ על אישור ליד הנח את הצ'אק על הבמה. לאחר מכן הסורק יתחיל בשגרת איסוף הנתונים האוטומטית.
הערה: הנתונים מוכנים לניתוח ברגע שהתוכנה מדווחת על מצב הסריקה של כל שבב כמוצלח.
9. ניתוח נתונים
- לחץ פעמיים על מנתח ExoView בשולחן העבודה של מחשב הבקרה. לאחר אתחול התוכנה, לחץ על כפתור Prescan Data ובחר את מיקום התיקיה עבור מערך הנתונים Prescan בסעיף 8.3.
- לחץ על כפתור נתוני Postscan ובחר את מיקום התיקייה עבור מערך הנתונים לאחר הסריקה בסעיף 8.3.
הערה: אם נתונים מזוהים כראוי בשתי התיקיות, לפחות עבור חלק מהשבבים, התוכנה תציג מספר "X" של שבבים שזוהו ליד כפתור המיקום של Chipfile. כאשר נשמרים כראוי, השבבים שזוהו צריכים להתאים למספר השבבים שנסרקו. - לחץ על הבא בתחתית אזור טעינת הנתונים.
- בתפריט הנפתח של טבלת המטא, לכל שבב יהיה תא. הזן את שמות הדגימה, גורמי הדילול והסמנים המוכתמים בכל ערוץ זיהוי על ידי לחיצה בתיבה המתאימה והקלדת המידע; בסיום, לחץ שוב על הבא .
הערה: מטא-נתונים אלה יישמרו עם הנתונים. - בצע בקרת איכות (QC) על ידי לחיצה על הכרטיסייה השבת מתחת ללחצן QC בפינה השמאלית העליונה.
הערה: תכונה זו מאפשרת לכבות בדיקות ושבבים ספציפיים לצורך ניתוח. התוכנה תספק שתי אזהרות שונות לגבי הנתונים, אחת לספירות גבוהות שיכולות להצביע על כך שלכידה מסוימת רוויה, ומקדם וריאציה גבוה (CV) המזהה מתי אחד מהשכפולים של סוג לכידה שונה מהאחרים.- לחץ על נקודת אזהרה עבור קורות חיים גבוהים, בדוק את הנקודות הנקראות על ידי התוכנה, וראה אם יש נזק פיזי ברור; כאשר הנזק מזוהה, לחץ על מספר הספוט כדי לכבות את הנקודה הזו בניתוח.
- חזור על הפעולה עד להערכת כל האזהרות ולאחר מכן לחץ על הבא.
- בצע ניתוח חיתוך על ידי לחיצה על הכרטיסייה חיתוך הממוקמת ליד הכרטיסייה השבת מתחת לכפתור QC בפינה השמאלית העליונה.
הערה: זה יציג את תגובת הפלואורסצנט של נקודת הבקרה בתרשים, ויציע למשתמש שתי הגדרות, מינימום ומקסימום עבור כל ערוץ צבע (אדום/ירוק/כחול). הגדרת ערך החיתוך עבור כל ערוץ זיהוי פלואורסצנטי היא התאמת הנתונים היחידה שיש לבצע והיא פשוטה יחסית. חשוב לציין, החתך הסופי שנבחר צריך להיות עקבי לאורך הניסוי וכאמור צריך להיות בכללי האצבע עבור 300-400 a.u. באדום וירוק, ו-400-700 a.u. עבור כחול בניסויים טיפוסיים; לתוכנה יש מדריכים מקודדים בצבע למשתמש גם לטווחים אלה.- לחץ על הכרטיסייה צבע ירוק כדי לטעון את הנתונים עבור ערוץ זה ולהציג את החיתוכים הנוכחיים. בבחינת נקודת הבקרה השלילית של האיזוטיפ ניתן לראות אירועי זיהוי עם עוצמות פלואורסצנטיות נמוכות מאוד; במקרה זה, משתמשים צריכים בדרך כלל לצפות להגדיר את הירוק בין 300-400 A.U.
- הגדל את המינימום עבור כל אחד מערוצי הזיהוי עד של-"Avg % Inc" (האחוז הממוצע של החלקיקים שזוהו בנקודת הבקרה הכלולה מעל החיתוך) מתחת לתצוגת הנתונים אין אזהרות כפי שמצוין על ידי הדגשה אדומה או צהובה בתא זה.
הערה: בדרך כלל אין צורך לכוונן את הערך המקסימלי מברירת המחדל, אך ניתן להוריד אותו כדי לסנן חלקיקים בהירים או להגביל את זיהוי הפלואורסצנט לטווח צר יותר. - לחץ על כפתור הבא .
- הגדל את המינימום עבור כל אחד מערוצי הזיהוי עד של-"Avg % Inc" (האחוז הממוצע של החלקיקים שזוהו בנקודת הבקרה הכלולה מעל החיתוך) מתחת לתצוגת הנתונים אין אזהרות כפי שמצוין על ידי הדגשה אדומה או צהובה בתא זה.
- לחץ על הכרטיסייה צבע אדום כדי לטעון את הנתונים עבור ערוץ זה ולהציג את החיתוכים הנוכחיים. שוב, בבחינת נקודת הבקרה השלילית של האיזוטיפ, ניתן לראות אירועי זיהוי עם עוצמות פלואורסצנטיות נמוכות מאוד; במקרה זה, משתמשים צריכים בדרך כלל לצפות להגדיר את האדום בין 300-400 a.u. בצע את אותם הליכים שצוינו בשלבים 9.6.1.1-9.6.1.3.
- לחץ על הכרטיסייה צבע כחול כדי לטעון את הנתונים עבור ערוץ זה ולהציג את החיתוכים הנוכחיים. שוב, בבחינת נקודת הבקרה השלילית של האיזוטיפ, בצע את אותם נהלים שצוינו בשלבים 9.6.1.1-9.6.1.3.
הערה: הערוץ הכחול ייחודי בכך שהנוגדן בנקודה הוא פלואורסצנטי אוטומטי ברמות מספיקות כדי לדרוש חתך מעט גבוה יותר של 400-700 a.u. אפילו עבור שבבים ריקים.
- לחץ על הכרטיסייה צבע ירוק כדי לטעון את הנתונים עבור ערוץ זה ולהציג את החיתוכים הנוכחיים. בבחינת נקודת הבקרה השלילית של האיזוטיפ ניתן לראות אירועי זיהוי עם עוצמות פלואורסצנטיות נמוכות מאוד; במקרה זה, משתמשים צריכים בדרך כלל לצפות להגדיר את הירוק בין 300-400 A.U.
- לאחר השלמת ניתוח ה-Cutoff, לחץ על הבא ותופיע תרשים מפת החום, שייתן סקירה כללית של קשירת החלקיקים על פני הניסוי. ישנם מספר כלי הדמיה שבהם המשתמש יכול להשתמש כדי להתוות את הנתונים. ניתן להוסיף עלילות אלו ונתונים גולמיים משויכים לדוח על ידי לחיצה על כפתור הוסף דוח עלילה .
הערה: מפת החום היא תצוגת הנתונים המוגדרת כברירת מחדל המציגה את כל הדגימות, כל הלכידה view של זיהוי מסוים. תצוגת ברירת המחדל היא הסכום הכולל שמכמת את הרכבים החשמליים הייחודיים הקשורים לכל נקודת לכידה. - לחץ על כפתור ייצוא דוח ובחר מיקום שמירה לדוח.
הערה: הדוח ייפתח בדפדפן. מצא את קובץ הגיליון האלקטרוני של רשימת החלקיקים המסוננים המצורף לדוח; זה המידע שיש להגיש ל-EVTRACK. - המשך להוסיף דוחות באמצעות הבחירות השונות של סוגי הדוגמאות והלכידה בתפריטים הנפתחים בחלק העליון של המסך. הנתונים המוצגים נשלטים על ידי בחירת כפתורי זיהוי הערוצים בפינה השמאלית העליונה. הגדר פנוטיפ/בחירת דגימה/ערוץ לכידה מסוים על ידי לחיצה על כל כפתור צבע כדי לכבות את ערוץ הזיהוי (אפור) או להפעיל (צבעוני).
- לחץ על כפתור הוסף עלילה לדוח כדי לצבור את החלקות הרצויות לדוח.
- לחץ על ייצוא דוח והגדר את מיקום השמירה של הדוח הסופי.
Access restricted. Please log in or start a trial to view this content.
תוצאות
איור 7 (פאנל שמאלי) מציג תמונה מורכבת בשלושה צבעים של רכבי EV שמקורם במדיה מותנית HEK293 הקשורה לנקודת CD63 על השבב ומוכתמת עבור CD81, CD63 ו-CD9 בערוצים הבאים ירוק, אדום וכחול, בהתאמה. איור 7 (הפאנל הימני העליון) הוא תמונה מוגדלת המראה שכל אחד מהרכבים הח...
Access restricted. Please log in or start a trial to view this content.
Discussion
שיטות האפיון הנוכחיות של EV מסתמכות במידה רבה על EVs מטוהרים, אשר מוגבלים על ידי המגבלות הניסיוניות הנוכחיות של שיטות טיהור EV 9,10,11,12,13. הדמיית החזר אינטרפרומטרי של חלקיק יחיד (SP-IRIS) היא ...
Access restricted. Please log in or start a trial to view this content.
Disclosures
קלייטון דייגן וג'ורג' דאבול הם עובדים ובעלי מניות ב-NanoView Biosciences Inc.
Acknowledgements
עבודה זו מומנה בחלקה על ידי בית הספר לרפואה של אוניברסיטת קנזס, תוכנית פרסי ציוד מחקר ורכש משאבים. PCG, LKC, FD ו-AR נתמכו במימון NIA R21 AG066488-01.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 10-cm sterile Petri dish | Fisher | FB0875712 | |
| 15mL sterile tube | n/a | various | |
| 24-well cell culture plate, flat bottom | Fisher | 08-772-1 | |
| Blocking Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chipfiles | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chips | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chuck | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 250 ml | Fisher | 09-761-4 | |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 500 ml | Fisher | 09-761-10 | |
| Deionized (DI) water | Fisher | LC267404 | |
| EMS style tweezers with Carbon Fiber tips | Fisher | 50-193-0842 | |
| ExoView Human Tetraspanin Kit | NanoView Biosciences | EV-TETRA-C | Capture for hCD81, hCD9, hCD63, IgG Control + stains for hEV-A (hEV-CD63-647, hEV-CD81-555, hEV-CD9-488) 16 Chips per kit |
| ExoView R100 Imager | NanoView Biosciences | EV-R100 | Interferometric microscope including high specification camera including 3 color fluorescence and label free sizing and counting extracellular vesicles |
| Fluorescently labled huma CD9 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD63 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD81 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Incubation Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Orbital shaker or microplate shaker with digital settings capable of shaking at 500 rpm | n/a | various | |
| Plate Seal | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution A | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution B | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution C | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution D | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Square/flat tip tweezer | Fisher | 50-239-62 | |
| Straight strong point Boley style tweezers | Fisher | 16-100-124 | |
| Thermo Scientific Adhesive PCR Plate Seals | Fisher | AB-0558 |
References
- Maas, S. L. N., Breakefield, X. O., Weaver, A. M. Extracellular vesicles: Unique intercellular delivery vehicles. Trends in Cell Biology. 27 (3), 172-188 (2017).
- Shah, R., Patel, T., Freedman, J. E. Circulating extracellular vesicles in human disease. The New England Journal of Medicine. 379 (10), 958-966 (2018).
- Deng, F., Miller, J. A review on protein markers of exosome from different bio-resources and the antibodies used for characterization. Journal of Histotechnology. 42 (4), 226-239 (2019).
- Cohen, O., et al. ’Golden’ exosomes as delivery vehicles to target tumors and overcome intratumoral barriers: in vivo tracking in a model for head and neck cancer. Biomaterials Science. 9 (6), 2103-2114 (2021).
- Han, Y., et al. Overview and update on methods for cargo loading into extracellular vesicles. Processes (Basel). 9 (2), 356(2021).
- Lee, M., Im, W., Kim, M. Exosomes as a potential messenger unit during heterochronic parabiosis for amelioration of Huntington's disease. Neurobiology of Disease. 155, 105374(2021).
- Sun, B., et al. Characterization and biomarker analyses of post-COVID-19 complications and neurological manifestations. Cells. 10 (2), 386(2021).
- Jiang, L., Gu, Y., Du, Y., Liu, J. Exosomes: Diagnostic biomarkers and therapeutic delivery vehicles for cancer. Molecular Pharmaceutics. 16 (8), 3333-3349 (2019).
- Bachurski, D., et al. Extracellular vesicle measurements with nanoparticle tracking analysis - An accuracy and repeatability comparison between NanoSight NS300 and ZetaView. Journal of Extracellular Vesicles. 8 (1), 1596016(2019).
- Carmicheal, J., et al. Label-free characterization of exosome via surface enhanced Raman spectroscopy for the early detection of pancreatic cancer. Nanomedicine. 16, 88-96 (2019).
- Greening, D. W., Xu, R., Ji, H., Tauro, B. J., Simpson, R. J. A protocol for exosome isolation and characterization: evaluation of ultracentrifugation, density-gradient separation, and immunoaffinity capture methods. Methods in Molecular Biology. 1295, 179-209 (2015).
- Wu, Y., Deng, W., Klinke, D. J. Exosomes: improved methods to characterize their morphology, RNA content, and surface protein biomarkers. The Analyst. 140 (19), 6631-6642 (2015).
- van de Vlekkert, D., Qiu, X., Annunziata, I., d'Azzo, A. Isolation and characterization of exosomes from skeletal muscle fibroblasts. Journal of Visualized Experiments: JoVE. (159), (2020).
- Ayala-Mar, S., Donoso-Quezada, J., Gallo-Villanueva, R. C., Perez-Gonzalez, V. H., Gonzalez-Valdez, J. Recent advances and challenges in the recovery and purification of cellular exosomes. Electrophoresis. 40 (23-24), 3036-3049 (2019).
- Daaboul, G. G., et al. Digital detection of exosomes by interferometric imaging. Scientific Reports. 6, 37246(2016).
- Pohler, K. G., et al. Circulating microRNA as candidates for early embryonic viability in cattle. NMolecular Reproduction and Development. 84 (8), 731-743 (2017).
- NanoView Biosciences. ExoView R100 User Guide. v240.4. NanoView Biosciences. , 202(2021).
- Daaboul, G. G., et al. Enhanced light microscopy visualization of virus particles from Zika virus to filamentous ebolaviruses. PLoS One. 12, 0179728(2017).
- ET Consortium. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14, 228-232 (2017).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved