È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Caratterizzazione dell'imaging interferometrico a riflettanza a singola particella di singole vescicole extracellulari e dinamica di popolazione
In questo articolo
Riepilogo
Questo protocollo presenta l'imaging interferometrico a riflettanza a singola particella progettato per le misurazioni multilivello e complete delle dimensioni delle vescicole extracellulari (EV), della conta EV, del fenotipo EV e della colocalizzazione dei biomarcatori EV.
Abstract
Le vescicole extracellulari (EV) sono vescicole di dimensioni nanometriche con un doppio strato lipidico che vengono secrete dalla maggior parte delle cellule. Le vescicole extracellulari trasportano una moltitudine di molecole biologiche diverse, tra cui proteine, lipidi, DNA e RNA, e si ipotizza che facilitino la comunicazione da cellula a cellula in diversi tessuti e organi. Recentemente, le vescicole extracellulari hanno attirato un'attenzione significativa come biomarcatori per la diagnostica e gli agenti terapeutici per varie malattie. Sono stati sviluppati molti metodi per la caratterizzazione delle vescicole extracellulari. Tuttavia, i metodi attuali per l'analisi delle EV hanno tutti limiti diversi. Pertanto, lo sviluppo di metodi efficienti ed efficaci per l'isolamento e la caratterizzazione delle vescicole extracellulari rimane uno dei passi cruciali per questo campo di ricerca all'avanguardia man mano che matura. Qui, forniamo un protocollo dettagliato che delinea un sensore di imaging interferometrico a riflettanza a singola particella (SP-IRIS), come metodo in grado di rilevare e caratterizzare le vescicole extracellulari da fonti biologiche non purificate e le vescicole extracellulari purificate con altre metodologie. Questa tecnica avanzata può essere utilizzata per misurazioni multilivello e complete per l'analisi delle dimensioni delle vescicole extracellulari, della conta delle vescicole extracellulari, del fenotipo delle vescicole extracellulari e della colocalizzazione dei biomarcatori.
Introduzione
Le vescicole extracellulari (EV) sono vescicole di membrana di dimensioni nanometriche di origine cellulare che possono essere isolate da numerosi fluidi biologici, tra cui sangue, latte materno, saliva, urina, bile, succo pancreatico e liquidi cerebrospinali e peritoneali. La derivazione delle vescicole extracellulari avviene attraverso tre meccanismi principali: apoptosi, rilascio attraverso la fusione di corpi multivescicolari con la membrana plasmatica e blebbing della membrana plasmatica1. L'evidenza del trasferimento di EV di componenti cellulari di donatori a cellule e tessuti vicini o distanti suggerisce che questi pacchetti racchiusi nella membrana possono svolgere un ruolo importante nelle cascate di segnalazione paracrina e a lunga distanza o endocrina 1,2,3. Poiché le vescicole extracellulari possono fornire un'istantanea del fenotipo di una cellula, il potenziale per il loro utilizzo come strumenti diagnostici e terapeutici per il trattamento di varie malattie è diventato un'area attiva di ricerca 4,5,6,7,8.
Sono stati sviluppati molti metodi finalizzati alla caratterizzazione delle EV 9,10,11,12,13. La maggior parte di questi metodi fornisce informazioni uniche e preziose sulle popolazioni di vescicole extracellulari principalmente in massa. Mentre un sottoinsieme di queste tecniche può fornire dettagli sulle sostanze all'interno o su singole vescicole extracellulari, ci possono essere limitazioni alla caratterizzazione delle vescicole extracellulari a livello di singola vescicola extravenosa. Ad esempio, la microscopia immunoelettronica può essere utilizzata per comprendere le singole vescicole extracellulari e la loro composizione, ma questa tecnica è a bassa produttività, fortemente limitata nella sua capacità di essere utilizzata per descrivere la dinamica della popolazione e richiede lo sviluppo di metodi significativi14.
Recentemente, lo sviluppo e la commercializzazione della tecnica del sensore di imaging interferometrico a riflettanza a singola particella (SP-IRIS), tramite la piattaforma ExoView, ha aperto la caratterizzazione delle singole vescicole extracellulari utilizzando un metodo di raccolta dati automatizzato semplice e di routine. Il cuore di questa tecnologia è il chip, un doppio strato Si/SiO2 di 1 cm x 1 cm, che consente la misurazione interferometrica di singole nanoparticelle biologiche. Il chip è lavorato con un microarray di singoli spot di anticorpi funzionalizzati, consentendo il rilevamento multiplex di un massimo di sei diversi tipi di cattura. I chip standard includono i comuni marcatori di tetraspanina (CD81, CD63 e CD9) per la cattura durante la fase di incubazione e l'utente può aggiungere ulteriori punti di cattura personalizzati per isolare popolazioni distinte di vescicole extracellulari separate dalle tetraspanine. Dopo la fase di incubazione, ogni punto di cattura ha legato ad esso molte vescicole extracellulari che esprimono il marcatore corrispondente. Queste vescicole extracellulari catturate possono quindi essere semplicemente lavate, asciugate e scansionate nel lettore per quantificare le dimensioni delle vescicole legate al punto di cattura tra 50 e 200 nm per fornire una distribuzione dimensionale ponderata tramite SP-IRIS15. Il sistema offre anche tre canali di rilevamento fluorescenti per l'immunomarcatura delle vescicole extracellulari catturate e fornisce sia l'intensità media della fluorescenza, che non è limitata dalle dimensioni come le misurazioni SP-IRIS, sia gli aspetti di colocalizzazione per ogni colorazione fluorescente. Ciò consente all'utente di definire popolazioni di singole vescicole extracellulari in base alla visualizzazione di quattro diversi biomarcatori per vescicola extracellulare (cattura più tre etichette immunofluorescenti). Il sistema può andare oltre la misurazione delle proteine di superficie con l'immunofluorescenza, poiché un protocollo di carico opzionale consente all'utente di sondare le proteine interne delle vescicole extracellulari catturate e gli epitopi luminali dei marcatori di superficie che attraversano la membrana, oltre a consentire all'utente di verificare l'integrità della membrana delle vescicole extracellulari. In questo articolo, forniamo un protocollo dettagliato che delinea i passaggi necessari per ottenere dati coerenti riguardo alle dimensioni e al numero di EV, con un massimo di quattro diversi biomarcatori a livello di EV su grandi popolazioni di EV. Questa tecnica può essere utilizzata sia su fluidi biologici non trattati che su vescicole extracellulari isolate utilizzando un numero qualsiasi di tecniche, come l'ultracentrifugazione, l'ultrafiltrazione, gli agenti precipitanti, la cattura di immunoaffinità, la microfluidica e la cromatografia ad esclusione dimensionale.
Il protocollo descritto di seguito utilizza vescicole extracellulari (EV) derivate da terreni di coltura cellulare HEK 293 e dal siero di topo utilizzando un metodo di isolamento stabilito16. Il protocollo è stato applicato a numerosi altri fluidi biologici, terreni di coltura cellulare e vescicole extracellulari purificate isolate da fluidi biologici. Questo protocollo è suddiviso in una procedura di due giorni con il flusso di lavoro per un tipico esperimento mostrato nella Figura 1.
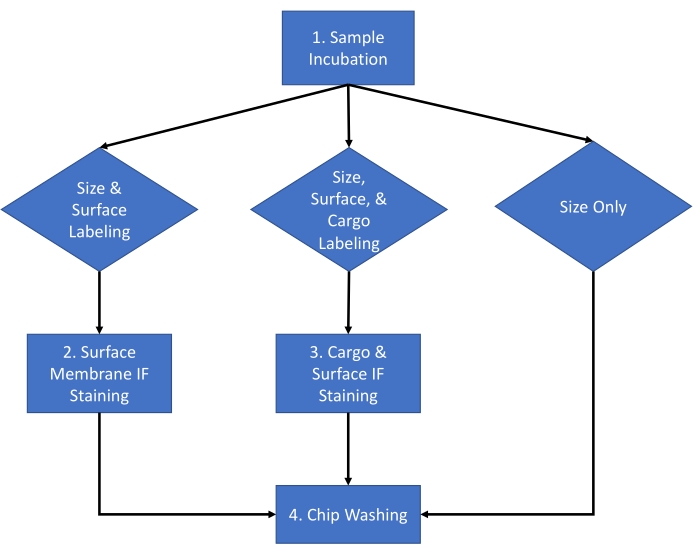
Figura 1: Flusso di lavoro del saggio. Flusso di lavoro del saggio per la scelta del tipo di analisi da completare per il campione tra dimensione e conteggio, conteggio delle dimensioni e colorazione della superficie, conteggio delle dimensioni e colorazione del carico. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
I campioni di siero sono stati raccolti dai topi secondo un protocollo approvato dai comitati istituzionali per la cura e l'uso degli animali (IACUC) presso il Centro medico dell'Università del Kansas (KUMC). L'uso di questi campioni biologici in questi esperimenti è stato approvato anche dal KUMC.
1. Preparazione del campione (giorno 1)
- Determinare la concentrazione di EV utilizzando il tracciamento delle nanoparticelle o una tecnica equivalente.
- Diluire il campione con la soluzione di incubazione ad una concentrazione di 5 x 107-5 x 108 EV/mL; è necessario un minimo di 50 μl.
NOTA: Per i campioni in cui la concentrazione di EV è sconosciuta, le proteine totali a 1 μg/mL possono essere utilizzate come misura sostitutiva. Se si prevede che il campione sia a bassa concentrazione, eseguire almeno una diluizione 1:1 nella soluzione di incubazione prima del caricamento. - Posizionare una piastra a 24 pozzetti su una superficie piana, priva di vibrazioni e movimenti bruschi.
NOTA: Per migliorare il contrasto, è possibile posizionare un foglio di carta bianco sotto la lastra. - Aggiungere acqua alle aree circostanti i pozzi (Figura 2).
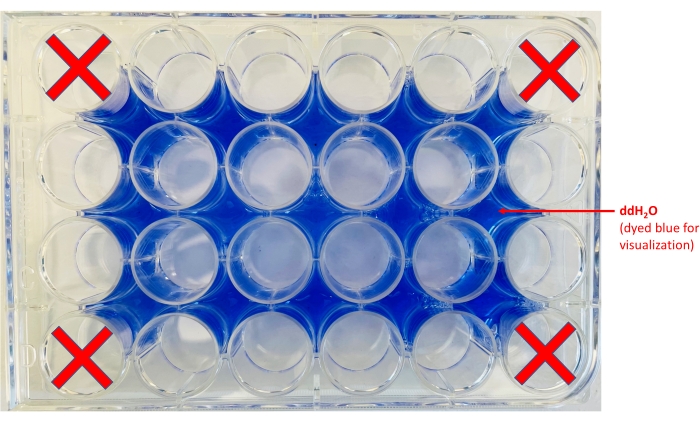
Figura 2: Layout della piastra a 24 pozzetti. Vengono mostrate le posizioni in cui aliquotare ddH2O (il colorante blu è stato aggiunto solo a scopo di visualizzazione) e i pozzetti in cui verranno conservati i chip. Clicca qui per visualizzare una versione più grande di questa figura.
2. Preparazione e prescansione dei chip
- Rimuovere la piastra sigillata a 48 pozzetti contenente le patatine dal frigorifero a 4-8 °C e lasciarle raggiungere la temperatura ambiente (~15 min) prima di aprire la guarnizione.
NOTA: Questo è essenziale per evitare la formazione di condensa sui trucioli che può danneggiare le macchie. Se si nota della condensa sulla superficie dopo aver rimosso il chip dalla confezione, attendere più a lungo prima di rimuoverne altri. - Procedere al passaggio 8 per recuperare il mandrino per prepararsi alla corsa pre-scansione (Figura 3).
NOTA: I dati pre-scansione verranno utilizzati per identificare eventuali particelle rilevabili sui punti di cattura prima dell'incubazione con il campione, in modo che possano essere rimosse durante la fase di analisi.

Figura 3: Immagine del mandrino utilizzato per caricare il truciolo nella macchina. Clicca qui per visualizzare una versione più grande di questa figura.
- Utilizzando una pinzetta, rimuovere il numero desiderato di trucioli (un truciolo per campione) dalla piastra a 48 pozzetti e caricare i trucioli direttamente nel mandrino per l'esecuzione prescan. Quando tutti i chip che si intende utilizzare per l'esperimento sono stati caricati e sottoposti a scansione preliminare come descritto nel passaggio 8, procedere al passaggio 2.4.
NOTA: Quando si maneggiano i chip, assicurarsi di non toccare i quadrati al centro, poiché i punti di cattura degli anticorpi si trovano in questa regione e si danneggiano se toccati dalla pinzetta (Figura 4).

Figura 4: Chip e corretta manipolazione del chip. (A) La linea tratteggiata gialla indica la posizione degli anticorpi macchiati o il lato funzionale del chip. L'ID del chip si trova sotto la riga ("58"). La figura mostra anche la corretta manipolazione. (B) Dimostra una manipolazione inappropriata del chip. (C) Lato non funzionante del chip. Clicca qui per visualizzare una versione più grande di questa figura.
- Posizionare ogni chip pre-scansionato con la superficie funzionalizzata rivolta verso l'alto nella piastra a 24 pozzetti pre-preparata. La superficie funzionalizzata è facilmente riconoscibile dalla numerazione distinta, dalle griglie di allineamento e dalle tre caselle nere al centro. Il lato non funzionalizzato è una superficie piatta e uniforme in argento.
- Utilizzare una pinzetta a punta fine per assicurarsi che ogni chip sia centrato all'interno del pozzetto (Figura 5).
NOTA: Centrare il chip all'interno del pozzetto è fondamentale perché se il chip tocca la parete del pozzetto, il campione può essere espulso dopo il caricamento.

Figura 5: Dimostrazione del corretto posizionamento dei trucioli nel pozzo. (A) I trucioli devono essere posizionati al centro del pozzo, senza che gli angoli tocchino i lati del pozzo. (B) Rappresentazione di un posizionamento improprio del chip, dove gli angoli toccano i lati del pozzo. Clicca qui per visualizzare una versione più grande di questa figura.
3. Caricamento e incubazione del chip e del campione
- Pipettare 35 μl del campione preparato (dalla fase 1) sul chip.
NOTA: Fare attenzione a non aggiungere bolle o toccare il chip con la punta della pipetta, poiché può impedire la distribuzione uniforme del campione sul chip o danneggiare le macchie di anticorpi sul chip. Il campione dovrebbe diffondersi su tutta la superficie del chip. Nel caso in cui il campione si stacchi dal truciolo durante il caricamento, aggiungere ulteriore campione al pozzetto fino a un volume di 250 μl o aggiungere una soluzione di incubazione fino a 250 μl e annotare il fattore di diluizione modificato per quel campione. - Sigillare la piastra, utilizzando la pellicola per piastre inclusa nel kit per evitare l'evaporazione del campione.
- Incubare il campione/chip per una notte (~16 h) nella piastra sigillata a temperatura ambiente in un'area priva di vibrazioni o movimenti.
NOTA: Dopo l'incubazione notturna, l'utente sceglierà l'opzione del Giorno 2 appropriata per la sua domanda di ricerca. Se si desidera solo la dimensione e il conteggio delle EV, procedere con il passaggio 4 (Giorno 2). Se si desidera l'analisi di più marcatori di superficie, andare al passaggio 5 (Giorno 2).
4. Determinazione delle dimensioni e del conteggio delle EV (Giorno 2)
- Aggiungere 1 mL di soluzione A sul lato di ciascun pozzetto contenente un chip, facendo attenzione a non aggiungere direttamente la soluzione sul chip o a non graffiare il chip con la punta della pipetta.
- Posizionare la piastra su un agitatore orbitale che ruota a ~500 giri/min per 3 minuti a temperatura ambiente.
NOTA: Se le patatine sbattono sulla piastra, diminuire immediatamente la velocità in modo che il liquido turbini ma non ci siano tintinnii. - Rimuovere 750 μl di liquido. Evitare di inclinare la piastra durante la rimozione del liquido per evitare l'essiccazione accidentale del truciolo.
- Aggiungere 750 μl di soluzione B utilizzando la tecnica descritta al punto 4.1 e agitare a ~500 giri/min per 3 minuti a temperatura ambiente.
- Ripetere i passaggi 4.3-4.4 altre due volte per un totale di tre lavaggi con la soluzione B.
- Al termine dell'ultimo lavaggio, rimuovere 750 μL della soluzione, lasciando 250 μL di Soluzione B nel pozzetto.
- Aggiungere 750 μl di acqua distillata doppia (ddH2O) e agitare a ~500 giri/min per 3 minuti a temperatura ambiente.
- Riempire una capsula di Petri da 10 cm con 50 ml di ddH2O e trasferire una fiches alla volta dal pozzetto nella piastra utilizzando una pinzetta.
NOTA: Fare attenzione a trasferire il truciolo orizzontalmente e assicurarsi che non si asciughi. È possibile lavare fino a otto trucioli prima di sostituire il ddH2O. - Nel ddH2O, tenere il chip per i bordi usando una pinzetta e agitarlo nel piatto per tre giri per rimuovere i detriti.
- Rimuovere il chip con un angolo di 45° fuori dall'acqua e posizionarlo su carta assorbente con l'ID del chip rivolto verso l'alto (Figura 6).
NOTA: Le fiches sono ora pronte per essere lette. Andare al passaggio 8 per la scansione sul lettore SP-IRIS.

Figura 6: Modo corretto per rimuovere il truciolo dall'acqua ddH2O con un angolo di 45°. (A) Vista dall'alto e (B) Vista laterale che dimostra l'angolo con cui rimuovere il truciolo. Clicca qui per visualizzare una versione più grande di questa figura.
5. Preparazione della soluzione anticorpale (Giorno 2)
- Aggiungere 300 μl di soluzione bloccante per chip in una provetta di dimensioni adeguate (da 0,5 a 3 mL).
- Aggiungere 0,6 μl di anticorpo per 300 μl di soluzione bloccante. Mescolare delicatamente picchiettando il tubo e girare rapidamente.
NOTA: Gli anticorpi per CD9, CD81 e CD63 umani o CD9, CD81 e CD63 murini sono inclusi nei kit appropriati. Se si desiderano altri anticorpi coniugati fluorescenti, determinare la concentrazione di colorazione ottimale utilizzando una titolazione tipica in un intervallo compreso tra 0,1 e 10 μg/mL con un carico costante del campione come si farebbe per la citometria a flusso.
6. Determinazione delle dimensioni, del conteggio e della fenotipizzazione delle EV con la colorazione immunofluorescente
- Aggiungere 1 mL di soluzione A sul lato di ciascun pozzetto contenente un chip, facendo attenzione a non aggiungere direttamente la soluzione sul chip o a non graffiare il chip con la punta della pipetta.
- Posizionare la piastra su un agitatore orbitale che ruota a ~500 giri/min per 3 minuti a temperatura ambiente.
NOTA: Se le patatine sbattono sulla piastra, diminuire immediatamente la velocità in modo che il liquido turbini ma non ci siano tintinnii. - Rimuovere 750 μl di liquido. Evitare di inclinare la piastra durante la rimozione del liquido per evitare l'essiccazione accidentale del truciolo.
- Aggiungere 750 μL di soluzione A utilizzando la tecnica descritta al punto 6.1 e agitare a ~500 giri/min per 3 minuti a temperatura ambiente.
- Ripetere due volte i passaggi 6.3 e 6.4 per un totale di tre lavaggi con la soluzione A.
- Al termine dell'ultimo lavaggio, rimuovere 750 μL della soluzione, lasciando 250 μL di soluzione A nel pozzetto.
NOTA: Se l'utente desidera la colorazione del carico, procedere al passaggio 7 a questo punto. In caso contrario, procedere al passaggio 6.7 - Aggiungere 250 μl della soluzione anticorpale (fase 5) a ciascun chip nel pozzetto. Coprire la piastra con un foglio di alluminio per proteggerla dalla luce e agitare per 1 ora sull'agitatore orbitale a temperatura ambiente.
- Aggiungere 500 μl di soluzione A, in modo che il volume totale per pozzetto sia ~1.000 μl.
- Rimuovere immediatamente 750 μL e aggiungere 750 μL di soluzione A e agitare a ~500 giri/min sull'agitatore orbitale per 3 minuti a temperatura ambiente.
- Rimuovere 750 μl della soluzione e aggiungere 750 μl di soluzione B e agitare a ~500 giri/min per 3 minuti a temperatura ambiente.
- Ripetere il passaggio 6.10 altre due volte per un totale di tre lavaggi.
- Aggiungere 750 μl di acqua distillata doppia (ddH2O) e agitare a ~500 giri/min per 3 minuti a temperatura ambiente.
- Riempire una capsula di Petri da 10 cm con 50 ml di ddH2O e trasferire una fiocca dal pozzetto nella piastra utilizzando una pinzetta.
NOTA: Fare attenzione a trasferire il truciolo orizzontalmente e assicurarsi che non si asciughi. È possibile lavare fino a otto trucioli prima di essere sostituiti con ddH2O freschi. - Nel ddH2O, tenere il chip per i bordi usando una pinzetta e far roteare il piatto per tre giri per rimuovere i detriti.
- Rimuovere il chip con un angolo di 45° fuori dall'acqua e posizionarlo su carta assorbente con l'ID del chip rivolto verso l'alto (Figura 6).
NOTA: Le fiches sono ora pronte per essere lette. Andare al passaggio 8 (Raccolta dati) per istruzioni su come configurare i chip per la scansione sul lettore.
7. Colorazione del carico opzionale
NOTA: Questo protocollo consente l'etichettatura simultanea di marcatori interni e di superficie.
- Con 250 μL di soluzione A rimanenti in ciascun pozzetto, aggiungere 250 μL di soluzione C in ciascun pozzetto.
- Posizionare immediatamente sull'agitatore orbitale e impostare a ~200 giri/min per esattamente 10 minuti.
NOTA: In questo passaggio e nel passaggio 7.8 di seguito, sia il cronometraggio che la velocità di agitazione più lenta sono fondamentali; Assicurati che la velocità dia solo un lento vortice nei pozzi e usa un timer. - Dopo 10 minuti di incubazione con la soluzione C, aggiungere 500 μl di soluzione A a ciascun pozzetto.
- Rimuovere immediatamente 750 μl e aggiungere 750 μl di soluzione A e agitare a ~500 giri/min sull'agitatore orbitale per 3 minuti a temperatura ambiente.
- Ripetere il passaggio 7.4 altre due volte per un totale di tre lavaggi.
- Al termine dell'ultimo lavaggio, rimuovere 750 μL della soluzione, lasciando ~250 μL della soluzione nel pozzetto con il chip.
- Aggiungere 250 μl di soluzione D a ciascun pozzetto.
- Posizionare immediatamente sull'agitatore orbitale impostato a ~200 giri/min per esattamente 10 minuti.
- Dopo 10 minuti di incubazione con la soluzione C, aggiungere 500 μl di soluzione A a ciascun pozzetto.
- Rimuovere immediatamente 750 μL e aggiungere 750 μL di soluzione A e agitare a ~500 giri/min sull'agitatore orbitale per 3 minuti a temperatura ambiente.
- Ripetere il passaggio 7.10 due volte per un totale di tre lavaggi.
- Rimuovere 750 μl di soluzione dopo l'ultimo lavaggio e tornare al passaggio 6.7 precedente per la colorazione e il completamento del protocollo di analisi.
8. Raccolta dei dati
NOTA: La procedura per la raccolta dei dati dai chip utilizzando ExoView R100 è automatizzata e non richiede alcun input da parte dell'utente. Istruzioni dettagliate sono disponibili nella Guida per l'utente e nel video corrispondente per il caricamento del portatrucioli, o "mandrino", e l'acquisizione dei dati17.
- Accendere la piattaforma di caratterizzazione EV utilizzando i due interruttori di alimentazione sul retro dello strumento, quindi avviare il software dello scanner facendo doppio clic sull'icona del desktop.
NOTA: All'avvio del software dello scanner, il lettore passerà automaticamente alla schermata iniziale, quindi chiederà all'utente di "Aprire lo sportello per caricare i chip". - Aprire lo sportello sulla parte anteriore del lettore sollevando la maniglia argentata. Questo espellerà il tavolino, consentendo all'utente di accedere al mandrino e impostare una nuova scansione nel software.
- Identificare la posizione in cui l'utente desidera salvare i dati facendo clic sulla cartella Salva e selezionando la posizione desiderata per i dati da salvare.
NOTA: Spesso è utile assegnare alla posizione di salvataggio un nome approfondito come il nome dell'esperimento, quindi creare due sottocartelle, una per le pre-scansioni e una per le post-scansioni. Ciò semplifica la ricerca e l'abbinamento dei dati durante l'analisi. - Per eseguire la scansione dei chip, individuare i chipfile sul computer di controllo.
NOTA: I chipfile sono mappe della disposizione dei punti dell'anticorpo sul chip in uso e consentono allo scanner di sapere dove eseguire la scansione su ciascun chip. In ogni kit è presente una chiavetta USB con all'interno i file dei chip. Questi devono essere salvati in una posizione sul computer di controllo al ricevimento del kit in cui gli utenti possano trovarli in modo affidabile. - Caricare i chip sul mandrino con il numero sui chip rivolto verso l'impugnatura del mandrino, quindi dal menu a discesa Chip sul computer, selezionare ciascun chip dall'elenco dei chipfile e posizionarlo nella posizione corrispondente appropriata nel mandrino virtuale nel software dello scanner. Una volta completato, apparirà un messaggio sullo schermo che chiede all'utente di posizionare il mandrino sul palco.
- Posizionare il mandrino carico sul tavolino; l'allineamento magnetico sul mandrino lo sposterà automaticamente nella posizione corretta sul tavolino, quindi fare clic su OK accanto a Posiziona il mandrino sul tavolino. Lo scanner inizierà quindi la routine di raccolta automatica dei dati.
NOTA: I dati sono pronti per essere analizzati una volta che il software segnala lo stato di scansione di ciascun chip come riuscito.
9. Analisi dei dati
- Fare doppio clic su ExoView Analyzer sul desktop del PC di controllo. Dopo l'avvio del software, fare clic sul pulsante Prescan Data e selezionare la posizione della cartella per il set di dati di prescan nella sezione 8.3.
- Fare clic sul pulsante Dati postscan e selezionare la posizione della cartella per il set di dati postscan nella sezione 8.3.
NOTA: Se i dati vengono rilevati correttamente in entrambe le cartelle, almeno per alcuni chip, il software visualizzerà il numero "X" di chip rilevati accanto al pulsante di posizione Chipfile. Se salvati correttamente, i chip rilevati dovrebbero corrispondere al numero di chip scansionati. - Fare clic su Avanti nella parte inferiore dell'area di caricamento dei dati.
- Nel menu a tendina della meta tabella, ogni avrà una cella. Inserire i nomi dei campioni, i fattori di diluizione e i marcatori che vengono colorati in ciascun canale di rilevamento facendo clic nella casella corrispondente e digitando le informazioni; al termine, fare nuovamente clic su Avanti .
NOTA: questi metadati verranno salvati con i dati. - Controllo qualità delle preforme (QC) facendo clic sulla scheda Disabilita sotto il pulsante QC in alto a sinistra.
NOTA: Questa funzione consente di disattivare sonde e chip specifici per l'analisi. Il software fornirà due diversi avvisi relativi ai dati, uno per conteggi elevati che possono indicare che una particolare cattura è satura, e un alto coefficiente di variazione (CV) che identifica quando una delle repliche di un tipo di cattura è diversa dalle altre.- Fare clic su un punto di avviso per un CV elevato, esaminare i punti che vengono richiamati dal software e vedere se ci sono evidenti danni fisici; quando il danno viene identificato, fare clic sul numero del punto per disattivare quel punto nell'analisi.
- Ripetere l'operazione fino a quando tutti gli avvisi sono stati valutati, quindi fare clic su Avanti.
- Preformare l'analisi del taglio facendo clic sulla scheda Taglio situata accanto alla scheda Disabilita sotto il pulsante QC in alto a sinistra.
NOTA: Questo presenterà la risposta fluorescente del punto di controllo in un grafico e offrirà all'utente due impostazioni, un minimo e un massimo per ciascun canale di colore (rosso/verde/blu). L'impostazione del valore di cutoff per ciascun canale di rilevamento fluorescente è l'unica regolazione dei dati che deve essere effettuata ed è relativamente semplice. È importante sottolineare che il cutoff finale scelto dovrebbe essere coerente in un esperimento e, come notato, dovrebbe rientrare nelle regole empiriche per 300-400 u.a. in rosso e verde e 400-700 u.a. per il blu negli esperimenti tipici; Il software dispone di guide codificate a colori per l'utente anche per queste gamme.- Fare clic sulla scheda Colore verde per caricare i dati per quel canale e visualizzare i tagli correnti. Esaminando il punto di controllo negativo dell'isotipo si potrebbero vedere eventi di rivelazione con intensità di fluorescenza molto basse; In questo caso, gli utenti dovrebbero in genere aspettarsi di impostare il verde tra 300-400 U.A.
- Aumentare il minimo per ciascuno dei canali di rilevamento fino a quando la "Media % Inc" (la % media delle particelle rilevate sul punto di controllo incluso sopra il cutoff) sotto il display dei dati non ha avvisi come indicato dall'evidenziazione rossa o gialla in quella cella.
NOTA: Il valore massimo in genere non deve essere regolato rispetto a quello predefinito, ma può essere abbassato per filtrare le particelle luminose o limitare il rilevamento fluorescente a un intervallo più ristretto. - Fare clic sul pulsante Avanti .
- Aumentare il minimo per ciascuno dei canali di rilevamento fino a quando la "Media % Inc" (la % media delle particelle rilevate sul punto di controllo incluso sopra il cutoff) sotto il display dei dati non ha avvisi come indicato dall'evidenziazione rossa o gialla in quella cella.
- Fare clic sulla scheda Colore rosso per caricare i dati per quel canale e visualizzare i cutoff correnti. Anche in questo caso, esaminando il punto di controllo negativo dell'isotipo, si potrebbero vedere eventi di rilevamento con intensità di fluorescenza molto basse; In questo caso, gli utenti dovrebbero in genere aspettarsi di impostare il rosso tra 300-400 A.U. Seguire le stesse procedure indicate nei passaggi 9.6.1.1-9.6.1.3.
- Fare clic sulla scheda Colore blu per caricare i dati per quel canale e visualizzare i tagli correnti. Anche in questo caso, esaminando il punto di controllo negativo dell'isotipo, seguire le stesse procedure indicate nei passaggi 9.6.1.1-9.6.1.3.
NOTA: Il canale blu è unico in quanto l'anticorpo nello spot è autofluorescente a livelli sufficienti da richiedere un cutoff leggermente più alto di 400-700 u.a. anche per chip bianchi.
- Fare clic sulla scheda Colore verde per caricare i dati per quel canale e visualizzare i tagli correnti. Esaminando il punto di controllo negativo dell'isotipo si potrebbero vedere eventi di rivelazione con intensità di fluorescenza molto basse; In questo caso, gli utenti dovrebbero in genere aspettarsi di impostare il verde tra 300-400 U.A.
- Dopo aver completato l'analisi Cutoff, fare clic su Avanti e apparirà il grafico della mappa di calore, che fornirà una panoramica del legame delle particelle durante l'esperimento. Esistono diversi strumenti di visualizzazione che l'utente può utilizzare per tracciare i dati. Questi grafici e i dati grezzi associati possono essere aggiunti a un report facendo clic sul pulsante Aggiungi report di stampa .
NOTA: La mappa di calore è la visualizzazione predefinita dei dati che mostra una vista di tutti i campioni, tutte le acquisizioni di un particolare rilevamento. La visualizzazione predefinita è il totale che quantifica le EV univoche associate a ciascun punto di cattura. - Fare clic sul pulsante Esporta report e selezionare una posizione di salvataggio per il report.
NOTA: il rapporto si aprirà in un browser. Trova il file del foglio di calcolo dell'elenco delle particelle filtrate allegato al rapporto; queste sono le informazioni da inviare a EVTRACK. - Continua ad aggiungere report utilizzando le diverse selezioni di Campioni e Tipi di acquisizione nei menu a discesa nella parte superiore dello schermo. I dati visualizzati sono controllati con la selezione dei pulsanti di rilevamento del canale in alto a sinistra. Imposta un particolare fenotipo/canale di selezione/acquisizione del campione facendo clic su ciascun pulsante del colore per disattivare (in grigio) o attivare (colorato) il canale di rilevamento.
- Fare clic sul pulsante Aggiungi grafico al rapporto per aggregare i grafici desiderati in un rapporto.
- Fare clic su Esporta rapporto e definire la posizione di salvataggio per il rapporto finale.
Risultati
La Figura 7 (pannello di sinistra) mostra un'immagine composita a tre colori di vescicole extracellulari derivate da supporti condizionati HEK293 legati al punto CD63 sul chip e colorati per CD81, CD63 e CD9 rispettivamente nei seguenti canali verde, rosso e blu. La Figura 7 (pannello in alto a destra) è un'immagine ingrandita che mostra ciascuna delle EV catturate in grado di visualizzare la co-localizzazione di uno o più col...
Discussione
Gli attuali metodi di caratterizzazione delle vescicole extracellulari si basano in gran parte su vescicole extracellulari purificate, che sono limitate dalle attuali limitazioni sperimentali dei metodi di purificazione delle vescicole extracellulari 9,10,11,12,13. L'imaging interferometrico a riflettanza a singola particella ...
Divulgazioni
Clayton Deighan e George Daaboul sono dipendenti e azionisti di NanoView Biosciences Inc.
Riconoscimenti
Questo lavoro è stato sponsorizzato in parte dal programma di premi per l'approvvigionamento di attrezzature e risorse per la ricerca della School of Medicine dell'Università del Kansas. PCG, LKC, FD e AR sono stati supportati con fondi da NIA R21 AG066488-01.
Materiali
| Name | Company | Catalog Number | Comments |
| 10-cm sterile Petri dish | Fisher | FB0875712 | |
| 15mL sterile tube | n/a | various | |
| 24-well cell culture plate, flat bottom | Fisher | 08-772-1 | |
| Blocking Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chipfiles | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chips | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chuck | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 250 ml | Fisher | 09-761-4 | |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 500 ml | Fisher | 09-761-10 | |
| Deionized (DI) water | Fisher | LC267404 | |
| EMS style tweezers with Carbon Fiber tips | Fisher | 50-193-0842 | |
| ExoView Human Tetraspanin Kit | NanoView Biosciences | EV-TETRA-C | Capture for hCD81, hCD9, hCD63, IgG Control + stains for hEV-A (hEV-CD63-647, hEV-CD81-555, hEV-CD9-488) 16 Chips per kit |
| ExoView R100 Imager | NanoView Biosciences | EV-R100 | Interferometric microscope including high specification camera including 3 color fluorescence and label free sizing and counting extracellular vesicles |
| Fluorescently labled huma CD9 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD63 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD81 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Incubation Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Orbital shaker or microplate shaker with digital settings capable of shaking at 500 rpm | n/a | various | |
| Plate Seal | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution A | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution B | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution C | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution D | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Square/flat tip tweezer | Fisher | 50-239-62 | |
| Straight strong point Boley style tweezers | Fisher | 16-100-124 | |
| Thermo Scientific Adhesive PCR Plate Seals | Fisher | AB-0558 |
Riferimenti
- Maas, S. L. N., Breakefield, X. O., Weaver, A. M. Extracellular vesicles: Unique intercellular delivery vehicles. Trends in Cell Biology. 27 (3), 172-188 (2017).
- Shah, R., Patel, T., Freedman, J. E. Circulating extracellular vesicles in human disease. The New England Journal of Medicine. 379 (10), 958-966 (2018).
- Deng, F., Miller, J. A review on protein markers of exosome from different bio-resources and the antibodies used for characterization. Journal of Histotechnology. 42 (4), 226-239 (2019).
- Cohen, O., et al. ’Golden’ exosomes as delivery vehicles to target tumors and overcome intratumoral barriers: in vivo tracking in a model for head and neck cancer. Biomaterials Science. 9 (6), 2103-2114 (2021).
- Han, Y., et al. Overview and update on methods for cargo loading into extracellular vesicles. Processes (Basel). 9 (2), 356 (2021).
- Lee, M., Im, W., Kim, M. Exosomes as a potential messenger unit during heterochronic parabiosis for amelioration of Huntington's disease. Neurobiology of Disease. 155, 105374 (2021).
- Sun, B., et al. Characterization and biomarker analyses of post-COVID-19 complications and neurological manifestations. Cells. 10 (2), 386 (2021).
- Jiang, L., Gu, Y., Du, Y., Liu, J. Exosomes: Diagnostic biomarkers and therapeutic delivery vehicles for cancer. Molecular Pharmaceutics. 16 (8), 3333-3349 (2019).
- Bachurski, D., et al. Extracellular vesicle measurements with nanoparticle tracking analysis - An accuracy and repeatability comparison between NanoSight NS300 and ZetaView. Journal of Extracellular Vesicles. 8 (1), 1596016 (2019).
- Carmicheal, J., et al. Label-free characterization of exosome via surface enhanced Raman spectroscopy for the early detection of pancreatic cancer. Nanomedicine. 16, 88-96 (2019).
- Greening, D. W., Xu, R., Ji, H., Tauro, B. J., Simpson, R. J. A protocol for exosome isolation and characterization: evaluation of ultracentrifugation, density-gradient separation, and immunoaffinity capture methods. Methods in Molecular Biology. 1295, 179-209 (2015).
- Wu, Y., Deng, W., Klinke, D. J. Exosomes: improved methods to characterize their morphology, RNA content, and surface protein biomarkers. The Analyst. 140 (19), 6631-6642 (2015).
- van de Vlekkert, D., Qiu, X., Annunziata, I., d'Azzo, A. Isolation and characterization of exosomes from skeletal muscle fibroblasts. Journal of Visualized Experiments: JoVE. (159), (2020).
- Ayala-Mar, S., Donoso-Quezada, J., Gallo-Villanueva, R. C., Perez-Gonzalez, V. H., Gonzalez-Valdez, J. Recent advances and challenges in the recovery and purification of cellular exosomes. Electrophoresis. 40 (23-24), 3036-3049 (2019).
- Daaboul, G. G., et al. Digital detection of exosomes by interferometric imaging. Scientific Reports. 6, 37246 (2016).
- Pohler, K. G., et al. Circulating microRNA as candidates for early embryonic viability in cattle. NMolecular Reproduction and Development. 84 (8), 731-743 (2017).
- NanoView Biosciences. ExoView R100 User Guide. v240.4. NanoView Biosciences. , 202 (2021).
- Daaboul, G. G., et al. Enhanced light microscopy visualization of virus particles from Zika virus to filamentous ebolaviruses. PLoS One. 12, 0179728 (2017).
- ET Consortium. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14, 228-232 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon