JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
Single-Particle Interferometric Reflectance Imaging Characterization of individual extracellular vesicles and population dynamics(개별 세포외 소포체 및 집단 역학의 단일 입자 간섭계 반사율 이미징 특성화)
요약
이 프로토콜은 세포외 소포체(EV) 크기, EV 수, EV 표현형 및 EV 바이오마커 공동 국소화의 다단계 및 포괄적인 측정을 위해 설계된 단일 입자 간섭계 반사 이미징을 제공합니다.
초록
세포외 소포체(EV)는 대부분의 세포에서 분비되는 지질 이중층이 있는 나노미터 크기의 소포입니다. EV는 단백질, 지질, DNA 및 RNA를 포함한 다양한 생물학적 분자를 운반하며 다양한 조직과 기관에서 세포 간 통신을 촉진하는 것으로 가정됩니다. 최근 EV는 다양한 질병에 대한 진단 및 치료제를 위한 바이오마커로 큰 주목을 받고 있습니다. EV 특성화를 위해 많은 방법이 개발되었습니다. 그러나 현재의 EV 분석 방법에는 모두 다른 제한 사항이 있습니다. 따라서 EV 절연 및 특성화를 위한 효율적이고 효과적인 방법을 개발하는 것은 이 최첨단 연구 분야가 성숙해짐에 따라 중요한 단계 중 하나로 남아 있습니다. 여기에서는 정제되지 않은 생물학적 소스의 EV와 다른 방법론으로 정제된 EV를 검출하고 특성화할 수 있는 방법으로서 단일 입자 간섭계 반사 이미징 센서(SP-IRIS)를 간략하게 설명하는 자세한 프로토콜을 제공합니다. 이 고급 기술은 EV 크기, EV 수, EV 표현형 및 바이오마커 공동 국소화 분석을 위한 다단계 및 포괄적인 측정에 사용할 수 있습니다.
서문
세포외 소포체(EV)는 혈액, 모유, 타액, 소변, 담즙, 췌장액, 뇌척수액 및 복막액을 포함한 수많은 생물학적 액체에서 분리할 수 있는 세포 기원의 나노미터 크기의 막 소포체입니다. EV의 유도는 세 가지 주요 메커니즘을 통해 발생합니다: 세포사멸(apoptosis), 다소포체(multivesicular body)와 원형질막(plasma membrane)의 융합을 통한 방출(release), 원형질막(plasma membrane)의 블리빙(blebbing)1. 공여체 세포 구성 요소가 인접 또는 먼 세포 및 조직으로 EV로 전달된다는 증거는 이러한 막으로 둘러싸인 패키지가 부분비뿐만 아니라 장거리 또는 내분비 신호 전달 캐스케이드에서 중요한 역할을 할 수 있음을 시사합니다 1,2,3. EV는 세포의 표현형에 대한 스냅샷을 제공할 수 있기 때문에 다양한 질병 치료를 위한 진단 및 치료 도구로 사용할 수 있는 잠재력이 활발한 연구 분야가 되었습니다 4,5,6,7,8.
EV 특성화를 목표로 하는 많은 방법이 개발되었습니다 9,10,11,12,13. 이러한 방법의 대부분은 주로 대량으로 EV 모집단에 대한 고유하고 가치 있는 정보를 제공합니다. 이러한 기술의 하위 집합은 단일 EV 내부 또는 단일 EV의 물질에 대한 세부 정보를 제공할 수 있지만 단일 EV 수준에서 EV를 특성화하는 데는 제한이 있을 수 있습니다. 예를 들어, 면역-전자 현미경은 단일 EV와 그 구성을 이해하는 데 사용할 수 있지만, 이 기술은 처리량이 적고 인구 역학을 설명하는 데 사용되는 능력이 심각하게 제한되며 상당한 방법 개발이 필요합니다14.
최근 ExoView 플랫폼을 통한 SP-IRIS(Single-Particle, 간섭계 반사 이미징 센서) 기술의 개발 및 상용화로 인해 일상적이고 간단한 자동 데이터 수집 방법을 사용하여 개별 EV 특성화가 가능해졌습니다. 이 기술의 핵심은 단일 생물학적 나노 입자의 간섭계 측정을 가능하게 하는 1cm x 1cm Si/SiO2 이중층인 칩입니다. 이 칩은 개별 기능화된 항체 스폿의 마이크로어레이로 경작되어 최대 6개의 서로 다른 포획 유형을 다중으로 검출할 수 있습니다. 표준 칩에는 배양 단계 중 포획을 위한 일반적인 테트라스파닌 마커(CD81, CD63 및 CD9)가 포함되어 있으며, 사용자는 테트라스파닌과 별도로 EV의 고유한 집단을 분리하기 위해 추가 사용자 지정 포획 지점을 추가할 수 있습니다. 인큐베이션 단계 후, 각 포획 지점에는 해당 마커를 표현하는 많은 EV가 바인딩되어 있습니다. 그런 다음 이러한 캡처된 EV는 리더에서 간단히 세척, 건조 및 스캔하여 50-200nm 사이의 캡처 지점에 결합된 소포의 크기를 정량화하여 SP-IRIS15를 통해 숫자 가중치 크기 분포를 제공할 수 있습니다. 이 시스템은 또한 포획된 EV의 면역 표지를 위한 3개의 형광 검출 채널을 제공하며, SP-IRIS 측정과 같은 크기에 의해 제한되지 않는 평균 형광 강도와 각 형광 염색에 대한 공동 국소화 측면을 모두 제공합니다. 이를 통해 사용자는 EV당 4개의 서로 다른 바이오마커(캡처 및 3개의 면역형광 라벨)의 표시를 기반으로 단일 EV의 모집단을 정의할 수 있습니다. 이 시스템은 면역 형광으로 표면 단백질을 측정하는 것 이상의 기능을 제공할 수 있으며, 옵션인 화물 프로토콜을 통해 사용자는 캡처된 EV의 내부 단백질과 멤브레인 표면 마커의 내강 항원결정기를 프로브할 수 있을 뿐만 아니라 EV 멤브레인 무결성을 확인할 수 있습니다. 이 기사에서는 대규모 EV 모집단에 대한 단일 EV 수준에서 최대 4개의 서로 다른 바이오마커를 사용하여 EV 크기 및 수에 대한 일관된 데이터를 얻는 데 필요한 단계를 설명하는 자세한 프로토콜을 제공합니다. 이 기술은 초원심분리, 한외여과, 침전제, 면역친화성 캡처, 미세유체역학 및 크기 배제 크로마토그래피와 같은 다양한 기술을 사용하여 분리된 처리되지 않은 생물학적 유체와 EV 모두에 사용할 수 있습니다.
아래에 설명된 프로토콜은 확립된 분리 방법을 사용하여 HEK 293 세포 배양 배지 및 마우스 혈청에서 유래한 세포외 소포체(EV)를 사용합니다16. 이 프로토콜은 수많은 다른 생물학적 유체, 세포 배양 배지 및 생물학적 유체에서 분리된 정제된 세포외 소포체에 적용되었습니다. 이 프로토콜은 그림 1에 표시된 일반적인 실험에 대한 워크플로우와 함께 2일 절차로 나뉩니다.
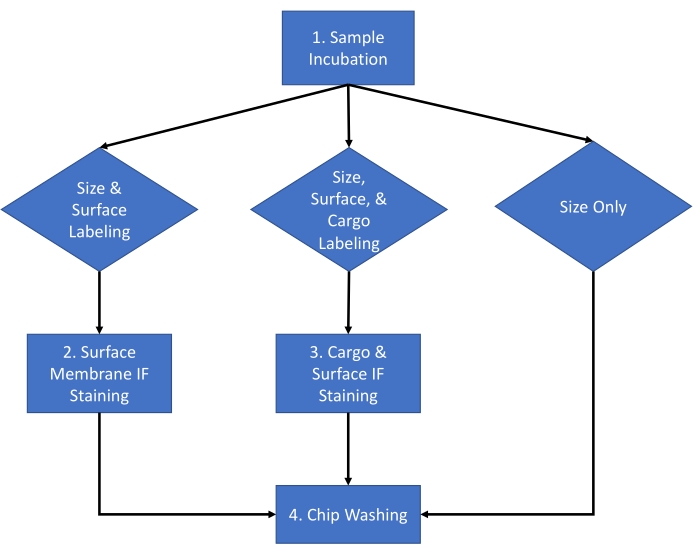
그림 1: 분석 워크플로우. 크기와 개수, 크기 개수와 표면 염색, 크기 개수와 화물 염색 사이에서 샘플에 대해 완료할 분석 유형을 선택하기 위한 분석 워크플로우. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Access restricted. Please log in or start a trial to view this content.
프로토콜
혈청 샘플은 캔자스 대학교 메디컬 센터(KUMC) 프로토콜에서 승인된 IACUC(Institutional Animal Care and Use Committees)에 따라 마우스에서 수집되었습니다. 이러한 실험에서 이러한 생물학적 시료를 사용하는 것도 KUMC의 승인을 받았습니다.
1. 시료 준비(1일차)
- 나노 입자 추적 또는 이에 상응하는 기술을 사용하여 EV 농도를 측정합니다.
- 배양 용액으로 샘플을 5 x 10,7-5 x 10,8 EVs/mL의 농도로 희석합니다. 최소 50μL가 필요합니다.
참고: EV 농도를 알 수 없는 샘플의 경우 1μg/mL의 총 단백질을 대체 측정으로 사용할 수 있습니다. 샘플의 농도가 낮을 것으로 예상되는 경우 로딩하기 전에 배양 용액에서 최소 1:1 희석을 수행합니다. - 진동과 무뚝뚝한 움직임이 없는 평평한 표면에 24웰 플레이트를 놓습니다.
참고: 대비를 향상시키기 위해 접시 아래에 흰색 종이를 놓을 수 있습니다. - 우물 주변 지역에 물을 추가합니다(그림 2).
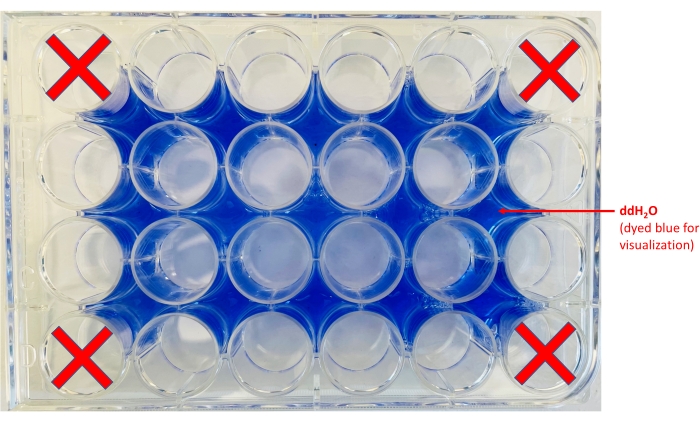
그림 2: 24웰 플레이트 레이아웃. ddH2O를 분주할 위치(파란색 염료는 시각화 목적으로만 추가됨)와 칩이 보관될 웰이 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
2. 칩 준비 및 사전 스캐닝
- 48-4°C 냉장고에서 칩이 들어 있는 밀봉된 8웰 플레이트를 제거하고 밀봉을 열기 전에 실온(~15분)에 도달하도록 합니다.
알림: 이것은 반점을 손상시킬 수 있는 칩의 결로 현상을 방지하는 데 필수적입니다. 팩에서 칩을 제거할 때 표면에 응결이 관찰되면 다른 칩을 제거하기 전에 더 오래 기다리십시오. - 8단계로 진행하여 척을 회수하고 사전 스캔 실행을 준비합니다(그림 3).
참고: 사전 스캔 데이터는 샘플과 함께 배양하기 전에 포집 지점에서 검출할 수 있는 입자를 식별하는 데 사용되므로 분석 단계에서 제거할 수 있습니다.

그림 3: 칩을 기계에 로드하는 데 사용되는 척 이미지. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 핀셋을 사용하여 48웰 플레이트에서 원하는 수의 칩(샘플당 칩 1개)을 제거하고 프리스캔 실행을 위해 칩을 척에 직접 로드합니다. 실험에 사용할 모든 칩이 8단계에서 설명한 대로 로드되고 사전 스캔되면 2.4단계로 진행합니다.
참고: 칩을 다룰 때는 항체 포획 지점이 이 영역에 있고 핀셋으로 건드리면 손상되므로 중앙의 사각형을 만지지 마십시오(그림 4).

그림 4: 칩 및 적절한 칩 처리. (A) 노란색 점선은 점박이 항체의 위치 또는 칩의 기능적 측면을 나타냅니다. 칩 ID는 줄 아래("58")에 있습니다. 그림에서는 적절한 취급을 보여줍니다. (B) 칩의 부적절한 취급을 보여줍니다. (C) 칩의 작동하지 않는 면. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 기능화된 표면이 위로 향하도록 사전 스캔된 각 칩을 사전 준비된 24웰 플레이트에 놓습니다. 기능화된 표면은 뚜렷한 번호 매기기, 정렬 그리드 및 중간에 있는 세 개의 검은색 상자로 쉽게 인식됩니다. 기능화되지 않은 면은 평평한 은색의 균일한 표면입니다.
- 미세 포인트 핀셋을 사용하여 각 칩이 웰 내 중앙에 오도록 합니다(그림 5).
참고: 칩이 웰 벽에 닿으면 로딩 후 샘플이 썩을 수 있기 때문에 웰 내의 칩을 중앙에 배치하는 것이 중요합니다.

그림 5: 우물에 적절한 칩 배치를 보여주는 것. (A) 칩은 우물의 측면에 모서리가 닿지 않도록 우물의 중앙에 설치해야 합니다. (B) 모서리가 우물의 측면에 닿는 칩의 부적절한 배치에 대한 묘사. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. 칩과 샘플의 적재 및 배양
- 준비된 시료 35μL(1단계)를 칩에 피펫팅합니다.
알림: 기포를 추가하거나 피펫 팁으로 칩을 만지지 않도록 주의하십시오.ample의 균일한 분포를 방해할 수 있습니다.amp칩에 있거나 칩의 항체 반점을 손상시킵니다. 샘플은 전체 칩 표면에 퍼져야 합니다. 로딩 중 샘플이 칩을 흡수하는 경우 최대 250μL 용량까지 웰에 추가 샘플을 추가하거나 최대 250μL의 배양 용액을 추가하고 해당 샘플의 변경된 희석 계수를 기록하십시오. - 샘플의 증발을 방지하기 위해 키트에 포함된 플레이트 필름을 사용하여 플레이트를 밀봉합니다.
- 시료/칩을 진동이나 움직임이 없는 실온의 밀봉된 플레이트에서 밤새(~16시간) 배양합니다.
참고: 하룻밤 배양 후 사용자는 연구 질문에 적합한 Day 2 옵션을 선택합니다. EV 크기와 개수만 원하는 경우 4단계(2일차)를 진행합니다. 다중 표면 마커 분석이 필요한 경우 5단계(2일차)로 건너뜁니다.
4. EV 크기 및 개수 결정(2일차)
- 칩이 들어 있는 각 웰의 측면에 용액 A를 1mL 추가하되, 칩에 용액을 직접 추가하거나 피펫 팁으로 칩을 긁지 않도록 주의하십시오.
- 실온에서 500분 동안 ~3rpm으로 회전하는 궤도 셰이커에 플레이트를 놓습니다.
알림: 칩이 접시에서 덜거덕거리면 액체가 소용돌이치지만 덜거덕거림이 없도록 즉시 속도를 줄이십시오. - 750μL의 액체를 제거합니다. 칩이 우발적으로 건조되는 것을 방지하기 위해 액체를 제거하는 동안 플레이트를 기울이지 마십시오.
- 4.1단계에서 설명한 기법을 사용하여 용액 B 750μL를 추가하고 실온에서 3분 동안 ~500rpm으로 흔듭니다.
- 4.3-4.4단계를 두 번 더 반복하여 용액 B로 총 3회 세척합니다.
- 마지막 세척이 끝나면 용액 750μL를 제거하고 웰에 용액 B 250μL를 남깁니다.
- 750μL의 이중 증류수(ddH2O)를 넣고 실온에서 ~500rpm으로 3분 동안 흔듭니다.
- 10cm 페트리 접시에 50mL의 ddH2O를 채우고 핀셋을 사용하여 우물에서 한 번에 하나의 칩을 접시로 옮깁니다.
알림: 칩을 수평으로 옮기도록 주의하고 건조되지 않도록 하십시오. ddH2O를 교체하기 전에 최대 8개의 칩을 세척할 수 있습니다. - ddH2O에서 핀셋을 사용하여 칩의 가장자리를 잡고 접시를 3바퀴 회전하여 이물질을 제거합니다.
- 물 밖으로 45° 각도로 칩을 제거하고 칩 ID가 위를 향하도록 하여 흡수지에 칩을 놓습니다(그림 6).
알림: 이제 칩을 읽을 준비가 되었습니다. SP-IRIS 리더에서 스캔하기 위한 8단계로 건너뜁니다.

그림 6: ddH2O 물에서 칩을 45° 각도로 제거하는 올바른 방법. (A) 위에서 보기 및 (B) 측면에서 칩을 제거하는 각도를 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
5. 항체 용액 준비 (Day 2)
- 칩당 300μL의 차단 용액을 적절한 크기의 튜브(0.5 - 3mL)에 추가합니다.
- 차단 용액 300μL당 0.6μL의 항체를 추가합니다. 튜브를 두드려 부드럽게 섞고 빠르게 회전합니다.
참고: 인간 CD9, CD81 및 CD63 또는 쥐 CD9, CD81 및 CD63에 대한 항체는 해당 키트에 포함되어 있습니다. 다른 형광 접합 항체가 필요한 경우, 유세포 분석에서와 마찬가지로 일정한 시료 로딩으로 0.1 - 10 μg/mL 범위의 일반적인 적정을 사용하여 최적의 염색 농도를 측정하십시오.
6. 면역형광 염색을 통한 EV 크기, 개수 및 표현형 결정
- 칩이 들어 있는 각 웰의 측면에 용액 A 1mL를 추가하고, 칩에 용액을 직접 추가하거나 피펫 팁으로 칩을 긁지 않도록 주의하십시오.
- 실온에서 500분 동안 ~3rpm으로 회전하는 궤도 셰이커에 플레이트를 놓습니다.
알림: 칩이 접시에서 덜거덕거리면 액체가 소용돌이치지만 덜거덕거림이 없도록 즉시 속도를 줄이십시오. - 액체 750μL를 제거합니다. 칩이 우발적으로 건조되는 것을 방지하기 위해 액체를 제거하는 동안 플레이트를 기울이지 마십시오.
- 6.1단계에서 설명한 기법을 사용하여 750μL의 용액 A를 추가하고 실온에서 3분 동안 ~500rpm으로 흔듭니다.
- 6.3단계와 6.4단계를 두 번 반복하여 용액 A로 총 3회 세척합니다.
- 마지막 세척이 끝나면 용액 750μL를 제거하고 용액 A 250μL를 웰에 남깁니다.
참고: 사용자가 화물 염색을 원하는 경우 이 시점에서 7단계로 진행하십시오. 그렇지 않은 경우 6.7단계로 진행합니다. - 250μL의 항체 용액을 well의 각 칩에 추가합니다(5단계). 빛으로부터 보호하기 위해 플레이트를 호일로 덮고 실온에서 오비탈 셰이커에서 1시간 동안 흔듭니다.
- 용액 A를 500μL씩 추가하면 웰당 총 부피는 ~1,000μL가 됩니다.
- 즉시 750μL를 제거하고 용액 A 750μL를 첨가한 후 실온에서 3분 동안 오비탈 셰이커에서 ~500rpm으로 흔듭니다.
- 용액 750μL를 제거하고 용액 B 750μL를 첨가한 후 실온에서 3분 동안 ~500rpm으로 흔듭니다.
- 6.10단계를 두 번 더 반복하여 총 세 번 세탁합니다.
- 750μL의 이중 증류수(ddH2O)를 넣고 실온에서 ~500rpm으로 3분 동안 흔듭니다.
- 10cm 페트리 접시에 50mL의 ddH2O를 채우고 핀셋을 사용하여 웰에서 접시로 칩 하나를 옮깁니다.
알림: 칩을 수평으로 옮기도록 주의하고 건조되지 않도록 하십시오. 새 ddH2O로 교환하기 전에 최대 8개의 칩을 세척할 수 있습니다. - ddH2O에서 핀셋을 사용하여 칩의 가장자리를 잡고 접시를 3바퀴 돌려 이물질을 제거합니다.
- 물에서 45° 각도로 칩을 제거하고 칩 ID가 위를 향하도록 하여 흡수지에 칩을 놓습니다(그림 6).
알림: 이제 칩을 읽을 준비가 되었습니다. 리더에서 스캔하기 위해 칩을 설정하는 방법에 대한 지침은 8단계(데이터 수집)로 건너뜁니다.
7. 선택적인 화물 얼룩이 지게 하기
참고: 이 프로토콜을 사용하면 내부 및 표면 마커에 동시에 레이블을 지정할 수 있습니다.
- 각 웰에 250μL의 용액 A가 남아 있는 상태에서 각 웰에 250μL의 용액 C를 추가합니다.
- 즉시 오비탈 셰이커에 놓고 정확히 10분 동안 ~200rpm으로 설정합니다.
참고: 이 단계와 아래 7.8단계에서는 타이밍과 느린 흔들기 속도가 모두 중요합니다. 속도가 우물에서 천천히 소용돌이치는 것만 제공하는지 확인하고 타이머를 사용하십시오. - 용액 C로 10분 동안 배양한 후 각 웰에 용액 A 500μL를 추가합니다.
- 즉시 750μL를 제거하고 750μL의 용액 A를 추가한 후 실온에서 3분 동안 오비탈 셰이커에서 ~500rpm으로 흔듭니다.
- 7.4단계를 두 번 더 반복하여 총 3회 세탁합니다.
- 마지막 세척이 끝나면 용액 750μL를 제거하고 칩과 함께 웰에 ~250μL의 용액을 남깁니다.
- 각 웰에 250μL의 용액 D를 추가합니다.
- 즉시 ~200rpm으로 설정된 오비탈 셰이커에 정확히 10분 동안 놓습니다.
- 용액 C로 10분 동안 배양한 후 각 웰에 용액 A 500μL를 추가합니다.
- 즉시 750μL를 제거하고 용액 A 750μL를 첨가한 후 실온에서 3분 동안 오비탈 셰이커에서 ~500rpm으로 흔듭니다.
- 7.10단계를 두 번 반복하여 총 세 번 세탁합니다.
- 마지막 세척 후 용액 750μL를 제거하고 위의 6.7단계로 돌아가 염색하고 분석 프로토콜을 완료합니다.
8. 데이터 수집
알림: Exo를 사용하여 칩에서 데이터를 수집하는 절차View R100은 자동화되어 있으며 사용자 입력이 필요하지 않습니다. 자세한 지침은 사용자 가이드 및 칩 캐리어 또는 "척" 로드 및 데이터 수집17에 대한 해당 비디오에서 찾을 수 있습니다.
- 기기 후면에 있는 두 개의 전원 스위치를 사용하여 EV 특성화 플랫폼의 전원을 켠 다음 바탕 화면 아이콘을 두 번 클릭하여 스캐너 소프트웨어를 시작합니다.
알림: 스캐너 소프트웨어가 시작되면 리더가 자동으로 홈 화면으로 설정되고 사용자에게 "칩을 로드하려면 문을 엽니다"라는 메시지가 표시됩니다. - 은색 손잡이를 들어 올려 리더기 전면에 있는 문을 엽니다. 이렇게 하면 s가 배출됩니다.tage, 사용자가 척에 액세스하고 소프트웨어에서 새 스캔을 설정할 수 있습니다.
- 사용자가 저장 폴더를 클릭하고 데이터를 저장할 원하는 위치를 선택하여 데이터를 저장하려는 위치를 식별합니다.
참고: 실험 이름과 같이 통찰력 있는 이름으로 저장 위치의 이름을 지정한 다음, 사전 스캔용과 사후 스캔용의 두 개의 하위 폴더를 만드는 것이 유용한 경우가 많습니다. 이렇게 하면 분석 중에 데이터를 더 쉽게 찾고 일치시킬 수 있습니다. - 칩을 스캔하려면 제어 컴퓨터에서 칩 파일을 찾으십시오.
참고: 칩 파일은 사용 중인 칩의 항체 스폿 레이아웃에 대한 맵이며 스캐너가 각 칩에서 스캔할 위치를 알 수 있도록 합니다. 모든 키트에는 내부 칩용 칩 파일이 있는 USB 키가 있습니다. 이러한 정보는 사용자가 안정적으로 찾을 수 있도록 키트를 수령하는 즉시 제어 컴퓨터의 위치에 저장해야 합니다. - 칩의 번호가 척의 핸들을 향하도록 하여 칩을 척에 로드한 다음 컴퓨터의 칩 드롭다운 메뉴에서 chipfiles 목록에서 각 칩을 선택하고 스캐너 소프트웨어의 가상 chuck에서 일치하는 적절한 위치에 놓습니다. 완료되면 사용자에게 Chuck을 스테이지에 배치하도록 요청하는 화면 프롬프트가 나타납니다.
- 적재된 척을 무대에 놓습니다. 척의 자기 정렬은 자동으로 s의 올바른 위치로 이동합니다.tage, 그런 다음 Place the Chuck on Stage 옆에 있는 OK(확인 )를 클릭합니다. 그런 다음 스캐너는 자동화된 데이터 수집 루틴을 시작합니다.
참고: 소프트웨어가 각 칩의 스캔 상태를 성공적으로 보고하면 데이터를 분석할 준비가 된 것입니다.
9. 데이터 분석
- 제어 PC의 바탕 화면에 있는 ExoView Analyzer 를 두 번 클릭합니다. 소프트웨어가 부팅된 후 Prescan Data(데이터 사전 검사 ) 버튼을 클릭하고 섹션 8.3에서 사전 검사 데이터 세트의 폴더 위치를 선택합니다.
- Postscan Data 버튼을 클릭하고 섹션 8.3에서 postscan 데이터 세트의 폴더 위치를 선택합니다.
참고: 데이터가 두 폴더 모두에서 제대로 감지되면 적어도 일부 칩에 대해 소프트웨어는 칩파일 위치 버튼 옆에 감지된 칩의 "X" 수를 표시합니다. 제대로 저장되면 감지된 칩이 스캔된 칩의 수와 일치해야 합니다. - 데이터 로딩 영역 하단의 다음을 클릭합니다.
- 메타 테이블 드롭다운 메뉴에서 각 칩에는 셀이 있습니다. 샘플 이름, 희석 계수 및 각 검출 채널에서 염색되고 있는 마커를 해당 상자를 클릭하고 정보를 입력하여 입력합니다. 완료되면 다음을 다시 클릭합니다.
참고: 이 메타데이터는 데이터와 함께 저장됩니다. - Preform 품질 관리(QC)는 왼쪽 상단의 QC 버튼 아래에 있는 Disable 탭을 클릭하여 수행합니다.
참고: 이 기능을 사용하면 분석을 위해 특정 프로브와 칩을 끌 수 있습니다. 소프트웨어는 데이터와 관련하여 두 가지 경고를 제공하는데, 하나는 특정 캡처가 포화 상태임을 나타낼 수 있는 높은 개수에 대한 경고이고, 다른 하나는 캡처 유형의 반복 실험 중 하나가 다른 복제와 다를 때 식별하는 높은 변동 계수(CV)에 대한 경고입니다.- 높은 CV에 대한 경고 지점을 클릭하고, 소프트웨어에 의해 호출되는 지점을 검사하고, 명백한 물리적 손상이 있는지 확인합니다. 손상이 확인되면 Spot Number(스팟 번호 )를 클릭하여 분석에서 해당 지점을 끕니다.
- 모든 경고가 평가될 때까지 반복하고 다음을 클릭합니다.
- 왼쪽 상단의 QC 버튼 아래에 있는 Disable 탭 옆에 있는 Cutoff 탭을 클릭하여 컷오프 분석을 미리 형성합니다.
참고: 이렇게 하면 제어 지점 형광 반응이 차트에 표시되고 사용자에게 각 색상 채널(빨간색/녹색/파란색)에 대한 최소 및 최대 두 가지 설정을 제공합니다. 각 형광 검출 채널에 대한 차단 값을 설정하는 것은 데이터 조정이 필요한 유일한 방법이며 비교적 간단합니다. 중요한 것은 선택되는 최종 컷오프는 실험 전반에 걸쳐 일관되어야 하며 언급한 바와 같이 빨간색과 녹색의 경우 300-400 au, 일반적인 실험의 경우 파란색의 경우 400-700 au에 대한 경험 법칙에 속해야 합니다. 이 소프트웨어에는 이러한 범위에 대한 사용자를 위한 색상으로 구분된 가이드도 있습니다.- Green Color 탭을 클릭하여 해당 채널에 대한 데이터를 로드하고 현재 컷오프를 표시합니다. isotype negative control spot을 검사하면 매우 낮은 형광 강도로 검출 이벤트를 볼 수 있습니다. 이 경우 사용자는 일반적으로 300-400 AU 사이에서 녹색을 설정할 것으로 예상해야 합니다.
- 데이터 디스플레이 아래의 "Avg % Inc"(컷오프 위에 포함된 제어 지점에서 감지된 입자의 평균 %)에 해당 셀에 빨간색 또는 노란색 강조 표시로 표시된 경고가 없을 때까지 각 감지 채널의 최소값을 높입니다.
참고: 최대값은 일반적으로 기본값에서 조정할 필요가 없지만 밝은 입자를 걸러내거나 형광 검출을 더 좁은 범위로 제한하기 위해 낮출 수 있습니다. - 다음 버튼을 클릭합니다.
- 데이터 디스플레이 아래의 "Avg % Inc"(컷오프 위에 포함된 제어 지점에서 감지된 입자의 평균 %)에 해당 셀에 빨간색 또는 노란색 강조 표시로 표시된 경고가 없을 때까지 각 감지 채널의 최소값을 높입니다.
- Red Color 탭을 클릭하여 해당 채널에 대한 데이터를 로드하고 현재 컷오프를 표시합니다. 다시 말하지만, isotype negative control spot을 검사하면 매우 낮은 형광 강도로 검출 이벤트를 볼 수 있습니다. 이 경우 사용자는 일반적으로 빨간색을 300-400 AU 사이로 설정할 것으로 예상해야 합니다. 9.6.1.1-9.6.1.3 단계에서 설명한 것과 동일한 절차를 따릅니다.
- Blue Color 탭을 클릭하여 해당 채널에 대한 데이터를 로드하고 현재 컷오프를 표시합니다. 다시 isotype 음성 대조 지점을 검사하여 9.6.1.1-9.6.1.3 단계에서 설명한 것과 동일한 절차를 따릅니다.
참고: 블루 채널은 해당 지점의 항체가 블랭크 칩의 경우에도 400-700 a.u.의 약간 더 높은 컷오프를 필요로 할 만큼 충분한 수준에서 자동 형광성이라는 점에서 독특합니다.
- Green Color 탭을 클릭하여 해당 채널에 대한 데이터를 로드하고 현재 컷오프를 표시합니다. isotype negative control spot을 검사하면 매우 낮은 형광 강도로 검출 이벤트를 볼 수 있습니다. 이 경우 사용자는 일반적으로 300-400 AU 사이에서 녹색을 설정할 것으로 예상해야 합니다.
- 컷오프 분석이 완료된 후 다음을 클릭하면 실험 전반의 입자 결합에 대한 개요를 제공하는 히트맵 플롯이 나타납니다. 사용자가 데이터를 그리는 데 사용할 수 있는 몇 가지 시각화 도구가 있습니다. 이러한 플롯 및 관련 원시 데이터는 Add Plot Report 버튼을 클릭하여 보고서에 추가할 수 있습니다.
참고: 히트맵은 특정 검출에 대한 모든 샘플, 모든 캡처 보기를 보여주는 기본 데이터 표시입니다. 기본 디스플레이는 각 캡처 지점에 바인딩된 고유한 EV를 정량화하는 합계입니다. - Export Report( 보고서 내보내기 ) 버튼을 클릭하고 보고서의 저장 위치를 선택합니다.
참고: 보고서가 브라우저에서 열립니다. 보고서에 첨부된 필터링된 파티클 목록 스프레드시트 파일을 찾습니다. EVTRACK에 제출할 정보입니다. - 화면 맨 위에 있는 드롭다운 메뉴에서 다양한 샘플 및 캡처 유형을 선택하여 보고서를 계속 추가합니다. 표시되는 데이터는 왼쪽 상단의 채널 감지 버튼을 선택하여 제어됩니다. 각 색상 버튼을 클릭하여 특정 표현형/샘플 선택/캡처 채널을 설정하여 해당 검출 채널을 끄거나(회색으로 표시) 켭니다(색상).
- Add Plot to Report 버튼을 클릭하여 원하는 플롯을 보고서로 집계합니다.
- Export Report(보고서 내보내기)를 클릭하고 최종 보고서의 저장 위치를 정의합니다.
Access restricted. Please log in or start a trial to view this content.
결과
그림 7 (왼쪽 패널)은 칩의 CD63 스폿에 결합되고 각각 녹색, 적색, 청색 채널에서 CD81, CD63 및 CD9에 대해 염색된 HEK293 컨디셔닝 미디어에서 파생된 EV의 3색 합성 이미지를 보여줍니다. 그림 7 (오른쪽 상단 패널)은 캡처된 각 EV가 각 채널에서 다양한 강도로 하나 이상의 색상의 공동 위치 파악을 표시할 수 있음을 보여주는 확대 이...
Access restricted. Please log in or start a trial to view this content.
토론
현재의 EV 특성화 방법은 주로 정제된 EV에 의존하며, 이는 EV 정제 방법의 현재 실험적 한계에 의해 제한됩니다 9,10,11,12,13. SP-IRIS(Single-particle interferometric reflectance imaging)는 시료 분석에 필요한 정제 단계를 제거하여 일반적인 EV 워크플로우와 관련된 ...
Access restricted. Please log in or start a trial to view this content.
공개
Clayton Deighan과 George Daaboul은 NanoView Biosciences Inc.의 직원이자 주주입니다.
감사의 말
이 작업은 University of Kansas School of Medicine Research Equipment and Resource Procurement Award Program의 일부 후원을 받았습니다. PCG, LKC, FD 및 AR은 NIA R21 AG066488-01의 자금으로 지원되었습니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 10-cm sterile Petri dish | Fisher | FB0875712 | |
| 15mL sterile tube | n/a | various | |
| 24-well cell culture plate, flat bottom | Fisher | 08-772-1 | |
| Blocking Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chipfiles | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chips | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chuck | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 250 ml | Fisher | 09-761-4 | |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 500 ml | Fisher | 09-761-10 | |
| Deionized (DI) water | Fisher | LC267404 | |
| EMS style tweezers with Carbon Fiber tips | Fisher | 50-193-0842 | |
| ExoView Human Tetraspanin Kit | NanoView Biosciences | EV-TETRA-C | Capture for hCD81, hCD9, hCD63, IgG Control + stains for hEV-A (hEV-CD63-647, hEV-CD81-555, hEV-CD9-488) 16 Chips per kit |
| ExoView R100 Imager | NanoView Biosciences | EV-R100 | Interferometric microscope including high specification camera including 3 color fluorescence and label free sizing and counting extracellular vesicles |
| Fluorescently labled huma CD9 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD63 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD81 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Incubation Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Orbital shaker or microplate shaker with digital settings capable of shaking at 500 rpm | n/a | various | |
| Plate Seal | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution A | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution B | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution C | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution D | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Square/flat tip tweezer | Fisher | 50-239-62 | |
| Straight strong point Boley style tweezers | Fisher | 16-100-124 | |
| Thermo Scientific Adhesive PCR Plate Seals | Fisher | AB-0558 |
참고문헌
- Maas, S. L. N., Breakefield, X. O., Weaver, A. M. Extracellular vesicles: Unique intercellular delivery vehicles. Trends in Cell Biology. 27 (3), 172-188 (2017).
- Shah, R., Patel, T., Freedman, J. E. Circulating extracellular vesicles in human disease. The New England Journal of Medicine. 379 (10), 958-966 (2018).
- Deng, F., Miller, J. A review on protein markers of exosome from different bio-resources and the antibodies used for characterization. Journal of Histotechnology. 42 (4), 226-239 (2019).
- Cohen, O., et al. ’Golden’ exosomes as delivery vehicles to target tumors and overcome intratumoral barriers: in vivo tracking in a model for head and neck cancer. Biomaterials Science. 9 (6), 2103-2114 (2021).
- Han, Y., et al. Overview and update on methods for cargo loading into extracellular vesicles. Processes (Basel). 9 (2), 356(2021).
- Lee, M., Im, W., Kim, M. Exosomes as a potential messenger unit during heterochronic parabiosis for amelioration of Huntington's disease. Neurobiology of Disease. 155, 105374(2021).
- Sun, B., et al. Characterization and biomarker analyses of post-COVID-19 complications and neurological manifestations. Cells. 10 (2), 386(2021).
- Jiang, L., Gu, Y., Du, Y., Liu, J. Exosomes: Diagnostic biomarkers and therapeutic delivery vehicles for cancer. Molecular Pharmaceutics. 16 (8), 3333-3349 (2019).
- Bachurski, D., et al. Extracellular vesicle measurements with nanoparticle tracking analysis - An accuracy and repeatability comparison between NanoSight NS300 and ZetaView. Journal of Extracellular Vesicles. 8 (1), 1596016(2019).
- Carmicheal, J., et al. Label-free characterization of exosome via surface enhanced Raman spectroscopy for the early detection of pancreatic cancer. Nanomedicine. 16, 88-96 (2019).
- Greening, D. W., Xu, R., Ji, H., Tauro, B. J., Simpson, R. J. A protocol for exosome isolation and characterization: evaluation of ultracentrifugation, density-gradient separation, and immunoaffinity capture methods. Methods in Molecular Biology. 1295, 179-209 (2015).
- Wu, Y., Deng, W., Klinke, D. J. Exosomes: improved methods to characterize their morphology, RNA content, and surface protein biomarkers. The Analyst. 140 (19), 6631-6642 (2015).
- van de Vlekkert, D., Qiu, X., Annunziata, I., d'Azzo, A. Isolation and characterization of exosomes from skeletal muscle fibroblasts. Journal of Visualized Experiments: JoVE. (159), (2020).
- Ayala-Mar, S., Donoso-Quezada, J., Gallo-Villanueva, R. C., Perez-Gonzalez, V. H., Gonzalez-Valdez, J. Recent advances and challenges in the recovery and purification of cellular exosomes. Electrophoresis. 40 (23-24), 3036-3049 (2019).
- Daaboul, G. G., et al. Digital detection of exosomes by interferometric imaging. Scientific Reports. 6, 37246(2016).
- Pohler, K. G., et al. Circulating microRNA as candidates for early embryonic viability in cattle. NMolecular Reproduction and Development. 84 (8), 731-743 (2017).
- NanoView Biosciences. ExoView R100 User Guide. v240.4. NanoView Biosciences. , 202(2021).
- Daaboul, G. G., et al. Enhanced light microscopy visualization of virus particles from Zika virus to filamentous ebolaviruses. PLoS One. 12, 0179728(2017).
- ET Consortium. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14, 228-232 (2017).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유