このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
単一粒子干渉反射率イメージング:個々の細胞外小胞と集団動態の特性評価
要約
このプロトコルは、細胞外小胞(EV)サイズ、EVカウント、EV表現型、およびEVバイオマーカーの共局在のマルチレベルかつ包括的な測定のために設計された単一粒子干渉反射率イメージングを提供します。
要約
細胞外小胞(EV)は、ほとんどの細胞から分泌される脂質二重層を持つナノメートルサイズの小胞です。EVは、タンパク質、脂質、DNA、RNAなど、さまざまな生体分子を運び、さまざまな組織や臓器での細胞間コミュニケーションを促進すると考えられています。近年、EVは様々な疾患の診断薬や治療薬のバイオマーカーとして大きな注目を集めています。EVの特性評価のために、多くの方法が開発されてきました。しかし、現在のEV解析手法には、それぞれ異なる制限があります。したがって、EVの絶縁と特性評価のための効率的で効果的な方法を開発することは、この最先端の研究分野が成熟するにつれて、依然として重要なステップの1つです。ここでは、未精製の生物学的源からのEVと他の方法論によって精製されたEVを検出および特性評価できる方法として、単一粒子干渉反射率イメージングセンサー(SP-IRIS)の概要を示す詳細なプロトコルを提供します。この高度な手法は、EVサイズ、EV数、EV表現型、およびバイオマーカーの共局在を分析するためのマルチレベルかつ包括的な測定に使用できます。
概要
細胞外小胞(EV)は、血液、母乳、唾液、尿、胆汁、膵液、脳脊髄液、腹膜液など、多数の生体液から単離できるナノメートルサイズの細胞起源の膜小胞です。EVの導出は、アポトーシス、多胞体と原形質膜との融合による放出、原形質膜のブレブ1という3つの主要なメカニズムによって発生します。ドナー細胞成分のEV移動が隣接または離れた細胞や組織に転移するという証拠は、これらの膜封入パッケージがパラクリンだけでなく、長距離または内分泌シグナル伝達カスケードでも重要な役割を果たす可能性があることを示唆しています1,2,3。EVは細胞の表現型のスナップショットを提供できるため、さまざまな疾患の治療のための診断および治療ツールとしての使用の可能性は、研究の活発な領域となっています4,5,6,7,8。
EVの特性評価を目的とした多くの方法が開発されてきた9,10,11,12,13。これらの方法のほとんどは、主にバルクのEVの個体数に関するユニークで貴重な情報を提供します。これらの手法のサブセットでは、単一のEV内または単一のEV上の物質に関する詳細を提供できますが、単一のEVレベルでEVを特性評価するには制限がある場合があります。例えば、免疫電子顕微鏡は、単一のEVとその組成を理解するために使用できるが、この技術はスループットが低く、集団動態の記述に使用する能力が著しく制限されており、かなりの方法開発が必要である14。
最近、ExoViewプラットフォームを介した単一粒子干渉反射率イメージングセンサー(SP-IRIS)技術の開発と商品化により、ルーチンで簡単な自動データ収集方法を使用した個々のEVの特性評価が可能になりました。この技術の中核となるのは、1cm×1cmのSi/SiO2二重層チップで、単一の生体ナノ粒子の干渉測定を可能にします。チップは、個々の機能化された抗体スポットのマイクロアレイで耕され、最大6つの異なる捕捉タイプのマルチプレックス検出が可能になります。標準チップには、インキュベーションステップ中に捕捉するための一般的なテトラスパニンマーカー(CD81、CD63、およびCD9)が含まれており、ユーザーは追加のカスタム捕捉スポットを追加して、テトラスパニンとは別のEVの異なる集団を分離できます。インキュベーションステップの後、各キャプチャースポットは、対応するマーカーを発現する多くのEVをそれに結合させます。次に、これらの捕捉されたEVをリーダーで簡単に洗浄、乾燥、およびスキャンして、捕捉スポットに結合した小胞のサイズを50〜200 nmで定量化し、SP-IRIS15を介して数個の重み付けサイズ分布を得ることができます。また、このシステムは、捕捉されたEVを免疫標識するための3つの蛍光検出チャンネルを提供し、SP-IRIS測定などのサイズに制限されない平均蛍光強度と、各蛍光染色の共局在の側面の両方を提供します。これにより、ユーザーはEVごとに4つの異なるバイオマーカー(キャプチャーと3つの免疫蛍光標識)の表示に基づいて、単一のEVの集団を定義することができます。このシステムは、免疫蛍光法による表面タンパク質の測定にとどまらず、オプションのカーゴプロトコルにより、捕捉されたEVの内部タンパク質や膜スパン表面マーカーの管腔エピトープをプローブしたり、EV膜の完全性を確認したりできます。この記事では、大規模なEV集団の1つのEVレベルで最大4つの異なるバイオマーカーを使用して、EVのサイズと数に関する一貫したデータを取得するために必要な手順を概説した詳細なプロトコルを提供します。この技術は、超遠心分離、限外ろ過、沈殿剤、免疫親和性捕捉、マイクロ流体工学、サイズ排除クロマトグラフィーなど、さまざまな技術を使用して単離された未処理の生体液とEVの両方に使用できます。
以下に述べるプロトコールは、確立された単離法16を用いて、HEK293細胞培養培地およびマウス血清に由来する細胞外小胞(EV)を使用する。このプロトコルは、他の多くの生体液、細胞培養培地、および生体液から単離された精製細胞外小胞に適用されています。このプロトコルは、 図 1 に示す一般的な実験のワークフローを使用して 2 日間の手順に分かれています。
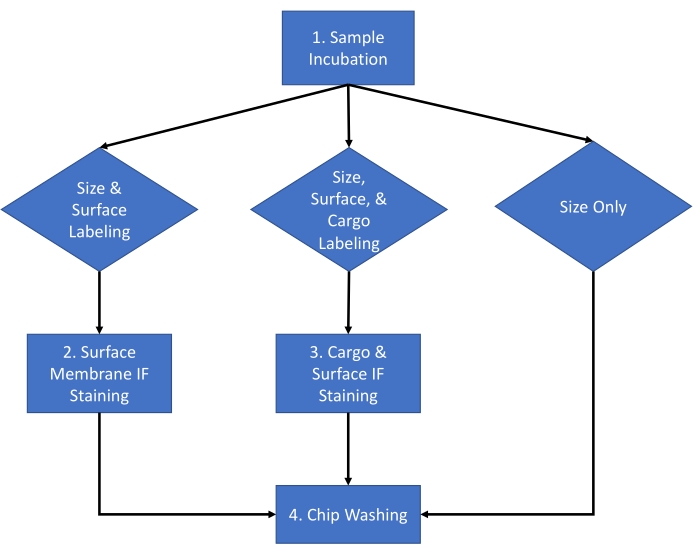
図1:アッセイワークフロー。 サンプルに対して完了する分析のタイプを、サイズとカウント、サイズカウントと表面染色、サイズカウントとカーゴ染色から選択するためのアッセイワークフロー。 この図の拡大版を表示するには、ここをクリックしてください。
Access restricted. Please log in or start a trial to view this content.
プロトコル
血清サンプルは、カンザス大学医療センター(KUMC)の承認された動物施設管理および使用委員会(IACUC)のプロトコルに従ってマウスから収集されました。これらの実験にこれらの生体試料を使用することも、建国大学工学院によって承認されています。
1. サンプル調製(1日目)
- ナノ粒子トラッキングまたは同等の技術を使用してEV濃度を決定します。
- インキュベーション溶液でサンプルを5 x 107-5 x 108 EV/mLの濃度に希釈します。最低50μLが必要です。
注:EV濃度が不明なサンプルの場合、1 μg/mLの総タンパク質を代用手段として使用できます。サンプルの濃度が低いと予想される場合は、インキュベーション溶液で少なくとも1:1の希釈を行ってからロードしてください。 - 24ウェルプレートを、振動や無愛想な動きのない平らな面に置きます。
注意: コントラストを高めるために、プレートの下に白い紙を置くことができます。 - 井戸の周辺に水を追加します(図2)。
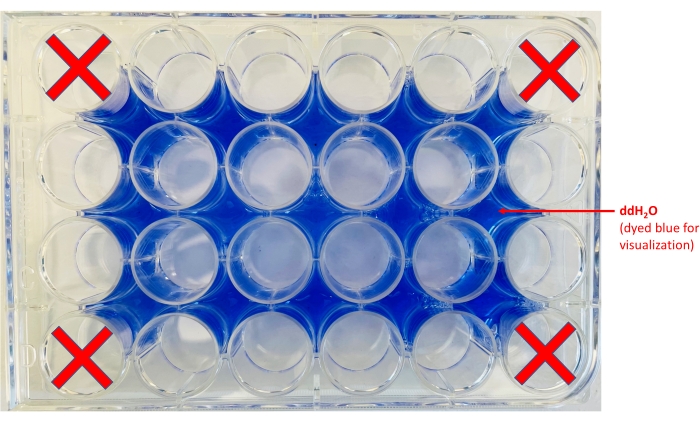
図2:24ウェルプレートのレイアウト。 ddH2O(青色色素は可視化のみを目的として添加)を分注する場所の位置と、チップが保持されるウェルが示されています。 この図の拡大版を表示するには、ここをクリックしてください。
2. チップの準備と事前スキャン
- チップが入った密封された48ウェルプレートを4〜8°Cの冷蔵庫から取り出し、室温(~15分)まで到達させてからシールを開けます。
注意: これは、スポットを損傷する可能性のあるチップ上の結露を避けるために不可欠です。チップをパックから取り出すときに表面に結露が見られる場合は、他のチップを取り除く前にもっと待ってください。 - ステップ8に進み、プリスキャン実行の準備をするためにチャックを取り出します(図3)。
注:プレスキャンデータは、サンプルとのインキュベーション前にキャプチャースポットで検出可能な粒子を特定するために使用され、分析ステップ中にそれらを除去できます。

図3:チップを機械にロードするために使用されるチャックの画像。 この図の拡大版を表示するには、ここをクリックしてください。
- ピンセットを使用して、必要な数のチップ(サンプルごとに1チップ)を48ウェルプレートから取り出し、チップを直接チャックにロードしてプレスキャンを実行します。実験に使用する予定のすべてのチップをロードし、ステップ8で説明したようにプレスキャンしたら、ステップ2.4に進みます。
注意:チップを取り扱う際は、抗体捕捉スポットがこの領域にあり、ピンセットで触れると損傷を受けるため、中央の四角に触れないように注意してください(図4)。

図4:チップとチップの適切な取り扱い (A) 黄色の点線は、スポット抗体の位置、またはチップの機能面を示しています。チップIDは線(「58」)の下にあります。図は適切な取り扱いも示しています。(B)チップの不適切な取り扱いを示しています。(C)チップの非機能的な側面。 この図の拡大版を表示するには、ここをクリックしてください。
- 事前にスキャンした各チップを、機能化された表面を上にして、事前に調製した24ウェルプレートに入れます。機能化された表面は、明確な番号付け、整列グリッド、および中央の3つのブラックボックスで簡単に認識できます。非機能化側は、平坦な銀色の均一な表面です。
- ファインポイントピンセットを使用して、各チップがウェルの中央に配置されていることを確認します(図5)。
注:チップがウェル壁に接触すると、ロード後にサンプルが吸い取られる可能性があるため、チップをウェル内にセンタリングすることが重要です。

図5:ウェル内のチップの適切な配置のデモンストレーション (A)チップは、ウェルの側面に接触する角がないように、ウェルの中央にセットする必要があります。(B)角がウェルの側面に接触するチップの不適切な配置の描写。 この図の拡大版を表示するには、ここをクリックしてください。
3. チップとサンプルのローディングとインキュベーション
- 調製したサンプル(ステップ1)35μLをチップにピペットで移します。
注:気泡を追加したり、チップに触れたりすると、チップ上のサンプルが均一に分布しなくなったり、チップ上の抗体スポットが損傷したりする可能性があるため、注意してください。サンプルはチップ表面全体に広がる必要があります。ローディング中にサンプルがチップからウィッシングされた場合は、250 μLまでウェルにサンプルを追加するか、最大250 μLのインキュベーション溶液を追加して、そのサンプルの希釈係数の変化に注意してください。 - サンプルの蒸発を防ぐために、キットに含まれているプレートフィルムを使用してプレートを密封します。
- サンプル/チップを密閉プレート内で一晩(~16時間)インキュベートし、室温で振動や動きのない場所でインキュベートします。
注:一晩のインキュベーション後、ユーザーは自分の研究課題に適したDay 2オプションを選択します。EVのサイズと個数のみをご希望の場合は、 ステップ4(Day 2)に進んでください。複数の表面マーカー分析が必要な場合は、 ステップ5(2日目)にスキップしてください。
4. EVのサイズと台数の決定(2日目)
- チップが入った各ウェルの側面に溶液Aを1 mL加え、チップに直接溶液を加えたり、ピペットチップでチップを傷つけたりしないように注意してください。
- プレートを~500rpmで回転するオービタルシェーカーにプレートを載せ、室温で3分間待ちます。
注意: チップがプレート上でガタガタする場合は、すぐに速度を下げて、液体が渦巻いているがガタガタ音がしないようにします。 - 750μLの液体を取り除きます。チップの偶発的な乾燥を防ぐために、液体の除去中にプレートを傾けないでください。
- ステップ 4.1 で説明した手法を使用して 750 μL の溶液 B を加え、室温で ~500 rpm で 3 分間振とうします。
- 手順4.3〜4.4をさらに2回繰り返し、溶液Bで合計3回洗浄します。
- 最後の洗浄の終わりに、750 μLの溶液を取り出し、250 μLの溶液Bをウェルに残します。
- 750 μL の二重蒸留水 (ddH2O) を加え、室温で ~500 rpm で 3 分間振とうします。
- 10 cmのシャーレに50 mLのddH2Oを入れ、ピンセットを使用してウェルから一度に1チップずつディッシュに移します。
注意: チップを水平に転写し、乾燥しないように注意してください。ddH2Oを交換する前に、最大8個のチップを洗浄できます。 - ddH2Oでは、ピンセットを使用してチップの端を保持し、皿内で3回転して破片を取り除きます。
- チップを水から45°の角度で取り出し、チップIDを上に向けてチップを吸収紙の上に置きます(図6)。
注: これで、チップを読み取る準備が整いました。SP-IRISリーダーでのスキャンについては、手順8に進みます。

図6:ddH2Oの水から45°の角度で切りくずを取り除く正しい方法(A)上面からの図と(B)側面からの図(切りくずを取り除く角度を示す図)。この図の拡大版を表示するには、ここをクリックしてください。
5. 抗体溶液の調製(2日目)
- チップあたり300 μLのブロッキング溶液を適切なサイズのチューブ(0.5〜3 mL)に加えます。
- ブロッキング溶液300μLあたり0.6μLの抗体を添加します。チューブを軽くたたいて素早く回転させて優しく混ぜます。
注:ヒトCD9、CD81、およびCD63またはマウスCD9、CD81、およびCD63に対する抗体は、適切なキットに含まれています。他の蛍光標識抗体が必要な場合は、フローサイトメトリーの場合と同様に、サンプル負荷を一定にしながら0.1〜10 μg/mLの範囲で一般的な滴定を行い、最適な染色濃度を決定します。
6. 免疫蛍光染色によるEVサイズ、カウント、表現型の決定
- チップが入った各ウェルの側面に溶液Aを1 mL加え、チップに直接溶液を加えたり、ピペットチップでチップを傷つけたりしないように注意してください。
- プレートを~500rpmで回転するオービタルシェーカーにプレートを載せ、室温で3分間待ちます。
注意: チップがプレート上でガタガタする場合は、すぐに速度を下げて、液体が渦巻いているがガタガタ音がしないようにします。 - 液体750μLを取り除きます。チップの偶発的な乾燥を防ぐために、液体の除去中にプレートを傾けないでください。
- ステップ 6.1 で説明した手法を使用して 750 μL の溶液 A を加え、室温で ~500 rpm で 3 分間振とうします。
- 手順6.3と6.4を2回繰り返し、溶液Aで合計3回洗浄します。
- 最後の洗浄の終わりに、750 μLの溶液を取り出し、250 μLの溶液Aをウェルに残します。
注:ユーザーがカーゴステインを希望する場合は、この時点でステップ7に進んでください。そうでない場合は、手順 6.7 に進みます - 250 μLの抗体溶液(ステップ5)をウェルの各チップに加えます。光から保護するためにプレートをホイルで覆い、オービタルシェーカーで室温で1時間振とうします。
- 溶液Aを500μL加えると、ウェルあたりの総容量は~1,000μLになります。
- 直ちに750μLを取り出し、750μLの溶液Aを加え、オービタルシェーカーで~500rpmで室温で3分間振とうします。
- 溶液750μLを取り出し、溶液Bを750μL加え、室温で~500rpmで3分間振とうします。
- 手順6.10をさらに2回繰り返し、合計3回の洗浄を行います。
- 750 μL の二重蒸留水 (ddH2O) を加え、室温で ~500 rpm で 3 分間振とうします。
- 10 cmのシャーレに50 mLのddH2Oを入れ、ピンセットを使用してウェルから1チップをディッシュに移します。
注意: チップを水平に転写し、乾燥しないように注意してください。新鮮なddH2Oに交換する前に、最大8個のチップを洗浄できます。 - ddH2Oでは、ピンセットを使用してチップの端を保持し、皿内で3回転して破片を取り除きます。
- 水から45°の角度でチップを取り出し、チップIDを上に向けてチップを吸収紙の上に置きます(図6)。
注: これで、チップを読み取る準備が整いました。手順8(データ収集)に進み、リーダーでスキャンするためのチップの設定方法を確認してください。
7. オプションのカーゴ染色
注:このプロトコルでは、内部マーカーと表面マーカーを同時にラベリングできます。
- 各ウェルに250 μLの溶液Aが残っている状態で、250 μLの溶液Cを各ウェルに加えます。
- すぐにオービタルシェーカーに置き、~200rpmに正確に10分間設定します。
注意: この手順と以下の手順 7.8 では、タイミングと遅い振とう速度の両方が重要です。速度が井戸内でゆっくりと渦巻くだけであることを確認し、タイマーを使用してください。 - 溶液Cで10分間インキュベートした後、各ウェルに500μLの溶液Aを加えます。
- 直ちに750μLを取り出し、750μLの溶液Aを加え、オービタルシェーカーで~500rpmで室温で3分間振とうします。
- 手順7.4をさらに2回繰り返し、合計3回の洗浄を行います。
- 最後の洗浄の終わりに、750 μL の溶液を取り出し、チップと共に ~250 μL の溶液をウェルに残します。
- 各ウェルに250 μLの溶液Dを加えます。
- すぐに~200rpmに設定したオービタルシェーカーに正確に10分間置きます。
- 溶液Cで10分間インキュベートした後、各ウェルに500μLの溶液Aを加えます。
- 直ちに750μLを取り出し、750μLの溶液Aを加え、オービタルシェーカーで~500rpmで室温で3分間振とうします。
- 手順7.10を2回繰り返し、合計3回の洗浄を行います。
- 最後の洗浄後に750 μLの溶液を取り出し、上記のステップ6.7に戻って染色し、アッセイプロトコルを完了します。
8. データ収集
注:ExoView R100を使用してチップからデータを収集する手順は自動化されており、ユーザー入力は必要ありません。詳細な手順は、チップキャリア、または「チャック」のロード、およびデータ収集17のユーザーガイドと対応するビデオに記載されています。
- 機器の背面にある2つの電源スイッチを使用してEV特性評価プラットフォームの電源を入れ、デスクトップアイコンをダブルクリックしてスキャナーソフトウェアを起動します。
注意: スキャナーソフトウェアが起動すると、リーダーは自動的にホーム画面に設定され、ユーザーに「ドアを開けてチップをロードしてください」と促します。 - 銀色のハンドルを持ち上げて、リーダーの前面にあるドアを開きます。これによりステージが排出され、ユーザーはチャックにアクセスして、ソフトウェアで新しいスキャンをセットアップできます。
- ユーザーがデータを保存する場所を特定するには、[ 保存 ] フォルダーをクリックし、データを保存する場所を選択します。
注: 多くの場合、保存場所に実験名のような洞察力のある名前を付けてから、事前スキャン用と事後スキャン用の 2 つのサブフォルダーを作成すると便利です。これにより、分析中のデータの検索と照合が容易になります。 - チップをスキャンするには、制御コンピュータでチップファイルを見つけます。
注:チップファイルは、使用中のチップ上の抗体スポットレイアウトのマップであり、スキャナーが各チップ上のどこをスキャンするかを知ることができます。すべてのキットには、内部のチップファイルが入ったUSBキーがあります。これらは、キットの受領時に、ユーザーが確実に見つけることができる制御コンピューター上の場所に保存する必要があります。 - チップの番号がチャックのハンドルに面している状態でチップをチャックにロードし、コンピューターの[チップ]ドロップダウンメニューからチップファイルリストから各チップを選択し、スキャナーソフトウェアの仮想チャックの適切な一致する場所に配置します。完了すると、チャックをステージに配置するようにユーザーに求める画面上のプロンプトが表示されます。
- ロードされたチャックをステージに置きます。チャックの磁気位置合わせにより、チャックはステージ上の正しい位置に自動的に移動し、[チャックをステージに配置する]の横にある [OK ]をクリックします。その後、スキャナーは自動データ収集ルーチンを開始します。
注意: ソフトウェアが各チップのスキャンステータスを成功として報告すると、データを分析する準備が整います。
9. データ分析
- コントロールPCのデスクトップで ExoView Analyzer をダブルクリックします。ソフトウェアが起動したら、[ Prescan Data ]ボタンをクリックし、セクション8.3でプレスキャンデータセットのフォルダの場所を選択します。
- Postscan Dataボタンをクリックし、セクション8.3でPostscanデータセットのフォルダの場所を選択します。
メモ: 両方のフォルダでデータが正しく検出されると、少なくとも一部のチップについて、ソフトウェアによって検出されたチップの数が [X] 個と表示されます。[チップファイルの場所] ボタンの横には、検出されたチップの数が表示されます。適切に保存されると、検出されたチップはスキャンされたチップの数と一致するはずです。 - データ読み込み領域の下部にある [次へ ]をクリックします。
- メタテーブルのドロップダウンメニューでは、各チップにセルがあります。サンプル名、希釈係数、および各検出チャネルで染色されているマーカーを入力するには、対応するボックスをクリックして情報を入力します。完了したら、もう一度[ 次へ ]をクリックします。
注: このメタデータはデータと共に保存されます。 - 品質管理(QC)をプリフォームするには、左上のQCボタンの下にある Disable(無効化 )タブをクリックします。
注意: この機能により、特定のプローブとチップをオフにして分析することができます。ソフトウェアは、データに関して 2 つの異なる警告を提供します。1 つは、特定のキャプチャが飽和していることを示す可能性のある高カウント、およびキャプチャ タイプの反復の 1 つが他のレプリケートと異なることを識別する高変動係数 (CV) です。- CVが高い警告 スポット をクリックし、ソフトウェアによって呼び出されているスポットを調べて、明らかな物理的損傷があるかどうかを確認します。損傷が特定されたら、 スポット番号 をクリックして、分析でそのスポットをオフにします。
- すべての警告が評価されるまで繰り返し、[ 次へ] をクリックします。
- カットオフ解析をプリフォームするには、左上のQCボタンの下にある「Disable」タブの隣にある「Cutoff」タブをクリックします。
注:これにより、コントロールスポットの蛍光応答がチャートに表示され、各カラーチャネル(赤/緑/青)の最小値と最大値の2つの設定が提供されます。各蛍光検出チャンネルのカットオフ値を設定することは、行う必要がある唯一のデータ調整であり、比較的簡単です。重要なことは、選択される最終的なカットオフは実験全体で一貫している必要があり、前述のように、赤と緑では300〜400 a.u.、典型的な実験では青では400〜700 a.u.の経験則に収まる必要があります。ソフトウェアには、これらの範囲のユーザー向けの色分けされたガイドもあります。- [Green Color] タブをクリックして、そのチャンネルのデータをロードし、現在のカットオフを表示します。アイソタイプネガティブコントロールスポットを調べると、非常に低い蛍光強度で検出イベントが見られる場合があります。この場合、ユーザーは通常、緑を300〜400AUに設定することを期待する必要があります。
- データディスプレイの下の「Avg % Inc」(カットオフより上に含まれるコントロールスポット上の検出粒子の平均%)に警告がなくなるまで、各検出チャネルの最小値を増やします(そのセルで赤または黄色の強調表示で示されています)。
注:通常、最大値をデフォルトから調整する必要はありませんが、明るい粒子をフィルタリングしたり、蛍光検出を狭い範囲に制限したりするために下げることができます。 - 「次へ」ボタンをクリックします。
- データディスプレイの下の「Avg % Inc」(カットオフより上に含まれるコントロールスポット上の検出粒子の平均%)に警告がなくなるまで、各検出チャネルの最小値を増やします(そのセルで赤または黄色の強調表示で示されています)。
- [Red Color] タブをクリックして、そのチャンネルのデータをロードし、現在のカットオフを表示します。繰り返しになりますが、アイソタイプネガティブコントロールスポットを調べると、非常に低い蛍光強度で検出イベントが見られる場合があります。この場合、ユーザーは通常、赤を300〜400AUに設定することを期待する必要があります。手順 9.6.1.1 から 9.6.1.3 に記載されているのと同じ手順に従います。
- [Blue Color] タブをクリックして、そのチャネルのデータをロードし、現在のカットオフを表示します。再度、アイソタイプネガティブコントロールスポットを調べるには、ステップ9.6.1.1-9.6.1.3で説明したのと同じ手順に従います。
注:青色チャンネルは、スポット内の抗体が、ブランクチップの場合でも400-700 a.u.のわずかに高いカットオフを必要とするのに十分なレベルで自己蛍光性であるという点で独特です。
- [Green Color] タブをクリックして、そのチャンネルのデータをロードし、現在のカットオフを表示します。アイソタイプネガティブコントロールスポットを調べると、非常に低い蛍光強度で検出イベントが見られる場合があります。この場合、ユーザーは通常、緑を300〜400AUに設定することを期待する必要があります。
- カットオフ解析が完了したら、[ Next ]をクリックすると、ヒートマッププロットが表示され、実験全体の粒子結合の概要が表示されます。ユーザーがデータをプロットするために使用できる視覚化ツールがいくつかあります。これらのプロットと関連する生データは、[ プロットレポートの追加 ]ボタンをクリックしてレポートに追加できます。
注:ヒートマップは、特定の検出のすべてのサンプル、すべてのキャプチャビューを表示するデフォルトのデータ表示です。デフォルトの表示は、各キャプチャ スポットにバインドされた一意の EV を定量化した合計です。 - [ レポートのエクスポート ]ボタンをクリックして、レポートの保存場所を選択します。
注: レポートはブラウザで開きます。レポートに添付されているフィルター パーティクル リスト スプレッドシート ファイルを見つけます。EVTRACKに提出する情報です。 - 画面上部のドロップダウンメニューで「サンプル」と「キャプチャタイプ」のさまざまな選択を使用して、レポートの追加を続けます。表示されるデータは、左上のチャンネル検出ボタンを選択して制御されます。特定の表現型/サンプル選択/キャプチャチャンネルを設定するには、各カラーボタンをクリックして、その検出チャンネルをオフ(グレー表示)またはオン(色付き)に切り替えます。
- [ レポートにプロットを追加 ]ボタンをクリックして、目的のプロットをレポートに集約します。
- [Export Report] をクリックし、最終レポートの保存場所を定義します。
Access restricted. Please log in or start a trial to view this content.
結果
図7 (左パネル)は、HEK293でコンディショニングされた培地をチップ上のCD63スポットに結合させ、CD81、CD63、CD9を染色したEVの3色合成画像です(それぞれ緑、赤、青)。 図7 (右上のパネル)は、キャプチャされた各EVが、各チャネルでさまざまな強度の1つ以上の色の共局在を表示できることを示すズームイン画像です。捕捉され?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
現在のEV特性評価法は、精製されたEVに大きく依存しており、これはEV精製法9,10,11,12,13の現在の実験的制限によって制限されています。単粒子干渉反射率イメージング(SP-IRIS)は、サンプル分析に必要な精製ステップを省くことができる効果的な技...
Access restricted. Please log in or start a trial to view this content.
開示事項
Clayton Deighan氏とGeorge Daaboul氏は、NanoView Biosciences Inc.の従業員であり、株主です。
謝辞
この研究は、カンザス大学医学部の研究機器およびリソース調達賞プログラムによって部分的に後援されました。PCG、LKC、FD、およびARは、NIA R21 AG066488-01からの資金で支援されました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 10-cm sterile Petri dish | Fisher | FB0875712 | |
| 15mL sterile tube | n/a | various | |
| 24-well cell culture plate, flat bottom | Fisher | 08-772-1 | |
| Blocking Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chipfiles | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chips | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chuck | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 250 ml | Fisher | 09-761-4 | |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 500 ml | Fisher | 09-761-10 | |
| Deionized (DI) water | Fisher | LC267404 | |
| EMS style tweezers with Carbon Fiber tips | Fisher | 50-193-0842 | |
| ExoView Human Tetraspanin Kit | NanoView Biosciences | EV-TETRA-C | Capture for hCD81, hCD9, hCD63, IgG Control + stains for hEV-A (hEV-CD63-647, hEV-CD81-555, hEV-CD9-488) 16 Chips per kit |
| ExoView R100 Imager | NanoView Biosciences | EV-R100 | Interferometric microscope including high specification camera including 3 color fluorescence and label free sizing and counting extracellular vesicles |
| Fluorescently labled huma CD9 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD63 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD81 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Incubation Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Orbital shaker or microplate shaker with digital settings capable of shaking at 500 rpm | n/a | various | |
| Plate Seal | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution A | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution B | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution C | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution D | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Square/flat tip tweezer | Fisher | 50-239-62 | |
| Straight strong point Boley style tweezers | Fisher | 16-100-124 | |
| Thermo Scientific Adhesive PCR Plate Seals | Fisher | AB-0558 |
参考文献
- Maas, S. L. N., Breakefield, X. O., Weaver, A. M. Extracellular vesicles: Unique intercellular delivery vehicles. Trends in Cell Biology. 27 (3), 172-188 (2017).
- Shah, R., Patel, T., Freedman, J. E. Circulating extracellular vesicles in human disease. The New England Journal of Medicine. 379 (10), 958-966 (2018).
- Deng, F., Miller, J. A review on protein markers of exosome from different bio-resources and the antibodies used for characterization. Journal of Histotechnology. 42 (4), 226-239 (2019).
- Cohen, O., et al. ’Golden’ exosomes as delivery vehicles to target tumors and overcome intratumoral barriers: in vivo tracking in a model for head and neck cancer. Biomaterials Science. 9 (6), 2103-2114 (2021).
- Han, Y., et al. Overview and update on methods for cargo loading into extracellular vesicles. Processes (Basel). 9 (2), 356(2021).
- Lee, M., Im, W., Kim, M. Exosomes as a potential messenger unit during heterochronic parabiosis for amelioration of Huntington's disease. Neurobiology of Disease. 155, 105374(2021).
- Sun, B., et al. Characterization and biomarker analyses of post-COVID-19 complications and neurological manifestations. Cells. 10 (2), 386(2021).
- Jiang, L., Gu, Y., Du, Y., Liu, J. Exosomes: Diagnostic biomarkers and therapeutic delivery vehicles for cancer. Molecular Pharmaceutics. 16 (8), 3333-3349 (2019).
- Bachurski, D., et al. Extracellular vesicle measurements with nanoparticle tracking analysis - An accuracy and repeatability comparison between NanoSight NS300 and ZetaView. Journal of Extracellular Vesicles. 8 (1), 1596016(2019).
- Carmicheal, J., et al. Label-free characterization of exosome via surface enhanced Raman spectroscopy for the early detection of pancreatic cancer. Nanomedicine. 16, 88-96 (2019).
- Greening, D. W., Xu, R., Ji, H., Tauro, B. J., Simpson, R. J. A protocol for exosome isolation and characterization: evaluation of ultracentrifugation, density-gradient separation, and immunoaffinity capture methods. Methods in Molecular Biology. 1295, 179-209 (2015).
- Wu, Y., Deng, W., Klinke, D. J. Exosomes: improved methods to characterize their morphology, RNA content, and surface protein biomarkers. The Analyst. 140 (19), 6631-6642 (2015).
- van de Vlekkert, D., Qiu, X., Annunziata, I., d'Azzo, A. Isolation and characterization of exosomes from skeletal muscle fibroblasts. Journal of Visualized Experiments: JoVE. (159), (2020).
- Ayala-Mar, S., Donoso-Quezada, J., Gallo-Villanueva, R. C., Perez-Gonzalez, V. H., Gonzalez-Valdez, J. Recent advances and challenges in the recovery and purification of cellular exosomes. Electrophoresis. 40 (23-24), 3036-3049 (2019).
- Daaboul, G. G., et al. Digital detection of exosomes by interferometric imaging. Scientific Reports. 6, 37246(2016).
- Pohler, K. G., et al. Circulating microRNA as candidates for early embryonic viability in cattle. NMolecular Reproduction and Development. 84 (8), 731-743 (2017).
- NanoView Biosciences. ExoView R100 User Guide. v240.4. NanoView Biosciences. , 202(2021).
- Daaboul, G. G., et al. Enhanced light microscopy visualization of virus particles from Zika virus to filamentous ebolaviruses. PLoS One. 12, 0179728(2017).
- ET Consortium. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14, 228-232 (2017).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved